このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
多細胞スフェロイドの作製およびマルチパラメーター生細胞蛍光寿命イメージング顕微鏡(FLIM)
* これらの著者は同等に貢献しました
要約
ここでは、フォローアップのマルチパラメーター生細胞顕微鏡検査を実施するためのさまざまな多細胞スフェロイド形成方法について説明します。蛍光寿命イメージング顕微鏡(FLIM)、細胞自家蛍光、染色色素、およびナノ粒子を使用して、生きた三次元(3D)がんおよび幹細胞由来スフェロイドにおける細胞代謝、低酸素症、および細胞死の解析のためのアプローチを実証します。
要約
多細胞腫瘍スフェロイドは、腫瘍の微小環境を再現し、薬物療法を試験および最適化し、3Dコンテキストでバイオセンサーとナノセンサーを使用するための一般的な3D組織微小凝集体モデルです。それらの生産の容易さ、予測可能なサイズ、成長、および観察された栄養素と代謝物の勾配は、3Dニッチのような細胞微小環境を再現するために重要です。しかし、スフェロイドの不均一性やその作製方法のばらつきは、細胞全体の代謝、生存率、薬物応答に影響を与える可能性があります。そのため、サイズ、ばらつき、バイオファブリケーションのニーズ、幹細胞生物学やがん細胞生物学における in vitro 3D組織モデルとしての使用など、最適な方法論を選択することは困難です。特に、スフェロイドの産生は、光学代謝イメージング、蛍光寿命イメージング顕微鏡(FLIM)、ナノセンサーによるスフェロイド低酸素症のモニタリング、生存率などの定量的生顕微鏡との適合性に影響を与える可能性があります。ここでは、従来のスフェロイド形成プロトコルをいくつか紹介し、生体広視野顕微鏡、共焦点顕微鏡、および2光子顕微鏡との適合性を強調しています。マルチプレックス自家蛍光FLIMを使用した、さまざまな種類のがんおよび幹細胞スフェロイドを使用した解析パイプラインへのフォローアップイメージングも紹介します。
概要
多細胞スフェロイドは、細胞の自己凝集によって取得され、球形を示す3D組織モデルのグループを表します。これらは、in vitroで細胞間および細胞とマトリックスの相互作用を模倣し、多数のがんおよび幹細胞由来のコンストラクト内で3Dコンテキストを再現するために広く使用されています。細胞の付着を減らし、凝集を促進するために、いくつかの技術が採用されています。これらには、表面張力1に依存する吊り下げ式が含まれます。超低付着プレート、マイクロモールド、マイクロウェルなどの細胞付着をはじく方法2,3;音波ベースのアプローチ4;流動誘起凝集法(スピナーフラスコ、バイオリアクター、マイクロ流体デバイス)5;磁性粒子支援形成6と凝集促進合成およびECMベースのマトリックスおよび足場の使用7,8,9。
癌の研究、開発、および新薬療法の検証において、スフェロイドは、栄養素、老廃物、およびO2の空間拡散制限勾配を再現する能力により魅力的なモデルであり、しばしば固形腫瘍に典型的な壊死性コアの形成につながる10,11。これらのより信頼性が高く洗練されたin vitroモデルは、動物研究の3Rの原則(置換、削減、改良)に従って、動物モデルの広範な使用の必要性に挑戦します(食品医薬品局[FDA]近代化法2.012)。がんに加えて、スフェロイドは幹細胞研究に応用されています。例えば、多能性幹細胞は胚様体(EB)を形成する能力を有しており、これは人工多能性幹細胞(iPSC)を、神経前駆細胞13や卵巣顆粒膜細胞13,14など、患者から直接入手することが困難な特殊な細胞型に分化させるために使用できる。さらに、EBの形成は、しばしば、より複雑なオルガノイドモデル、例えば、神経15、網膜16、心臓17、肝臓18、胃19、および腸オルガノイド20の開発における最初のステップである。実験に適したスフェロイド形成法を選択する際には、サイズ、再現性、スループット、ダウンストリームアプリケーションなどの要素を考慮する必要があります。
3D培養は複雑化するため、2D培養に比べてばらつきが大きくなる可能性があります。栄養組成物21、培地蒸発22、粘度23、pH制御24、スフェロイド形成方法、さらには培養25,26の時間などの要因により、さまざまな形態、サイズ、生存率、および異なる化学療法抵抗性27,28のスフェロイドを得ることができる.最近の研究では、スフェロイドの酸素勾配は常に静的ではなく、形成方法、スフェロイドサイズ、および細胞外粘度の影響を受け、スフェロイドの不均一性に影響を与えることが示された29。スフェロイドの再現性とデータへのアクセス性を向上させるために、MISpheroIDナレッジベースが開発され26、細胞株、培地、形成方法、およびスフェロイドサイズが再現性のある結果のための最小限の情報として特定されています。したがって、複数のハイスループット法(SphericalPlate 5D、ラボ製マイクロモールド、およびマイクロティッシュモールド)および低アタッチメント法(すなわち、BiofloatおよびLipidureコーティングされた96ウェルプレート、足場フリーおよびスキャフォールドベース)(図1および表1)について、ウェルサイズ(最大スフェロイドサイズの推定値)、使用した消耗品、調製時間、およびスフェロイドを顕微鏡ディッシュに輸送せずにスフェロイドをモニタリングする可能性を含む詳細な比較が行われました。 後者では長期的な研究が可能ですが、ハイスループット法で作製したスフェロイドは、多くの場合、エンドポイント実験につながります。5DspheriPlateのグリッドを除くすべての方法は、不要な自家蛍光をもたらさないため、顕微鏡で直接使用できます。
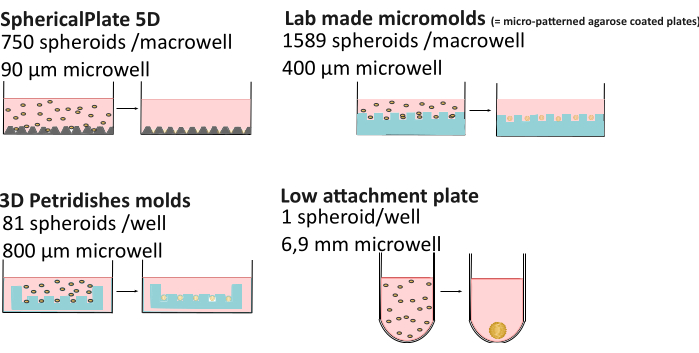
図1:スフェロイド形成方法の説明。 特許取得済みのマイクロウェルをプレートに統合したSphericalPlate 5Dなどのハイスループット法や、ラボで製造されたマイクロモールドやMicroTissueモールドは、スタンプを使用してアガロース(青)で複数のマイクロウェルを作製します。Lipidure(Amsbio)やBiofloat(Sarstedt)などの低接着性プレートは、細胞表面の接着を阻害し、細胞の自己凝集を促進する非接着性コーティングを使用しています。 この図の拡大版を表示するには、ここをクリックしてください。
| 5Dスフェリプレート | 自社製作のマイクロモールド | マイクロティッシュ | 低アタッチメント方式 | |
| スフェロイド数/ウェル | 750 | 1589 | 81 | 1 |
| 直径井戸 | 90μm | 400μm | 800μm | 1ミリメートル |
| 培養量 | 1 mLの | 5ミリリットル | 1 mLの | 200μL |
| その他の消耗品 | / | 3%アガロース7mL | 500 μL の 2% アガロース | / * |
| 準備時間 | 10分間 | 2時間+3日間のメディア適応 | 0.5時間+15分メディア適応 | 10〜30分+1時間の乾燥 |
| モニタリング | はい | いいえ** | はい | はい |
| オート蛍光 | はい | いいえ | いいえ | いいえ |
| 再 利用 | いいえ | はい | はい | いいえ** |
| 費用 | €€ | € | €€€€ | €€€€:コーティングとマトリゲル |
| €€:市販の96ウェルプレート | ||||
| *一部の細胞株では、コンパクトなスフェロイドを形成するためにECM(2%–5%マトリゲル)を添加する必要があります。 | ||||
| **コーティングは消耗するまで再利用できます。ただし、各プレートは少量のメディアを消費し、時間の経過とともにほこりが蓄積する可能性があります。フィルター滅菌は定期的に必要です。 | ||||
表1:複数のスフェロイド形成方法の比較29.「モニタリング」:顕微鏡ディッシュに移すことなくスフェロイドをモニタリングする能力。€:0-50€、€€:50-150€、€€:150-500€、€€€:>500€
蛍光顕微鏡法は、細胞死、生存率、増殖、代謝、粘度、さらには機械的特性など、スフェロイド内の主要な生物学的側面を直接モニタリングすることを可能にする30。蛍光寿命イメージング顕微鏡(FLIM)は、それらの(マイクロ)環境31,32,33,34内での蛍光プローブ相互作用を研究するための追加の定量的次元を提供し、異なる発光寿命35,36に従って重なり合う発光スペクトルを解像することを可能にする内因性細胞自家蛍光に基づく細胞代謝の調査。したがって、ニコチンアミドアデニンジヌクレオチドリン酸(NAD(P)H)、フラビンモノヌクレオチド(FMN)、フラビンアデニンジヌクレオチド(FAD)、プロトポルフィリンIXなどの広範な細胞内自己蛍光化合物は、1光子および2光子FLIMで測定でき、グルコース異化作用、酸化的リン酸化(OxPhos)の固有の「センサー」として機能し、細胞の酸化還元状態の一般的な概要を提供します。NAD(P)Hは、遊離細胞質、またはタンパク質結合ミトコンドリア型で存在する37,38。同様に、FADの酸化状態は蛍光性であり、遊離型の寿命が長くなります。NAD(P)HおよびFAD顕微鏡法は、通常、試料の光損傷39を防ぐことを目的として、2光子励起FLIMを使用する。多くの場合、「光学代謝イメージング」FLIMは、色素ベースのプローブ、遺伝的にコードされたバイオセンサー、リン光寿命イメージング顕微鏡(PLIM)、およびレシオメトリック強度ベースの測定と組み合わせて使用することで、スフェロイドまたはオルガノイドの代謝、酸素化、増殖、および細胞生存率のより完全な画像を提供することができます29,30,31.さらに、FLIMは、フェルスター共鳴エネルギー移動(FRET)法と組み合わせて、アクセプターと密接に接触したときのドナー蛍光色素の寿命変動を測定し、薬物とその標的ドメインとの結合を調査することもできます33,40,41。
取得したFLIM画像は、通常、ピクセルごとに寿命を計算するために分析されます。現在、蛍光寿命を得るためには、少なくとも3つの一般的な戦略が用いられています:半定量的な「高速FLIM」42(「タウセンス」と呼ばれることもあります)43,44、1指数関数的、2指数関数的、または3指数関数的フィッティングを使用した減衰曲線フィッティング、およびフェーザ変換とフェーザプロット解析による「フィッティングフリー」アプローチです。ベンダーに応じて、提供されているソフトウェア(LAS X、Symphotime、SPCImageなど)またはオープンソースソフトウェア(FLIMfit45、FLIMJ46、またはその他47など)のいずれかを使用して、測定されたFLIMデータを処理できます。通常、ベンダーが提供するソフトウェアは予備的なデータ分析に役立ちますが、オープンソースのソリューションは、フェーザプロットや3Dビジュアライゼーションなどを使用して、より正確な研究を提供できます。
スフェロイドの研究法としてのFLIMの有用性と魅力にもかかわらず、利用可能な実験プロトコルは非常に少なく、FLIMを含むライブマルチパラメトリック顕微鏡実験を成功させるための最も適切な形成方法を選択するための知識が一般的に不足しています。ここでは、一般的に使用されるスフェロイド形成プロトコルの詳細な比較を、その形態、生存率、および酸素化に基づいて、最近検証され特性評価された遠赤および近赤外(NIR)酸素感知ナノセンサー(MMIR1)と示します。カチオン性ナノ粒子には、参照O2-非感受性aza-BODIPY(励起650 nm、発光675 nm)とNIR O2感受性メタロポルフィリンPtTPTBPF(励起620 nm、発光760 nm)の2つのレポーター色素を含浸させます。MMIR1は、細胞毒性を導入することなく、従来の蛍光顕微鏡(レシオメトリック分析を使用)またはリン光寿命顕微鏡(PLIM)で酸素勾配のリアルタイム分析を可能にし、安定したシグナル、長期モニタリング、およびマルチプレックス25,29を可能にします。色素やナノセンサー、スフェロイドのスループット、または細胞タイプで染色する必要性に応じて、最も適切な形成プロトコルを選択できます。スフェロイドの生存率と酸素化の研究は、がんおよび幹細胞由来のスフェロイドの研究に関連しているため、提示されたプロトコルには、これらのモデルを使用したNAD(P)H-FLIMおよびFAD-FLIMの例と予想される典型的な結果も含まれています。提示されたイメージングおよび分析パイプラインは、最も一般的な時間相関のある単一光子計数ベースのFLIM顕微鏡プラットフォームを対象としています。
Access restricted. Please log in or start a trial to view this content.
プロトコル
1. 多細胞スフェロイドの作製
- 細胞培養
注:細胞培養は、American Type Culture Collection(ATCC)、Lonza、Sigma-Aldrich、またはその他のベンダーから収集できます。ATCCは、優先増殖培地、継代培養手順、バイオセーフティレベル、成長率、STRプロファイルなど、必要なすべての取り扱い情報を提供します。ここでは、ヒト結腸癌細胞株HCT116の500細胞/スフェロイドを、10%FBSと1mMピルビン酸ナトリウムを添加したMcCoyの5A培地(VWR、392-0420)に使用しています。毎日モニタリングする長期実験では、10 mM HEPES、pH 7.2を培地に添加できます。- 細胞培養を70%〜90%のコンフルエント度に達するように増殖させます。
- 予熱(37°C)滅菌PBS(25 cm2 の場合は5 mL、フラスコ75 cm2 あたり10 mL)で細胞をすすぎます。
- 0.05%トリプシン-1 mM EDTA溶液(25 cm²の場合は0.5 mL、フラスコ75 cm2 フラスコあたり1 mL)を添加し、5%CO2、湿度95%の37°Cで5〜10分間インキュベートして細胞剥離に到達します。
注:透過光(明視野)顕微鏡下で細胞の剥離を制御します。解離酵素溶液への細胞の過剰曝露は、細胞の生存率に影響を与える可能性があります。 - 10%FBSを含む過剰な細胞培養培地(解離液1mLあたり少なくとも5mLの培地)を添加してトリプシンを中和します。
注:低FBSまたはFBSフリーの培養培地で培養した細胞の場合、トリプシン処理した細胞培養物に0.5mLの100%FBSを添加し、続いて遠心分離して細胞を元の培養培地に移すことでトリプシン中和を行うことができます。 - ピペッティングにより細胞凝集体を解離し、培地中の単一細胞懸濁液を取得します。
注:上部に1000 μLのピペットチップが付いた血清学的ピペットでピペッティングすると、大量の浮遊細胞培養での単一細胞懸濁液の生成が大幅に改善されます。 - 計数チャンバー(ノイバウアールール血球計算盤または代替品)を使用して、細胞懸濁液1 mLあたりの細胞数をカウントします。
- 細胞懸濁液を希釈して、ミリリットルあたり必要な細胞数を得ます。
- 濃縮されたO2 プローブ(ナノ粒子)溶液を最終濃度10 μg/mLで細胞懸濁液に加え、O2のレシオメトリック分析を行います。
注:細胞懸濁液を(プローブで)均一にするために、スフェロイド形成前に複数回再懸濁してください。O2 プローブが不要な場合は、このステップをスキップしてスフェロイドの形成を進めてください。iPS細胞の取り扱いには、修正したプロトコルを適用する必要があります。簡単に説明すると、iPSCはGeltrexコーティングされたプレート上のコロニーで増殖し、ベンダー提供のプロトコル48に記載されているようにReLeSRを使用して継代します。回転楕円体が形成される日には、コロニーは大きく、コンパクトで、明確な境界を持つ多層の中心を示す必要があります。予熱した滅菌PBSで細胞をすすぎます。1 mLの穏やかな細胞解離試薬(GCDR)を加え、37°Cで8〜10分間インキュベートします。1000 μLチップを使用して、ウェルから細胞を穏やかに剥離し、単一細胞懸濁液を取得します。シングルセル懸濁液を滅菌済みの50 mLコニカルチューブに移し、予熱したDMEM-F12培地4 mLを添加してGCDRを中和します。1 mLのDMEM-F12でウェルを洗浄し、残りの細胞懸濁液に移します。300 x g で5分間遠心分離します。さらなる実験のために、1 mLの適切な培地に再懸濁してください。この原稿に記載されている実験には、mTeSR + 10 μM Rock Inhibitorを使用しました。細胞懸濁液をカウントして希釈し、ミリリットルあたり必要な細胞数を得ます。
- スフェロイド形成法
- 3Dシャーレマイクロモールド
注:このハイスループット法は、直径800μm、深さ800μmの9×9マイクロモールドアレイで多数のスフェロイド(81スフェロイド)を同時に生成するために使用されます。- 3Dペトリ皿をdH2Oで鋳造するためのマイクロモールドをすすぎ、オートクレーブ可能な容器に入れます。
- 電気泳動グレードのアガロース粉末2 gを測定し、乾燥した200 mLオートクレーブ対応ガラス瓶に入れます。
注:ボトルとアガロースパウダーが液体や湿気がなく乾燥していることを確認してください。 - 3Dシャーレとボトルをアガロース粉末でドライサイクルで30分間鋳造するためのオートクレーブマイクロモールド。
- 100mLの超純水に0.9gのNaClを添加して0.9 w/v%生理食塩水を作り、オートクレーブ滅菌します。
注:NaClはメーカーによって推奨されています。アガロースの安定性を高めます。 - 滅菌したアガロース粉末に滅菌生理食塩水を加えて、アガロース溶液を調製します。圧力が溜まらないように、蓋を緩く取り付けます。ボトルを回してアガロースパウダーを混ぜます。
- アガロースパウダーを電子レンジで沸騰させて溶かします。電子レンジを頻繁に停止します(~10秒ごと)。ボトルを渦巻き、アガロースが溶解するまで繰り返します。
注意:アガロース溶液は高温で、取り扱いには注意が必要です。溶融手順の直後に振とうすると、溶液が容器から破裂する可能性があります。事故を避けるために、容量の50%以下に満たされた十分に大きな容器を使用し、適切な個人用保護具(手袋、オーブンミット、目の保護具、白衣)を使用してください。 - 溶解したアガロース溶液を60〜70°Cまで冷まします。 アセプティック技術と条件を使用して、溶融アガロースをマイクロモールドにピペットで取り込みます(12シリーズの場合は500 μL、24シリーズの場合は330 μL)。
注:アガロースの混合またはピペッティング中の気泡形成は避けてください。アガロースが固化する前に、ピペッティングまたは穏やかにスクラップすることにより、マイクロモールドの小さな特徴に閉じ込められた小さな気泡を取り除きます。 - アガロースを約2〜3分間固めます。その後、マイクロモールドを慎重に曲げて3Dシャーレを取り出し、12ウェル組織培養プレートに移します。
注:マイクロモールドが過度に曲がると、アガロースモールド内に亀裂が形成される可能性があります。 - 3Dシャーレを平衡化するには、2.5 mL/ウェル細胞培養培地を加えます。15分以上インキュベートします。培地を取り出し、新しい培地と交換します。もう一度繰り返して、3Dシャーレを培地と平衡化します。
注:プロトコルは、細胞播種までここで中断できます。長期保存(4°Cで最大2週間)の場合は、中程度ではなくPBS溶液を使用してください。 - 培地(またはPBS)を囲む3Dペトリ皿を完全に取り外し、組織培養プレートを傾けて3Dペトリ皿内の培地を正確に取り出します。
- 40,500個の細胞を含む細胞懸濁液190 μLを細胞播種チャンバーに慎重に播種します(ステップ1.1を参照)。
注:アガローススタンプのマイクロウェルの数によって、スタンプごとに生成されるスフェロイドの数が決まります。この場合、このアガロース型には81個のマイクロウェル(81 x 500細胞/スフェロイド)が含まれています。マクロウェルに添加した懸濁液中の細胞濃度の変動により、スフェロイドあたりの細胞数を変更できるため、スフェロイドのサイズを制御できます。 - 細胞が3D Petri dshの特徴に落ち着くまで~10分待ちます。次に、2Dペトリ皿の外側に3mLの培地を追加します。
- 組織培養プレートを細胞培養インキュベーターに置き、必要に応じて3Dペトリ皿を囲む培地を交換します。
- 低アタッチメントプレート
注:この方法は、ウェルごとに1つのスフェロイドを生成するために使用されます。コーティングされたプレート(リピジュアまたはバイオフロート)は市販されています(ステップ1.2.2.1-1.2.2.4をスキップ)。あるいは、コーティングを別途購入して、未処理のマルチウェルプレートのコーティングに使用することもできます。96ウェルプレートの端にあるウェルは、これらのウェルでの蒸発が速いため、滅菌水またはPBSで満たすことが推奨されます。これにより、スフェロイドのウェルの数が60に制限されます。必要なスフェロイドの数が少ない場合は、周囲の空の井戸に水またはPBSを入れます。液体の取り扱いにはほこりのないチップを使用して、小さな粒子がスフェロイドの形成を妨げるため、ウェルに持ち込まれないようにします。- ガラス瓶容器内の50mLのエタノールに0.25gのポリマー粉末を溶解することにより、0.5 w / v%コーティング溶液を調製します。コーティングをフィルター滅菌します。
注:フィルター滅菌とすべての次のステップは、層流下で滅菌条件で実施する必要があります。 - 200 μLのコーティング溶液を96 U底培養プレートの各ウェルに加えます。
- 1分間インキュベートし、余分なコーティングを取り除きます。
注:コーティング液は複数回使用できます。ガラス容器に入れて室温(RT)で保存してください。プラスチック容器は、プラスチックが部分的に溶解して溶液の一部になる可能性があるため、お勧めしません。粉塵が存在する場合は、0.22μmのポリエーテルスルホン(PES)またはナイロンシリンジフィルターでフィルター滅菌します。 - 96ウェルプレートを約1時間風乾させます。
注:細胞が細胞外マトリックスを必要とする場合は、ステップ1.2.3に進みます。コーティングされたプレートは、アルミニウムで包むとRTで最大1か月間保存できます。少量の細胞/ウェルを播種する場合は、プレートを300 gで5分間遠心分離すると、細胞を引き下げるのに役立ちます。
- ガラス瓶容器内の50mLのエタノールに0.25gのポリマー粉末を溶解することにより、0.5 w / v%コーティング溶液を調製します。コーティングをフィルター滅菌します。
- 細胞外マトリックス支援形成プロトコール
注:一部の細胞株は、それ自体で十分な細胞外マトリックス(ECM)を産生せず、コンパクトなスフェロイド49,50,51を形成するために、Matrigel、Cultrex、またはGeltrexなどのECMの添加を必要とする。乳癌MDA−MB-231、ヒト皮膚乳頭、前立腺癌細胞などの細胞型については、ECMの追加を必要とする以下の修飾52でステップ1.2.2を使用することが可能である。粉塵がスフェロイド形成に干渉しないように、液体の取り扱いには粉塵のないチップを使用することが望ましいです。ステップ1および4-7は、生物学的安全キャビネット(クラスII)の下で実行する必要があります。- 表面処理の手順1.2.2.1-1.2.2.4(低アタッチメントプレート)に進みます。
- 96ウェルプレートを4°Cで冷蔵庫で予冷します。
- 96ウェルプレートに適したアダプターを使用して遠心分離機を準備し、4°Cで予冷します。
- 予冷(4°C)した細胞培養培地に基底膜マトリックス(BMM)の5%溶液を調製します。
注:BMMはRTで迅速に架橋します。取り扱いの際は、ストックと溶液を氷上に保管してください。 - 細胞懸濁液を調製します(ステップ1.1を参照)。
- 各ウェルに50 μLのBMM溶液を加えます。
- BMM溶液(25,000細胞/ウェル)の上に、各ウェルに50 μLの細胞懸濁液を静かに加えます。
注:このボリュームをウェルにブラストしないでください、そうしないと、細胞がウェルの側面に広がり、底に集まらなくなります。ウェルあたりの細胞数が少なくなるのは、それに応じて細胞密度を調整することによって得られます。すべての細胞がすべての播種密度でスフェロイドを形成するわけではありません。最適化は、セルタイプと目的の次元で実行する必要があります。提供された量で、BMMの最終濃度は2.5%です。異なる濃度が必要な場合は、原液を低濃度/高濃度で調製する必要があります。 - 96ウェルプレートを300 x g 、4°Cで5分間遠心分離します。
注:この手順を行わないと、細胞がウェルの底に適切に凝集せず、複数の小さな凝集体が形成されます。この段階での架橋を避けるために、遠心分離機を冷却する必要があります。 - プレートを細胞培養インキュベーターに入れます。凝集体は、播種後4日目に成熟したと見なされます。
注:スフェロイド形成に関するその他のプロトコルについては、 補足ファイル1を参照してください。
- 3Dシャーレマイクロモールド
2. スフェロイドの生顕微鏡観察
- ライブイメージング解析のためのスフェロイドの調製
注:実験デザイン(例:長期モニタリングまたはエンドポイント分析、顕微鏡のセットアップ、または測定された蛍光のスペクトル特性)またはスフェロイド製造方法と顕微鏡法の非互換性(例:サンプルの厚さ、材料の自家蛍光、イメージング中のスフェロイドの浮遊)により、スフェロイドが製造されたプレート内のスフェロイドの直接モニタリング、 できないかもしれません。このプロトコールでは、イメージング用のスフェロイドの調製について説明しており、これはほとんどの倒立広視野顕微鏡および共焦点顕微鏡に適しています。- 調製および予温(37°C)イメージング培地:DMEMにHEPES-Na、pH 7.2(10 mM)、ピルビン酸ナトリウム(1 mM)、L-グルタミン(2 mM)、およびグルコース(5 mM)を添加し、フェノールレッドを含まない。
注:重曹単独またはHEPES-Naと組み合わせて使用 することができる、イメージング中にCO2 制御が提供されている場合24。一部の細胞培養タイプは、HEPESの存在に耐えられません。実験デザインに応じて、ピルビン酸、グルタミン、およびグルコース含有量を変更することができます。 - コーティングされた(スフェロイドの接着力が強い)またはコーティングされていない(スフェロイドの接着力が低い)カバーガラス表面(厚さ#1.5)を備えた滅菌顕微鏡ディッシュ(市販またはラボ製)を準備します。
注:コーティングの必要性と種類は、細胞の種類、スフェロイドの接着特性、および3Dから2Dへの細胞移動速度によって異なります。コーティングは3D組織の喪失を促進し、スフェロイドの微小勾配の形状を変化させ、その結果、細胞の挙動を変化させる可能性があるため、これを考慮することが重要です。いくつかの実験(例えば、薬物刺激に対する応答を解析したイメージング)では、表面への強いスフェロイド接着が必要であり、ゼラチン、BMM、コラーゲン、コラーゲン/ポリ−D−リジン、またはポリ−D−リジンによる被膜が好ましい。 - O2 プローブで染色したスフェロイドをマイクロパターン化したアガロースプレートまたは96ウェルプレートのマイクロウェルから静かに洗い流し、まだ浮いているスフェロイドを2 mLバイアルに移します。
注:ハイスループット法からすべてのスフェロイドを確実に収集するために、追加の容量の培養培地で型を1〜3回すすぎ、すべてのスフェロイド懸濁液を1つのバイアルに組み合わせます。低アタッチメントプレートのスフェロイドの場合、個々のウェルからスフェロイドを1つずつバイアルに収集するか、実験に少数のスフェロイドで十分な場合は顕微鏡ディッシュに直接収集します。大きなスフェロイドを移し替えるときは、ピペットチップの端をカットして、ピペッティング中に損傷を与えないようにしてください。 - バイアルを最大5分間垂直位置に置いて、スフェロイドをバイアルの底に落ち着かせ、目に見えるペレットを形成します。
- スフェロイドをそのままにしてチューブから培地を取り出し、十分な量の新鮮な培養物に静かに再懸濁します。
注:スフェロイドの取り扱いの便宜上、この段階でイメージング媒体へのスフェロイドの移し替えも小さなバッチで行うことができます。ステップ 2.1.7 と 2.1.8 を参照してください。異なる実験群のスフェロイドは、培地組成と培地のプレコンディショニング時間が代謝に影響を与える可能性があるため、同等に扱う必要があります。 - スフェロイドが浮いている間に、顕微鏡ディッシュのウェルごとに等量のスフェロイド懸濁液を移します。
- スフェロイドをCO2 インキュベーターで37°Cで1〜2時間インキュベートし、顕微鏡ディッシュ/ウェルの表面にスフェロイドが付着することを確認します。イメージングの場合は、手順2.1.9に進みます。追加のプローブによるスフェロイドの染色については、ステップ2.1.8に進みます。
注記:3Dスフェロイドから2D表面界面への細胞移動速度は、時間の関数です。3D組織の喪失を避けるためには、細胞の種類、表面コーティングの種類、実験のデザインに関してインキュベーション時間を最適化する必要があります。例えば、HCT116は、スフェロイドのサイズにもよりますが、コラーゲンIV/ポリD-リジンコーティング表面への適切なスフェロイドの付着には少なくとも2時間かかりますが、hDPSCの付着と2D界面への移行は非常に速く、1〜2時間で3D組織が失われます。過度の拡散による「2Dスフェロイド」のイメージングを避けるために、インキュベーション時間を短縮したコーティングされていないガラス表面が使用されます。 - 推奨濃度または経験的に最適化された濃度の蛍光プローブを、既知の量のスフェロイド懸濁液に添加します。イメージング前に、37 °C CO2 インキュベーターで1時間インキュベートします。
注:生死アッセイには、ヨウ化プロピジウムとCalcein Green-AMを標準最終濃度1μg/mLで使用します。iPSC胚様体に対するヨウ化プロピジウム染色の毒性影響を避けるため、最終的なヨウ化プロピジウム濃度は0.5 μg/mLでした。スフェロイドサイズが大きいためにプローブの拡散が効率的でない場合、プローブのロード時間が長くなる可能性があります。ロード時間は常に、表面へのスフェロイドの付着に必要な総インキュベーション時間の一部として考慮する必要があります。スフェロイドの付着に長い時間が必要な場合は、この期間の終わりに染色手順を調整する必要があります。インキュベーション時間が長くなると、3D組織が失われる可能性があることに注意してください。 - 細胞培養培地または蛍光プローブを含む培地を取り出し、必要な量のイメージング培地と交換してください。スフェロイドイメージングの途中で培地蛍光バックグラウンドが存在しないことを確認するために、培地交換(洗浄)ステップを最大5回繰り返します。
注:培地交換中のスフェロイドの除去を避けるために、顕微鏡ディッシュの端から200μLピペットで培地を慎重に吸引し、壁面または顕微鏡ディッシュ内で横向きに培地を添加することをお勧めします。 - ただちにイメージングプロトコルのステップ2.2.1に進みます。
注:調製からイメージング、実際のイメージング取得までの間隔が長すぎると、細胞代謝(培地組成の変更など)、生存率(エンドポイント分析に使用される一部の蛍光プローブには毒性作用があり、長いインキュベーション期間後に細胞死を刺激する可能性があります)に影響を与えるだけでなく、3D組織の喪失につながる可能性があります。スフェロイドの複数のグループまたは実験条件を比較する必要がある場合は、分析されたグループ間で治療、プレコンディショニング、およびイメージング手順のタイミングをできるだけ均等に保つために、それに応じて実験デザインを開発する必要があります。
- 調製および予温(37°C)イメージング培地:DMEMにHEPES-Na、pH 7.2(10 mM)、ピルビン酸ナトリウム(1 mM)、L-グルタミン(2 mM)、およびグルコース(5 mM)を添加し、フェノールレッドを含まない。
- 画像取得
注:このプロトコルは、Stellaris 8 Falcon(ライカ)共焦点顕微鏡およびLeica Application Suite X(LAS X)ソフトウェアバージョン4.7を使用したライブスフェロイドのマルチパラメトリックイメージングについて説明しています。ただし、このような分析を代替の顕微鏡プラットフォームで実行するために必要なのは、わずかな変更だけです。- イメージングの30〜60分前に温度気候制御ユニットをオンにします。必要な換気速度と温度(35-37°C)を設定します。
注:温度制御に加えて、イメージング中にガス濃度(CO2 またはO2など)を制御する必要がある場合は、イメージング前に必要な条件に到達するために、対応するデバイスも事前に起動する必要があります。 - 顕微鏡と接続されたデバイス(WLLレーザー、コンピューター、水浸対物レンズ用のウォーターポンプ、およびその他の操作電子ブロック)の電源を入れます。正確な顕微鏡セットアップに付属の顕微鏡制御ソフトウェア(LAS X マシンモード や 環境制御付きマシンモードなど)を起動し、ステージキャリブレーションを初期化します。
- ソフトウェアで必要な対物レンズを選択し、必要に応じて浸漬液を塗布します。
注:生顕微鏡では、HC Fluotar L 25x/0.95 W VIS IR(2.4 mm作動距離)、HC PL Apo 40x/1.25 GLYC(0.35 mm作動距離)、または空気対物レンズに少なくともNA = 0.4以上など、十分な(「長い」)作動距離を持つ水またはグリセロール浸対物レンズを使用することをお勧めします。倍率および作動距離の選択は、画像化されたサンプルおよび測定された蛍光シグナル(輝度、量子収率、染色効率、例えば、色素およびナノ粒子に関する議論53を参照)の性質およびサイズに依存する。大きな物体(スフェロイドまたはオルガノイド、>500 μmサイズ)、「バイオリアクター」、またはマイクロ流体チップは、長い作動距離の対物レンズと低い倍率を必要としますが、個々の細胞または細胞小器官の分析には高倍率が必要であり、多くの場合、「モザイク」イメージングによって達成されます。 - ステージ上にスフェロイドを装着した顕微鏡ディッシュを設置します。フォーカスを調整し、対象物/関心領域(ROI)を見つけます。
注:小さく、蛍光が弱い、コントラストが悪い、または珍しい物体を見つける必要があり、焦点を見つけるのが難しい場合は、顕微鏡ディッシュの壁に事前に焦点を合わせ、ウェルの角の1つから始めて、蛇行した物体の表面を「スクリーニング」することをお勧めします。 - 「 プロジェクトを開く 」ウィンドウを選択し、対応するアイコン「 新規プロジェクトを作成」をクリックします。研究プロジェクトファイルに標準名(例:「YY-MM-DD+説明」から始まる)を付けます。イメージング中、生成されたすべての画像は、作成されたプロジェクトの .lif ファイルに自動的に保存されます。
- 「Acquisition」ウィンドウを開きます。白色光レーザー(WLL)の励起波長と、測定された蛍光の既知のスペクトル特性(励起/吸光度および発光スペクトル)に基づいて、ハイブリッドまたは共鳴走査検出器(HyD S、HyD X、またはHyD Rタイプ)の必要な範囲を設定します。スキャンの種類として [ライン] または [フレーム] を選択します。
注:市販の蛍光色素のほとんどでは、スペクトル特性はLAS X Dye Assistantパッケージに含まれています(または追加されています)。プローブのスペクトル特性に適合し、FLIMの場合はフォトンカウンティング(HyD XまたはHyD R)と互換性のある適切なスペクトル感度範囲を持つ検出器を選択してください。マルチパラメトリックイメージングの場合、WLLを複数の励起位置に設定します(例:FAD/FlavinsとレシオメトリックMMIR1 O2 プローブの2つの蛍光チャネルの同時イメージングの場合-参照および高感度の場合、励起/発光設定は、1つまたは2つのシーケンシャルスキャンシーケンスでそれぞれ460 nm/510-590 nm HyD X1および614 nm/631-690 HyD X3および724-800 nm HyD Rになります)。検出器は異なるスペクトル感度を持つことができるので、放出を収集するために適切な検出器を割り当てることが重要である54。 - (FLIMの場合はオプション) 「Acquisition 」ウィンドウで、「 FLIM 」モードを選択して、フォトンカウンティング(減衰収集)と組み合わせたイメージングを実行します。すぐに、FLIMデータをナビゲートおよび分析するための追加の「LAS XソフトウェアのFLIMモジュール」が開きます。
- (FLIMの場合はオプション)WLLパルスの繰り返し率は、蛍光色素分子の予想平均寿命に基づいて選択します。
注:レーザーパルスの周波数は、完全な蛍光減衰を収集するために調整する必要があります。これは、顕微鏡にインストールされた パルスピッカー 機能を使用して行うことができます。蛍光減衰がレーザーパルスと重なると、推定蛍光寿命が短くなります。パルス間隔は、予想される平均蛍光寿命の4〜5倍長くすることをお勧めします(たとえば、最大5nsの寿命の場合は25 ns / 40 MHz)。多くのパルスレーザーは、80MHzの固定繰り返し周波数を持っています(最大2〜3nsの範囲にのみ理想的です)。これは、実験に適した蛍光色素を選択するために重要です。 - FAST LIVEモードを使用してプレビューイメージングを開始し、関心のあるセクションのイメージングオブジェクトのファインフォーカスを調整します。
注意: レーザーの安全規則に厳密に従ってください。常にレーザーの安全ルールを考慮し、イメージングが停止するまで待ってから、透過灯をオンにし、接眼レンズを覗き込むか、サンプルを確認してください。
注: FAST LIVE モードでは、600 Hzの高速スキャン(双方向Xスキャンモードが使用されている場合は最大フレームレート4.43 / sに相当)、256 x 256ピクセルの解像度が画像に自動的に適用され、蛍光を光退色から保護します。ピンホールを開く(例:3〜4 AU)および/または蛍光信号が弱すぎて物体に焦点を合わせられない場合は、レーザー強度を上げます。不完全な減衰収集は避けてください。 - (FLIMの場合はオプション) FLIMモジュール ウィンドウ(ライブ モード)でのイメージング中に表示されるピクセル強度ヒストグラムを見て、適切なレーザー強度/ピンホールサイズと解像度を調整して、カウントレート~1光子/レーザーパルス制限(赤線)を達成します。パイルアップ効果のリスクを排除するために、1を大幅に超えることは避けてください。必要に応じて、減衰ウィンドウに完全な減衰コレクションを持つようにWLLパルス繰り返しレートを調整します(不完全な減衰コレクションを回避するには、手順2.2.8を参照してください)。
注:フォトンの数(強度)が、フィッティング分析またはフェーザプロットクラウドの信頼性の高い減衰を再構築するのに十分でない場合は、いくつかのスキャンリピート(フレームまたはライン、またはスキャン時間の設定)を適用し、レーザー強度を上げるか、解像度(スキャンされたROIサイズ)を犠牲にします。レーザーの繰り返し率を下げると、信頼性の高い崩壊再構成のためにより多くの光子を収集する必要があり、イメージングパラメータの追加補正が必要になる場合があることに注意してください。強い光と長時間の連続照明が細胞の生存率と代謝に及ぼす潜在的な影響に注意してください55。生存率と代謝への悪影響は、励起光の強度、持続時間、波長、およびイメージングモダリティ(例えば、一光子共焦点イメージングと多光子イメージング)によって、個々のケースで異なる可能性があります。それに応じてイメージングパラメータを調整し、必要に応じて、パイロット実験56でCalcein Green-AMまたはヨウ化プロピジウム強度による細胞生存率/死を制御します。可能であれば、蛍光プローブ染色プロトコルのさらなる最適化を検討して、生顕微鏡観察中に適切な蛍光シグナルに到達する必要があります。 - (3D zスタックの場合はオプション)Fast Liveでは、Zスタックウィンドウ(XYZスキャンモード)で座標、スキャン方向を設定し、それらを開始と終了に属性付けします。Z ステップのサイズまたはステップ数を選択します。
注:ソフトウェアは、使用された解像度とスキャンパラメータに基づいて「最適な」ステップ数を自動的に計算しますが、ライブ3D再構成は通常、高速取得を達成するために必要なステップ数が少なくて済みます(例:1〜2μmのステップサイズ、50〜100μmのスタックサイズ、双方向スキャン、合計スキャン時間の2〜3分が必要です)。細胞内小器官、細胞、および3D細胞モデルも測定中に動く可能性があることに注意してください。さらに、光の透過深度と散乱限界により、共焦点FLIMでは通常、50〜100μmのスキャン深度しか達成できません。 - 必要な設定がすべて適用されたら、イメージングを開始します。
- 画像に適切な名前を付けます。
- 透過光モードで次の撮像対象物を検索し、以前に最適化した撮像設定で撮像手順を繰り返します(手順2.2.8-2.2.12)。
注:強度ベースの比較または強度比分析(MMIR O2プローブベースの酸素化分析など)では、分析されたすべてのオブジェクト(倍率と対物レンズの種類、レーザー強度、出力とパルス周波数、励起波長、検出器の範囲、ピンホール、スキャン速度、ピクセル滞留時間、解像度)で常に同じイメージング設定を維持してください。ただし、蛍光寿命は蛍光強度に依存せず、信頼性の高い計算のために適切な数の光子を収集する必要があるため、FLIMイメージングパラメータは実験の過程で再調整して、収集された光子数を異なる処理または実験条件間で一致させることができます。したがって、強度ベースの分析と蛍光寿命ベースの分析の両方が必要なマルチパラメトリック分析では、比較した実験グループのすべてのオブジェクトに最適化されたユニバーサルイメージング設定を適用する必要があります。「FLIMのみ」の比較では、LAS Xソフトウェアが個々の画像測定42に対するIRFの計算を提供するので、わずかに異なるイメージング設定で取得された画像を比較することが可能である。ただし、LAS X(FLIMfit45など)以外のFLIMフィッティング解析では、イメージングソフトウェアからエクスポートできないため、機器応答関数(IRF)は異なるイメージング条件ごとに測定する必要があります。したがって、実験デザインとワークロードを簡素化するために、データセット内のすべての画像に同じイメージング設定を適用することをお勧めします。次いで、対応するIRF測定は、測定されたスペクトルチャネル57,58,59の発光特性を有する消光または高速蛍光寿命蛍光蛍光色素(ps範囲内)を使用して、金ナノ粒子発光60または多光子FLIM61の第2高調波発生信号によって行うことができる.LAS Xソフトウェアでは、関心のあるファイルを右クリックして[画像設定の適用]を選択することで、以前に最適化されたイメージングパラメータを新しいプロジェクトにロードできます。 - イメージングセッションが終了したら、イメージングプロジェクトを保存します。イメージングセッションを終了するには、イメージング施設に実装されている標準的な手順に従って、顕微鏡ステージからサンプルを取り出し、浸漬液(使用する場合)から対物レンズを洗浄します。プロジェクトとソフトウェアを閉じます。顕微鏡、レーザー、および接続されているすべてのデバイスの電源を切ります。
- イメージングデータの解析(手順2.3)を進めます。
- イメージングの30〜60分前に温度気候制御ユニットをオンにします。必要な換気速度と温度(35-37°C)を設定します。
- LAS X FLIMモジュールとFIJIによるNAD(P)H/FAD-FLIMフェーザ画像処理
注:このプロトコルは、NAD(P)HおよびFAD/Flavins自家蛍光FLIMの例に関する周波数領域データのための画像化されたスフェロイドの蛍光寿命分析について説明しています。NAD(P)H自家蛍光測定は代謝分析のゴールドスタンダードとなり、NAD(P)H自家蛍光寿命の短い成分と長いNAD(P)H自家蛍光寿命成分がそれぞれ解糖または酸化的リン酸化(OxPhos)に関連しています。これは、遊離NAD(P)Hまたはタンパク質結合NAD(P)Hの測定標準に向けた代謝軌道に沿ったフェーザープロットのシフトによって分析できます31,62.代謝シフトの軌跡を解析するため、および実験グループ間のプロット上のフェーザ雲の位置(ステップ2.3.6以下の注を参照)を比較するために、フェーザ雲の幾何学的中心(重心)の計算に基づく簡略化されたフェーザ座標分析が実装されました29.記載されているプロトコルは、フィジーにおける重心座標の計算と、座標(例えば、回転楕円体NAD(P)H自家蛍光フェーザ雲の重心から「自由NAD(P)H」理論点までの距離)を使用してフェーザプロット上の2点間の距離を測定する方法を示しています。同様に、FADやその他の自家蛍光シグナルも解析できます。データセット 1 と .lif (LAS Xソフトウェアが必要)または .ptu この手順を学習するためのファイル形式が提供されています(増補ファイル2、増補ファイル3、増補ファイル4、増補ファイル5、増補ファイル6、増補ファイル7、増補ファイル8、増補ファイル9、 そして 補遺ファイル10).- LAS X で FLIM モジュールを開き、[Open Project] を選択して、自家蛍光 NAD(P)H/FAD 解析用のスフェロイド画像ファイル (.lif) を読み込みます。
注:FLIMモジュールにはバグや中間的なデータ損失が発生する可能性があるため、NAD(P)H/FAD解析には元の回転楕円体画像ファイル(.lif)のコピーを使用し、RAWファイルは変更しないでください。 - 1 つの画像を選択し、FLIM 解析インターフェイスに移動します。 [フェーザ ]をクリックしてフェーザプロットにアクセスし、フェーザ解析モードをアクティブにします。フィルター(中央値 または ウェーブレット)を適用し、 しきい値 を設定して、ノイズを最小限に抑え、すべてのフェーザ解析のデータの可視性を向上させます。倍音を選択します。NAD(P)Hデータに基づく代謝シフトの解析は、手順2.3.3-2.3.5に進みます。フェーザプロットの一般的な比較については、ステップ2.3.6から進んでください。
注:分析設定(フィルタータイプ、高調波、しきい値、ビニング、およびフェーザROI)を、比較したデータセット内のすべての画像に等しく適用します。 - (NAD(P)H解析ではオプション)データセット関連の画像を選択し、[ Draw Ratio Cursor for Two Components ] オプションを使用して、標準フェーザ プロット空間のユニバーサル円上の 0.45 ns の位置を正確に特定します。この位置は、遊離NAD(P)Hの純粋な均質溶液の平均蛍光寿命に割り当てられ、これは通常、単指数関数的崩壊62に近い。
注:遊離NADHおよび遊離NAD(P)Hは、水溶液中での類似のスペクトル特性および蛍光寿命の類似した値を有し、2つの短い寿命成分、0.3 nsおよび0.7 ns63を有する。したがって、フェーザベースの分析を単純化するために、また寿命成分間のわずかな違いにより、それらの蛍光減衰は単指数関数的であると受け入れられ、これによりフェーザクラウドを普遍的な円に割り当てることができます。free-NAD(P)H形状の参照平均寿命も測定し、フェーザ空間にプロットして同様の解析を行うことができます。参照寿命は、文献62に基づいて選択されました。他のソースでは、溶液中の自由NAD(P)Hの値がわずかに異なる(0.4 ns64)ことが見つかることに注意してください。 - (NAD(P)H解析ではオプション)free-NAD(P)H のライフタイムが割り当てられたフェーザ プロット (手順 2.3.3 を参照) をエクスポートします。これには、プロットを右クリックして [データのエクスポート] を選択します。フェーザプロットを .tiff 形式のファイルとして指定の保存フォルダにエクスポートします。
注:LAS X FLIMモジュールからエクスポートされたフェーザプロット画像の元のピクセルサイズは常に1024 x 600ピクセルです。データのエクスポートと事前分析に別のソフトウェアを使用する場合は、すべてのフェーザプロット画像が同じサイズと解像度でエクスポートされていることを確認してください。 - 回転楕円体関連のフェーザ雲をエクスポートするには、LAS X FLIMモジュールの Draw Cursor ツールを使用して、画像上の回転楕円体ROIを選択します。生成されたフェーザ プロットを手順 2.3.4 で説明したとおりにエクスポートします。
注:フェーザ空間の対応するgおよびs(xおよびyと同様)座標は、選択されたROIのすべてのピクセルに、それらの寿命に従って割り当てられ、周波数領域データセット64,65に変換される。tf (位相寿命) と tm (変調寿命) の値が類似するピクセルのクラスターは、プロット上に雲パターン (フェーザ クラウド) を形成し、色分け (濃い青から赤の範囲) は豊富な寿命値を反映します。普遍的な円または内側の雲の位置によって、単指数関数的または多指数関数的な崩壊を区別できます。一部の測定媒体は強い(自己)蛍光を示し、フェーザプロット上に対応する雲が現れますが、これは強度の閾値では簡単に除去できません。このパターンは重心座標の計算に影響を与えるため、エクスポートされたフェーザ プロットから除外する必要があります。スフェロイドROIを操作すると、無関係なピクセルをさらなるフェーザ解析から除外できます。 - データセット内のすべてのスフェロイドROIに対して、フェーザプロットのエクスポート手順を繰り返します(手順2.3.4および2.3.6を参照)。さらに、エクスポートされた.tiff画像のセットをチェックして、さらに比較分析するためのデータの完全なセットを保証し、エクスポートされたすべての画像のピクセルサイズが同じであることを確認します(手順2.3.4の注を参照)。
注:プロトコルのこの段階では、画像セットには、free-NAD(P)H位置(文献または経験的に得られたデータに基づく)とすべてのスフェロイドROI(または特定の分析に必要な場合は代替ROIパターン)のフェーザプロットを持つフェーザを含める必要があります。このステップから、FIJIでさらに分析が行われ、その後スプレッドシートで分析が行われます。 分析 ツールウィンドウを使用して、FIJIの スケールオプションを設定し 、すべてのフェーザプロット画像が同じ単位タイプ(ピクセル単位のみ)でキャリブレーションされていることを確認します。そうでない場合は、選択した スケール の設定ウィンドウで単位の長さを設定します(たとえば、ピクセルベースのスケールの場合は、 ピクセル単位 の距離フィールドに1を入力し、長さの単位を ピクセルに設定します)。さらに比較するには、エクスポートされたすべてのデータを同じ単位タイプを使用して測定します。 - (NAD(P)H解析ではオプション)対応するエクスポートされたフェーザプロット画像上の自由NAD(P)H平均蛍光寿命のピクセル点位置を決定します(ステップ2.3.3を参照)FIJIでフェーザ画像を開き、画像を拡大して、ユニバーサル半円と 2つのコンポーネントの比率カーソル ラインとの間の交点をピクセル解像度で明確に視覚化します。 長方形 ROIツールを使用して、交差を選択します。
注:次のステップ(ステップ2.3.9)でその座標を正確に決定するために、長方形の選択が交点の周りの小さな領域であることを確認してください。 - (NAD(P)H解析ではオプション)[解析] ツールを開き、[測定値の設定] ウィンドウを選択し、測定パラメーターとして [重心] を選択します。解析ツールウィンドウで測定をクリックして、自由NAD(P)H参照点の重心座標を決定します。これらの座標をスプレッドシートにエクスポートします。
注:Free-NAD(P)H座標は、この点からデータセット内の回転楕円体フェーザー雲の位置までの距離を比較するための基準点として使用されます(NAD(P)H FLIM自家蛍光分析で解糖とOxPosの間の代謝シフトを特徴付ける方法) - FIJIを使用して、回転楕円体フェーザ雲の画像を開きます。 イメージ ウィンドウツールを開き、「 調整」を選択して、ツールバーの 「カラーしきい値 」を選択します。選択した しきい値処理方法 (Otsu など) を選択し、 色相値 と 明るさ値 を設定して、ピクセル座標が最も豊富なフェーザ クラウド部分を選択するためのパラメーターを絞り込みます。 「選択 」をクリックして、クラスター領域を定義します。
注:分析する必要があるすべてのフェーザプロット画像に同じしきい値パラメータを保持します。提示されたNAD(P)H-FLIMデータでは、色相値9と明るさ値160を設定した閾値法Otsuを選択し、すべてのフェーザ画像に適用しました。または、選択した領域をROIマネージャーにコピーし([編集]>[選択]>[マネージャーに追加]のパスをたどります)、フェーザROIのライブラリを作成してさらに分析することもできます。 - 選択を保持したまま、ステップ 2.3.9 で説明した手順に従って、選択した領域の重心座標を計算します。これらの座標をスプレッドシート ファイルにエクスポートします。
- 手順 2.3.9 と 2.3.10 を繰り返して、すべての ROI フェーザ イメージの重心座標を決定し、スプレッドシートにデータ セットを作成します。
注: ROI Manager ROIライブラリを使用すると、ROI分析を簡素化および整理するのに役立ちます(ステップ2.3.10を参照) - (NAD(P)H解析ではオプション)異なる比較グループからエクスポートされた参照 free-NAD(P)H 座標とスフェロイド ROI 座標を使用してスプレッドシートを開きます。決定された座標と次の式を使用して、個々の回転楕円体フェーザ重心から参照自由 NAD(P)H 位置までの距離を計算します。
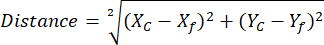
ここで、Xc と Yc は重心座標、Xf と Yf は参照座標です。
注:参照寿命へのシフトを決定するための重心パラメータの適用は、データセットのすべての重心が参照点に向かって同じ線形軌道上にある場合にのみ適切です。これを確認するには、データセットのすべての重心点を同じ座標空間の参照点と一緒にプロットし、線形トレンドの位置合わせを実行する必要があります。すべての点を通る線形トレンド ラインの R2 係数が 1 に近い場合 (たとえば、R2 が 0.8 から 0.99 の場合)、距離分析は適切であると想定されます。 - 比較のためにすべてのデータを適切に整理し、対応するソフトウェア(Origin、MatLabなど)を使用して統計分析を実行します。データセットの特性(分布の正規性、統計単位の数など)に応じて、適切な統計的検定を選択してください。
注:NAD(P)H解析では、距離値を比較して、実験条件に応じて代謝シフトを特徴付けます。実験グループ間でフェーザプロットを比較するには、ROIフェーザ雲重心座標の比較を実行します。
- LAS X で FLIM モジュールを開き、[Open Project] を選択して、自家蛍光 NAD(P)H/FAD 解析用のスフェロイド画像ファイル (.lif) を読み込みます。
Access restricted. Please log in or start a trial to view this content.
結果
適切なスフェロイド形成法の選択
選択したスフェロイド形成法は、スフェロイドのサイズ、形状、細胞密度、生存率、および薬剤感受性に大きく影響します(図2)。以前は、複数のハイスループット(SphericalPlate 5D、ラボ製マイクロモールド、およびMicroTissueモールド)と「中スループット」の低アタッチメント(BiofloatおよびLipidureコーティングされた96ウェル...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
多細胞スフェロイドは、腫瘍および幹細胞ニッチ微小環境の研究、創薬、およびバイオファブリケーションのための「組織ビルディングブロック」の開発において選択される方法になりつつあります。スフェロイドの不均一な内部構造、栄養素の勾配、および酸素化は、比較的単純化されたアクセス可能な環境で、in vivo組織や腫瘍のそれらを模倣することができます。方法論の透明性...
Access restricted. Please log in or start a trial to view this content.
開示事項
開示するものはありません。
謝辞
この研究は、ゲント大学の特別研究基金(BOF)助成金(BOF/STA/202009/003;BOF/IOP/2022/058)、フランダース研究財団(FWO、I001922N)、欧州連合、fliMAGIN3D-DN Horizon Europe-MSCA-DN No. 101073507.
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| 0.05% Trypsin-EDTA | Gibco | 25300-054 | Also available from Sigma |
| 10 mL serological pipets | VWR | 612-3700 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 12 well cell-culture plates, sterile | Greiner bio-one | 665-180 | Similar products are also available from Sarstedt, Corning and other companies. |
| 12 Well Chamber slide, removable | Ibidi | 81201 | Also available from Grace Bio-Labs, ThermoFisher Scientific and others |
| 15 mL centrifuge tubes | Nerbe plus | 02-502-3001 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 3D Petri Dish micromolds | Microtissue | Z764000-6EA | |
| 6 well cell-culture plates, sterile | Greiner bio-one | 657160 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| 70% ethanol | ChemLab | CL02.0537.5000 | |
| Biofloat | Sarstedt | 83.3925.400 | Commercial available coated 96-well plate for spheroid formation |
| Calcein Green-AM | Tebubio | AS-89201 | Apply in dilution 1:1000 |
| CellSens Dimension software | Olympus | version 3 | |
| Collagen from human placenta, type IV | Sigma | C5533 | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Confocal FLIM Microscope | Leica Microsystems | N/A | Stellaris 8 Falcon inverted microscope with white-light laser, HyD X detectors, climate / T control chamber (OkoLab), 25x/0.95 W objective |
| D(+)-Glucose | Merck | 8342 | Prepare 1 M stock solution, 1:100 for preparation of imaging medium (final concentration 10 mM) |
| Dulbecco's modified Eagle's medium (DMEM), phenol red-, glucose-, pyruvate- and glutamine-free | Sigma-Aldrich | D5030-10X1L | For preparation of imaging medium |
| Fetal Bovine Serum (FBS) | Gibco | 10270-098 | Also available from Sigma. Needs to be heat-inactivated before use. |
| HEPES (1M) | Gibco | 15630-080 | Dilution 1/100 for preparation of imaging medium (final concentration 10 mM) |
| Human colon cancer cells HCT116 | ATCC | ||
| ImageJ | NIH | version 1.54f | |
| Leica Application Suite X (LAS X) | Leica Microsystems | version 4.6.1.27508 | |
| L-glutamine | Gibco | 25030 | Also available from Sigma. Apply in dilution 1:100. |
| Lipidure-CM5206 | Amsbio | AMS.52000034GB1G | |
| McCoy's 5A, need addition of 1 mM Sodium Pyruvate and 10 mM HEPES | VWR | 392-0420 | Standard growth medium for HCT116 cells |
| micro-patterned 3D-printed PDMS stamps | N/A | N/A | Provided by the Centre for Microsystems Technology, Professor Dr. Jan Vanfleteren, Ghent University |
| NaCl | Chemlab | CL00.1429.100 | |
| Neubauer couting chamber | Fisher Scientific | 15980396 | |
| O2 probes: MMIR1 | N/A | N/A | Full characterization, validation and some applications can be found at: https://www.biorxiv.org/content/10.1101/2023.12.11.571110 v1 |
| PBS | Fisher scientific | Gibco18912014 | Dissolve PBS tablet in 500 mL of distilled water. |
| Pen Strep :Penicillin (10,000 U/mL) / streptomycin (10,000 μg/mL) 100x solution | Gibco | 15140-122 | Also available from Sigma. Apply in dilution 1:100. |
| Poly-D-lysine | Sigma | P6407-5mg | For the preparation of 0.07 mg/mL Collagen and 0.03 mg/mL Poly-D-lysine coated microscopy dishes |
| Propidium Iodide | Sigma-Aldrich | 25535-16-4 | Cell death staining, use 1 µg/mL at 1h incubation |
| PVDF syringe filter 0.22 µm | Novolab | A35149 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Sodium pyruvate (100 mM) | Gibco | 11360-070 | Dilution 1/100 for preparation of imaging medium (final concentration 1mM) |
| SphericalPlate 5D 24-well | Kugelmeiers | SP5D-24W | |
| sterile petridish | Greiner bio-one | 633181 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (25 cm² ) | VWR | 734-2311 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| Tissue culture flask (75 cm²) | VWR | 734-2313 | Similar products are also available from Sarstedt, Corning, VWR and other companies |
| U-bottom 96-well plate | VWR | 10062-900 | Similar products are also available from Sarstedt, Corning, Greiner Bio-one and other companies |
| Ultrapure Agarose | Invitrogen (Life Technologies) | 16500-500 | Other types of Agarose such as Agarose low melting point (A-9414, Sigma), Agarose for routine use (A-9539, Sigma) |
| Widefield fluorescence inverted microscope | Olympus | N/A | Inverted fluorescence microscope IX81, with motorised Z-axis control, CoolLED pE4000 (16 channels, 365-770 nm), ORCA-Flash4.0LT (Hamamatsu) cMOS camera, glass warming plate Okolab, CellSens Dimension v.3 software and air objectives 4x/0.13 UPlanFLN and 40x/0.6 LUCPlanFLN. (Optional, for high-resolution imaging) 60x/1.0 LUMPLFLN water |
参考文献
- Foty, R. A simple hanging drop cell culture protocol for generation of 3d spheroids. J Vis Exp. (51), e2720(2011).
- Moskovits, N., et al. Establishing 3-dimensional spheroids from patient-derived tumor samples and evaluating their sensitivity to drugs. J Vis Exp. (190), e64564(2022).
- Griner, L. M., et al. Generation of high-throughput three-dimensional tumor spheroids for drug screening. J Vis Exp. (139), e57476(2018).
- Qian, Y., Wei, X., Chen, K., Xu, M. Three-dimensional acoustic assembly device for mass manufacturing of cell spheroids. J Vis Exp. (200), e66078(2023).
- He, H., et al. Dynamic formation of cellular aggregates of chondrocytes and mesenchymal stem cells in spinner flask. Cell Prolif. 52 (4), e12587(2019).
- Perez, J. E., Nagle, I., Wilhelm, C. Magnetic molding of tumor spheroids: Emerging model for cancer screening. Biofabrication. 13 (1), 015018(2020).
- Kingsley, D. M., et al. Laser-based 3d bioprinting for spatial and size control of tumor spheroids and embryoid bodies. Acta Biomater. 95, 357-370 (2019).
- Guillaume, O., et al. Hybrid spheroid microscaffolds as modular tissue units to build macro-tissue assemblies for tissue engineering. Acta Biomater. 165, 72-85 (2023).
- Danilevicius, P., et al. laser-made 3D microscaffolds for tissue spheroid encagement. Biointerphases. 10 (2), 021011(2015).
- Jamieson, L. E., Harrison, D. J., Campbell, C. Chemical analysis of multicellular tumour spheroids. Analyst. 140 (12), 3910-3920 (2015).
- Dmitriev, R., Borisov, S., Jenkins, J., Papkovsky, D. Multiparametric imaging of tumor spheroids with ultra-bright and tunable nanoparticle O2 probes. SPIE BiOS. 9328, (2015).
- Zushin, P. -J. H., Mukherjee, S., Wu, J. C. FDA Modernization Act 2.0: transitioning beyond animal models with human cells, organoids, and AI/ML-based approaches. J Clin Invest. 133 (21), e175824(2023).
- Kim, D. -S., et al. Robust enhancement of neural differentiation from human es and ips cells regardless of their innate difference in differentiation propensity. Stem Cell Rev Rep. 6 (2), 270-281 (2010).
- Hart, D., Gutiérrez, D. R., Biason-Lauber, A. Generation of a human ovarian granulosa cell model from induced pluripotent stem cells. bioRxiv. , 2022(2022).
- Chiaradia, I., et al. Tissue morphology influences the temporal program of human brain organoid development. Cell Stem Cell. 30 (10), 1351-1367 (2023).
- Wagstaff, E. L., Ten Asbroek, A. L., Ten Brink, J. B., Jansonius, N. M., Bergen, A. A. An alternative approach to produce versatile retinal organoids with accelerated ganglion cell development. Sci Rep. 11 (1), 1101(2021).
- Thavandiran, N., et al. Design and formulation of functional pluripotent stem cell-derived cardiac microtissues. Proc Natl Acad Sci U S A. 110 (49), E4698-E4707 (2013).
- Harrison, S. P., et al. Scalable production of tissue-like vascularized liver organoids from human pscs. Exp Mol Med. 55 (9), 2005-2024 (2023).
- Noguchi, T. -A. K., Kurisaki, A. Formation of stomach tissue by organoid culture using mouse embryonic stem cells. Methods Mol Biol. 2017, 217-228 (2017).
- Takahashi, J., et al. Suspension culture in a rotating bioreactor for efficient generation of human intestinal organoids. Cell Reports Methods. 2 (11), 100337(2022).
- Lagziel, S., Gottlieb, E., Shlomi, T. Mind your media. Nat Metab. 2 (12), 1369-1372 (2020).
- Das, V., Fürst, T., Gurská, S., Džubák, P., Hajdúch, M. Reproducibility of uniform spheroid formation in 384-well plates: The effect of medium evaporation. J Biomol Screen. 21 (9), 923-930 (2016).
- Bera, K., et al. Extracellular fluid viscosity enhances cell migration and cancer dissemination. Nature. 611 (7935), 365-373 (2022).
- Michl, J., Park, K. C., Swietach, P. Evidence-based guidelines for controlling ph in mammalian live-cell culture systems. Commun Biol. 2, 144(2019).
- Okkelman, I. A., Vercruysse, C., Kondrashina, A. V., Borisov, S. M., Dmitriev, R. I. Affordable oxygen microscopy-assisted biofabrication of multicellular spheroids. J Vis Exp. (182), e63403(2022).
- Peirsman, A., et al. Mispheroid: A knowledgebase and transparency tool for minimum information in spheroid identity. Nat Methods. 18 (11), 1294-1303 (2021).
- Raghavan, S., et al. Comparative analysis of tumor spheroid generation techniques for differential in vitro drug toxicity. Oncotarget. 7 (13), 16948(2016).
- Froehlich, K., et al. Generation of multicellular breast cancer tumor spheroids: Comparison of different protocols. J Mammary Gland Biol Neoplasia. 21 (3-4), 89-98 (2016).
- Debruyne, A. C., et al. Live microscopy of multicellular spheroids with the multimodal near-infrared nanoparticles reveals differences in oxygenation gradients. ACS Nano. 18 (19), 12168-12186 (2024).
- Debruyne, A. C., Okkelman, I. A., Dmitriev, R. I. Balance between the cell viability and death in 3D. Semin Cell Dev Biol. 144, 55-66 (2023).
- Barroso, M., Monaghan, M. G., Niesner, R., Dmitriev, R. I. Probing organoid metabolism using fluorescence lifetime imaging microscopy (flim): The next frontier of drug discovery and disease understanding. Adv Drug Deliv Rev. 201, 115081(2023).
- Becker, W. Fluorescence lifetime imaging-techniques and applications. J Microsc. 247 (2), 119-136 (2012).
- Dmitriev, R. I., Intes, X., Barroso, M. M. Luminescence lifetime imaging of three-dimensional biological objects. J Cell Sci. 134 (9), 1-17 (2021).
- Sarder, P., Maji, D., Achilefu, S. Molecular probes for fluorescence lifetime imaging. Bioconjug Chem. 26 (6), 963-974 (2015).
- Alfonso-Garcia, A., et al. Mesoscopic fluorescence lifetime imaging: Fundamental principles, clinical applications and future directions. J Biophotonics. 14 (6), e202000472(2021).
- Datta, R., Heaster, T. M., Sharick, J. T., Gillette, A. A., Skala, M. C. Fluorescence lifetime imaging microscopy: Fundamentals and advances in instrumentation, analysis, and applications. J Biomed Opt. 25 (7), 1-43 (2020).
- Yellen, G. Fueling thought: Management of glycolysis and oxidative phosphorylation in neuronal metabolism. J Cell Biol. 217 (7), 2235-2246 (2018).
- Heikal, A. A. Intracellular coenzymes as natural biomarkers for metabolic activities and mitochondrial anomalies. Biomark Med. 4 (2), 241-263 (2010).
- Kolenc, O. I., Quinn, K. P. Evaluating cell metabolism through autofluorescence imaging of nad (p) h and fad. Antioxid Redox Signal. 30 (6), 875-889 (2019).
- Verma, A., et al. Fluorescence lifetime imaging for quantification of targeted drug delivery in varying tumor microenvironments. bioRxiv. , 2024(2024).
- Smith, J. T., et al. In vivo quantitative fret small animal imaging: Intensity versus lifetime-based fret. Biophys Rep. 3 (2), 100110(2023).
- Alvarez, L. A., et al. Application Note: SP8 Falcon: A novel concept in fluorescence lifetime imaging enabling video-rate confocal flim. Nat Methods. , (2019).
- Roberti, M. J., et al. TauSense: A fluorescence lifetime-based tool set for everyday imaging. Nat. Methods. , https://www.nature.com/articles/d42473-020-00364-w.pdf (2020).
- Auer, J. M. T., Murphy, L. C., Xiao, D., Li, D. U., Wheeler, A. P. Non-fitting flim-fret facilitates analysis of protein interactions in live zebrafish embryos. J Microsc. 291 (1), 43-56 (2023).
- Warren, S. C., et al. Rapid global fitting of large fluorescence lifetime imaging microscopy datasets. PLoS One. 8 (8), e70687(2013).
- Gao, D., et al. Flimj: An open-source imagej toolkit for fluorescence lifetime image data analysis. PloS One. 15 (12), e0238327(2020).
- Tullis, I. D. C., Ameer-Beg, S. M., Barber, P. R., Rankov, V., Vojnovic, B. Mapping femtosecond pulse front distortion and group velocity dispersion in multiphoton microscopy. Proc. SPIE 6089, Multiphoton Microscopy in the Biomedical Sciences VI. , 60890(2006).
- Zhou, Y., et al. One-step derivation of functional mesenchymal stem cells from human pluripotent stem cells. Bio Protoc. 8 (22), e3080(2018).
- Benton, G., Arnaoutova, I., George, J., Kleinman, H. K., Koblinski, J. Matrigel: From discovery and ECM mimicry to assays and models for cancer research. Adv Drug Deliv Rev. 79-80, 3-18 (2014).
- Badea, M. A., et al. Influence of matrigel on single-and multiple-spheroid cultures in breast cancer research. SLAS Discov. 24 (5), 563-578 (2019).
- Lang, S., Sharrard, R., Stark, M., Villette, J., Maitland, N. Prostate epithelial cell lines form spheroids with evidence of glandular differentiation in three-dimensional matrigel cultures. Br J Cancer. 85 (4), 590-599 (2001).
- Barra, J., et al. DMT1-dependent endosome-mitochondria interactions regulate mitochondrial iron translocation and metastatic outgrowth. Oncogene. 43 (9), 650-667 (2024).
- Dmitriev, R. I., Papkovsky, D. B. Intracellular probes for imaging oxygen concentration: How good are they. Methods Appl Fluoresc. 3 (3), 034001(2015).
- Schweikhard, V., et al. Application Note: The power HyD family of detectors for confocal microscopy. Nat Methods. , https://www.nature.com/articles/d42473-020-00398-0 (2020).
- Alam, S. R., Wallrabe, H., Christopher, K. G., Siller, K. H., Periasamy, A. Characterization of mitochondrial dysfunction due to laser damage by 2-photon flim microscopy. Sci Rep. 12 (1), 11938(2022).
- Bush, P. G., Wokosin, D. L., Hall, A. C. Two-versus one photon excitation laser scanning microscopy: Critical importance of excitation wavelength. Front Biosci. 12, 2646-2657 (2007).
- Liu, M., et al. Instrument response standard in time-resolved fluorescence spectroscopy at visible wavelength: Quenched fluorescein sodium. Appl Spectrosc. 68 (5), 577-583 (2014).
- Szabelski, M., et al. Collisional quenching of erythrosine b as a potential reference dye for impulse response function evaluation. Appl Spectrosc. 63 (3), 363-368 (2009).
- Chib, R., et al. Standard reference for instrument response function in fluorescence lifetime measurements in visible and near infrared. Meas Sci Technol. 27 (2), 027001(2015).
- Talbot, C. B., et al. Application of ultrafast gold luminescence to measuring the instrument response function for multispectral multiphoton fluorescence lifetime imaging. Opt Express. 19 (15), 13848-13861 (2011).
- Becker, W. Recording the instrument response function of a multiphoton flim system. Becker & Hickl. , Available from: https://www.becker-hickl.com/literature/application-notes/recording-the-instrument-response-function-of-a-multiphoton-flim-system/ (2007).
- Leben, R., Köhler, M., Radbruch, H., Hauser, A. E., Niesner, R. A. Systematic enzyme mapping of cellular metabolism by phasor-analyzed label-free nad (p) h fluorescence lifetime imaging. Int J Mol Sci. 20 (22), 5565(2019).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using nadh and nadph autofluorescence. Free Radic Biol Med. 100, 53-65 (2016).
- Gottlieb, D., Asadipour, B., Kostina, P., Ung, T. P. L., Stringari, C. FLUTE: A python gui for interactive phasor analysis of flim data. Biol Imaging. 3, e21(2023).
- Malacrida, L., Ranjit, S., Jameson, D. M., Gratton, E. The phasor plot: A universal circle to advance fluorescence lifetime analysis and interpretation. Annu Rev Biophys. 50, 575-593 (2021).
- Okkelman, I., Vandenberghe, W., Dmitriev, R. Role of preconditioning with oxygen and glucose deprivation in promoting differentiation of dental pulp stem cells in 3D culture. Mol Biol Cell. 34 (2), 1212(2022).
- Park, J. H., et al. The effect of bmp-mimetic peptide tethering bioinks on the differentiation of dental pulp stem cells (DPSCs) in 3D bioprinted dental constructs. Biofabrication. 12 (3), 035029(2020).
- Pașca, S. P. The rise of three-dimensional human brain cultures. Nature. 553 (7689), 437-445 (2018).
- Dmitriev, R. I., Zhdanov, A. V., Nolan, Y. M., Papkovsky, D. B. Imaging of neurosphere oxygenation with phosphorescent probes. Biomaterials. 34 (37), 9307-9317 (2013).
- Bryanskaya, E. O., et al. High levels of FAD autofluorescence indicate pathology preceding cell death. Biochim Biophys Acta Gen Subj. 1868 (1), 130520(2024).
- Han, S. J., Kwon, S., Kim, K. S. Challenges of applying multicellular tumor spheroids in preclinical phase. Cancer Cell Int. 21 (1), 152(2021).
- Okkelman, I. A., Neto, N., Papkovsky, D. B., Monaghan, M. G., Dmitriev, R. I. A deeper understanding of intestinal organoid metabolism revealed by combining fluorescence lifetime imaging microscopy (flim) and extracellular flux analyses. Redox Biol. 30, 101420(2020).
- Gstraunthaler, G., Seppi, T., Pfaller, W. Impact of culture conditions, culture media volumes, and glucose content on metabolic properties of renal epithelial cell cultures: Are renal cells in tissue culture hypoxic. Cell Physiol Biochem. 9 (3), 150-172 (1999).
- Glickman, R. D. Phototoxicity to the retina: Mechanisms of damage. Int J Toxicol. 21 (6), 473-490 (2002).
- Golub, A. S., Pittman, R. N. Monitoring Parameters of Oxygen Transport to Cells in the Microcirculation. Quenched-Phosphorescence Detection of Molecular Oxygen: Applications in Life Sciences. , The Royal Society of Chemistry. (2018).
- Nishigaki, T., Wood, C. D., Shiba, K., Baba, S. A., Darszon, A. Stroboscopic illumination using light-emitting diodes reduces phototoxicity in fluorescence cell imaging. Biotechniques. 41 (2), 191-197 (2006).
- Penjweini, R., Loew, H. G., Hamblin, M. R., Kratky, K. W. Long-term monitoring of live cell proliferation in presence of pvp-hypericin: A new strategy using ms pulses of led and the fluorescent dye cfse. J Microsc. 245 (1), 100-108 (2012).
- Carrasco Kind, M., et al. flimview: A software framework to handle, visualize and analyze flim data. F1000Research. 9, 574(2020).
- Schrimpf, W., Barth, A., Hendrix, J., Lamb, D. C. Pam: A framework for integrated analysis of imaging, single-molecule, and ensemble fluorescence data. Biophys J. 114 (7), 1518-1528 (2018).
- Chen, S. -J., Sinsuebphon, N., Barroso, M., Intes, X., Michalet, X. Optical Molecular Probes, Imaging and Drug Delivery. , Optica Publishing Group. (2021).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved