Method Article
Desarrollo de ensayos para el cribado de fármacos de alto rendimiento contra micobacterias
En este artículo
Resumen
Este estudio desarrolló un ensayo para el cribado de alto rendimiento contra Mycobacterium abscessus con una cepa doble reportera de reciente creación, utilizando fluorescencia y luminiscencia para evaluar rápidamente la viabilidad bacteriana. Este protocolo será relevante para los investigadores que buscan cribar nuevos fármacos contra esta bacteria resistente a los fármacos.
Resumen
Las infecciones por Mycobacterium abscessus (Mab) son difíciles de tratar debido a la alta resistencia intrínseca a los medicamentos, comparable a la tuberculosis multirresistente. Los tratamientos son extremadamente ineficaces y se basan en un régimen de múltiples fármacos, lo que resulta en un bajo cumplimiento por parte del paciente. En consecuencia, se insta a la comunidad científica a identificar fármacos nuevos y eficaces para tratar estas infecciones. Una de las estrategias empleadas para este fin es la readaptación de fármacos, es decir, el proceso de identificación de nuevas oportunidades terapéuticas para los fármacos existentes en el mercado, evitando el tiempo necesario para establecer los perfiles farmacocinéticos y de seguridad de los nuevos fármacos. Dado que la mayoría de los estudios sobre el desarrollo de fármacos contra el Mab se basan en métodos tradicionales y lentos, se desarrolló un ensayo para el cribado de fármacos de alto rendimiento contra las micobacterias utilizando una cepa de Mab doble reportera desarrollada internamente. Mediante el uso de robótica de manejo de líquidos, microscopía automatizada y análisis, junto con cepas reporteras dobles desarrolladas internamente, la viabilidad bacteriana se puede medir rápidamente utilizando dos lecturas diferentes, luminiscencia y fluorescencia, sin agregar reactivos ni realizar ningún paso adicional. Esto reduce el tiempo y la variabilidad entre ensayos, una gran ventaja para los cribados de alto rendimiento. El protocolo descrito se validó mediante el cribado de una biblioteca de 1280 compuestos. Los resultados obtenidos fueron corroborados por la literatura, con detección eficiente de compuestos activos. De esta forma, este trabajo cumplió el objetivo de dotar al campo de una nueva herramienta que ayudara a combatir esta bacteria extremadamente resistente a los fármacos.
Introducción
Mycobacterium abscessus (Mab) es un patógeno oportunista responsable de infecciones pulmonares, especialmente en personas con fibrosis quística y otros trastornos pulmonares. Las infecciones causadas por el Mab son infamemente difíciles de tratar debido a la exquisita resistencia intrínseca a los medicamentos, comparable a la tuberculosis multirresistente1. Los fármacos disponibles son en gran medida ineficaces debido a la envoltura celular micobacteriana altamente impermeable y a un genoma que codifica varias enzimas que desactivan losantibióticos. Por lo tanto, el tratamiento incluye la combinación de varios medicamentos que tarda meses o años. Este desafiante régimen de múltiples fármacos, combinado con un bajo cumplimiento por parte de los pacientes, da como resultado una tasa de curación promedio del 30 % al 50 %3. Además, la prevalencia de infecciones pulmonares causadas por micobacterias no tuberculosas ha aumentado en las últimas décadas, incluidas las causadas por Mab 1,4. En consecuencia, la comunidad científica está compitiendo para desarrollar nuevos compuestos para tratar las infecciones por Mab.
Una de las estrategias que se persiguen con este objetivo es la reutilización de fármacos, es decir, el proceso de identificación de nuevas oportunidades terapéuticas para los fármacos existentes. De este modo, se evita el mayor reto que acompaña a la fase de descubrimiento y desarrollo de un nuevo fármaco: el tiempo5. Este sencillo concepto aprovecha los perfiles farmacocinéticos y de seguridad ya establecidos de varios fármacos para reducir los costes de desarrollo y acortar el tiempo que se tarda en llevar un fármaco desde el laboratorio hasta la cabecera delpaciente. Por lo tanto, se han compilado bibliotecas que combinan cientos o miles de dichos compuestos, lo que permite a los investigadores probar rápidamente la posibilidad de reutilizar medicamentos contra el patógeno de interés.
La mayoría de los estudios sobre el desarrollo de fármacos contra el Mab se basan en un ensayo tradicional que evalúa la actividad in vitro de un compuesto frente a las micobacterias, es decir, las unidades formadoras de colonias7. A pesar de su precisión, este procedimiento requiere mucho tiempo, y rápidamente se vuelve inviable cuando alguien pretende probar bibliotecas que contienen miles de compuestos. Con este fin, se han integrado en el desarrollo de fármacos cribados de alto rendimiento (HTS), ensayos robustos que aprovechan la robótica y los dispositivos de manipulación de líquidos, lo que permite cribar rápidamente miles de compuestos en paralelo8. Por lo general, esto se hace probando una sola concentración inicialmente, utilizando placas de microtitulación de formatos de 96, 384, 1536 o 3456 pocillos, que actúan como punto de partida para identificar los resultados y optimizarlos aún más en el proceso para su uso clínico.
Los ensayos basados en reporteros proporcionan una ventaja significativa para la robustez de HTS debido a su simplicidad y sensibilidad en comparación con otros ensayos basados en colorantes y absorbancias 7,9. Sin embargo, hasta donde sabemos, solo unos pocos estudios han optimizado un cribado de alto rendimiento contra Mab9.
Recientemente, nuestro laboratorio ha desarrollado cepas reporteras dobles capaces de emitir luminiscencia y fluorescencia10 de forma concomitante. La Mab operon_mScarlet es una de esas variedades. Es autoluminiscente debido a la expresión del operón LuxABCDE , que incluye una luciferasa bacteriana (por expresión de los genes luxAB ) y un sustrato de aldehído de cadena larga (por expresión de los genes luxCDE ). Por otro lado, la lectura fluorescente se obtiene a través de la expresión de una proteína fluorescente roja recientemente desarrollada, mScarlet, que supera a las proteínas eGFP y mCherry más utilizadas, proporcionando una señal más potente11. El uso de esta cepa nos permite evaluar la viabilidad bacteriana en cultivo líquido midiendo la señal luminiscente en un lector de microplacas sin añadir reactivos ni realizar pasos adicionales. En términos de detección, la fluorescencia intrínseca permite la visualización al microscopio en células vivas o fijas sin utilizar colorantes ni anticuerpos. Tener una sola cepa con ambas lecturas proporciona a los investigadores una ventaja significativa cuando se utiliza en ensayos HTS: la variabilidad reducida entre ensayos con diferentes lecturas, ya que no es necesario intercambiar cepas en función de la naturaleza del ensayo.
Por lo tanto, este trabajo desarrolló un ensayo de alto rendimiento contra Mab utilizando una deformación doble reportera diseñada internamente (Figura 1). Esto permitió una evaluación rápida de la actividad in vitro de los fármacos 1280 destinados a la reutilización utilizando una biblioteca comercial (véase la Tabla de Materiales). En primer lugar, las actividades se evaluaron en un ensayo de cultivo en caldo utilizando luminiscencia y, en segundo lugar, utilizando macrófagos infectados con Mab aprovechando la señal fluorescente, emulando mejor el proceso de infección observado in vivo12.
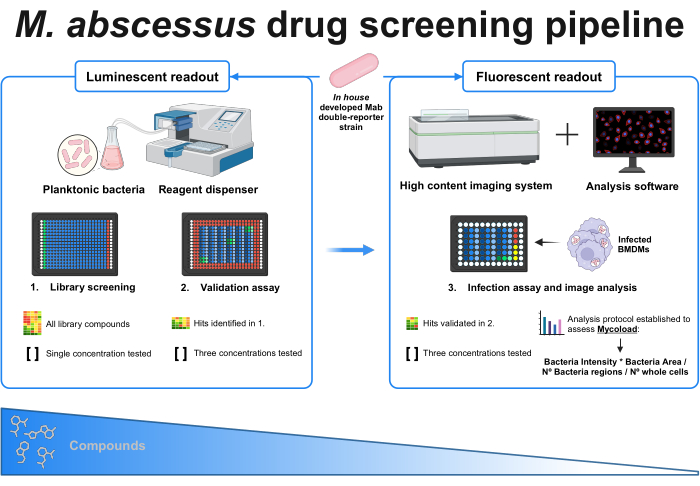
Figura 1: Resumen gráfico del protocolo establecido. El actor clave en este cribado es una cepa de micobacterias doble reportera desarrollada internamente , que se utiliza en todos los experimentos. En primer lugar, utilizando un dosificador de reactivos y bacterias planctónicas, se realiza un cribado inicial probando los compuestos en una sola concentración. Los resultados identificados continúan en el ensayo de validación, donde se prueban tres concentraciones diferentes. Ambos experimentos se realizan utilizando la lectura luminiscente. Los resultados validados continúan en el ensayo de infección, donde también se prueban tres concentraciones diferentes, y los macrófagos derivados de la médula ósea se infectan en MOI 1. Un sistema de imágenes de alto contenido adquiere la señal fluorescente bacteriana y se utiliza un software de análisis para evaluar la carga intracelular a través de la fórmula Mycoload. El número de compuestos se reduce a medida que aumenta la complejidad del ensayo. Creado con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Todos los procedimientos con animales fueron aprobados por el Comité Local de Ética Animal de i3S y autorizados por la Dirección General de Alimentación y Veterinaria de la Autoridad Portuguesa (DGAV), siguiendo la Directiva del Consejo Europeo (2010/63/UE) y la ley portuguesa (DL 113/2013) para el bienestar animal en experimentación.
1. Evaluación del factor Z'
NOTA: Para evaluar el rendimiento de un ensayo HTS, se pueden determinar varios parámetros. El parámetro más utilizado es el factor primo Z (factor Z'). Esta métrica mide el tamaño del efecto estadístico de un experimento determinado, lo que indica qué tan separados están los controles positivos y negativos. Si un factor Z' oscila entre 0,5 y 1, la separación entre ambas poblaciones es excelente, y estas condiciones se pueden utilizar en el ensayo HTS. Si Z' < 0,5, la separación es solo marginal y no se recomienda realizar el cribado13. Para ello, se diseñó un experimento utilizando la mitad de una placa con bacterias no tratadas y la otra mitad con un testigo positivo para calcular el factor Z' (Figura 1 suplementaria).
- A los 2-3 días antes del ensayo, prepare los precultivos de Mab inoculando bacterias en un medio de crecimiento líquido (aproximadamente 1 x 107 UFC/mL) en un matraz Erlenmeyer. Deje que las bacterias crezcan a 37 °C en una incubadora con agitación (90 rpm).
PRECAUCIÓN: Se requiere una cabina de bioseguridad cuando se trabaja con cultivos Mab. - Siga el crecimiento del precultivo del Mab mediante la medición de la densidad óptica a 600 nm utilizando un espectrofotómetro. Cuando la cultura alcance la fase de crecimiento exponencial, continúe con el paso 1.3.
- Utilizando el precultivo exponencial, prepare una suspensión bacteriana de aproximadamente 5 x 105 UFC/mL (correlacionada con los valores de densidad óptica)14 en medio de crecimiento líquido.
- Prepare una solución de control positivo de inhibición del crecimiento bacteriano en un medio de crecimiento líquido. Para este ensayo, el control positivo fue claritromicina (10 mg/mL en DMSO) a 4 μg/mL. La claritromicina debe prepararse 2 veces la concentración final deseada.
- Utilizando un dispensador de reactivo de microplaca equipado con un casete pequeño, dispense 15 μL de medio de crecimiento líquido a las columnas en blanco y no tratadas de una placa de polipropileno negro de 384 pocillos. Para las columnas en blanco, dispense 15 μL adicionales de medio de crecimiento líquido.
NOTA: Las columnas en blanco evitan la evaporación de los pozos interiores a 37 °C. - Con el dispensador de reactivo de microplaca equipado con un casete pequeño, dispense 15 μL de la solución de control positivo preparada en el paso 1.4 a las columnas de control positivo.
- Con un dispensador de reactivos de microplaca equipado con un casete grande, dispense 15 μL de la suspensión bacteriana preparada en el paso 1.3 a todas las columnas excepto a los blancos, diluyendo todo 1:2. El volumen final de los 384 pocillos es de 30 μL.
NOTA: El dispensador de reactivo de microplaca puede equiparse con un casete grande o pequeño, que difiere en el tamaño del tubo (0,5 y 0,22 mm, respectivamente). Es preferible utilizar un casete más grande para evitar la agregación bacteriana en el sistema de tubos, aunque resulte en un mayor desperdicio de líquido. No obstante, se puede utilizar el mismo casete durante todo el ensayo. - Selle cada placa con un sellador de placas que permita el intercambio continuo de gases. Incubar a 37 °C en una incubadora sin agitación dentro de una cámara húmeda durante 48 h.
- A las 48 h, lea la luminiscencia emitida por las bacterias en cada placa utilizando un lector de placas con un tiempo de integración de 1 s.
- Para determinar el factor Z', utilice los valores obtenidos en el paso 1.9 y calcule el promedio y la desviación estándar de los controles positivo (AVGP y SDP) y negativo (AVGN y SDN). Después, aplica la siguiente fórmula:
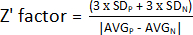
NOTA: 3 x SD es un umbral comúnmente utilizado; Sin embargo, se puede ajustar para que se adapte mejor a la detección, ya sea aumentando o disminuyendo el factor por el cual se multiplica la desviación estándar.
2. Cribado de éxitos en la biblioteca
NOTA: Los primeros cribados se realizaron contra bacterias que crecían en cultivo líquido. La configuración fue diseñada para placas de 384 pocillos con 30 μL de volumen final. Cada placa contenía pocillos en blanco (solo medio de crecimiento líquido), controles negativos (bacterias no tratadas) y control de solvente (bacterias más solvente de compuesto, en este caso, DMSO; Figura complementaria 2). Los compuestos o el disolvente se añadieron a la placa a 2 veces la concentración final deseada, y las bacterias se añadieron 1:1 (diluyendo todo a la mitad).
- Prepare la suspensión bacteriana como se describe en los pasos 1.1 a 1.3.
PRECAUCIÓN: Se requiere una cabina de bioseguridad cuando se trabaja con cultivos Mab. - Prepare una solución de solvente en un medio de crecimiento líquido. La cantidad de disolvente debe ser 2 veces la concentración final deseada.
- Utilizando un dispensador de reactivo de microplacas equipado con un casete pequeño, dispense 15 μL de medio de crecimiento líquido a cada columna de placas de polipropileno negro de 384 pocillos, excepto a la columna de control de solventes. En el caso de las columnas en blanco, dispense 15 μl adicionales de medio de crecimiento líquido.
- Con el dispensador de reactivo de microplacas equipado con un pequeño casete, dispense 15 μL de la solución de disolvente previamente preparada a la columna de control de disolvente.
- Usando un manipulador de líquidos, transfiera 200 nL de los compuestos de las placas de 96 pocillos (1 mM en DMSO) a las placas de 384 pocillos, excepto las columnas en blanco, control de solventes y no tratadas. Los compuestos se encuentran ahora en una concentración de 13,3 μM.
- Con un dispensador de reactivos de microplaca equipado con un casete grande, dispense 15 μL de la suspensión bacteriana preparada en el paso 2.1 a todas las columnas excepto a los blancos, diluyendo todo 1:2. El volumen final de los 384 pozos es de 30 μL, y cada compuesto es de 6,66 μM.
- Selle cada placa con un sellador de placas que permita el intercambio continuo de gases.
NOTA: El ensayo se puede realizar con tapas regulares; Sin embargo, existe una mayor posibilidad de efectos de borde más fuertes. - Incubar a 37 °C en una incubadora sin agitación dentro de una cámara húmeda durante 48 h.
- A las 48 h, lea la luminiscencia emitida por las bacterias en cada placa utilizando un lector de placas con un tiempo de integración de 1 s.
3. Análisis de datos de detección de resultados
NOTA: Para identificar los aciertos con los datos de luminiscencia adquiridos, los valores deben normalizarse al disolvente en el que se disuelven los compuestos y, si se produce, al efecto de borde verificado en las placas de 384 microtítulos.
- Para la normalización del disolvente de los datos obtenidos en el paso 2.9, divida las unidades ligeras relativas (RLU) de cada pocillo por la RLU media de los pocillos de control del disolvente (en la placa correspondiente). En este ensayo, se verificó un fuerte efecto de borde hacia el centro de las placas de cribado; Por lo tanto, se decidió normalizar todos los datos para ello. Para lograr esto, el proceso se realizó en dos pasos, como se describe a continuación.
- Cree una máscara de efecto de borde dividiendo los valores obtenidos en el paso 3.1 para las posiciones correspondientes, excluyendo los golpes, en todas las placas (placa 1 - P1, placa 2 - P2, placa 3 - P3, placa 4 - P4). Por ejemplo, para las posiciones A1 y A2, Mask_A1 = Promedio (P1_A1; P2_A1; P3_A1; P4_A1); Mask_A2 = Promedio (P1_A2; P2_A2; P4_A2) [P3_A2 fue excluido porque fue un éxito].
- Divida los valores obtenidos en el paso 3.1. por su máscara de efecto de borde obtenida en el paso 3.1.1. Por ejemplo,
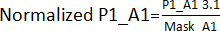 . A continuación, calcule el AVG y el SD de cada placa para establecer un umbral (AVG ± 3SD) para cada placa. Cualquier compuesto fuera de este umbral se considera un resultado positivo y se incluye en el ensayo de validación de impacto.
. A continuación, calcule el AVG y el SD de cada placa para establecer un umbral (AVG ± 3SD) para cada placa. Cualquier compuesto fuera de este umbral se considera un resultado positivo y se incluye en el ensayo de validación de impacto.
4. Ensayo de validación de aciertos
NOTA: Después de identificar los resultados, estos deben validarse utilizando diferentes concentraciones. El ensayo de validación utiliza una configuración similar con los mismos controles, pero cada golpe se someterá a diluciones en serie 1:2. Para este ensayo, se utilizaron tres concentraciones: 13,3 μM, 6,66 μM y 3,3 μM. Los controles de solvente contendrán el mismo % de solvente que cada pozo compuesto (Figura suplementaria 3).
- Prepare la suspensión bacteriana como se describe en los pasos 1.1 a 1.3.
PRECAUCIÓN: Se requiere una cabina de bioseguridad cuando se trabaja con cultivos Mab. - Utilizando un dispensador de reactivo de microplaca equipado con un casete pequeño, dispense 15 μL de medio de crecimiento líquido a cada columna de una placa de polipropileno negro de 384 pocillos. En el caso de los pocillos en blanco, dispense 15 μL adicionales de medio de crecimiento líquido a la primera y última columna.
- Con el dispensador de reactivo de microplaca, dispense 15 μL adicionales de medio de crecimiento líquido a las columnas que contengan la mayor cantidad de [μM] de cada compuesto.
- De las placas de 96 pocillos originales, pipetee manualmente 0,8 μL de cada golpe (1 mM en DMSO) en sus pocillos respectivos desde el paso 4.3, por duplicado. Los compuestos se encuentran ahora en una concentración de 26,6 μM.
- Pipetear manualmente 0,8 μL de disolvente en los pocillos de control por duplicado (igual que en el paso 4.4).
- Con una micropipeta multicanal, realice dos diluciones en serie 1:2 para cada compuesto y control de disolventes. Deseche los 15 μL que excedan después de la última dilución en serie. Todos los pozos tienen ahora 15 μL y las concentraciones de los compuestos son de 26,6 μM, 13,3 μM y 6,66 μM.
- Utilizando un dispensador de reactivo de microplaca con un casete grande, dispense 15 μL de la suspensión bacteriana preparada en 4.1 a todas las columnas (excepto a los pocillos en blanco), diluyendo los compuestos 1:2. Todos los pozos tienen un volumen final de 30 μL y cada compuesto tiene 13,3 μM, 6,66 μM y 3,3 μM.
- Selle la placa con un sellador de placas que permita el intercambio continuo de gases. Incubar a 37 °C en una incubadora sin agitación dentro de una cámara húmeda durante 48 h.
- A las 48 h, leer la luminiscencia emitida por las bacterias en cada placa con un tiempo de integración de 1 s utilizando un lector de placas.
5. Ensayo de infección
NOTA: Mab es un patógeno facultativo intracelular, por lo que la actividad antimicrobiana y la toxicidad del huésped deben determinarse en ensayos de infección. Para ello, los macrófagos derivados de la médula ósea (BMM) se infectaron con Mab y se trataron con los hits validados en el paso 4 a diferentes concentraciones. Cada ensayo incluyó blanco (solo agua), controles negativos (macrófagos infectados no tratados), controles positivos (macrófagos infectados tratados con un antibiótico eficaz), controles con disolventes (en este caso, DMSO) y los compuestos a probar en tres concentraciones: 13,3 μM, 6,66 μM y 3,3 μM (Figura complementaria 4).
- Derivan macrófagos de la médula ósea murina de ratones adultos de tipo salvaje (BALB/c o C57BL/6), como se ha descrito anteriormente15. Coloque las celdas en una placa de fondo plano de 96 pocillos, negra, ópticamente transparente, a 2 x 105 celdas/mL, 200 μL por pocillo. Después de 10 días de diferenciación a 37 °C, 7% de CO215, las células están listas para infectar y continuar con el tratamiento.
NOTA: El protocolo para la obtención de macrófagos derivados de la médula ósea está bien establecido15; sin embargo, este protocolo de cribado puede adaptarse a otras células, como RAW 264.7, THP-1 y macrófagos derivados de PBMC. - Prepare la suspensión bacteriana como se describe en los pasos 1.1 a 1.3, ajustando la suspensión bacteriana en el medio de cultivo celular para una infección con una multiplicidad de infección (MOI) de 1.
PRECAUCIÓN: Se requiere una cabina de bioseguridad cuando se trabaja con cultivos Mab. - Aspire cuidadosamente el medio de cultivo celular suplementado de los pocillos utilizando una bomba de vacío conectada a una pipeta de vidrio. Este paso debe realizarse lentamente y con facilidad para evitar el desprendimiento de células.
- Pipetear manualmente 75 μL de la suspensión bacteriana preparada en el paso 5.2 en cada pocillo. Incubar la placa a 37 °C con 7% de CO2 durante 4 h en una incubadora de celdas.
- Preparar los compuestos para el tratamiento celular. Utilizando un dispensador de reactivos de microplacas equipado con un casete pequeño, dispense a polipropileno, fondo redondo, placas de 96 pocillos, 110 μL de medio de cultivo celular suplementado a las columnas donde se estarán los compuestos o el control de solventes (Figura complementaria 4).
- Utilizando un dispensador de reactivo de microplaca, dispense 104 μL adicionales de medio de cultivo celular suplementado a las columnas donde se encontrará la concentración más alta de compuesto o solvente.
- Pipetear manualmente 5,9 μL de los compuestos (1 mM en DMSO) o disolvente en sus pocillos designados (columnas en el paso 5.6). Los compuestos se encuentran ahora en una concentración de 26,6 μM.
- Con una micropipeta multicanal, realice dos diluciones en serie 1:2 para cada compuesto y disolvente. Deseche los 110 μL que excedan después de la última dilución en serie. Todos los pozos ahora tienen 110 μL y los compuestos están a una concentración de 26,6 μM, 13,3 μM y 6,66 μM.
- Con un dispensador de reactivo en microplaca, dispense 110 μL de medio de cultivo celular suplementado en todas las columnas (excluyendo los pocillos en blanco). Todos los pozos tienen ahora 220 μL y los compuestos están a una concentración de 13,3 μM, 6,66 μM y 3,3 μM.
- Prepare una solución de claritromicina (control positivo) a 2 μg/mL en un medio de cultivo celular suplementado.
- A las 4 h después del paso 5.4, utilizando un arandeplatos, lavar 3 veces con 200 μL de solución de lavado para infecciones la placa de 96 pocillos infectada. Mantenga las velocidades de aspiración y dispensación al mínimo posible para evitar cualquier desprendimiento de células.
- Después de la aspiración final, transfiera 200 μL de los compuestos preparados en los pasos 5.5-5.9 a la placa de 96 pocillos que contiene los macrófagos infectados.
- Transfiera 200 μL de medio de cultivo celular suplementado a los pocillos no tratados. Transfiera 200 μL de la solución de claritromicina preparada en el paso 5.10 a sus pocillos respectivos.
- Incubar la placa en una incubadora de celdas a 37 °C, con 7% de CO2, durante 48 h.
- Después de 48 h, lavar la placa de 96 pocillos infectada con un arandela de placas con la solución de lavado del compuesto (3x con 200 μL por pocillo).
- Con una micropipeta multicanal, dispense 200 μL de la solución de fijación a todos los pocillos y déjela actuar durante 10 minutos.
- Después de la fijación, utilizando un arandela de placas, lavar las células con la solución de lavado de los compuestos (3x con 200 μL por pocillo).
NOTA: El protocolo se puede pausar aquí. Las células pueden almacenarse en la solución de lavado de los compuestos a 4 °C durante varios días o semanas si se evita la contaminación y la sequedad de las células. La solución de lavado de los compuestos debe aspirarse antes del paso 5.18. - Con una micropipeta multicanal, dispense 200 μL de la solución permeable a todos los pocillos y déjela actuar durante 15 min. Con una arandela de placas, aspire la solución permeable.
- Con una micropipeta multicanal, dispense manualmente 100 μL de la solución de tinción e incube las placas a temperatura ambiente durante 30 minutos.
- Con una arandela de placas, lave las placas con la solución de lavado de los compuestos (3x con 200 μL por pocillo). Al final, deje las células teñidas en la solución de lavado de los compuestos.
- Filtre las placas en un sistema de imágenes de alto contenido con los siguientes ajustes:
Objetivo: 20x Air/0.4NA (un pocillo de una placa de 96 microtítulos, 57 campos de visión (FOV)
Modo: Confocal
Láser 405 nm para DAPI (núcleos)
Láser 561 nm para mScarlet (bacterias)
Láser de 640 nm para HCS Cell Mask Deep Red (citoplasma)
Sin pila z
NOTA: Las células se pueden visualizar con un sistema de imágenes de campo amplio, lo que disminuye la definición. - Configure un robot de manejo de equipos para la adquisición de imágenes durante la noche. Este brazo robótico intercambia las placas en el sistema de imágenes de alto contenido sin intervención humana.
NOTA: Esto no es obligatorio; Las placas se pueden cambiar manualmente.
6. Análisis de imágenes
NOTA: El análisis de imágenes se realiza con el software de análisis, que es el mismo que el sistema de imágenes de alto contenido utilizado para adquirir las imágenes. Se debe crear una canalización de análisis utilizando imágenes de muestra (Figura complementaria 5). Posteriormente, se aplicará a todo el pozo (57 FOV) y al conjunto de datos (todas las placas). El software de análisis sigue una secuencia lógica de pasos, comenzando con la segmentación de las diferentes regiones de interés (núcleos, citoplasma, células y bacterias), relacionándolas (por ejemplo, bacterias dentro de las células) y luego extrayendo las propiedades morfológicas y de intensidad (por ejemplo, área, intensidad). A continuación se describen los pasos esenciales del análisis de imágenes. El protocolo completo utilizado se puede encontrar en la Figura complementaria 5.
- Segmente los núcleos usando la señal DAPI, el citoplasma usando la señal DeepRed y las bacterias usando la señal mScarlet.
- Relacionar los núcleos y la segmentación del citoplasma para crear una máscara para las células.
- Elimine todos los objetos de borde. Solo se considerarán las celdas que aparezcan como llenas en el campo de visión (celdas completas).
- Defina la población de salida de bacterias en células enteras relacionando células enteras y bacterias.
- Defina las células enteras infectadas como aquellas que tienen más de una región bacteriana y las células enteras no infectadas como aquellas que tienen menos de una región bacteriana.
- Una vez segmentadas todas las regiones de interés y creadas las poblaciones, calcule y extraiga sus propiedades. Se extraen las siguientes propiedades para cada pocillo.
- Para células enteras/ Células enteras infectadas/ Células enteras no infectadas, extracto
Número de celdas (suma por pocillo)
Área [μm2] (Media + Desviación estándar por pocillo)
Relación entre la anchura y la longitud de la celda (media + desviación estándar por pocillo) - Para las bacterias en células enteras, extracto
Número de regiones bacterianas identificadas (Suma por pocillo)
Intensidad (suma por pocillo de la intensidad media de cada región)
Área de la región (Suma por pozo)
- Para células enteras/ Células enteras infectadas/ Células enteras no infectadas, extracto
- Después de exportar los datos y seleccionarlos en una hoja de cálculo, cuantifique la carga intracelular (Mycoload) de acuerdo con la siguiente fórmula:
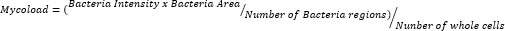
Resultados
Evaluación del factor Z'
La Figura 2 muestra los datos de uno de los dos experimentos realizados para calcular el factor Z'. El control positivo fue claritromicina a 4 μg/mL. Ambos experimentos arrojaron factores Z' de 0,64 y 0,62, lo que significa que las condiciones y la lectura utilizadas para este ensayo se pueden aplicar al ensayo de cribado posterior (Z' > 0,5). No obstante, se calculó el factor Z' para todos los experimentos restantes (ensayo de infección) para controlar el rendimiento de cada experimento.
Como prueba de concepto del ensayo HTS diseñado, se probó una biblioteca de compuestos destinados a la reutilización de fármacos. Comprende 1280 moléculas diversas y pequeñas, el 95% de las cuales son medicamentos aprobados por la FDA y la EMA. Estas moléculas ofrecen una alta diversidad química y farmacológica.
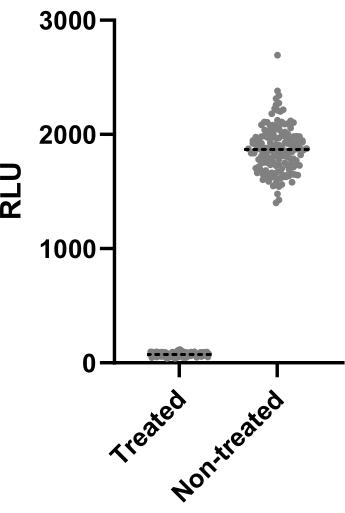
Figura 2: Resultados de la evaluación del factor Z'. Mab a 2,5 x 105 UFC/mL se incubó con y sin claritromicina a 4 μg/mL durante 48 h a 37 °C. Después del período de incubación, se midió la luminiscencia para evaluar la viabilidad de las micobacterias. El gráfico muestra los valores de luminiscencia individuales de las micobacterias viables tratadas y no tratadas en un experimento independiente. Haga clic aquí para ver una versión más grande de esta figura.
Proyección de éxitos en la biblioteca
Todos los compuestos se probaron a 6,66 μM frente a Mab. Debido al tamaño de la biblioteca, los compuestos se dividieron en cuatro placas de 384 pocillos de microtitulación diferentes (Figura 3A-D). Los resultados se normalizaron tanto para el solvente como para el efecto de borde verificado.
En este ensayo de cribado, se identificaron treinta y tres aciertos, treinta de los cuales disminuyeron significativamente la emisión de luminiscencia, reduciendo la viabilidad de las micobacterias (Figura 3). Curiosamente, tres compuestos condujeron a una mayor emisión de luminiscencia, posiblemente relacionada con un mayor metabolismo o proliferación bacteriana (Figura 3). Los 33 resultados se llevaron al ensayo de validación, incluidos los tres compuestos que aumentaron la luminiscencia, para probar si se mantendría este perfil.
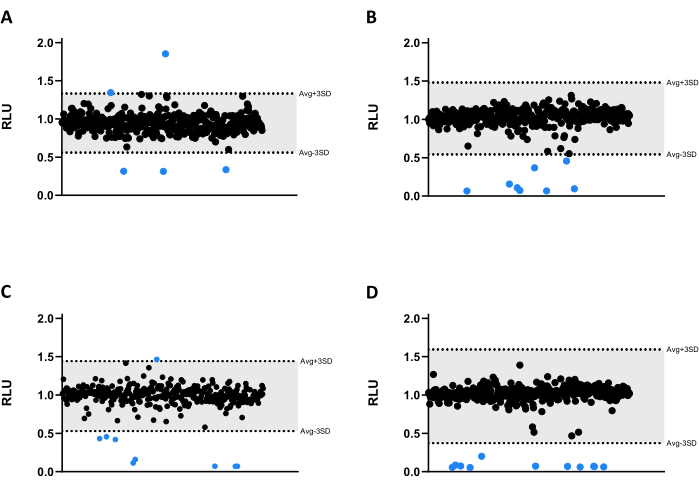
Figura 3: Resultados del cribado en la biblioteca. (A-D) Mab a 2,5 x 105 UFC/mL se incubó con 1280 compuestos a 6,66 μM durante 48 h a 37 °C. Después de la incubación, se midió la luminiscencia para evaluar la viabilidad de las micobacterias. Los gráficos muestran la RLU de un experimento independiente, presentada como unidades arbitrarias después de la normalización del efecto solvente y del borde. Se calculó la RLU AVG y su SD correspondiente para cada placa para determinar un umbral (en gris; AVG ± 3SD). Los símbolos redondos representan un compuesto probado. En azul, cualquier compuesto fuera de ese umbral se considera un éxito. Haga clic aquí para ver una versión más grande de esta figura.
Ensayo de validación de aciertos
Las concentraciones utilizadas para este ensayo fueron 13,3 μM, 6,66 μM y 3,3 μM. Cada compuesto se probó por duplicado para aumentar la robustez.
Es importante destacar que los compuestos 30, 31 y 32, previamente asociados con una RLU más alta (Figura 3A, C), mantuvieron este perfil, con más del 200% de viabilidad micobacteriana lograda en los pozos tratados con el compuesto 32 (Figura 4).
Los compuestos 20 y 33 se consideraron inactivos, ya que la viabilidad micobacteriana es cercana al 100% en todas las concentraciones analizadas (Figura 4).
Los compuestos 9 y 21 mostraron una inactividad similar en las dos concentraciones más bajas; sin embargo, a diferencia de 20, permanecen activos en el nivel más alto, 13,3 μM, y el compuesto 21 muestra una mayor potencia (Figura 4).
El compuesto 3 también muestra inactividad en la concentración más baja y una pérdida de actividad a 6,66 μM, aunque menor que el compuesto 21 (Figura 4). Solo en la concentración más baja, los compuestos 2, 7, 8, 10, 16, 18, 24 y 28 mostraron un menor potencial terapéutico. Sin embargo, 2, 10 y 28 aún conducen a un <50% de la viabilidad de las micobacterias.
A nivel mundial, los catorce compuestos restantes estaban activos en todas las concentraciones analizadas.
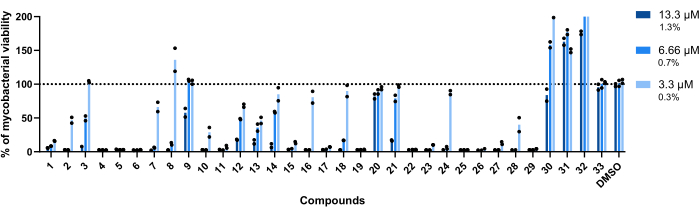
Figura 4: Resultados del ensayo de validación de aciertos. Se incubó Mab a 2,5 x 105 UFC/mL con cada golpe previamente identificado a 13,3 μM, 6,66 μM y 3,3 μM (1,3%, 0,7% y 0,3% de DMSO, respectivamente) durante 48 h a 37 °C. Después del período de incubación, se midió la luminiscencia para evaluar la viabilidad de las micobacterias. El gráfico muestra los porcentajes de micobacterias viables tratadas en relación con las micobacterias no tratadas de un experimento independiente, con cada compuesto probado por duplicado. Los símbolos redondos representan los duplicados de cada compuesto. Las barras representan el promedio de esos duplicados. Haga clic aquí para ver una versión más grande de esta figura.
Ensayo de infección
De los treinta y tres compuestos probados, se seleccionaron veintiocho para el ensayo de infección (Figura 4). Los compuestos 7, 8, 9, 20 y 33 no fueron seleccionados para este ensayo. Mientras que el 9, el 20 y el 33 quedaron fuera por su inactividad al validarse (Figura 4), los dos primeros quedaron fuera por razones técnicas. Sin embargo, estos compuestos fueron identificados como rifampicina y linezolid, antibióticos ya utilizados para el tratamiento de las infecciones por Mab12. Se identificaron todos los compuestos probados en el ensayo de infección y se enumeran en la Tabla 1. La actividad antimicrobiana de los compuestos contra los macrófagos infectados con Mab se evaluó utilizando la fluorescencia intrínseca de las bacterias como lectura.
| Compuesto | Nombre | Compuesto | Nombre |
| 1 | Sulfathiazol | 18 | Cefuroxima |
| 2 | Ciprofloxacino | 19 | Rifaximina |
| 3 | Cefotaxima | 21 | Cefdinir |
| 4 | Daunorrubicina | 22 | Claritromicina |
| 5 | Doxorrubicina | 23 | Besifloxacina |
| 6 | Thiostrepton | 24 | Levofloxacino |
| 10 | Amikacina | 25 | Rifabutin |
| 11 | Moxalactama | 26 | Gatifloxacina |
| 12 | Sulfamethizole | 27 | Epirubicina |
| 13 | Sulfamonometoxina | 28 | Pamoato de pirvinio |
| 14 | Cefoxitin | 29 | Moxifloxacina |
| 15 | Novobiocina | 30 | Troleandomicina |
| 16 | Cefmetazol | 31 | Lincomicina |
| 17 | Roxitromicina | 32 | Espiramicina |
Tabla 1: Lista de los compuestos probados en el ensayo de infección. Los compuestos validados en el paso 4 se probaron en el ensayo de infección (paso 5).
La toxicidad de los compuestos frente a los macrófagos infectados con Mab fue el primer parámetro evaluado. El umbral establecido para considerar un compuesto como tóxico o no tóxico fue del 85% de macrófagos viables (Figura 5). De los veintiocho compuestos analizados, 4, 5, 6, 27 y 28 se consideraron tóxicos. (Figura 5). Por lo tanto, estos cinco compuestos se excluyeron de la siguiente evaluación de actividad intramacrofágica.
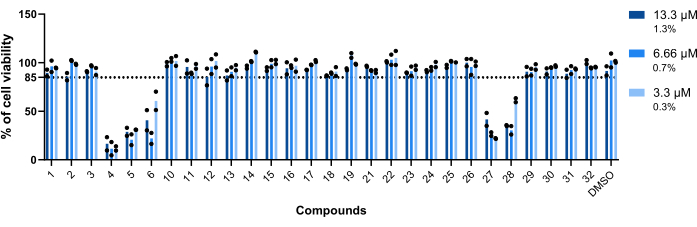
Figura 5: Toxicidad de Hits hacia macrófagos infectados con Mab. Los ratones BALB/c BMM se infectaron con Mab (MOI=1) y se incubaron con cada golpe previamente identificado a 13,3 μM, 6,66 μM y 3,3 μM (1,3%, 0,7% y 0,3% de DMSO, respectivamente) durante 48 h a 37 °C con 7% de CO2. Las células se observaron en un microscopio de fluorescencia de cribado de alto contenido, utilizando el número de núcleos (teñidos con DAPI) para medir la viabilidad celular. El gráfico muestra los porcentajes de macrófagos infectados viables tratados en relación con los macrófagos infectados viables no tratados de dos experimentos independientes. Los símbolos redondos representan la viabilidad de la célula para cada ensayo. Las barras representan el promedio de dos experimentos independientes. Haga clic aquí para ver una versión más grande de esta figura.
Para inferir la actividad intramacrofágica de los veintitrés compuestos restantes frente al Mab intracelular (Figura 6), se utilizó la fórmula Mycoload (explicada previamente en el paso 6) para obtener el porcentaje de viabilidad micobacteriana normalizada a los macrófagos infectados no tratados. La mayoría de los compuestos perdieron su potencial terapéutico frente a las micobacterias internalizadas (Figura 6) en comparación con el ensayo de validación de aciertos (Figura 4), ya que el número de aciertos se redujo de veinticinco a seis en la concentración más alta probada. Sorprendentemente, los tres compuestos que aumentaron la viabilidad del Mab planctónico en comparación con las bacterias no tratadas (30, 31 y 32; Figura 3 y Figura 4) mostraron actividad antimicobacteriana frente al Mab intracelular, presentándose los compuestos 30 y 32 una diferencia estadísticamente significativa en comparación con el DMSO, incluso a 3,3 μM en el caso del compuesto 32 (Figura 6). Las micobacterias tratadas con los compuestos 11 y 23 mostraron una viabilidad <50% a 13,3 μM; sin embargo, esto no fue significativamente diferente del control DMSO (Figura 6). Los compuestos 21, 26 y 29 fueron lo suficientemente potentes a 13,3 μM como para justificar una diferencia estadística significativa, siendo 29 el más activo (Figura 6). Por último, los compuestos 17, 22 y 25 fueron extremadamente potentes en todas las concentraciones probadas contra micobacterias internalizadas. Estos se identificaron como roxitromicina, claritromicina y rifabutina, respectivamente (Tabla 1). De los tres compuestos, la claritromicina fue el más activo frente al Mab, con una viabilidad micobacteriana que nunca superó el 10%, presentando un valor p <0,0001 en todas las concentraciones ensayadas en comparación con el DMSO (Figura 6).
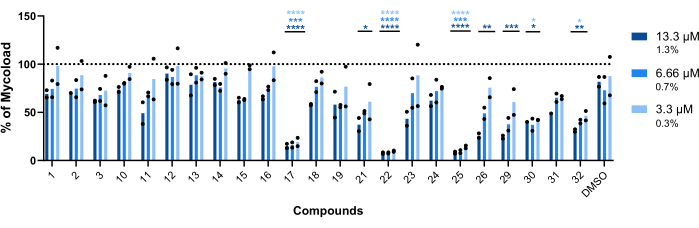
Figura 6: Actividad intramacrofágica de Hits frente a macrófagos infectados con Mab. Los BMM de ratones BALB/c se infectaron con Mab (MOI=1) y se incubaron con cada golpe previamente identificado a 13,3, 6,66 y 3,3 μM (1,3, 0,7 y 0,3% de DMSO, respectivamente) durante 48 h a 37 °C con 7% de CO2. Las células se obtuvieron en un microscopio de fluorescencia de cribado de alto contenido y se utilizó la señal fluorescente para calcular la micocarga (véase el paso 6). El gráfico muestra los porcentajes de Mycoload encontrados en los macrófagos infectados tratados en relación con los macrófagos infectados no tratados en dos experimentos independientes. La estadística se realizó mediante ANOVA de dos vías con la prueba de comparación múltiple de Dunnet; *, p < 0,05; **, p < 0,01; , p < 0,001; , p < 0,0001 en comparación con el DMSO (control de disolventes). Los asteriscos siguen el mismo código de color para cada concentración que la leyenda del gráfico. Los símbolos redondos representan la Mycoload para cada ensayo. Las barras representan el promedio de dos experimentos independientes. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Ejemplo de un diseño de placa de 384 pocillos utilizado en la evaluación del factor Z'. Blanco: pozos en blanco (solo medio de crecimiento líquido); amarillo - control positivo (bacterias tratadas con un antibiótico); Rojo - control negativo (bacterias no tratadas). Haga clic aquí para descargar este archivo.
Figura complementaria 2: Ejemplo de un diseño de placa de 384 pocillos utilizado en el cribado de la biblioteca. Blanco: pozos en blanco (solo medio de crecimiento líquido); verde - control de solventes; rojo - control negativo (bacterias no tratadas); Azul: compuestos que se van a examinar. Haga clic aquí para descargar este archivo.
Figura complementaria 3: Ejemplo de un diseño de placa de 384 pocillos utilizado para la validación de impactos. Blanco: pozos en blanco (solo medio de crecimiento líquido); verde - control de disolventes (por duplicados); rojo - control negativo (bacterias no tratadas); Azul: compuestos que se van a cribar (por duplicado). Los colores difuminados representan diluciones en serie 1:2. Haga clic aquí para descargar este archivo.
Figura complementaria 4: Ejemplo de un diseño de placa de 96 pocillos utilizado para un ensayo de infección. Blanco - pozos en blanco (agua para evitar la evaporación); rojo - control negativo (macrófagos no tratados); amarillo - control positivo (macrófagos tratados con un antibiótico); verde - control de solventes; Azul: compuestos que se van a probar. Los colores difuminados representan diluciones en serie 1:2. Haga clic aquí para descargar este archivo.
Figura complementaria 5: Protocolo de análisis de imágenes. En este trabajo se utiliza un protocolo detallado de análisis de imágenes, que puede adaptarse a software de código abierto. Haga clic aquí para descargar este archivo.
Discusión
Este protocolo describe una línea de cribado de fármacos contra Mab utilizando en cepas desarrolladas internamente 10. Mediante el uso de robótica de manejo de líquidos, microscopía y análisis automatizados y cepas reporteras dobles, la viabilidad bacteriana se evalúa rápidamente mediante luminiscencia o fluorescencia sin agregar reactivos ni realizar pasos adicionales. Este enfoque reduce el tiempo y la variabilidad entre ensayos, lo cual es una ventaja significativa cuando se considera el propósito de los ensayos HTS.
Al analizar miles de compuestos, un fármaco eficaz debe identificarse fácilmente. Con este fin, el factor Z' se utiliza a menudo para medir el tamaño del efecto estadístico, inferiendo el rendimiento del ensayo. Si el factor Z' > 0,5, las condiciones ensayadas son óptimas para distinguir entre poblaciones tratadas y no tratadas13. Las condiciones ensayadas arrojaron factores Z' > 0,6 (Figura 2), demostrando estadísticamente que podrían aplicarse a una campaña de cribado. Este paso es crucial para garantizar la eficacia del cribado.
Así, se desarrolló un protocolo HTS para detectar la actividad antimicrobiana de miles de compuestos frente al Mab que crece planctónicamente. Dado que Mab es un patógeno facultativo intracelular, el protocolo diseñado también examina la actividad antimicrobiana intracelular contra la misma cepa bacteriana, una ventaja fundamental. Además, también se puede evaluar la toxicidad para las células huésped. Por lo tanto, se describe un enfoque de varios pasos para el cribado de fármacos contra Mab, utilizando diferentes configuraciones experimentales para evaluar la actividad antimicrobiana junto con la citotoxicidad, lo que aumenta las posibilidades de éxito. Como prueba de concepto, se seleccionó una biblioteca química compuesta por 1280 compuestos.
Se identificaron un total de treinta y tres aciertos (Figura 3). De ellos, tres aumentaron significativamente la viabilidad micobacteriana en cultivos líquidos (compuestos 30, 31 y 32; Figura 3 y Figura 4). Cabe señalar que estos compuestos pueden interferir con la emisión de luminiscencia sin afectar la viabilidad bacteriana. Cuando se probaron contra micobacterias internalizadas, estos compuestos mostraron actividad antimicrobiana (Figura 6), demostrando una mayor eficacia contra Mab después de la internalización de la célula huésped. Estos compuestos se identificaron como troleandomicina (30), espiramicina (32) y lincomicina (31; Tabla 1). Los dos primeros son macrólidos, una clase de antibióticos utilizados para tratar infecciones micobacterianas16, y el último es una lincosamida, un antibiótico con un mecanismo de acción similar a los macrólidos17. Se ha reportado que el Mab es particularmente resistente a otra lincosamida, la clindamicina, tanto en cultivos líquidos como sólidos18. No obstante, se han asociado propiedades inmunomoduladoras y antiinflamatorias con macrólidos19,20 y lincosamidas21, lo que podría explicar el aumento de la actividad antimicrobiana frente a micobacterias internalizadas (Figura 6).
De los treinta impactos restantes, once reducen en un >90% la viabilidad micobacteriana en todas las concentraciones (Figura 4). Dado que, un ensayo HTS típico tiene una tasa de aciertos esperada de ~1%22, el protocolo desarrollado está de acuerdo con lo que se observa habitualmente. No obstante, varios otros compuestos seguían activos, y veintiocho continuaron con el ensayo de infección.
De los compuestos identificados, cinco compuestos se consideraron tóxicos (Figura 5): daunorrubicina (4), doxorrubicina (5), epirrubicina (27), tiostreptón (6) y pamoato de pirvinio (28; Tabla 1). Los tres primeros son agentes antineoplásicos 23,24,25, por lo que, como era de esperar, son tóxicos para las células de mamíferos utilizadas en este ensayo. El pamoato de pirvinio se utilizó durante muchos años como un antihelmíntico eficaz; Sin embargo, desde 2004, también se ha relacionado con actividades antineoplásicas26. Por último, el tiostrepton es un oligopéptido utilizado a menudo en medicina veterinaria, nunca aprobado para su uso en humanos27. Se ha reportado la actividad de este fármaco contra las células de cáncer de mama28. No se evaluó la actividad intramacrofágica del tiostrepton debido a su toxicidad sobre los macrófagos derivados de la médula ósea (Figura 5). Sin embargo, se ha demostrado que el tiostrepton es efectivo a 5 μM en células THP-1 infectadas con Mab29. Los resultados reportados contra las bacterias planctónicas29 son similares a los obtenidos en este cribado, siendo el tiostrepton extremadamente potente (Figura 4).
La mayoría de los compuestos analizados para la actividad intramacrófica no mostraron potencial terapéutico (Figura 6). El crecimiento intracelular bacteriano es considerablemente diferente del de los cultivos planctónicos. En este último, siempre es posible el contacto directo entre bacterias y fármacos. En el primero, debido a la internalización por parte de las células huésped, varias membranas del huésped actúan como barreras físicas que los fármacos deben transponer para llegar al objetivo, lo que puede ayudar a explicar la disminución de la actividad antimicrobiana de la mayoría de los golpes. A la concentración más alta, 13,3 μM, tres compuestos mostraron suficiente potencia para ser estadísticamente diferentes del control de DMSO (Figura 6): cefdinir (21), gatifloxacino (26) y moxifloxacina (29; Tabla 1). Mientras que la moxifloxacina ya se utiliza como fármaco antituberculoso de segunda línea16, el cefdinir se utiliza habitualmente para tratar varias infecciones bacterianas del tracto respiratorio, como la neumonía30. Sin embargo, se ha reportado su actividad contra M. tuberculosis31 y Mab, mostrando un potente efecto sinérgico con un carbapenémico contra este último32. La gatifloxacina es una fluoroquinolona, y su actividad se ha reportado contra varias micobacterias en los últimos 33,34 años. Los tres compuestos más activos en este ensayo (Figura 6) fueron la roxitromicina (17), la claritromicina (22) y la rifabutina (25; Tabla 1), que son extremadamente potentes en todas las concentraciones. Los dos primeros son macrólidos, mientras que la rifabutina es una rifamicina, y ambas clases sirven como base para el tratamiento contra muchas infecciones micobacterianas16.
Este cribado se basa en equipos de manipulación de líquidos específicos y costosos para reducir la variabilidad entre ensayos. A pesar de ser extremadamente reproducible, el manipulador de líquidos no tiene una transferencia de compuestos sin contacto, como una acústica. Por lo tanto, ciertos compuestos adherentes pueden permanecer adheridos a los pines metálicos entre transferencias, trasladándose a los pozos siguientes, lo que resulta en falsos positivos durante la fase de cribado: esto fue lo que sucedió con el compuesto 33. Esta es la razón por la que la validación del cribado es tan importante para su éxito, ya que garantiza que ningún falso positivo pase a los siguientes pasos del proceso de cribado de fármacos. El ensayo de infección utiliza un sistema de imágenes de alto contenido con un modo confocal para obtener la mejor definición posible de las regiones bacterianas. No obstante, todavía es posible utilizarlo sin un modo confocal, aunque con una pérdida de definición y posiblemente dificultando la identificación de regiones bacterianas. Este protocolo aprovecha el software de análisis simple y fácil de usar integrado con el sistema de imágenes; sin embargo, el software de código abierto se puede utilizar siguiendo el protocolo (Figura complementaria 5). En última instancia, a pesar de no reutilizar fármacos para tratar las infecciones por Mab, estos resultados son extremadamente importantes porque validan el protocolo establecido, que puede ampliarse a bibliotecas más grandes. Es importante destacar que creemos que este protocolo se puede adaptar a cualquier bacteria, siempre que se disponga de lecturas fluorescentes o luminiscentes. Así, este trabajo contribuye significativamente al campo del descubrimiento de fármacos, proporcionando las herramientas necesarias para ayudar a combatir uno de los mayores problemas de salud pública, las bacterias resistentes a los antibióticos, y en particular un patógeno casi intratable, Mycobacterium abscessus.
Divulgaciones
Los autores declaran no tener ningún conflicto de intereses.
Agradecimientos
Este trabajo está financiado por fondos nacionales portugueses a través de FCT - Fundação para a Ciência e a Tecnologia, I.P, dentro del proyecto PTDC/BIA-MIC/3458/2020 (DOI: 10.54499/PTDC/BIA-MIC/3458/2020) y becas de doctorado 2021.07335.BD a GSO y UI/BD/150830/2021 a CMB; FWO - Fundación de Investigación de Flandes, beca n° 1S68720N; Iniciativa de Medicina Innovadora 2 Convocatoria 16 (IMI2-Convocatoria 16) propuesta RespiriTB bajo el acuerdo número 853903. Los autores agradecen el apoyo de la Plataforma Científica i3S BioSciences Screening, miembro de las infraestructuras nacionales PT-OPENSCREEN (NORTE-01-0145-FEDER-085468) y PPBI - Plataforma Portuguesa de Bioimagen (PPBI-POCI-01-0145-FEDER-022122).
Materiales
| Name | Company | Catalog Number | Comments |
| 10% Neutral buffered formalin | Bio Optica | 05-01005Q | fixation solution |
| 384-well black round bottom polypropylene not treated microplate | Corning | 3658 | White microplates can be used; polystyrene can be used |
| 50 TS Washer | BioTek | ||
| Breathe-Easy sealing membrane | Sigma-Aldrich | Z380059-1PAK | |
| cell::explorer | Revvity | equipment handling robot | |
| Clarithromycin | Sigma-Aldrich | A3487-100MG | Dissolved in DMSO to the desired concentration |
| DMSO | Fisher Scientific | D/4121/PB15 | Dilute in liquid growth medium to the desired concentration |
| Harmony 5.1 | Revvity | analysis software | |
| Heracell VIOS 160i CO2 Incubator, 165 L | Thermo Scientific | cell incubator | |
| JANUS with 4 Tip Arm and MDT Arm | Revvity | liquid handler | |
| KB 8182 | Termaks | incubator with agitation | |
| Libra S4+ | Biochrom | ||
| Multidrop Combi Reagent Dispenser | Thermo Scientific | ||
| Opera Phenix Plus | Revvity | imaging system | |
| PhenoPlate | Revvity | 6055302 | Necessary for high-content microscopes |
| Phosphate Buffer Saline (PBS) 1x | NA | NA | washing solution |
| Prestwick Chemical libraries | Commercial chemical library intended for drug repurposing | ||
| Shaking incubator 3031 | GFL | ||
| VICTOR Nivo | Revvity | ||
| Cell culture medium: | When needed, add 10% of supplement | ||
| DMEM | Gibco | 10938-025 | |
| Fetal Bovine Serum (FBS) | Biowest | S1810 | |
| HEPES Buffer Solution (1 M) | Gibco | 15630-056 | |
| LCCM | NA | NA | Conditioned media from L929 cell line that releases Macrophage colony stimulating factor (M-CSF); cell culture medium supplement |
| L-glutamin (200 mM) | Thermo Scientific | 25030024 | |
| Sodium Pyruvate 100 mM (100X) | Gibco | 11360-039 | |
| Infection washing solution | |||
| Apirogenic water | Multiple suppliers available | ||
| Hank's Balanced Salt Solution 10x | Gibco | 14060-040 | Dilute to 1x with apirogenic water |
| Permeabilising Solution | PBS with 0.1% Triton X100 | ||
| PBS 1x | NA | NA | |
| Triton X100 | Sigma-Aldrich | T8787 | Other suppliers available |
| Staining solution | PBS with DAPI (500 ng/mL) and HCS Far Red (10 ng/mL) | ||
| DAPI | Sigma-Aldrich | D9564 | |
| HCS Far Red | Invitrogen | H32721 | |
| PBS 1x | NA | NA | Multiple suppliers available |
| Liquid growth medium | When needed, add 10% supplement | ||
| Middlebrook 7H9 Broth | BD Difco | 271310 | |
| Tween80 | Sigma-Aldrich | P4780 | 0.05% final concentration. Other suppliers available |
| Liquid growth medium supplement: | |||
| Bovine Serum Albumin | Fisher Scientific | BP9702-100 | |
| D-Glucose anhydrous | Fisher Scientific | G/0450/60 |
Referencias
- Victoria, L., Gupta, A., Gomez, J. L., Robledo, J. Mycobacterium abscessus complex: A review of recent developments in an emerging pathogen. Front Cell Infect Microbiol. 11, 659997 (2021).
- Luthra, S., Rominski, A., Sander, P. The role of antibiotic-target-modifying and antibiotic-modifying enzymes in Mycobacterium abscessus drug resistance. Front Microbiol. 9, 2179 (2018).
- Nathavitharana, R. R., Strnad, L., Lederer, P. A., Shah, M., Hurtado, R. M. Top questions in the diagnosis and treatment of pulmonary M. abscessus disease. Open Forum Infect Dis. 6 (7), ofz221 (2019).
- Cristancho-Rojas, C., et al. Epidemiology of Mycobacterium abscessus. Clin Microbiol Infect. 30 (6), 712-717 (2024).
- Hua, Y., et al. Drug repositioning: Progress and challenges in drug discovery for various diseases. Eur J Med Chem. 234, 114239 (2022).
- Doan, T. L., Pollastri, M., Walters, M. A., Georg, G. I., Macor, J. E. The future of drug repositioning. Annual reports in medicinal chemistry. , 385-401 (2011).
- Bento, C. M., Gomes, M. S., Silva, T. Evolution of antibacterial drug screening methods: Current prospects for mycobacteria. Microorganisms. 9 (12), 2562 (2021).
- Attene-Ramos, M. S., Austin, C. P., Xia, M. High throughput screening. Encycl Toxicol. 3, 916-917 (2014).
- Gupta, R., Netherton, M., Byrd, T. F., Rohde, K. H. Reporter-based assays for high-throughput drug screening against Mycobacterium abscessus. Front Microbiol. 8, 2204 (2017).
- Bento Clara, M., et al. Characterization of novel double-reporter strains of Mycobacterium abscessus for drug discovery: A study in mScarlet. Microbiol Spectr. , e0036224 (2024).
- Kolbe, K., et al. Development and optimization of chromosomally-integrated fluorescent Mycobacterium tuberculosis reporter constructs. Front Microbiol. 11, 591866 (2020).
- Lopez-Roa, P., Esteban, J., Munoz-Egea, M. C. Updated review on the mechanisms of pathogenicity in Mycobacterium abscessus, a rapidly growing emerging pathogen. Microorganisms. 11 (1), 90 (2022).
- Bray, M. A., Carpenter, A., Markossian, S., et al. . Advanced assay development guidelines for image-based high content screening and analysis in Assay guidance manual. , (2004).
- Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard. CLSI document M07-A10. , (2015).
- Silva, T., et al. Lactoferricin peptides increase macrophages' capacity to kill Mycobacterium avium. mSphere. 2 (4), e00301-e00317 (2017).
- Bento, C. M., Gomes, M. S., Silva, T. Looking beyond typical treatments for atypical mycobacteria. Antibiotics (Basel). 9 (1), 18 (2020).
- Spizek, J., Rezanka, T. Lincosamides: Chemical structure, biosynthesis, mechanism of action, resistance, and applications. Biochem Pharmacol. 133, 20-28 (2017).
- Hurst-Hess, K. R., Rudra, P., Ghosh, P. Ribosome protection as a mechanism of lincosamide resistance in Mycobacterium abscessus. Antimicrob Agents Chemother. 65 (11), e0118421 (2021).
- Kang, J. K., Kang, H. K., Hyun, C. G. Anti-inflammatory effects of spiramycin in lps-activated raw 264.7 macrophages. Molecules. 27 (10), 3202 (2022).
- Zimmermann, P., Ziesenitz, V. C., Curtis, N., Ritz, N. The immunomodulatory effects of macrolides-a systematic review of the underlying mechanisms. Front Immunol. 9, 302 (2018).
- Pradhan, S., Madke, B., Kabra, P., Singh, A. L. Anti-inflammatory and immunomodulatory effects of antibiotics and their use in dermatology. Indian J Dermatol. 61 (5), 469-481 (2016).
- Volochnyuk, D. M., et al. Evolution of commercially available compounds for hts. Drug Discov Today. 24 (2), 390-402 (2019).
- Sritharan, S., Sivalingam, N. A comprehensive review on time-tested anticancer drug doxorubicin. Life Sci. 278, 119527 (2021).
- Arwanih, E. Y., Louisa, M., Rinaldi, I., Wanandi, S. I. Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: A literature review. Cureus. 14 (12), e33165 (2022).
- Khasraw, M., Bell, R., Dang, C. Epirubicin: Is it like doxorubicin in breast cancer? A clinical review. Breast. 21 (2), 142-149 (2012).
- Schultz, C. W., Nevler, A. Pyrvinium pamoate: Past, present, and future as an anti-cancer drug. Biomedicines. 10 (12), 3249 (2022).
- Bailly, C. The bacterial thiopeptide thiostrepton. An update of its mode of action, pharmacological properties and applications. Eur J Pharmacol. 914, 174661 (2022).
- Kwok, J. M., et al. Thiostrepton selectively targets breast cancer cells through inhibition of forkhead box m1 expression. Mol Cancer Ther. 7 (7), 2022-2032 (2008).
- Kim, T. H., et al. Thiostrepton: A novel therapeutic drug candidate for Mycobacterium abscessus infection. Molecules. 24 (24), 4511 (2019).
- Perry, C. M., Scott, L. J. Cefdinir: A review of its use in the management of mild-to-moderate bacterial infections. Drugs. 64 (13), 1433-1464 (2004).
- Ramon-Garcia, S., et al. Repurposing clinically approved cephalosporins for tuberculosis therapy. Sci Rep. 6, 34293 (2016).
- Kumar, P., et al. Mycobacterium abscessus l,d-transpeptidases are susceptible to inactivation by carbapenems and cephalosporins but not penicillins. Antimicrob Agents Chemother. 61 (10), e00866-e00917 (2017).
- Alvirez-Freites, E. J., Carter, J. L., Cynamon, M. H. In vitro and in vivo activities of gatifloxacin against Mycobacterium tuberculosis. Antimicrob Agents Chemother. 46 (4), 1022-1025 (2002).
- Braback, M., Riesbeck, K., Forsgren, A. Susceptibilities of Mycobacterium marinum to gatifloxacin, gemifloxacin, levofloxacin, linezolid, moxifloxacin, telithromycin, and quinupristin-dalfopristin (synercid) compared to its susceptibilities to reference macrolides and quinolones. Antimicrob Agents Chemother. 46 (4), 1114-1116 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados