Method Article
Desenvolvimento de ensaio para triagem de medicamentos de alto rendimento contra micobactérias
Neste Artigo
Resumo
Este estudo desenvolveu um ensaio para triagem de alto rendimento contra Mycobacterium abscessus com uma cepa repórter dupla recém-criada, usando fluorescência e luminescência para avaliar a viabilidade bacteriana rapidamente. Este protocolo será relevante para pesquisadores que pretendem rastrear novos medicamentos contra essa bactéria resistente a medicamentos.
Resumo
As infecções por Mycobacterium abscessus (Mab) são difíceis de tratar devido à alta resistência intrínseca aos medicamentos, comparável à tuberculose multirresistente. Os tratamentos são extremamente ineficazes e baseados em um regime multimedicamentoso, resultando em baixa adesão do paciente. Consequentemente, a comunidade científica é instada a identificar medicamentos novos e eficazes para tratar essas infecções. Uma das estratégias empregadas para esse fim é o reaproveitamento de medicamentos - o processo de identificação de novas oportunidades terapêuticas para medicamentos existentes no mercado, contornando o tempo necessário para estabelecer perfis farmacocinéticos e de segurança de novos medicamentos. Com a maioria dos estudos sobre o desenvolvimento de medicamentos contra Mab contando com métodos tradicionais e demorados, um ensaio para triagem de medicamentos de alto rendimento foi desenvolvido contra micobactérias usando uma cepa de Mab de repórter duplo desenvolvida internamente. Usando robótica de manuseio de líquidos, microscopia automatizada e análise, juntamente com cepas repórter duplas desenvolvidas internamente, a viabilidade bacteriana pode ser medida rapidamente usando duas leituras diferentes, luminescência e fluorescência, sem adicionar reagentes ou realizar etapas extras. Isso reduz o tempo e a variabilidade entre os ensaios, uma grande vantagem para triagens de alto rendimento. O protocolo descrito foi validado por meio da triagem de uma biblioteca de 1280 compostos. Os resultados obtidos foram corroborados pela literatura, com detecção eficiente de compostos ativos. Assim, este trabalho cumpriu o objetivo de fornecer ao campo uma nova ferramenta para ajudar a combater essa bactéria extremamente resistente a medicamentos.
Introdução
Mycobacterium abscessus (Mab) é um patógeno oportunista responsável por infecções pulmonares, especialmente em pessoas com fibrose cística e outras doenças pulmonares. As infecções causadas por Mab são notoriamente difíceis de tratar devido à requintada resistência intrínseca aos medicamentos, comparável à tuberculose multirresistente1. Os medicamentos disponíveis são amplamente ineficazes devido ao envelope celular micobacteriano altamente impermeável e a um genoma que codifica várias enzimas que desativam os antibióticos2. Assim, o tratamento inclui a combinação de vários medicamentos levando meses a anos. Esse regime desafiador de múltiplas drogas, combinado com a baixa adesão do paciente, resulta em uma taxa média de cura de 30% a 50%3. Além disso, a prevalência de infecções pulmonares causadas por micobactérias não tuberculosas aumentou nas últimas décadas, incluindo as causadas por Mab 1,4. Consequentemente, a comunidade científica está correndo para desenvolver novos compostos para tratar infecções por Mab.
Uma das estratégias perseguidas com esse objetivo é o reaproveitamento de medicamentos - o processo de identificação de novas oportunidades terapêuticas para medicamentos existentes. Isso contorna o maior desafio que acompanha o pipeline de descoberta e desenvolvimento de um novo medicamento - o tempo5. Esse conceito simples aproveita os perfis farmacocinéticos e de segurança já estabelecidos de vários medicamentos para reduzir os custos de desenvolvimento e encurtar o prazo necessário para levar um medicamento da bancada à beira do leito6. Assim, bibliotecas que combinam centenas a milhares de tais compostos foram compiladas, permitindo que os pesquisadores testem rapidamente a possibilidade de redirecionar medicamentos contra seu patógeno de interesse.
A maioria dos estudos sobre o desenvolvimento de medicamentos contra o Mab é baseada em um ensaio padrão-ouro, mas tradicional, que avalia a atividade in vitro de um composto contra micobactérias - contagem de unidades formadoras de colônias7. Apesar de sua precisão, esse procedimento é extremamente demorado, tornando-se rapidamente inviável quando alguém pretende testar bibliotecas contendo milhares de compostos. Para esse fim, as triagens de alto rendimento (HTS) foram integradas ao desenvolvimento de medicamentos - ensaios robustos que aproveitam a robótica e os dispositivos de manuseio de líquidos, permitindo que milhares de compostos sejam rapidamente rastreados em paralelo8. Isso normalmente é feito testando uma única concentração inicialmente, usando placas de microtitulação de formatos de 96, 384, 1536 ou 3456 poços, atuando como ponto de partida para identificar acertos e otimizá-los ainda mais no pipeline para serem usados clinicamente.
Os ensaios baseados em repórteres fornecem uma vantagem significativa para a robustez do HTS devido à sua simplicidade e sensibilidade em comparação com outros ensaios baseados em corantes e absorbâncias 7,9. No entanto, até onde sabemos, apenas alguns estudos otimizaram uma triagem de alto rendimento contra o Mab9.
Recentemente, nosso laboratório desenvolveu cepas repórter duplas capazes de emitir concomitantemente luminescência e fluorescência10. Mab operon_mScarlet é uma dessas variedades. É autoluminescente devido à expressão do operon LuxABCDE , que inclui uma luciferase bacteriana (pela expressão dos genes luxAB ) e um substrato aldeído de cadeia longa (pela expressão dos genes luxCDE ). Por outro lado, a leitura fluorescente é obtida através da expressão de uma proteína fluorescente vermelha recentemente desenvolvida, a mScarlet, que supera as proteínas eGFP e mCherry mais comumente usadas, fornecendo um sinal mais potente11. O uso dessa cepa nos permite avaliar a viabilidade bacteriana em cultura líquida, medindo o sinal luminescente em um leitor de microplacas sem adicionar reagentes ou realizar etapas adicionais. Em termos de detecção, a fluorescência intrínseca permite a visualização microscópica em células vivas ou fixas sem o uso de corantes ou anticorpos. Ter uma única cepa com ambas as leituras fornece aos pesquisadores uma vantagem significativa ao usá-la em ensaios HTS - a variabilidade reduzida entre ensaios com leituras diferentes, pois não há necessidade de trocar cepas com base na natureza do ensaio.
Assim, este trabalho desenvolveu um ensaio de alto rendimento contra Mab usando uma cepa repórter dupla projetada internamente (Figura 1). Isso permitiu uma avaliação rápida da atividade in vitro de 1280 medicamentos destinados ao reaproveitamento usando uma biblioteca comercial (ver Tabela de Materiais). Em primeiro lugar, as atividades foram avaliadas em ensaio de cultura em caldo usando luminescência e, em segundo lugar, usando macrófagos infectados com Mab aproveitando o sinal fluorescente, emulando melhor o processo de infecção visto in vivo12.
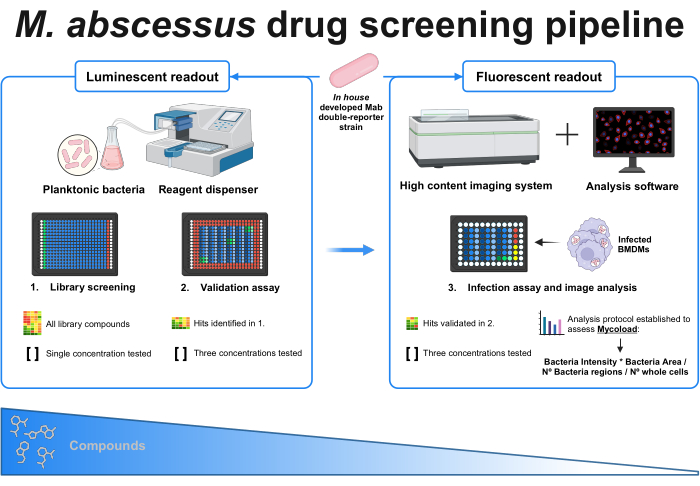
Figura 1: Resumo gráfico do protocolo estabelecido. O principal participante nesta triagem é uma cepa micobacteriana repórter dupla desenvolvida internamente , que é usada em todos os experimentos. Em primeiro lugar, usando um dispensador de reagentes e bactérias planctônicas, uma triagem inicial é realizada testando os compostos em uma única concentração. Os acertos identificados continuam para o ensaio de validação, onde três concentrações diferentes são testadas. Ambos os experimentos são feitos usando a leitura luminescente. Os acertos validados continuam para o ensaio de infecção, onde três concentrações diferentes também são testadas, e macrófagos derivados da medula óssea são infectados no MOI 1. Um sistema de imagem de alto conteúdo adquire o sinal fluorescente bacteriano e o software de análise é usado para avaliar a carga intracelular por meio da fórmula Mycoload. O número de compostos diminui à medida que a complexidade do ensaio aumenta. Criado com BioRender.com. Clique aqui para ver uma versão maior desta figura.
Protocolo
Todos os procedimentos em animais foram aprovados pela Comissão Local de Ética Animal do i3S e licenciados pela Direção-Geral de Alimentação e Veterinária (DGAV), seguindo a Diretiva do Conselho Europeu (2010/63/UE) e a lei portuguesa (DL 113/2013) para o bem-estar animal na experimentação.
1. Avaliação do fator Z'
NOTA: Para avaliar o desempenho de um ensaio HTS, vários parâmetros podem ser determinados. O parâmetro mais utilizado é o fator Z primo (fator Z'). Essa métrica mede o tamanho do efeito estatístico de um determinado experimento, indicando o quão bem separados estão os controles positivos e negativos. Se um fator Z' variar entre 0,5 e 1, a separação entre as duas populações é excelente, e essas condições podem ser usadas no ensaio HTS. Se Z' < 0,5, a separação é apenas marginal, não sendo recomendada a realização da triagem13. Assim, foi projetado um experimento usando metade de uma placa com bactérias não tratadas e a outra metade com um controle positivo para calcular o fator Z' (Figura Suplementar 1).
- 2-3 dias antes do ensaio, preparar pré-culturas de Mab inoculando bactérias num meio de cultura líquido (cerca de 1 x 107 UFC/ml) num balão Erlenmeyer. Deixar a bactéria crescer a 37 °C numa incubadora com agitação (90 rpm).
CUIDADO: Um gabinete de biossegurança é necessário ao trabalhar com culturas Mab. - Acompanhe o crescimento da pré-cultura do Mab por medição da densidade óptica a 600 nm usando um espectrofotômetro. Quando a cultura atingir a fase de crescimento exponencial, prosseguir para a etapa 1.3.
- Usando a pré-cultura exponencial, prepare uma suspensão bacteriana de aproximadamente 5 x 105 UFC/mL (correlacionada com os valores de densidade óptica)14 em meio de crescimento líquido.
- Preparar uma solução de controlo positivo da inibição do crescimento bacteriano num meio de crescimento líquido. Para este ensaio, o controle positivo foi claritromicina (10 mg/mL em DMSO) a 4 μg/mL. A claritromicina deve ser preparada 2x a concentração final desejada.
- Usando um dispensador de reagente de microplaca equipado com um pequeno, dispense 15 μL de meio de crescimento líquido para as colunas em branco e não tratadas de uma placa de polipropileno preto de 384 poços. Para as colunas em branco, dispensar mais 15 μL de meio de crescimento líquido.
NOTA: As colunas em branco evitam a evaporação dos poços internos a 37 °C. - Com o dispensador de reagentes de microplacas equipado com um pequeno, dispense 15 μL da solução de controle positivo preparada na etapa 1.4 para as colunas de controle positivo.
- Com um dispensador de reagente de microplaca equipado com um grande, dispense 15 μL da suspensão bacteriana preparada na etapa 1.3 para todas as colunas, exceto os brancos, diluindo tudo 1:2. O volume final de todos os 384 poços é de 30 μL.
NOTA: O dispensador de reagentes de microplacas pode ser equipado com um grande ou pequeno, diferindo no tamanho da tubulação (0.5 e 0.22 mm, respectivamente). É preferível usar um maior para evitar a agregação bacteriana no sistema de tubulação, mesmo que resulte em mais desperdício de líquido. No entanto, o mesmo pode ser usado em todo o ensaio. - Sele cada placa com um selador de placas que permita a troca gasosa contínua. Incubar a 37 °C numa incubadora sem agitação dentro de uma câmara húmida durante 48 h.
- Após 48 h, leia a luminescência emitida pelas bactérias em cada placa usando um leitor de placas com um tempo de integração de 1 s.
- Para determinar o fator Z', utilizar os valores obtidos na etapa 1.9 e calcular a média e o desvio padrão dos controles positivo (AVGP e DPP) e negativo (AVGN e DPN). Depois, aplique a seguinte fórmula:
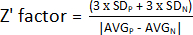
NOTA: 3 x DP é um limite comumente usado; no entanto, ele pode ser ajustado para melhor se adequar à triagem - aumentando ou diminuindo o fator pelo qual multiplicar o desvio padrão.
2. Triagem de acessos na biblioteca
NOTA: Os primeiros rastreios foram realizados contra bactérias que crescem em cultura líquida. A configuração foi projetada para placas de 384 poços com 30 μL de volume final. Cada placa continha poços em branco (apenas meio de crescimento líquido), controles negativos (bactérias não tratadas) e controle de solvente (bactérias mais solvente do composto - neste caso, DMSO; Figura suplementar 2). Os compostos ou solventes foram adicionados à placa a 2x a concentração final desejada e as bactérias foram adicionadas 1:1 (diluindo tudo pela metade).
- Preparar a suspensão bacteriana conforme descrito nos passos 1.1 a 1.3.
CUIDADO: Um gabinete de biossegurança é necessário ao trabalhar com culturas Mab. - Preparar uma solução de solvente num meio de cultura líquido. A quantidade de solvente deve ser 2x a concentração final desejada.
- Usando um dispensador de reagente de microplacas equipado com um pequeno, dispense 15 μL de meio de crescimento líquido em cada coluna de placas de polipropileno preto de 384 poços, exceto na coluna de controle de solvente. Para colunas em branco, dispense mais 15 μL de meio de crescimento líquido.
- Com o dispensador de reagente de microplacas equipado com um pequeno, dispense 15 μL da solução de solvente previamente preparada para a coluna de controle de solvente.
- Usando um manipulador de líquidos, transfira 200 nL dos compostos das placas de 96 poços de estoque (1 mM em DMSO) para as placas de 384 poços, exceto para os brancos, controle de solvente e colunas não tratadas. Os compostos estão agora em uma concentração de 13,3 μM.
- Com um dispensador de reagente de microplaca equipado com um grande, dispense 15 μL da suspensão bacteriana preparada na etapa 2.1 para todas as colunas, exceto os brancos, diluindo tudo 1:2. O volume final de todos os 384 poços é de 30 μL e cada composto está em 6,66 μM.
- Sele cada placa com um selador de placas que permita a troca gasosa contínua.
NOTA: O ensaio pode ser feito com tampas regulares; no entanto, há uma possibilidade maior de efeitos de borda mais fortes. - Incubar a 37 °C numa incubadora sem agitação dentro de uma câmara húmida durante 48 h.
- Após 48 h, leia a luminescência emitida pelas bactérias em cada placa usando um leitor de placas com um tempo de integração de 1 s.
3. Análise de dados de triagem de acertos
NOTA: Para identificar acertos com os dados de luminescência adquiridos, os valores devem ser normalizados para o solvente no qual os compostos são dissolvidos e, se ocorrer, para o efeito de borda verificado nas placas de 384 microtitulação.
- Para a normalização do solvente dos dados obtidos na etapa 2.9, divida as unidades de luz relativa (RLU) de cada poço pela RLU média dos poços de controle de solvente (na placa correspondente). Neste ensaio, um forte efeito de borda foi verificado no meio das placas de triagem; assim, decidiu-se normalizar todos os dados para ele. Para isso, o processo foi feito em duas etapas, conforme descrito a seguir.
- Crie uma máscara de efeito de borda dividindo os valores obtidos na etapa 3.1 para as posições correspondentes, excluindo acertos, em todas as placas (placa 1 - P1, placa 2 - P2, placa 3 - P3, placa 4 - P4). Por exemplo, para as posições A1 e A2, Mask_A1 = Média (P1_A1; P2_A1; P3_A1; P4_A1); Mask_A2 = Média (P1_A2; P2_A2; P4_A2) [P3_A2 foi excluído porque foi um sucesso].
- Divida os valores obtidos na etapa 3.1. por sua máscara de efeito de borda obtida na etapa 3.1.1. Por exemplo,
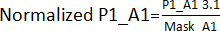 . Depois, calcule o AVG e o DP de cada placa para estabelecer um limite (AVG ± 3SD) para cada placa. Qualquer composto fora desse limite é considerado um acerto e continua para o ensaio de validação de acerto.
. Depois, calcule o AVG e o DP de cada placa para estabelecer um limite (AVG ± 3SD) para cada placa. Qualquer composto fora desse limite é considerado um acerto e continua para o ensaio de validação de acerto.
4. Ensaio de validação de acertos
NOTA: Depois de identificar os acertos, eles precisam ser validados usando diferentes concentrações. O ensaio de validação usa uma configuração semelhante com os mesmos controles, mas cada acerto será submetido a diluições seriais de 1:2. Para este ensaio, foram utilizadas três concentrações - 13,3 μM, 6,66 μM e 3,3 μM. Os controles de solvente conterão a mesma % de solvente que cada composto (Figura Suplementar 3).
- Preparar a suspensão bacteriana conforme descrito nos passos 1.1 a 1.3.
CUIDADO: Um gabinete de biossegurança é necessário ao trabalhar com culturas Mab. - Usando um dispensador de reagente de microplaca equipado com um pequeno, dispense 15 μL de meio de crescimento líquido para cada coluna de uma placa de polipropileno preto de 384 poços. Para poços em branco, dispense mais 15 μL de meio de crescimento líquido na primeira e na última coluna.
- Com o dispensador de reagente de microplaca, dispense mais 15 μL de meio de crescimento líquido nas colunas que contêm o [μM] mais alto de cada composto.
- A partir das placas de 96 poços, pipetar manualmente 0,8 μL de cada acerto (1 mM em DMSO) em seus respectivos poços da etapa 4.3, em duplicata. Os compostos estão agora em uma concentração de 26,6 μM.
- Pipetar manualmente 0,8 μL de solvente para os alvéolos de controlo em duplicado (o mesmo que o passo 4.4).
- Usando uma micropipeta multicanal, execute duas diluições seriais 1:2 para cada composto e controle de solvente. Rejeitar os 15 μl excedentários após a última diluição em série. Todos os poços agora têm 15 μL e as concentrações dos compostos são 26,6 μM, 13,3 μM e 6,66 μM.
- Utilizando um dispensador de reagentes em microplacas com uma grande, distribuir 15 μL da suspensão bacteriana preparada em 4.1 para todas as colunas (exceto os poços em branco), diluindo os compostos 1:2. Todos os poços têm um volume final de 30 μL e cada composto está em 13,3 μM, 6,66 μM e 3,3 μM.
- Sele a placa com um selador de placas que permita a troca gasosa contínua. Incubar a 37 °C numa incubadora sem agitação dentro de uma câmara húmida durante 48 h.
- Após 48 h, leia a luminescência emitida pelas bactérias em cada placa com um tempo de integração de 1 s usando um leitor de placas.
5. Ensaio de infecção
NOTA: Mab é um patógeno facultativo intracelular, portanto, a atividade antimicrobiana dos hits e a toxicidade do hospedeiro devem ser determinadas em ensaios de infecção. Para isso, macrófagos derivados da medula óssea (BMM) foram infectados com Mab e tratados com os hits validados na etapa 4 em diferentes concentrações. Cada ensaio incluiu branco (apenas água), controles negativos (macrófagos infectados não tratados), controles positivos (macrófagos infectados tratados com um antibiótico eficaz), controles de solvente (neste caso, DMSO) e os compostos a serem testados em três concentrações - 13,3 μM, 6,66 μM e 3,3 μM (Figura Suplementar 4).
- Derivar macrófagos da medula óssea murina de camundongos adultos do tipo selvagem (BALB/c ou C57BL/6), conforme descrito anteriormente15. Coloque as células em uma placa de fundo plano de 96 poços, preta e opticamente transparente a 2 x 105 células/mL, 200 μL por poço. Após 10 dias de diferenciação a 37 °C, 7% CO215, as células estão prontas para infectar e prosseguir com o tratamento.
NOTA: O protocolo para obtenção de macrófagos derivados da medula óssea está bem estabelecido15; no entanto, este protocolo de triagem pode ser adaptado a outras células, como RAW 264.7, THP-1 e macrófagos derivados de PBMC. - Preparar a suspensão bacteriana conforme descrito nos passos 1.1 a 1.3, ajustando a suspensão bacteriana em meio de cultura celular para uma infecção com uma multiplicidade de infecção (MOI) de 1.
CUIDADO: Um gabinete de biossegurança é necessário ao trabalhar com culturas Mab. - Aspirar cuidadosamente o meio de cultura de células suplementado dos alvéolos utilizando uma bomba de vácuo ligada a uma pipeta de vidro. Esta etapa precisa ser realizada lentamente e com facilidade para evitar o descolamento celular.
- Pipetar manualmente 75 μL da suspensão bacteriana preparada no passo 5.2 para cada alvéolo. Incubar a placa a 37 °C com CO2 a 7% durante 4 h numa incubadora de células.
- Prepare os compostos para o tratamento celular. Usando um dispensador de reagente de microplaca equipado com um pequeno, dispense em polipropileno, fundo redondo, placas de 96 poços, 110 μL de meio de cultura de células suplementado para colunas onde estarão os compostos ou o controle de solvente (Figura Suplementar 4).
- Usando um dispensador de reagente de microplaca, dispense mais 104 μL de meio de cultura de células suplementado nas colunas onde estará a maior concentração de composto ou solvente.
- Pipetar manualmente 5,9 μL dos compostos (1 mM em DMSO) ou solvente nos seus poços designados (colunas no passo 5.6). Os compostos estão agora em uma concentração de 26,6 μM.
- Usando uma micropipeta multicanal, execute duas diluições seriadas 1:2 para cada composto e solvente. Rejeitar os 110 μl excedidos, após a última diluição em série. Todos os poços agora têm 110 μL e os compostos estão em uma concentração de 26,6 μM, 13,3 μM e 6,66 μM.
- Usando um dispensador de reagente de microplaca, dispense 110 μL de meio de cultura de células suplementado em todas as colunas (excluindo poços em branco). Todos os poços agora têm 220 μL e os compostos estão em uma concentração de 13,3 μM, 6,66 μM e 3,3 μM.
- Preparar uma solução de claritromicina (controlo positivo) a 2 μg/ml num meio de cultura celular suplementado.
- Às 4 h após a etapa 5.4, usando uma lavadora de placas, lave 3x com 200 μL de solução de lavagem de infecção a placa infectada de 96 poços. Mantenha as velocidades de aspiração e distribuição o mais lentas possível para evitar qualquer descolamento celular.
- Após a aspiração final, transfira 200 μL dos compostos preparados nas etapas 5.5-5.9 para a placa de 96 poços contendo os macrófagos infectados.
- Transfira 200 μL de meio de cultura celular suplementado para os poços não tratados. Transferir 200 μL da solução de claritromicina preparada na etapa 5.10 para seus respectivos poços.
- Incubar a placa numa incubadora de células a 37 °C, com 7% de CO2, durante 48 h.
- Após 48 h, lave a placa infectada de 96 poços usando uma lavadora de placas com a solução de lavagem do composto (3x com 200 μL por poço).
- Usando uma micropipeta multicanal, dispense 200 μL da solução de fixação em todos os alvéolos e deixe agir por 10 minutos.
- Após a fixação, utilizando uma lavadora de placas, lavar as células com a solução de lavagem dos compostos (3x com 200 μL por alvéolo).
NOTA: O protocolo pode ser pausado aqui. As células podem ser armazenadas na solução de lavagem dos compostos a 4 °C durante vários dias ou semanas, se forem evitadas a contaminação e a secura celular. A solução de lavagem dos compostos deve ser aspirada antes da etapa 5.18. - Usando uma micropipeta multicanal, dispense 200 μL da solução permeabilizante em todos os poços e deixe agir por 15 min. Usando uma lavadora de placas, aspire a solução permeabilizante.
- Usando uma micropipeta multicanal, dispense manualmente 100 μL da solução de coloração e incube as placas em temperatura ambiente por 30 min.
- Usando uma lavadora de placas, lave as placas com a solução de lavagem dos compostos (3x com 200 μL por poço). No final, deixe as células coradas na solução de lavagem dos compostos.
- Filtre as placas em um sistema de imagem de alto conteúdo com as seguintes configurações:
Objetiva: 20x Ar/0,4NA (um poço de uma placa de 96 microtitulação, 57 campos de visão (FOV)
Modo: Confocal
Laser 405 nm para DAPI (núcleos)
Laser 561 nm para mScarlet (bactérias)
Laser 640 nm para HCS Cell Mask Deep Red (citoplasma)
Sem pilha z
NOTA: As células podem ser visualizadas com um sistema de imagem de campo amplo, diminuindo a definição. - Configure um robô de manuseio de equipamentos para aquisição de imagens durante a noite. Este braço robótico troca as placas no sistema de imagem de alto conteúdo sem intervenção humana.
NOTA: Isso não é obrigatório; As placas podem ser trocadas manualmente.
6. Análise de imagem
NOTA: A análise da imagem é realizada com o software de análise, que é o mesmo que o sistema de imagem de alto conteúdo usado para adquirir as imagens. Um pipeline de análise deve ser criado usando imagens de amostra (Figura Suplementar 5). Posteriormente, será aplicado a todo o poço (57 FOV) e conjunto de dados (todas as placas). O software de análise segue uma sequência lógica de etapas, começando com a segmentação das diferentes regiões de interesse (núcleos, citoplasma, células e bactérias), relacionando-as (por exemplo, bactérias dentro das células) e, em seguida, extraindo propriedades morfológicas e de intensidade (por exemplo, área, intensidade). As etapas essenciais da análise da imagem são descritas abaixo. O protocolo completo usado pode ser encontrado na Figura Suplementar 5.
- Segmente os núcleos usando o sinal DAPI, o citoplasma usando o sinal DeepRed e as bactérias usando o sinal mScarlet.
- Relacione os núcleos e a segmentação do citoplasma para criar uma máscara para as células.
- Remova todos os objetos de borda. Apenas as células que aparecem como cheias no FOV serão consideradas (células inteiras).
- Defina a população de saída Bactérias em células inteiras relacionando células inteiras e bactérias.
- Defina células inteiras infectadas como tendo mais de uma região bacteriana e células inteiras não infectadas como tendo menos de uma região bacteriana.
- Depois que todas as regiões de interesse forem segmentadas e as populações forem criadas, calcule e extraia suas propriedades. As propriedades a seguir são extraídas para cada poço.
- Para células inteiras/células inteiras infectadas/células inteiras não infectadas, extrato
Número de células (soma por poço)
Área [μm2] (Média + Desvio padrão por poço)
Relação largura-comprimento da célula (média + desvio padrão por poço) - Para bactérias em células inteiras, extrair
Número de regiões bacterianas identificadas (soma por poço)
Intensidade (soma por poço da intensidade média de cada região)
Área da região (soma por poço)
- Para células inteiras/células inteiras infectadas/células inteiras não infectadas, extrato
- Depois de exportar os dados e selecioná-los em uma planilha, quantifique a carga intracelular (Mycoload) de acordo com a seguinte fórmula:
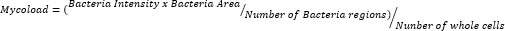
Resultados
Avaliação fatorial Z'
A Figura 2 mostra os dados de um dos dois experimentos realizados para calcular o fator Z'. O controle positivo foi claritromicina a 4 μg/mL. Ambos os experimentos produziram fatores Z 'de 0,64 e 0,62, o que significa que as condições e a leitura usadas para este ensaio podem ser aplicadas ao ensaio de triagem que se seguiu (Z '> 0,5). No entanto, o fator Z' foi calculado para todos os experimentos restantes (ensaio de infecção) para controlar o desempenho de cada experimento.
Como prova de conceito do ensaio HTS projetado, uma biblioteca de compostos destinados ao reaproveitamento de medicamentos foi testada. É composto por 1280 moléculas diversas e pequenas, 95% das quais são medicamentos aprovados pela FDA e EMA. Essas moléculas oferecem alta diversidade química e farmacológica.
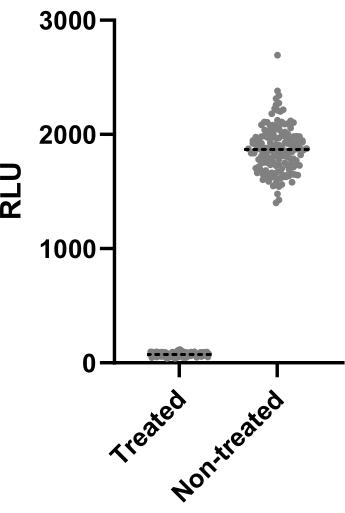
Figura 2: Resultados da avaliação fatorial Z'. Mab a 2,5 x 105 UFC/mL foi incubado com e sem claritromicina a 4 μg/mL por 48 h a 37 °C. Após o período de incubação, a luminescência foi medida para avaliar a viabilidade micobacteriana. O gráfico mostra os valores individuais de luminescência de micobactérias viáveis tratadas e não tratadas em um experimento independente. Clique aqui para ver uma versão maior desta figura.
Triagem de acertos na biblioteca
Todos os compostos foram testados a 6,66 μM contra Mab. Devido ao tamanho da biblioteca, os compostos foram divididos em quatro diferentes placas de microtitulação de 384 poços (Figura 3A-D). Os resultados foram normalizados tanto para o solvente quanto para o efeito de borda verificado.
Neste ensaio de triagem, trinta e três acertos foram identificados, trinta dos quais diminuíram significativamente a emissão de luminescência, reduzindo a viabilidade micobacteriana (Figura 3). Curiosamente, três compostos levaram a uma maior emissão de luminescência, possivelmente relacionada ao aumento do metabolismo ou proliferação bacteriana (Figura 3). Todos os 33 acertos foram levados para o ensaio de validação, incluindo os três compostos que aumentaram a luminescência, para testar se esse perfil seria mantido.
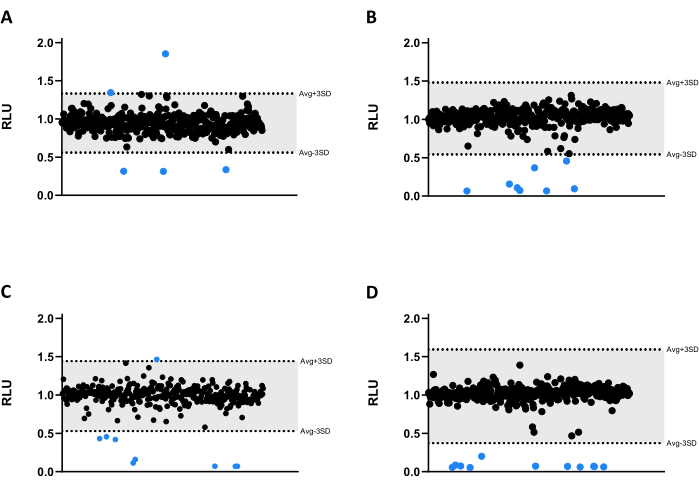
Figura 3: Resultados da triagem da biblioteca. (A-D) Mab a 2,5 x 105 UFCs/mL foi incubado com 1280 compostos a 6,66 μM por 48 h a 37 °C. Após a incubação, a luminescência foi medida para avaliar a viabilidade micobacteriana. Os gráficos mostram a RLU de um experimento independente, apresentada como unidades arbitrárias após a normalização do solvente e do efeito de borda. O AVG RLU e seu DP correspondente foram calculados para cada placa para determinar um limite (em cinza; AVG ± 3SD). Os símbolos redondos representam um composto testado. Em azul, qualquer composto fora desse limite é considerado um acerto. Clique aqui para ver uma versão maior desta figura.
Ensaio de validação de acertos
As concentrações usadas para este ensaio foram 13,3 μM, 6,66 μM e 3,3 μM. Cada composto foi testado em duplicata para aumentar a robustez.
É importante ressaltar que os compostos 30, 31 e 32, anteriormente associados a maior RLU (Figura 3A,C), mantiveram esse perfil, com mais de 200% de viabilidade micobacteriana alcançada em poços tratados com o composto 32 (Figura 4).
Os compostos 20 e 33 foram considerados inativos, pois a viabilidade micobacteriana é próxima de 100% em todas as concentrações testadas (Figura 4).
Os compostos 9 e 21 apresentaram inatividade semelhante nas duas concentrações mais baixas; no entanto, ao contrário do 20, eles permanecem ativos no máximo, 13,3 μM, com o composto 21 exibindo maior potência (Figura 4).
O composto 3 também apresenta inatividade na concentração mais baixa e uma perda de atividade em 6,66 μM, embora menor que o composto 21 (Figura 4). Apenas na menor concentração, os compostos 2, 7, 8, 10, 16, 18, 24 e 28 apresentaram menor potencial terapêutico. No entanto, 2, 10 e 28 ainda levam a <50% da viabilidade micobacteriana.
Globalmente, os quatorze compostos restantes foram ativos em todas as concentrações testadas.
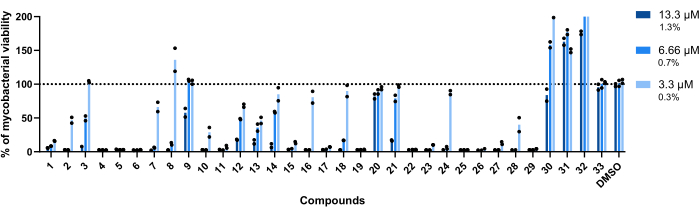
Figura 4: Resultados do ensaio de validação de acerto. Mab a 2,5 x 105 UFC/mL foi incubado com cada hit previamente identificado a 13,3 μM, 6,66 μM e 3,3 μM (1,3%, 0,7% e 0,3% de DMSO, respectivamente) por 48 h a 37 °C. Após o período de incubação, a luminescência foi medida para avaliar a viabilidade micobacteriana. O gráfico mostra as porcentagens de micobactérias viáveis tratadas em relação às micobactérias não tratadas de um experimento independente, com cada composto testado em duplicata. Os símbolos redondos representam as duplicatas de cada composto. As barras representam a média dessas duplicatas. Clique aqui para ver uma versão maior desta figura.
Ensaio de infecção
Dos trinta e três compostos testados, vinte e oito foram selecionados para o ensaio de infecção (Figura 4). Os compostos 7, 8, 9, 20 e 33 não foram selecionados para este ensaio. Enquanto 9, 20 e 33 foram deixados de fora por inatividade quando validados (Figura 4), os dois primeiros foram deixados de fora por motivos técnicos. No entanto, esses compostos foram identificados como rifampicina e linezolida, antibióticos já utilizados para tratar infecções por Mab12. Todos os compostos testados no ensaio de infecção foram identificados e estão listados na Tabela 1. A atividade antimicrobiana dos compostos contra macrófagos infectados com Mab foi avaliada usando a fluorescência intrínseca da bactéria como leitura.
| Composto | Nome | Composto | Nome |
| 1 | Sulfatiazol | 18 | Cefuroxima |
| 2 | Ciprofloxacina | 19 | Rifaximina |
| 3 | Cefotaxima | 21 | Cefdinir |
| 4 | Daunorrubicina | 22 | Claritromicina |
| 5 | Doxorrubicina | 23 | Besifloxacina |
| 6 | Thiostrepton | 24 | Levofloxacina |
| 10 | Amicacina | 25 | Rifabutina |
| 11 | Moxalactama | 26 | Gatifloxacina |
| 12 | Sulfametizol | 27 | Epirrubicina |
| 13 | Sulfamenometoxina | 28 | Pamoato de pirvinio |
| 14 | Cefoxitina | 29 | Moxifloxacina |
| 15 | Novobiocina | 30 | Troleandomicina |
| 16 | Cefmetazol | 31 | Lincomycin |
| 17 | Roxitromicina | 32 | Espiramicina |
Tabela 1: Lista dos compostos testados no ensaio de infecção. Os compostos validados na etapa 4 foram testados no ensaio de infecção (etapa 5).
A toxicidade dos compostos para macrófagos infectados com Mab foi o primeiro parâmetro avaliado. O limite estabelecido para considerar um composto como tóxico ou não tóxico foi de 85% de macrófagos viáveis (Figura 5). Dos vinte e oito compostos testados, 4, 5, 6, 27 e 28 foram considerados tóxicos. (Figura 5). Assim, esses cinco compostos foram excluídos da seguinte avaliação da atividade intramacrofágica.
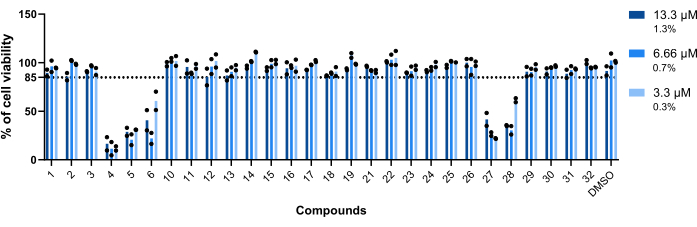
Figura 5: Toxicidade de hits para macrófagos infectados com Mab. BMMs de camundongos BALB/c foram infectados com Mab (MOI=1) e incubados com cada hit previamente identificado a 13,3 μM, 6,66 μM e 3,3 μM (1,3%, 0,7% e 0,3% de DMSO, respectivamente) por 48 h a 37 °C com 7% de CO2. As células foram visualizadas em um microscópio de fluorescência de triagem de alto conteúdo, usando o número de núcleos (corados com DAPI) para medir a viabilidade celular. O gráfico mostra as porcentagens de macrófagos infectados tratados viáveis em relação aos macrófagos infectados viáveis não tratados de dois experimentos independentes. Os símbolos redondos representam a viabilidade da célula para cada ensaio. As barras representam a média de dois experimentos independentes. Clique aqui para ver uma versão maior desta figura.
Para inferir a atividade intramacrofágica dos vinte e três compostos restantes contra o Mab intracelular (Figura 6), a fórmula Mycoload (explicada anteriormente na etapa 6) foi usada para obter a porcentagem de viabilidade micobacteriana normalizada para macrófagos infectados não tratados. A maioria dos compostos perdeu seu potencial terapêutico contra micobactérias internalizadas (Figura 6) em comparação com o ensaio de validação de acertos (Figura 4), pois o número de acertos reduziu de vinte e cinco para seis na concentração mais alta testada. Surpreendentemente, todos os três compostos que aumentaram a viabilidade do Mab planctônico em comparação com bactérias não tratadas (30, 31 e 32; Figura 3 e Figura 4) apresentaram atividade antimicobacteriana contra Mab intracelular, com os compostos 30 e 32 apresentando diferença estatisticamente significativa quando comparados ao DMSO, mesmo a 3,3 μM no caso do composto 32 (Figura 6). As micobactérias tratadas com os compostos 11 e 23 apresentaram viabilidade <50% a 13,3 μM; no entanto, isso não foi significativamente diferente do controle DMSO (Figura 6). Os compostos 21, 26 e 29 foram potentes o suficiente a 13,3 μM para justificar uma diferença estatística significativa, sendo 29 o mais ativo (Figura 6). Por fim, os compostos 17, 22 e 25 foram extremamente potentes em todas as concentrações testadas contra micobactérias internalizadas. Estes foram identificados como roxitromicina, claritromicina e rifabutina, respectivamente (Tabela 1). Dos três compostos, a claritromicina foi a mais ativa contra o Mab, com viabilidade micobacteriana nunca ultrapassando 10%, apresentando um valor de p <0,0001 em todas as concentrações testadas em comparação com o DMSO (Figura 6).
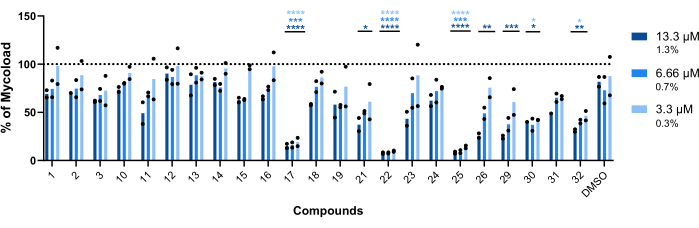
Figura 6: Atividade intramacrofágica de Hits contra macrófagos infectados com Mab. BMMs de camundongos BALB/c foram infectados com Mab (MOI=1) e incubados com cada acerto previamente identificado a 13,3, 6,66 e 3,3 μM (1,3, 0,7 e 0,3% de DMSO, respectivamente) por 48 h a 37 °C com 7% de CO2. As células foram visualizadas em um microscópio de fluorescência de triagem de alto conteúdo e o sinal fluorescente foi usado para calcular o Mycoload (consulte a etapa 6). O gráfico mostra as porcentagens de Mycoload encontradas em macrófagos infectados tratados em relação a macrófagos infectados não tratados em dois experimentos independentes. A estatística foi realizada por meio de ANOVA de duas vias com teste de comparações múltiplas de Dunnet; *, p < 0,05; **, p < 0,01; , p < 0,001; , p < 0,0001 em comparação com o DMSO (controle de solventes). Os asteriscos seguem o mesmo código de cores para cada concentração que a legenda do gráfico. Os símbolos redondos representam o Mycoload para cada ensaio. As barras representam a média de dois experimentos independentes. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Exemplo de um layout de placa de 384 poços usado na avaliação do fator Z'. White - poços em branco (apenas meio de crescimento líquido); amarelo - controle positivo (bactérias tratadas com antibiótico); vermelho - controle negativo (bactérias não tratadas). Clique aqui para baixar este arquivo.
Figura suplementar 2: Exemplo de um layout de placa de 384 poços usado na triagem da biblioteca. White - poços em branco (apenas meio de crescimento líquido); verde - controle de solventes; vermelho - controle negativo (bactérias não tratadas); azul - compostos a serem rastreados. Clique aqui para baixar este arquivo.
Figura suplementar 3: Exemplo de um layout de placa de 384 poços usado para validação de acerto. White - poços em branco (apenas meio de crescimento líquido); verde - controle de solventes (em duplicatas); vermelho - controle negativo (bactérias não tratadas); azul - compostos a serem rastreados (em duplicatas). As cores desbotadas representam diluições seriais 1:2. Clique aqui para baixar este arquivo.
Figura suplementar 4: Exemplo de um layout de placa de 96 poços usado para um ensaio de infecção. Branco - poços em branco (água para evitar a evaporação); vermelho - controle negativo (macrófagos não tratados); amarelo - controle positivo (macrófagos tratados com antibiótico); verde - controle de solventes; azul - compostos a serem testados. As cores desbotadas representam diluições seriais 1:2. Clique aqui para baixar este arquivo.
Figura suplementar 5: Protocolo de análise de imagem. Um protocolo detalhado de análise de imagens é utilizado neste trabalho, que pode ser adaptado para software de código aberto. Clique aqui para baixar este arquivo.
Discussão
Este protocolo descreve um pipeline de triagem de drogas contra Mab usando em cepas desenvolvidas internamente 10. Usando robótica de manuseio de líquidos, microscopia e análise automatizadas e cepas repórter duplas, a viabilidade bacteriana é avaliada rapidamente usando luminescência ou fluorescência sem adição de reagentes ou execução de etapas extras. Essa abordagem reduz o tempo e a variabilidade entre os ensaios, o que é uma vantagem significativa quando se considera o objetivo dos ensaios HTS.
Ao rastrear milhares de compostos, um medicamento eficaz deve ser facilmente identificado. Para tanto, o fator Z' é frequentemente utilizado para medir o tamanho do efeito estatístico, inferindo o desempenho do ensaio. Se o fator Z' > 0,5, as condições testadas são ótimas para distinguir entre populações tratadas e não tratadas13. As condições testadas produziram fatores Z' > 0,6 (Figura 2), comprovando estatisticamente que poderiam ser aplicados a uma campanha de rastreamento. Esta etapa é crucial para garantir a eficácia da triagem.
Assim, um protocolo HTS foi desenvolvido para detectar a atividade antimicrobiana de milhares de compostos contra o Mab crescendo plancttonicamente. Como o Mab é um patógeno facultativo intracelular, o protocolo projetado também rastreia a atividade antimicrobiana intracelular contra a mesma cepa bacteriana - uma vantagem crítica. Além disso, a toxicidade para as células hospedeiras também pode ser avaliada. Assim, uma abordagem de várias etapas para a triagem de medicamentos contra Mab é descrita, usando diferentes configurações experimentais para avaliar a atividade antimicrobiana junto com a citotoxicidade, aumentando as chances de sucesso. Como prova de conceito, foi selecionada uma biblioteca química composta por 1280 compostos.
Foram identificados trinta e três acertos (Figura 3). Destes, três aumentaram significativamente a viabilidade micobacteriana em culturas líquidas (compostos 30, 31 e 32; Figura 3 e Figura 4). Deve-se notar que esses compostos podem interferir na emissão de luminescência sem afetar a viabilidade bacteriana. Quando testados contra micobactérias internalizadas, esses compostos apresentaram atividade antimicrobiana (Figura 6), demonstrando maior eficácia contra Mab após a internalização da célula hospedeira. Esses compostos foram identificados como troleandomicina (30), espiramicina (32) e lincomicina (31; Tabela 1). Os dois primeiros são os macrolídeos, uma classe de antibióticos usados para tratar infecções micobacterianas16, e o segundo é a lincosamida, um antibiótico com mecanismo de ação semelhante aos macrolídeos17. Foi relatado que o Mab é particularmente resistente a outra lincosamida, a clindamicina, em culturas líquidas e sólidas18. No entanto, propriedades imunomoduladoras e anti-inflamatórias têm sido associadas a macrolídeos19,20 e lincosamidas21, o que poderia explicar o aumento da atividade antimicrobiana contra micobactérias internalizadas (Figura 6).
Dos trinta acertos restantes, onze reduzem em >90% a viabilidade micobacteriana em todas as concentrações (Figura 4). Dado que, um ensaio HTS típico tem uma taxa de acerto esperada de ~1%22, o protocolo desenvolvido está de acordo com o que geralmente é observado. No entanto, vários outros compostos ainda estavam ativos e vinte e oito continuaram para o ensaio de infecção.
Dos compostos identificados, cinco compostos foram considerados tóxicos (Figura 5) - daunorrubicina (4), doxorrubicina (5), epirrubicina (27), tiostreptônio (6) e pamoato de pirvinio (28; Tabela 1). Os três primeiros são agentes antineoplásicos 23,24,25, portanto, sem surpresa, são tóxicos para as células de mamíferos usadas neste ensaio. O pamoato de pirvinio foi usado por muitos anos como um anti-helmíntico eficaz; no entanto, desde 2004, também tem sido associada a atividades antineoplásicas26. Por fim, o tiostrepto é um oligopeptídeo frequentemente usado em medicina veterinária, nunca aprovado para uso em humanos27. A atividade dessa droga contra células de câncer de mama tem sidorelatada 28. A atividade intramacrofágica do tiostreptônio não foi avaliada devido à sua toxicidade em macrófagos derivados da medula óssea (Figura 5). No entanto, foi demonstrado que o tiostreptônio é eficaz a 5 μM em células THP-1 infectadas com Mab29. Os resultados relatados contra bactérias planctônicas29 são semelhantes aos obtidos nesta triagem, sendo o tiostrepton extremamente potente (Figura 4).
A maioria dos compostos rastreados para atividade intramacrofágica não apresentou potencial terapêutico (Figura 6). O crescimento intracelular bacteriano é consideravelmente diferente das culturas planctônicas. Neste último, o contato direto entre bactérias e drogas é sempre possível. No primeiro, devido à internalização pelas células hospedeiras, várias membranas do hospedeiro atuam como barreiras físicas que os fármacos precisam transpor para atingir o alvo, o que pode ajudar a explicar a diminuição da atividade antimicrobiana da maioria dos hits. Na concentração mais alta, 13,3 μM, três compostos apresentaram potência suficiente para serem estatisticamente diferentes do controle DMSO (Figura 6) - cefdinir (21), gatifloxacino (26) e moxifloxacino (29; Tabela 1). Enquanto o moxifloxacino já é usado como medicamento antituberculose de segunda linha16, o cefdinir é comumente usado para tratar várias infecções bacterianas do trato respiratório, como pneumonia30. No entanto, sua atividade contra M. tuberculosis31 e Mab foi relatada, exibindo um potente efeito sinérgico com um carbapenêmico contra este último32. A gatifloxacina é uma fluoroquinolona, e sua atividade foi relatada contra várias micobactérias no passado33,34. Os três compostos mais ativos neste ensaio (Figura 6) foram roxitromicina (17), claritromicina (22) e rifabutina (25; Tabela 1), que são extremamente potentes em todas as concentrações. Os dois primeiros são macrolídeos, enquanto a rifabutina é uma rifamicina, com ambas as classes servindo de base para o tratamento contra muitas infecções micobacterianas16.
Essa triagem depende de equipamentos específicos e caros de manuseio de líquidos para reduzir a variabilidade entre os ensaios. Apesar de ser extremamente reprodutível, o manipulador de líquidos não possui transferência de compostos sem contato, como a acústica. Assim, certos compostos aderentes podem permanecer presos aos pinos de metal entre as transferências, transportando-se para os poços seguintes, resultando em falsos positivos durante a fase de triagem - foi o que aconteceu com o composto 33. É por isso que validar a triagem é tão importante para seu sucesso, garantindo que nenhum falso positivo continue nas próximas etapas do pipeline de triagem de drogas. O ensaio de infecção utiliza um sistema de imagem de alto conteúdo com modo confocal para obter a melhor definição possível das regiões bacterianas. No entanto, ainda é possível usá-lo sem um modo confocal, embora com perda de definição e possivelmente dificultando a identificação de regiões bacterianas. Este protocolo aproveita o software de análise simples e fácil de usar integrado ao sistema de imagem; no entanto, o software de código aberto pode ser usado seguindo o protocolo (Figura Suplementar 5). Em última análise, apesar de não redirecionar medicamentos para tratar infecções por Mab, esses resultados são extremamente importantes porque validam o protocolo estabelecido, que pode ser expandido para bibliotecas maiores. É importante ressaltar que acreditamos que este protocolo pode ser adaptado a qualquer bactéria, desde que leituras fluorescentes ou luminescentes estejam disponíveis. Assim, este trabalho contribui significativamente para o campo da descoberta de fármacos, fornecendo as ferramentas necessárias para ajudar a combater um dos maiores problemas de saúde pública - bactérias resistentes a antibióticos - e particularmente um patógeno quase intratável - Mycobacterium abscessus.
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Este trabalho é financiado por fundos nacionais portugueses através da FCT - Fundação para a Ciência e a Tecnologia, I.P, no âmbito do projeto PTDC/BIA-MIC/3458/2020 (DOI: 10.54499/PTDC/BIA-MIC/3458/2020) e bolsas de doutoramento 2021.07335.BD ao GSO e UI/BD/150830/2021 ao CMB; FWO - Fundação de Pesquisa Flandres, concessão n° 1S68720N; Iniciativa de Medicina Inovadora 2 Chamada 16 (IMI2-Chamada 16) proposta RespiriTB sob o número de acordo 853903. Os autores agradecem o apoio da Plataforma Científica BioSciences Screening do i3S, membro das infraestruturas nacionais PT-OPENSCREEN (NORTE-01-0145-FEDER-085468) e PPBI - Plataforma Portuguesa de Bioimagem (PPBI-POCI-01-0145-FEDER-022122).
Materiais
| Name | Company | Catalog Number | Comments |
| 10% Neutral buffered formalin | Bio Optica | 05-01005Q | fixation solution |
| 384-well black round bottom polypropylene not treated microplate | Corning | 3658 | White microplates can be used; polystyrene can be used |
| 50 TS Washer | BioTek | ||
| Breathe-Easy sealing membrane | Sigma-Aldrich | Z380059-1PAK | |
| cell::explorer | Revvity | equipment handling robot | |
| Clarithromycin | Sigma-Aldrich | A3487-100MG | Dissolved in DMSO to the desired concentration |
| DMSO | Fisher Scientific | D/4121/PB15 | Dilute in liquid growth medium to the desired concentration |
| Harmony 5.1 | Revvity | analysis software | |
| Heracell VIOS 160i CO2 Incubator, 165 L | Thermo Scientific | cell incubator | |
| JANUS with 4 Tip Arm and MDT Arm | Revvity | liquid handler | |
| KB 8182 | Termaks | incubator with agitation | |
| Libra S4+ | Biochrom | ||
| Multidrop Combi Reagent Dispenser | Thermo Scientific | ||
| Opera Phenix Plus | Revvity | imaging system | |
| PhenoPlate | Revvity | 6055302 | Necessary for high-content microscopes |
| Phosphate Buffer Saline (PBS) 1x | NA | NA | washing solution |
| Prestwick Chemical libraries | Commercial chemical library intended for drug repurposing | ||
| Shaking incubator 3031 | GFL | ||
| VICTOR Nivo | Revvity | ||
| Cell culture medium: | When needed, add 10% of supplement | ||
| DMEM | Gibco | 10938-025 | |
| Fetal Bovine Serum (FBS) | Biowest | S1810 | |
| HEPES Buffer Solution (1 M) | Gibco | 15630-056 | |
| LCCM | NA | NA | Conditioned media from L929 cell line that releases Macrophage colony stimulating factor (M-CSF); cell culture medium supplement |
| L-glutamin (200 mM) | Thermo Scientific | 25030024 | |
| Sodium Pyruvate 100 mM (100X) | Gibco | 11360-039 | |
| Infection washing solution | |||
| Apirogenic water | Multiple suppliers available | ||
| Hank's Balanced Salt Solution 10x | Gibco | 14060-040 | Dilute to 1x with apirogenic water |
| Permeabilising Solution | PBS with 0.1% Triton X100 | ||
| PBS 1x | NA | NA | |
| Triton X100 | Sigma-Aldrich | T8787 | Other suppliers available |
| Staining solution | PBS with DAPI (500 ng/mL) and HCS Far Red (10 ng/mL) | ||
| DAPI | Sigma-Aldrich | D9564 | |
| HCS Far Red | Invitrogen | H32721 | |
| PBS 1x | NA | NA | Multiple suppliers available |
| Liquid growth medium | When needed, add 10% supplement | ||
| Middlebrook 7H9 Broth | BD Difco | 271310 | |
| Tween80 | Sigma-Aldrich | P4780 | 0.05% final concentration. Other suppliers available |
| Liquid growth medium supplement: | |||
| Bovine Serum Albumin | Fisher Scientific | BP9702-100 | |
| D-Glucose anhydrous | Fisher Scientific | G/0450/60 |
Referências
- Victoria, L., Gupta, A., Gomez, J. L., Robledo, J. Mycobacterium abscessus complex: A review of recent developments in an emerging pathogen. Front Cell Infect Microbiol. 11, 659997 (2021).
- Luthra, S., Rominski, A., Sander, P. The role of antibiotic-target-modifying and antibiotic-modifying enzymes in Mycobacterium abscessus drug resistance. Front Microbiol. 9, 2179 (2018).
- Nathavitharana, R. R., Strnad, L., Lederer, P. A., Shah, M., Hurtado, R. M. Top questions in the diagnosis and treatment of pulmonary M. abscessus disease. Open Forum Infect Dis. 6 (7), ofz221 (2019).
- Cristancho-Rojas, C., et al. Epidemiology of Mycobacterium abscessus. Clin Microbiol Infect. 30 (6), 712-717 (2024).
- Hua, Y., et al. Drug repositioning: Progress and challenges in drug discovery for various diseases. Eur J Med Chem. 234, 114239 (2022).
- Doan, T. L., Pollastri, M., Walters, M. A., Georg, G. I., Macor, J. E. The future of drug repositioning. Annual reports in medicinal chemistry. , 385-401 (2011).
- Bento, C. M., Gomes, M. S., Silva, T. Evolution of antibacterial drug screening methods: Current prospects for mycobacteria. Microorganisms. 9 (12), 2562 (2021).
- Attene-Ramos, M. S., Austin, C. P., Xia, M. High throughput screening. Encycl Toxicol. 3, 916-917 (2014).
- Gupta, R., Netherton, M., Byrd, T. F., Rohde, K. H. Reporter-based assays for high-throughput drug screening against Mycobacterium abscessus. Front Microbiol. 8, 2204 (2017).
- Bento Clara, M., et al. Characterization of novel double-reporter strains of Mycobacterium abscessus for drug discovery: A study in mScarlet. Microbiol Spectr. , e0036224 (2024).
- Kolbe, K., et al. Development and optimization of chromosomally-integrated fluorescent Mycobacterium tuberculosis reporter constructs. Front Microbiol. 11, 591866 (2020).
- Lopez-Roa, P., Esteban, J., Munoz-Egea, M. C. Updated review on the mechanisms of pathogenicity in Mycobacterium abscessus, a rapidly growing emerging pathogen. Microorganisms. 11 (1), 90 (2022).
- Bray, M. A., Carpenter, A., Markossian, S., et al. . Advanced assay development guidelines for image-based high content screening and analysis in Assay guidance manual. , (2004).
- Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard. CLSI document M07-A10. , (2015).
- Silva, T., et al. Lactoferricin peptides increase macrophages' capacity to kill Mycobacterium avium. mSphere. 2 (4), e00301-e00317 (2017).
- Bento, C. M., Gomes, M. S., Silva, T. Looking beyond typical treatments for atypical mycobacteria. Antibiotics (Basel). 9 (1), 18 (2020).
- Spizek, J., Rezanka, T. Lincosamides: Chemical structure, biosynthesis, mechanism of action, resistance, and applications. Biochem Pharmacol. 133, 20-28 (2017).
- Hurst-Hess, K. R., Rudra, P., Ghosh, P. Ribosome protection as a mechanism of lincosamide resistance in Mycobacterium abscessus. Antimicrob Agents Chemother. 65 (11), e0118421 (2021).
- Kang, J. K., Kang, H. K., Hyun, C. G. Anti-inflammatory effects of spiramycin in lps-activated raw 264.7 macrophages. Molecules. 27 (10), 3202 (2022).
- Zimmermann, P., Ziesenitz, V. C., Curtis, N., Ritz, N. The immunomodulatory effects of macrolides-a systematic review of the underlying mechanisms. Front Immunol. 9, 302 (2018).
- Pradhan, S., Madke, B., Kabra, P., Singh, A. L. Anti-inflammatory and immunomodulatory effects of antibiotics and their use in dermatology. Indian J Dermatol. 61 (5), 469-481 (2016).
- Volochnyuk, D. M., et al. Evolution of commercially available compounds for hts. Drug Discov Today. 24 (2), 390-402 (2019).
- Sritharan, S., Sivalingam, N. A comprehensive review on time-tested anticancer drug doxorubicin. Life Sci. 278, 119527 (2021).
- Arwanih, E. Y., Louisa, M., Rinaldi, I., Wanandi, S. I. Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: A literature review. Cureus. 14 (12), e33165 (2022).
- Khasraw, M., Bell, R., Dang, C. Epirubicin: Is it like doxorubicin in breast cancer? A clinical review. Breast. 21 (2), 142-149 (2012).
- Schultz, C. W., Nevler, A. Pyrvinium pamoate: Past, present, and future as an anti-cancer drug. Biomedicines. 10 (12), 3249 (2022).
- Bailly, C. The bacterial thiopeptide thiostrepton. An update of its mode of action, pharmacological properties and applications. Eur J Pharmacol. 914, 174661 (2022).
- Kwok, J. M., et al. Thiostrepton selectively targets breast cancer cells through inhibition of forkhead box m1 expression. Mol Cancer Ther. 7 (7), 2022-2032 (2008).
- Kim, T. H., et al. Thiostrepton: A novel therapeutic drug candidate for Mycobacterium abscessus infection. Molecules. 24 (24), 4511 (2019).
- Perry, C. M., Scott, L. J. Cefdinir: A review of its use in the management of mild-to-moderate bacterial infections. Drugs. 64 (13), 1433-1464 (2004).
- Ramon-Garcia, S., et al. Repurposing clinically approved cephalosporins for tuberculosis therapy. Sci Rep. 6, 34293 (2016).
- Kumar, P., et al. Mycobacterium abscessus l,d-transpeptidases are susceptible to inactivation by carbapenems and cephalosporins but not penicillins. Antimicrob Agents Chemother. 61 (10), e00866-e00917 (2017).
- Alvirez-Freites, E. J., Carter, J. L., Cynamon, M. H. In vitro and in vivo activities of gatifloxacin against Mycobacterium tuberculosis. Antimicrob Agents Chemother. 46 (4), 1022-1025 (2002).
- Braback, M., Riesbeck, K., Forsgren, A. Susceptibilities of Mycobacterium marinum to gatifloxacin, gemifloxacin, levofloxacin, linezolid, moxifloxacin, telithromycin, and quinupristin-dalfopristin (synercid) compared to its susceptibilities to reference macrolides and quinolones. Antimicrob Agents Chemother. 46 (4), 1114-1116 (2002).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados