Method Article
Sviluppo di saggi per lo screening di farmaci ad alto rendimento contro i micobatteri
In questo articolo
Riepilogo
Questo studio ha sviluppato un test per lo screening ad alto rendimento contro Mycobacterium abscessus con un ceppo doppio reporter di recente creazione, utilizzando la fluorescenza e la luminescenza per valutare rapidamente la vitalità batterica. Questo protocollo sarà rilevante per i ricercatori che mirano a selezionare nuovi farmaci contro questo batterio resistente ai farmaci.
Abstract
Le infezioni da Mycobacterium abscessus (Mab) sono difficili da trattare a causa dell'elevata resistenza intrinseca ai farmaci, paragonabile alla tubercolosi multiresistente. I trattamenti sono estremamente inefficaci e si basano su un regime multifarmaco, con conseguente bassa compliance del paziente. Di conseguenza, la comunità scientifica è sollecitata a identificare nuovi ed efficaci farmaci per trattare queste infezioni. Una delle strategie impiegate a tal fine è il riposizionamento dei farmaci, ovvero il processo di identificazione di nuove opportunità terapeutiche per i farmaci esistenti sul mercato, eludendo il tempo necessario per stabilire i profili farmacocinetici e di sicurezza dei nuovi farmaci. Poiché la maggior parte degli studi sullo sviluppo di farmaci contro il Mab si basa su metodi tradizionali e dispendiosi in termini di tempo, è stato sviluppato un test per lo screening dei farmaci ad alto rendimento contro i micobatteri utilizzando un ceppo di Mab a doppio reporter sviluppato internamente. Utilizzando la robotica per la manipolazione dei liquidi, la microscopia automatizzata e l'analisi, oltre a ceppi a doppio reporter sviluppati internamente, la vitalità batterica può essere misurata rapidamente utilizzando due diverse letture, luminescenza e fluorescenza, senza l'aggiunta di reagenti o l'esecuzione di ulteriori passaggi. Ciò riduce i tempi e la variabilità tra i saggi, un grande vantaggio per gli screening ad alta produttività. Il protocollo descritto è stato convalidato mediante lo screening di una libreria di 1280 composti. I risultati ottenuti sono stati corroborati dalla letteratura, con un'efficiente rilevazione dei composti attivi. Pertanto, questo lavoro ha raggiunto l'obiettivo di fornire al settore un nuovo strumento per aiutare a combattere questo batterio estremamente resistente ai farmaci.
Introduzione
Il Mycobacterium abscessus (Mab) è un patogeno opportunista responsabile delle infezioni polmonari, specialmente nelle persone con fibrosi cistica e altre malattie polmonari. Le infezioni causate da Mab sono notoriamente difficili da trattare a causa della squisita resistenza intrinseca ai farmaci, paragonabile alla tubercolosi multiresistente1. I farmaci disponibili sono in gran parte inefficaci a causa dell'involucro cellulare micobatterico altamente impermeabile e di un genoma che codifica diversi enzimi che disattivano gli antibiotici2. Pertanto, il trattamento include la combinazione di diversi farmaci che richiede mesi o anni. Questo impegnativo regime multifarmacologico, combinato con una bassa compliance del paziente, si traduce in un tasso medio di guarigione compreso tra il 30% e il 50%3. Inoltre, negli ultimi decenni è aumentata la prevalenza delle infezioni polmonari causate da micobatteri non tubercolari, comprese quelle causate da Mab 1,4. Di conseguenza, la comunità scientifica sta correndo per sviluppare nuovi composti per trattare le infezioni da Mab.
Una delle strategie perseguite a questo scopo è il riposizionamento dei farmaci, il processo di identificazione di nuove opportunità terapeutiche per i farmaci esistenti. In questo modo si elude la sfida più grande che accompagna la pipeline di scoperta e sviluppo di un nuovo farmaco: il tempo5. Questo semplice concetto sfrutta i profili farmacocinetici e di sicurezza già stabiliti di diversi farmaci per ridurre i costi di sviluppo e abbreviare i tempi necessari per portare un farmaco dal laboratorio al letto del paziente6. Pertanto, sono state compilate librerie che combinano centinaia o migliaia di tali composti, consentendo ai ricercatori di testare rapidamente la possibilità di riutilizzare i farmaci contro il loro agente patogeno di interesse.
La maggior parte degli studi sullo sviluppo di farmaci contro il Mab si basa su un test gold standard ma tradizionale che valuta l'attività in vitro di un composto contro i micobatteri - unità formanticolonie che contano 7. Nonostante la sua accuratezza, questa procedura richiede molto tempo, diventando rapidamente irrealizzabile quando qualcuno mira a testare librerie contenenti migliaia di composti. A tal fine, nello sviluppo dei farmaci sono stati integrati screening ad alto rendimento (HTS), saggi robusti che sfruttano la robotica e i dispositivi di manipolazione dei liquidi, consentendo di eseguire rapidamente lo screening di migliaia di composti in parallelo8. Questo viene in genere fatto testando inizialmente una singola concentrazione, utilizzando piastre per microtitolazione di formati a 96, 384, 1536 o 3456 pozzetti, fungendo da punto di partenza per identificare i risultati e ottimizzarli ulteriormente lungo la pipeline per essere utilizzati clinicamente.
I saggi basati su reporter offrono un vantaggio significativo per la robustezza degli HTS grazie alla loro semplicità e sensibilità rispetto ad altri saggi basati su coloranti e assorbanza 7,9. Tuttavia, per quanto ne sappiamo, solo pochi studi hanno ottimizzato uno screening ad alto rendimento contro Mab9.
Recentemente, il nostro laboratorio ha sviluppato ceppi reporter doppi in grado di emettere contemporaneamente luminescenza e fluorescenza10. Il Mab operon_mScarlet è una di queste varietà. È autoluminescente grazie all'espressione dell'operone LuxABCDE , che include una luciferasi batterica (per espressione dei geni luxAB ) e un substrato aldeidico a catena lunga (per espressione dei geni luxCDE ). D'altra parte, la lettura fluorescente è ottenuta attraverso l'espressione di una proteina fluorescente rossa di recente sviluppo, mScarlet, che supera le proteine eGFP e mCherry più comunemente utilizzate, fornendo un segnale più potente11. L'utilizzo di questo ceppo ci consente di valutare la vitalità batterica in coltura liquida misurando il segnale luminescente in un lettore di micropiastre senza aggiungere reagenti o eseguire ulteriori passaggi. In termini di rilevamento, la fluorescenza intrinseca consente la visualizzazione al microscopio in cellule vive o fissate senza l'uso di coloranti o anticorpi. Avere un singolo ceppo con entrambe le letture offre ai ricercatori un vantaggio significativo quando lo utilizzano nei saggi HTS: la ridotta variabilità tra i saggi con letture diverse, poiché non è necessario scambiare i ceppi in base alla natura del test.
Pertanto, questo lavoro ha sviluppato un test ad alto rendimento contro Mab utilizzando un ceppo doppio reporter progettato internamente (Figura 1). Ciò ha permesso una rapida valutazione dell'attività in vitro dei farmaci 1280 destinati al riposizionamento utilizzando una libreria commerciale (vedi Tabella dei materiali). In primo luogo, le attività sono state valutate in un saggio di coltura in brodo utilizzando la luminescenza e, in secondo luogo, utilizzando macrofagi infettati da Mab sfruttando il segnale fluorescente, emulando meglio il processo di infezione osservato nel vivo12.
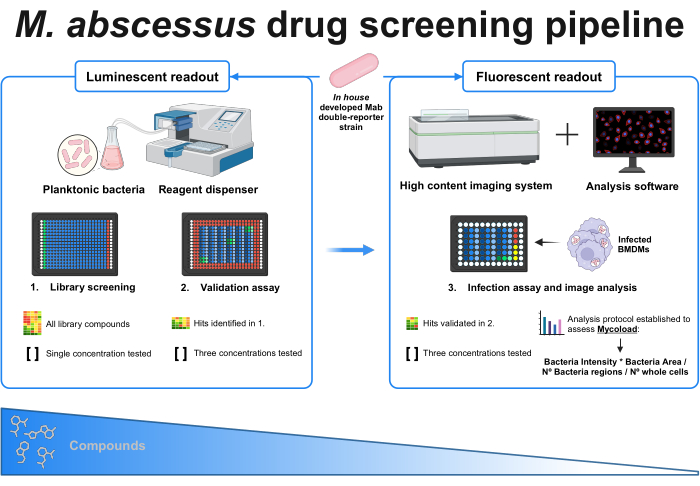
Figura 1: Abstract grafico del protocollo stabilito. L'attore chiave in questo screening è un ceppo micobatterico doppio reporter sviluppato internamente , che viene utilizzato in tutti gli esperimenti. In primo luogo, utilizzando un dosatore di reagenti e batteri planctonici, viene eseguito uno screening iniziale testando i composti a una singola concentrazione. I risultati identificati proseguono con il test di convalida, in cui vengono testate tre diverse concentrazioni. Entrambi gli esperimenti vengono eseguiti utilizzando la lettura luminescente. I risultati convalidati proseguono con il test di infezione, dove vengono testate anche tre diverse concentrazioni e i macrofagi derivati dal midollo osseo vengono infettati al MOI 1. Un sistema di imaging ad alto contenuto acquisisce il segnale fluorescente batterico e il software di analisi viene utilizzato per valutare il carico intracellulare attraverso la formula Mycoload. Il numero di composti si riduce all'aumentare della complessità del saggio. Creato con BioRender.com. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
Tutte le procedure per gli animali sono state approvate dal Comitato Etico Animale Locale di i3S e autorizzate dalla Direzione Generale per l'Alimentazione e la Veterinaria dell'Autorità Portoghese (DGAV), in conformità alla Direttiva del Consiglio Europeo (2010/63/UE) e alla legge portoghese (DL 113/2013) per il benessere degli animali sulla sperimentazione.
1. Valutazione del fattore Z'
NOTA: Per valutare le prestazioni di un test HTS, è possibile determinare diversi parametri. Il parametro più utilizzato è il fattore primo Z (fattore Z'). Questa metrica misura la dimensione dell'effetto statistico di un determinato esperimento, indicando quanto sono ben separati i controlli positivi e negativi. Se un fattore Z' è compreso tra 0,5 e 1, la separazione tra le due popolazioni è eccellente e queste condizioni possono essere utilizzate nel test HTS. Se Z' < 0,5, la separazione è solo marginale e non è consigliabile eseguire lo screening13. Pertanto, è stato progettato un esperimento utilizzando metà di una piastra con batteri non trattati e l'altra metà con un controllo positivo per calcolare il fattore Z' (Figura supplementare 1).
- A 2-3 giorni prima del saggio, preparare le pre-colture di Mab inoculando i batteri in un terreno di crescita liquido (circa 1 x 107 CFU/mL) in un matraccio di Erlenmeyer. Lasciare crescere i batteri a 37 °C in un'incubatrice con agitazione (90 giri/min).
ATTENZIONE: Quando si lavora con le colture Mab, è necessaria una cappa di biosicurezza. - Seguire la crescita della precoltura Mab misurando la densità ottica a 600 nm utilizzando uno spettrofotometro. Quando la cultura raggiunge la fase di crescita esponenziale, procedere con il passaggio 1.3.
- Utilizzando la precoltura esponenziale, preparare una sospensione batterica di circa 5 x 105 CFU/mL (correlata ai valori di densità ottica)14 in terreno di crescita liquido.
- Preparare una soluzione di controllo positivo dell'inibizione della crescita batterica in un terreno di coltura liquido. Per questo test, il controllo positivo era la claritromicina (10 mg/mL in DMSO) a 4 μg/mL. La claritromicina deve essere preparata 2 volte la concentrazione finale desiderata.
- Utilizzando un dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare 15 μl di terreno di crescita liquido nelle colonne bianche e non trattate di una piastra nera in polipropilene a 384 pozzetti. Per le colonne bianche, erogare altri 15 μL di terreno di coltura liquido.
NOTA: Le colonne cieche impediscono l'evaporazione dei pozzetti interni a 37 °C. - Con il dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare 15 μl della soluzione di controllo positivo preparata al punto 1.4 alle colonne di controllo positivo.
- Con un dispenser di reagenti per micropiastre dotato di una cassetta di grandi dimensioni, erogare 15 μl della sospensione batterica preparata nella fase 1.3 in tutte le colonne tranne i bianchi, diluendo il tutto 1:2. Il volume finale di tutti i 384 pozzetti è di 30 μL.
NOTA: Il dispenser di reagenti per micropiastre può essere dotato di una cassetta grande o piccola, che differisce per le dimensioni del tubo (0,5 e 0,22 mm, rispettivamente). È preferibile utilizzare una cassetta più grande per evitare l'aggregazione batterica nel sistema di tubi, anche se si verifica un maggiore spreco di liquidi. Tuttavia, la stessa cassetta può essere utilizzata per l'intero test. - Sigillare ogni piastra con un sigillante per piastre che consenta lo scambio continuo di gas. Incubare a 37 °C in un'incubatrice senza agitazione all'interno di una camera umida per 48 ore.
- Dopo 48 ore, leggere la luminescenza emessa dai batteri in ogni piastra utilizzando un lettore di piastre con un tempo di integrazione di 1 s.
- Per determinare il fattore Z', utilizzare i valori ottenuti al punto 1.9 e calcolare la media e la deviazione standard dei controlli positivi (AVGP e SDP) e negativi (AVGN e SDN). Successivamente, applica la seguente formula:
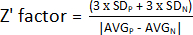
NOTA: 3 x SD è una soglia comunemente usata; Tuttavia, può essere regolato per adattarsi meglio allo screening, aumentando o diminuendo il fattore per il quale moltiplicare la deviazione standard.
2. Screening della libreria per i risultati
NOTA: I primi screening sono stati eseguiti contro i batteri che crescono in coltura liquida. La configurazione è stata progettata per piastre da 384 pozzetti con 30 μL di volume finale. Ogni piastra conteneva pozzetti vuoti (solo terreno di crescita liquido), controlli negativi (batteri non trattati) e controllo del solvente (batteri più solvente del composto - in questo caso, DMSO; Figura supplementare 2). I composti o il solvente sono stati aggiunti alla piastra a una dose 2 volte superiore alla concentrazione finale desiderata e i batteri sono stati aggiunti 1:1 (diluendo tutto a metà).
- Preparare la sospensione batterica come descritto nei passaggi da 1.1 a 1.3.
ATTENZIONE: Quando si lavora con le colture Mab, è necessaria una cappa di biosicurezza. - Preparare una soluzione di solvente in un terreno di coltura liquido. La quantità di solvente deve essere 2 volte la concentrazione finale desiderata.
- Utilizzando un dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare 15 μl di terreno di crescita liquido in ogni colonna di piastre in polipropilene nero a 384 pozzetti, ad eccezione della colonna di controllo del solvente. Per le colonne vuote, erogare altri 15 μl di terreno di crescita liquido.
- Con il dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare 15 μl della soluzione solvente precedentemente preparata nella colonna di controllo del solvente.
- Utilizzando un gestore di liquidi, trasferire 200 nL di composti dalle piastre stock a 96 pozzetti (1 mM in DMSO) alle piastre a 384 pozzetti, ad eccezione dei bianchi, del controllo del solvente e delle colonne non trattate. I composti sono ora a una concentrazione di 13,3 μM.
- Con un dispenser di reagenti per micropiastre dotato di una cassetta di grandi dimensioni, erogare 15 μl della sospensione batterica preparata al punto 2.1 in tutte le colonne tranne i bianchi, diluendo il tutto 1:2. Il volume finale di tutti i 384 pozzetti è di 30 μl e ogni composto è a 6,66 μM.
- Sigillare ogni piastra con un sigillante per piastre che consenta lo scambio continuo di gas.
NOTA: Il test può essere eseguito con coperchi normali; Tuttavia, c'è una maggiore possibilità di effetti di spigolo più forti. - Incubare a 37 °C in un'incubatrice senza agitazione all'interno di una camera umida per 48 ore.
- Dopo 48 ore, leggere la luminescenza emessa dai batteri in ogni piastra utilizzando un lettore di piastre con un tempo di integrazione di 1 s.
3. Analisi dei dati di screening dei risultati
NOTA: Per identificare i colpi con i dati di luminescenza acquisiti, i valori devono essere normalizzati al solvente in cui i composti sono disciolti e, se si verifica, all'effetto bordo verificato sulle piastre da 384 microtitolazioni.
- Per la normalizzazione con solvente dei dati ottenuti al punto 2.9, dividere le unità di luce relativa (RLU) di ciascun pozzetto per l'RLU medio dei pozzetti di controllo del solvente (nella piastra corrispondente). In questo test è stato verificato un forte effetto spigolo verso il centro delle piastre di vagliatura; Pertanto, si è deciso di normalizzare tutti i dati per IT. Per raggiungere questo obiettivo, il processo è stato eseguito in due fasi, come descritto di seguito.
- Create una maschera effetto bordo dividendo i valori ottenuti nel passaggio 3.1 per le posizioni corrispondenti, esclusi i colpi, su tutte le piastre (piastra 1 - P1, piastra 2 - P2, piastra 3 - P3, piastra 4 - P4). Ad esempio, per le posizioni A1 e A2, Mask_A1 = Media (P1_A1; P2_A1; P3_A1; P4_A1); Mask_A2 = Media (P1_A2; P2_A2; P4_A2) [P3_A2 è stato escluso perché è stato un successo].
- Dividi i valori ottenuti nel passaggio 3.1. dalla sua maschera effetto bordo ottenuta al punto 3.1.1. Ad esempio,
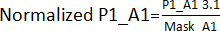 . Successivamente, calcola l'AVG e l'SD di ciascuna lastra per stabilire una soglia (AVG ± 3SD) per ciascuna lastra. Qualsiasi composto al di fuori di questa soglia è considerato un hit e passa al test di convalida dell'hit.
. Successivamente, calcola l'AVG e l'SD di ciascuna lastra per stabilire una soglia (AVG ± 3SD) per ciascuna lastra. Qualsiasi composto al di fuori di questa soglia è considerato un hit e passa al test di convalida dell'hit.
4. Saggio di convalida dei risultati
NOTA: Dopo aver identificato i risultati, questi devono essere convalidati utilizzando diverse concentrazioni. Il test di convalida utilizza una configurazione simile con gli stessi controlli, ma ogni hit sarà sottoposto a diluizioni seriali 1:2. Per questo test sono state utilizzate tre concentrazioni: 13,3 μM, 6,66 μM e 3,3 μM. I controlli del solvente conterranno la stessa % di solvente di ciascun pozzetto del composto (Figura supplementare 3).
- Preparare la sospensione batterica come descritto nei passaggi da 1.1 a 1.3.
ATTENZIONE: Quando si lavora con le colture Mab, è necessaria una cappa di biosicurezza. - Utilizzando un dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare 15 μl di terreno di crescita liquido su ogni colonna di una piastra nera in polipropilene a 384 pozzetti. Per i pozzetti bianchi, erogare altri 15 μL di terreno di crescita liquido nella prima e nell'ultima colonna.
- Con il dosatore di reagenti per micropiastre, erogare altri 15 μL di terreno di crescita liquido nelle colonne contenenti il massimo [μM] di ciascun composto.
- Dalle piastre a 96 pozzetti di serie, pipettare manualmente 0,8 μL di ciascun colpo (1 mM in DMSO) nei rispettivi pozzetti dal passaggio 4.3, in duplicato. I composti sono ora a una concentrazione di 26,6 μM.
- Pipettare manualmente 0,8 μL di solvente nei pozzetti di controllo in duplicato (come al passaggio 4.4).
- Utilizzando una micropipetta multicanale, eseguire due diluizioni seriali 1:2 per ciascun composto e il controllo del solvente. Scartare i 15 μl eccedenti dopo l'ultima diluizione seriale. Tutti i pozzetti hanno ora 15 μL e le concentrazioni dei composti sono 26,6 μM, 13,3 μM e 6,66 μM.
- Utilizzando un dispenser di reagenti per micropiastre con una cassetta grande, erogare 15 μl della sospensione batterica preparata in 4.1 in tutte le colonne (ad eccezione dei pozzetti bianchi), diluendo i composti 1:2. Tutti i pozzetti hanno un volume finale di 30 μL e ogni composto è a 13,3 μM, 6,66 μM e 3,3 μM.
- Sigillare la piastra con un sigillante per piastre che consenta lo scambio continuo di gas. Incubare a 37 °C in un'incubatrice senza agitazione all'interno di una camera umida per 48 ore.
- Dopo 48 ore, leggere la luminescenza emessa dai batteri in ogni piastra con un tempo di integrazione di 1 s utilizzando un lettore di piastre.
5. Dosaggio dell'infezione
NOTA: Il Mab è un patogeno facoltativo intracellulare, quindi l'attività antimicrobica dei colpi e la tossicità dell'ospite devono essere determinate nei test di infezione. Per questo, i macrofagi derivati dal midollo osseo (BMM) sono stati infettati con Mab e trattati con i risultati convalidati nella fase 4 a diverse concentrazioni. Ogni test includeva bianco (solo acqua), controlli negativi (macrofagi infetti non trattati), controlli positivi (macrofagi infetti trattati con un antibiotico efficace), controlli con solvente (in questo caso, DMSO) e i composti da testare in tre concentrazioni: 13,3 μM, 6,66 μM e 3,3 μM (Figura 4 supplementare).
- Derivare macrofagi dal midollo osseo murino di topi adulti wild-type (BALB/c o C57BL/6), come descritto in precedenza15. Piastra le cellule su una piastra a fondo piatto a 96 pozzetti, nera, otticamente trasparente, a 2 x 105 cellule/mL, 200 μL per pozzetto. Dopo 10 giorni di differenziazione a 37 °C, 7% CO215, le cellule sono pronte per infettare e procedere con il trattamento.
NOTA: Il protocollo per ottenere macrofagi derivati dal midollo osseo è ben consolidato15; tuttavia, questo protocollo di screening può essere adattato ad altre cellule, come RAW 264.7, THP-1 e macrofagi derivati da PBMC. - Preparare la sospensione batterica come descritto nei passaggi da 1.1 a 1.3, regolando la sospensione batterica nel terreno di coltura cellulare per un'infezione con una molteplicità di infezioni (MOI) di 1.
ATTENZIONE: Quando si lavora con le colture Mab, è necessaria una cappa di biosicurezza. - Aspirare con cura il terreno di coltura cellulare integrato dei pozzetti utilizzando una pompa a vuoto collegata a una pipetta di vetro. Questo passaggio deve essere eseguito lentamente e con facilità per prevenire il distacco delle cellule.
- Pipettare manualmente 75 μl della sospensione batterica preparata al punto 5.2 in ciascun pozzetto. Incubare la piastra a 37 °C con CO2 al 7% per 4 ore in un incubatore cellulare.
- Preparare i composti per il trattamento cellulare. Utilizzando un dispenser di reagenti per micropiastre dotato di una piccola cassetta, erogare su polipropilene, fondo rotondo, piastre a 96 pozzetti, 110 μl di terreno di coltura cellulare integrato su colonne in cui si troveranno i composti o il controllo del solvente (Figura 4 supplementare).
- Utilizzando un dispenser di reagenti per micropiastre, erogare altri 104 μl di terreno di coltura cellulare integrato nelle colonne in cui si troverà la massima concentrazione di composto o solvente.
- Pipettare manualmente 5,9 μl dei composti (1 mM in DMSO) o del solvente nei pozzetti designati (colonne al passaggio 5.6). I composti sono ora a una concentrazione di 26,6 μM.
- Utilizzando una micropipetta multicanale, eseguire due diluizioni seriali 1:2 per ciascun composto e solvente. Scartare i 110 μl eccedenti dopo l'ultima diluizione in serie. Tutti i pozzetti hanno ora 110 μL e i composti sono a una concentrazione di 26,6 μM, 13,3 μM e 6,66 μM.
- Utilizzando un dispenser di reagenti per micropiastre, erogare 110 μl di terreno di coltura cellulare integrato in tutte le colonne (esclusi i pozzetti bianchi). Tutti i pozzetti hanno ora 220 μL e i composti sono a una concentrazione di 13,3 μM, 6,66 μM e 3,3 μM.
- Preparare una soluzione di claritromicina (controllo positivo) a 2 μg/mL in un terreno di coltura cellulare integrato.
- A 4 ore dopo il passaggio 5.4, utilizzando una lavatrice per piastre, lavare 3 volte con 200 μL di soluzione di lavaggio per infezioni la piastra infetta a 96 pozzetti. Mantenere le velocità di aspirazione e dispensazione al minimo possibile per evitare il distacco delle cellule.
- Dopo l'aspirazione finale, trasferire 200 μl dei composti preparati nei passaggi 5.5-5.9 nella piastra a 96 pozzetti contenente i macrofagi infetti.
- Trasferire 200 μl di terreno di coltura cellulare integrato nei pozzetti non trattati. Trasferire 200 μl della soluzione di claritromicina preparata al punto 5.10 nei rispettivi pozzetti.
- Incubare la piastra in un incubatore cellulare a 37 °C, con CO2 al 7%, per 48 ore.
- Dopo 48 ore, lavare la piastra infetta a 96 pozzetti utilizzando una lavatrice per piastre con la soluzione di lavaggio del composto (3 volte con 200 μl per pozzetto).
- Utilizzando una micropipetta multicanale, erogare 200 μl della soluzione di fissazione in tutti i pozzetti e lasciarla agire per 10 minuti.
- Dopo il fissaggio, utilizzando una lavatrice per piastre, lavare le cellule con la soluzione di lavaggio dei composti (3 volte con 200 μL per pozzetto).
NOTA: Il protocollo può essere messo in pausa qui. Le celle possono essere conservate nella soluzione di lavaggio dei composti a 4 °C per diversi giorni o settimane se si evitano la contaminazione e la secchezza delle cellule. La soluzione di lavaggio dei composti deve essere aspirata prima del passaggio 5.18. - Utilizzando una micropipetta multicanale, erogare 200 μl di soluzione permeabilizzante in tutti i pozzetti e lasciarla agire per 15 minuti. Utilizzando una rondella per piastre, aspirare la soluzione permeabilizzante.
- Utilizzando una micropipetta multicanale, erogare manualmente 100 μl di soluzione colorante e incubare le piastre a temperatura ambiente per 30 minuti.
- Utilizzando una lavatrice per piastre, lavare le piastre con la soluzione di lavaggio dei composti (3 volte con 200 μl per pozzetto). Alla fine, lasciare le cellule colorate nella soluzione di lavaggio dei composti.
- Screening delle lastre in un sistema di imaging ad alto contenuto con le seguenti impostazioni:
Obiettivo: 20x Air/0.4NA (un pozzetto di una piastra da 96 microtitolazioni, 57 campi visivi (FOV)
Modalità: Confocale
Laser 405 nm per DAPI (nuclei)
Laser 561 nm per mScarlet (batteri)
Laser 640 nm per maschera cellulare HCS Deep Red (citoplasma)
Niente z-stack
NOTA: Le cellule possono essere visualizzate con un sistema di imaging a campo largo, diminuendo la definizione. - Configurare un robot di movimentazione delle apparecchiature per l'acquisizione delle immagini durante la notte. Questo braccio robotico sostituisce le piastre nel sistema di imaging ad alto contenuto senza l'intervento umano.
NOTA: Questo non è obbligatorio; Le piastre possono essere sostituite manualmente.
6. Analisi delle immagini
NOTA: L'analisi delle immagini viene eseguita con il software di analisi, che è lo stesso del sistema di imaging ad alto contenuto utilizzato per acquisire le immagini. È necessario creare una pipeline di analisi utilizzando immagini di esempio (Figura 5 supplementare). Successivamente, verrà applicato all'intero pozzetto (57 FOV) e al set di dati (tutte le piastre). Il software di analisi segue una sequenza logica di passaggi, a partire dalla segmentazione delle diverse regioni di interesse (nuclei, citoplasma, cellule e batteri), mettendole in relazione (ad esempio, batteri all'interno delle cellule) e quindi estraendo le proprietà morfologiche e di intensità (ad esempio, area, intensità). Di seguito sono descritte le fasi essenziali dell'analisi delle immagini. Il protocollo completo utilizzato è disponibile nella Figura 5 supplementare.
- Segmentare i nuclei utilizzando il segnale DAPI, il citoplasma utilizzando il segnale DeepRed e i batteri utilizzando il segnale mScarlet.
- Correlare i nuclei e la segmentazione del citoplasma per creare una maschera per le cellule.
- Rimuovi tutti gli oggetti bordo. Saranno prese in considerazione solo le cellule che appaiono come complete sul FOV (celle intere).
- Definire la popolazione di output Batteri in cellule intere mettendo in relazione cellule intere e batteri.
- Definisci le cellule intere infette come aventi più di una regione batterica e le cellule intere non infette come aventi meno di una regione batterica.
- Dopo che tutte le regioni di interesse sono state segmentate e le popolazioni sono state create, calcola ed estrai le loro proprietà. Per ogni pozzetto vengono estratte le seguenti proprietà.
- Per Cellule intere/ Cellule intere infette/ Cellule intere non infette, estratto
Numero di celle (Somma per pozzetto)
Area [μm2] (Media + Deviazione standard per pozzetto)
Rapporto larghezza-lunghezza delle celle (media + deviazione standard per pozzetto) - Per i batteri in cellule intere, estrarre
Numero di regioni batteriche identificate (Somma per pozzetto)
Intensità (Somma per pozzo dell'intensità media di ciascuna regione)
Superficie della regione (Somma per pozzo)
- Per Cellule intere/ Cellule intere infette/ Cellule intere non infette, estratto
- Dopo aver esportato i dati e averli curati in un foglio di calcolo, quantificare il carico intracellulare (Mycoload) secondo la seguente formula:
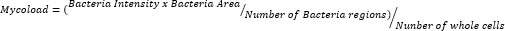
Risultati
Valutazione del fattore Z'
La Figura 2 illustra i dati di uno dei due esperimenti eseguiti per calcolare il fattore Z'. Il controllo positivo era la claritromicina a 4 μg/mL. Entrambi gli esperimenti hanno prodotto fattori Z' di 0,64 e 0,62, il che significa che le condizioni e la lettura utilizzate per questo test possono essere applicate al test di screening successivo (Z' > 0,5). Tuttavia, il fattore Z' è stato calcolato per tutti gli esperimenti rimanenti (test di infezione) per controllare le prestazioni di ciascun esperimento.
Come prova di concetto del test HTS progettato, è stata testata una libreria di composti destinati al riposizionamento dei farmaci. Comprende 1280 molecole diverse e di piccole dimensioni, il 95% delle quali sono farmaci approvati sia dalla FDA che dall'EMA. Queste molecole offrono un'elevata diversità chimica e farmacologica.
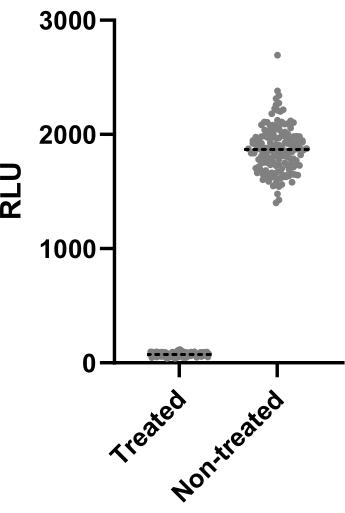
Figura 2: Risultati della valutazione del fattore Z'. Mab a 2,5 x 105 CFU/mL è stato incubato con e senza claritromicina a 4 μg/mL per 48 ore a 37 °C. Dopo il periodo di incubazione, la luminescenza è stata misurata per valutare la vitalità dei micobatteri. Il grafico mostra i singoli valori di luminescenza dei micobatteri vitali trattati e non trattati in un esperimento indipendente. Clicca qui per visualizzare una versione più grande di questa figura.
Screening della libreria per i risultati
Tutti i composti sono stati testati a 6,66 μM contro Mab. A causa delle dimensioni della libreria, i composti sono stati divisi in quattro diverse piastre per microtitolazione a 384 pozzetti (Figura 3A-D). I risultati sono stati normalizzati sia per il solvente che per l'effetto bordo verificato.
In questo test di screening sono stati identificati trentatré risultati, trenta dei quali hanno diminuito significativamente l'emissione di luminescenza, riducendo la vitalità dei micobatteri (Figura 3). È interessante notare che tre composti hanno portato a una maggiore emissione di luminescenza, probabilmente correlata all'aumento del metabolismo o della proliferazione batterica (Figura 3). Tutti i 33 risultati sono stati sottoposti al test di convalida, compresi i tre composti che aumentavano la luminescenza, per verificare se questo profilo sarebbe stato mantenuto.
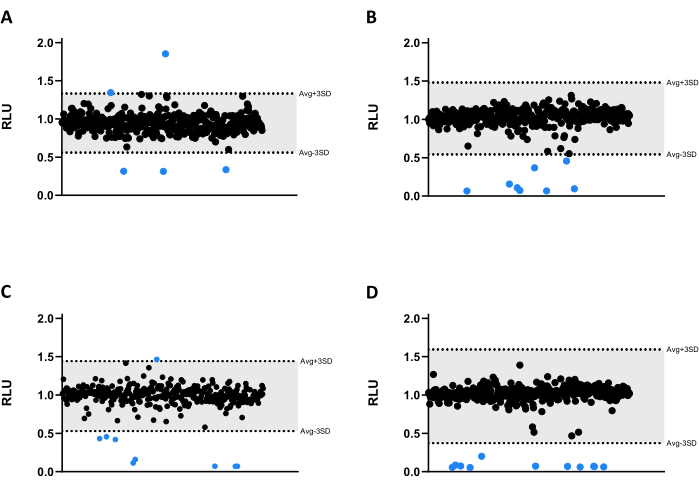
Figura 3: Risultati dello screening della libreria. (A-D) Il Mab a 2,5 x 105 CFU/mL è stato incubato con 1280 composti a 6,66 μM per 48 ore a 37 °C. Dopo l'incubazione, la luminescenza è stata misurata per valutare la vitalità dei micobatteri. I grafici mostrano l'RLU di un esperimento indipendente, presentato come unità arbitrarie dopo la normalizzazione dell'effetto solvente e dell'effetto spigolo. L'RLU AVG e la SD corrispondente sono stati calcolati per ciascuna targa per determinare una soglia (in grigio; AVG ± 3SD). I simboli rotondi rappresentano un composto testato. In blu, qualsiasi composto al di fuori di tale soglia è considerato un hit. Clicca qui per visualizzare una versione più grande di questa figura.
Saggio di convalida dei risultati
Le concentrazioni utilizzate per questo test sono state 13,3 μM, 6,66 μM e 3,3 μM. Ogni composto è stato testato in duplicato per aumentarne la robustezza.
È importante sottolineare che i composti 30, 31 e 32, precedentemente associati a RLU più elevati (Figura 3A, C), hanno mantenuto questo profilo, con oltre il 200% di vitalità micobatterica raggiunta nei pozzetti trattati con il composto 32 (Figura 4).
I composti 20 e 33 sono stati considerati inattivi, poiché la vitalità dei micobatteri è vicina al 100% in tutte le concentrazioni testate (Figura 4).
I composti 9 e 21 hanno mostrato un'inattività simile alle due concentrazioni più basse; tuttavia, contrariamente al 20, rimangono attivi al massimo, 13,3 μM, con il composto 21 che mostra una potenza maggiore (Figura 4).
Il composto 3 mostra anche inattività alla concentrazione più bassa e una perdita di attività a 6,66 μM, anche se inferiore al composto 21 (Figura 4). Solo nella concentrazione più bassa, i composti 2, 7, 8, 10, 16, 18, 24 e 28 hanno mostrato un potenziale terapeutico inferiore. Tuttavia, 2, 10 e 28 portano ancora al <50% della vitalità micobatterica.
A livello globale, i restanti quattordici composti erano attivi in tutte le concentrazioni testate.
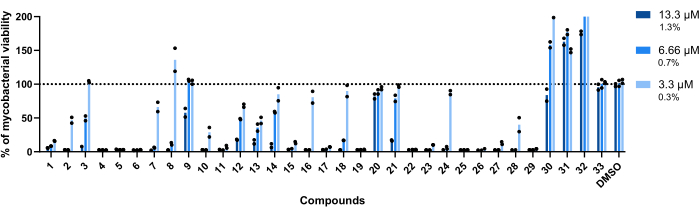
Figura 4: Risultati del test di convalida dell'hit. Il Mab a 2,5 x 105 CFU/mL è stato incubato con ogni hit precedentemente identificato a 13,3 μM, 6,66 μM e 3,3 μM (1,3%, 0,7% e 0,3% di DMSO, rispettivamente) per 48 ore a 37 °C. Dopo il periodo di incubazione, la luminescenza è stata misurata per valutare la vitalità dei micobatteri. Il grafico mostra le percentuali di micobatteri vitali trattati rispetto ai micobatteri non trattati di un esperimento indipendente, con ogni composto testato in duplicato. I simboli rotondi rappresentano i duplicati per ogni composto. Le barre rappresentano la media di questi duplicati. Clicca qui per visualizzare una versione più grande di questa figura.
Test di infezione
Su trentatré composti testati, ventotto sono stati selezionati per il test dell'infezione (Figura 4). I composti 7, 8, 9, 20 e 33 non sono stati selezionati per questo test. Mentre 9, 20 e 33 sono stati esclusi a causa della loro inattività al momento della convalida (Figura 4), i primi due sono stati esclusi per motivi tecnici. Ciononostante, questi composti sono stati identificati come rifampicina e linezolid, antibiotici già usati per trattare le infezioni da Mab12. Tutti i composti testati nel test dell'infezione sono stati identificati e sono elencati nella Tabella 1. L'attività antimicrobica dei composti contro i macrofagi infetti da Mab è stata valutata utilizzando la fluorescenza intrinseca dei batteri come lettura.
| Composto | Nome | Composto | Nome |
| 1 | Sulfatiazolo | 18 | Cefuroxima |
| 2 | Ciprofloxacina | 19 | Rifaximina |
| 3 | Cefotaxime | 21 | Cefdinir |
| 4 | Daunorubicina | 22 | Claritromicina |
| 5 | Doxorubicina | 23 | Besifloxacina |
| 6 | Thiostrepton | 24 | Levofloxacina |
| 10 | Amikacina | 25 | Rifabutina |
| 11 | Moxalattamo | 26 | Gatifloxacina |
| 12 | Sulfametismo | 27 | Epirubicina |
| 13 | Sulfamonometossina | 28 | Pirvinio pamoato |
| 14 | Cefoxitina | 29 | Moxifloxacina |
| 15 | Novobiocin | 30 | Troleandomicina |
| 16 | Cefmetazolo | 31 | Lincomicina |
| 17 | Roxitromicina | 32 | Spiramicina |
Tabella 1: Elenco dei composti testati nel test di infezione. I composti convalidati nella fase 4 sono stati testati nel test di infezione (fase 5).
La tossicità dei composti nei confronti dei macrofagi infetti da Mab è stato il primo parametro valutato. La soglia stabilita per ritenere un composto tossico o non tossico era l'85% dei macrofagi vitali (Figura 5). Dei ventotto composti testati, 4, 5, 6, 27 e 28 sono stati ritenuti tossici. (Figura 5). Pertanto, questi cinque composti sono stati esclusi dalla seguente valutazione dell'attività intramacrofagica.
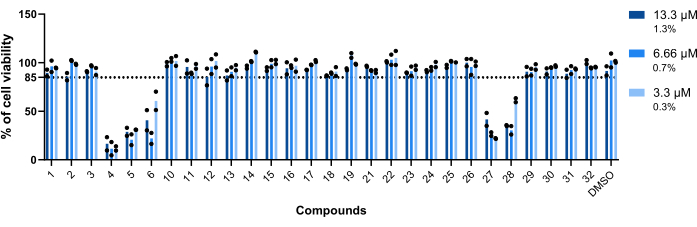
Figura 5: Tossicità dei colpi nei confronti dei macrofagi infetti da Mab. I BMM di topi BALB/c sono stati infettati con Mab (MOI=1) e incubati con ogni hit precedentemente identificato a 13,3 μM, 6,66 μM e 3,3 μM (1,3%, 0,7% e 0,3% di DMSO, rispettivamente) per 48 ore a 37 °C con 7% di CO2. Le cellule sono state visualizzate in un microscopio a fluorescenza ad alto contenuto, utilizzando il numero di nuclei (colorati con DAPI) per misurare la vitalità cellulare. Il grafico mostra le percentuali di macrofagi infetti vitali trattati rispetto ai macrofagi infetti vitali non trattati di due esperimenti indipendenti. I simboli rotondi rappresentano la vitalità della cellula per ogni test. Le barre rappresentano la media di due esperimenti indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
Per dedurre l'attività intramacrofagica dei restanti ventitré composti contro il Mab intracellulare (Figura 6), la formula Mycoload (precedentemente spiegata nella fase 6) è stata utilizzata per ottenere la percentuale di vitalità micobatterica normalizzata ai macrofagi infetti non trattati. La maggior parte dei composti ha perso il proprio potenziale terapeutico contro i micobatteri internalizzati (Figura 6) rispetto al test di convalida dei risultati (Figura 4), poiché il numero di risultati si è ridotto da venticinque a sei alla massima concentrazione testata. Sorprendentemente, tutti e tre i composti che hanno aumentato la vitalità del Mab planctonico rispetto ai batteri non trattati (30, 31 e 32; Figura 3 e Figura 4) hanno mostrato attività antimicobatterica contro il Mab intracellulare, con i composti 30 e 32 che presentano una differenza statistica significativa rispetto al DMSO, anche a 3,3 μM nel caso del composto 32 (Figura 6). I micobatteri trattati con i composti 11 e 23 hanno mostrato vitalità <50% a 13,3 μM; tuttavia, questo non era significativamente diverso dal controllo DMSO (Figura 6). I composti 21, 26 e 29 erano abbastanza potenti a 13,3 μM da giustificare una differenza statistica significativa, con 29 che era il più attivo (Figura 6). Infine, i composti 17, 22 e 25 sono risultati estremamente potenti in tutte le concentrazioni testate contro i micobatteri internalizzati. Questi sono stati identificati rispettivamente come roxitromicina, claritromicina e rifabutina (Tabella 1). Dei tre composti, la claritromicina è risultata la più attiva contro il Mab, con una vitalità micobatterica che non ha mai superato il 10%, presentando un valore p di <0,0001 in tutte le concentrazioni testate rispetto al DMSO (Figura 6).
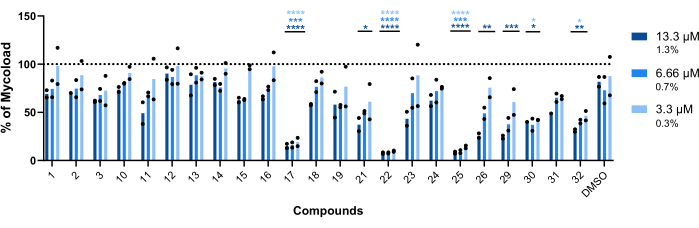
Figura 6: Attività intramacrofagica dei Hits contro i macrofagi infetti da Mab. I BMM dei topi BALB/c sono stati infettati con Mab (MOI=1) e incubati con ogni hit precedentemente identificato a 13,3, 6,66 e 3,3 μM (1,3, 0,7 e 0,3% di DMSO, rispettivamente) per 48 ore a 37 °C con CO2 al 7%. Le cellule sono state visualizzate in un microscopio a fluorescenza ad alto contenuto e il segnale fluorescente è stato utilizzato per calcolare il Mycoload (vedi passaggio 6). Il grafico mostra le percentuali di Mycoload riscontrate nei macrofagi infetti trattati rispetto ai macrofagi infetti non trattati in due esperimenti indipendenti. Le statistiche sono state eseguite utilizzando l'ANOVA a due vie con il test di confronto multiplo di Dunnet; *, p < 0,05; **, p < 0,01; , p < 0,001; , p < 0,0001 rispetto al DMSO (controllo del solvente). Gli asterischi seguono lo stesso codice colore per ogni concentrazione della legenda del grafico. I simboli rotondi rappresentano il Mycoload per ogni test. Le barre rappresentano la media di due esperimenti indipendenti. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 1 supplementare: Esempio di layout di una piastra a 384 pozzetti utilizzato nella valutazione del fattore Z'. Bianco - pozzetti vuoti (solo terreno di crescita liquido); giallo - controllo positivo (batteri trattati con un antibiotico); rosso - controllo negativo (batteri non trattati). Clicca qui per scaricare questo file.
Figura 2 supplementare: Esempio di layout di piastre a 384 pozzetti utilizzato nello screening della libreria. Bianco - pozzetti vuoti (solo terreno di crescita liquido); verde - controllo dei solventi; rosso - controllo negativo (batteri non trattati); blu - composti da vagliare. Clicca qui per scaricare questo file.
Figura 3 supplementare: Esempio di layout di piastra a 384 pozzetti utilizzato per la convalida dell'hit. Bianco - pozzetti vuoti (solo terreno di crescita liquido); verde - controllo del solvente (in duplicati); rosso - controllo negativo (batteri non trattati); blu - composti da vagliare (in duplicati). I colori sbiaditi rappresentano diluizioni seriali 1:2. Clicca qui per scaricare questo file.
Figura 4 supplementare: Esempio di layout di piastra a 96 pozzetti utilizzato per un test di infezione. Bianco - pozzetti vuoti (acqua per prevenire l'evaporazione); rosso - controllo negativo (macrofagi non trattati); giallo - controllo positivo (macrofagi trattati con un antibiotico); verde - controllo dei solventi; blu - composti da testare. I colori sbiaditi rappresentano diluizioni seriali 1:2. Clicca qui per scaricare questo file.
Figura supplementare 5: Protocollo di analisi delle immagini. In questo lavoro viene utilizzato un protocollo dettagliato di analisi delle immagini, che può essere adattato a software open source. Clicca qui per scaricare questo file.
Discussione
Questo protocollo descrive una pipeline di screening farmacologico contro Mab utilizzando ceppi sviluppati internamente 10. Utilizzando la robotica per la manipolazione dei liquidi, la microscopia e l'analisi automatizzate e i ceppi a doppio reporter, la vitalità batterica viene valutata rapidamente utilizzando la luminescenza o la fluorescenza senza l'aggiunta di reagenti o l'esecuzione di passaggi aggiuntivi. Questo approccio riduce il tempo e la variabilità tra i saggi, il che rappresenta un vantaggio significativo se si considera lo scopo dei saggi HTS.
Quando si esaminano migliaia di composti, un farmaco efficace deve essere facilmente identificato. A tal fine, il fattore Z' viene spesso utilizzato per misurare la dimensione statistica dell'effetto, deducendo le prestazioni del test. Se il fattore Z' > 0,5, le condizioni testate sono ottimali per distinguere tra popolazioni trattate e non trattate13. Le condizioni testate hanno prodotto fattori Z' > 0,6 (Figura 2), dimostrando statisticamente che potrebbero essere applicati a una campagna di screening. Questo passaggio è fondamentale per garantire l'efficacia dello screening.
Pertanto, è stato sviluppato un protocollo HTS per rilevare l'attività antimicrobica di migliaia di composti contro il Mab che cresce planctonicamente. Poiché il Mab è un patogeno facoltativo intracellulare, il protocollo progettato esamina anche l'attività antimicrobica intracellulare contro lo stesso ceppo batterico, un vantaggio fondamentale. Inoltre, può essere valutata anche la tossicità nei confronti delle cellule ospiti. Pertanto, viene descritto un approccio in più fasi allo screening farmacologico contro Mab, utilizzando diverse configurazioni sperimentali per valutare l'attività antimicrobica insieme alla citotossicità, aumentando le possibilità di successo. Come prova di concetto, è stata vagliata una libreria chimica comprendente 1280 composti.
Sono stati identificati un totale di trentatré riscontri positivi (Figura 3). Di questi, tre hanno aumentato significativamente la vitalità micobatterica nelle colture liquide (composti 30, 31 e 32; Figura 3 e Figura 4). Va notato che questi composti possono interferire con l'emissione di luminescenza senza influire sulla vitalità batterica. Quando testati contro micobatteri internalizzati, questi composti hanno mostrato attività antimicrobica (Figura 6), dimostrando una maggiore efficacia contro Mab dopo l'internalizzazione della cellula ospite. Questi composti sono stati identificati come troleandomicina (30), spiramicina (32) e lincomicina (31; Tabella 1). I primi due sono macrolidi, una classe di antibiotici usati per trattare le infezioni micobatteriche16, e il secondo è una lincosamide, un antibiotico con un meccanismo d'azione simile ai macrolidi17. È stato riportato che il Mab è particolarmente resistente a un'altra lincosamide, la clindamicina, sia in colture liquide che solide18. Tuttavia, proprietà immunomodulanti e antinfiammatorie sono state associate ai macrolidi19,20 e ai lincosamidi21, il che potrebbe spiegare l'aumentata attività antimicrobica contro i micobatteri internalizzati (Figura 6).
Dei restanti trenta risultati, undici riducono del >90% la vitalità dei micobatteri in tutte le concentrazioni (Figura 4). Dato che un tipico test HTS ha un tasso di successo previsto di ~1%22, il protocollo sviluppato è in accordo con ciò che viene solitamente osservato. Ciononostante, molti altri composti erano ancora attivi e ventotto sono passati al test dell'infezione.
Dei composti identificati, cinque sono stati ritenuti tossici (Figura 5): daunorubicina (4), doxorubicina (5), epirubicina (27), tiostrepton (6) e pirvinio pamoato (28; Tabella 1). I primi tre sono agenti antineoplastici 23,24,25, quindi non sorprende che siano tossici per le cellule di mammifero utilizzate in questo test. Il pirvinio pamoato è stato usato per molti anni come efficace antielmintico; Tuttavia, dal 2004, è stato collegato anche alle attività antineoplastiche26. Infine, il tiostrepton è un oligopeptide spesso utilizzato in medicina veterinaria, mai approvato per l'uso nell'uomo27. L'attività di questo farmaco contro le cellule del cancro al seno è stata riportata28. L'attività intramacrofagica di thiostrepton non è stata valutata a causa della sua tossicità sui macrofagi derivati dal midollo osseo (Figura 5). Tuttavia, è stato dimostrato che il tiostrepton è efficace a 5 μM sulle cellule THP-1 infettate da Mab29. I risultati riportati contro i batteri planctonici29 sono simili a quelli ottenuti in questo screening, con il tiostrepton estremamente potente (Figura 4).
La maggior parte dei composti sottoposti a screening per l'attività intramacrofagica non ha mostrato alcun potenziale terapeutico (Figura 6). La crescita intracellulare batterica è notevolmente diversa dalle colture planctoniche. In quest'ultimo caso, il contatto diretto tra batteri e farmaci è sempre possibile. Nel primo, a causa dell'internalizzazione da parte delle cellule ospiti, diverse membrane dell'ospite fungono da barriere fisiche che i farmaci devono trasporre per raggiungere il bersaglio, il che può aiutare a spiegare la diminuzione dell'attività antimicrobica della maggior parte dei colpi. Alla concentrazione più alta, 13,3 μM, tre composti hanno mostrato una potenza sufficiente per essere statisticamente diversi dal controllo DMSO (Figura 6) - cefdinir (21), gatifloxacina (26) e moxifloxacina (29; Tabella 1). Mentre la moxifloxacina è già utilizzata come farmaco antitubercolare di seconda linea16, il cefdinir è comunemente usato per trattare diverse infezioni batteriche del tratto respiratorio, come la polmonite30. Tuttavia, è stata riportata la sua attività contro M. tuberculosis31 e Mab, mostrando un potente effetto sinergico con un carbapenema contro quest'ultimo32. La gatifloxacina è un fluorochinolone e la sua attività è stata riportata contro diversi micobatteri negli ultimi 33,34. I tre composti più attivi in questo test (Figura 6) erano la roxitromicina (17), la claritromicina (22) e la rifabutina (25; Tabella 1), che sono estremamente potenti in tutte le concentrazioni. I primi due sono macrolidi, mentre la rifabutina è una rifamicina, ed entrambe le classi servono come base per il trattamento contro molte infezioni micobatteriche16.
Questo screening si basa su attrezzature specifiche e costose per la manipolazione dei liquidi per ridurre la variabilità tra i saggi. Nonostante sia estremamente riproducibile, il liquid handler non ha un trasferimento senza contatto di composti, come ad esempio quello acustico. Pertanto, alcuni composti aderenti possono rimanere attaccati ai perni metallici tra un trasferimento e l'altro, trasferendoli ai pozzetti successivi, con conseguente falsi positivi durante la fase di screening - questo è ciò che è accaduto con il composto 33. Questo è il motivo per cui la convalida dello screening è così fondamentale per il suo successo, garantendo che nessun falso positivo continui alle fasi successive della pipeline di screening dei farmaci. Il test dell'infezione utilizza un sistema di imaging ad alto contenuto con modalità confocale per ottenere la migliore definizione possibile delle regioni batteriche. Ciononostante, è ancora possibile utilizzarlo senza una modalità confocale, anche se con una perdita di definizione e possibilmente ostacolando l'identificazione delle regioni batteriche. Questo protocollo sfrutta il software di analisi semplice e intuitivo integrato con il sistema di imaging; tuttavia, il software open source può essere utilizzato seguendo il protocollo (Figura supplementare 5). In definitiva, nonostante non ripropongano farmaci per il trattamento delle infezioni da Mab, questi risultati sono estremamente importanti perché convalidano il protocollo stabilito, che può essere esteso a librerie più ampie. È importante sottolineare che riteniamo che questo protocollo possa essere adattato a qualsiasi batterio, a condizione che siano disponibili letture fluorescenti o luminescenti. Pertanto, questo lavoro contribuisce in modo significativo al campo della scoperta di farmaci, fornendo gli strumenti necessari per aiutare a combattere uno dei maggiori problemi di salute pubblica: i batteri resistenti agli antibiotici, e in particolare un agente patogeno quasi intrattabile: il Mycobacterium abscessus.
Divulgazioni
Gli autori dichiarano di non avere conflitti di interesse.
Riconoscimenti
Questo lavoro è finanziato da fondi nazionali portoghesi attraverso FCT - Fundação para a Ciência e a Tecnologia, I.P, nell'ambito del progetto PTDC/BIA-MIC/3458/2020 (DOI: 10.54499/PTDC/BIA-MIC/3458/2020) e borse di dottorato 2021.07335.BD a GSO e UI/BD/150830/2021 a CMB; FWO - Fondazione per la Ricerca nelle Fiandre, sovvenzione n° 1S68720N; Iniziativa di medicina innovativa 2 Call 16 (IMI2-Call 16) proposta RespiriTB nell'ambito dell'accordo numero 853903. Gli autori riconoscono il supporto della piattaforma scientifica i3S BioSciences Screening, membro delle infrastrutture nazionali PT-OPENSCREEN (NORTE-01-0145-FEDER-085468) e PPBI - Piattaforma portoghese di bioimaging (PPBI-POCI-01-0145-FEDER-022122).
Materiali
| Name | Company | Catalog Number | Comments |
| 10% Neutral buffered formalin | Bio Optica | 05-01005Q | fixation solution |
| 384-well black round bottom polypropylene not treated microplate | Corning | 3658 | White microplates can be used; polystyrene can be used |
| 50 TS Washer | BioTek | ||
| Breathe-Easy sealing membrane | Sigma-Aldrich | Z380059-1PAK | |
| cell::explorer | Revvity | equipment handling robot | |
| Clarithromycin | Sigma-Aldrich | A3487-100MG | Dissolved in DMSO to the desired concentration |
| DMSO | Fisher Scientific | D/4121/PB15 | Dilute in liquid growth medium to the desired concentration |
| Harmony 5.1 | Revvity | analysis software | |
| Heracell VIOS 160i CO2 Incubator, 165 L | Thermo Scientific | cell incubator | |
| JANUS with 4 Tip Arm and MDT Arm | Revvity | liquid handler | |
| KB 8182 | Termaks | incubator with agitation | |
| Libra S4+ | Biochrom | ||
| Multidrop Combi Reagent Dispenser | Thermo Scientific | ||
| Opera Phenix Plus | Revvity | imaging system | |
| PhenoPlate | Revvity | 6055302 | Necessary for high-content microscopes |
| Phosphate Buffer Saline (PBS) 1x | NA | NA | washing solution |
| Prestwick Chemical libraries | Commercial chemical library intended for drug repurposing | ||
| Shaking incubator 3031 | GFL | ||
| VICTOR Nivo | Revvity | ||
| Cell culture medium: | When needed, add 10% of supplement | ||
| DMEM | Gibco | 10938-025 | |
| Fetal Bovine Serum (FBS) | Biowest | S1810 | |
| HEPES Buffer Solution (1 M) | Gibco | 15630-056 | |
| LCCM | NA | NA | Conditioned media from L929 cell line that releases Macrophage colony stimulating factor (M-CSF); cell culture medium supplement |
| L-glutamin (200 mM) | Thermo Scientific | 25030024 | |
| Sodium Pyruvate 100 mM (100X) | Gibco | 11360-039 | |
| Infection washing solution | |||
| Apirogenic water | Multiple suppliers available | ||
| Hank's Balanced Salt Solution 10x | Gibco | 14060-040 | Dilute to 1x with apirogenic water |
| Permeabilising Solution | PBS with 0.1% Triton X100 | ||
| PBS 1x | NA | NA | |
| Triton X100 | Sigma-Aldrich | T8787 | Other suppliers available |
| Staining solution | PBS with DAPI (500 ng/mL) and HCS Far Red (10 ng/mL) | ||
| DAPI | Sigma-Aldrich | D9564 | |
| HCS Far Red | Invitrogen | H32721 | |
| PBS 1x | NA | NA | Multiple suppliers available |
| Liquid growth medium | When needed, add 10% supplement | ||
| Middlebrook 7H9 Broth | BD Difco | 271310 | |
| Tween80 | Sigma-Aldrich | P4780 | 0.05% final concentration. Other suppliers available |
| Liquid growth medium supplement: | |||
| Bovine Serum Albumin | Fisher Scientific | BP9702-100 | |
| D-Glucose anhydrous | Fisher Scientific | G/0450/60 |
Riferimenti
- Victoria, L., Gupta, A., Gomez, J. L., Robledo, J. Mycobacterium abscessus complex: A review of recent developments in an emerging pathogen. Front Cell Infect Microbiol. 11, 659997(2021).
- Luthra, S., Rominski, A., Sander, P. The role of antibiotic-target-modifying and antibiotic-modifying enzymes in Mycobacterium abscessus drug resistance. Front Microbiol. 9, 2179(2018).
- Nathavitharana, R. R., Strnad, L., Lederer, P. A., Shah, M., Hurtado, R. M. Top questions in the diagnosis and treatment of pulmonary M. abscessus disease. Open Forum Infect Dis. 6 (7), ofz221(2019).
- Cristancho-Rojas, C., et al. Epidemiology of Mycobacterium abscessus. Clin Microbiol Infect. 30 (6), 712-717 (2024).
- Hua, Y., et al. Drug repositioning: Progress and challenges in drug discovery for various diseases. Eur J Med Chem. 234, 114239(2022).
- Doan, T. L., Pollastri, M., Walters, M. A., Georg, G. I. The future of drug repositioning. Annual reports in medicinal chemistry. Macor, J. E. , Academic Press. 385-401 (2011).
- Bento, C. M., Gomes, M. S., Silva, T. Evolution of antibacterial drug screening methods: Current prospects for mycobacteria. Microorganisms. 9 (12), 2562(2021).
- Attene-Ramos, M. S., Austin, C. P., Xia, M. High throughput screening. Encycl Toxicol. 3, 916-917 (2014).
- Gupta, R., Netherton, M., Byrd, T. F., Rohde, K. H. Reporter-based assays for high-throughput drug screening against Mycobacterium abscessus. Front Microbiol. 8, 2204(2017).
- Bento Clara, M., et al. Characterization of novel double-reporter strains of Mycobacterium abscessus for drug discovery: A study in mScarlet. Microbiol Spectr. , e0036224(2024).
- Kolbe, K., et al. Development and optimization of chromosomally-integrated fluorescent Mycobacterium tuberculosis reporter constructs. Front Microbiol. 11, 591866(2020).
- Lopez-Roa, P., Esteban, J., Munoz-Egea, M. C. Updated review on the mechanisms of pathogenicity in Mycobacterium abscessus, a rapidly growing emerging pathogen. Microorganisms. 11 (1), 90(2022).
- Bray, M. A., Carpenter, A. Advanced assay development guidelines for image-based high content screening and analysis in Assay guidance manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences, Bethesda (MD). (2004).
- Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard. CLSI document M07-A10. , 10th ed, Wayne, PA. (2015).
- Silva, T., et al. Lactoferricin peptides increase macrophages' capacity to kill Mycobacterium avium. mSphere. 2 (4), e00301-e00317 (2017).
- Bento, C. M., Gomes, M. S., Silva, T. Looking beyond typical treatments for atypical mycobacteria. Antibiotics (Basel). 9 (1), 18(2020).
- Spizek, J., Rezanka, T. Lincosamides: Chemical structure, biosynthesis, mechanism of action, resistance, and applications. Biochem Pharmacol. 133, 20-28 (2017).
- Hurst-Hess, K. R., Rudra, P., Ghosh, P. Ribosome protection as a mechanism of lincosamide resistance in Mycobacterium abscessus. Antimicrob Agents Chemother. 65 (11), e0118421(2021).
- Kang, J. K., Kang, H. K., Hyun, C. G. Anti-inflammatory effects of spiramycin in lps-activated raw 264.7 macrophages. Molecules. 27 (10), 3202(2022).
- Zimmermann, P., Ziesenitz, V. C., Curtis, N., Ritz, N. The immunomodulatory effects of macrolides-a systematic review of the underlying mechanisms. Front Immunol. 9, 302(2018).
- Pradhan, S., Madke, B., Kabra, P., Singh, A. L. Anti-inflammatory and immunomodulatory effects of antibiotics and their use in dermatology. Indian J Dermatol. 61 (5), 469-481 (2016).
- Volochnyuk, D. M., et al. Evolution of commercially available compounds for hts. Drug Discov Today. 24 (2), 390-402 (2019).
- Sritharan, S., Sivalingam, N. A comprehensive review on time-tested anticancer drug doxorubicin. Life Sci. 278, 119527(2021).
- Arwanih, E. Y., Louisa, M., Rinaldi, I., Wanandi, S. I. Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: A literature review. Cureus. 14 (12), e33165(2022).
- Khasraw, M., Bell, R., Dang, C. Epirubicin: Is it like doxorubicin in breast cancer? A clinical review. Breast. 21 (2), 142-149 (2012).
- Schultz, C. W., Nevler, A. Pyrvinium pamoate: Past, present, and future as an anti-cancer drug. Biomedicines. 10 (12), 3249(2022).
- Bailly, C. The bacterial thiopeptide thiostrepton. An update of its mode of action, pharmacological properties and applications. Eur J Pharmacol. 914, 174661(2022).
- Kwok, J. M., et al. Thiostrepton selectively targets breast cancer cells through inhibition of forkhead box m1 expression. Mol Cancer Ther. 7 (7), 2022-2032 (2008).
- Kim, T. H., et al. Thiostrepton: A novel therapeutic drug candidate for Mycobacterium abscessus infection. Molecules. 24 (24), 4511(2019).
- Perry, C. M., Scott, L. J. Cefdinir: A review of its use in the management of mild-to-moderate bacterial infections. Drugs. 64 (13), 1433-1464 (2004).
- Ramon-Garcia, S., et al. Repurposing clinically approved cephalosporins for tuberculosis therapy. Sci Rep. 6, 34293(2016).
- Kumar, P., et al. Mycobacterium abscessus l,d-transpeptidases are susceptible to inactivation by carbapenems and cephalosporins but not penicillins. Antimicrob Agents Chemother. 61 (10), e00866-e00917 (2017).
- Alvirez-Freites, E. J., Carter, J. L., Cynamon, M. H. In vitro and in vivo activities of gatifloxacin against Mycobacterium tuberculosis. Antimicrob Agents Chemother. 46 (4), 1022-1025 (2002).
- Braback, M., Riesbeck, K., Forsgren, A. Susceptibilities of Mycobacterium marinum to gatifloxacin, gemifloxacin, levofloxacin, linezolid, moxifloxacin, telithromycin, and quinupristin-dalfopristin (synercid) compared to its susceptibilities to reference macrolides and quinolones. Antimicrob Agents Chemother. 46 (4), 1114-1116 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon