Method Article
Développement d’un test pour le criblage de médicaments à haut débit contre les mycobactéries
Dans cet article
Résumé
Cette étude a mis au point un test de criblage à haut débit contre Mycobacterium abscessus avec une souche à double rapporteur récemment créée, en utilisant la fluorescence et la luminescence pour évaluer rapidement la viabilité bactérienne. Ce protocole sera pertinent pour les chercheurs qui souhaitent cribler de nouveaux médicaments contre cette bactérie résistante aux médicaments.
Résumé
Les infections à Mycobacterium abscessus (Mab) sont difficiles à traiter en raison de leur forte résistance intrinsèque aux médicaments, comparable à celle de la tuberculose multirésistante. Les traitements sont extrêmement inefficaces et basés sur un régime multimédicamenteux, ce qui entraîne une faible observance des patients. Par conséquent, la communauté scientifique est invitée à identifier de nouveaux médicaments efficaces pour traiter ces infections. L’une des stratégies employées à cette fin est la réorientation des médicaments, c’est-à-dire le processus d’identification de nouvelles opportunités thérapeutiques pour les médicaments existants sur le marché, en contournant le temps nécessaire à l’établissement des profils pharmacocinétiques et d’innocuité des nouveaux médicaments. La plupart des études sur le développement de médicaments contre Mab reposant sur des méthodes traditionnelles et chronophages, un test de criblage de médicaments à haut débit a été mis au point contre les mycobactéries à l’aide d’une souche à double rapporteur de Mab développée en interne. À l’aide de la robotique de manipulation des liquides, de la microscopie automatisée et de l’analyse, ainsi que de souches à double rapporteur développées en interne, la viabilité bactérienne peut être rapidement mesurée à l’aide de deux lectures différentes, la luminescence et la fluorescence, sans ajouter de réactifs ni effectuer d’étapes supplémentaires. Cela réduit le temps et la variabilité entre les essais, un avantage majeur pour les criblages à haut débit. Le protocole décrit a été validé par le criblage d’une bibliothèque de 1280 composés. Les résultats obtenus ont été corroborés par la littérature, avec une détection efficace des composés actifs. Ainsi, ces travaux ont atteint l’objectif de doter le domaine d’un nouvel outil pour aider à lutter contre cette bactérie extrêmement résistante aux médicaments.
Introduction
Mycobacterium abscessus (Mab) est un agent pathogène opportuniste responsable d’infections pulmonaires, en particulier chez les personnes atteintes de mucoviscidose et d’autres troubles pulmonaires. Les infections causées par l’épi mab sont tristement difficiles à traiter en raison de leur résistance intrinsèque aux médicaments, comparable à celle de la tuberculose multirésistante1. Les médicaments disponibles sont largement inefficaces en raison de l’enveloppe cellulaire mycobactérienne hautement imperméable et d’un génome qui code pour plusieurs enzymes qui désactivent les antibiotiques2. Ainsi, le traitement comprend la combinaison de plusieurs médicaments prenant des mois à des années. Ce schéma thérapeutique multimédicamenteux complexe, combiné à une faible observance du patient, se traduit par un taux de guérison moyen de 30 % à 50 %3. De plus, la prévalence des infections pulmonaires causées par des mycobactéries non tuberculeuses a augmenté au cours des dernières décennies, y compris celles causées par Mab 1,4. Par conséquent, la communauté scientifique s’efforce de développer de nouveaux composés pour traiter les infections à Mab.
L’une des stratégies poursuivies dans ce but est la réaffectation des médicaments, c’est-à-dire le processus d’identification de nouvelles opportunités thérapeutiques pour les médicaments existants. Cela permet de contourner le plus grand défi accompagnant le pipeline de découverte et de développement d’un nouveau médicament - temps5. Ce concept simple tire parti des profils pharmacocinétiques et d’innocuité déjà établis de plusieurs médicaments pour réduire les coûts de développement et raccourcir le délai nécessaire pour acheminer un médicament du laboratoire au chevet du patient6. Ainsi, des banques qui combinent des centaines à des milliers de ces composés ont été compilées, ce qui permet aux chercheurs de tester rapidement la possibilité de réorienter les médicaments contre leur agent pathogène d’intérêt.
La plupart des études sur le développement de médicaments contre le Mab sont basées sur un test traditionnel de référence qui évalue l’activité in vitro d’un composé contre les mycobactéries - les unités formant des colonies comptant7. Malgré sa précision, cette procédure prend énormément de temps, devenant rapidement irréalisable lorsque quelqu’un vise à tester des banques contenant des milliers de composés. À cette fin, les criblages à haut débit (HTS) ont été intégrés dans le développement de médicaments - des tests robustes qui tirent parti de la robotique et des dispositifs de manipulation des liquides, permettant à des milliers de composés d’être rapidement criblés en parallèle8. Cela se fait généralement en testant initialement une seule concentration, à l’aide de plaques de microtitration de formats 96, 384, 1536 ou 3456 puits, servant de point de départ pour identifier les résultats et les optimiser davantage en cours de développement pour être utilisés cliniquement.
Les dosages basés sur le rapporteur offrent un avantage significatif pour la robustesse des SHD en raison de leur simplicité et de leur sensibilité par rapport à d’autres dosages basés sur les colorants et l’absorbance 7,9. Cependant, à notre connaissance, seules quelques études ont permis d’optimiser un dépistage à haut débit contre Mab9.
Récemment, notre laboratoire a mis au point des souches à double rapporteur capables d’émettre simultanément une luminescence et une fluorescence10. Mab operon_mScarlet est l’une de ces souches. Il est autoluminescent en raison de l’expression de l’opéron LuxABCDE , qui comprend une luciférase bactérienne (par expression des gènes luxAB ) et un substrat aldéhyde à longue chaîne (par expression des gènes luxCDE ). D’autre part, la lecture fluorescente est obtenue par l’expression d’une protéine fluorescente rouge récemment développée, mScarlet, qui surpasse les protéines eGFP et mCherry plus couramment utilisées, fournissant un signal plus puissant11. L’utilisation de cette souche nous permet d’évaluer la viabilité bactérienne en culture liquide en mesurant le signal luminescent dans un lecteur de microplaques sans ajouter de réactifs ni effectuer d’étapes supplémentaires. En termes de détection, la fluorescence intrinsèque permet une visualisation au microscope dans des cellules vivantes ou fixes sans utiliser de colorants ou d’anticorps. Le fait d’avoir une seule souche avec les deux lectures offre aux chercheurs un avantage significatif lorsqu’ils l’utilisent dans les tests HTS - la variabilité réduite entre les tests avec des lectures différentes, car il n’est pas nécessaire d’échanger les souches en fonction de la nature du test.
Ainsi, ce travail a mis au point un test à haut débit contre Mab à l’aide d’une souche double rapporteure conçue en interne (Figure 1). Cela a permis d’évaluer rapidement l’activité in vitro des médicaments 1280 destinés à être repositionnés à l’aide d’une bibliothèque commerciale (voir la table des matériaux). Tout d’abord, les activités ont été évaluées dans un essai de culture en bouillon utilisant la luminescence, et deuxièmement, en utilisant des macrophages infectés par Mab tirant parti du signal fluorescent, imitant mieux le processus d’infection observé in vivo12.
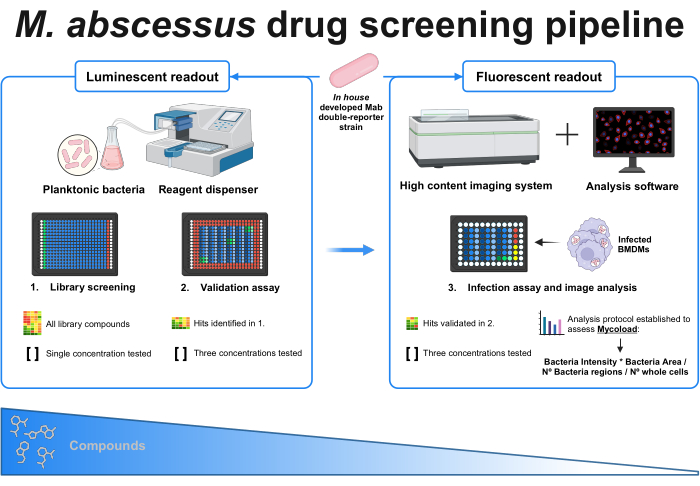
Figure 1 : Résumé graphique du protocole établi. L’acteur clé de ce criblage est une souche mycobactérienne à double rapporteur développée en interne, qui est utilisée dans toutes les expériences. Tout d’abord, à l’aide d’un distributeur de réactifs et de bactéries planctoniques, un premier criblage est effectué en testant les composés à une concentration unique. Les résultats positifs identifiés se poursuivent lors de l’essai de validation, où trois concentrations différentes sont testées. Les deux expériences sont réalisées à l’aide de la lecture luminescente. Les résultats validés se poursuivent dans le test d’infection, où trois concentrations différentes sont également testées, et où des macrophages dérivés de la moelle osseuse sont infectés au MOI 1. Un système d’imagerie à haut contenu acquiert le signal fluorescent bactérien, et un logiciel d’analyse est utilisé pour évaluer la charge intracellulaire à l’aide de la formule de la mycocharge. Le nombre de composés diminue à mesure que la complexité du test augmente. Créé avec BioRender.com. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Toutes les procédures animales ont été approuvées par le Comité local d’éthique animale d’i3S et autorisées par la Direction générale de l’alimentation et des affaires vétérinaires (DGAV), conformément à la directive du Conseil européen (2010/63/UE) et à la loi portugaise (DL 113/2013) sur le bien-être animal en matière d’expérimentation.
1. L’appréciation du facteur Z
REMARQUE : Pour évaluer les performances d’un test HTS, plusieurs paramètres peuvent être déterminés. Le paramètre le plus utilisé est le facteur premier Z (facteur Z'). Cette mesure mesure l’ampleur de l’effet statistique d’une expérience donnée, indiquant dans quelle mesure les contrôles positifs et négatifs sont bien séparés. Si un facteur Z' est compris entre 0,5 et 1, la séparation entre les deux populations est excellente, et ces conditions peuvent être utilisées dans le test HTS. Si Z' < 0,5, la séparation n’est que marginale, et il n’est pas recommandé d’effectuer le dépistage13. C’est pourquoi une expérience utilisant la moitié d’une plaque avec des bactéries non traitées et l’autre moitié avec un témoin positif pour calculer le facteur Z' a été conçue (figure supplémentaire 1).
- 2 à 3 jours avant l’essai, préparer les précultures d’anticorps monoclonaux en inoculant des bactéries dans un milieu de croissance liquide (environ 1 x 107 UFC/mL) dans un erlenmeyer. Laissez les bactéries se développer à 37 °C dans un incubateur avec agitation (90 tr/min).
ATTENTION : Une enceinte de biosécurité est nécessaire lorsque l’on travaille avec des cultures d’épis. - Suivez la croissance de la préculture de Mab par mesure optique de la densité à 600 nm à l’aide d’un spectrophotomètre. Lorsque la culture atteint la phase de croissance exponentielle, passez à l’étape 1.3.
- À l’aide de la pré-culture exponentielle, préparer une suspension bactérienne d’environ 5 x 105 UFC/mL (corrélée aux valeurs de densité optique)14 dans un milieu de croissance liquide.
- Préparez une solution de contrôle positif de l’inhibition de la croissance bactérienne dans un milieu de croissance liquide. Pour ce dosage, le témoin positif était la clarithromycine (10 mg/mL dans le DMSO) à 4 μg/mL. La clarithromycine doit être préparée 2 fois la concentration finale souhaitée.
- À l’aide d’un distributeur de réactifs sur microplaque équipé d’une petite cassette, distribuer 15 μL de milieu de croissance liquide dans les colonnes vierges et non traitées d’une plaque en polypropylène noir à 384 puits. Pour les colonnes vierges, distribuer 15 μL supplémentaires de milieu de croissance liquide.
REMARQUE : Les colonnes vierges empêchent l’évaporation des puits intérieurs à 37 °C. - À l’aide du distributeur de réactifs sur microplaques équipé d’une petite cassette, versez 15 μL de la solution de contrôle positif préparée à l’étape 1.4 dans les colonnes de contrôle positif.
- À l’aide d’un distributeur de réactifs sur microplaques équipé d’une grande cassette, distribuer 15 μL de la suspension bactérienne préparée à l’étape 1.3 dans toutes les colonnes, à l’exception des blancs, en diluant le tout 1:2. Le volume final de l’ensemble des 384 puits est de 30 μL.
REMARQUE : Le distributeur de réactifs pour microplaques peut être équipé d’une grande ou d’une petite cassette, différant par la taille du tube (0,5 et 0,22 mm, respectivement). Il est préférable d’utiliser une cassette plus grande pour éviter l’agrégation bactérienne dans le système de tuyauterie, même si cela entraîne plus de gaspillage de liquide. Néanmoins, la même cassette peut être utilisée tout au long du test. - Scellez chaque plaque avec une scelleuse de plaques qui permet un échange gazeux continu. Incuber à 37 °C dans un incubateur sans agitation à l’intérieur d’une chambre humide pendant 48 h.
- Après 48 h, lire la luminescence émise par les bactéries dans chaque plaque à l’aide d’un lecteur de plaques avec un temps d’intégration de 1 s.
- Pour déterminer le facteur Z', utilisez les valeurs obtenues à l’étape 1.9 et calculez la moyenne et l’écart-type des commandes positives (AVG P et SDP) et négatives (AVGN et SDN). Ensuite, appliquez la formule suivante :
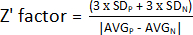
REMARQUE : 3 x SD est un seuil couramment utilisé ; Cependant, il peut être ajusté pour mieux s’adapter au dépistage, soit en augmentant soit en diminuant le facteur par lequel multiplier l’écart-type.
2. Sélection des visites en bibliothèque
REMARQUE : Les premiers criblages ont été effectués contre des bactéries se développant en culture liquide. La configuration a été conçue pour des plaques de 384 puits avec un volume final de 30 μL. Chaque plaque contenait des puits vierges (milieu de croissance liquide uniquement), des témoins négatifs (bactéries non traitées) et un contrôle de solvant (bactéries plus solvant du composé - dans ce cas, DMSO ; Figure supplémentaire 2). Les composés ou le solvant ont été ajoutés à la plaque à 2 fois la concentration finale souhaitée, et les bactéries ont été ajoutées 1:1 (diluant le tout à la moitié).
- Préparez la suspension bactérienne comme décrit aux étapes 1.1 à 1.3.
ATTENTION : Une enceinte de biosécurité est nécessaire lorsque l’on travaille avec des cultures d’épis. - Préparez une solution de solvant dans un milieu de croissance liquide. La quantité de solvant doit être 2 fois supérieure à la concentration finale souhaitée.
- À l’aide d’un distributeur de réactifs sur microplaques équipé d’une petite cassette, distribuer 15 μL de milieu de croissance liquide dans chaque colonne de plaques de polypropylène noir à 384 puits, à l’exception de la colonne de contrôle du solvant. Pour les colonnes blanches, distribuer 15 μL supplémentaires de milieu de croissance liquide.
- À l’aide du distributeur de réactifs sur microplaques équipé d’une petite cassette, versez 15 μL de la solution de solvant préalablement préparée dans la colonne de contrôle du solvant.
- À l’aide d’un manipulateur de liquides, transférez 200 nL des composés des plaques de stock à 96 puits (1 mM dans le DMSO) vers les plaques à 384 puits, à l’exception des blancs, du témoin de solvant et des colonnes non traitées. Les composés sont maintenant à une concentration de 13,3 μM.
- À l’aide d’un distributeur de réactifs sur microplaques équipé d’une grande cassette, distribuer 15 μL de la suspension bactérienne préparée à l’étape 2.1 dans toutes les colonnes, à l’exception des blancs, en diluant le tout 1:2. Le volume final de l’ensemble des 384 puits est de 30 μL, et chaque composé est à 6,66 μM.
- Scellez chaque plaque avec une scelleuse de plaques qui permet un échange gazeux continu.
REMARQUE : Le test peut être effectué avec des couvercles réguliers ; Cependant, il y a une plus grande possibilité d’effets de bord plus forts. - Incuber à 37 °C dans un incubateur sans agitation à l’intérieur d’une chambre humide pendant 48 h.
- Après 48 h, lire la luminescence émise par les bactéries dans chaque plaque à l’aide d’un lecteur de plaques avec un temps d’intégration de 1 s.
3. Analyse des données de dépistage des correspondances
REMARQUE : Pour identifier les résultats positifs avec les données de luminescence acquises, les valeurs doivent être normalisées en fonction du solvant dans lequel les composés sont dissous et, le cas échéant, en fonction de l’effet de bord vérifié sur les plaques de 384 microtitres.
- Pour la normalisation du solvant des données obtenues à l’étape 2.9, divisez les unités légères relatives (RLU) de chaque puits par la RLU moyenne des puits de contrôle du solvant (dans la plaque correspondante). Dans ce test, un fort effet de bord a été vérifié vers le milieu des plaques de criblage ; Ainsi, il a été décidé de normaliser toutes les données pour celui-ci. Pour ce faire, le processus s’est déroulé en deux étapes, comme décrit ci-dessous.
- Créez un masque d’effet de bord en divisant les valeurs obtenues à l’étape 3.1 pour les positions correspondantes, à l’exclusion des coups, sur toutes les plaques (plaque 1 - P1, plaque 2 - P2, plaque 3 - P3, plaque 4 - P4). Par exemple, pour les positions A1 et A2, Mask_A1 = Moyen (P1_A1 ; P2_A1 ; P3_A1 ; P4_A1) ; Mask_A2 = Moyen (P1_A2 ; P2_A2 ; P4_A2) [P3_A2 a été exclu parce que c’était un succès].
- Divisez les valeurs obtenues à l’étape 3.1. par son masque d’effet de bord obtenu à l’étape 3.1.1. Par exemple,
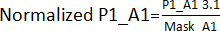 . Ensuite, calculez l’AVG et l’ET de chaque plaque pour établir un seuil (AVG ± 3SD) pour chaque plaque. Tout composé en dehors de ce seuil est considéré comme un résultat positif et est poursuivi dans le test de validation des résultats.
. Ensuite, calculez l’AVG et l’ET de chaque plaque pour établir un seuil (AVG ± 3SD) pour chaque plaque. Tout composé en dehors de ce seuil est considéré comme un résultat positif et est poursuivi dans le test de validation des résultats.
4. Test de validation des résultats
REMARQUE : Après avoir identifié les résultats, ceux-ci doivent être validés à l’aide de différentes concentrations. Le test de validation utilise une configuration similaire avec les mêmes contrôles, mais chaque résultat sera soumis à des dilutions en série de 1:2. Pour cet essai, trois concentrations ont été utilisées : 13,3 μM, 6,66 μM et 3,3 μM. Les témoins de solvant contiendront le même pourcentage de solvant que chaque puits composé (figure supplémentaire 3).
- Préparez la suspension bactérienne comme décrit aux étapes 1.1 à 1.3.
ATTENTION : Une enceinte de biosécurité est nécessaire lorsque l’on travaille avec des cultures d’épis. - À l’aide d’un distributeur de réactifs sur microplaque équipé d’une petite cassette, distribuez 15 μL de milieu de croissance liquide dans chaque colonne d’une plaque en polypropylène noir à 384 biens. Pour les puits vierges, versez 15 μL supplémentaires de milieu de croissance liquide dans la première et la dernière colonne.
- À l’aide du distributeur de réactifs sur microplaques, versez 15 μL supplémentaires de milieu de croissance liquide dans les colonnes contenant le [μM] le plus élevé de chaque composé.
- À partir des plaques de 96 puits, pipeter manuellement 0,8 μL de chaque coup (1 mM dans le DMSO) dans leurs puits respectifs à partir de l’étape 4.3, en double. Les composés sont maintenant à une concentration de 26,6 μM.
- Pipeter manuellement 0,8 μL de solvant dans les puits de contrôle en double exemplaire (comme à l’étape 4.4).
- À l’aide d’une micropipette multicanaux, effectuez deux dilutions en série 1:2 pour chaque composé et contrôle du solvant. Jeter les 15 μL excédant après la dernière dilution en série. Tous les puits ont maintenant 15 μL, et les concentrations des composés sont de 26,6 μM, 13,3 μM et 6,66 μM.
- À l’aide d’un distributeur de réactifs sur microplaques doté d’une grande cassette, distribuer 15 μL de la suspension bactérienne préparée en 4.1 dans toutes les colonnes (à l’exception des puits vierges), en diluant les composés à l’ordre de 1:2. Tous les puits ont un volume final de 30 μL, et chaque composé est à 13,3 μM, 6,66 μM et 3,3 μM.
- Scellez la plaque avec une scelleuse de plaques qui permet un échange gazeux continu. Incuber à 37 °C dans un incubateur sans agitation à l’intérieur d’une chambre humide pendant 48 h.
- Après 48 h, lire la luminescence émise par les bactéries dans chaque plaque avec un temps d’intégration de 1 s à l’aide d’un lecteur de plaques.
5. Dosage de l’infection
REMARQUE : Le Mab est un agent pathogène facultatif intracellulaire, de sorte que l’activité antimicrobienne et la toxicité de l’hôte doivent être déterminées dans des essais d’infection. Pour cela, des macrophages dérivés de la moelle osseuse (BMM) ont été infectés par Mab et traités avec les résultats validés à l’étape 4 à différentes concentrations. Chaque essai comprenait des témoins à blanc (uniquement de l’eau), des témoins négatifs (macrophages infectés non traités), des témoins positifs (macrophages infectés traités avec un antibiotique efficace), des témoins de solvant (dans ce cas, du DMSO) et les composés à tester à trois concentrations - 13,3 μM, 6,66 μM et 3,3 μM (figure supplémentaire 4).
- Dériver des macrophages de la moelle osseuse murine de souris adultes de type sauvage (BALB/c ou C57BL/6), comme décrit précédemment15. Plaquez les cellules sur une plaque à fond plat noire de 96 puits, optiquement transparente, à raison de 2 x 105 cellules/mL, 200 μL par puits. Après 10 jours de différenciation à 37 °C, 7% CO215, les cellules sont prêtes à infecter et à poursuivre le traitement.
REMARQUE : Le protocole d’obtention de macrophages dérivés de la moelle osseuse est bien établi15 ; cependant, ce protocole de criblage peut être adapté à d’autres cellules, telles que RAW 264.7, THP-1 et les macrophages dérivés de PBMC. - Préparez la suspension bactérienne comme décrit aux étapes 1.1 à 1.3, en ajustant la suspension bactérienne dans le milieu de culture cellulaire pour une infection avec une multiplicité d’infection (MOI) de 1.
ATTENTION : Une enceinte de biosécurité est nécessaire lorsque l’on travaille avec des cultures d’épis. - Aspirer soigneusement le milieu de culture cellulaire complété des puits à l’aide d’une pompe à vide fixée à une pipette en verre. Cette étape doit être effectuée lentement et facilement pour éviter le détachement des cellules.
- Pipeter manuellement 75 μL de la suspension bactérienne préparée à l’étape 5.2 dans chaque puits. Incuber la plaque à 37 °C avec 7% de CO2 pendant 4 h dans un incubateur de cellules.
- Préparez les composés pour le traitement cellulaire. À l’aide d’un distributeur de réactifs sur microplaques équipé d’une petite cassette, distribuer sur des plaques de polypropylène, à fond rond, à 96 puits, 110 μL de milieu de culture cellulaire supplémenté dans des colonnes où se trouveront des composés ou un témoin de solvant (figure supplémentaire 4).
- À l’aide d’un distributeur de réactifs sur microplaques, versez 104 μL supplémentaires de milieu de culture cellulaire supplémenté dans les colonnes où se trouvera la concentration la plus élevée de composé ou de solvant.
- Pipeter manuellement 5,9 μL des composés (1 mM dans le DMSO) ou du solvant dans les puits désignés (colonnes à l’étape 5.6). Les composés sont maintenant à une concentration de 26,6 μM.
- À l’aide d’une micropipette multicanaux, effectuez deux dilutions en série 1:2 pour chaque composé et solvant. Jeter les 110 μL excédant après la dernière dilution en série. Tous les puits ont maintenant 110 μL, et les composés sont à une concentration de 26,6 μM, 13,3 μM et 6,66 μM.
- À l’aide d’un distributeur de réactifs sur microplaques, distribuer 110 μL de milieu de culture cellulaire supplémenté dans toutes les colonnes (à l’exception des puits vierges). Tous les puits ont maintenant 220 μL, et les composés sont à une concentration de 13,3 μM, 6,66 μM et 3,3 μM.
- Préparer une solution de clarithromycine (témoin positif) à 2 μg/mL dans un milieu de culture cellulaire supplémenté.
- 4 h après l’étape 5.4, à l’aide d’un laveur de plaques, laver 3 fois avec 200 μL de solution de lavage infectieuse la plaque infectée à 96 puits. Maintenez les vitesses d’aspiration et de distribution au plus lent possible pour éviter tout détachement cellulaire.
- Après l’aspiration finale, transférez 200 μL des composés préparés aux étapes 5.5 à 5.9 dans la plaque à 96 puits contenant les macrophages infectés.
- Transférer 200 μL de milieu de culture cellulaire supplémenté dans les puits non traités. Transférez 200 μL de la solution de clarithromycine préparée à l’étape 5.10 dans ses puits respectifs.
- Incuber la plaque dans un incubateur cellulaire à 37 °C, avec 7% de CO2, pendant 48 h.
- Après 48 h, laver la plaque infectée à 96 puits à l’aide d’un laveur de plaques avec la solution de lavage du composé (3x avec 200 μL par puits).
- À l’aide d’une micropipette multicanaux, versez 200 μL de la solution de fixation dans tous les puits et laissez-la agir pendant 10 minutes.
- Après la fixation, à l’aide d’un laveur de plaques, laver les cellules avec la solution de lavage des composés (3x avec 200 μL par puits).
REMARQUE : Le protocole peut être mis en pause ici. Les cellules peuvent être stockées dans la solution de lavage des composés à 4 °C pendant plusieurs jours ou semaines si la contamination et la sécheresse des cellules sont évitées. La solution de lavage des composés doit être aspirée avant l’étape 5.18. - À l’aide d’une micropipette multicanaux, distribuer 200 μL de la solution de perméabilisation dans tous les puits et laisser agir pendant 15 min. À l’aide d’un rond-plat, aspirez la solution de perméabilisation.
- À l’aide d’une micropipette multicanaux, distribuez manuellement 100 μL de la solution de coloration et incubez les plaques à température ambiante pendant 30 min.
- À l’aide d’un laveur de plaques, lavez les plaques avec la solution de lavage des composés (3x avec 200 μL par puits). À la fin, laissez les cellules colorées dans la solution de lavage des composés.
- Criblez les plaques dans un système d’imagerie à haut contenu avec les paramètres suivants :
Objectif : 20x Air/0,4NA (un puits d’une plaque de 96 microtitres, 57 champs de vision (FOV))
Mode : Confocal
Laser 405 nm pour DAPI (noyaux)
Laser 561 nm pour mScarlet (bactéries)
Laser 640 nm pour HCS Cell Mask Deep Red (cytoplasme)
Pas de pile z
REMARQUE : Les cellules peuvent être imagées avec un système d’imagerie à grand champ, ce qui diminue la définition. - Mettre en place un robot de manutention d’équipements pour l’acquisition d’images pendant la nuit. Ce bras robotique remplace les plaques dans le système d’imagerie à haut contenu sans intervention humaine.
REMARQUE : Ce n’est pas obligatoire ; Les plaques peuvent être remplacées manuellement.
6. Analyse d’images
REMARQUE : L’analyse d’image est effectuée à l’aide du logiciel d’analyse, qui est le même que le système d’imagerie à haut contenu utilisé pour acquérir les images. Un pipeline d’analyse doit être créé à l’aide d’exemples d’images (Figure supplémentaire 5). Par la suite, il sera appliqué à l’ensemble du puits (57 FOV) et de l’ensemble de données (toutes les plaques). Le logiciel d’analyse suit une séquence logique d’étapes, en commençant par la segmentation des différentes régions d’intérêt (noyaux, cytoplasme, cellules et bactéries), en les reliant (par exemple, les bactéries à l’intérieur des cellules), puis en extrayant les propriétés morphologiques et d’intensité (par exemple, l’aire, l’intensité). Les étapes essentielles de l’analyse d’image sont décrites ci-dessous. Le protocole complet utilisé se trouve à la figure supplémentaire 5.
- Segmentez les noyaux à l’aide du signal DAPI, le cytoplasme à l’aide du signal DeepRed et les bactéries à l’aide du signal mScarlet.
- Relier les noyaux et la segmentation du cytoplasme pour créer un masque pour les cellules.
- Retirez tous les objets de bordure. Seules les cellules qui apparaissent comme pleines sur le FOV seront prises en compte (cellules entières).
- Définissez la population de sortie Bactéries dans des cellules entières en reliant les cellules entières et les bactéries.
- Définissez les cellules entières infectées comme ayant plus d’une région bactérienne et les cellules entières non infectées comme ayant moins d’une région bactérienne.
- Une fois que toutes les régions d’intérêt ont été segmentées et que les populations ont été créées, calculez et extrayez leurs propriétés. Les propriétés suivantes sont extraites pour chaque puits.
- Pour les cellules entières/ Cellules entières infectées/ Cellules entières non infectées, extrait
Nombre de cellules (somme par puits)
Aire [μm2] (Moyenne + Écart-type par puits)
Rapport largeur/longueur des cellules (moyenne + écart-type par puits) - Pour les bactéries dans des cellules entières, extrayez
Nombre de régions bactériennes identifiées (somme par puits)
Intensité (somme par puits de l’intensité moyenne de chaque région)
Superficie de la région (somme par puits)
- Pour les cellules entières/ Cellules entières infectées/ Cellules entières non infectées, extrait
- Après avoir exporté les données et les avoir organisées dans une feuille de calcul, quantifiez la charge intracellulaire (Mycoload) selon la formule suivante :
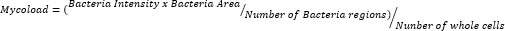
Résultats
Évaluation du facteur Z
La figure 2 représente les données d’une des deux expériences réalisées pour calculer le facteur Z'. Le témoin positif était la clarithromycine à 4 μg/mL. Les deux expériences ont donné des facteurs Z' de 0,64 et 0,62, ce qui signifie que les conditions et la lecture utilisées pour ce test peuvent être appliquées au test de dépistage qui a suivi (Z' > 0,5). Néanmoins, le facteur Z a été calculé pour toutes les expériences restantes (test d’infection) afin de contrôler les performances de chaque expérience.
Comme preuve de concept du test HTS conçu, une bibliothèque de composés destinés à la réaffectation de médicaments a été testée. Il comprend 1280 molécules diverses et petites, dont 95% sont des médicaments approuvés par la FDA et l’EMA. Ces molécules offrent une grande diversité chimique et pharmacologique.
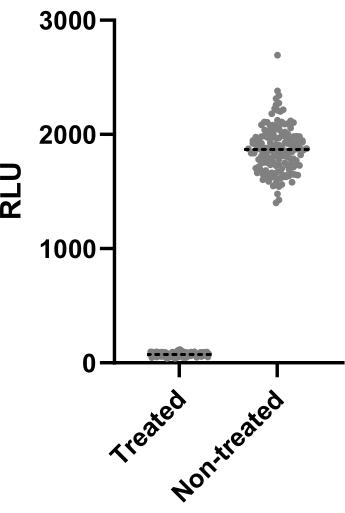
Figure 2 : Résultats de l’évaluation du facteur Z. L’anticorps monoclonal à 2,5 x10 5 UFC/mL a été incubé avec et sans clarithromycine à 4 μg/mL pendant 48 h à 37 °C. Après la période d’incubation, la luminescence a été mesurée pour évaluer la viabilité mycobactérienne. Le graphique montre les valeurs de luminescence individuelles des mycobactéries viables traitées et non traitées dans une expérience indépendante. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Projection de la bibliothèque pour les visites
Tous les composés ont été testés à 6,66 μM contre Mab. En raison de la taille de la bibliothèque, les composés ont été divisés en quatre plaques de microtitration différentes à 384 puits (figure 3A-D). Les résultats ont été normalisés pour le solvant et l’effet de bord vérifié.
Dans cet essai de dépistage, trente-trois résultats ont été identifiés, dont trente ont considérablement diminué l’émission de luminescence, réduisant ainsi la viabilité mycobactérienne (figure 3). Il est intéressant de noter que trois composés ont conduit à une émission de luminescence plus élevée, peut-être liée à une augmentation du métabolisme bactérien ou à une prolifération (Figure 3). Les 33 résultats ont été retenus pour l’essai de validation, y compris les trois composés qui augmentaient la luminescence, afin de vérifier si ce profil serait conservé.
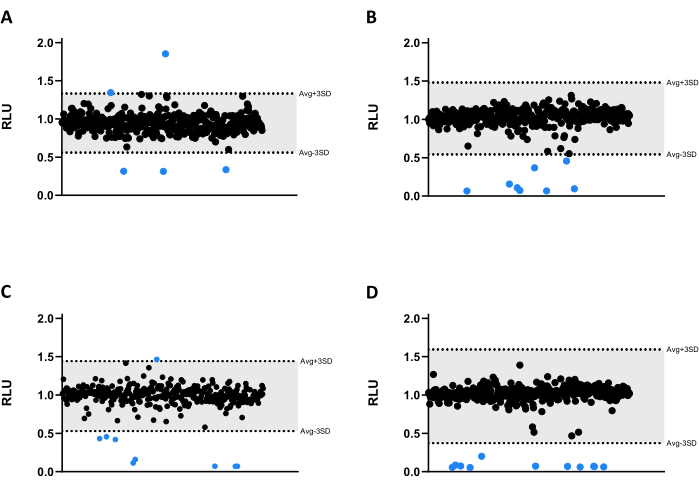
Figure 3 : Résultats du criblage en banque. (A-D) Le Mab à 2,5 x 105 UFC/mL a été incubé avec 1280 composés à 6,66 μM pendant 48 h à 37 °C. Après l’incubation, la luminescence a été mesurée pour évaluer la viabilité mycobactérienne. Les graphiques montrent le RLU d’une expérience indépendante, présenté sous forme d’unités arbitraires après normalisation du solvant et de l’effet de bord. L’AVG RLU et l’écart-type correspondant ont été calculés pour chaque plaque afin de déterminer un seuil (en gris ; AVG ± 3SD). Les symboles ronds représentent un composé testé. En bleu, tout composé en dehors de ce seuil est considéré comme un succès. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Test de validation des résultats positifs
Les concentrations utilisées pour ce test étaient de 13,3 μM, 6,66 μM et 3,3 μM. Chaque composé a été testé en double pour augmenter la robustesse.
Il est important de noter que les composés 30, 31 et 32, précédemment associés à des RLU plus élevés (figures 3A, C), ont conservé ce profil, avec plus de 200 % de la viabilité mycobactérienne obtenue dans les puits traités avec le composé 32 (figure 4).
Les composés 20 et 33 ont été jugés inactifs, car la viabilité mycobactérienne est proche de 100 % dans toutes les concentrations testées (figure 4).
Les composés 9 et 21 présentaient une inactivité similaire aux deux concentrations les plus faibles ; cependant, contrairement à 20, ils restent actifs au plus haut, 13,3 μM, le composé 21 affichant une puissance plus élevée (figure 4).
Le composé 3 présente également une inactivité à la concentration la plus faible et une perte d’activité à 6,66 μM, bien que inférieure à celle du composé 21 (figure 4). Ce n’est qu’à la concentration la plus faible que les composés 2, 7, 8, 10, 16, 18, 24 et 28 ont montré un potentiel thérapeutique plus faible. Cependant, 2, 10 et 28 conduisent toujours à <50% de la viabilité mycobactérienne.
À l’échelle mondiale, les quatorze composés restants étaient actifs dans toutes les concentrations testées.
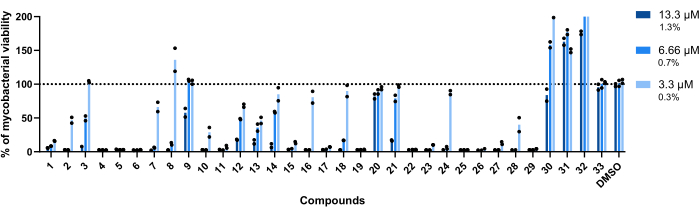
Figure 4 : Résultats du test de validation des résultats. À raison de 2,5 x 105 UFC/mL, chaque occurrence précédemment identifiée étant à 13,3 μM, 6,66 μM et 3,3 μM (1,3 %, 0,7 % et 0,3 % de DMSO, respectivement) pendant 48 h à 37 °C. Après la période d’incubation, la luminescence a été mesurée pour évaluer la viabilité mycobactérienne. Le graphique montre les pourcentages de mycobactéries viables traitées par rapport aux mycobactéries non traitées d’une expérience indépendante, chaque composé étant testé en double. Les symboles ronds représentent les doublons de chaque composé. Les barres représentent la moyenne de ces doublons. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Dosage de l’infection
Sur trente-trois composés testés, vingt-huit ont été sélectionnés pour l’essai d’infection (figure 4). Les composés 7, 8, 9, 20 et 33 n’ont pas été retenus pour cet essai. Alors que 9, 20 et 33 ont été laissés de côté en raison de leur inactivité lors de la validation (figure 4), les deux premiers ont été laissés de côté pour des raisons techniques. Néanmoins, ces composés ont été identifiés comme étant la rifampicine et le linézolide, des antibiotiques déjà utilisés pour traiter les infections à Mab12. Tous les composés testés dans le cadre de l’essai d’infection ont été identifiés et sont énumérés dans le tableau 1. L’activité antimicrobienne des composés contre les macrophages infectés par Mab a été évaluée en utilisant la fluorescence intrinsèque de la bactérie comme lecture.
| Composé | Nom | Composé | Nom |
| 1 | Le sulfathiazole | 18 | Cévuroxime |
| 2 | Ciprofloxacine | 19 | La rifaximine |
| 3 | Céfotaxime | 21 | Cefdinir |
| 4 | Daunorubicine | 22 | Clarithromycine |
| 5 | Doxorubicine | 23 | Bésifloxacine |
| 6 | Thiostrepton | 24 | Lévofloxacine |
| 10 | Amikacine | 25 | La rifabutine |
| 11 | Moxalactam | 26 | Gatifloxacine |
| 12 | Sulfaméthizole | 27 | Épirubicine |
| 13 | Sulfamonométhoxine | 28 | Pamoate de pyrvinium |
| 14 | Céfoxitine | 29 | Moxifloxacine |
| 15 | Novobiocine | 30 | Troleandomycine |
| 16 | Cefmétazole | 31 | Lincomycine |
| 17 | Roxithromycine | 32 | La spiramycine |
Tableau 1 : Liste des composés testés dans le test d’infection. Les composés validés à l’étape 4 ont été testés dans le test d’infection (étape 5).
La toxicité des composés vis-à-vis des macrophages infectés par Mab a été le premier paramètre évalué. Le seuil établi pour juger un composé toxique ou non toxique était de 85 % de macrophages viables (figure 5). Sur les vingt-huit composés testés, 4, 5, 6, 27 et 28 ont été jugés toxiques. (Figure 5). Par conséquent, ces cinq composés ont été exclus de l’évaluation suivante de l’activité intramacrophage.
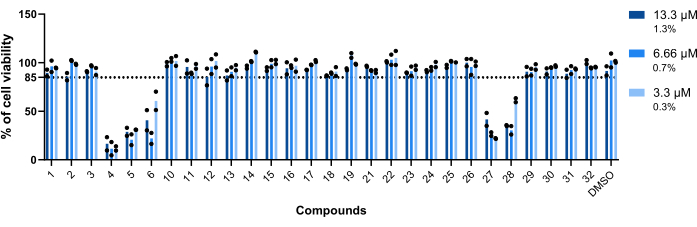
Figure 5 : Toxicité des occurrences vis-à-vis des macrophages infectés par Mab. Les BMM de souris BALB/c ont été infectées par Mab (MOI = 1) et incubées avec chaque coup précédemment identifié à 13,3 μM, 6,66 μM et 3,3 μM (1,3 %, 0,7 % et 0,3 % de DMSO, respectivement) pendant 48 h à 37 °C avec 7 % de CO2. Les cellules ont été imagées à l’aide d’un microscope à fluorescence de dépistage à haut contenu, en utilisant le nombre de noyaux (colorés avec du DAPI) pour mesurer la viabilité cellulaire. Le graphique montre les pourcentages de macrophages infectés viables traités par rapport aux macrophages infectés viables non traités de deux expériences indépendantes. Des symboles ronds représentent la viabilité de la cellule pour chaque essai. Les barres représentent la moyenne de deux expériences indépendantes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Pour inférer l’activité intramacrophagique des vingt-trois composés restants contre les anticorps mabiques intracellulaires (figure 6), la formule de la mycocharge (expliquée précédemment à l’étape 6) a été utilisée pour obtenir le pourcentage de viabilité mycobactérienne normalisé par rapport aux macrophages infectés non traités. La plupart des composés ont perdu leur potentiel thérapeutique contre les mycobactéries internalisées (figure 6) par rapport au test de validation des résultats (figure 4), car le nombre de résultats est passé de vingt-cinq à six à la concentration testée la plus élevée. Étonnamment, les trois composés qui ont augmenté la viabilité du Mab planctonique par rapport aux bactéries non traitées (30, 31 et 32 ; Les figures 3 et 4) ont montré une activité antimycobactérienne contre les anticorps monoclonaux intracellulaires, les composés 30 et 32 présentant une différence statistique significative par rapport au DMSO, même à 3,3 μM dans le cas du composé 32 (figure 6). Les mycobactéries traitées avec les composés 11 et 23 ont montré une viabilité <50 % à 13,3 μM ; cependant, ce n’était pas significativement différent de celui du témoin DMSO (figure 6). Les composés 21, 26 et 29 étaient suffisamment puissants à 13,3 μM pour justifier une différence statistique significative, 29 étant les plus actifs (Figure 6). Enfin, les composés 17, 22 et 25 étaient extrêmement puissants dans toutes les concentrations testées contre les mycobactéries internalisées. Il s’agit de la roxithromycine, de la clarithromycine et de la rifabutine, respectivement (tableau 1). Des trois composés, la clarithromycine était le plus actif contre Mab, la viabilité mycobactérienne n’ayant jamais dépassé 10 %, présentant une valeur p <0,0001 dans toutes les concentrations testées par rapport au DMSO (figure 6).
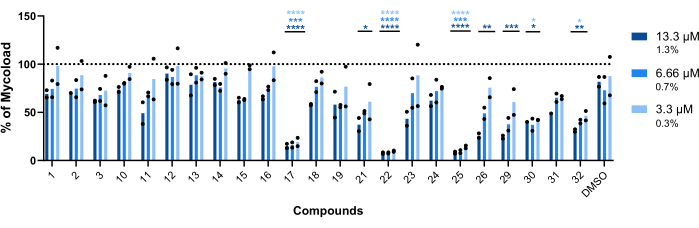
Figure 6 : Activité intramacrophagique des occurrences contre les macrophages infectés par Mab. Les BMM de souris BALB/c ont été infectées par Mab (MOI = 1) et incubées avec chaque résultat précédemment identifié à 13,3, 6,66 et 3,3 μM (1,3, 0,7 et 0,3 % de DMSO, respectivement) pendant 48 h à 37 °C avec 7 % de CO2. Les cellules ont été imagées à l’aide d’un microscope à fluorescence de dépistage à haut contenu, et le signal fluorescent a été utilisé pour calculer la mycocharge (voir l’étape 6). Le graphique montre les pourcentages de Mycoload trouvés dans les macrophages infectés traités par rapport aux macrophages infectés non traités dans deux expériences indépendantes. Les statistiques ont été réalisées à l’aide de l’ANOVA à deux facteurs avec le test de comparaison multiple de Dunnet ; *, p < 0,05 ; **, p < 0,01 ; , p. < 0,001 ; , p < 0,0001 par rapport au DMSO (contrôle des solvants). Les astérisques suivent le même code couleur pour chaque concentration que la légende du graphique. Des symboles ronds représentent la Mycoload pour chaque dosage. Les barres représentent la moyenne de deux expériences indépendantes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire 1 : Exemple de disposition d’une plaque à 384 puits utilisée dans l’évaluation du facteur Z. Blanc - puits vides (milieu de croissance liquide uniquement) ; jaune - contrôle positif (bactéries traitées avec un antibiotique) ; rouge - contrôle négatif (bactéries non traitées). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Exemple d’une disposition de plaque à 384 puits utilisée dans le criblage de bibliothèque. Blanc - puits vides (milieu de croissance liquide uniquement) ; vert - contrôle des solvants ; rouge - contrôle négatif (bactéries non traitées) ; bleu - composés à cribler. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Exemple d’une disposition de plaque de 384 puits utilisée pour la validation des résultats. Blanc - puits vides (milieu de croissance liquide uniquement) ; vert - contrôle des solvants (en doubles) ; rouge - contrôle négatif (bactéries non traitées) ; bleu - composés à cribler (en doublets). Les couleurs délavées représentent des dilutions en série de 1:2. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4 : Exemple d’une disposition de plaque à 96 puits utilisée pour un test d’infection. Blanc - puits vides (eau pour éviter l’évaporation) ; rouge - contrôle négatif (macrophages non traités) ; jaune - contrôle positif (macrophages traités avec un antibiotique) ; vert - contrôle des solvants ; bleu - composés à tester. Les couleurs délavées représentent des dilutions en série de 1:2. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5 : Protocole d’analyse d’images. Un protocole détaillé d’analyse d’images est utilisé dans ce travail, qui peut être adapté à des logiciels open source. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ce protocole décrit un pipeline de dépistage de médicaments contre le Mab à l’aide de souches 10 développées en interne. À l’aide de la robotique de manipulation des liquides, de la microscopie et de l’analyse automatisées et des souches à double rapporteur, la viabilité bactérienne est rapidement évaluée à l’aide de la luminescence ou de la fluorescence sans ajouter de réactifs ni effectuer d’étapes supplémentaires. Cette approche réduit le temps et la variabilité entre les essais, ce qui est un avantage significatif si l’on considère l’objectif des tests HTS.
Lors du criblage de milliers de composés, un médicament efficace doit être facilement identifié. À cette fin, le facteur Z' est souvent utilisé pour mesurer la taille de l’effet statistique, en déduisant les performances du test. Si le facteur Z > de 0,5, les conditions testées sont optimales pour distinguer les populations traitées des populations non traitées13. Les conditions testées ont donné des facteurs Z > 0,6 (figure 2), prouvant statistiquement qu’ils pouvaient être appliqués à une campagne de dépistage. Cette étape est cruciale pour garantir l’efficacité du dépistage.
Ainsi, un protocole HTS a été développé pour détecter l’activité antimicrobienne de milliers de composés contre le Mab en croissance planctonique. Comme le Mab est un agent pathogène facultatif intracellulaire, le protocole conçu permet également de cribler l’activité antimicrobienne intracellulaire contre la même souche bactérienne - un avantage essentiel. De plus, la toxicité envers les cellules hôtes peut également être évaluée. Ainsi, une approche en plusieurs étapes du criblage des médicaments contre le Mab est décrite, en utilisant différents dispositifs expérimentaux pour évaluer l’activité antimicrobienne ainsi que la cytotoxicité, augmentant ainsi les chances de succès. En guise de preuve de concept, une chimiothèque comprenant 1280 composés a été criblée.
Au total, trente-trois résultats positifs ont été recensés (figure 3). Parmi ceux-ci, trois ont considérablement augmenté la viabilité mycobactérienne dans les cultures liquides (composés 30, 31 et 32 ; Figure 3 et Figure 4). Il convient de noter que ces composés peuvent interférer avec l’émission de luminescence sans affecter la viabilité bactérienne. Lorsqu’ils ont été testés contre des mycobactéries internalisées, ces composés ont montré une activité antimicrobienne (Figure 6), démontrant une efficacité plus élevée contre les Mab après l’internalisation de la cellule hôte. Ces composés ont été identifiés comme étant la troleandomycine (30), la spiramycine (32) et la lincomycine (31 ; Tableau 1). Les deux premiers sont des macrolides, une classe d’antibiotiques utilisés pour traiter les infections mycobactériennes16, et le second est un lincosamide, un antibiotique dont le mécanisme d’action est similaire à celui des macrolides17. Il a été signalé que Mab est particulièrement résistant à un autre lincosamide, la clindamycine, dans les cultures liquides et solides18. Néanmoins, des propriétés immunomodulatrices et anti-inflammatoires ont été associées aux macrolides19,20 et aux lincosamides21, ce qui pourrait expliquer l’augmentation de l’activité antimicrobienne contre les mycobactéries internalisées (Figure 6).
Sur les trente occurrences restantes, onze réduisent de >90 % la viabilité mycobactérienne à toutes les concentrations (figure 4). Étant donné qu’un test HTS typique a un taux de réussite attendu de ~1%22, le protocole développé est en accord avec ce qui est généralement observé. Néanmoins, plusieurs autres composés étaient encore actifs, et vingt-huit ont poursuivi le test d’infection.
Parmi les composés identifiés, cinq ont été jugés toxiques (figure 5) : la daunorubicine (4), la doxorubicine (5), l’épirubicine (27), le thiostrepton (6) et le pamoate de pyrvinium (28 ; Tableau 1). Les trois premiers sont des agents antinéoplasiques 23,24,25, donc sans surprise, ils sont toxiques pour les cellules de mammifères utilisées dans cet essai. Le pamoate de pyrvinium a été utilisé pendant de nombreuses années comme anthelminthique efficace ; Cependant, depuis 2004, il a également été associé à des activités antinéoplasiques26. Enfin, le thiostrepton est un oligopeptide souvent utilisé en médecine vétérinaire, jamais approuvé pour une utilisation chez l’homme27. L’activité de ce médicament contre les cellules cancéreuses du sein a été rapportée28. L’activité intramacrophagique du thiostrepton n’a pas été évaluée en raison de sa toxicité sur les macrophages dérivés de la moelle osseuse (figure 5). Cependant, il a été démontré que le thiostrepton est efficace à 5 μM sur les cellules THP-129 infectées par Mab. Les résultats rapportés contre les bactéries planctoniques29 sont similaires à ceux obtenus dans ce dépistage, le thiostrepton étant extrêmement puissant (Figure 4).
La plupart des composés dont l’activité intramacrophagique a été testée n’ont montré aucun potentiel thérapeutique (Figure 6). La croissance intracellulaire bactérienne est considérablement différente de celle des cultures planctoniques. Dans ce dernier, un contact direct entre les bactéries et les médicaments est toujours possible. Dans le premier cas, en raison de l’internalisation par les cellules hôtes, plusieurs membranes de l’hôte agissent comme des barrières physiques que les médicaments doivent transposer pour atteindre la cible, ce qui peut aider à expliquer la diminution de l’activité antimicrobienne de la plupart des hits. À la concentration la plus élevée, soit 13,3 μM, trois composés présentaient une puissance suffisante pour être statistiquement différents du témoin DMSO (figure 6) : le cefdinir (21), la gatifloxacine (26) et la moxifloxacine (29 ; Tableau 1). Alors que la moxifloxacine est déjà utilisée comme médicament antituberculeux de deuxième intention16, le cefdinir est couramment utilisé pour traiter plusieurs infections bactériennes des voies respiratoires, telles que la pneumonie30. Cependant, son activité contre M. tuberculosis31 et Mab a été signalée, montrant un puissant effet synergique avec un carbapénème contre ce dernier32. La gatifloxacine est une fluoroquinolone, et son activité a été signalée contre plusieurs mycobactéries dans le passé33,34. Les trois composés les plus actifs de cet essai (figure 6) étaient la roxithromycine (17), la clarithromycine (22) et la rifabutine (25 ; Tableau 1), qui sont extrêmement puissants à toutes les concentrations. Les deux premiers sont des macrolides, tandis que la rifabutine est une rifamycine, les deux classes servant de base au traitement contre de nombreuses infections mycobactériennes16.
Ce criblage s’appuie sur un équipement de manipulation de liquides spécifique et coûteux pour réduire la variabilité entre les dosages. Bien qu’il soit extrêmement reproductible, le manipulateur de liquides ne dispose pas d’un transfert sans contact de composés, comme un transfert acoustique. Ainsi, certains composés adhérents peuvent rester attachés aux broches métalliques entre les transferts, se répercutant sur les puits suivants, entraînant des faux positifs pendant la phase de criblage - c’est ce qui s’est passé avec le composé 33. C’est pourquoi la validation du dépistage est si essentielle à son succès, car elle permet de s’assurer qu’aucun faux positif ne se poursuit aux étapes suivantes du processus de dépistage des médicaments. Le test d’infection utilise un système d’imagerie à haut contenu avec un mode confocal pour obtenir la meilleure définition possible des régions bactériennes. Néanmoins, il est toujours possible de l’utiliser sans mode confocal, bien qu’avec une perte de définition et éventuellement un obstacle à l’identification des régions bactériennes. Ce protocole tire parti du logiciel d’analyse simple et convivial intégré au système d’imagerie ; cependant, des logiciels open source peuvent être utilisés selon le protocole (Figure supplémentaire 5). En fin de compte, bien qu’il n’y ait pas de réorientation des médicaments pour traiter les infections à Mab, ces résultats sont extrêmement importants car ils valident le protocole établi, qui peut être étendu à de plus grandes bibliothèques. Il est important de noter que nous pensons que ce protocole peut être adapté à n’importe quelle bactérie, à condition que des lectures fluorescentes ou luminescentes soient disponibles. Ainsi, ce travail contribue de manière significative au domaine de la découverte de médicaments, en fournissant les outils nécessaires pour aider à lutter contre l’un des plus grands problèmes de santé publique - les bactéries résistantes aux antibiotiques - et en particulier un agent pathogène presque intraitable - Mycobacterium abscessus.
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Ce travail est financé par des fonds nationaux portugais par l’intermédiaire de FCT - Fundação para a Ciência e a Tecnologia, I.P, dans le cadre du projet PTDC/BIA-MIC/3458/2020 (DOI : 10.54499/PTDC/BIA-MIC/3458/2020) et des bourses de doctorat 2021.07335.BD à GSO et UI/BD/150830/2021 à CMB ; FWO - Fondation pour la recherche Flandre, subvention n° 1S68720N ; Innovative Medicine Initiative 2 Call 16 (IMI2-Call 16) proposition RespiriTB sous le numéro d’accord 853903. Les auteurs remercient le soutien de la Plateforme Scientifique i3S BioSciences Screening, membre des infrastructures nationales PT-OPENSCREEN (NORTE-01-0145-FEDER-085468) et PPBI - Plateforme Portugaise de Bioimagerie (PPBI-POCI-01-0145-FEDER-022122).
matériels
| Name | Company | Catalog Number | Comments |
| 10% Neutral buffered formalin | Bio Optica | 05-01005Q | fixation solution |
| 384-well black round bottom polypropylene not treated microplate | Corning | 3658 | White microplates can be used; polystyrene can be used |
| 50 TS Washer | BioTek | ||
| Breathe-Easy sealing membrane | Sigma-Aldrich | Z380059-1PAK | |
| cell::explorer | Revvity | equipment handling robot | |
| Clarithromycin | Sigma-Aldrich | A3487-100MG | Dissolved in DMSO to the desired concentration |
| DMSO | Fisher Scientific | D/4121/PB15 | Dilute in liquid growth medium to the desired concentration |
| Harmony 5.1 | Revvity | analysis software | |
| Heracell VIOS 160i CO2 Incubator, 165 L | Thermo Scientific | cell incubator | |
| JANUS with 4 Tip Arm and MDT Arm | Revvity | liquid handler | |
| KB 8182 | Termaks | incubator with agitation | |
| Libra S4+ | Biochrom | ||
| Multidrop Combi Reagent Dispenser | Thermo Scientific | ||
| Opera Phenix Plus | Revvity | imaging system | |
| PhenoPlate | Revvity | 6055302 | Necessary for high-content microscopes |
| Phosphate Buffer Saline (PBS) 1x | NA | NA | washing solution |
| Prestwick Chemical libraries | Commercial chemical library intended for drug repurposing | ||
| Shaking incubator 3031 | GFL | ||
| VICTOR Nivo | Revvity | ||
| Cell culture medium: | When needed, add 10% of supplement | ||
| DMEM | Gibco | 10938-025 | |
| Fetal Bovine Serum (FBS) | Biowest | S1810 | |
| HEPES Buffer Solution (1 M) | Gibco | 15630-056 | |
| LCCM | NA | NA | Conditioned media from L929 cell line that releases Macrophage colony stimulating factor (M-CSF); cell culture medium supplement |
| L-glutamin (200 mM) | Thermo Scientific | 25030024 | |
| Sodium Pyruvate 100 mM (100X) | Gibco | 11360-039 | |
| Infection washing solution | |||
| Apirogenic water | Multiple suppliers available | ||
| Hank's Balanced Salt Solution 10x | Gibco | 14060-040 | Dilute to 1x with apirogenic water |
| Permeabilising Solution | PBS with 0.1% Triton X100 | ||
| PBS 1x | NA | NA | |
| Triton X100 | Sigma-Aldrich | T8787 | Other suppliers available |
| Staining solution | PBS with DAPI (500 ng/mL) and HCS Far Red (10 ng/mL) | ||
| DAPI | Sigma-Aldrich | D9564 | |
| HCS Far Red | Invitrogen | H32721 | |
| PBS 1x | NA | NA | Multiple suppliers available |
| Liquid growth medium | When needed, add 10% supplement | ||
| Middlebrook 7H9 Broth | BD Difco | 271310 | |
| Tween80 | Sigma-Aldrich | P4780 | 0.05% final concentration. Other suppliers available |
| Liquid growth medium supplement: | |||
| Bovine Serum Albumin | Fisher Scientific | BP9702-100 | |
| D-Glucose anhydrous | Fisher Scientific | G/0450/60 |
Références
- Victoria, L., Gupta, A., Gomez, J. L., Robledo, J. Mycobacterium abscessus complex: A review of recent developments in an emerging pathogen. Front Cell Infect Microbiol. 11, 659997(2021).
- Luthra, S., Rominski, A., Sander, P. The role of antibiotic-target-modifying and antibiotic-modifying enzymes in Mycobacterium abscessus drug resistance. Front Microbiol. 9, 2179(2018).
- Nathavitharana, R. R., Strnad, L., Lederer, P. A., Shah, M., Hurtado, R. M. Top questions in the diagnosis and treatment of pulmonary M. abscessus disease. Open Forum Infect Dis. 6 (7), ofz221(2019).
- Cristancho-Rojas, C., et al. Epidemiology of Mycobacterium abscessus. Clin Microbiol Infect. 30 (6), 712-717 (2024).
- Hua, Y., et al. Drug repositioning: Progress and challenges in drug discovery for various diseases. Eur J Med Chem. 234, 114239(2022).
- Doan, T. L., Pollastri, M., Walters, M. A., Georg, G. I. The future of drug repositioning. Annual reports in medicinal chemistry. Macor, J. E. , Academic Press. 385-401 (2011).
- Bento, C. M., Gomes, M. S., Silva, T. Evolution of antibacterial drug screening methods: Current prospects for mycobacteria. Microorganisms. 9 (12), 2562(2021).
- Attene-Ramos, M. S., Austin, C. P., Xia, M. High throughput screening. Encycl Toxicol. 3, 916-917 (2014).
- Gupta, R., Netherton, M., Byrd, T. F., Rohde, K. H. Reporter-based assays for high-throughput drug screening against Mycobacterium abscessus. Front Microbiol. 8, 2204(2017).
- Bento Clara, M., et al. Characterization of novel double-reporter strains of Mycobacterium abscessus for drug discovery: A study in mScarlet. Microbiol Spectr. , e0036224(2024).
- Kolbe, K., et al. Development and optimization of chromosomally-integrated fluorescent Mycobacterium tuberculosis reporter constructs. Front Microbiol. 11, 591866(2020).
- Lopez-Roa, P., Esteban, J., Munoz-Egea, M. C. Updated review on the mechanisms of pathogenicity in Mycobacterium abscessus, a rapidly growing emerging pathogen. Microorganisms. 11 (1), 90(2022).
- Bray, M. A., Carpenter, A. Advanced assay development guidelines for image-based high content screening and analysis in Assay guidance manual. Markossian, S., et al. , Eli Lilly & Company and the National Center for Advancing Translational Sciences, Bethesda (MD). (2004).
- Clinical and Laboratory Standards Institute. Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically; approved standard. CLSI document M07-A10. , 10th ed, Wayne, PA. (2015).
- Silva, T., et al. Lactoferricin peptides increase macrophages' capacity to kill Mycobacterium avium. mSphere. 2 (4), e00301-e00317 (2017).
- Bento, C. M., Gomes, M. S., Silva, T. Looking beyond typical treatments for atypical mycobacteria. Antibiotics (Basel). 9 (1), 18(2020).
- Spizek, J., Rezanka, T. Lincosamides: Chemical structure, biosynthesis, mechanism of action, resistance, and applications. Biochem Pharmacol. 133, 20-28 (2017).
- Hurst-Hess, K. R., Rudra, P., Ghosh, P. Ribosome protection as a mechanism of lincosamide resistance in Mycobacterium abscessus. Antimicrob Agents Chemother. 65 (11), e0118421(2021).
- Kang, J. K., Kang, H. K., Hyun, C. G. Anti-inflammatory effects of spiramycin in lps-activated raw 264.7 macrophages. Molecules. 27 (10), 3202(2022).
- Zimmermann, P., Ziesenitz, V. C., Curtis, N., Ritz, N. The immunomodulatory effects of macrolides-a systematic review of the underlying mechanisms. Front Immunol. 9, 302(2018).
- Pradhan, S., Madke, B., Kabra, P., Singh, A. L. Anti-inflammatory and immunomodulatory effects of antibiotics and their use in dermatology. Indian J Dermatol. 61 (5), 469-481 (2016).
- Volochnyuk, D. M., et al. Evolution of commercially available compounds for hts. Drug Discov Today. 24 (2), 390-402 (2019).
- Sritharan, S., Sivalingam, N. A comprehensive review on time-tested anticancer drug doxorubicin. Life Sci. 278, 119527(2021).
- Arwanih, E. Y., Louisa, M., Rinaldi, I., Wanandi, S. I. Resistance mechanism of acute myeloid leukemia cells against daunorubicin and cytarabine: A literature review. Cureus. 14 (12), e33165(2022).
- Khasraw, M., Bell, R., Dang, C. Epirubicin: Is it like doxorubicin in breast cancer? A clinical review. Breast. 21 (2), 142-149 (2012).
- Schultz, C. W., Nevler, A. Pyrvinium pamoate: Past, present, and future as an anti-cancer drug. Biomedicines. 10 (12), 3249(2022).
- Bailly, C. The bacterial thiopeptide thiostrepton. An update of its mode of action, pharmacological properties and applications. Eur J Pharmacol. 914, 174661(2022).
- Kwok, J. M., et al. Thiostrepton selectively targets breast cancer cells through inhibition of forkhead box m1 expression. Mol Cancer Ther. 7 (7), 2022-2032 (2008).
- Kim, T. H., et al. Thiostrepton: A novel therapeutic drug candidate for Mycobacterium abscessus infection. Molecules. 24 (24), 4511(2019).
- Perry, C. M., Scott, L. J. Cefdinir: A review of its use in the management of mild-to-moderate bacterial infections. Drugs. 64 (13), 1433-1464 (2004).
- Ramon-Garcia, S., et al. Repurposing clinically approved cephalosporins for tuberculosis therapy. Sci Rep. 6, 34293(2016).
- Kumar, P., et al. Mycobacterium abscessus l,d-transpeptidases are susceptible to inactivation by carbapenems and cephalosporins but not penicillins. Antimicrob Agents Chemother. 61 (10), e00866-e00917 (2017).
- Alvirez-Freites, E. J., Carter, J. L., Cynamon, M. H. In vitro and in vivo activities of gatifloxacin against Mycobacterium tuberculosis. Antimicrob Agents Chemother. 46 (4), 1022-1025 (2002).
- Braback, M., Riesbeck, K., Forsgren, A. Susceptibilities of Mycobacterium marinum to gatifloxacin, gemifloxacin, levofloxacin, linezolid, moxifloxacin, telithromycin, and quinupristin-dalfopristin (synercid) compared to its susceptibilities to reference macrolides and quinolones. Antimicrob Agents Chemother. 46 (4), 1114-1116 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon