Method Article
Obtención de imágenes de microdominios Ca2+ iniciales en células T primarias
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, proporcionamos un protocolo integral para resolver las señales locales iniciales de Ca2+ , conocidas como microdominios de Ca2+ , en células T primarias murinas y humanas mediante microscopía de fluorescencia. Este protocolo sirve como un recurso valioso para los investigadores que examinan las vías de señalización de Ca2+ dentro de las células inmunitarias y para desentrañar aún más su función.
Resumen
Las señales locales de Ca2+ en menos de un segundo, denominadas microdominios Ca2+ , son señales de Ca2+ altamente dinámicas y de corta duración, que dan lugar a una elevación global [Ca2+]i y ya podrían determinar el destino de una célula T. Tras la activación del receptor de células T, el NAADP se forma rápidamente, uniéndose a las proteínas de unión al NAADP (HN1L/JPT2, LSM12) y sus respectivos receptores (RyR1, TPC2) que se encuentran en las reservas intracelulares deCa2+ , como el RE y los lisosomas, y conduce a la posterior liberación y elevación de [Ca2+]i. Para capturar estas señales de Ca2+ rápidas y dinámicas, desarrollamos una técnica de imagen de alta resolución utilizando una combinación de dos indicadores de Ca2+ , Fluo-4 AM y FuraRed AM. Para el posprocesamiento, se desarrolló un enfoque de detección de microdominios Ca2+ semiautomatizado de código abierto basado en el lenguaje de programación Python. Con este flujo de trabajo, somos capaces de detectar de forma fiable microdominios Ca2+ a nivel subcelular en células T primarias murinas y humanas en vídeos de fluorescencia de alta resolución temporal y espacial. Este método también se puede aplicar a otros tipos de células, como las células NK y las líneas celulares neuronales murinas.
Introducción
La técnica de microscopía de fluorescencia presentada permite la visualización de señales iniciales locales y temporales de calcio (Ca2+) en células T primarias de ratón, denominadas microdominios Ca2+ . Los microdominios de Ca2+ representan eventos de señalización de Ca2+ altamente dinámicos y de corta duración, lo que plantea desafíos para la obtención de imágenes y el análisis efectivos de células vivas1.
Las células T son un desafío para la obtención de imágenes de células vivas debido a las diferencias relativas en la intensidad de la fluorescencia central y periférica, que se pueden atribuir a su forma esférica y su pequeño diámetro de ~ 6-8 μm. Tras la estimulación y la formación de sinapsis inmunitaria, las células T experimentan cambios morfológicos, lo que complica aún más la obtención de imágenes de las células T1. Por lo tanto, se vuelve imperativo emplear un análisis radiométrico, que se logra grabando dos imágenes que representan diferentes propiedades de un colorante Ca2+ o utilizando una combinación de dos tintes Ca2+ . Las características exigentes de los microdominios Ca2+ incluyen su naturaleza rápida, temporal y espacialmente limitada. Para capturar esto, los tintes Ca2+ utilizados deben poseer un alto brillo basal y una alta relación señal-ruido (SNR) para obtener la mayor resolución temporal y espacial posible. Se han logrado resultados óptimos utilizando una combinación del colorante de doble longitud de onda Fura Red y el colorante de una sola longitud de onda Fluo-4. Las células de cocarga con Fluo-4 y Fura Red mitigan los desafíos planteados por el fotoblanqueo fuerte de los tintes de doble longitud de onda de emisión y el retraso temporal asociado con los tintes de doble excitación, lo que garantiza la idoneidad para la adquisición rápida de imágenes. Este enfoque facilita aún más la visualización de cambios en la forma y movimientos sutiles. También se imponen exigencias especiales al sistema de imágenes en términos de resolución espacial para permitir la visualización de señales de Ca2+ que se originan en la apertura de pequeños grupos de canales o incluso canales individuales1.
La señalización de Ca2+ desempeña un papel fundamental en la activación de las funciones inmunitarias dentro de las células T, incluida la formación de sinapsis y la producción y liberación de citocinas 2,3. El destino específico de la célula se regula a través de las señales de Ca2+ pronunciadas y distribuidas localmente, los microdominios Ca2+ 3. En particular, esas señales locales de Ca2+ preceden a un aumento generalizado de los niveles intracelulares de Ca2+ en las células T y la formación de microdominios de Ca2+ depende tanto de la entrada como de la liberación de Ca2+ 1,4,5. Tras la estimulación del receptor de células T (TCR)/CD3, se desencadena la formación de segundos mensajeros liberadores de Ca2+, como el ácido nicotínico adenina dinucleótido fosfato (NAADP), D-mio-inositol 1,4,5-trifosfato (IP3) y ADP ribosa cíclica (cADPR), lo que conduce a un aumento de los niveles intracelulares de Ca2+ hasta 1 μM 6,7. Los eventos tempranos de señalización de Ca2+ están relacionados con la liberación de Ca2+ de las reservas intracelulares de Ca2+, como el retículo endoplásmico (RE), con canales como el receptor de rianodina 1 (RyR1) y el receptor IP3 (IP3R) siendo predominantemente responsables de esta señalización. Posteriormente, esto desencadena la afluencia extracelular de Ca2+ y da como resultado una señal global de Ca2+ a través de la entrada de Ca2+ operada por la tienda (SOCE)8. Además, hay otros canales implicados en la señalización deCa-2+ durante la activación de los linfocitos T9, por ejemplo, los canales P2X4 y P2X7 aseguran la entrada de cationes dependientes de trifosfato de adenosina (ATP), lo que contribuye al aumento del Ca2+ intracelular. Sorprendentemente, los microdominios iniciales de Ca2+ dependientes de la adherencia (ADCM) ya se forman antes de la estimulación de TCR, pero con amplitudes y frecuencias de Ca2+ más bajas. Lo más probable es que estas señales iniciales de Ca2+ independientes del TCR sirvan para la migración de las células T al sitio de la inflamación y preparen a las células T para la reestimulación en el sitio de la infección10,11.
Al desarrollar el método descrito para la obtención de imágenes locales de Ca2+ , hemos obtenido una herramienta adicional para explorar el origen y la importancia de las señales tempranas de Ca2+ en la activación de las células T. Este método permite al usuario detectar señales de Ca2+ más pequeñas, de corta duración y de aparición más rápida de lo que era posible anteriormente. Además, Deconvolución, Análisis, Registro, Seguimiento y Normalización de Formas (DARTS), la canalización de análisis basada en Python, permite compartir las herramientas de análisis con un público más amplio12.
Protocolo
Todos los experimentos con animales fueron aprobados y realizados de acuerdo con las directrices de bienestar animal del Comité Institucional de Cuidado y Uso de Animales del Centro Médico Universitario de Hamburgo-Eppendorf.
1. Aislamiento de células T primarias de ratón de ganglios linfáticos y bazo
- Extraiga los bazos/ganglios linfáticos en condiciones estériles de acuerdo con las pautas éticas y colóquelos en un tubo con medio Clicks helado (10% Suero fetal de ternero (FCS), 2 mM L-Glutamina, Penicilina/Estreptomicina (P/S) 100 U/mL, 50 nM β-Mercaptoetanol).
NOTA: Las células T de ratón tipo salvaje y KO se aíslan de la misma manera. - Enfríe la centrífuga a 4 °C.
- Coloque el bazo y los ganglios linfáticos en un filtro de células de 70 μm en una placa de Petri estéril y agregue medio de aislamiento del bazo (medio RPMI + 7,5 % NCS + 1 % P/S) hasta un volumen total de 20 mL.
- Rompe el bazo a fondo pero con suavidad con un mortero plástico. Posteriormente, transfiera las células a un tubo de centrífuga de 50 ml.
NOTA: Ahora debe haber un volumen total de 20 mL en el tubo de centrífuga de 50 mL. El bazo y los ganglios linfáticos deben estar completamente alterados. Trabaja en hielo a partir de este punto. Mantener las células en hielo ralentiza su metabolismo y evita la muerte celular, asegurando que permanezcan viables y funcionalmente intactas. Esto ayuda a preservar su estado fisiológico durante un período de tiempo más largo, lo que es especialmente importante para experimentos posteriores precisos. - Centrifugar la suspensión celular a 300 x g durante 5 min a 4 °C.
- Deseche el sobrenadante y asegúrese de resuspender el pellet resultante en 2 mL de solución salina tamponada con fosfato (DPBS) de Dulbecco (sin Ca2+ y Mg2+). Transfiera a un nuevo tubo de centrífuga de 12 mL.
2. Selección negativa de linfocitos T CD4+
NOTA: Para la selección negativa de linfocitos T CD4+ , se utiliza un kit de aislamiento de linfocitos T que contiene un bloqueador de FcR, anticuerpos biotinilados contra linfocitos T no CD4+ y partículas magnéticas recubiertas de estreptavidina.
- Añada 20 μL/mL de bloqueador de FcR de ratón del kit de aislamiento de células T a la suspensión celular.
- Además, agregue 100 μL de los cócteles de aislamiento de células T CD4+ de ratón a una concentración de 50 μL/mL, mezcle bien e incube durante 10 minutos a temperatura ambiente (RT).
- Partículas magnéticas recubiertas de estreptavidina durante 30 s, añadir 150 μL a la suspensión celular, mezclar bien e incubar durante 2 min 30 s a RT.
- Agregue DPBS a la suspensión celular, llenándola hasta un volumen total de 7 mL. Asegúrese de pipetear hacia arriba y hacia abajo suavemente, luego incube en el imán durante 2 minutos y 30 segundos en RT.
- A continuación, recoja suavemente las células T enriquecidas en el sobrenadante y transfiéralas a un nuevo tubo de centrífuga de 12 ml.
- Centrifugar las celdas a 300 x g en RT durante 5 min.
- Deseche el sobrenadante.
- Vuelva a suspender las células T CD4+ aisladas y enriquecidas en 1 ml de DPBS, pipeteando minuciosamente hacia arriba y hacia abajo. Incubar la mezcla durante 2 min 30 s en el imán en RT para asegurarse de que no se contaminen las perlas.
- Recoja el sobrenadante, transfiéralo a un nuevo tubo de centrífuga y cuente las células T enriquecidas.
- Para contar las células, tiña las células con azul de tripán y cuenta con un contador de células automatizado.
NOTA: Es mejor cargar directamente las células T CD4+ aisladas y obtener imágenes de ellas el mismo día del aislamiento. Sin embargo, los linfocitos T murinos primarios pueden cultivarse sin ningún tipo de estimulación durante la noche en una incubadora a 37 °C con 5 % de CO2 en un medio de aislamiento (RPMI 1640 + 10% FCS + P/S).
3. Carga de células T CD4+ primarias de ratón
NOTA: Para medir las concentraciones de Ca2+ citosólico libre, se emplean colorantes fluorescentes y permeables a la membrana en los experimentos de imágenes de Ca2+ . Una combinación de éster fluo-4-acetoximetilo (AM) y el colorante radiométrico Fura Red-AM sirve como indicador para la detección rápida de señales locales de Ca2+ . Asegúrate de trabajar en la oscuridad mientras usas tintes fluorescentes.
- Centrifugar 2-5 x 106 celdas durante 5 min, 300 x g, desechar el sobrenadante y resuspender en 480 μL de medio de celdas T (RPMI 1640, 10% FCS) con 10 μM Fluo-4-AM (Stock: [1 mM]), y 20 μM Fura Red-AM (Stock: [4 mM]).
- Incubar inicialmente durante 20 minutos en RT debajo del banco en la oscuridad, cubriendo el halcón con papel de aluminio.
NOTA: No exceda el primer tiempo de incubación, ya que los indicadores de Ca2+ están diluidos en DMSO, lo que podría ser perjudicial para las células a un tiempo de exposición más largo en el volumen total bajo. Mantenga las celdas protegidas de la luz después de la carga debido a la sensibilidad a la luz de los tintes. - Al final de la incubación inicial de 20 minutos, agregue 2 ml de medio de células T a las células y continúe la incubación durante 30 minutos adicionales en la oscuridad a RT.
- Centrifugar la suspensión celular a 300 x g durante 5 min en RT.
- Deseche el sobrenadante y limpie el pellet lavándolo con 2 mL de tampón de medición de Ca2+ (140 mM de NaCl, 5 mM de KCl, 1 mM de MgSO4, 1 mM de CaCl2, 20 mM de HEPES, 1 mM de NaH2PO4, 5 mM de glucosa, pH 7,4) seguido de una centrifugación a 300 x g durante 5 min a RT.
- Por último, deseche el sobrenadante y vuelva a suspenderlo con el tampón de medición Ca2+ . Ajuste el número de celdas a ~ 100,000 celdas por 10 μL.
NOTA: Se utiliza un volumen de 10 μL para una medición. Evite diluir demasiado las células, ya que es posible que sea necesario ajustar aún más el recuento de células bajo el microscopio para alcanzar un óptimo para las condiciones de medición. - Deje que las células se incuben durante ~ 20 minutos para asegurar la infiltración celular completa y la desesterificación de los tintes. Guarde las celdas en hielo y en la oscuridad hasta la medición.
NOTA: Las células T murinas primarias cargadas se pueden usar hasta ~ 4 h después de la carga.
4. Imágenes locales de Ca2+
- Preparación de portaobjetos
- Recubra los cubreobjetos del microscopio (24 mm x 46 mm) con albúmina sérica bovina (BSA, 5 mg/mL) y poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Deje reposar BSA durante ~ 20 minutos antes de aplicar PLL.
NOTA: El recubrimiento facilita la adhesión de las células. Para una adhesión adecuada de las células, extienda con precisión tanto BSA como PLL hasta que no se vean más rayas. - Para crear cámaras de reacción, pegue juntas tóricas de goma reutilizables a los portaobjetos con una pasta de silicona (Figura 1B, C).
NOTA: Para la adquisición de imágenes se pueden utilizar otros portaobjetos/cámaras o placas, como portaobjetos de 35 mm, cámaras metálicas reutilizables o placas de 8, 24 o 48 pocillos.
- Recubra los cubreobjetos del microscopio (24 mm x 46 mm) con albúmina sérica bovina (BSA, 5 mg/mL) y poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Deje reposar BSA durante ~ 20 minutos antes de aplicar PLL.
- Recubrimiento de perlas magnéticas de proteína G (10 μm de diámetro) con anticuerpos
- Mezcle suavemente la suspensión de perlas magnéticas de proteínas y transfiera 12,5 μL a un tubo nuevo.
- Coloque el tubo en un soporte magnético para eliminar el búfer de almacenamiento y deje que las cuentas migren al imán. Retire suavemente el tampón de almacenamiento pipeteándolo en el lado opuesto del imán.
- Para eliminar cualquier tampón de almacenamiento restante, lave las perlas añadiendo 500 μL de PBS-T (PBS, 0,1% Tween) y vórtice durante 10 s. Vuelva a colocar el tubo en el soporte magnético y retire el tampón.
- Para recubrir las perlas con anticuerpos, resuspenderlas en 7,5 μL de PBS-T y añadir 5 μL de anti-CD3 (0,5 mg/mL) y anti-CD28 (0,5 mg/mL), respectivamente. Incubar durante 30-60 min con mezcla continua en RT.
- Lave las perlas recubiertas tres veces con 500 μL de PBS-T, seguido de un lavado una vez con 500 μL de tampón de medición de Ca2+ utilizando el soporte magnético.
- Vuelva a suspender las perlas en 200-400 μL de tampón de medición de Ca2+.
NOTA: Verifique la densidad del cordón durante las mediciones, diluya aún más si es necesario.
- Microscopía de imagen local de Ca2+
NOTA: Las imágenes se realizan utilizando un microscopio de luz de campo claro con un aumento de 100 veces, equipado con una lámpara de arco de xenón como fuente de luz. Los fotogramas se capturan en modo de 14 bits con agrupación doble utilizando una cámara acoplada a la carga que multiplica los electrones. Para registrar y dividir las longitudes de onda de emisión de ambos colorantes, se emplea un módulo de doble vista, con los siguientes filtros en nm (ex, 480/40; bs, 495; em1, 542/50; em2, 650/57). La configuración de imágenes incluye un centro de adquisición y software de imágenes para la adquisición de imágenes.- Coloque 10 μL de las células cargadas en el portaobjetos preparado y deje que se adhieran al portaobjetos durante 3-5 minutos.
- Añada suavemente 80 μL de tampón de medición de Ca2+ al portaobjetos.
- Seleccione la lente de inmersión en aceite 100x y aplique una pequeña gota de aceite de inmersión. Coloque el portaobjetos sobre la mesa del microscopio.
- Ajuste el enfoque en el modo de campo claro, seleccione cuidadosamente un campo de visión con hasta 10 celdas que no estén en contacto entre sí y adquiera una imagen.
NOTA: Las celdas superpuestas y en contacto serán difíciles de analizar más adelante.- Encienda la lámpara, compruebe la fluorescencia y la carga de los linfocitos T, y compare los pilos de ambos canales para comprobar que no están preactivados.
- Asegúrese de tomar fotografías del campo de visión de los canales de campo claro y fluorescencia antes y después de la medición para comprobar el movimiento y la carga de las células.
- Inicie la medición y capture la actividad basal durante 1 minuto con una velocidad de adquisición de 1 fotograma por 5 s.
- Añada 10 μL de compuesto/estimulante (perla o estimulador/inhibidor) después de 1 minuto y mida durante un total de 3 minutos utilizando 40 fotogramas por segundo o la velocidad de fotogramas máxima posible.
NOTA: La adición de cuentas es el paso crucial; Asegúrese de que las cuentas se agreguen cerca de la luz de excitación en el portaobjetos sin mover el portaobjetos. En la Figura 2 se muestran ejemplos representativos de microdominios Ca2+ en linfocitos T CD4+ primarios al contacto con perlas de ratones WT y P2x4-/- y P2x7-/-.
5. Postprocesamiento/análisis de datos
NOTA: Para el procesamiento de imágenes y el análisis de datos, se utiliza la canalización de código abierto DARTS basada en Python. Ha sido desarrollado por Woelk et al.12 a partir de los trabajos de Diercks et al.13.
- Instalación del gasoducto DARTS
- Instale Python 3.10.014, anaconda15 y git16, y clone el repositorio de GitHub usando el comando de terminal git clone: https://github.com/IPMI-ICNS-UKE/DARTS.git
- Cree un entorno de conda con conda create --name DARTS e instale todos los paquetes de Python necesarios con pip install <package>.
NOTA: Siga las instrucciones de instalación en el repositorio de GitHub de DARTS17,18 para obtener información más detallada. - Antes de utilizar Bioformatos, asegúrese de que un entorno de ejecución de Java esté instalado correctamente19.
- Uso de la canalización DARTS
NOTA: Una vez instalado, DARTS se puede iniciar desde la ventana del terminal.- Navegue hasta la copia local del repositorio DARTS (cd path/to/DARTS) que contiene el archivo main.py. Asegúrese de que el entorno de Conda esté activado (Conda activate DARTS).
- Ejecute DARTS escribiendo python main.py en la ventana del terminal y pulsando Intro.
- En la interfaz gráfica de usuario (GUI), especifique el directorio de origen y de resultados, así como el formato de imagen en la sección superior izquierda (consulte la Figura 3).
- Defina las propiedades de medición, como la escala (píxeles por micrómetro), la velocidad de fotogramas, el tipo de celda, etc. La canalización de procesamiento se puede ensamblar en función de los datos de imagen específicos y la pregunta de investigación.
NOTA: Lea más sobre los parámetros de deconvolución, por ejemplo, en la documentación correspondiente20. - Guarde la configuración en la computadora antes de hacer clic en Inicio. Se puede encontrar una descripción más detallada de la GUI en la documentación de DARTS.
- Imágenes de microdominio Ca2+ y contactos de perlas
NOTA: Para la obtención de imágenes locales y la estimulación con perlas y el posterior análisis de imágenes, el usuario debe definir los contactos de perlas específicas de la célula para identificar el punto de inicio (t = 0) del período de medición de interés (ver Figura 4). Un contacto de perla se define como el sitio de contacto de una perla (estimuladora) con una celda de interés. Consiste en la especificación de (1) el tiempo de contacto del cordón, por ejemplo, el marco 300, y (2) la posición de contacto del cordón en relación con el centro de la celda, por ejemplo, 1 en punto o 12 en punto. Con la versión actual de DARTS, el usuario debe definir e ingresar manualmente la información para cada contacto de cordón. Utilice el siguiente procedimiento para definir los contactos del cordón:- Utilice el control deslizante para determinar el tiempo de contacto entre una cuenta y una celda de interés.
- En el menú de opciones de la derecha, seleccione el contacto del talón: x, y, t.
- Seleccione el punto en el lado izquierdo de la imagen donde la celda y la cuenta hacen contacto.
- Seleccione Elegir celda haciendo clic en un punto dentro. Haga clic en la celda estimulada por esta cuenta, preferiblemente en el centro.
- Haga clic en AGREGAR contacto de cuenta.
- Repita los pasos 5.3.1 a 5.3.5 para cada contacto de cordón adicional en este archivo. Una vez que todos los contactos del cordón estén definidos, haga clic en el botón Continuar .
- Continúe con los archivos adicionales. Una vez alcanzado el último archivo, el script iniciará automáticamente el análisis de todos los archivos.
- Análisis de datos
- Encuentre las imágenes de proporción procesadas, los datos de microdominio para cada celda a lo largo del tiempo (incluida la localización, la amplitud, el tamaño), los datos de origen para la proyección de la diana y otros recursos en la carpeta de resultados.
- Para crear dianas, navegue hasta la carpeta /src/analysis/ dentro de la carpeta DARTS (patrón: cd path/to/DARTS/src/analysis) que contiene el script DartboardPlotGUI.py. A continuación, escriba python DartboardPlotGUI.py y pulse Intro.
- Asegúrese de que se proporciona la información necesaria y de que los archivos de hoja de cálculo de la carpeta de resultados se seleccionan con precisión como archivos de origen para la generación de la diana.
Resultados
En este protocolo, describimos un método actualizado para obtener imágenes y analizar los microdominios iniciales de Ca2+ en células T primarias de ratón basado en el trabajo previo de nuestro grupo 1,13. Este enfoque fue fundamental para desentrañar la participación de los canales CRAC como ORAI1, STIM1 y STIM2, así como los canales de liberación intracelulares de Ca2+ como RyR1 en los eventos de señalización tempranos de Ca2+ 4.
Para ello, investigamos la formación espontánea de microdominios Ca2+ mediante imágenes de ratones primarios no estimulados Orai1-/-, Stim1-/-, Stim2-/-, y Ryr1-/- y los comparamos con células T primarias de ratón WT. El análisis de la formación de microdominios Ca2+ abarcó la velocidad de inicio de la señal, la amplitud de Ca2+ y el número de señales por plano confocal. En particular, a excepción de las células T Stim2-/- , todas las células T KO mostraron una disminución significativa de las señales locales de Ca2+ y una concentración basal reducida de Ca2+ citosólico libre basal en comparación con las células WT. Esto nos llevó a concluir que la formación de microdominios Ca2+ está estrechamente relacionada con la interacción de ORAI1, STIM1 y RyR14. Además, identificamos y caracterizamos con éxito microdominios espontáneos de Ca2+ en la membrana plasmática. Estos microdominios de Ca2+ se caracterizaron por una amplitud de Ca2+ de 290 nm ± 12 nm. El uso de un enfoque codificado por colores para las señales de Ca2+ permitió la visualización de microdominios de Ca2+ en toda la célula. Los resultados destacaron aún más la rápida aparición de microdominios de Ca2+ , visibles en milisegundos, y la capacidad de este método para detectar señales de Ca2+ con una longevidad de unos pocos milisegundos4. Estos microdominios espontáneos de Ca2+ se identificaron posteriormente como microdominios de Ca2+ dependientes de la adhesión (ADCM), dependientes no solo de SOCE, sino que también actúan a través de la cascada de señalización FAK/PLC-γ/IP3 10 y la participación de P2X49. Además, esta técnica fue fundamental para confirmar las oxidasas duales 1 y 2 (DUOX1/2) como enzimas productoras de NAADP21 y HN1L/JPT222 como una de las proteínas de unión a NAADP recientemente descubiertas23.
La Figura 2 muestra ejemplos representativos de microdominios Ca2+ en células T CD4+ primarias al entrar en contacto con las perlas de WT, así como en ratones P2x4-/- y P2x7-/-. Las células se cargaron con los colorantes Ca2+ Fluo-4 AM y Fura Red AM y se obtuvieron imágenes a una velocidad de adquisición de 25 ms (40 fotogramas/s). Para imitar la formación de sinapsis de células T, las células se estimularon con perlas recubiertas de anti-CD3/anti-CD28. La formación inicial del microdominio Ca2+ se analizó 1 s antes y hasta 15 s después del contacto con el cordón utilizando la tubería DARTS. Al entrar en contacto con el cordón, la célula WT mostró una rápida formación de microdominios Ca2+ en el primer segundo después de la estimulación en el sitio de contacto con el cordón (Figura 2A). Estos microdominios Ca2+ se expandieron aún más por toda la célula en los siguientes 15 s después del contacto con las perlas. A diferencia de la célula WT, las células P2x4-/- y P2x7-/- (Figura 2B) mostraron una disminución de la formación de microdominiosCa2+ tras la estimulación de las perlas, así como para las P2x4-/-un nivel basal más bajo antes del contacto con las perlas. Estos hallazgos representativos están en línea con los resultados publicados previamente por Brock et al.9, que indican la formación de microdominios Ca2+ en las células WT T directamente después del contacto con las cuentas durante 15 s y señales más bajas por trama en las células P2x4-/- y P2x7-/- . Además, la amplitud en las células P2x4-/-se redujo significativamente, lo que establece aún más el papel de la señalización purinérgica en los microdominios Ca2+ dependientes de la adhesión.
Además, este método también se puede utilizar para visualizar los microdominios Ca2+ iniciales en las células T CD4+ humanas primarias (Figura 3). De acuerdo con los linfocitos T murinos primarios, los microdominios Ca2+ iniciales se evocan en el sitio de contacto con la perla. Sin embargo, la respuesta general de Ca2+ parece ocurrir en una escala de tiempo diferente en comparación con las células T CD4+ murinas.
El análisis de las señales locales de Ca2+ de forma manual no es factible, ya que es bastante laborioso y subjetivo para el investigador individual. Por lo tanto, previamente desarrollamos un algoritmo en MATLAB Simulink utilizando sus cajas de herramientas de procesamiento y optimización de imágenes para el postprocesamiento13 para analizar microdominios locales de Ca2+ .
Recientemente, desarrollamos una nueva línea de posprocesamiento de código abierto llamada DARTS para el análisis de microdominios Ca2+ en imágenes de células vivas de alta resolución utilizando la plataforma de software Python12. Aquí, se pueden seleccionar diferentes algoritmos de deconvolución, dependiendo de la preferencia del usuario, se puede realizar una normalización de la forma de la celda para compensar los cambios morfológicos en la forma de la celda, y se pueden definir parámetros específicos del microscopio y la medición (por ejemplo, escala, velocidad de fotogramas, tiempo medido) (Figura 4).
Después de seleccionar los parámetros para el análisis de microdominios Ca2+ , se abre una segunda ventana emergente para cada medición individual para definir el contacto del cordón (Figura 5). Para definir el contacto del cordón, el usuario puede desplazarse manualmente por el archivo tiff utilizando el control deslizante y seleccionar el marco de contacto del cordón individualmente. El contacto del cordón se selecciona haciendo clic en el sitio de contacto del cordón (Figura 5, el contacto del cordón y el cordón se indica con un anillo y una flecha amarillas), así como la selección de celdas. Este paso debe repetirse para cada celda de interés. Finalmente, se aplica el posprocesamiento automatizado de imágenes y los datos de resultados se resumen y guardan en una hoja de cálculo.
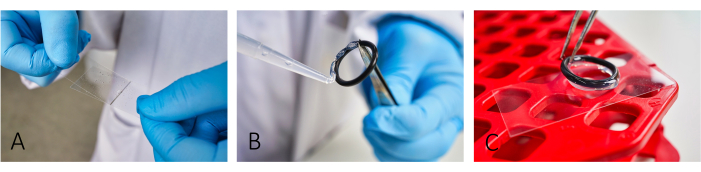
Figura 1: Flujo de trabajo de la preparación de portaobjetos para la obtención de imágenes. (A) Agregue y extienda BSA y PLL en el portaobjetos usando un segundo cubreobjetos de vidrio. (B,C) Para construir una cámara, pegue las juntas tóricas de goma con grasa de silicona en la corredera. Asegúrese de que todo el anillo esté cubierto con una fina capa de grasa para tener un aislamiento adecuado de la cámara. Haga clic aquí para ver una versión más grande de esta figura.
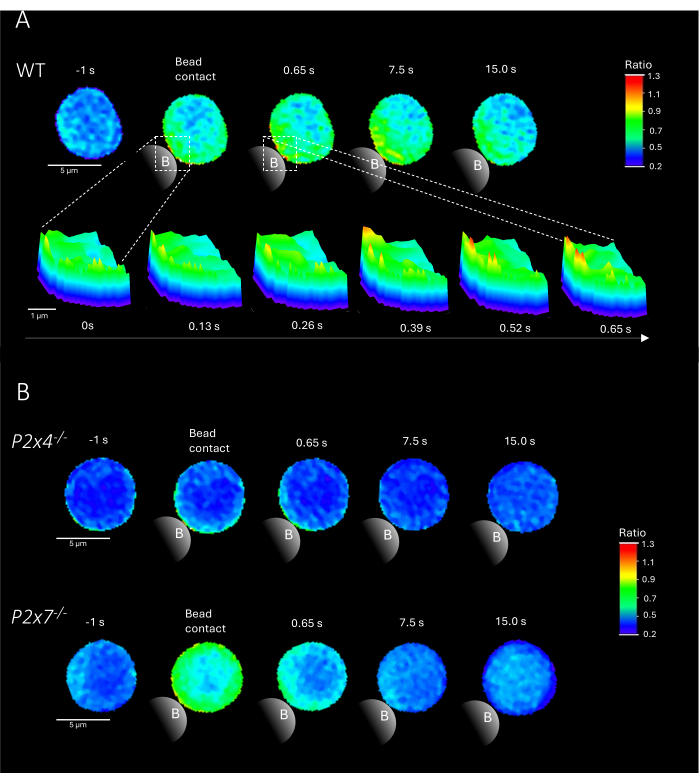
Figura 2: Células representativas de microdominios Ca2+ dependientes del receptor de células T en un linfocito T CD4+ de tipo salvaje (WT) (A) murino primario de tipo salvaje (W), P2x4-/- o P2x7-/-(B). Las células T CD4+ se aislaron negativamente y se cargaron con Fluo-4 AM y Fura Red, como se describió anteriormente. Las células T se analizaron utilizando la tubería DARTS, lo que dio como resultado imágenes de células comparables a los resultados publicados anteriormente9. (A) Linfocitos T primarios WT 1 s antes de la estimulación con perlas recubiertas anti-CD3/anti-CD28 y hasta 15 s después de la estimulación (barra de escala de 5 μm), así como un gráfico de superficie en 3D de un zoom de 0 s a 0,65 s en la región de contacto de la perla (barra de escala de 1 μm). (B) Carril superior: linfocito T primario P2x4-/- representativo 1 s antes y hasta 15 s después de la estimulación del cordón. Carril inferior: linfocito T primario P2x7-/- representativo 1 s antes y hasta 15 s después de la estimulación con perlas. Haga clic aquí para ver una versión más grande de esta figura.
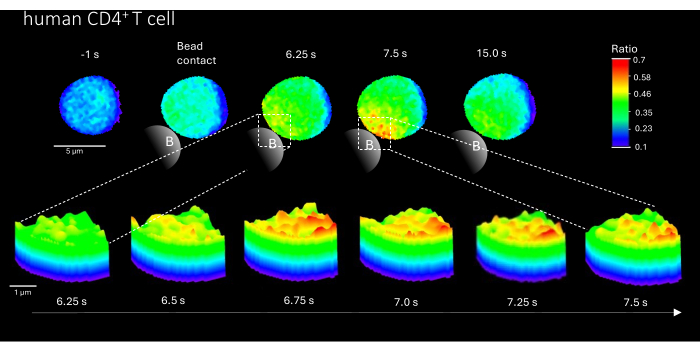
Figura 3: Microdominios Ca2+ en una célula T humana primaria representativa después de la estimulación de TCR. Las células T CD4+ humanas primarias se aislaron de células mononucleares de sangre periférica (PBMC) mediante clasificación celular activada por fluorescencia (FACS) de capas leucocitarias y se cargaron con Fluo-4 AM y Fura Red, como se describió anteriormente. La figura muestra un linfocito T humano primario 1 s antes de la estimulación con perlas recubiertas de anti-CD3 y hasta 15 s después de la estimulación (barra de escala de 5 μm), así como un gráfico de superficie en 3D de un zoom de 6,25 s a 7,5 s en la región de contacto de la perla (barra de escala de 1 μm). Haga clic aquí para ver una versión más grande de esta figura.
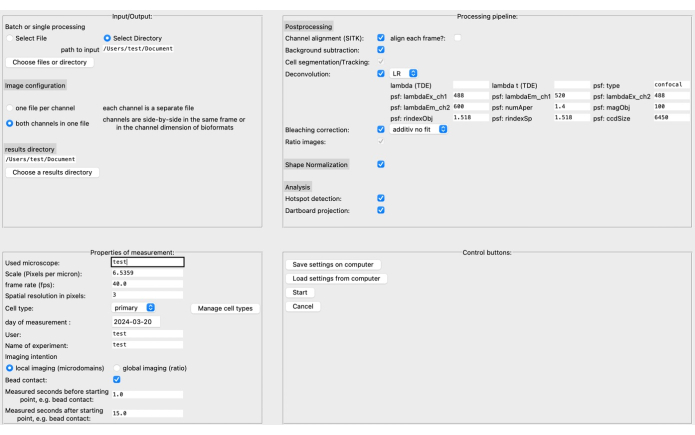
Figura 4: La interfaz gráfica de usuario (GUI) de DARTS. La interfaz gráfica de usuario se divide en cuatro áreas. En el área Entrada/Salida, debe proporcionar información sobre los datos sin procesar, incluido el directorio de origen y la configuración de la imagen (dos canales por archivo o canales separados), así como el directorio de resultados. En el área Propiedades de medición, el experimento debe describirse con toda su información relevante, como la escala (micras por píxel), la velocidad de fotogramas y el intervalo de medición en relación con el punto de inicio determinado posteriormente. A continuación, se puede ensamblar una canalización de procesamiento que consta de pasos de posprocesamiento, normalización de formas y el análisis real (detección de microdominios y acumulación de datos de diana). Finalmente, la configuración se puede guardar o cargar desde la computadora. Una vez configurado el análisis, haga clic en Iniciar para continuar. Para obtener más información sobre la configuración, visite https://ipmi-icns-uke.github.io/DARTS/General/Usage.html. Haga clic aquí para ver una versión más grande de esta figura.
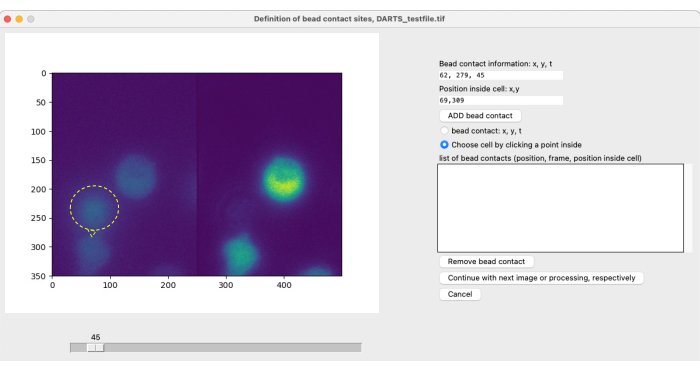
Figura 5: Definición manual de los contactos del cordón. Si se añaden cuentas a las celdas durante el experimento, el tiempo de contacto inicial de la cuenta con una celda de interés y la ubicación del contacto deben definirse manualmente. Esto se hace desplazándose por los fotogramas con el control deslizante y encontrando una posición (x,y) en un punto de tiempo t. Para rellenar automáticamente el campo de información de contacto del cordón, el usuario hace clic en la mitad izquierda de la imagen del microscopio en la posición de contacto del cordón. A continuación, para asociar una celda con el contacto del cordón, el usuario hace clic en una posición dentro de la celda que tiene un contacto del cordón. La información debe confirmarse seleccionando ADD bead. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Describimos un protocolo extenso para la obtención de imágenes de células vivas de alta resolución de microdominios locales Ca2+ en células T primarias murinas y humanas desencadenadas por la estimulación de TCR/CD3 a través de perlas recubiertas de anticuerpos. Además, implementamos un algoritmo basado en Python fácil de usar y de código abierto para identificar y analizar señales locales de Ca2+. En particular, el protocolo no se limita a la detección de microdominios Ca2+ en el contexto de la estimulación de TCR/CD3, sino que es adaptable a otros tipos de células (inmunes) como las líneas celulares NK (KHYG-1)12 o los microdominios Ca2+ independientes de TCR10,11.
Un paso crítico dentro del protocolo es el tamaño y el número de perlas estimulantes. Para imitar una sinapsis inmunológica, las perlas deben ser similares en tamaño a las células. Por lo tanto, para las células T primarias murinas y humanas, así como para las líneas celulares (Jurkat y KHYG1), utilizamos perlas magnéticas con un diámetro de 10 μm. Además, cada célula debe ser estimulada por una sola perla. Por lo tanto, la cantidad de cuentas agregadas a cada diapositiva debería ser suficiente por un lado, pero si hay demasiadas cuentas en el campo de visión, el fondo aumenta y no es posible detectar un solo punto de tiempo de activación y lado de contacto.
El protocolo utiliza los colorantes fluorescentes Ca2+ Fluo-4 AM y FuraRed AM de manera radiométrica, lo que permite la calibración de los datos13. Además, el protocolo podría adaptarse a otros pares de indicadores de Ca2+ , pero se debe tener precaución en el proceso de selección en términos de cinética de unión a Ca2+ , distribución subcelular y fotoblanqueo1. Además, las condiciones de carga deben desarrollarse y optimizarse para cada tipo de célula individual, pero las concentraciones indicadas aquí son un buen punto de partida. Para visualizar los microdominios Ca2+ , el Kd de los colorantes Ca2+ debe estar en el rango de 300-1200 nM y el tiempo de adquisición por fotograma debe ser de ≤60 ms. Si la intensidad de fluorescencia es demasiado baja, se debe verificar el conjunto de filtros, pero también es posible cargar una cantidad doble de colorante Ca2+ en las células T. Sin embargo, el colorante Ca2+ podría extrapolarse a otros orgánulos o secuestrarse en vesículas, pero también podría actuar como un amortiguador de Ca2+ y afectar las respuestas de Ca2+ .
Una limitación del algoritmo de análisis es que se asume una forma esférica de la célula; Por lo tanto, es posible que los tipos de células con diferentes morfologías necesiten una adaptación de la caja de herramientas de análisis. El algoritmo se ha utilizado para analizar microdominios locales de Ca2+ en células T murinas primarias, así como células T Jurkat y una línea celular NK (KHYG-1)12 y tuvo éxito en el análisis de microdominios Ca2+ para una línea celular neuronal murina (N2a, datos no publicados). En principio, el protocolo y la caja de herramientas de análisis podrían utilizarse para analizar tipos de células no esféricas como las células HEK293 o HeLa, pero para estos tipos de células, la proyección de la diana no se puede adaptar porque se basa en una normalización de la estructura y la forma redondas de las células. Además, el protocolo para detectar microdominios iniciales localizados de Ca2+ tras la estimulación con perlas se puede adaptar para analizar señales locales de Ca2+ derivadas de otros estímulos, como compuestos activadores o inhibidores solubles, así como microdominios de Ca2+ dependientes de adherencia e independientes de TCR/CD310,11. Cabe destacar que es más fácil definir un solo contacto de cordón en términos de tiempo y ubicación que determinar el punto de inicio de la activación después de los compuestos solubles.
Una limitación general para la detección de la formación de microdominios Ca2+ radica en la alta resolución espacio-temporal requerida y la alta relación señal-ruido (SNR) necesaria. Actualmente, la resolución derivada de nuestra configuración alcanza una resolución espacial calculada de ~0,368 μm y una resolución temporal de ~40 fotogramas por segundo (fps)1. Los avances recientes en el desarrollo de cámaras y detectores, así como la mejora de los tintes fluorescentes, podrían conducir a la posibilidad de alcanzar grabaciones ópticas de un solo canal tal como se han descrito para las construcciones ORAI-GECI (indicadores de Ca2+ expresados genéticamente)24 para la obtención de imágenes de células vivas utilizando indicadores de Ca2+ con mayor resolución temporal y espacial en el futuro.
En conjunto, el protocolo y la herramienta de análisis para la obtención de imágenes de microdominio Ca2+ de alta resolución descritos aquí se pueden utilizar no solo para analizar las señales iniciales de Ca2+ local en las células T, sino que también se pueden adaptar a otros tipos de células para descifrar la importancia de la señalización local de Ca2+ en estas.
Divulgaciones
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera considerarse un potencial conflicto de intereses.
Agradecimientos
Este trabajo contó con el apoyo de la Deutsche Forschungsgemeinschaft (DFG) (proyecto número 335447717; SFB1328, A02 a B-PD y RW; A14 a ET; número de proyecto 516286863 a B-PD). Los autores agradecen a los donantes de sangre y al Departamento de Medicina Transfusional de la UKE por su cooperación.
Materiales
| Name | Company | Catalog Number | Comments |
| α-CD3 | BD Pharmingen | 553058 | |
| α-CD28 | BD Pharmingen | 553295 | |
| Anaconda | Anaconda | https://docs.anaconda.com/free/anaconda/install/index.html | |
| Bovine serum albumin | Sigma-Aldrich | A2058 | |
| Cell strainer | Biofil | CSS-013-070 | 70 µm diameter |
| Countess Automated Cell Counter | Invitrogen | A49865 | |
| Cover slips | Paul Marienfeld GmbH | 101202 | |
| DARTS | GitHub | https://github.com/IPMI-ICNS-UKE/DARTS | |
| Dulbecco’s Phosphate buffered saline | Gibco, Thermo Fisher | 14190144 | |
| EasySep CD4+ T cell Isolation Kit | Stemcell | 19852 | |
| EasySep Magnetic Multistand | Stemcell | 18010 | |
| Fluo 4-AM | Invitrogen | F14201 | |
| Fura Red-AM | Invitrogen | F3020 | |
| Gibco RPMI 1640 medium | Gibco, Thermo Fisher | 11875093 | |
| Git | GitHub | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git | |
| immersion oil | Leica | 11513859 | |
| Newborn calf serum | Sigma-Aldrich | N4637 | |
| Oracle | Oracle | https://www.oracle.com/de/java/technologies/downloads/ | |
| Penicillin/Streptamycin | Gibco, Thermo Fisher | 15240062 | |
| Poly-L-lysine | Sigma-Aldrich | P4707 | |
| Protein G magnetic beads | Merck | LSKMAGG02 | 10 µM diameter |
| Python | Python | https://www.python.org/downloads/ | |
| Silicon grease (basylone) | Bayer | 291-1220 | |
| Time-Dependent Entropy Deconvolution | GitHub | https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters | |
| Tween | q-biogene | TWEEN201 |
Referencias
- Wolf, I. M. A., et al. Frontrunners of T cell activation: Initial, localized Ca2+ signals mediated by NAADP and the type 1 ryanodine receptor. Sci Signal. 8 (398), 102(2015).
- Taguchi, T., Mukai, K. Innate immunity signalling and membrane trafficking. Curr Opin Cell Biol. 59, 1-7 (2019).
- Trebak, M., Kinet, J. -P. Calcium signalling in T cells. Nat Rev Immunol. 19 (3), 154-169 (2019).
- Diercks, B. -P., et al. ORAI1, STIM1/2, and RYR1 shape subsecond Ca2+ microdomains upon T cell activation. Sci Signal. 11, 0358(2018).
- Cheng, H., Lederer, W. J., Cannell, M. B. Calcium sparks: Elementary events underlying excitation-contraction coupling in heart muscle. Science. 262 (5134), 740-744 (1993).
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Berridge, M. J., Lipp, P., Bootman, M. D. The versatility and universality of calcium signalling. Nat Rev Mol Cell Biol. 1 (1), 11-21 (2000).
- Kummerow, C., Junker, C., Kruse, K., Rieger, H., Quintana, A., Hoth, M. The immunological synapse controls local and global calcium signals in T lymphocytes. Immunol Rev. 231 (1), 132-147 (2009).
- Brock, V. J., et al. P2X4 and P2X7 are essential players in basal T cell activity and Ca2+ signaling milliseconds after T cell activation. Sci Adv. 8 (5), 9770(2022).
- Weiß, M., et al. Adhesion to laminin-1 and collagen IV induces the formation of Ca2+ microdomains that sensitize mouse T cells for activation. Sci Signal. 16 (790), 9405(2023).
- Diercks, B. -P. The importance of Ca2+ microdomains for the adaptive immune response. Biochim Biophys Acta Mol Cell Res. 1871 (5), 119710(2024).
- Woelk, L. -M., et al. DARTS: an open-source Python pipeline for Ca2+ microdomain analysis in live cell imaging data. Front Immunol. 14, 1299435(2024).
- Diercks, B. -P., Werner, R., Schetelig, D., Wolf, I. M. A., Guse, A. H. High-resolution calcium imaging method for local calcium signaling. Methods Mol Biol. 1929, 27-39 (2019).
- Python. Python. , Available from: https://www.python.org/downloads/ (2024).
- Anaconda. Anaconda. , Available from: https://docs.anaconda.com/free/anaconda/install/index.html (2024).
- git. git. , Available from: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git (2024).
- DARTS. DARTS. , Available from: https://github.com/IPMI-ICNS-UKE/DARTS (2024).
- DARTS. DARTS GitHub Repository. , Available from: https://ipmi-icns-uke.github.io/DARTS/ (2024).
- Oracle. , Available from: https://www.oracle.com/de/java/technologies/downloads/ (2024).
- Time-Dependent Entropy Deconvolution. , Available from: https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters (2024).
- Gu, F., et al. Dual NADPH oxidases DUOX1 and DUOX2 synthesize NAADP and are necessary for Ca2+ signaling during T cell activation. Sci Signal. 14 (709), 3800(2021).
- Roggenkamp, H. G., et al. HN1L/JPT2: A signaling protein that connects NAADP generation to Ca2+ microdomain formation. Sci Signal. 14 (675), 5647(2021).
- Heßling, L. D., Troost-Kind, B., Weiß, M. NAADP-binding proteins - Linking NAADP signaling to cancer and immunity. Biochim Biophy. Acta Mol Cell Res. 1870 (7), 119531(2023).
- Dynes, J. L., Amcheslavsky, A., Cahalan, M. D. Genetically targeted single-channel optical recording reveals multiple Orai1 gating states and oscillations in calcium influx. Proc Natl Acad Sci U S A. 113 (2), 440-445 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados