Method Article
Imagerie des microdomaines Ca2+ initiaux dans les lymphocytes T primaires
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici, nous fournissons un protocole complet pour résoudre les signaux locaux initiaux Ca2+ , connus sous le nom de microdomaines Ca2+ , dans les cellules T primaires murines et humaines à l’aide de la microscopie à fluorescence. Ce protocole constitue une ressource précieuse pour les chercheurs qui examinent les voies de signalisation Ca2+ au sein des cellules immunitaires et pour mieux comprendre leur fonction.
Résumé
Les signaux Ca2+ locaux inférieurs à la seconde, appelés microdomaines Ca2+ , sont des signaux Ca2+ hautement dynamiques et de courte durée, qui entraînent une élévation globale de [Ca2+]i et pourraient déjà déterminer le destin d’un lymphocyte T. Lors de l’activation des récepteurs des lymphocytes T, le NAADP se forme rapidement, se liant aux protéines de liaison au NAADP (HN1L/JPT2, LSM12) et à leurs récepteurs respectifs (RyR1, TPC2) situés sur les réserves intracellulaires de Ca2+ , comme le RE et les lysosomes, et conduisant à la libération et à l’élévation ultérieures de [Ca2+]i. Pour capturer ces signaux Ca2+ rapides et dynamiques, nous avons développé une technique d’imagerie haute résolution utilisant une combinaison de deux indicateurs Ca2+ , Fluo-4 AM et FuraRed AM. Pour le post-traitement, une approche open source et semi-automatisée de détection de microdomaines Ca2+ a été développée sur la base du langage de programmation Python. À l’aide de ce flux de travail, nous sommes en mesure de détecter de manière fiable les microdomaines Ca2+ au niveau subcellulaire dans les cellules T primaires murines et humaines dans des vidéos de fluorescence à haute résolution temporelle et spatiale. Cette méthode peut également être appliquée à d’autres types de cellules, comme les cellules NK et les lignées cellulaires neuronales murines.
Introduction
La technique de microscopie à fluorescence présentée permet de visualiser les signaux calciques initiaux locaux et temporels (Ca2+) dans les lymphocytes T primaires de souris, appelés microdomaines Ca2+ . Les microdomaines Ca2+ représentent des événements de signalisation Ca2+ hautement dynamiques et de courte durée, ce qui pose des défis pour l’imagerie et l’analyse efficaces des cellules vivantes1.
Les lymphocytes T sont difficiles pour l’imagerie des cellules vivantes en raison des différences relatives d’intensité de fluorescence centrale et périphérique, qui peuvent être attribuées à leur forme sphérique et à leur petit diamètre de ~6-8 μm. Lors de la stimulation et de la formation de synapses immunitaires, les lymphocytes T subissent des changements morphologiques, ce qui complique davantage l’imagerie des lymphocytes T1. Par conséquent, l’utilisation d’une analyse ratiométrique devient impérative, réalisée soit en enregistrant deux images représentant des propriétés différentes d’un colorant Ca2+ , soit en utilisant une combinaison de deux colorants Ca2+ . Les caractéristiques exigeantes des microdomaines Ca2+ comprennent leur nature rapide, limitée dans le temps et dans l’espace. Pour capturer cela, les colorants Ca2+ utilisés doivent posséder à la fois une luminosité basale élevée et un rapport signal/bruit (SNR) élevé pour obtenir la résolution temporelle et spatiale la plus élevée possible. Des résultats optimaux ont été obtenus en utilisant une combinaison du colorant à double longueur d’onde Fura Red et du colorant à longueur d’onde unique Fluo-4. Les cellules de co-chargement avec Fluo-4 et Fura Red atténuent les défis posés par le fort photoblanchiment des colorants à double longueur d’onde d’émission et le retard temporel associé aux colorants à double excitation, garantissant ainsi une acquisition d’image rapide. Cette approche facilite encore la visualisation des changements de forme et des mouvements subtils. Le système d’imagerie est également soumis à des exigences particulières en termes de résolution spatiale pour permettre la visualisation des signaux Ca2+ provenant de l’ouverture de petits groupes de canaux ou même de canaux uniques1.
La signalisation Ca2+ joue un rôle central dans l’activation des fonctions immunitaires au sein des lymphocytes T, y compris la formation des synapses et la production et la libération de cytokines 2,3. Le destin spécifique de la cellule est régulé par les signaux Ca2+ différemment prononcés et distribués localement, les microdomaines Ca2+ 3. Notamment, ces signaux locaux Ca2+ précèdent une augmentation généralisée des niveaux intracellulaires de Ca2+ dans les cellules T et la formation de microdomaines Ca2+ dépend à la fois de l’entrée et de la libération de Ca2+ 1,4,5. Lors de la stimulation du récepteur des lymphocytes T (TCR)/CD3, la formation de Ca2+ libérant des seconds messagers, comme l’acide nicotinique adénine dinucléotide phosphate (NAADP), le D-myo-inositol 1,4,5-trisphosphate (IP3) et l’ADP ribose cyclique (cADPR) est déclenchée, entraînant une augmentation des niveaux intracellulaires de Ca2+ jusqu’à 1 μM 6,7. Les événements précoces de signalisation Ca2+ sont liés à la libération de Ca2+ à partir des réserves intracellulaires de Ca2+ telles que le réticulum endoplasmique (RE), des canaux tels que le récepteur de la ryanodine 1 (RyR1) et le récepteur IP3 (IP3R) étant principalement responsables de cette signalisation. Cela déclenche ensuite un afflux extracellulaire de Ca2+ et aboutit à un signal global de Ca2+ via l’entrée de Ca2+ opérée en magasin (SOCE)8. De plus, d’autres canaux sont impliqués dans la signalisation Ca2+ lors de l’activation des lymphocytes T9, par exemple, les canaux P2X4 et P2X7 assurent l’afflux cationique dépendant de l’adénosine triphosphate (ATP), contribuant ainsi à l’augmentation du Ca2+ intracellulaire. Remarquablement, les microdomaines Ca2+ dépendants de l’adhésion initiale (ADCM) sont déjà formés avant la stimulation TCR, mais avec des amplitudes et des fréquences Ca2+ plus faibles. Ces signaux Ca2+ initiaux indépendants du TCR servent très probablement à la migration des lymphocytes T vers le site de l’inflammation et préparent les lymphocytes T à la restimulation au site de l’infection10,11.
En développant la méthode décrite pour l’imagerie locale Ca2+ , nous avons obtenu un outil supplémentaire pour explorer l’origine et la signification des signaux précoces Ca2+ dans l’activation des lymphocytes T. Cette méthode permet à l’utilisateur de détecter des signaux Ca2+ plus petits, de courte durée et plus rapides qu’auparavant. De plus, la déconvolution, l’analyse, l’enregistrement, le suivi et la normalisation de forme (DARTS), le pipeline d’analyse basé sur Python, permet de partager les outils d’analyse avec un public plus large12.
Protocole
Toutes les expériences sur les animaux ont été approuvées et réalisées conformément aux directives en matière de bien-être animal de la commission institutionnelle de soin et d’utilisation des animaux du centre médical universitaire de Hambourg-Eppendorf.
1. Isolement des lymphocytes T primaires de souris dans les ganglions lymphatiques et la rate
- Prélever les rates/ganglions lymphatiques dans des conditions stériles conformément aux directives éthiques et les placer dans un tube avec un milieu Clicks glacé (10 % de sérum de veau fœtal (FCS), 2 mM de L-Glutamine, Pénicilline/Streptomycine (P/S) 100 U/mL, 50 nM β-Mercaptoéthanol).
REMARQUE : Les lymphocytes T de souris de type sauvage et KO sont isolés de la même manière. - Refroidissez la centrifugeuse à 4 °C.
- Placez la rate et les ganglions lymphatiques dans une passoire cellulaire de 70 μm dans une boîte de Pétri stérile et ajoutez un milieu d’isolement de la rate (milieu RPMI + 7,5 % NCS + 1 % P/S) jusqu’à un volume total de 20 mL.
- Perturbez soigneusement mais doucement la rate à l’aide d’un mortier plastique. Transférez ensuite les cellules dans un tube à centrifuger de 50 ml.
REMARQUE : Un volume total de 20 ml doit maintenant se trouver dans le tube de centrifugation de 50 ml. La rate et les ganglions lymphatiques doivent être complètement perturbés. Travaillez sur glace à partir de ce moment. Garder les cellules sur la glace ralentit leur métabolisme et empêche la mort cellulaire, garantissant qu’elles restent viables et fonctionnellement intactes. Cela permet de préserver leur état physiologique pendant une plus longue période, ce qui est particulièrement important pour des expériences précises en aval. - Centrifuger la suspension cellulaire à 300 x g pendant 5 min à 4 °C.
- Jeter le surnageant et assurer la remise en suspension de la pastille obtenue dans 2 mL de solution saline tamponnée au phosphate (DPBS) de Dulbecco (sans Ca2+ et Mg2+). Transférer dans un nouveau tube à centrifuger de 12 ml.
2. Sélection négative des lymphocytes T CD4+
REMARQUE : Pour la sélection négative des lymphocytes T CD4+ , un kit d’isolement des lymphocytes T contenant un bloqueur de FcR, des anticorps biotinylés contre les lymphocytes T non CD4+ et des particules magnétiques enrobées de streptavidine est utilisé.
- Ajouter 20 μL/mL d’inhibiteur FcR de souris du kit d’isolement des lymphocytes T à la suspension cellulaire.
- De plus, ajoutez 100 μL de cocktails d’isolement de lymphocytes T CD4+ de souris à une concentration de 50 μL/mL, mélangez bien et incubez pendant 10 min à température ambiante (RT).
- Des particules magnétiques enrobées de streptavidine vortex pendant 30 s, ajoutent 150 μL à la suspension cellulaire, mélangent bien et incuberont pendant 2 min 30 s à RT.
- Ajouter le DPBS à la suspension cellulaire et la remplir jusqu’à un volume total de 7 ml. Assurez-vous de pipeter doucement de haut en bas, puis incubez dans l’aimant pendant 2 min 30 s à RT.
- Par la suite, recueillez délicatement les lymphocytes T enrichis dans le surnageant et transférez-les dans un nouveau tube à centrifuger de 12 ml.
- Cellules de centrifugation à 300 x g à RT pendant 5 min.
- Jetez le surnageant.
- Remettre en suspension les lymphocytes T CD4+ isolés et enrichis dans 1 mL de DPBS, en pipetant soigneusement de haut en bas. Incuber le mélange pendant 2 min 30 s dans l’aimant à RT pour s’assurer qu’il n’y a pas de contamination des billes.
- Récupérez le surnageant, transférez-le dans un nouveau tube à centrifuger et comptez les lymphocytes T enrichis.
- Pour compter les cellules, colorez-les avec du bleu trypan et comptez à l’aide d’un compteur de cellules automatisé.
REMARQUE : Il est préférable de charger directement les lymphocytes T CD4+ isolés et de les imager le jour même de l’isolement. Cependant, les lymphocytes T murins primaires peuvent être cultivés sans aucune stimulation pendant la nuit dans un incubateur à 37 °C avec 5 % de CO2 dans un milieu d’isolement (RPMI 1640 + 10 % FCS + P/S).
3. Chargement des lymphocytes T CD4+ primaires de souris
REMARQUE : Pour mesurer les concentrations cytosoliques libres de Ca2+ , des colorants fluorescents et perméables à la membrane sont utilisés dans les expériences d’imagerie Ca2+ . Une combinaison d’ester acétoxyméthylique de fluo-4 (AM) et du colorant ratiométrique Fura Red-AM sert d’indicateur pour la détection rapide des signaux locaux de Ca2+ . Assurez-vous de travailler dans l’obscurité tout en utilisant des colorants fluorescents.
- Centrifuger 2-5 x 106 cellules pendant 5 min, 300 x g, jeter le surnageant et remettre en suspension dans 480 μL de milieu de cellules T (RPMI 1640, 10 % FCS) avec 10 μM Fluo-4-AM (stock : [1 mM]) et 20 μM Fura Red-AM (stock : [4 mM]).
- Incuber d’abord pendant 20 min à RT sous le banc dans l’obscurité, en recouvrant le faucon de papier d’aluminium.
REMARQUE : Ne pas dépasser le premier temps d’incubation, car les indicateurs Ca2+ sont dilués dans du DMSO, ce qui pourrait être nocif pour les cellules à un temps d’exposition plus long dans le faible volume total. Gardez les cellules à l’abri de la lumière après le chargement en raison de la sensibilité à la lumière des colorants. - À la fin de l’incubation initiale de 20 minutes, ajouter 2 mL de milieu à cellules T dans les cellules et poursuivre l’incubation pendant 30 minutes supplémentaires dans l’obscurité à l’heure de la RT.
- Centrifuger la suspension cellulaire à 300 x g pendant 5 min à RT.
- Jeter le surnageant et nettoyer la pastille en la lavant avec 2 mL de tampon de mesure Ca2+ (140 mM NaCl, 5 mM KCl, 1 mM MgSO4, 1 mM CaCl2, 20 mM HEPES, 1 mM NaH2PO4, 5 mM de glucose, pH 7,4) suivie d’une centrifugation à 300 x g pendant 5 min à RT.
- Enfin, jetez le surnageant et remettez-le en suspension avec le tampon de mesure Ca2+ . Ajustez le nombre de cellules à ~100 000 cellules par 10 μL.
REMARQUE : Un volume de 10 μL est utilisé pour une mesure. Évitez de trop diluer les cellules, car il pourrait être nécessaire d’ajuster davantage le nombre de cellules au microscope pour atteindre un optimum pour les conditions de mesure. - Laissez les cellules incuber pendant ~20 min pour assurer une infiltration cellulaire complète et une désestérification des colorants. Conservez les cellules sur de la glace et dans l’obscurité jusqu’à la mesure.
REMARQUE : Les cellules T murines primaires chargées peuvent être utilisées jusqu’à ~4 h après le chargement.
4. Imagerie locale Ca2+
- Préparation des lames
- Enrober les lamelles de microscope (24 mm x 46 mm) d’albumine sérique bovine (BSA, 5 mg/mL) et de poly-L-lysine (PLL, 0,1 mg/mL) (figure 1A). Laissez BSA reposer pendant ~20 min avant d’appliquer la PLL.
REMARQUE : Le revêtement facilite l’adhésion des cellules. Pour une bonne adhérence cellulaire, répartissez avec précision le BSA et le PLL jusqu’à ce qu’il n’y ait plus de traînées visibles. - Pour créer des chambres de réaction, collez des joints toriques en caoutchouc réutilisables sur les lames à l’aide d’une pâte de silicone (Figure 1B,C).
REMARQUE : D’autres diapositives/chambres ou plaques telles que des lames de 35 mm, des chambres métalliques réutilisables ou des plaques à 8, 24 ou 48 puits peuvent être utilisées pour l’acquisition d’images.
- Enrober les lamelles de microscope (24 mm x 46 mm) d’albumine sérique bovine (BSA, 5 mg/mL) et de poly-L-lysine (PLL, 0,1 mg/mL) (figure 1A). Laissez BSA reposer pendant ~20 min avant d’appliquer la PLL.
- Enrobage de billes magnétiques de protéine G (10 μm de diamètre) avec des anticorps
- Mélangez délicatement la suspension de billes magnétiques de protéines et transférez 12,5 μL dans un nouveau tube.
- Placez le tube sur un support magnétique pour retirer le tampon de stockage et laissez les billes migrer vers l’aimant. Retirez délicatement la mémoire tampon de stockage en la pipetant sur le côté opposé de l’aimant.
- Pour éliminer toute mémoire tampon de stockage restante, lavez les billes en ajoutant 500 μL de PBS-T (PBS, 0,1 % Tween) et en faisant tourbillonner pendant 10 s. Replacez le tube dans le support magnétique et retirez le tampon.
- Pour enrober les billes d’anticorps, il faut les remettre en suspension dans 7,5 μL de PBS-T et ajouter 5 μL d’anti-CD3 (0,5 mg/mL) et d’anti-CD28 (0,5 mg/mL), respectivement. Incuber pendant 30 à 60 min avec un mélange continu à RT.
- Lavez les billes enrobées trois fois avec 500 μL de PBS-T, puis lavez une fois avec 500 μL de tampon de mesure Ca2+ à l’aide du support magnétique.
- Remettre les billes en suspension dans 200-400 μL de tampon de mesure Ca2+.
REMARQUE : Vérifiez la densité du cordon pendant les mesures, diluez davantage si nécessaire.
- Microscopie d’imagerie locale Ca2+
REMARQUE : L’imagerie est réalisée à l’aide d’un microscope optique à fond clair avec un grossissement de 100, équipé d’une lampe à arc au xénon comme source lumineuse. Les images sont capturées en mode 14 bits avec double binning à l’aide d’une caméra couplée à la charge multiplicatrice d’électrons. Pour enregistrer et diviser les longueurs d’onde d’émission des deux colorants, un module à double vue est utilisé, doté des filtres suivants en nm (ex, 480/40 ; bs, 495 ; em1, 542/50 ; em2, 650/57). La configuration d’imagerie comprend un hub d’acquisition et un logiciel d’imagerie pour l’acquisition d’images.- Placez 10 μL des cellules chargées sur la lame préparée et laissez-les se fixer à la lame pendant 3 à 5 minutes.
- Ajoutez délicatement 80 μL de tampon de mesure Ca2+ sur la lame.
- Sélectionnez l’objectif d’immersion dans l’huile 100x et appliquez une petite goutte d’huile d’immersion. Placez la lame sur la table du microscope.
- Ajustez la mise au point en mode fond clair, sélectionnez soigneusement un champ de vision avec jusqu’à 10 cellules qui ne sont pas en contact les unes avec les autres et acquérez une image.
REMARQUE : Les cellules qui se chevauchent et se touchent seront difficiles à analyser ultérieurement.- Allumez la lampe, vérifiez la fluorescence et la charge des lymphocytes T, et comparez les cellules des deux canaux pour vérifier qu’elles ne sont pas préactivées.
- Assurez-vous de prendre des photos du champ de vision des canaux en fond clair et en fluorescence avant et après la mesure pour vérifier le mouvement et la charge des cellules.
- Démarrez la mesure et capturez l’activité basale pendant 1 min avec un taux d’acquisition de 1 image toutes les 5 s.
- Ajoutez 10 μL de composé/stimulant (bille ou stimulateur/inhibiteur) après 1 min et mesurez pendant un total de 3 min en utilisant 40 images par s ou la fréquence d’images maximale possible.
REMARQUE : L’ajout de perles est l’étape cruciale ; Assurez-vous que les perles sont ajoutées près de la lumière d’excitation sur la diapositive sans déplacer la diapositive. La figure 2 montre des exemples représentatifs de microdomaines Ca2+ dans les lymphocytes T CD4+ primaires au contact des billes de souris WT et P2x4-/- et P2x7-/-.
5. Post-traitement/analyse des données
REMARQUE : Pour le traitement d’images et l’analyse de données, le pipeline open source DARTS basé sur Python est utilisé. Il a été développé par Woelk et al.12 sur la base des travaux de Diercks et al.13.
- Installation de la canalisation DARTS
- Installez Python 3.10.014, anaconda15 et git16, et clonez le dépôt GitHub à l’aide de la commande terminal git clone : https://github.com/IPMI-ICNS-UKE/DARTS.git
- Créez un environnement conda avec conda create --name DARTS et installez tous les packages Python nécessaires avec pip install <package>.
REMARQUE : Suivez les instructions d’installation dans le référentiel GitHub DARTS 17,18 pour plus d’informations. - Avant d’utiliser Bioformats, assurez-vous qu’un environnement d’exécution Java est correctement installé19.
- Utilisation du pipeline DARTS
REMARQUE : Une fois installé, DARTS peut être démarré à partir de la fenêtre du terminal.- Accédez à la copie locale du référentiel DARTS (cd path/to/DARTS) qui contient le fichier main.py. Assurez-vous que l’environnement conda est activé (conda activate DARTS).
- Exécutez DARTS en tapant python main.py dans la fenêtre du terminal et en appuyant sur Entrée.
- Dans l’interface utilisateur graphique (GUI), spécifiez le répertoire source et results ainsi que le format de l’image dans la section supérieure gauche (voir Figure 3).
- Définissez les propriétés de mesure, telles que l’échelle (pixels par micromètre), la fréquence d’images, le type de cellule, etc. Le pipeline de traitement peut être assemblé en fonction des données d’image spécifiques et de la question de recherche.
REMARQUE : Pour en savoir plus sur les paramètres de déconvolution, par exemple, consultez la documentation correspondante20. - Enregistrez les paramètres sur l’ordinateur avant de cliquer sur Démarrer. Une description plus détaillée de l’interface graphique peut être trouvée dans la documentation DARTS.
- Imagerie microdomaine Ca2+ et contacts de billes
REMARQUE : Pour l’imagerie et la stimulation locales avec des billes et l’analyse d’image ultérieure, les contacts de billes spécifiques à la cellule doivent être définis par l’utilisateur pour identifier le point de départ (t = 0) de la période de mesure d’intérêt (voir Figure 4). Un contact de bille est défini comme le site de contact d’une bille (stimulative) avec une cellule d’intérêt. Il s’agit de la spécification de (1) le temps de contact du talon, par exemple, le cadre 300, et (2) la position de contact du talon par rapport au centre de la cellule, par exemple, 1 heure ou 12 heures. Avec la version actuelle de DARTS, l’utilisateur doit définir et saisir manuellement les informations pour chaque contact de bille. Pour définir les contacts de talon, procédez comme suit :- Utilisez le curseur pour localiser le temps de contact entre une bille et une cellule d’intérêt.
- Dans le menu des options sur le côté droit, sélectionnez contact de perle : x, y, t.
- Sélectionnez l’endroit sur le côté gauche de l’image où la cellule et la perle entrent en contact.
- Sélectionnez Choisir une cellule en cliquant sur un point à l’intérieur. Cliquez sur la cellule stimulée par cette perle, de préférence au centre.
- Cliquez sur AJOUTER le contact de la perle.
- Répétez les étapes 5.3.1 à 5.3.5 pour chaque contact de talon supplémentaire dans ce fichier. Une fois que tous les contacts de la perle sont définis, cliquez sur le bouton Continuer .
- Passez aux fichiers supplémentaires. Une fois le dernier fichier atteint, le script lance automatiquement l’analyse de tous les fichiers.
- Analyse des données
- Vous trouverez les images de rapport traitées, les données de microdomaine pour chaque cellule au fil du temps (y compris la localisation, l’amplitude, la taille), les données sources pour la projection du jeu de fléchettes et d’autres ressources dans le dossier des résultats.
- Pour créer des jeux de fléchettes, accédez au dossier /src/analysis/ à l’intérieur du dossier DARTS (motif : chemin du cd/vers/DARTS/src/analysis) contenant le script DartboardPlotGUI.py. Ensuite, tapez python DartboardPlotGUI.py et appuyez sur Entrée.
- Assurez-vous que les informations nécessaires sont fournies et que les fichiers de feuille de calcul du dossier de résultats sont correctement sélectionnés comme fichiers sources pour la génération du jeu de fléchettes.
Résultats
Dans ce protocole, nous avons décrit une méthode mise à jour pour imager et analyser les microdomaines Ca2+ initiaux dans les lymphocytes T primaires de souris, basée sur des travaux antérieurs de notre groupe 1,13. Cette approche a permis de démêler l’implication des canaux CRAC tels que ORAI1, STIM1 et STIM2, ainsi que des canaux de libération intracellulaires de Ca2+ comme RyR1 dans les événements de signalisation précoces Ca2+ 4.
Pour ce faire, nous avons étudié la formation spontanée de microdomaines Ca2+ en imageant des souris primaires non stimulées Orai1-/-, Stim1-/-, Stim2-/- et Ryr1-/- et les avons comparées à des lymphocytes T primaires de souris WT. L’analyse de la formation des microdomaines Ca2+ a englobé la vitesse d’apparition du signal, l’amplitude Ca2+ et le nombre de signaux par plan confocal. Notamment, à l’exception des lymphocytes T Stim2-/-, tous les lymphocytes T KO ont montré une diminution significative des signaux locaux Ca2+ et une concentration cytosolique libre basale Ca2+ réduite par rapport aux cellules WT. Cela nous a amenés à conclure que la formation de microdomaines Ca2+ est intimement liée à l’interaction d’ORAI1, STIM1 et RyR14. De plus, nous avons réussi à identifier et à caractériser des microdomaines Ca2+ spontanés au niveau de la membrane plasmique. Ces microdomaines Ca2+ ont été caractérisés par une amplitude Ca2+ de 290 nm ± 12 nm. L’utilisation d’une approche codée par couleur pour les signaux Ca2+ a permis de visualiser les microdomaines Ca2+ à travers la cellule. Les résultats ont également mis en évidence l’apparition rapide de microdomaines Ca2+, visibles en quelques millisecondes, et la capacité de cette méthode à détecter les signaux Ca2+ avec une longévité de quelques millisecondes4. Ces microdomaines Ca2+ spontanés ont ensuite été identifiés comme des microdomaines Ca2+ dépendants de l’adhésion (ADCM), dépendant non seulement du SOCE, mais agissant également via la cascade de signalisation FAK/PLC-γ/IP3 10 et l’implication de P2X49. De plus, cette technique a été fondamentale pour confirmer que les oxydases doubles 1 et 2 (DUOX1/2) sont les enzymes productrices de NAADP21 et que HN1L/JPT222 est l’une des protéines de liaison à NAADPnouvellement découvertes 23.
La figure 2 montre des exemples représentatifs de microdomaines Ca2+ dans les lymphocytes T CD4+ primaires au contact des billes des souris WT ainsi que des souris P2x4-//- et P2x7-//. Les cellules ont été chargées avec les colorants Ca2+ Fluo-4 AM et Fura Red AM et imagées à une vitesse d’acquisition de 25 ms (40 images/s). Pour imiter la formation des synapses des lymphocytes T, les cellules ont été stimulées avec des billes enrobées d’anti-CD3/anti-CD28. La formation initiale du microdomaine Ca2+ a été analysée 1 s avant et jusqu’à 15 s après le contact avec le talon à l’aide du pipeline DARTS. Au contact des billes, la cellule WT a montré une formation rapide de microdomaines Ca2+ dans la première seconde après la stimulation au site de contact des billes (Figure 2A). Ces microdomaines Ca2+ se sont encore étendus dans toute la cellule dans les 15 secondes suivant le contact avec les billes. Contrairement à la cellule WT, les cellules P2x4-/- et P2x7-/- (Figure 2B) ont montré une diminution de la formation de microdomaines Ca2+ lors de la stimulation des billes, ainsi que pour le niveau basal inférieur de P2x4-/-a avant le contact avec les billes. Ces résultats représentatifs sont conformes aux résultats précédemment publiés par Brock et al.9, indiquant la formation de microdomaines Ca2+ dans les cellules T WT directement après le contact des billes sur 15 s et des signaux plus faibles par image dans les cellules P2x4-/- et P2x7-/- . De plus, l’amplitude dans les cellules P2x4-/-a été significativement réduite, établissant davantage le rôle de la signalisation purinergique dans les microdomaines Ca2+ dépendants de l’adhésion.
De plus, cette méthode peut également être utilisée pour visualiser les microdomaines Ca2+ initiaux dans les lymphocytes T CD4+ humains primaires (Figure 3). Dans la lignée des lymphocytes T murins primaires, les microdomaines initiaux Ca2+ sont évoqués au site de contact de la bille. Cependant, la réponse globale au Ca2+ semble se produire à une échelle de temps différente de celle des lymphocytes T CD4+ murins.
L’analyse manuelle des signaux locaux Ca2+ n’est pas réalisable car elle est assez laborieuse et subjective pour l’enquêteur individuel. Par conséquent, nous avons précédemment développé un algorithme dans MATLAB Simulink en utilisant ses boîtes de traitement et d’optimisation d’images pour le post-traitement13 afin d’analyser les microdomaines locaux Ca2+ .
Récemment, nous avons développé un nouveau pipeline de post-traitement open source appelé DARTS pour l’analyse de microdomaines Ca2+ dans l’imagerie haute résolution de cellules vivantes à l’aide de la plate-forme logicielle Python12. Ici, différents algorithmes de déconvolution peuvent être sélectionnés, en fonction des préférences de l’utilisateur, une normalisation de la forme des cellules effectuée pour compenser les changements morphologiques de forme des cellules, et des paramètres spécifiques au microscope et à la mesure définis (par exemple, l’échelle, la fréquence d’images, le temps mesuré) (Figure 4).
Après avoir sélectionné les paramètres pour l’analyse du microdomaine Ca2+ , une deuxième fenêtre contextuelle s’ouvre pour chaque mesure individuelle afin de définir le contact du cordon (Figure 5). Pour définir le contact du talon, l’utilisateur peut faire défiler manuellement le fichier tiff à l’aide du curseur et sélectionner le cadre de contact du talon individuellement. Le contact de la perle est sélectionné en cliquant sur le site de contact de la perle (Figure 5, perle et contact de la perle indiqués par un anneau et une flèche jaunes) ainsi que la sélection de cellule. Cette étape doit être répétée pour chaque cellule d’intérêt. Enfin, le post-traitement automatisé des images est appliqué et les données des résultats sont résumées et enregistrées dans une feuille de calcul.
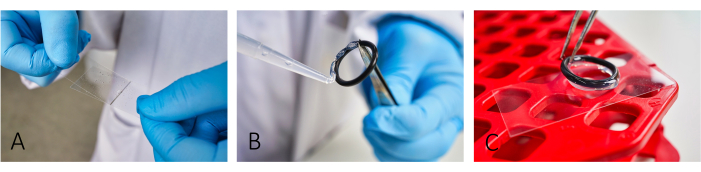
Figure 1 : Flux de travail de préparation des lames pour l’imagerie. (A) Ajoutez et étalez à la fois le BSA et le PLL sur la lame à l’aide d’une deuxième lamelle en verre. (B, C) Pour construire une chambre, collez les joints toriques en caoutchouc avec de la graisse de silicone sur la glissière. Assurez-vous que l’ensemble de l’anneau est recouvert d’une fine couche de graisse pour bien isoler la chambre. Veuillez cliquer ici pour voir une version agrandie de cette figure.
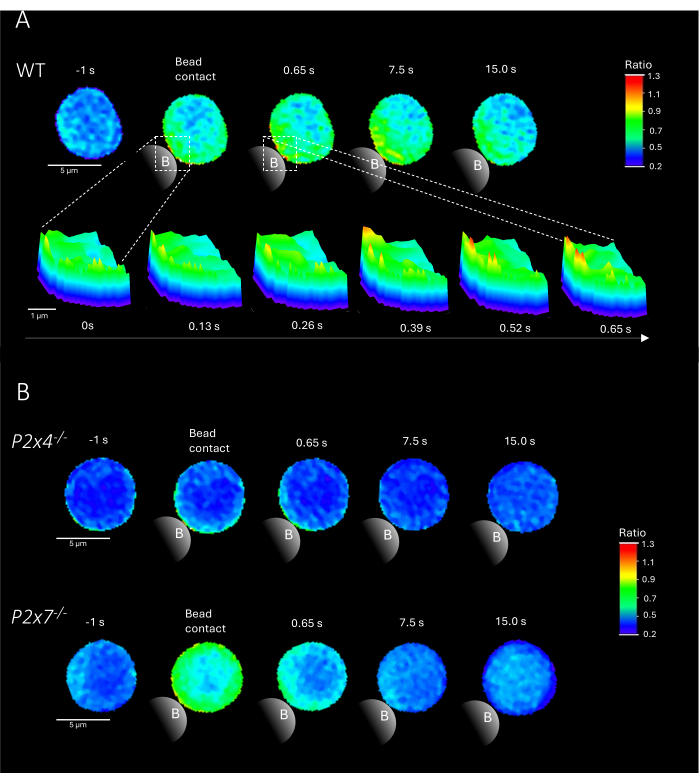
Figure 2 : Cellules représentatives de microdomaines Ca2+ dépendants des récepteurs des lymphocytes T dans un lymphocyte T CD4+ primaire de type sauvage (WT) (A), P2x4-/- ou P2x7-/-(B). Les lymphocytes T CD4+ ont été isolés négativement et chargés en Fluo-4 AM et en Fura Red, comme décrit ci-dessus. Les lymphocytes T ont été analysés à l’aide du pipeline DARTS, ce qui a permis d’obtenir des images cellulaires comparables aux résultats précédemment publiés9. (A) Cellule T primaire WT 1 s avant stimulation avec des billes enrobées d’anti-CD3/anti-CD28 et jusqu’à 15 s après stimulation (barre d’échelle 5 μm), ainsi qu’un tracé de surface 3D d’un zoom avant de 0 s à 0,65 s dans la zone de contact de la bille (barre d’échelle 1 μm). (B) Voie supérieure : cellule T primaire représentative P2x4-/- 1 s avant et jusqu’à 15 s après la stimulation par billes. Voie inférieure : cellule T primaire représentative P2x7-/- 1 s avant et jusqu’à 15 s après la stimulation par billes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
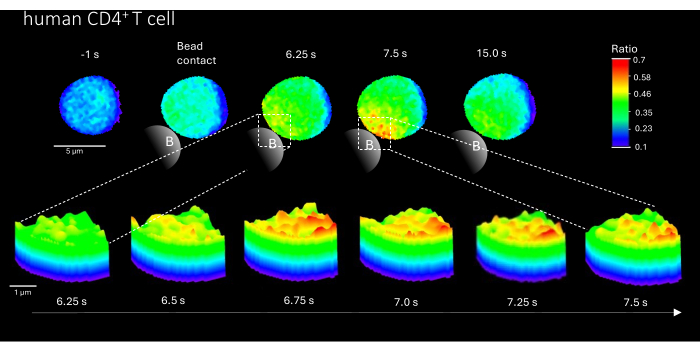
Figure 3 : Microdomaines Ca2+ dans une cellule T humaine primaire représentative après stimulation TCR. Des lymphocytes T CD4+ humains primaires ont été isolés à partir de cellules mononucléées du sang périphérique (PBMC) par tri cellulaire activé par fluorescence (FACS) à partir de couches leucocytaires et chargés de Fluo-4 AM et de Fura Red, comme décrit ci-dessus. La figure montre un lymphocyte T humain primaire 1 s avant la stimulation avec des billes enrobées d’anti-CD3 et jusqu’à 15 s après la stimulation (barre d’échelle de 5 μm), ainsi qu’un tracé de surface 3D d’un zoom avant de 6,25 s à 7,5 s dans la région de contact de la bille (barre d’échelle de 1 μm). Veuillez cliquer ici pour voir une version agrandie de cette figure.
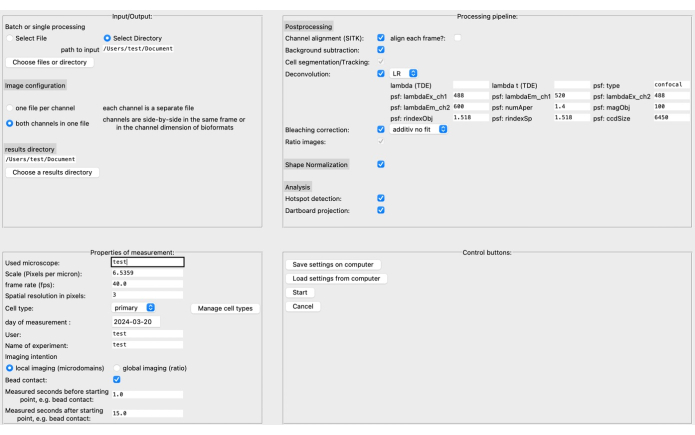
Figure 4 : Interface utilisateur graphique (GUI) de DARTS. L’interface graphique est divisée en quatre zones. Dans la zone Entrée/Sortie, vous devez fournir des informations sur les données brutes, y compris le répertoire source et la configuration de l’image (soit deux canaux par fichier, soit des canaux distincts), ainsi que le répertoire des résultats. Dans la zone Propriétés de mesure, l’expérience doit être décrite avec toutes les informations pertinentes, telles que l’échelle (microns par pixel), la fréquence d’images et l’intervalle de mesure par rapport au point de départ déterminé ultérieurement. Ensuite, un pipeline de traitement composé d’étapes de post-traitement, de normalisation de forme et d’analyse réelle (détection de microdomaines et accumulation de données de jeu de fléchettes) peut être assemblé. Enfin, les paramètres peuvent être enregistrés ou chargés à partir de l’ordinateur. Une fois l’analyse mise en place, cliquez sur Démarrer pour continuer. Pour en savoir plus sur la configuration, rendez-vous sur https://ipmi-icns-uke.github.io/DARTS/General/Usage.html. Veuillez cliquer ici pour voir une version agrandie de cette figure.
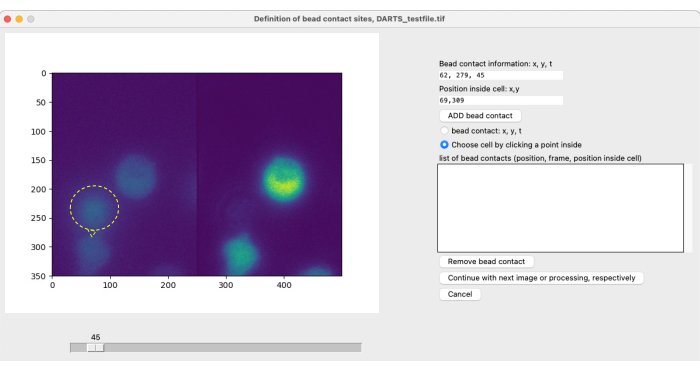
Figure 5 : Définition manuelle des contacts de talon. Si des billes sont ajoutées aux cellules pendant l’expérience, le temps de contact initial de la bille avec une cellule d’intérêt et l’emplacement de contact doivent être définis manuellement. Pour ce faire, il suffit de faire défiler les cadres à l’aide du curseur et de trouver une position (x,y) à un point temporel t. Pour remplir automatiquement le champ des informations de contact de la bille, l’utilisateur clique sur la moitié gauche de l’image du microscope à la position de contact de la bille. Ensuite, pour associer une cellule au contact de bille, l’utilisateur clique sur une position dans la cellule qui a un contact de bille. Les informations doivent être confirmées en sélectionnant ADD bering. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Nous avons décrit un protocole étendu pour l’imagerie cellulaire vivante à haute résolution des microdomaines locaux Ca2+ dans les cellules T primaires murines et humaines déclenchées par la stimulation TCR/CD3 à travers des billes enrobées d’anticorps. De plus, nous avons mis en œuvre un algorithme convivial et open-source basé sur Python pour identifier et analyser les signaux locaux Ca2+. Notamment, le protocole ne se limite pas à la détection des microdomaines Ca2+ dans le contexte de la stimulation TCR/CD3, mais est adaptable à d’autres types de cellules (immunitaires) telles que les lignées cellulaires NK (KHYG-1)12 ou les microdomaines Ca2+ indépendants du TCR10,11.
Une étape critique du protocole est la taille et le nombre de billes de stimulation. Pour imiter une synapse immunitaire, les billes doivent être de taille similaire à celle des cellules. Par conséquent, pour les lymphocytes T primaires murins et humains ainsi que pour les lignées cellulaires (Jurkat et KHYG1), nous utilisons des billes magnétiques d’un diamètre de 10 μm. De plus, chaque cellule ne doit être stimulée que par une seule bille. Par conséquent, le nombre de perles ajoutées à chaque diapositive doit être suffisant d’une part, mais s’il y a trop de perles dans le champ de vision, l’arrière-plan augmente et il n’est pas possible de détecter un seul point de temps d’activation et du côté du contact.
Le protocole utilise les colorants fluorescents Ca2+ Fluo-4 AM et FuraRed AM de manière ratiométrique, permettant ainsi l’étalonnage des données13. De plus, le protocole pourrait être adapté à d’autres paires d’indicateurs Ca2+ , mais il faut faire preuve de prudence dans le processus de sélection en termes de cinétique de liaison Ca2+ , de distribution subcellulaire et de photoblanchiment1. De plus, les conditions de charge doivent être développées et optimisées pour chaque type de cellule, mais les concentrations indiquées ici constituent un bon point de départ. Pour visualiser les microdomaines Ca2+ , le Kd des colorants Ca2+ doit être compris entre 300 et 1200 nM et le temps d’acquisition par trame doit être de ≤60 ms. Si l’intensité de fluorescence est trop faible, le jeu de filtres doit être vérifié, mais il est également possible de charger une double quantité de colorant Ca2+ dans les cellules T. Cependant, le colorant Ca2+ pourrait se déplacer à d’autres organites ou séquestrer dans des vésicules, mais il pourrait également agir comme un tampon Ca2+ et affecter les réponses Ca2+ .
L’une des limites de l’algorithme d’analyse est qu’une forme sphérique de la cellule est supposée ; Par conséquent, des types de cellules avec des morphologies différentes peuvent nécessiter l’adaptation de la boîte à outils d’analyse. L’algorithme a été utilisé pour analyser les microdomaines locaux Ca2+ dans les lymphocytes T murins primaires, ainsi que les lymphocytes T Jurkat et une lignée cellulaire NK (KHYG-1)12 et a été couronné de succès dans l’analyse des microdomaines Ca2+ pour une lignée cellulaire neuronale murine (N2a, données non publiées). En principe, le protocole et la boîte à outils d’analyse pourraient être utilisés pour analyser des types de cellules non sphériques telles que les cellules HEK293 ou HeLa, mais pour ces types de cellules, la projection du jeu de fléchettes ne peut pas être adaptée car elle est basée sur une structure ronde et une normalisation de forme des cellules. De plus, le protocole de détection des microdomaines Ca2+ initiaux localisés lors de la stimulation de billes peut être adapté pour analyser les signaux locaux Ca2+ dérivés d’autres stimuli, tels que les composés activateurs ou inhibiteurs solubles, ainsi que les microdomaines Ca2+ dépendants de l’adhésion et indépendants du TCR/CD310,11. Il est à noter qu’il est plus facile de définir un contact de bille unique en termes de temps et d’emplacement que de déterminer le point de départ de l’activation après des composés solubles.
Une limitation générale pour la détection de la formation de microdomaines Ca2+ réside dans la haute résolution temporelle et spatiale requise et le rapport signal/bruit (SNR) élevé nécessaire. Actuellement, la résolution dérivée de notre configuration atteint une résolution spatiale calculée de ~0,368 μm et une résolution temporelle de ~40 images par seconde (fps)1. Les progrès récents dans le développement de caméras et de détecteurs, ainsi que l’amélioration des colorants fluorescents, pourraient conduire à la possibilité d’obtenir des enregistrements optiques monocanaux tels qu’ils ont été décrits pour les constructions ORAI-GECI (indicateurs Ca2+ génétiquement exprimés)24 pour l’imagerie de cellules vivantes utilisant des indicateurs Ca2+ avec une résolution temporelle et spatiale plus élevée à l’avenir.
Dans l’ensemble, le protocole et l’outil d’analyse pour l’imagerie à haut niveau du microdomaine Ca2+ décrits ici peuvent être utilisés non seulement pour analyser les signaux locaux initiaux Ca2+ dans les lymphocytes T, mais peuvent également être adaptés à d’autres types de cellules pour déchiffrer l’importance de la signalisation locale Ca2+ dans ceux-ci.
Déclarations de divulgation
Les auteurs déclarent que la recherche a été menée en l’absence de toute relation commerciale ou financière qui pourrait être considérée comme un conflit d’intérêts potentiel.
Remerciements
Ce travail a été soutenu par la Deutsche Forschungsgemeinschaft (DFG) (numéro de projet 335447717 ; SFB1328, A02 à B-et RW ; A14 à ET ; le numéro de projet 516286863 à B-). Les auteurs remercient les donneurs de sang et le Département de médecine transfusionnelle de l’UKE pour leur coopération.
matériels
| Name | Company | Catalog Number | Comments |
| α-CD3 | BD Pharmingen | 553058 | |
| α-CD28 | BD Pharmingen | 553295 | |
| Anaconda | Anaconda | https://docs.anaconda.com/free/anaconda/install/index.html | |
| Bovine serum albumin | Sigma-Aldrich | A2058 | |
| Cell strainer | Biofil | CSS-013-070 | 70 µm diameter |
| Countess Automated Cell Counter | Invitrogen | A49865 | |
| Cover slips | Paul Marienfeld GmbH | 101202 | |
| DARTS | GitHub | https://github.com/IPMI-ICNS-UKE/DARTS | |
| Dulbecco’s Phosphate buffered saline | Gibco, Thermo Fisher | 14190144 | |
| EasySep CD4+ T cell Isolation Kit | Stemcell | 19852 | |
| EasySep Magnetic Multistand | Stemcell | 18010 | |
| Fluo 4-AM | Invitrogen | F14201 | |
| Fura Red-AM | Invitrogen | F3020 | |
| Gibco RPMI 1640 medium | Gibco, Thermo Fisher | 11875093 | |
| Git | GitHub | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git | |
| immersion oil | Leica | 11513859 | |
| Newborn calf serum | Sigma-Aldrich | N4637 | |
| Oracle | Oracle | https://www.oracle.com/de/java/technologies/downloads/ | |
| Penicillin/Streptamycin | Gibco, Thermo Fisher | 15240062 | |
| Poly-L-lysine | Sigma-Aldrich | P4707 | |
| Protein G magnetic beads | Merck | LSKMAGG02 | 10 µM diameter |
| Python | Python | https://www.python.org/downloads/ | |
| Silicon grease (basylone) | Bayer | 291-1220 | |
| Time-Dependent Entropy Deconvolution | GitHub | https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters | |
| Tween | q-biogene | TWEEN201 |
Références
- Wolf, I. M. A., et al. Frontrunners of T cell activation: Initial, localized Ca2+ signals mediated by NAADP and the type 1 ryanodine receptor. Sci Signal. 8 (398), 102(2015).
- Taguchi, T., Mukai, K. Innate immunity signalling and membrane trafficking. Curr Opin Cell Biol. 59, 1-7 (2019).
- Trebak, M., Kinet, J. -P. Calcium signalling in T cells. Nat Rev Immunol. 19 (3), 154-169 (2019).
- Diercks, B. -P., et al. ORAI1, STIM1/2, and RYR1 shape subsecond Ca2+ microdomains upon T cell activation. Sci Signal. 11, 0358(2018).
- Cheng, H., Lederer, W. J., Cannell, M. B. Calcium sparks: Elementary events underlying excitation-contraction coupling in heart muscle. Science. 262 (5134), 740-744 (1993).
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Berridge, M. J., Lipp, P., Bootman, M. D. The versatility and universality of calcium signalling. Nat Rev Mol Cell Biol. 1 (1), 11-21 (2000).
- Kummerow, C., Junker, C., Kruse, K., Rieger, H., Quintana, A., Hoth, M. The immunological synapse controls local and global calcium signals in T lymphocytes. Immunol Rev. 231 (1), 132-147 (2009).
- Brock, V. J., et al. P2X4 and P2X7 are essential players in basal T cell activity and Ca2+ signaling milliseconds after T cell activation. Sci Adv. 8 (5), 9770(2022).
- Weiß, M., et al. Adhesion to laminin-1 and collagen IV induces the formation of Ca2+ microdomains that sensitize mouse T cells for activation. Sci Signal. 16 (790), 9405(2023).
- Diercks, B. -P. The importance of Ca2+ microdomains for the adaptive immune response. Biochim Biophys Acta Mol Cell Res. 1871 (5), 119710(2024).
- Woelk, L. -M., et al. DARTS: an open-source Python pipeline for Ca2+ microdomain analysis in live cell imaging data. Front Immunol. 14, 1299435(2024).
- Diercks, B. -P., Werner, R., Schetelig, D., Wolf, I. M. A., Guse, A. H. High-resolution calcium imaging method for local calcium signaling. Methods Mol Biol. 1929, 27-39 (2019).
- Python. Python. , Available from: https://www.python.org/downloads/ (2024).
- Anaconda. Anaconda. , Available from: https://docs.anaconda.com/free/anaconda/install/index.html (2024).
- git. git. , Available from: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git (2024).
- DARTS. DARTS. , Available from: https://github.com/IPMI-ICNS-UKE/DARTS (2024).
- DARTS. DARTS GitHub Repository. , Available from: https://ipmi-icns-uke.github.io/DARTS/ (2024).
- Oracle. , Available from: https://www.oracle.com/de/java/technologies/downloads/ (2024).
- Time-Dependent Entropy Deconvolution. , Available from: https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters (2024).
- Gu, F., et al. Dual NADPH oxidases DUOX1 and DUOX2 synthesize NAADP and are necessary for Ca2+ signaling during T cell activation. Sci Signal. 14 (709), 3800(2021).
- Roggenkamp, H. G., et al. HN1L/JPT2: A signaling protein that connects NAADP generation to Ca2+ microdomain formation. Sci Signal. 14 (675), 5647(2021).
- Heßling, L. D., Troost-Kind, B., Weiß, M. NAADP-binding proteins - Linking NAADP signaling to cancer and immunity. Biochim Biophy. Acta Mol Cell Res. 1870 (7), 119531(2023).
- Dynes, J. L., Amcheslavsky, A., Cahalan, M. D. Genetically targeted single-channel optical recording reveals multiple Orai1 gating states and oscillations in calcium influx. Proc Natl Acad Sci U S A. 113 (2), 440-445 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon