Method Article
Bildgebung von anfänglichen Ca2+ -Mikrodomänen in primären T-Zellen
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein umfassendes Protokoll zur Verfügung, um anfängliche lokale Ca2+ -Signale, sogenannte Ca2+ -Mikrodomänen, in primären murinen und humanen T-Zellen mittels Fluoreszenzmikroskopie aufzuklären. Dieses Protokoll dient als wertvolle Ressource für Forscher, die die Ca2+ -Signalwege in Immunzellen untersuchen und ihre Funktion weiter entschlüsseln können.
Zusammenfassung
Lokale, sub-zweite Ca2+ -Signale, die als Ca2+ -Mikrodomänen bezeichnet werden, sind hochdynamische und kurzlebige Ca2+ -Signale, die zu einer globalen [Ca2+]i-Erhöhung führen und möglicherweise bereits das Schicksal einer T-Zelle bestimmen. Bei Aktivierung des T-Zell-Rezeptors wird NAADP schnell gebildet und bindet an NAADP-Bindungsproteine (HN1L/JPT2, LSM12) und ihre jeweiligen Rezeptoren (RyR1, TPC2), die auf intrazellulären Ca2+ -Speichern wie dem ER und den Lysosomen sitzen, und führt zu einer anschließenden Freisetzung und Erhöhung von [Ca2+]i. Um diese schnell und dynamisch ablaufenden Ca2+ -Signale zu erfassen, haben wir eine hochauflösende Bildgebungstechnik entwickelt, die eine Kombination aus zwei Ca2+ -Indikatoren, Fluo-4 AM und FuraRed AM, verwendet. Für die Nachbearbeitung wurde ein quelloffener, halbautomatisierter Ca2+ Microdomain-Detektionsansatz entwickelt, der auf der Programmiersprache Python basiert. Mit diesem Workflow sind wir in der Lage, Ca2+ -Mikrodomänen auf subzellulärer Ebene in primären murinen und humanen T-Zellen in Fluoreszenzvideos mit hoher zeitlicher und räumlicher Auflösung zuverlässig zu detektieren. Diese Methode kann auch auf andere Zelltypen angewendet werden, wie z.B. NK-Zellen und murine neuronale Zelllinien.
Einleitung
Die vorgestellte Fluoreszenzmikroskopietechnik ermöglicht die Visualisierung lokaler, temporaler initialer Calciumsignale (Ca2+) in primären T-Zellen der Maus, den sogenannten Ca2+ -Mikrodomänen. Ca2+ -Mikrodomänen stellen hochdynamische und kurzlebige Ca2+ -Signalereignisse dar, die eine Herausforderung für eine effektive Bildgebung und Analyse lebender Zellen darstellen1.
T-Zellen stellen aufgrund der relativen Unterschiede in der zentralen und peripheren Fluoreszenzintensität, die auf ihre kugelförmige Form und ihren geringen Durchmesser von ~6-8 μm zurückzuführen sind, eine Herausforderung für die Bildgebung von lebenden Zellen dar. Nach der Stimulation und der Bildung von Immunsynapsen erfahren T-Zellen morphologische Veränderungen, was die Bildgebung von T-Zellen weiter erschwert1. Daher ist die Anwendung einer ratiometrischen Analyse unerlässlich, die entweder durch die Aufnahme von zwei Bildern, die unterschiedliche Eigenschaften eines Ca2+ -Farbstoffs darstellen, oder durch die Verwendung einer Kombination aus zwei Ca2+ -Farbstoffen erreicht wird. Zu den anspruchsvollen Eigenschaften von Ca2+ -Mikrodomänen gehört ihre schnelle, zeitliche und räumlich begrenzte Natur. Um dies zu erfassen, müssen die verwendeten Ca2+ -Farbstoffe sowohl eine hohe basale Helligkeit als auch ein hohes Signal-Rausch-Verhältnis (SNR) aufweisen, um eine möglichst hohe zeitliche und räumliche Auflösung zu erhalten. Optimale Ergebnisse wurden mit einer Kombination aus dem Dual-Wellenlängen-Farbstoff Fura Red und dem Single-Wellenlängen-Farbstoff Fluo-4 erzielt. Co-Loading-Zellen mit Fluo-4 und Fura Red mildern die Herausforderungen, die sich aus dem starken Photobleaching von Dual-Emissions-Wellenlängenfarbstoffen und der zeitlichen Verzögerung von Dual-Excitation-Farbstoffen ergeben, und gewährleisten so die Eignung für eine schnelle Bildaufnahme. Dieser Ansatz erleichtert außerdem die Visualisierung von Formveränderungen und subtilen Bewegungen. Besondere Anforderungen werden auch an das abbildende System in Bezug auf die räumliche Auflösung gestellt, um die Visualisierung von Ca2+ -Signalen zu ermöglichen, die aus der Öffnung kleiner Kanalcluster oder sogar einzelner Kanäle1 stammen.
Der Ca2+-Signalweg spielt eine zentrale Rolle bei der Aktivierung von Immunfunktionen in T-Zellen, einschließlich der Synapsenbildung und der Zytokinproduktion und -freisetzung 2,3. Das spezifische Schicksal der Zelle wird durch die unterschiedlich ausgeprägten und lokal verteilten Ca2+-Signale, dieCa2+-Mikrodomänen3, reguliert. Bemerkenswert ist, dass diese lokalen Ca2+-Signale einem weit verbreiteten Anstieg der intrazellulären Ca2+-Spiegel in T-Zellen vorausgehen und die Bildung von Ca2+-Mikrodomänen sowohl vom Eintritt als auchvon der Freisetzung von Ca2+-1,4,5 abhängt. Bei Stimulation des T-Zell-Rezeptors (TCR)/CD3 wird die Bildung von Ca2+-freisetzenden Botenstoffen wie Nikotinsäure, Adenindinukleotidphosphat (NAADP), D-Myo-Inositol-1,4,5-trisphosphat (IP3) und zyklischer ADP-Ribose (cADPR) ausgelöst, was zu einem Anstieg der intrazellulären Ca2+-Spiegel auf bis zu 1 μM 6,7 führt. Frühe Ca2+-Signalereignisse sind mit der Freisetzung von Ca2+ aus intrazellulären Ca2+-Speichern wie dem endoplasmatischen Retikulum (ER) verbunden, wobei Kanäle wie der Ryanodinrezeptor 1 (RyR1) und derIP3-Rezeptor (IP3R) überwiegend für diese Signalübertragung verantwortlich sind. Dies löst anschließend einen extrazellulären Ca2+-Einstrom aus und führt zu einem globalen Ca2+-Signal über den speicherbetriebenen Ca2+-Eintrag (SOCE)8. Darüber hinaus gibt es andere Kanäle, die an der Ca2+-Signalgebung während der T-Zell-Aktivierung beteiligt sind9, z. B. P2X4- und P2X7-Kanäle sorgen für einen Adenosintriphosphat (ATP)-abhängigen Kationeneinstrom, der zum Anstieg des intrazellulären Ca2+-Gehalts beiträgt. Bemerkenswert ist, dass anfängliche adhäsionsabhängige Ca2+-Mikrodomänen (ADCMs) bereits vor der TCR-Stimulation gebildet werden, jedoch mit niedrigeren Ca2+-Amplituden und -Frequenzen. Diese initialen TCR-unabhängigen Ca2+-Signale dienen höchstwahrscheinlich der Migration der T-Zellen zum Entzündungsort und bereiten die T-Zellen auf die Restimulation am Infektionsort vor10,11.
Durch die Entwicklung der beschriebenen Methode zur lokalen Ca2+ -Bildgebung haben wir ein zusätzliches Werkzeug zur Erforschung des Ursprungs und der Bedeutung früher Ca2+ -Signale bei der T-Zell-Aktivierung gewonnen. Diese Methode ermöglicht es dem Anwender, kleinere, kurzlebige und schneller auftretende Ca2+ -Signale zu erkennen, als dies bisher möglich war. Darüber hinaus ermöglicht Deconvolution, Analysis, Registration, Tracking, and Shape normalization (DARTS), die Python-basierte Analysepipeline, die gemeinsame Nutzung der Analysewerkzeuge mit einem breiteren Publikum12.
Protokoll
Alle Tierversuche wurden nach den Tierschutzrichtlinien des Institutional Animal Care and Use Committee des Universitätsklinikums Hamburg-Eppendorf genehmigt und durchgeführt.
1. Isolierung von primären Maus-T-Zellen aus Lymphknoten und Milz
- Entnehmen Sie die Milz/Lymphknoten unter sterilen Bedingungen gemäß den ethischen Richtlinien und legen Sie sie in ein Röhrchen mit eiskaltem Clicks Medium (10% Fetal Calf Serum (FCS), 2 mM L-Glutamin, Penicillin/Streptomycin (P/S) 100 U/mL, 50 nM β-Mercaptoethanol).
HINWEIS: Wildtyp- und KO-Maus-T-Zellen werden auf die gleiche Weise isoliert. - Die Zentrifuge auf 4 °C abkühlen lassen.
- Legen Sie die Milz und die Lymphknoten in ein 70-μm-Zellsieb in einer sterilen Petrischale und fügen Sie Milzisolationsmedium (RPMI-Medium + 7,5 % NCS + 1 % P/S) zu einem Gesamtvolumen von 20 mL hinzu.
- Brechen Sie die Milz gründlich, aber vorsichtig mit einem Kunststoffmörtel auf. Übertragen Sie die Zellen anschließend in ein 50-ml-Zentrifugenröhrchen.
HINWEIS: Im 50-ml-Zentrifugenröhrchen sollte sich nun ein Gesamtvolumen von 20 mL befinden. Milz und Lymphknoten sollten vollständig zerstört werden. Ab diesem Zeitpunkt wird auf Eis gearbeitet. Die Zellen auf Eis zu halten, verlangsamt ihren Stoffwechsel und verhindert den Zelltod, wodurch sichergestellt wird, dass sie lebensfähig und funktionell intakt bleiben. Dies trägt dazu bei, ihren physiologischen Zustand über einen längeren Zeitraum zu erhalten, was besonders wichtig für genaue nachgelagerte Experimente ist. - Die Zellsuspension bei 300 x g für 5 min bei 4 °C zentrifugieren.
- Entsorgen Sie den Überstand und stellen Sie sicher, dass das resultierende Pellet in 2 ml Dulbecco-Phosphat-gepufferter Kochsalzlösung (DPBS) (ohne Ca2+ und Mg2+) resorbiert wird. In ein neues 12-ml-Zentrifugenröhrchen umfüllen.
2. Negative Selektion von CD4+ T-Zellen
HINWEIS: Für die negative Selektion von CD4+ T-Zellen wird ein T-Zell-Isolationskit verwendet, das einen FcR-Blocker, biotinylierte Antikörper gegen Nicht-CD4+ T-Zellen und Streptavidin-beschichtete Magnetpartikel enthält.
- Geben Sie 20 μl/ml Maus-FcR-Blocker aus dem T-Zell-Isolationskit in die Zellsuspension.
- Geben Sie außerdem 100 μl der CD4+ T-Zell-Isolationscocktails der Maus in einer Konzentration von 50 μl/ml hinzu, mischen Sie gut und inkubieren Sie sie 10 Minuten lang bei Raumtemperatur (RT).
- Streptavidin-beschichtete magnetische Partikel 30 s lang vortexen, 150 μl zur Zellsuspension geben, gut mischen und 2 min 30 s bei RT inkubieren.
- Geben Sie DPBS in die Zellsuspension und füllen Sie sie auf ein Gesamtvolumen von 7 ml. Stellen Sie sicher, dass Sie vorsichtig auf und ab pipettieren und dann 2 min 30 s bei RT im Magneten inkubieren.
- Anschließend werden die angereicherten T-Zellen vorsichtig im Überstand gesammelt und in ein neues 12-ml-Zentrifugenröhrchen überführt.
- Zellen bei 300 x g bei RT für 5 min zentrifugieren.
- Entsorgen Sie den Überstand.
- Resuspendieren Sie die isolierten und angereicherten CD4+ T-Zellen in 1 ml DPBS und pipetieren Sie gründlich auf und ab. Inkubieren Sie die Mischung für 2 min 30 s im Magneten bei RT, um sicherzustellen, dass keine Bead-Kontamination vorliegt.
- Sammeln Sie den Überstand, geben Sie ihn in ein neues Zentrifugenröhrchen und zählen Sie die angereicherten T-Zellen.
- Um die Zellen zu zählen, färben Sie die Zellen mit Trypanblau und zählen Sie mit einem automatisierten Zellzähler.
HINWEIS: Es ist am besten, die isolierten CD4+ T-Zellen direkt zu laden und am selben Tag der Isolierung abzubilden. Primäre murine T-Zellen können jedoch ohne jegliche Stimulation über Nacht in einem Inkubator bei 37 °C mit 5 % CO2 in einem Isolationsmedium (RPMI 1640 + 10 % FCS + P/S) kultiviert werden.
3. Beladung von primären CD4+ T-Zellen der Maus
HINWEIS: Zur Messung der freien zytosolischen Ca2+ -Konzentrationen werden in den Ca2+ -Bildgebungsexperimenten fluoreszierende und membranpermeable Farbstoffe verwendet. Eine Kombination aus Fluo-4-acetoxymethylester (AM) und dem ratiometrischen Farbstoff Fura Red-AM dient als Indikator für die schnelle Detektion lokaler Ca2+ -Signale. Stellen Sie sicher, dass Sie im Dunkeln arbeiten, während Sie fluoreszierende Farbstoffe verwenden.
- 2-5 x 106 Zellen für 5 min, 300 x g zentrifugieren, den Überstand verwerfen und in 480 μl T-Zellmedium (RPMI 1640, 10 % FCS) mit 10 μM Fluo-4-AM (Lager: [1 mM]) und 20 μM Fura Red-AM (Lager: [4 mM]) resuspendieren.
- Inkubieren Sie zunächst 20 Minuten lang bei RT unter der Bank im Dunkeln und decken Sie den Falken mit Alufolie ab.
HINWEIS: Überschreiten Sie nicht die erste Inkubationszeit, da die Ca2+ -Indikatoren in DMSO verdünnt sind, was bei längerer Einwirkzeit bei geringem Gesamtvolumen für die Zellen schädlich sein könnte. Halten Sie die Zellen nach dem Laden aufgrund der Lichtempfindlichkeit der Farbstoffe vor Licht geschützt. - Am Ende der anfänglichen 20-minütigen Inkubation fügen Sie den Zellen 2 ml T-Zellmedium hinzu und setzen Sie die Inkubation für weitere 30 Minuten im Dunkeln bei RT fort.
- Die Zellsuspension wird bei 300 x g 5 min lang bei RT zentrifugiert.
- Entsorgen Sie den Überstand und reinigen Sie das Pellet, indem Sie es mit 2 mL Ca2+ Messpuffer (140 mM NaCl, 5 mM KCl, 1 mM MgSO4, 1 mM CaCl2, 20 mM HEPES, 1 mM NaH2PO4, 5 mM Glukose, pH 7,4) waschen, gefolgt von einer Zentrifugation bei 300 x g für 5 min bei RT.
- Zum Schluss wird der Überstand verworfen und mit dem Messpuffer Ca2+ wieder suspendiert. Stellen Sie die Zellzahl auf ~100.000 Zellen pro 10 μl ein.
HINWEIS: Für eine Messung wird ein Volumen von 10 μl verwendet. Vermeiden Sie eine Überverdünnung der Zellen, da die Zellzahl unter dem Mikroskop möglicherweise weiter angepasst werden muss, um ein Optimum für die Messbedingungen zu erreichen. - Lassen Sie die Zellen ~20 Minuten lang inkubieren, um eine vollständige Zellinfiltration und Entesterung der Farbstoffe zu gewährleisten. Lagern Sie die Zellen bis zur Messung auf Eis und im Dunkeln.
HINWEIS: Geladene primäre murine T-Zellen können bis zu ~4 h nach dem Laden verwendet werden.
4. Lokale Ca2+ -Bildgebung
- Vorbereitung der Objektträger
- Deckgläser für Mikroskope (24 mm x 46 mm) mit Rinderserumalbumin (BSA, 5 mg/ml) und Poly-L-Lysin (PLL, 0,1 mg/ml) beschichten (Abbildung 1A). Lassen Sie BSA ~20 Minuten einwirken, bevor Sie PLL auftragen.
HINWEIS: Die Beschichtung erleichtert die Zelladhäsion. Für eine korrekte Zelladhäsion verteilen Sie sowohl BSA als auch PLL genau, bis keine Streifen mehr sichtbar sind. - Um Reaktionskammern zu erzeugen, kleben Sie wiederverwendbare Gummi-O-Ringe mit einer Silikonpaste auf die Objektträger (Abbildung 1B,C).
HINWEIS: Für die Bildaufnahme können auch andere Objektträger/Kammern oder Platten wie 35-mm-Objektträger, wiederverwendbare Metallkammern oder 8-, 24- oder 48-Well-Platten verwendet werden.
- Deckgläser für Mikroskope (24 mm x 46 mm) mit Rinderserumalbumin (BSA, 5 mg/ml) und Poly-L-Lysin (PLL, 0,1 mg/ml) beschichten (Abbildung 1A). Lassen Sie BSA ~20 Minuten einwirken, bevor Sie PLL auftragen.
- Beschichtung von Protein G Magnetkügelchen (10 μm Durchmesser) mit Antikörpern
- Mischen Sie vorsichtig die Protein-Magnetbead-Suspension und geben Sie 12,5 μl in ein neues Röhrchen.
- Stellen Sie das Röhrchen auf einen Magnetständer, um den Aufbewahrungspuffer zu entfernen, und lassen Sie die Kügelchen zum Magneten wandern. Entfernen Sie den Vorratspuffer vorsichtig, indem Sie ihn auf die gegenüberliegende Seite des Magneten pipettieren.
- Um den restlichen Lagerpuffer zu entfernen, waschen Sie die Kügelchen durch Zugabe von 500 μl PBS-T (PBS, 0,1 % Tween) und lassen Sie sie 10 s lang vortexen. Legen Sie das Rohr wieder in den Magnetständer und entfernen Sie den Puffer.
- Um die Kügelchen mit Antikörpern zu beschichten, resuspendieren Sie sie in 7,5 μl PBS-T und fügen Sie 5 μl Anti-CD3 (0,5 mg/ml) bzw. Anti-CD28 (0,5 mg/ml) hinzu. 30-60 min inkubieren und kontinuierlich bei RT mischen.
- Waschen Sie die beschichteten Kügelchen dreimal mit 500 μl PBS-T, anschließend einmal mit 500 μl Ca2+ Messpuffer mit dem Magnetständer.
- Resuspendieren Sie die Kügelchen in 200-400 μl Ca2+-Messpuffer.
HINWEIS: Überprüfen Sie die Raupendichte während der Messungen und verdünnen Sie sie bei Bedarf weiter.
- Lokale Ca2+ Bildgebungsmikroskopie
HINWEIS: Die Bildgebung erfolgt mit einem Hellfeld-Lichtmikroskop mit 100-facher Vergrößerung, das mit einer Xenon-Bogenlampe als Lichtquelle ausgestattet ist. Die Frames werden im 14-Bit-Modus mit zweifachem Binning unter Verwendung einer elektronenmultiplizierenden ladungsgekoppelten Kamera aufgenommen. Um die Emissionswellenlängen beider Farbstoffe aufzuzeichnen und aufzuteilen, wird ein Dual-View-Modul verwendet, das die folgenden Filter in nm aufweist (z. B. 480/40; bs, 495; em1, 542/50; em2, 650/57). Das Imaging-Setup umfasst einen Erfassungs-Hub und eine Imaging-Software für die Bildaufnahme.- Legen Sie 10 μL der geladenen Zellen auf den vorbereiteten Objektträger und lassen Sie sie 3-5 Minuten lang am Objektträger anhaften.
- Geben Sie vorsichtig 80 μl Ca2+ Messpuffer in den Objektträger.
- Wählen Sie die 100x Öl-Immersionslinse und tragen Sie einen kleinen Tropfen Immersionsöl auf. Legen Sie den Objektträger auf den Mikroskoptisch.
- Passen Sie den Fokus im Hellfeldmodus an, wählen Sie sorgfältig ein Sichtfeld mit bis zu 10 Zellen aus, die nicht miteinander in Kontakt stehen, und nehmen Sie ein Bild auf.
HINWEIS: Überlappende und sich berührende Zellen lassen sich später nur schwer analysieren.- Schalten Sie die Lampe ein, überprüfen Sie die Fluoreszenz und die Belastung der T-Zellen und vergleichen Sie die Zellen in beiden Kanälen, um sicherzustellen, dass sie nicht voraktiviert sind.
- Stellen Sie sicher, dass Sie vor und nach der Messung Bilder des Sichtfelds sowohl für Hellfeld- als auch für Fluoreszenzkanäle aufnehmen, um die Zellbewegung und -belastung zu überprüfen.
- Starten Sie die Messung und erfassen Sie die Basalaktivität für 1 Minute mit einer Erfassungsrate von 1 Bild pro 5 s.
- Fügen Sie nach 1 Minute 10 μl Verbindung/Stimulans (Kügelchen oder Stimulator/Inhibitor) hinzu und messen Sie insgesamt 3 Minuten lang mit 40 Bildern pro s oder der maximal möglichen Bildrate.
HINWEIS: Das Hinzufügen von Perlen ist der entscheidende Schritt; Stellen Sie sicher, dass die Perlen in der Nähe des Anregungslichts auf dem Objektträger angebracht werden, ohne den Objektträger zu bewegen. Repräsentative Beispiele für Ca2+ -Mikrodomänen in primären CD4+ T-Zellen nach Bead-Kontakt von WT und P2x4-/- und P2x7-/- Mäusen sind in Abbildung 2 dargestellt.
5. Nachbearbeitung/ Datenanalyse
HINWEIS: Für die Bildverarbeitung und Datenanalyse wird die Python-basierte Open-Source-Pipeline DARTS verwendet. Es wurde von Woelk et al.12 auf der Grundlage von Arbeiten von Diercks et al.13 entwickelt.
- Installation der DARTS-Pipeline
- Installieren Sie Python 3.10.014, anaconda15 und git16, und klonen Sie das GitHub-Repository mit dem Terminalbefehl git clone: https://github.com/IPMI-ICNS-UKE/DARTS.git
- Erstellen Sie eine Conda-Umgebung mit conda create --name DARTS und installieren Sie alle notwendigen Python-Pakete mit pip install <package>.
HINWEIS: Befolgen Sie die Installationsanweisungen im DARTS GitHub-Repository17,18, um weitere Informationen zu erhalten. - Stellen Sie vor der Verwendung von Bioformaten sicher, dass eine Java Runtime Environment korrekt installiert ist19.
- Nutzung der DARTS-Pipeline
HINWEIS: Nach der Installation kann DARTS über das Terminalfenster gestartet werden.- Navigieren Sie zur lokalen Kopie des DARTS-Repositorys (cd path/to/DARTS), die die main.py Datei enthält. Stellen Sie sicher, dass die Conda-Umgebung aktiviert ist (conda activate DARTS).
- Führen Sie DARTS aus, indem Sie python main.py in das Terminalfenster eingeben und die Eingabetaste drücken.
- Geben Sie in der grafischen Benutzeroberfläche (GUI) im oberen linken Bereich das Quell- und Ergebnisverzeichnis sowie das Bildformat an (siehe Abbildung 3).
- Definieren Sie die Eigenschaften der Messung, wie z. B. den Maßstab (Pixel pro Mikrometer), die Bildrate, den Zellentyp usw. Die Verarbeitungspipeline kann auf der Grundlage der spezifischen Bilddaten und der Forschungsfrage zusammengestellt werden.
HINWEIS: Lesen Sie mehr über die Dekonvolutionsparameter, z. B. in der entsprechenden Dokumentation20. - Speichern Sie die Einstellungen auf dem Computer, bevor Sie auf Start klicken. Eine detailliertere Beschreibung der GUI finden Sie in der DARTS Dokumentation.
- Ca2+ Mikrodomänen-Bildgebung und Perlenkontakte
HINWEIS: Für die lokale Bildgebung und Stimulation mit Kügelchen und die anschließende Bildanalyse müssen die zellspezifischen Kügelchenkontakte vom Benutzer definiert werden, um den Startpunkt (t = 0) des interessierenden Messzeitraums zu identifizieren (siehe Abbildung 4). Ein Kügelchenkontakt ist definiert als die Kontaktstelle einer (stimulierenden) Perle mit einer interessierenden Zelle. Es besteht aus der Angabe von (1) der Raupenkontaktzeit, z. B. Rahmen 300, und (2) der Raupenkontaktposition relativ zum Zentrum der Zelle, z. B. 1 Uhr oder 12 Uhr. Mit der aktuellen DART-Version muss der Benutzer die Informationen für jeden Raupenkontakt manuell definieren und eingeben. Gehen Sie wie folgt vor, um Raupenkontakte zu definieren:- Verwenden Sie den Schieberegler, um die Zeit des Kontakts zwischen einer Perle und einer interessierenden Zelle zu bestimmen.
- Wählen Sie im Optionsmenü auf der rechten Seite den Raupenkontakt: x, y, t.
- Wählen Sie die Stelle auf der linken Seite des Bildes aus, an der sich die Zelle und die Perle berühren.
- Wählen Sie Zelle auswählen, indem Sie auf einen Punkt darin klicken. Klicken Sie auf die Zelle, die durch diese Perle stimuliert wird, vorzugsweise in der Mitte.
- Klicken Sie auf BEAD-Kontakt hinzufügen.
- Wiederholen Sie die Schritte 5.3.1 bis 5.3.5 für jeden weiteren Raupenkontakt in dieser Datei. Sobald alle Raupenkontakte definiert sind, klicken Sie auf die Schaltfläche Weiter .
- Fahren Sie mit weiteren Dateien fort. Nachdem die letzte Datei erreicht wurde, initiiert das Skript automatisch die Analyse aller Dateien.
- Datenanalyse
- Im Ergebnisordner finden Sie die verarbeiteten Verhältnisbilder, Microdomain-Daten für jede Zelle im Zeitverlauf (inkl. Lokalisierung, Amplitude, Größe), Quelldaten für die Dartscheibeprojektion und weitere Ressourcen.
- Um Dartscheiben zu erstellen, navigieren Sie zu dem Ordner /src/analysis/ im DARTS-Ordner (Muster: cd path/to/DARTS/src/analysis), der das Skript DartboardPlotGUI.py enthält. Geben Sie dann python DartboardPlotGUI.py ein und drücken Sie die Eingabetaste.
- Stellen Sie sicher, dass die erforderlichen Informationen bereitgestellt werden und dass die Tabellenkalkulationsdateien aus dem Ergebnisordner korrekt als Quelldateien für die Erstellung der Dartscheibe ausgewählt sind.
Ergebnisse
In diesem Protokoll haben wir eine aktualisierte Methode zur Abbildung und Analyse anfänglicher Ca2+-Mikrodomänen in primären Maus-T-Zellen vorgestellt, die auf früheren Arbeiten unserer Gruppe 1,13 basiert. Dieser Ansatz war maßgeblich daran beteiligt, die Beteiligung von CRAC-Kanälen wie ORAI1, STIM1 und STIM2 sowie von intrazellulären Ca2+-Freisetzungskanälen wie RyR1 an frühen Ca2+-Signalereignissen zu entschlüsseln4.
Zu diesem Zweck untersuchten wir die spontane Bildung von Ca2+-Mikrodomänen, indem wir nicht-stimulierte primäre Maus-Orai1-/-, Stim1-/-, Stim2-/-- und Ryr1-/- abbildeten und sie mit WT-primären Maus-T-Zellen verglichen. Die Analyse der Ca2+-Mikrodomänenbildung umfasste die Signalbeginngeschwindigkeit, die Ca2+-Amplitude und die Anzahl der Signale pro konfokaler Ebene. Mit Ausnahme von Stim2-/- T-Zellen zeigten alle KO T-Zellen im Vergleich zu WT-Zellen eine signifikante Abnahme der lokalenCa2+-Signale und eine reduzierte basale freie zytosolischeCa2+-Konzentration. Dies führte uns zu dem Schluss, dass die Bildung von Ca2+-Mikrodomänen eng mit der Interaktion von ORAI1, STIM1 und RyR1verbunden ist 4. Darüber hinaus ist es uns gelungen, spontane Ca2+-Mikrodomänen an der Plasmamembran zu identifizieren und zu charakterisieren. Diese Ca2+-Mikrodomänen wurden durch eineCa2+-Amplitude von 290 nm ± 12 nm charakterisiert. Die Verwendung eines farbcodierten Ansatzes für Ca2+-Signale ermöglichte die Visualisierung von Ca2+-Mikrodomänen in der gesamten Zelle. Die Ergebnisse unterstreichen ferner das schnelle Einsetzen von Ca2+-Mikrodomänen, die innerhalb von Millisekunden sichtbar sind, und die Fähigkeit dieser Methode, Ca2+-Signale mit einer Langlebigkeit von wenigen Millisekunden zu detektieren4. Diese spontanen Ca2+-Mikrodomänen wurden später als adhäsionsabhängige Ca2+-Mikrodomänen (ADCM) identifiziert, die nicht nur von SOCE abhängen, sondern auch über die FAK/PLC-γ/IP 3-Signalkaskade10 und die Beteiligung von P2X49 wirken. Darüber hinaus war diese Technik von grundlegender Bedeutung für die Bestätigung der dualen Oxidasen 1 und 2 (DUOX1/2) als NAADP-produzierende Enzyme21 und HN1L/JPT222 als eines der neu entdeckten NAADP-bindenden Proteine23.
Abbildung 2 zeigt repräsentative Beispiele für Ca2+ -Mikrodomänen in primären CD4+ T-Zellen nach Bead-Kontakt von WT sowie P2x4-/- und P2x7-/- Mäusen. Die Zellen wurden mit denCa-2+ -Farbstoffen Fluo-4 AM und Fura Red AM beladen und mit einer Erfassungsrate von 25 ms (40 Frames/s) abgebildet. Um die Bildung von T-Zell-Synapsen nachzuahmen, wurden die Zellen mit anti-CD3/anti-CD28-beschichteten Kügelchen stimuliert. Die anfängliche Ca2+ -Mikrodomänenbildung wurde 1 s vor und bis zu 15 s nach dem Bead-Kontakt mit Hilfe der DART-Pipeline analysiert. Nach dem Kontakt mit der Bead zeigte die WT-Zelle in der ersten Sekunde nach der Stimulation an der Kontaktstelle der Beads eine schnelle Bildung von Ca2+ -Mikrodomänen (Abbildung 2A). Diese Ca2+ -Mikrodomänen dehnten sich in den folgenden 15 s nach dem Bead-Kontakt weiter in der Zelle aus. Im Gegensatz zur WT-Zelle zeigten die P2x4-/- und P2x7-/- Zellen (Abbildung 2B) eine verminderte Ca2+ -Mikrodomänenbildung nach Bead-Stimulation, ebenso wie die P2x4-/-a niedrigere Basalebene vor dem Bead-Kontakt. Diese repräsentativen Befunde stehen im Einklang mit den zuvor veröffentlichten Ergebnissen von Brock et al.9, die auf eine Ca2+ -Mikrodomänenbildung in WT-T-Zellen direkt nach dem Bead-Kontakt über 15 s und niedrigere Signale pro Frame in P2x4-/- und P2x7-/- Zellen hinweisen. Darüber hinaus wurde die Amplitude in P2x4-/-Zellen signifikant reduziert, was die Rolle des purinergen Signalwegs in adhäsionsabhängigen Ca2+ -Mikrodomänen weiter belegt.
Darüber hinaus kann diese Methode auch verwendet werden, um anfängliche Ca2+ -Mikrodomänen in primären humanen CD4+ T-Zellen sichtbar zu machen (Abbildung 3). In Übereinstimmung mit primären murinen T-Zellen werden anfängliche Ca2+ -Mikrodomänen an der Kontaktstelle der Kügelchen evoziert. Die gesamteCa2+ -Antwort scheint jedoch auf einer anderen Zeitskala zu erfolgen als bei murinen CD4+ T-Zellen.
Die manuelle Analyse lokaler Ca2+ -Signale ist nicht durchführbar, da sie recht mühsam und subjektiv für den einzelnen Untersucher ist. Aus diesem Grund haben wir zuvor einen Algorithmus in MATLAB Simulink entwickelt, der seine Bildverarbeitungs- und Optimierungs-Toolboxen für die Nachbearbeitung13 zur Analyse lokaler Ca2+ -Mikrodomänen verwendet.
Vor kurzem haben wir eine neue Open-Source-Postprocessing-Pipeline namens DARTS für die Ca2+ -Mikrodomänenanalyse in der hochauflösenden Lebendzellbildgebung mit der Softwareplattform Python12 entwickelt. Hier können je nach Präferenz des Benutzers verschiedene Dekonvolutionsalgorithmen ausgewählt, eine Zellformnormalisierung zur Kompensation morphologischer Zellformänderungen durchgeführt und mikroskop- und messspezifische Parameter definiert werden (z. B. Skala, Bildrate, gemessene Zeit) (Abbildung 4).
Nach der Auswahl der Parameter für die Ca2+ Microdomain-Analyse wird für jede einzelne Messung ein zweites Pop-up-Fenster geöffnet, in dem der Bead-Kontakt definiert werden kann (Abbildung 5). Um den Raupenkontakt zu definieren, kann der Benutzer mit dem Schieberegler manuell durch die TIFF-Datei scrollen und den Raupenkontaktrahmen einzeln auswählen. Der Raupenkontakt wird durch Klicken auf die Raupenkontaktstelle (Abbildung 5, Raupen- und Raupenkontakt durch gelben Ring und Pfeil gekennzeichnet) sowie durch Zellauswahl ausgewählt. Dieser Schritt muss für jede interessierende Zelle wiederholt werden. Abschließend wird die automatisierte Bildnachbearbeitung angewendet und die Ergebnisdaten werden zusammengefasst und in einer Tabelle gespeichert.
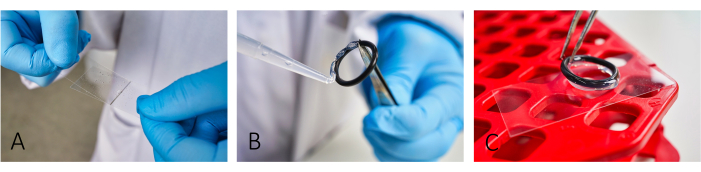
Abbildung 1: Arbeitsablauf bei der Vorbereitung des Objektträgers für die Bildgebung. (A) Fügen Sie sowohl BSA als auch PLL hinzu und verteilen Sie sie mit einem zweiten Glasabdeckglas auf dem Objektträger. (B,C) Um eine Kammer zu bauen, kleben Sie die Gummi-O-Ringe mit Silikonfett auf den Schlitten. Stellen Sie sicher, dass der gesamte Ring mit einer dünnen Fettschicht bedeckt ist, um eine ordnungsgemäße Isolierung der Kammer zu gewährleisten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
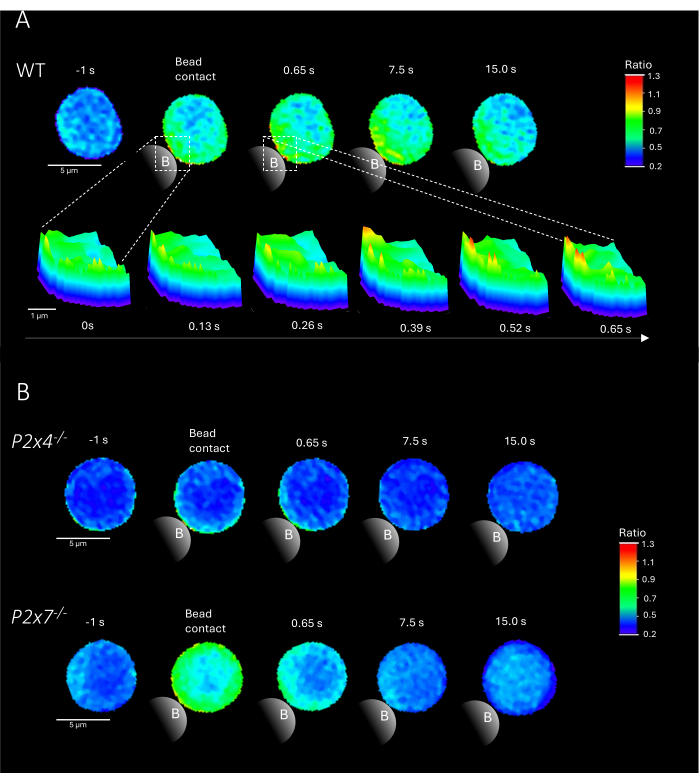
Abbildung 2: Repräsentative Zellen von T-Zell-Rezeptor-abhängigen Ca2+ -Mikrodomänen in einer primären murinen Wildtyp-(A), P2x4-/- oder P2x7-/-(B) CD4+ T-Zelle. CD4+ T-Zellen wurden negativ isoliert und mit Fluo-4 AM und Fura Red beladen, wie oben beschrieben. T-Zellen wurden mit der DARTS-Pipeline analysiert, was zu vergleichbaren Zellbildern mit zuvor veröffentlichten Ergebnissenführte 9. (A) Primäre WT-T-Zelle 1 s vor der Stimulation mit anti-CD3/anti-CD28-beschichteten Kügelchen und bis zu 15 s nach der Stimulation (Maßstabsbalken 5 μm), sowie 3D-Oberflächendiagramm eines Zoom-Hineinzooms von 0 s bis 0,65 s im Kugelkontaktbereich (Maßstabsbalken 1 μm). (B) Obere Spur: repräsentative P2x4-/- primäre T-Zelle 1 s vor und bis zu 15 s nach der Bead-Stimulation. Untere Spur: repräsentative P2x7-/- primäre T-Zelle 1 s vor und bis zu 15 s nach der Bead-Stimulation. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
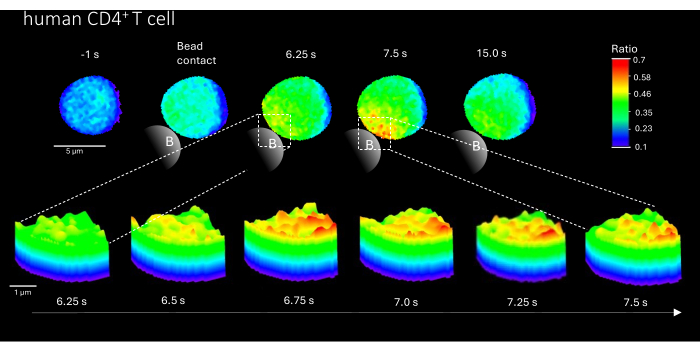
Abbildung 3: Ca2+ -Mikrodomänen in einer repräsentativen primären humanen T-Zelle nach TCR-Stimulation. Primär humane CD4+ T-Zellen wurden aus mononukleären Zellen des peripheren Blutes (PBMCs) durch Fluoreszenz-aktivierte Zellsortierung (FACS) aus Buffy-Coats isoliert und mit Fluo-4 AM und Fura Red beladen, wie oben beschrieben. Die Abbildung zeigt eine primäre humane T-Zelle 1 s vor der Stimulation mit anti-CD3-beschichteten Kügelchen und bis zu 15 s nach der Stimulation (Maßstabsbalken 5 μm) sowie einen 3D-Oberflächenplot eines Zoom-Ins von 6,25 s auf 7,5 s im Kugelkontaktbereich (Maßstabsbalken 1 μm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
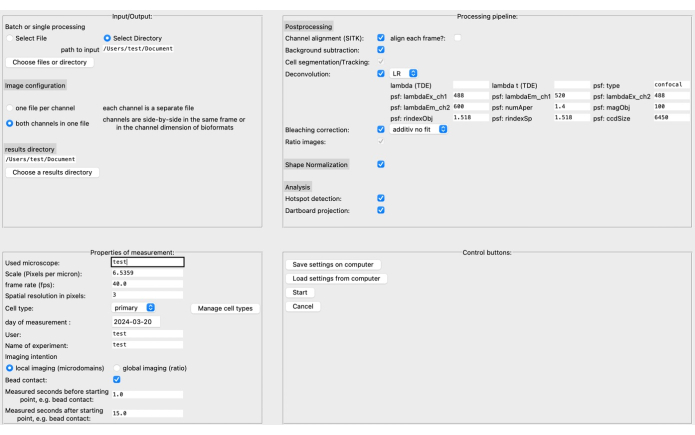
Abbildung 4: Die grafische Benutzeroberfläche (GUI) von DARTS. Die GUI ist in vier Bereiche unterteilt. Im Bereich Eingabe/Ausgabe müssen Sie Informationen über die Rohdaten angeben, einschließlich des Quellverzeichnisses und der Bildkonfiguration (entweder zwei Kanäle pro Datei oder separate Kanäle) sowie des Ergebnisverzeichnisses. Im Bereich Eigenschaften der Messung muss das Experiment mit allen relevanten Informationen beschrieben werden, wie z. B. Maßstab (Mikrometer pro Pixel), Bildrate und Messintervall relativ zum später ermittelten Startpunkt. Als nächstes kann eine Verarbeitungspipeline bestehend aus Nachbearbeitungsschritten, Formnormalisierung und der eigentlichen Analyse (Microdomain-Erkennung und Dartboard-Datenakkumulation) zusammengestellt werden. Schließlich können die Einstellungen auf dem Computer gespeichert oder von diesem geladen werden. Sobald die Analyse eingerichtet ist, klicken Sie auf Start , um fortzufahren. Um mehr über die Einrichtung zu erfahren, besuchen Sie https://ipmi-icns-uke.github.io/DARTS/General/Usage.html. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
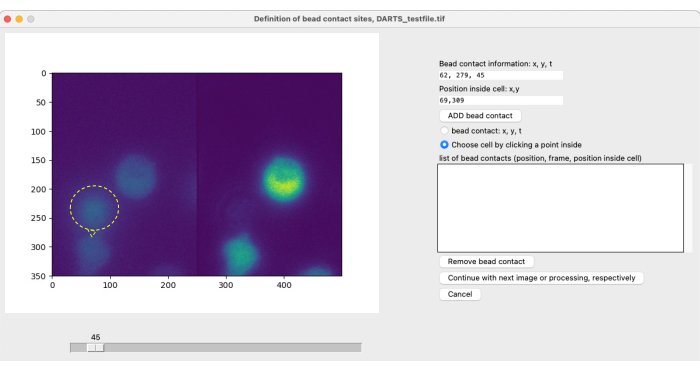
Abbildung 5: Manuelle Definition von Perlenkontakten. Wenn den Zellen während des Experiments Kügelchen hinzugefügt werden, müssen die anfängliche Kontaktzeit der Kügelchen mit einer interessierenden Zelle und die Kontaktstelle manuell definiert werden. Dazu scrollt man mit dem Schieberegler durch die Frames und findet eine Position (x,y) zu einem Zeitpunkt t. Um das Feld für die Kontaktinformationen der Perle automatisch auszufüllen, klickt der Benutzer auf die linke Hälfte des Mikroskopbildes an der Position des Perle-Kontakts. Um als Nächstes eine Zelle mit dem Raupenkontakt zu verknüpfen, klickt der Benutzer auf eine Position innerhalb der Zelle, die über einen Raupenkontakt verfügt. Die Angaben müssen durch Auswahl von ADD bead bestätigt werden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Wir haben ein umfangreiches Protokoll für die hochauflösende Lebendzellbildgebung von lokalen Ca2+-Mikrodomänen in primären murinen und humanen T-Zellen beschrieben, die durch TCR/CD3-Stimulation durch Antikörper-beschichtete Kügelchen ausgelöst werden. Darüber hinaus haben wir einen benutzerfreundlichen und quelloffenen Python-basierten Algorithmus implementiert, um lokale Ca2+-Signale zu identifizieren und zu analysieren. Bemerkenswert ist, dass das Protokoll nicht auf die Detektion von Ca2+-Mikrodomänen im Rahmen der TCR/CD3-Stimulation beschränkt ist, sondern auch auf andere (Immun-)Zelltypen wie NK-Zelllinien (KHYG-1)12 oder TCR-unabhängige Ca2+-Mikrodomänen10,11 adaptierbar ist.
Ein kritischer Schritt innerhalb des Protokolls ist die Größe und Anzahl der stimulierenden Kügelchen. Um eine Immunsynapse nachzuahmen, sollten die Kügelchen ähnlich groß wie die Zellen sein. Daher verwenden wir für primäre murine und humane T-Zellen sowie Zelllinien (Jurkat und KHYG1) magnetische Kügelchen mit einem Durchmesser von 10 μm. Darüber hinaus sollte jede Zelle nur durch eine einzige Kugel stimuliert werden. Daher sollte einerseits die Anzahl der Perlen, die zu jedem Objektträger hinzugefügt werden, ausreichend sein, aber wenn sich zu viele Perlen im Sichtfeld befinden, nimmt der Hintergrund zu, und es ist nicht möglich, einen einzigen Aktivierungszeitpunkt und eine Kontaktseite zu erkennen.
Das Protokoll verwendet die fluoreszierenden Ca2+ -Farbstoffe Fluo-4 AM und FuraRed AM auf ratiometrische Weise, was eine Kalibrierung der Daten13 ermöglicht. Darüber hinaus könnte das Protokoll an andere Ca2+ -Indikatorpaare angepasst werden, aber bei der Auswahl ist Vorsicht geboten, was die Ca2+ -Bindungskinetik, die subzelluläre Verteilung und das Photobleaching1 betrifft. Darüber hinaus müssen die Beladungsbedingungen für jeden einzelnen Zelltyp entwickelt und optimiert werden, aber die hier angegebenen Konzentrationen sind ein guter Ausgangspunkt. Um Ca2+ -Mikrodomänen sichtbar zu machen, sollte die Kd der Ca2+ -Farbstoffe im Bereich von 300-1200 nM liegen und die Erfassungszeit pro Frame sollte ≤60 ms betragen. Ist die Fluoreszenzintensität zu gering, muss das Filterset überprüft werden, es ist aber auch möglich, eine doppelte Menge Ca2+ Farbstoff in T-Zellen zu laden. Der Ca2+ -Farbstoff könnte sich jedoch zu anderen Organellen verlagern oder an Vesikel sequestrieren, aber er könnte auch alsCa2+ -Puffer fungieren und dieCa2+ -Reaktionen beeinflussen.
Eine Einschränkung des Analysealgorithmus besteht darin, dass eine kugelförmige Form der Zelle angenommen wird; Daher kann es sein, dass Zelltypen mit unterschiedlichen Morphologien eine Anpassung der Analyse-Toolbox erfordern. Der Algorithmus wurde zur Analyse lokaler Ca2+-Mikrodomänen in primären murinen T-Zellen sowie von Jurkat-T-Zellen und einer NK-Zelllinie (KHYG-1)12 verwendet und war erfolgreich bei der Analyse von Ca2+-Mikrodomänen für eine murine neuronale Zelllinie (N2a, unveröffentlichte Daten). Prinzipiell könnte der Protokoll- und Analysebaukasten verwendet werden, um nicht-sphärische Zelltypen wie HEK293- oder HeLa-Zellen zu analysieren, aber für diese Zelltypen kann die Darterscheibenprojektion nicht angepasst werden, da sie auf einer runden Struktur- und Formnormalisierung der Zellen basiert. Darüber hinaus kann das Protokoll zum Nachweis lokalisierter anfänglicher Ca2+-Mikrodomänen nach Bead-Stimulation angepasst werden, um lokale Ca2+-Signale zu analysieren, die von anderen Stimuli abgeleitet werden, wie z. B. löslichen aktivierenden oder hemmenden Verbindungen sowie adhäsionsabhängigen und TCR/CD3-unabhängigen Ca2+-Mikrodomänen10,11. Bemerkenswert ist, dass es einfacher ist, einen einzelnen Kügelchenkontakt in Bezug auf Zeit und Ort zu definieren, als den Startpunkt der Aktivierung nach löslichen Verbindungen zu bestimmen.
Eine generelle Einschränkung für die Detektion derCa2+ -Mikrodomänenbildung liegt in der geforderten hohen zeitlich-räumlichen Auflösung und dem notwendigen hohen Signal-Rausch-Verhältnis (SNR). Derzeit erreicht die aus unserem Aufbau abgeleitete Auflösung eine berechnete räumliche Auflösung von ~0,368 μm und eine zeitliche Auflösung von ~40 Bildern pro Sekunde (fps)1. Jüngste Fortschritte in der Kamera- und Detektorentwicklung sowie die Verbesserung von Fluoreszenzfarbstoffen könnten dazu führen, dass optische Einkanal-Aufzeichnungen, wie sie für ORAI-GECI (genetisch exprimierte Ca2+ -Indikatoren) beschrieben wurden, in Zukunft für die Lebendzellbildgebung unter Verwendung von Ca2+- Indikatoren mit höherer zeitlicher und räumlicher Auflösung erreicht werden können.
Zusammengenommen kann das hier beschriebene Protokoll und Analysewerkzeug für hochauflösende Ca2+ -Mikrodomänen-Bildgebung nicht nur zur Analyse anfänglicher lokaler Ca2+ -Signale in T-Zellen verwendet werden, sondern auch auf andere Zelltypen angepasst werden, um die Bedeutung der lokalen Ca2+ -Signalübertragung in diesen zu entschlüsseln.
Offenlegungen
Die Autoren erklären, dass die Untersuchung in Ermangelung einer kommerziellen oder finanziellen Beziehung durchgeführt wurde, die als potenzieller Interessenkonflikt angesehen werden könnte.
Danksagungen
Diese Arbeit wurde gefördert durch die Deutsche Forschungsgemeinschaft (DFG) (Projektnummer 335447717; SFB1328, A02 bis B-PD und RW; A14 nach ET; Projektnummer 516286863 zu B-PD). Die Autoren danken den Blutspendern und der Klinik für Transfusionsmedizin am UKE für die Zusammenarbeit.
Materialien
| Name | Company | Catalog Number | Comments |
| α-CD3 | BD Pharmingen | 553058 | |
| α-CD28 | BD Pharmingen | 553295 | |
| Anaconda | Anaconda | https://docs.anaconda.com/free/anaconda/install/index.html | |
| Bovine serum albumin | Sigma-Aldrich | A2058 | |
| Cell strainer | Biofil | CSS-013-070 | 70 µm diameter |
| Countess Automated Cell Counter | Invitrogen | A49865 | |
| Cover slips | Paul Marienfeld GmbH | 101202 | |
| DARTS | GitHub | https://github.com/IPMI-ICNS-UKE/DARTS | |
| Dulbecco’s Phosphate buffered saline | Gibco, Thermo Fisher | 14190144 | |
| EasySep CD4+ T cell Isolation Kit | Stemcell | 19852 | |
| EasySep Magnetic Multistand | Stemcell | 18010 | |
| Fluo 4-AM | Invitrogen | F14201 | |
| Fura Red-AM | Invitrogen | F3020 | |
| Gibco RPMI 1640 medium | Gibco, Thermo Fisher | 11875093 | |
| Git | GitHub | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git | |
| immersion oil | Leica | 11513859 | |
| Newborn calf serum | Sigma-Aldrich | N4637 | |
| Oracle | Oracle | https://www.oracle.com/de/java/technologies/downloads/ | |
| Penicillin/Streptamycin | Gibco, Thermo Fisher | 15240062 | |
| Poly-L-lysine | Sigma-Aldrich | P4707 | |
| Protein G magnetic beads | Merck | LSKMAGG02 | 10 µM diameter |
| Python | Python | https://www.python.org/downloads/ | |
| Silicon grease (basylone) | Bayer | 291-1220 | |
| Time-Dependent Entropy Deconvolution | GitHub | https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters | |
| Tween | q-biogene | TWEEN201 |
Referenzen
- Wolf, I. M. A., et al. Frontrunners of T cell activation: Initial, localized Ca2+ signals mediated by NAADP and the type 1 ryanodine receptor. Sci Signal. 8 (398), 102(2015).
- Taguchi, T., Mukai, K. Innate immunity signalling and membrane trafficking. Curr Opin Cell Biol. 59, 1-7 (2019).
- Trebak, M., Kinet, J. -P. Calcium signalling in T cells. Nat Rev Immunol. 19 (3), 154-169 (2019).
- Diercks, B. -P., et al. ORAI1, STIM1/2, and RYR1 shape subsecond Ca2+ microdomains upon T cell activation. Sci Signal. 11, 0358(2018).
- Cheng, H., Lederer, W. J., Cannell, M. B. Calcium sparks: Elementary events underlying excitation-contraction coupling in heart muscle. Science. 262 (5134), 740-744 (1993).
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Berridge, M. J., Lipp, P., Bootman, M. D. The versatility and universality of calcium signalling. Nat Rev Mol Cell Biol. 1 (1), 11-21 (2000).
- Kummerow, C., Junker, C., Kruse, K., Rieger, H., Quintana, A., Hoth, M. The immunological synapse controls local and global calcium signals in T lymphocytes. Immunol Rev. 231 (1), 132-147 (2009).
- Brock, V. J., et al. P2X4 and P2X7 are essential players in basal T cell activity and Ca2+ signaling milliseconds after T cell activation. Sci Adv. 8 (5), 9770(2022).
- Weiß, M., et al. Adhesion to laminin-1 and collagen IV induces the formation of Ca2+ microdomains that sensitize mouse T cells for activation. Sci Signal. 16 (790), 9405(2023).
- Diercks, B. -P. The importance of Ca2+ microdomains for the adaptive immune response. Biochim Biophys Acta Mol Cell Res. 1871 (5), 119710(2024).
- Woelk, L. -M., et al. DARTS: an open-source Python pipeline for Ca2+ microdomain analysis in live cell imaging data. Front Immunol. 14, 1299435(2024).
- Diercks, B. -P., Werner, R., Schetelig, D., Wolf, I. M. A., Guse, A. H. High-resolution calcium imaging method for local calcium signaling. Methods Mol Biol. 1929, 27-39 (2019).
- Python. Python. , Available from: https://www.python.org/downloads/ (2024).
- Anaconda. Anaconda. , Available from: https://docs.anaconda.com/free/anaconda/install/index.html (2024).
- git. git. , Available from: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git (2024).
- DARTS. DARTS. , Available from: https://github.com/IPMI-ICNS-UKE/DARTS (2024).
- DARTS. DARTS GitHub Repository. , Available from: https://ipmi-icns-uke.github.io/DARTS/ (2024).
- Oracle. , Available from: https://www.oracle.com/de/java/technologies/downloads/ (2024).
- Time-Dependent Entropy Deconvolution. , Available from: https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters (2024).
- Gu, F., et al. Dual NADPH oxidases DUOX1 and DUOX2 synthesize NAADP and are necessary for Ca2+ signaling during T cell activation. Sci Signal. 14 (709), 3800(2021).
- Roggenkamp, H. G., et al. HN1L/JPT2: A signaling protein that connects NAADP generation to Ca2+ microdomain formation. Sci Signal. 14 (675), 5647(2021).
- Heßling, L. D., Troost-Kind, B., Weiß, M. NAADP-binding proteins - Linking NAADP signaling to cancer and immunity. Biochim Biophy. Acta Mol Cell Res. 1870 (7), 119531(2023).
- Dynes, J. L., Amcheslavsky, A., Cahalan, M. D. Genetically targeted single-channel optical recording reveals multiple Orai1 gating states and oscillations in calcium influx. Proc Natl Acad Sci U S A. 113 (2), 440-445 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten