Method Article
Imaging iniziale di microdomini Ca2+ in cellule T primarie
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, forniamo un protocollo completo per risolvere i segnali locali iniziali di Ca2+ , noti come microdomini Ca2+ , in cellule T primarie murine e umane utilizzando la microscopia a fluorescenza. Questo protocollo è una risorsa preziosa per i ricercatori che esaminano le vie di segnalazione del Ca2+ all'interno delle cellule immunitarie e per svelare ulteriormente la loro funzione.
Abstract
I segnali locali di Ca2+ inferiori al secondo, chiamati microdomini di Ca2+ , sono segnali di Ca2+ altamente dinamici e di breve durata, che determinano un'elevazione globale di [Ca2+]i e potrebbero già determinare il destino di una cellula T. Dopo l'attivazione del recettore delle cellule T, NAADP si forma rapidamente, legandosi alle proteine leganti NAADP (HN1L/JPT2, LSM12) e ai rispettivi recettori (RyR1, TPC2) che si trovano su riserve intracellulari di Ca2+ , come l'ER e i lisosomi, e portando al successivo rilascio e all'elevazione di [Ca2+]i. Per catturare questi segnali di Ca2+ veloci e dinamici, abbiamo sviluppato una tecnica di imaging ad alta risoluzione utilizzando una combinazione di due indicatori di Ca2+ , Fluo-4 AM e FuraRed AM. Per la post-elaborazione, è stato sviluppato un approccio di rilevamento del microdominio Ca2+ open source e semi-automatizzato basato sul linguaggio di programmazione Python. Utilizzando questo flusso di lavoro, siamo in grado di rilevare in modo affidabile microdomini Ca2+ a livello subcellulare in cellule T primarie murine e umane in video di fluorescenza ad alta risoluzione temporale e spaziale. Questo metodo può essere applicato anche ad altri tipi di cellule, come le cellule NK e le linee cellulari neuronali murine.
Introduzione
La tecnica di microscopia a fluorescenza presentata consente la visualizzazione di segnali locali e temporali iniziali di calcio (Ca2+) in cellule T primarie di topo, denominate microdomini Ca2+ . I microdomini del Ca2+ rappresentano eventi di segnalazione del Ca2+ altamente dinamici e di breve durata, ponendo sfide per l'imaging e l'analisi efficaci delle cellule vive1.
Le cellule T sono impegnative per l'imaging di cellule vive a causa delle differenze relative nell'intensità della fluorescenza centrale e periferica, che possono essere attribuite alla loro forma sferica e al loro piccolo diametro di ~6-8 μm. Dopo la stimolazione e la formazione delle sinapsi immunitarie, le cellule T subiscono cambiamenti morfologici, complicando ulteriormente l'imaging delle cellule T1. Pertanto, l'impiego di un'analisi raziometrica diventa imperativo, ottenuto registrando due immagini che rappresentano diverse proprietà di un colorante Ca2+ o utilizzando una combinazione di due coloranti Ca2+ . Le caratteristiche impegnative dei microdomini Ca2+ includono la loro natura rapida, temporalmente e spazialmente limitata. Per catturare questo, i coloranti Ca2+ utilizzati devono possedere sia un'elevata luminosità basale che un alto rapporto segnale/rumore (SNR) per ottenere la massima risoluzione temporale e spaziale possibile. Risultati ottimali sono stati ottenuti utilizzando una combinazione del colorante a doppia lunghezza d'onda Fura Red e del colorante a lunghezza d'onda singola Fluo-4. Le celle di co-caricamento con Fluo-4 e Fura Red mitigano le sfide poste dal forte fotobleaching dei coloranti a doppia emissione e dal ritardo temporale associato ai coloranti a doppia eccitazione, garantendo l'idoneità per l'acquisizione rapida delle immagini. Questo approccio facilita ulteriormente la visualizzazione dei cambiamenti di forma e dei movimenti sottili. Al sistema di imaging vengono inoltre poste esigenze speciali in termini di risoluzione spaziale per consentire la visualizzazione di segnali di Ca2+ provenienti dall'apertura di piccoli gruppi di canali o anche di singoli canali1.
La segnalazione del Ca2+ svolge un ruolo fondamentale nell'attivazione delle funzioni immunitarie all'interno delle cellule T, tra cui la formazione delle sinapsi e la produzione e il rilascio di citochine 2,3. Il destino specifico della cellula è regolato attraverso i segnali del Ca2+ diversamente pronunciati e distribuiti localmente, i microdomini del Ca2+ 3. In particolare, questi segnali locali di Ca2+ precedono un aumento diffuso dei livelli intracellulari di Ca2+ nelle cellule T e la formazione di microdomini di Ca2+ dipende sia dall'ingresso chedal rilascio di Ca2+ 1,4,5. Dopo la stimolazione del recettore delle cellule T (TCR)/CD3, viene innescata la formazione di secondi messaggeri che rilasciano Ca2+, come l'acido nicotinico adenina dinucleotide fosfato (NAADP), il D-mio-inositolo 1,4,5-trisfosfato (IP3) e l'ADP ribosio ciclico (cADPR), portando ad un aumento dei livelli intracellulari di Ca2+ fino a 1 μM 6,7. Gli eventi precoci di segnalazione del Ca2+ sono legati al rilascio di Ca2+ da depositi intracellulari di Ca2+ come il reticolo endoplasmatico (ER), con canali come il recettore della rianodina 1 (RyR1) e il recettore IP3 (IP3R) che sono prevalentemente responsabili di questa segnalazione. Questo successivamente innesca l'afflusso extracellulare di Ca2+ e si traduce in un segnale globale di Ca2+ tramite l'ingresso di Ca2+ gestito dal negozio (SOCE)8. Inoltre, ci sono altri canali coinvolti nella segnalazione del Ca2+ durante l'attivazione delle cellule T9, ad esempio, i canali P2X4 e P2X7 assicurano l'afflusso di cationi dipendenti dall'adenosina trifosfato (ATP), contribuendo all'aumento del Ca2+ intracellulare. Sorprendentemente, i microdomini iniziali di Ca2+ dipendenti dall'adesione (ADCM) si formano già prima della stimolazione del TCR, ma con ampiezze e frequenze di Ca2+ inferiori. Questi segnali iniziali di Ca2+ indipendenti dal TCR molto probabilmente servono alla migrazione delle cellule T verso il sito dell'infiammazione e preparano le cellule T per la restimolazione nel sito di infezione10,11.
Sviluppando il metodo descritto per l'imaging locale del Ca2+ , abbiamo ottenuto uno strumento aggiuntivo per esplorare l'origine e il significato dei primi segnali del Ca2+ nell'attivazione delle cellule T. Questo metodo consente all'utente di rilevare segnali di Ca2+ più piccoli, di breve durata e più rapidi di quanto fosse possibile in precedenza. Inoltre, la pipeline di analisi basata su Python (DARTS), la deconvoluzione, l'analisi, la registrazione, il tracciamento e la normalizzazione delle forme, consente di condividere gli strumenti di analisi con un pubblico più ampio12.
Protocollo
Tutti gli esperimenti sugli animali sono stati approvati ed eseguiti in conformità con le linee guida sul benessere degli animali del Comitato istituzionale per la cura e l'uso degli animali presso il Centro medico universitario di Amburgo-Eppendorf.
1. Isolamento di cellule T primarie di topo da linfonodi e milza
- Raccogliere la milza/linfonodi in condizioni sterili secondo le linee guida etiche e metterli in una provetta con terreno Clicks ghiacciato (10% siero fetale di vitello (FCS), 2 mM di L-glutammina, penicillina/streptomicina (P/S) 100 U/mL, 50 nM β-mercaptoetanolo).
NOTA: Le cellule T di topo wild type e KO vengono isolate allo stesso modo. - Raffreddare la centrifuga a 4 °C.
- Collocare la milza e i linfonodi in un colino cellulare da 70 μm in una capsula di Petri sterile e aggiungere il terreno di isolamento della milza (terreno RPMI + 7,5 % NCS + 1% P/S) per un volume totale di 20 ml.
- Distruggere accuratamente ma delicatamente la milza usando un mortaio di plastica. Trasferire successivamente le cellule in una provetta da centrifuga da 50 mL.
NOTA: Un volume totale di 20 mL dovrebbe ora essere nella provetta da centrifuga da 50 mL. La milza e i linfonodi dovrebbero essere completamente distrutti. Da questo punto in poi lavora sul ghiaccio. Mantenere le cellule sul ghiaccio rallenta il loro metabolismo e previene la morte cellulare, assicurando che rimangano vitali e funzionalmente intatte. Questo aiuta a preservare il loro stato fisiologico per un periodo di tempo più lungo, il che è particolarmente importante per esperimenti accurati a valle. - Centrifugare la sospensione cellulare a 300 x g per 5 minuti a 4 °C.
- Scartare il surnatante e garantire la risospensione del pellet risultante in 2 mL di soluzione salina tamponata con fosfato di Dulbco (DPBS) (senza Ca2+ e Mg2+). Trasferire in una nuova provetta da centrifuga da 12 mL.
2. Selezione negativa delle cellule T CD4+
NOTA: Per la selezione negativa delle cellule T CD4+ , viene utilizzato un kit di isolamento delle cellule T contenente un bloccante FcR, anticorpi biotinilati contro le cellule T non CD4+ e particelle magnetiche rivestite di streptavidina.
- Aggiungere 20 μL/mL di bloccante FcR di topo dal kit di isolamento delle cellule T alla sospensione cellulare.
- Inoltre, aggiungere 100 μl di cocktail di isolamento delle cellule T CD4+ di topo a una concentrazione di 50 μl/mL, mescolare bene e incubare per 10 minuti a temperatura ambiente (RT).
- Vortex particelle magnetiche rivestite di streptavidina per 30 s, aggiungere 150 μl alla sospensione cellulare, mescolare bene e incubare per 2 min 30 s a RT.
- Aggiungere DPBS alla sospensione cellulare, riempiendola fino a un volume totale di 7 mL. Assicurarsi di pipettare delicatamente su e giù, quindi incubare nel magnete per 2 minuti e 30 secondi a RT.
- Successivamente, raccogliere delicatamente le cellule T arricchite nel surnatante e trasferirle in una nuova provetta da centrifuga da 12 mL.
- Centrifugare le celle a 300 x g a RT per 5 min.
- Scartare il surnatante.
- Risospendere le cellule T CD4+ isolate e arricchite in 1 mL di DPBS, pipettando accuratamente su e giù. Incubare la miscela per 2 minuti e 30 s nel magnete a RT per garantire l'assenza di contaminazione delle microsfere.
- Raccogliere il surnatante, trasferirlo in una nuova provetta da centrifuga e contare le cellule T arricchite.
- Per contare le cellule, colorare le cellule con il blu di tripano e contare utilizzando un contatore di celle automatizzato.
NOTA: È meglio caricare direttamente le cellule T CD4+ isolate e visualizzarle lo stesso giorno dell'isolamento. Tuttavia, le cellule T murine primarie possono essere coltivate senza alcuna stimolazione durante la notte in un incubatore a 37 °C con il 5% di CO2 in un terreno di isolamento (RPMI 1640 + 10% FCS + P/S).
3. Caricamento delle cellule T CD4+ primarie di topo
NOTA: Per misurare le concentrazioni di Ca2+ citosolico libero, negli esperimenti di imaging del Ca2+ vengono impiegati coloranti fluorescenti e permeabili alla membrana. Una combinazione di Fluo-4-acetossimetilestere (AM) e del colorante raziometrico Fura Red-AM funge da indicatore per il rilevamento rapido dei segnali locali di Ca2+ . Assicurati di lavorare al buio mentre usi coloranti fluorescenti.
- Centrifugare 2-5 x 106 celle per 5 minuti, 300 x g, scartare il surnatante e risospendere in 480 μL di terreno di coltura per cellule T (RPMI 1640, 10% FCS) con 10 μM di Fluo-4-AM (stock: [1 mM]) e 20 μM di Fura Red-AM (stock: [4 mM]).
- Incubare inizialmente per 20 minuti a RT sotto la panca al buio, coprendo il falco con un foglio di alluminio.
NOTA: Non superare il primo tempo di incubazione, poiché gli indicatori di Ca2+ sono diluiti in DMSO, il che potrebbe essere dannoso per le cellule in un tempo di esposizione più lungo nel basso volume totale. Mantenere le celle al riparo dalla luce dopo il caricamento a causa della sensibilità alla luce dei coloranti. - Al termine dell'incubazione iniziale di 20 minuti, aggiungere 2 mL di terreno di coltura a cellule T alle cellule e continuare l'incubazione per altri 30 minuti al buio a RT.
- Centrifugare la sospensione cellulare a 300 x g per 5 minuti a RT.
- Scartare il surnatante e pulire il pellet lavandolo con 2 mL di tampone dosatore Ca2+ (140 mM NaCl, 5 mM KCl, 1 mM MgSO4, 1 mM CaCl2, 20 mM HEPES, 1 mM NaH2PO4, 5 mM glucosio, pH 7,4) seguita da centrifugazione a 300 x g per 5 min a RT.
- Infine, scartare il surnatante e risospenderlo con il tampone di misurazione del Ca2+ . Regolare il numero di celle a ~100.000 celle per 10 μl.
NOTA: Per una misurazione viene utilizzato un volume di 10 μl. Evitare di diluire eccessivamente le cellule, poiché potrebbe essere necessario regolare ulteriormente il conteggio delle cellule al microscopio per raggiungere un valore ottimale per le condizioni di misurazione. - Lasciare incubare le cellule per ~20 minuti per garantire la completa infiltrazione cellulare e la deesterificazione dei coloranti. Conservare le cellule sul ghiaccio e al buio fino alla misurazione.
NOTA: Le cellule T murine primarie caricate possono essere utilizzate fino a ~4 ore dopo il caricamento.
4. Imaging locale di Ca2+
- Preparazione dei vetrini
- Rivestire i vetrini coprioggetti del microscopio (24 mm x 46 mm) con albumina sierica bovina (BSA, 5 mg/mL) e poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Lasciare riposare BSA per ~20 minuti prima di applicare il PLL.
NOTA: Il rivestimento facilita l'adesione delle cellule. Per una corretta adesione cellulare, stendere accuratamente sia BSA che PLL fino a quando non sono più visibili striature. - Per creare camere di reazione, incollare gli O-ring in gomma riutilizzabili ai vetrini utilizzando una pasta siliconica (Figura 1B, C).
NOTA: Per l'acquisizione delle immagini è possibile utilizzare altri vetrini/camere o piastre come vetrini da 35 mm, camere metalliche riutilizzabili o piastre da 8, 24 o 48 pozzetti.
- Rivestire i vetrini coprioggetti del microscopio (24 mm x 46 mm) con albumina sierica bovina (BSA, 5 mg/mL) e poli-L-lisina (PLL, 0,1 mg/mL) (Figura 1A). Lasciare riposare BSA per ~20 minuti prima di applicare il PLL.
- Rivestimento di sfere magnetiche di proteina G (10 μm di diametro) con anticorpi
- Miscelare delicatamente la sospensione di microsfere magnetiche proteiche e trasferire 12,5 μl in una nuova provetta.
- Posiziona il tubo su un supporto magnetico per rimuovere il buffer di stoccaggio e lascia che le perline migrino verso il magnete. Rimuovere delicatamente il tampone di conservazione pipettandolo sul lato opposto del magnete.
- Per rimuovere l'eventuale buffer di stoccaggio rimanente, lavare le perle aggiungendo 500 μL di PBS-T (PBS, 0,1% Tween) e agitare per 10 s. Riposizionare il tubo nel supporto magnetico e rimuovere il tampone.
- Per rivestire le perle con anticorpi, risospenderle in 7,5 μL di PBS-T e aggiungere 5 μL di anti-CD3 (0,5 mg/mL) e anti-CD28 (0,5 mg/mL), rispettivamente. Incubare per 30-60 minuti con miscelazione continua a RT.
- Lavare le perle rivestite tre volte con 500 μL di PBS-T, quindi lavare una volta con 500 μL di tampone di misura Ca2+ utilizzando il supporto magnetico.
- Risospendere le perle in 200-400 μL di tampone di misura Ca2+.
NOTA: Controllare la densità del cordone durante le misurazioni, diluire ulteriormente se necessario.
- Microscopia per immagini locale Ca2+
NOTA: L'imaging viene eseguito utilizzando un microscopio ottico a campo chiaro con un ingrandimento di 100 volte, dotato di una lampada ad arco allo xeno come sorgente luminosa. I fotogrammi vengono acquisiti in modalità a 14 bit con binning a due pieghe utilizzando una fotocamera ad accoppiamento di carica con moltiplicazione di elettroni. Per registrare e suddividere le lunghezze d'onda di emissione di entrambi i coloranti, viene utilizzato un modulo a doppia visualizzazione, dotato dei seguenti filtri in nm (es, 480/40; bs, 495; em1, 542/50; em2, 650/57). La configurazione di imaging include un hub di acquisizione e un software di imaging per l'acquisizione delle immagini.- Posizionare 10 μl delle celle caricate sul vetrino preparato e lasciarle attaccare al vetrino per 3-5 minuti.
- Aggiungere delicatamente 80 μl di tampone di misura Ca2+ al vetrino.
- Seleziona la lente da immersione in olio 100x e applica una piccola goccia di olio da immersione. Posizionare il vetrino sul tavolo del microscopio.
- Regola la messa a fuoco nella modalità campo chiaro, seleziona attentamente un campo visivo con un massimo di 10 celle che non sono in contatto tra loro e acquisisci un'immagine.
NOTA: Le celle sovrapposte e toccanti saranno difficili da analizzare in seguito.- Accendi la lampada, controlla la fluorescenza e il carico delle cellule T e confronta le cellule in entrambi i canali per verificare che non siano preattivate.
- Assicurarsi di scattare foto del campo visivo sia per il campo chiaro che per i canali di fluorescenza prima e dopo la misurazione per verificare il movimento e il carico delle celle.
- Avvia la misurazione e cattura l'attività basale per 1 minuto con una velocità di acquisizione di 1 fotogramma ogni 5 s.
- Aggiungere 10 μL di composto/stimolante (microsfere o stimolatore/inibitore) dopo 1 minuto e misurare per un totale di 3 minuti utilizzando 40 fotogrammi al s o il frame rate massimo possibile.
NOTA: L'aggiunta di perline è il passaggio cruciale; Assicurarsi che le perline vengano aggiunte vicino alla luce di eccitazione sul vetrino senza muovere il vetrino. Esempi rappresentativi di microdomini Ca2+ nelle cellule T CD4+ primarie dopo contatto con le perle da WT e topi P2x4-/- e P2x7-/- sono mostrati nella Figura 2.
5. Post-elaborazione/analisi dei dati
NOTA: per l'elaborazione delle immagini e l'analisi dei dati, viene utilizzata la pipeline open source DARTS basata su Python. È stato sviluppato da Woelk et al.12 sulla base del lavoro di Diercks et al.13.
- Installazione del gasdotto DARTS
- Installa Python 3.10.014, anaconda15 e git16 e clona il repository GitHub utilizzando il comando del terminale git clone: https://github.com/IPMI-ICNS-UKE/DARTS.git
- Crea un ambiente conda con conda create --name DARTS e installa tutti i pacchetti Python necessari con pip install <package>.
NOTA: Segui le istruzioni di installazione nel repository GitHub DARTS17,18 per informazioni più dettagliate. - Prima di utilizzare i bioformati, assicurarsi che sia installato correttamente un Java Runtime Environment19.
- Utilizzo della pipeline DARTS
NOTA: Una volta installato, DARTS può essere avviato dalla finestra del terminale.- Passa alla copia locale del repository DARTS (cd path/to/DARTS) che contiene il file main.py. Assicurati che l'ambiente conda sia attivato (conda activate DARTS).
- Esegui DARTS digitando python main.py nella finestra del terminale e premendo Invio.
- Nell'interfaccia utente grafica (GUI), specificare la directory di origine e dei risultati, nonché il formato dell'immagine nella sezione in alto a sinistra (vedere la Figura 3).
- Definisci le proprietà della misurazione, come la scala (pixel per micrometro), la frequenza dei fotogrammi, il tipo di cella, ecc. La pipeline di elaborazione può essere assemblata in base ai dati dell'immagine specifici e alla domanda di ricerca.
NOTA: Per ulteriori informazioni sui parametri di deconvoluzione, ad esempio, consultare la documentazione corrispondente20. - Salvare le impostazioni sul computer prima di fare clic su Avvia. Una descrizione più dettagliata della GUI è disponibile nella documentazione DARTS.
- Imaging a microdominio Ca2+ e contatti a microsfere
NOTA: Per l'imaging locale e la stimolazione con le perle e la successiva analisi dell'immagine, i contatti delle perle specifici della cellula devono essere definiti dall'utente per identificare il punto di partenza (t = 0) del periodo di misurazione di interesse (vedere la Figura 4). Un contatto con la perlina è definito come il sito di contatto di una perlina (stimolante) con una cella di interesse. Consiste nella specificazione di (1) il tempo di contatto del cordone, ad esempio il fotogramma 300, e (2) la posizione del contatto del cordone rispetto al centro della cella, ad esempio le ore 1 o le ore 12. Con l'attuale versione DARTS, l'utente deve definire e inserire manualmente le informazioni per ogni contatto del tallone. Per definire i contatti del cordone, attenersi alla procedura seguente:- Usa il cursore per individuare il tempo di contatto tra una perlina e una cella di interesse.
- Nel menu delle opzioni sul lato destro, selezionare il contatto del cordone: x, y, t.
- Seleziona il punto sul lato sinistro dell'immagine in cui la cella e il cordone entrano in contatto.
- Seleziona Scegli cella facendo clic su un punto all'interno. Fare clic sulla cellula stimolata da questa perlina, preferibilmente al centro.
- Fare clic su AGGIUNGI contatto perlina.
- Ripetere i passaggi 5.3.1- 5.3.5 per ogni contatto aggiuntivo del tallone in questo file. Una volta definiti tutti i contatti del tallone, fare clic sul pulsante Continua .
- Procedi con file aggiuntivi. Dopo aver raggiunto l'ultimo file, lo script avvierà automaticamente l'analisi di tutti i file.
- Analisi dei dati
- Trova le immagini del rapporto elaborate, i dati del microdominio per ogni cella nel tempo (incl. localizzazione, ampiezza, dimensione), i dati di origine per la proiezione del bersaglio e altre risorse nella cartella dei risultati.
- Per creare freccette, vai alla cartella /src/analysis/ all'interno della cartella DARTS (pattern: cd path/to/DARTS/src/analysis) contenente lo script DartboardPlotGUI.py. Quindi, digita python DartboardPlotGUI.py e premi invio.
- Assicurarsi che vengano fornite le informazioni necessarie e che i file del foglio di calcolo dalla cartella dei risultati siano accuratamente selezionati come file di origine per la generazione del bersaglio.
Risultati
In questo protocollo, abbiamo delineato un metodo aggiornato per visualizzare e analizzare i microdomini iniziali di Ca2+ nelle cellule T primarie di topo sulla base di precedenti lavori del nostro gruppo 1,13. Questo approccio è stato determinante per svelare il coinvolgimento dei canali CRAC come ORAI1, STIM1 e STIM2, così come dei canali di rilascio intracellulare di Ca2+ come RyR1 nei primi eventi di segnalazione del Ca2+ 4.
Per fare ciò, abbiamo studiato la formazione spontanea di microdomini Ca2+ mediante imaging di topi primari non stimolati Orai1-/-, Stim1-/-, Stim2-/- e Ryr1-/- e li abbiamo confrontati con le cellule T primarie di topo WT. L'analisi della formazione di microdomini Ca2+ comprendeva la velocità di insorgenza del segnale, l'ampiezza del Ca2+ e il numero di segnali per piano confocale. In particolare, ad eccezione delle cellule T Stim2-/-, tutte le cellule T KO hanno mostrato una significativa diminuzione dei segnali locali di Ca2+ e una ridotta concentrazione di Ca2+ citosolico libero basale rispetto alle cellule WT. Questo ci ha portato a concludere che la formazione di microdomini di Ca2+ è strettamente legata all'interazione di ORAI1, STIM1 e RyR14. Inoltre, abbiamo identificato e caratterizzato con successo microdomini spontanei di Ca2+ sulla membrana plasmatica. Questi microdomini di Ca2+ erano caratterizzati da un'ampiezza di Ca2+ di 290 nm ± 12 nm. L'utilizzo di un approccio codificato a colori per i segnali di Ca2+ ha permesso la visualizzazione di microdomini di Ca2+ in tutta la cellula. I risultati hanno ulteriormente evidenziato la rapida insorgenza di microdomini Ca2+, visibili entro millisecondi, e la capacità di questo metodo di rilevare segnali Ca2+ con una longevità di pochi millisecondi4. Questi microdomini spontanei del Ca2+ sono stati successivamente identificati come microdomini del Ca2+ dipendenti dall'adesione (ADCM), dipendenti non solo da SOCE, ma anche agenti attraverso la cascata di segnalazione FAK/PLC-γ/IP3 10 e il coinvolgimento di P2X49. Inoltre, questa tecnica è stata fondamentale per confermare le doppie ossidasi 1 e 2 (DUOX1/2) come enzimi21 produttori di NAADP e HN1L/JPT222 come una delle proteine leganti NAADP23 recentemente scoperte.
La Figura 2 mostra esempi rappresentativi di microdomini Ca2+ nelle cellule T CD4+ primarie dopo il contatto con le perline da WT e nei topi P2x4-/- e P2x7-/-. Le cellule sono state caricate con i coloranti Ca2+ Fluo-4 AM e Fura Red AM e sono state sottoposte a imaging a una velocità di acquisizione di 25 ms (40 fotogrammi/s). Per imitare la formazione delle sinapsi delle cellule T, le cellule sono state stimolate con perle rivestite anti-CD3/anti-CD28. La formazione iniziale del microdominio Ca2+ è stata analizzata 1 s prima e fino a 15 s dopo il contatto con il cordone utilizzando la pipeline DARTS. Al contatto con la perlina, la cellula WT ha mostrato una rapida formazione di microdomini Ca2+ nel primo secondo dopo la stimolazione nel sito di contatto della perlina (Figura 2A). Questi microdomini di Ca2+ si sono ulteriormente espansi in tutta la cellula nei successivi 15 s dopo il contatto con la perlina. A differenza delle cellule WT, le cellule P2x4-/- e P2x7-/- (Figura 2B) hanno mostrato una ridotta formazione di microdomini Ca2+ dopo la stimolazione della perlina, così come per il livello basale P2x4-/-a inferiore prima del contatto con la perlina. Questi risultati rappresentativi sono in linea con i risultati precedentemente pubblicati da Brock et al.9, che indicano la formazione di microdomini Ca2+ nelle cellule T WT direttamente dopo il contatto con le perline oltre 15 s e segnali inferiori per frame nelle cellule P2x4-/- e P2x7-/- . Inoltre, l'ampiezza nelle cellule P2x4-/-è risultata significativamente ridotta, stabilendo ulteriormente il ruolo della segnalazione purinergica nei microdomini Ca2+ dipendenti dall'adesione.
Inoltre, questo metodo può essere utilizzato anche per visualizzare i microdomini iniziali di Ca2+ in cellule T CD4+ umane primarie (Figura 3). In linea con le cellule T murine primarie, i microdomini iniziali di Ca2+ sono evocati nel sito di contatto della sfera. Tuttavia, la risposta complessiva al Ca2+ sembra verificarsi su una scala temporale diversa rispetto alle cellule T CD4+ murine.
L'analisi dei segnali locali di Ca2+ in modo manuale non è fattibile in quanto piuttosto laboriosa e soggettiva per il singolo investigatore. Pertanto, abbiamo precedentemente sviluppato un algoritmo in MATLAB Simulink utilizzando i suoi toolbox di elaborazione e ottimizzazione delle immagini per la post-elaborazione13 per l'analisi di microdomini Ca2+ locali.
Recentemente, abbiamo sviluppato una nuova pipeline di post-elaborazione open source chiamata DARTS per l'analisi di microdomini Ca2+ nell'imaging di cellule vive ad alta risoluzione utilizzando la piattaforma software Python12. Qui, è possibile selezionare diversi algoritmi di deconvoluzione, a seconda delle preferenze dell'utente, eseguire una normalizzazione della forma della cella per compensare i cambiamenti morfologici della forma della cellula e definire parametri specifici del microscopio e della misurazione (ad esempio, scala, frequenza dei fotogrammi, tempo misurato) (Figura 4).
Dopo aver selezionato i parametri per l'analisi del microdominio Ca2+ , viene aperta una seconda finestra pop-up per ogni singola misurazione per definire il contatto del cordone (Figura 5). Per definire il contatto del cordone, l'utente può scorrere manualmente il file tiff utilizzando il cursore e selezionare il telaio del contatto del cordone individualmente. Il contatto del cordone viene selezionato facendo clic sul sito di contatto del cordone (Figura 5, perlina e contatto del cordone indicati da anello e freccia gialli) e selezionando la cella. Questo passaggio deve essere ripetuto per ogni cella di interesse. Infine, viene applicata la post-elaborazione automatizzata delle immagini e i dati dei risultati vengono riassunti e salvati in un foglio di calcolo.
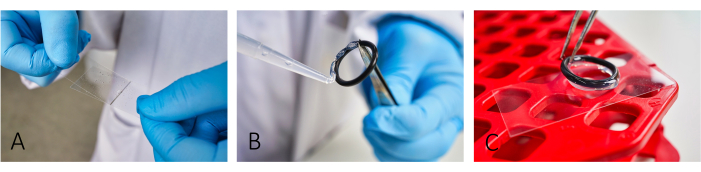
Figura 1: Flusso di lavoro della preparazione dei vetrini per l'imaging. (A) Aggiungere e stendere sia BSA che PLL sul vetrino utilizzando un secondo vetrino coprioggetti. (B,C) Per costruire una camera, incollare gli o-ring in gomma usando grasso siliconico sul vetrino. Assicurarsi che l'intero anello sia coperto da un sottile strato di grasso per avere un adeguato isolamento della camera. Clicca qui per visualizzare una versione più grande di questa figura.
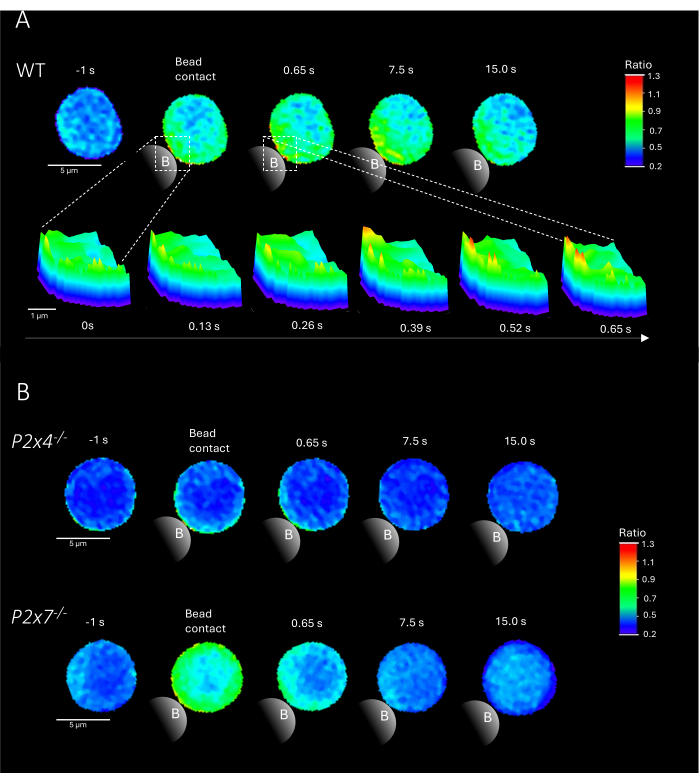
Figura 2: Cellule rappresentative di microdomini Ca2+ dipendenti dal recettore delle cellule T in una cellula T CD4+ murina primaria di tipo selvatico (WT) (A), P2x4-/- o P2x7-/-(B). Le cellule T CD4+ sono state isolate negativamente e caricate con Fluo-4 AM e Fura Red, come descritto sopra. Le cellule T sono state analizzate utilizzando la pipeline DARTS, ottenendo immagini cellulari comparabili con i risultati precedentemente pubblicati9. (A) Cellula T primaria WT 1 s prima della stimolazione con perle rivestite anti-CD3/anti-CD28 e fino a 15 s dopo la stimolazione (barra della scala 5 μm), nonché grafico della superficie 3D di uno zoom da 0 s a 0,65 s nella regione di contatto della perlina (barra della scala 1 μm). (B) Corsia superiore: cellula T primaria P2x4-/- rappresentativa 1 s prima e fino a 15 s dopo la stimolazione con microsfere. Corsia inferiore: cellula T primaria P2x7-/- rappresentativa 1 s prima e fino a 15 s dopo la stimolazione con perline. Clicca qui per visualizzare una versione più grande di questa figura.
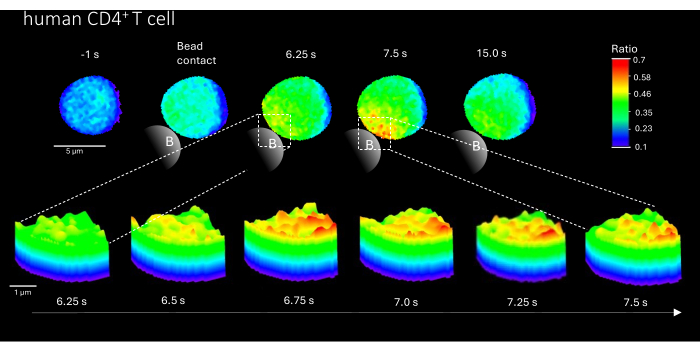
Figura 3: Microdomini Ca2+ in una cellula T umana primaria rappresentativa dopo stimolazione con TCR. Le cellule T CD4+ umane primarie sono state isolate da cellule mononucleate del sangue periferico (PBMC) mediante smistamento cellulare attivato dalla fluorescenza (FACS) da buffy coat e caricate con Fluo-4 AM e Fura Red, come descritto sopra. La figura mostra una cellula T umana primaria 1 s prima della stimolazione con perle rivestite anti-CD3 e fino a 15 s dopo la stimolazione (barra della scala 5 μm), nonché un grafico della superficie 3D di uno zoom da 6,25 s a 7,5 s nella regione di contatto della perlina (barra della scala 1 μm). Clicca qui per visualizzare una versione più grande di questa figura.
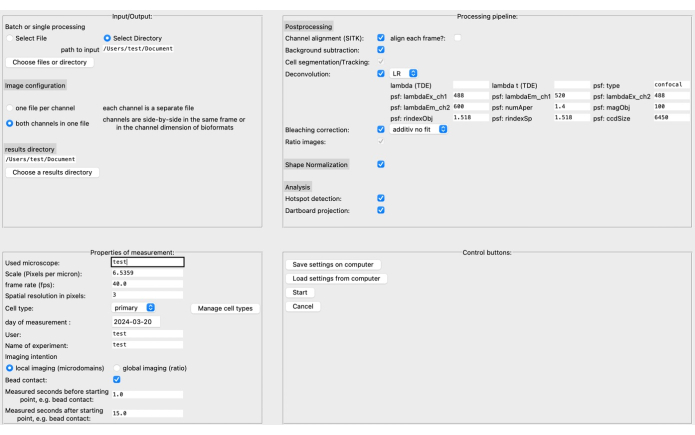
Figura 4: L'interfaccia utente grafica (GUI) DARTS. La GUI è suddivisa in quattro aree. Nell'area Input/Output, è necessario fornire informazioni sui dati grezzi, inclusa la directory di origine e la configurazione dell'immagine (due canali per file o canali separati), nonché la directory dei risultati. Nell'area Proprietà di misurazione, l'esperimento deve essere descritto con tutte le informazioni pertinenti, come la scala (micron per pixel), la frequenza dei fotogrammi e l'intervallo di misurazione relativo al punto di partenza determinato in seguito. Successivamente, è possibile assemblare una pipeline di elaborazione composta da fasi di post-elaborazione, normalizzazione della forma e analisi effettiva (rilevamento del microdominio e accumulo di dati del bersaglio). Infine, le impostazioni possono essere salvate o caricate dal computer. Una volta impostata l'analisi, fare clic su Avvia per procedere. Per saperne di più sulla configurazione, visita https://ipmi-icns-uke.github.io/DARTS/General/Usage.html. Clicca qui per visualizzare una versione più grande di questa figura.
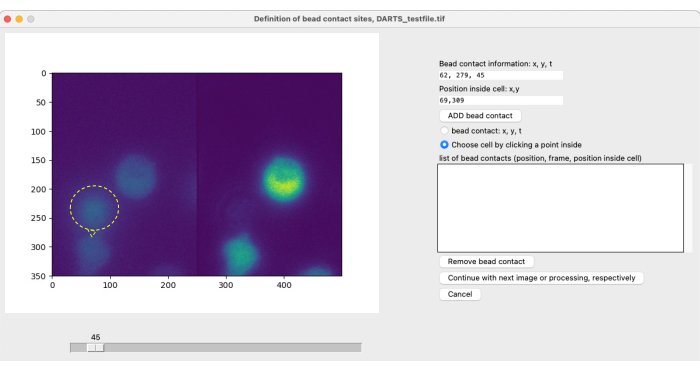
Figura 5: Definizione manuale dei contatti a cordona. Se le perle vengono aggiunte alle celle durante l'esperimento, il tempo iniziale di contatto della perlina con una cella di interesse e la posizione del contatto devono essere definiti manualmente. Questo viene fatto scorrendo i fotogrammi con il cursore e trovando una posizione (x,y) in un punto temporale t. Per compilare automaticamente il campo delle informazioni di contatto del cordone, l'utente fa clic sulla metà sinistra dell'immagine del microscopio nella posizione di contatto del cordone. Successivamente, per associare una cella al contatto del perlina, l'utente fa clic su una posizione all'interno della cella che ha un contatto del cordone. Le informazioni devono essere confermate selezionando AGGIUNGI perlina. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Abbiamo descritto un ampio protocollo per l'imaging ad alta risoluzione di microdomini locali di Ca2+ in cellule T primarie murine e umane attivate dalla stimolazione di TCR/CD3 attraverso perle rivestite di anticorpi. Inoltre, abbiamo implementato un algoritmo intuitivo e open-source basato su Python per identificare e analizzare i segnali locali di Ca2+. In particolare, il protocollo non si limita alla rilevazione di microdomini Ca2+ nel contesto della stimolazione TCR/CD3, ma è adattabile ad altri tipi di cellule (immunitarie) come le linee cellulari NK (KHYG-1)12 o i microdomini Ca2+ TCR-indipendenti10,11.
Un passaggio critico all'interno del protocollo è la dimensione e il numero delle perle stimolanti. Per imitare una sinapsi immunitaria, le perle dovrebbero essere di dimensioni simili alle cellule. Pertanto, per le cellule T primarie murine e umane, nonché per le linee cellulari (Jurkat e KHYG1), utilizziamo perline magnetiche con un diametro di 10 μm. Inoltre, ogni cellula dovrebbe essere stimolata da una singola perlina. Pertanto, il numero di perline aggiunte a ciascuna diapositiva dovrebbe essere sufficiente da un lato, ma se ci sono troppe perline nel campo visivo, lo sfondo aumenta e non è possibile rilevare un singolo punto di tempo di attivazione e lato di contatto.
Il protocollo utilizza i coloranti fluorescenti Ca2+ Fluo-4 AM e FuraRed AM in modo raziometrico, consentendo quindi la calibrazione dei dati13. Inoltre, il protocollo potrebbe essere adattato ad altre coppie di indicatori di Ca2+ , ma è necessario prestare attenzione nel processo di selezione in termini di cinetica di legame del Ca2+ , distribuzione subcellulare e fotobleaching1. Inoltre, le condizioni di carico devono essere sviluppate e ottimizzate per ogni singolo tipo di cellula, ma le concentrazioni qui indicate sono un buon punto di partenza. Per visualizzare i microdomini Ca2+ , il Kd dei coloranti Ca2+ dovrebbe essere compreso tra 300 e 1200 nM e il tempo di acquisizione per fotogramma dovrebbe essere di ≤60 ms. Se l'intensità della fluorescenza è troppo bassa, è necessario controllare il set di filtri, ma è anche possibile caricare una quantità doppia di colorante Ca2+ nelle cellule T. Tuttavia, il colorante Ca2+ potrebbe essere posizionato erroneamente in altri organelli o sequestrare le vescicole, ma potrebbe anche fungere da tampone per il Ca2+ e influenzare le risposte del Ca2+ .
Una limitazione dell'algoritmo di analisi è che si assume una forma sferica della cella; Pertanto, i tipi di cellule con morfologie diverse potrebbero richiedere l'adattamento del toolbox di analisi. L'algoritmo è stato utilizzato per analizzare microdomini locali di Ca2+ in cellule T murine primarie, così come in cellule T Jurkat e una linea cellulare NK (KHYG-1)12 e ha avuto successo nell'analisi di microdomini di Ca2+ per una linea cellulare neuronale murina (N2a, dati non pubblicati). In linea di principio, il protocollo e la cassetta degli attrezzi per l'analisi potrebbero essere utilizzati per analizzare tipi di cellule non sferiche come le cellule HEK293 o HeLa, ma per questi tipi di cellule, la proiezione del bersaglio non può essere adattata perché si basa su una struttura rotonda e sulla normalizzazione della forma delle cellule. Inoltre, il protocollo per rilevare i microdomini Ca2+ iniziali localizzati dopo la stimolazione delle perline può essere adattato per analizzare i segnali locali di Ca2+ derivati da altri stimoli, come i composti attivanti o inibitori solubili, nonché i microdomini Ca2+ dipendenti dall'adesione e indipendenti da TCR/CD310,11. Da notare che è più facile definire un singolo contatto della perlina in termini di tempo e posizione piuttosto che determinare il punto di inizio dell'attivazione dopo i composti solubili.
Una limitazione generale per la rilevazione della formazione di microdomini di Ca2+ risiede nell'elevata risoluzione spazio-temporale richiesta e nell'elevato rapporto segnale/rumore (SNR) necessario. Attualmente, la risoluzione derivata dalla nostra configurazione raggiunge una risoluzione spaziale calcolata di ~0,368 μm e una risoluzione temporale di ~40 fotogrammi al secondo (fps)1. I recenti progressi nello sviluppo di telecamere e rivelatori, così come il miglioramento dei coloranti fluorescenti, potrebbero portare alla possibilità di raggiungere registrazioni ottiche a canale singolo come sono state descritte per i costrutti ORAI-GECI (indicatori Ca2+ geneticamente espressi)24 per l'imaging di cellule vive utilizzando indicatori Ca2+ con una risoluzione temporale e spaziale più elevata in futuro.
Nel loro insieme, il protocollo e lo strumento di analisi per l'imaging del microdominio Ca2+ ad alta risoluzione qui descritti possono essere utilizzati non solo per analizzare i segnali locali iniziali del Ca2+ nelle cellule T, ma possono anche essere adattati ad altri tipi di cellule per decifrare il significato della segnalazione locale del Ca2+ in queste.
Divulgazioni
Gli autori dichiarano che la ricerca è stata condotta in assenza di qualsiasi rapporto commerciale o finanziario che possa essere considerato un potenziale conflitto di interessi.
Riconoscimenti
Questo lavoro è stato sostenuto dalla Deutsche Forschungsgemeinschaft (DFG) (progetto numero 335447717; SFB1328, A02 a B-PD e RW; A14 fino a ET; numero di progetto 516286863 a B-PD). Gli autori ringraziano i donatori di sangue e il Dipartimento di Medicina Trasfusionale dell'UKE per la loro collaborazione.
Materiali
| Name | Company | Catalog Number | Comments |
| α-CD3 | BD Pharmingen | 553058 | |
| α-CD28 | BD Pharmingen | 553295 | |
| Anaconda | Anaconda | https://docs.anaconda.com/free/anaconda/install/index.html | |
| Bovine serum albumin | Sigma-Aldrich | A2058 | |
| Cell strainer | Biofil | CSS-013-070 | 70 µm diameter |
| Countess Automated Cell Counter | Invitrogen | A49865 | |
| Cover slips | Paul Marienfeld GmbH | 101202 | |
| DARTS | GitHub | https://github.com/IPMI-ICNS-UKE/DARTS | |
| Dulbecco’s Phosphate buffered saline | Gibco, Thermo Fisher | 14190144 | |
| EasySep CD4+ T cell Isolation Kit | Stemcell | 19852 | |
| EasySep Magnetic Multistand | Stemcell | 18010 | |
| Fluo 4-AM | Invitrogen | F14201 | |
| Fura Red-AM | Invitrogen | F3020 | |
| Gibco RPMI 1640 medium | Gibco, Thermo Fisher | 11875093 | |
| Git | GitHub | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git | |
| immersion oil | Leica | 11513859 | |
| Newborn calf serum | Sigma-Aldrich | N4637 | |
| Oracle | Oracle | https://www.oracle.com/de/java/technologies/downloads/ | |
| Penicillin/Streptamycin | Gibco, Thermo Fisher | 15240062 | |
| Poly-L-lysine | Sigma-Aldrich | P4707 | |
| Protein G magnetic beads | Merck | LSKMAGG02 | 10 µM diameter |
| Python | Python | https://www.python.org/downloads/ | |
| Silicon grease (basylone) | Bayer | 291-1220 | |
| Time-Dependent Entropy Deconvolution | GitHub | https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters | |
| Tween | q-biogene | TWEEN201 |
Riferimenti
- Wolf, I. M. A., et al. Frontrunners of T cell activation: Initial, localized Ca2+ signals mediated by NAADP and the type 1 ryanodine receptor. Sci Signal. 8 (398), 102(2015).
- Taguchi, T., Mukai, K. Innate immunity signalling and membrane trafficking. Curr Opin Cell Biol. 59, 1-7 (2019).
- Trebak, M., Kinet, J. -P. Calcium signalling in T cells. Nat Rev Immunol. 19 (3), 154-169 (2019).
- Diercks, B. -P., et al. ORAI1, STIM1/2, and RYR1 shape subsecond Ca2+ microdomains upon T cell activation. Sci Signal. 11, 0358(2018).
- Cheng, H., Lederer, W. J., Cannell, M. B. Calcium sparks: Elementary events underlying excitation-contraction coupling in heart muscle. Science. 262 (5134), 740-744 (1993).
- Feske, S. Calcium signalling in lymphocyte activation and disease. Nat Rev Immunol. 7 (9), 690-702 (2007).
- Berridge, M. J., Lipp, P., Bootman, M. D. The versatility and universality of calcium signalling. Nat Rev Mol Cell Biol. 1 (1), 11-21 (2000).
- Kummerow, C., Junker, C., Kruse, K., Rieger, H., Quintana, A., Hoth, M. The immunological synapse controls local and global calcium signals in T lymphocytes. Immunol Rev. 231 (1), 132-147 (2009).
- Brock, V. J., et al. P2X4 and P2X7 are essential players in basal T cell activity and Ca2+ signaling milliseconds after T cell activation. Sci Adv. 8 (5), 9770(2022).
- Weiß, M., et al. Adhesion to laminin-1 and collagen IV induces the formation of Ca2+ microdomains that sensitize mouse T cells for activation. Sci Signal. 16 (790), 9405(2023).
- Diercks, B. -P. The importance of Ca2+ microdomains for the adaptive immune response. Biochim Biophys Acta Mol Cell Res. 1871 (5), 119710(2024).
- Woelk, L. -M., et al. DARTS: an open-source Python pipeline for Ca2+ microdomain analysis in live cell imaging data. Front Immunol. 14, 1299435(2024).
- Diercks, B. -P., Werner, R., Schetelig, D., Wolf, I. M. A., Guse, A. H. High-resolution calcium imaging method for local calcium signaling. Methods Mol Biol. 1929, 27-39 (2019).
- Python. Python. , Available from: https://www.python.org/downloads/ (2024).
- Anaconda. Anaconda. , Available from: https://docs.anaconda.com/free/anaconda/install/index.html (2024).
- git. git. , Available from: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git (2024).
- DARTS. DARTS. , Available from: https://github.com/IPMI-ICNS-UKE/DARTS (2024).
- DARTS. DARTS GitHub Repository. , Available from: https://ipmi-icns-uke.github.io/DARTS/ (2024).
- Oracle. , Available from: https://www.oracle.com/de/java/technologies/downloads/ (2024).
- Time-Dependent Entropy Deconvolution. , Available from: https://ipmi-icns-uke.github.io/TDEntropyDeconvolution/General/2-usage.html#input-parameters (2024).
- Gu, F., et al. Dual NADPH oxidases DUOX1 and DUOX2 synthesize NAADP and are necessary for Ca2+ signaling during T cell activation. Sci Signal. 14 (709), 3800(2021).
- Roggenkamp, H. G., et al. HN1L/JPT2: A signaling protein that connects NAADP generation to Ca2+ microdomain formation. Sci Signal. 14 (675), 5647(2021).
- Heßling, L. D., Troost-Kind, B., Weiß, M. NAADP-binding proteins - Linking NAADP signaling to cancer and immunity. Biochim Biophy. Acta Mol Cell Res. 1870 (7), 119531(2023).
- Dynes, J. L., Amcheslavsky, A., Cahalan, M. D. Genetically targeted single-channel optical recording reveals multiple Orai1 gating states and oscillations in calcium influx. Proc Natl Acad Sci U S A. 113 (2), 440-445 (2016).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon