Method Article
Aislamiento de microorganismos del suelo mediante la tecnología iChip
En este artículo
Resumen
La técnica iChip hace uso de un dispositivo de aislamiento in situ barato y sencillo que aumenta las tasas de descubrimiento de nuevos microorganismos en el suelo. Los nuevos microorganismos se pueden utilizar para estudios posteriores relacionados con el microbioma del suelo o el descubrimiento de productos naturales, entre otras aplicaciones.
Resumen
La técnica de aislamiento iChip utiliza un dispositivo de aislamiento in situ que aumenta la cultivabilidad de microorganismos previamente no cultivables. Los microorganismos son una fuente importante de nuevas sustancias químicas y moléculas potencialmente bioactivas. Sin embargo, solo el 1% de los microorganismos ambientales se pueden cultivar utilizando métodos convencionales de laboratorio. Con el aumento de la resistencia a los antimicrobianos, encontrar nuevos fármacos para combatir infecciones y enfermedades es de suma importancia, y un método fundamental para encontrar nuevas moléculas es el descubrimiento de nuevos microorganismos. Al incubar colonias de microorganismos del suelo en los pocillos de una placa de 96 pocillos, sellada con una membrana semipermeable e incubada en la parte superior del suelo, los microbios están en contacto con el agua y los factores de crecimiento del suelo, lo que permite el aislamiento de nuevos microbios en un entorno de laboratorio. Después de un período de domesticación en un iChip, los microorganismos pueden subcultivarse en medios convencionales y usarse para estudios posteriores. Este dispositivo es valioso para el descubrimiento de moléculas bioactivas y la investigación del microbioma del suelo, y se ha utilizado anteriormente en ambas aplicaciones.
Introducción
Las bacterias ambientales son una rica fuente de productos naturales (NP)1. Estos metabolitos no son vitales para la supervivencia de los microorganismos, sino que se producen para facilitar su colonización al superar a otros microorganismos en su entorno2. La evolución ha afinado las estructuras químicas y las actividades de las NP, convirtiéndolas en agentes antimicrobianos eficaces. Un ejemplo de ello es la daptomicina, un antibiótico aprobado por la FDA en 20033. En los últimos años, la resistencia microbiana ha aumentado en incidencia y gravedad, y los nuevos fármacos como la daptomicina se erigen como tratamientos de última línea contra las infecciones que han desarrollado resistencia a los antibióticos másantiguos. El desarrollo de nuevos antibióticos y otros medicamentos es esencial para mantener las infecciones y enfermedades comunes tratables.
Aunque las NP a menudo son buenas pistas para los medicamentos, la industria farmacéutica recurrió a métodos sintéticos para el descubrimiento de medicamentos después de la edad de oro de los antibióticos en las décadas de 1950 y 1960. En la década de 1970, los mismos microorganismos y metabolitos antimicrobianos continuaron siendo reaislados repetidamente, yse descubrieron cada vez menos nuevos candidatos a fármacos 1,6. El microbioma del suelo contiene una gran diversidad microbiana, pero solo se puede aislar un pequeño número de microorganismos en condiciones convencionales de laboratorio. La gran mayoría de los microorganismos del suelo o microorganismos detectados por genómica no se observan cuando se cultivan utilizando métodos de cultivo convencionales, lo que lleva a que el tema se acuñe como "la gran anomalía del recuento de placas"7. Estos microbios incultivables han sido llamados materia oscura microbiana, ya que se sabe que existen, pero no pueden ser estudiados in vitro hasta que puedan ser aislados como cultivos puros. Es probable que estos microorganismos incultivables puedan producir una gran cantidad de candidatos a fármacos, lo cual es de gran importancia en la era de la resistencia microbiana.
La técnica iChip es un método que se puede utilizar para aumentar la recuperación de nuevos microorganismos del medio ambiente 8,9. Esta tecnología ayuda a simular el entorno natural de las bacterias durante la incubación, aumentando así la cultivabilidad de microorganismos que de otro modo no podrían sobrevivir a las condiciones convencionales de laboratorio10. Los iChips modificados ya se han desarrollado y utilizado para aislar microorganismos de muchas fuentes diferentes, como el suelo, los sedimentos, los entornos marinos y los intestinos de los animales 11,12,13,14,15,16. Quizás el caso más impactante del uso de esta tecnología ha sido el de NovoBiotic Pharmaceuticals, donde se descubrió una nueva bacteria, Eleftheria terrae 17. Se descubrió que este microorganismo produce Teixobactina, un antibiótico de una nueva clase, que inhibe varias especies bacterianas resistentes pertinentes para la salud humana sin que se detecte el desarrollo de resistencia 5,18. Este fue un descubrimiento impactante, ya que la teixobactina es la primera nueva clase de antibióticos descubierta en décadas y es una señal de que esta técnica es una ruta prometedora para el descubrimiento de nuevos medicamentos y la superación de la gran anomalía del recuento de placas19. En este trabajo se presenta un iChip modificado basado en una publicación previa de Berdy et al. que fue optimizado para facilitar su uso y prevenir la contaminación9.
Protocolo
1. Preparación y esterilización de medios
- Prepare y esterilice los medios de sales mínimas de succinato (SMS), que consisten en 0,1 g de almidón de patata, 1 g de ácidos casamino, 0,125 g de caseína digestiva y 15 g de agar bacteriológico o técnico en 1 L de agua.
- Esterilizar en autoclave el medio y 100 mL de agua de grado molecular utilizando un ciclo de líquido de 20 min, 121 °C.
NOTA: Asegúrese de que todos los demás elementos que entrarán en contacto con la mezcla de agar y celda se compren estériles, incluidas las puntas de pipeta y los tubos de centrífuga, de lo contrario, también los esterilizará en autoclave.
2. Construcción iChip modificada
- Retire el fondo de los pocillos en cuatro placas de 96 pocillos con una herramienta perforadora de agar de 5 mm.
- Corte membranas de policarbonato de 0,05 μm en rectángulos de 7,6 cm x 11 cm, o iguales a las dimensiones de la parte inferior de la placa de 96 pocillos.
- Con un sellador de silicona, adhiera membranas de policarbonato de 0,05 μm a la parte inferior de las placas de 96 pocillos, asegurándose de que el adhesivo selle los pocillos, pero no cubra completamente las aberturas de los pocillos. Deje secar durante al menos 24 h o siguiendo las instrucciones del adhesivo.
NOTA: El sellador debe ser impermeable, no tóxico y 100% de silicona. La mayoría de los selladores de acuarios funcionarán bien.
3. Preparación de suspensiones celulares
- Etiquete cuatro tubos de centrífuga de 15 mL A-D y cuatro tubos de centrífuga de 50 mL E-H y agregue 4,5 mL de agua estéril a cada uno.
- Mida 1 g de tierra en un tubo de centrífuga de 50 ml, agregue 10 ml de agua estéril y vórtice durante 10 minutos.
- Deje que la suspensión del suelo se asiente durante 10 minutos.
- Pipetear 0,5 mL del sobrenadante que contiene células del suelo en el tubo A y mezclar bien.
- Agregue 0,5 mL de la suspensión celular en el tubo A al tubo B y mezcle bien. Transfiera 0,5 mL de solución B al tubo C y mezcle bien. Repita esto para todos los tubos de centrífuga restantes, completando una serie de diluciones de 10 veces a través de los ocho tubos de centrífuga.
- Retire 0,5 mL del tubo H para que tenga el mismo volumen en todos los tubos.
NOTA: Estas concentraciones están optimizadas para los suelos locales. Cuando intente este procedimiento por primera vez, lleve a cabo una gama más amplia de diluciones para encontrar la concentración de muestra adecuada.
4. Inoculación de iChip modificado
- Sumerja completamente los iChips en etanol al 95% durante al menos 15 minutos.
- Retire las placas del etanol y colóquelas sobre una toalla de papel estéril. Mientras deja que el etanol se evapore, encienda el esterilizador UV en la campana de flujo laminar durante 15 minutos para esterilizarlos aún más.
- Pipetear 360 μL de medios SMS estériles en la primera columna de la placa para que actúen como pocillos de control.
- Añadir 45 mL de SMS enfriado a 50 °C a la suspensión celular en el tubo E y mezclar bien para combinar la suspensión celular y el agar.
- Pipetear 360 μL de la mezcla de agar-célula del paso 4.4 en todos los demás pocillos de la placa de 96 pocillos.
NOTA: Se recomienda una pipeta multicanal ya que el agar se solidifica rápidamente. No se aconseja recalentar la mezcla de agar y célula una vez mezclada debido al riesgo de matar los microorganismos. - Una vez que el medio esté configurado, selle la parte superior de las placas con una cubierta de placa PCR.
- Repita los pasos 3.3 a 3.6 con tubos F-H para llenar un total de cuatro iChips con diferencias de concentración diez veces mayores.
NOTA: Los medios se pueden calentar en el microondas durante 30 s con una tapa aflojada cuando sea necesario o mantenerse en un baño de agua caliente a 60 °C entre las placas para garantizar que permanezcan fundidos para cada una.
5. Incubación
- Coloque las placas en una caja que contenga aproximadamente una pulgada de la tierra utilizada en el paso 3.2, con la membrana hacia abajo y coloque la cubierta.
- Incubar las placas en el recipiente tapado, en un lugar oscuro a 25 °C.
NOTA: Después de una semana, examine el contenido de los iChips modificados para asegurarse de que no se haya producido contaminación durante la configuración. La contaminación está indicada por el crecimiento excesivo de uno o pocos microorganismos en todos los pocillos de una placa. - Después de 6 semanas, examine los iChips. Utilice las placas que contienen crecimiento en menos del 25% de los pocillos para el aislamiento de colonias.
6. Aislamiento de colonias
- Enjuague los iChips modificados 3 veces con agua estéril para eliminar todas las partículas de tierra.
- Limpie la parte superior y los lados de la placa con etanol al 95%, evitando el lado con la membrana semipermeable.
- Con una cuchilla estéril, corte a través de la cubierta de la placa alrededor de un pocillo que contenga una colonia.
- Con una herramienta de rayado estéril, perfore la colonia y raye en agar SMS en placas de 100 x 15 mm utilizando el método de cuatro cuadrantes.
- Repita el procedimiento para todas las demás colonias de los iChips que estén creciendo una colonia por pocillo.
NOTA: Para colonias que son minúsculas, la placa se puede volver a colocar en el suelo e incubar hasta que sea lo suficientemente grande como para subcultivar, siempre y cuando no se haya retirado la cubierta de la placa alrededor del pozo. - Incubar las placas SMS a 25 °C y controlar el crecimiento.
- Examine las colonias para asegurarse de que se hayan aislado los cultivos axénicos.
7. Identificación de microorganismos
- Secuenciar los genomas de los aislados recuperados20.
- Evalúe los genomas utilizando JSpeciesWS u otra plataforma de identificación basada en genomas para determinar el grado de similitud de cada aislado con los microorganismos conocidos en GenomesDB.
- Categorizar los aislados por debajo de los umbrales aceptados por la comunidad para la identificación de especies como supuestamente nuevos.
Resultados
En la Figura 1 se muestra una descripción general visual del protocolo. Un experimento exitoso de iChip modificado da como resultado al menos una placa que contiene crecimiento en menos del 25% de los pocillos, sin que se produzca ningún crecimiento en los pozos de control. Este número de pozos con crecimiento generalmente asegura que las colonias individuales estén aisladas en los pozos. Además, el número de colonias, o el número de pozos que contienen colonias, debe disminuir diez veces con cada placa subsiguiente preparada a partir de la serie de diluciones. Los resultados representativos se muestran en la Figura 2. donde las placas 7 y 8 son iChips exitosos ya que contienen crecimiento en menos del 25% de los pocillos, y el porcentaje de pocillos que contienen colonias disminuye con cada placa, como se muestra en la Tabla 1. Si todas las placas de un ensayo contienen más de una colonia por pocillo o no hay colonias en ningún pocillo, eso constituye un resultado negativo.
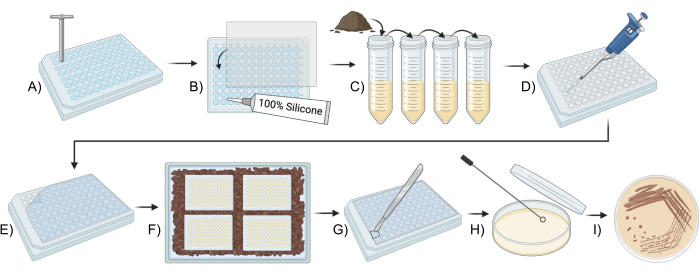
Figura 1: Resumen esquemático del uso de un iChip modificado para cultivar microbios del suelo. (A,B) Construcción de la placa (Paso 2). (C) Preparación del inóculo (Paso 3). (D,E) Configuración del iChip (Paso 4). (f) Incubación del iChip (Paso 5). (G-I) Transferencia de los aislados a medios convencionales (Paso 6). Haga clic aquí para ver una versión más grande de esta figura.
| Ichip | Concentración de suelo en suspensión de agar | Pozos con colonias | Unidades formadoras de colonias |
| E | 5,0 x 10-6 g/ml | Todo | Demasiados para contarlos |
| F | 5,0 x 10-7 g/ml | Todo | Demasiados para contarlos |
| G | 5,0 x 10-8 g/ml | 22 | 22 |
| H | 5,0 x 10-9 g/ml | 6 | 6 |
Tabla 1: Datos representativos de un experimento exitoso de iChip donde el número de pocillos que contienen colonias disminuye con cada dilución en serie.
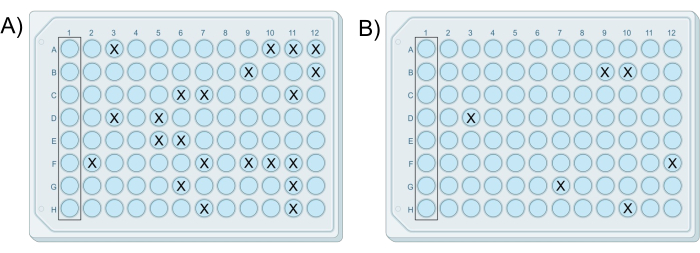
Figura 2: Resultados ejemplares de dos iChips modificados exitosos. Los iChips procedían del mismo experimento en el que (A) la placa preparada a partir de la mezcla de célula y agar G contiene 22 colonias, y (B) la placa preparada a partir de la mezcla de célula y agar H contiene seis colonias, lo que es coherente con la diferencia de dilución de 10 veces entre las mezclas. Haga clic aquí para ver una versión más grande de esta figura.
Se pueden visualizar las colonias a simple vista o al microscopio, como se muestra en la Figura 3A-G. Las colonias pueden estar creciendo en la superficie del agar, como se ve en la Figura 3B o incrustadas en el agar, como la colonia en la Figura 3F. Puede haber casos en los que haya dos colonias en el mismo pozo en un iChip modificado con éxito, como en la Figura 3G. En algunos casos, las colonias están lo suficientemente separadas como para que puedan ser perforadas con una aguja por separado y subcultivadas; sin embargo, es poco probable que los de la Figura 3G puedan cultivarse fácilmente como microorganismos axénicos. En la Figura 4 se muestran ejemplos de colonias transferidas desde el iChip a medios SMS convencionales.
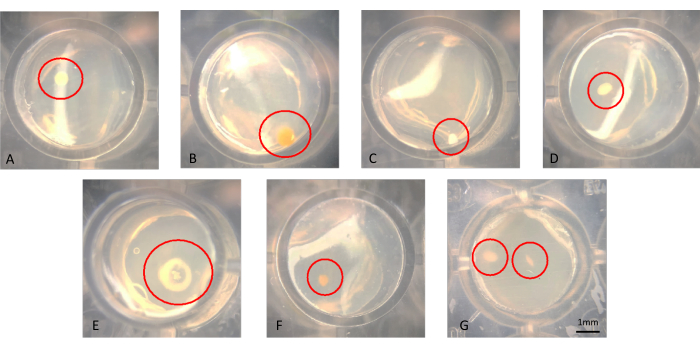
Figura 3: Vista microscópica representativa de las colonias que crecen en un iChip después de 6 semanas de incubación. (A-F) Se observan colonias individuales, a partir de las cuales se deben subcultivar, y en (G) se observan dos colonias en las proximidades, que son difíciles de obtener como cultivos axénicos. Haga clic aquí para ver una versión más grande de esta figura.
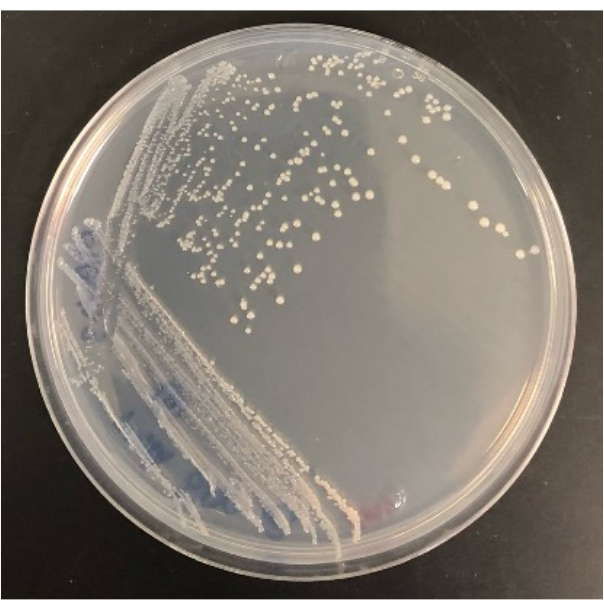
Figura 4: Resultado representativo de una bacteria aislada del iChip modificado una vez que crece en una placa de agar SMS. Haga clic aquí para ver una versión más grande de esta figura.
La capacidad de aislar colonias del iChip modificado en placas de agar convencionales también determina el éxito del experimento. No todas las colonias que crecen en el iChip modificado sobrevivirán a la transferencia a placas convencionales. En un iChip, las colonias están expuestas a factores de crecimiento y nutrientes del suelo, lo que aumenta su cultivabilidad. Para algunos microorganismos, un período de domesticación no es suficiente para hacerlos cultivables en condiciones convencionales. La tasa de recuperación de las colonias de un iChip a las placas variará de un experimento a otro, pero algunas deben crecer en placas, ya que este método también cultivará microorganismos fácilmente cultivables. Por ejemplo, los iChips modificados de la Figura 2 contenían 28 colonias en total, y el número de colonias que crecieron en placas de agar después del subcultivo fue de 16, mientras que 12 no sobrevivieron a la transferencia. Los aislados se pueden identificar y utilizar para estudios posteriores una vez cultivados en placas de agar de tamaño completo, como se muestra en la Figura 4.
Un experimento exitoso de iChip modificado debería resultar en el aislamiento de microorganismos que difieren de los microorganismos conocidos a nivel de especie. El grado de similitud de cada aislado con los microorganismos conocidos se encuentra comparando el ADN aislado con los genomas de los microorganismos conocidos. Las puntuaciones tetra Z de los microorganismos relacionados más cercanos a cada aislado describen el porcentaje de similitud. La Figura 5 muestra que de las doce bacterias aisladas, cuatro eran especies nuevas o superiores, dos eran probablemente cepas nuevas y seis eran microorganismos conocidos. La recuperación de varias especies o cepas nuevas es consistente con los experimentos de iChip publicados anteriormente, lo que indica que esta construcción modificada de iChip no impide el aumento en la recuperación de nuevas especies11.
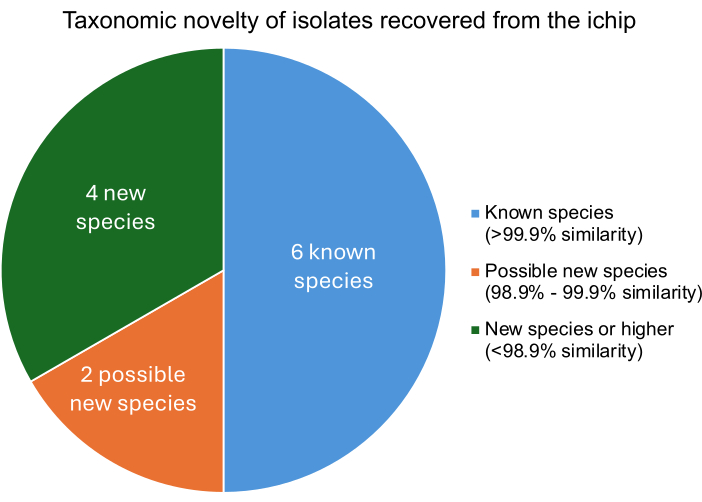
Figura 5: Nivel de clasificación taxonómica de los aislados recuperados de un experimento con iChip. El porcentaje de similitud se determinó con base en las puntuaciones tetra Z, que describen la similitud de los aislados con bacterias conocidas, donde la similitud del >99,9% indica una coincidencia de especies, la similitud del 98,9%-99,9% indica una posible nueva especie y la similitud del <98,9% indica una nueva especie o superior. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Numerosos métodos, como la minería del genoma y el examen de las vías biosintéticas silenciosas, han facilitado el descubrimiento de nuevas sustancias químicas bioactivas en los últimos años21,22. Sin embargo, las NP descubiertas utilizando tales métodos a menudo exhiben una alta similitud estructural con los compuestos conocidos. Obtener acceso a microorganismos previamente no cultivados desbloqueará una mayor diversidad química y NP con nuevos modos de acción que pueden ayudar mejor a combatir la resistencia microbiana. Se ha demostrado que la técnica de aislamiento iChip aumenta la cultivabilidad de nuevos microorganismos, que se pueden utilizar para construir bibliotecas de microorganismos para el descubrimiento de nuevas NP.
La primera conceptualización de un iChip se publicó en 2002, que consistía en anillos metálicos individuales que contenían una mezcla de células de agar sellada con membranas semipermeables e incubada in situ23. Más tarde, en 2010, se repitió para contener un pequeño chip con muchos pocillos11, seguido por el desarrollo de un iChip de bajo costo construido a partir de materiales de laboratorio comunes en una influyente publicación de Nature Protocols en 20179. En esta publicación se han realizado varias modificaciones al protocolo de Nature para mejorar su practicidad y facilidad de uso. El protocolo Nature consiste en pegar una membrana semipermeable a ambos lados de un chip en comparación con este método, que utiliza una cubierta de placa adhesiva en un lado. Pueden surgir problemas al usar pegamento de silicona para unir la membrana después de cargar el iChip. Hasta donde sabemos, todos los adhesivos de silicona no tóxicos emiten ácido acético a medida que se endurecen, lo que puede afectar la viabilidad celular24. El protocolo descrito en este video también reduce significativamente la cantidad de manipulación requerida después de la inoculación mediante el uso de la cubierta de la placa PCR para sellar la placa llena, lo que garantiza aún más la esterilidad y reduce el tiempo de configuración. Sobre la base de la identificación de múltiples aislados nuevos de este experimento, el uso de una sola membrana semipermeable inhibe el aumento en las tasas de descubrimiento de nuevos organismos que se ha informado para los iChips construidos con dos membranas semipermeables. Sin embargo, se necesitarían más experimentos con un tamaño de muestra mayor para cuantificar el impacto.
Otra modificación del método es solo subcultivos de los iChips modificados que contienen crecimiento en menos del 25% de los pozos. Si las colonias crecen en la mayoría de los pocillos de una placa, es probable que haya algunos pocillos que contengan múltiples microorganismos, aunque no sean fácilmente visibles. Durante el desarrollo del método, se descubrió que era en gran medida imposible obtener cultivos axénicos cuando se subcultivaba a partir de pozos que contenían más de una colonia. El aislamiento de cultivos no axénicos presenta problemas significativos aguas abajo en términos de ensayos de bioactividad e identificación. Por lo tanto, para simplificar, solo se recomienda subcultivar iChips modificados con colonias que crezcan en una parte de los pozos.
El problema más importante que puede surgir con los métodos iChip es la contaminación de pocillos o iChips completos con otros microorganismos. La contaminación se indica por las colonias que crecen en los pozos de control, o por el mismo microorganismo que crece en varios pozos o en toda un área del iChip modificado. La fuente de contaminación puede deberse a una esterilización inadecuada de los materiales durante la instalación o a una técnica aséptica inadecuada. En tales casos, asegúrese de que todos los materiales utilizados estén esterilizados en autoclave o etanol como se indica en el método, y asegúrese de que no se produzca contacto entre ningún elemento no estéril y el iChip modificado que no sea la mezcla de agar y celda. Si se observa un crecimiento excesivo de un solo microorganismo en la parte inferior o superior de varios pocillos iChip modificados, lo más probable es que sea el resultado de un sellado incompleto entre los pocillos y la membrana. En este caso, asegúrese de que el adhesivo utilizado sea 100% silicona, que no se degrada en etanol, y asegúrese de que la membrana semipermeable y la cubierta de la placa PCR estén completamente selladas alrededor de cada pozo durante la construcción.
La serie de diluciones utilizada en el protocolo actual debe proporcionar una dilución adecuada para la mayoría de los tipos de suelo con un período de almacenamiento de menos de una semana, ya que incluye un rango de diluciones mil veces. Sin embargo, puede haber una variación significativa en los recuentos de microorganismos entre los tipos de suelo. Si los pocillos de control no contienen crecimiento, pero hay múltiples colonias creciendo en cada pocillo de los cuatro iChips modificados, las concentraciones de celdas utilizadas no fueron lo suficientemente bajas. La serie de dilución debe modificarse para alcanzar concentraciones celulares más bajas en las mezclas de células y medios utilizadas para configurar los iChips modificados. Del mismo modo, si no se observa crecimiento en ninguna de estas placas, se deben aumentar las concentraciones utilizadas en las mezclas de células y medios. Alternativamente, es posible que la temperatura de los medios utilizados fuera demasiado alta para la supervivencia de los microorganismos. En tal caso, se debe permitir que el medio se enfríe tanto como sea posible sin solidificarse antes de agregarlo a la suspensión celular diluida.
La tecnología es un avance importante hacia la superación de la gran anomalía del recuento de placas. Sin embargo, todavía está limitado por la falta de idoneidad de las técnicas de cultivo convencionales, como lo indica el número de microorganismos que no sobreviven a la transferencia del iChip modificado a las placas de agar convencionales. Publicaciones anteriores han informado de que múltiples rondas de subcultivo e incubación en iChips aumentan aún más la cultivabilidad de los microorganismos. El tiempo de domesticación prolongado y la exposición a los factores de crecimiento del suelo mientras se está en agar en el iChip aumenta las posibilidades de que una colonia crezca solo con agar. Sin embargo, no se ha reportado que este enfoque produzca una mayor probabilidad de nuevos microorganismos que una sola incubación de iChip 25,26.
Se están explorando y modificando muchas otras tácticas para aumentar la cultivabilidad de nuevos microorganismos. Por ejemplo, se ha propuesto fabricar un dispositivo iChip completo a partir de un material semipermeable para facilitar el cocultivo de microorganismos en pozos vecinos27. Dicho esto, una ventaja de la construcción descrita en esta publicación es su bajo costo, ya que el costo de construir una placa equivale a aproximadamente $12 ($4 por placa de 96 pocillos, $8 por membrana, $2 por cubierta de PCR). Además, su construcción simple lo convierte en una herramienta sencilla cuando se usa como se describe y brinda muchas posibilidades de personalización. Aunque este protocolo utiliza un medio selectivo para las bacterias, la configuración experimental puede ajustarse teóricamente para dirigirse a una población microbiana deseada modificando el medio utilizado, como el uso de medios supresores de bacterias para dirigirse a los hongos, o agar bajo en nutrientes para los microorganismos esporulantes.
Divulgaciones
Los autores han declarado no tener conflictos de intereses.
Agradecimientos
Agradecemos la financiación de J-base (J-001757, J-001842) proporcionada a través de Agriculture and Agri-Food Canada, que hizo posible este proyecto. Agradecemos a Brett van Heyningen por filmar el contenido de video para este protocolo. También nos gustaría agradecer a Ron Matters por recolectar muestras de suelo utilizadas en los experimentos descritos en esta publicación.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
Referencias
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados