Method Article
使用 iChip 技术分离土壤微生物
摘要
iChip 技术利用一种廉价且简单的 原 位分离装置,可以提高从土壤中发现新型微生物的速度。新型微生物可用于与土壤微生物组或天然产物发现有关的进一步研究,以及其他应用。
摘要
iChip 分离技术使用 原位 分离装置,可增加以前无法培养的微生物的可培养性。微生物是新型化学和潜在生物活性分子的重要来源。然而,只有 1% 的环境微生物可以使用常规实验室方法进行培养。随着抗菌素耐药性的上升,寻找对抗感染和疾病的新药至关重要,而寻找新分子的关键方法是发现新的微生物。通过在 96 孔板的孔中培养土壤微生物菌落,用半透膜密封并在土壤顶部孵育,微生物与土壤中的水和生长因子接触,从而在实验室环境中分离新型微生物。在 iChip 中驯化一段时间后,微生物有可能在常规培养基上传代培养并用于进一步研究。该设备对于生物活性分子发现和土壤微生物组研究很有价值,并且之前已用于这两种应用。
引言
环境细菌是天然产物 (NP) 的丰富来源1。这些代谢物对微生物的生存并不重要,而是通过超越周围的其他微生物来促进它们的定植2。进化微调了 NP 的化学结构和活性,使其成为有效的抗菌剂。这样一个例子是达托霉素,一种于 2003 年获得 FDA 批准的抗生素3。近年来,微生物耐药性的发生率和严重程度都有所增加,像达托霉素这样的新药是治疗对旧抗生素产生耐药性的感染的最后一线药物4。开发新的抗生素和其他药物对于保持常见感染和疾病的可治疗至关重要。
尽管 NP 通常是良好的药物先导物,但在 1950 年代至 1960 年代抗生素的黄金时代之后,制药行业转向合成方法进行药物发现5。到 1970 年代,相同的微生物和抗菌代谢物继续被反复重新分离,发现的新型候选药物越来越少 1,6。土壤微生物组包含大量的微生物多样性,但在常规实验室条件下只能分离出少量微生物。使用常规培养方法培养时,基因组学检测到的微生物的绝大多数土壤微生物都无法观察到,因此该问题被称为“大平板计数异常”7。这些不可培养的微生物被称为微生物暗物质,因为它们已知存在,但在它们可以被分离为纯培养物之前,不能在体外进行研究。这些不可培养的微生物可能会产生大量的候选药物,这在微生物耐药性时代非常重要。
iChip 技术是一种可用于提高新型微生物从环境中恢复的方法 8,9。该技术有助于模拟细菌在孵化过程中的自然环境,从而提高微生物的培养能力,否则这些微生物将无法在常规实验室条件下存活10。改良的 iChips 已经被开发出来,用于从许多不同的来源分离微生物,例如土壤、沉积物、海洋环境和动物肠道 11,12,13,14,15,16。也许使用这项技术最具影响力的案例是 NovoBiotic Pharmaceuticals,在那里发现了一种新型细菌 Eleftheria terrae 17。发现这种微生物产生 Teixobactin,这是一种新型抗生素,可抑制与人类健康相关的几种耐药细菌物种,而不会检测到耐药性的发展 5,18。这是一个有影响力的发现,因为 Teixobactin 是几十年来发现的第一类新型抗生素,这表明该技术是发现新药和克服巨大板数异常19 的有前途的途径。在此,提出了一种基于 Berdy 等人先前出版物的改进型 iChip,该 iChip 针对易用性和污染预防进行了优化9。
研究方案
1. 培养基制备和灭菌
- 制备琥珀酸盐最低盐培养基 (SMS) 并对其进行消毒,该培养基由 0.1 g 马铃薯淀粉、1 g 酪蛋白氨基酸、0.125 g 酪蛋白消化物和 15 g 细菌学或技术琼脂溶于 1 L 水中组成。
- 使用 20 分钟、121 °C 的液体循环对培养基和 100 mL 分子级水进行高压灭菌。
注意:确保与琼脂细胞混合物接触的所有其他物品都是无菌的,包括移液器吸头和离心管,否则也要将它们高压灭菌。
2. 改进的 iChip 结构
- 使用 5 mm 琼脂打孔工具去除四个 96 孔板中的孔底部。
- 将 0.05 μm 聚碳酸酯膜切割成 7.6 cm x 11 cm 的矩形,或等于 96 孔板底部的尺寸。
- 使用有机硅密封剂,将 0.05 μm 聚碳酸酯膜粘附到 96 孔板的底部,确保胶粘剂密封孔,但不会完全覆盖孔的开口。晾干至少 24 小时或按照粘合剂上的说明进行作。
注意:密封胶必须是防水、无毒和 100% 硅胶的。大多数水族箱密封剂效果很好。
3. 细胞悬液的制备
- 标记 4 个 15 mL 离心管 A-D 和 4 个 50 mL 离心管 E-H,并向每个离心管中加入 4.5 mL 无菌水。
- 在 50 mL 离心管中量取 1 g 土壤,加入 10 mL 无菌水,涡旋 10 分钟。
- 让土壤悬浮液沉降 10 分钟。
- 从土壤中吸取 0.5 mL 含有细胞的上清液到试管 A 中并充分混合。
- 将 0.5 mL 试管 A 中的细胞悬液添加到试管 B 中并充分混合。将 0.5 mL 溶液 B 转移到试管 C 中并充分混合。对所有剩余的离心管重复此作,在 8 个离心管上完成一系列 10 倍稀释。
- 从试管 H 中取出 0.5 mL,使所有试管中的体积相等。
注意:这些浓度针对当地土壤进行了优化。首次尝试此程序时,请进行更大范围的稀释以找到合适的样品浓度。
4. 改良的 iChip 接种
- 将 iChips 完全浸入 95% 乙醇中至少 15 分钟。
- 从乙醇中取出板子,将它们放在无菌纸巾上。在让乙醇蒸发的同时,在层流罩中打开紫外线消毒器 15 分钟以进一步消毒。
- 将 360 μL 无菌 SMS 培养基移液到板的第一列中,作为对照孔。
- 将 45 mL 冷却至 50 °C 的 SMS 添加到管 E 中的细胞悬液中,并充分混合以混合细胞悬液和琼脂。
- 将步骤 4.4 中的 360 μL 琼脂细胞混合物移液到 96 孔板的所有其他孔中。
注意:建议使用多通道移液器,因为琼脂会快速凝固。不建议在混合后重新加热琼脂细胞混合物,因为有杀死微生物的风险。 - 培养基凝固后,用 PCR 板盖密封板的顶部。
- 用试管 F-H 重复步骤 3.3 至 3.6,以填充总共四个浓度差异为 10 倍的 iChChip。
注:培养基可在需要时用微波炉加热 30 秒,或保存在板之间的 60 °C 热水浴中,以确保每个板保持熔融。
5. 孵化
- 将板放在一个装有步骤 3.2 中使用的大约一英寸土壤的盒子中,膜面朝下并盖上盖子。
- 将板放在有盖的容器中,在 25 °C 的黑暗处孵育。
注意:一周后,检查修改后的 iChips 的内容物,以确保在设置过程中没有发生污染。污染表现为板的所有孔中单个或少量微生物的过度生长。 - 6 周后,检查 iChips。使用在不到 25% 的孔中含有生长物的板进行菌落分离。
6. 菌落分离
- 用无菌水冲洗改性的 iChips 3 次,以去除所有污垢颗粒。
- 用 95% 乙醇擦拭板的顶部和侧面,避开带有半透膜的一侧。
- 使用无菌刀片,切开一个包含菌落的孔周围的板盖。
- 使用无菌划线工具,刺穿菌落,并使用四象限法在 100 x 15 mm 板中的 SMS 琼脂上划线。
- 对 iChips 中每孔生长一个菌落的所有其他菌落重复此作。
注:对于微小的菌落,可以将板放回土壤上并孵育,直到足够大,可以进行传代培养,只要没有去除孔周围的板盖。 - 在 25 °C 下孵育 SMS 板并监测生长。
- 检查菌落以确保已分离出 axenic 培养物。
7. 微生物的鉴定
- 对回收的分离株的基因组进行测序20.
- 使用 JSpeciesWS 或其他基于基因组的鉴定平台评估基因组,以确定每个分离株与 GenomesDB 中已知微生物的相似程度。
- 将低于社区接受的物种鉴定阈值的分离株归类为推定的新分离株。
结果
该协议的直观概述如图 1 所示。成功的改良 iChip 实验导致至少一个板在不到 25% 的孔中含有生长,而对照孔中没有发生生长。这种生长的孔数通常确保在孔中分离出单个菌落。此外,菌落的数量或包含菌落的孔的数量应随着从一系列稀释液制备的每个后续板减少 10 倍。代表性结果如图 2 所示。其中板 7 和 8 是成功的 iChip,因为它们在不到 25% 的孔中含有生长,并且含有菌落的孔的百分比随着每个板的增加而降低,如 表 1 所示。 如果试验中的所有板每孔都包含一个以上的菌落,或者任何孔中都没有菌落,则构成负面结果。
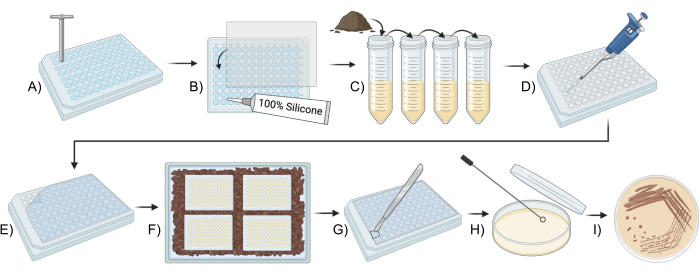
图 1:使用改良的 iChip 培养土壤微生物的示意图概述。 (A,B) 板的构造(步骤 2)。(C) 接种物的制备(步骤 3)。(D,E) iChip 设置(步骤 4)。(F) iChip 的孵育(步骤 5)。(G-I)将分离物转移到常规培养基中(步骤 6)。 请单击此处查看此图的较大版本。
| 芯片 | 琼脂悬浮液中的土壤浓度 | 有菌落的井 | 菌落形成单位 |
| E | 5.0 x 10-6 克/毫升 | 都 | 太多了,数不清 |
| F | 5.0 x 10-7 克/毫升 | 都 | 太多了,数不清 |
| G | 5.0 x 10-8 克/毫升 | 22 | 22 |
| H | 5.0 x 10-9 克/毫升 | 6 | 6 |
表 1:来自成功 iChip 实验的代表性数据,其中含有菌落的孔数随着每次串联稀释而减少。
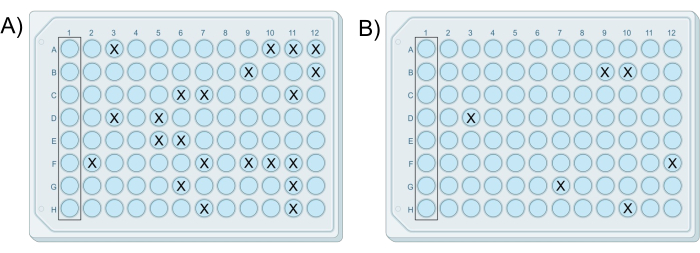
图 2:两个成功的改良 iChip 的示例结果。 iChips 来自同一实验,其中 (A) 由细胞-琼脂混合物 G 制备的平板包含 22 个菌落,以及 (B) 由细胞-琼脂混合物 H 制备的平板含有 6 个菌落,与混合物之间的 10 倍稀释差异一致。 请单击此处查看此图的较大版本。
可以用肉眼或显微镜观察菌落,如图 3A-G 所示。菌落可能在琼脂表面生长,如图 3B 所示或嵌入琼脂中,例如图 3F 中的菌落。在成功修饰的 iChip 中,可能存在同一孔中有两个菌落的情况,如图 3G 所示。在某些情况下,菌落相距足够远,可以分别用针刺穿并进行传代培养;然而,图 3G 中的那些不太可能很容易地作为轴系微生物培养。图 4 显示了从 iChip 转移到传统 SMS 培养基上的菌落示例。
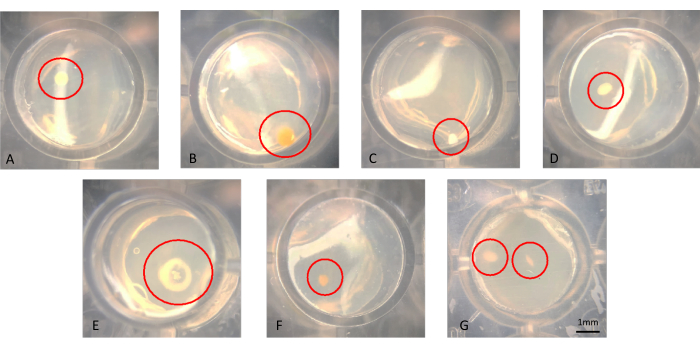
图 3:孵育 6 周后在 iChip 中生长的菌落的代表性显微镜视图。 (AF) 观察到单个菌落,应从中进行传代培养,在 (G) 中,近距离观察到两个菌落,这些菌落很难作为轴系培养物获得。 请单击此处查看此图的较大版本。
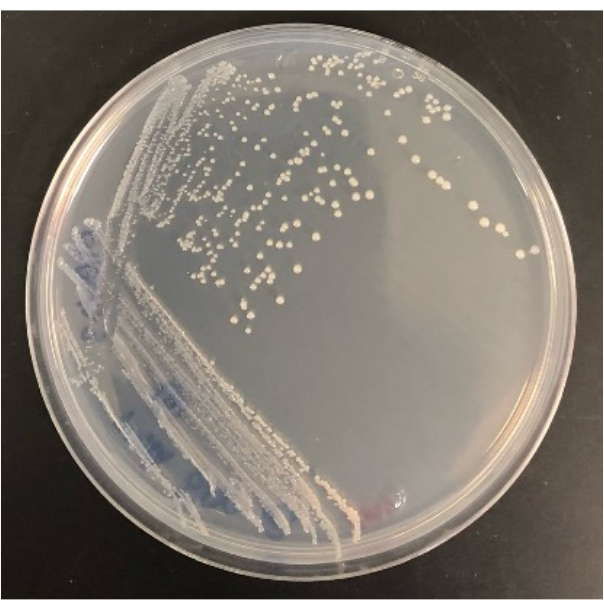
图 4:从 SMS 琼脂平板上生长的改良 iChip 中分离出的细菌的代表性结果。请单击此处查看此图的较大版本。
将克隆从修饰的 iChip 分离到常规琼脂平板上的能力也决定了实验的成功。并非所有在改良 iChip 中生长的菌落都能在转移到常规板后存活。在 iChip 中,菌落暴露于生长因子和土壤养分中,从而提高了其可培养性。对于某些微生物,一段时间的驯化不足以使它们在常规条件下进行培养。从 iChip 到平板的菌落回收率因实验而异,但有些应该在平板中生长,因为这种方法也会培养易于培养的微生物。例如, 图 2 中修饰的 iChips 总共包含 28 个菌落,传代培养后在琼脂平板上生长的菌落数为 16 个,而 12 个菌落在转移后没有存活。一旦在全尺寸琼脂平板上培养,就可以鉴定分离株并用于进一步研究,如图 4 所示。
成功的改良 iChip 实验应导致分离出与物种水平上的已知微生物不同的微生物。通过将分离的 DNA 与已知微生物的基因组进行比较,可以确定每个分离株与已知微生物的相似程度。与每个分离株最接近的相关微生物的 tetra Z 分数描述了相似性百分比。 图 5 显示,在分离的 12 种细菌中,4 种是新种或更高种,2 种可能是新菌株,6 种是已知微生物。几种新物种或菌株的回收率与先前发表的 iChip 实验一致,表明这种改进的 iChip 结构不会阻碍新物种11 回收率的增加。
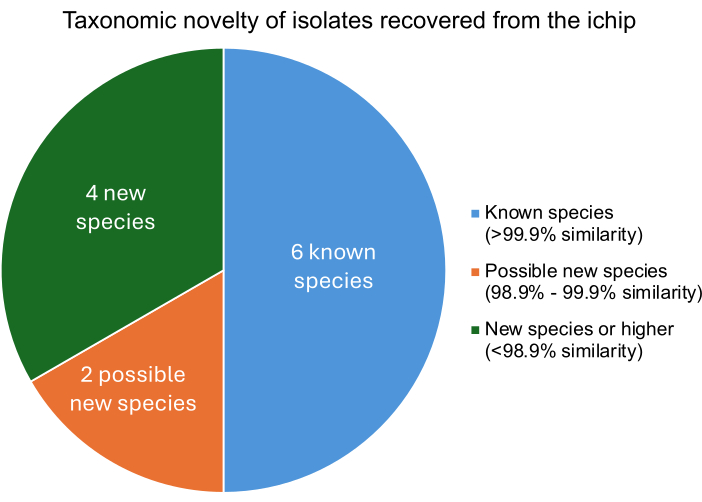
图 5:从 iChip 实验中回收的分离株的分类分类级别。 根据 tetra Z 分数确定百分比相似性,该分数描述了分离株与已知细菌的相似性,其中 >99.9% 相似性表示物种匹配,98.9%-99.9% 相似性表示可能的新物种,<98.9% 相似度表示新物种或更高。 请单击此处查看此图的较大版本。
讨论
近年来,许多方法,如基因组挖掘和检查沉默生物合成途径,都促进了新型生物活性化学物质的发现21,22。然而,使用这种方法发现的 NP 通常与已知化合物表现出高度的结构相似性。获得以前未培养的微生物将释放更大的化学多样性和具有新作用模式的 NP,从而更好地帮助对抗微生物耐药性。iChip 分离技术已被证明可以提高新型微生物的培养性,可用于构建微生物文库以发现新型 NP。
iChip 的最早概念化发表于 2002 年,它由单个金属环组成,其中包含用半透膜密封并原位孵育的琼脂细胞混合物23。后来在 2010 年迭代它包含一个具有许多孔的小芯片11,随后在 2017 年有影响力的 Nature Protocols 出版物中开发了一种由常见实验室材料制成的廉价 iChip9。本出版物对 Nature 协议进行了几处修改,以提高其实用性和易用性。与这种方法相比,Nature 协议涉及将半透膜粘合到芯片的两侧,而这种方法在一侧使用粘合板盖。加载 iChip 后使用硅胶连接膜时可能会出现问题。据我们所知,所有无毒有机硅胶粘剂在凝固时都会释放出乙酸,这可能会影响细胞活力24。本视频中概述的方案还通过使用 PCR 板盖密封填充板,显著减少了接种后所需的处理量,进一步确保无菌并缩短设置时间。根据本实验中多种新型分离株的鉴定,仅使用单个半透膜确实抑制了由两个半透膜构建的 iChips 的新型生物发现率的增加,而已报道这种发现率由两个半透膜构建。然而,需要使用更大样本量的进一步实验来量化影响。
另一种方法修改是仅从改良的 iChips 中传代培养,这些 iChips 在不到 25% 的孔中含有生长。如果菌落在板的大部分孔中生长,则可能有一些孔包含多种微生物,即使不容易看到。在方法开发过程中,发现从包含多个菌落的孔中传代培养时,基本上不可能获得轴系培养物。非轴系培养物的分离在下游的生物活性测定和鉴定方面存在重大问题。因此,为简单起见,建议仅对在部分孔中生长菌落的修饰 iChips 进行传代培养。
iChip 方法可能出现的最重要问题是孔或整个 iChips 被其他微生物污染。在对照孔中生长的菌落,或在多个孔中或修饰的 iChip 的整个区域中生长相同的微生物表明存在污染。污染源可能是由于设置过程中材料灭菌不充分或无菌技术不当。在这种情况下,请确保使用的所有材料都按照方法中的指示进行高压灭菌或乙醇灭菌,并确保除琼脂细胞混合物外,任何非无菌物品与改性的 iChip 之间不会发生接触。如果在多个改良的 iChip 孔的底部或顶部观察到单个微生物的过度生长,则很可能是孔和膜之间密封不完全的结果。在这种情况下,请确保使用的粘合剂是 100% 硅胶,在乙醇中不会降解,并确保在施工过程中每个孔周围半透膜和 PCR 板盖完全密封。
当前方案中使用的稀释系列应为大多数土壤类型提供足够的稀释,储存期少于一周,因为它包括一千倍的稀释范围。然而,不同土壤类型的微生物数量可能存在显著差异。如果对照孔中没有生长,但所有四种修饰的 iChIP 的每个孔中都有多个菌落生长,则使用的细胞浓度不够低。应修改稀释系列,以在用于设置改性 iChChips 的细胞-培养基混合物中达到较低的细胞浓度。同样,如果在这些板中的任何一个中都没有观察到生长,则应增加细胞 - 培养基混合物中使用的浓度。或者,所用培养基的温度可能太高,无法使微生物存活。在这种情况下,在添加到稀释的细胞悬液中之前,应让培养基尽可能冷却而不凝固。
该技术是克服巨大板数异常的重要进步。然而,它仍然受到常规培养技术不适用性的限制,从改良的 iChip 转移到常规琼脂板后无法存活的微生物数量就表明了这一点。以前的出版物报道,在 iChips 中进行多轮传代培养和孵育进一步提高了微生物的可培养性。在 iChip 中,在琼脂上驯化时间的延长和暴露于土壤生长因子会增加菌落仅在琼脂上生长的机会。然而,尚未报道这种方法产生比单个 iChip 孵育更高的新型微生物的可能性25,26。
正在探索和修改许多其他策略,以增加新型微生物的可培养性。例如,有人提议用半透性材料制造整个 iChip 设备,以促进微生物在相邻孔中的共培养27。话虽如此,本出版物中概述的结构优势是其成本低,构建一个板的成本相当于大约 12 美元(每个 96 孔板 4 美元,每个膜 8 美元,每个 PCR 盖 2 美元)。此外,其简单的结构使其成为按描述使用时的简单工具,并提供了许多定制的可能性。尽管该方案使用对细菌选择性的培养基,但理论上可以通过修改所使用的培养基来调整实验设置以针对所需的微生物种群,例如使用细菌抑制培养基来靶向真菌,或使用低营养琼脂来靶向孢子微生物。
披露声明
作者已声明没有利益冲突。
致谢
我们非常感谢加拿大农业和农业食品部提供的 J-base 资金(J-001757、J-001842),使该项目成为可能。我们感谢 Brett van Heyningen 为本协议拍摄视频内容。我们还要感谢 Ron Matters 收集本出版物中描述的实验中使用的土壤样本。
材料
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
参考文献
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。