Method Article
iChip技術を用いた土壌微生物の分離
要約
iChip技術は、安価でシンプルな in-situ 分離装置を利用しており、土壌からの新規微生物の発見率を高めます。新しい微生物は、他のアプリケーションの中でも、土壌マイクロバイオームや天然物の発見に関連するさらなる研究に使用できます。
要約
iChip単離技術は、これまで培養できなかった微生物の培養性を高める in-situ 単離装置を使用します。微生物は、新規の化学物質や潜在的に生理活性な分子の重要な供給源です。しかし、従来の実験室の方法で培養できる環境微生物はわずか1%です。薬剤耐性の増加に伴い、感染症や疾病に対抗するための新薬を見つけることが何よりも重要であり、新しい分子を見つけるための重要な方法は、新しい微生物の発見です。96ウェルプレートのウェルで土壌微生物のコロニーをインキュベートし、半透膜で密封し、土壌の上でインキュベートすることにより、微生物は土壌からの水および成長因子と接触し、実験室環境での新規微生物の分離が可能になります。iChipで一定期間家畜化した後、微生物を従来の培地に継代培養し、さらなる研究に使用できる可能性があります。このデバイスは、生理活性分子の発見や土壌マイクロバイオームの研究に価値があり、以前にも両方のアプリケーションで使用されてきました。
概要
環境細菌は、天然物(NP)1の豊富な供給源です。これらの代謝産物は、微生物の生存に不可欠ではなく、代わりに、周囲の他の微生物と競合することにより、微生物のコロニー形成を促進するために生成されます2。進化により、NPの化学構造と活性が微調整され、NPは効果的な抗菌剤になりました。そのような例は、2003年にFDAによって承認された抗生物質であるダプトマイシンです3。近年、微生物耐性の発生と重症度が増しており、ダプトマイシンのような新薬は、古い抗生物質に対する耐性を発達させた感染症に対する最終選択薬として立っています4。新しい抗生物質やその他の薬の開発は、一般的な感染症や病気を治療可能に保つために不可欠です。
NPはしばしば優れた医薬品のリードになりますが、製薬業界は1950年代から1960年代にかけての抗生物質の黄金時代以降、創薬のための合成法に目を向けました5。1970年代までに、同じ微生物と抗菌代謝物が繰り返し再分離され続け、発見される新薬候補はますます少なくなっていきました1,6。土壌マイクロバイオームには多様な微生物が含まれていますが、従来の実験室条件下では少数の微生物しか分離できません。ゲノミクスで検出される微生物の土壌微生物は、従来の培養方法では観察されないものが圧倒的多数を占めており、「グレートプレート数異常」と呼ばれる問題となっています7。これらの培養不可能な微生物は、存在することが知られているため、微生物暗黒物質と呼ばれていますが、純粋な培養物として分離できるようになるまで、in vitroで研究することはできません。これらの培養不可能な微生物は、微生物耐性の時代に非常に重要な、豊富な薬剤候補を生み出す可能性があります。
iChip技術は、環境からの新規微生物の回収率を高めるために使用できる1つの方法である8,9。この技術は、インキュベーション中の細菌の自然環境をシミュレートするのに役立ち、それによって、従来の実験室条件では生き残れない微生物の培養能力を向上させる10。改変iChipは、土壌、堆積物、海洋環境、および動物の腸など、多くの異なる供給源から微生物を分離するためにすでに開発され、使用されている11,12,13,14,15,16。おそらく、この技術の使用で最も影響力のあるケースは、NovoBiotic Pharmaceuticals社によるもので、新しい細菌であるEleftheria terraeが発見されました17。この微生物は、新しいクラスの抗生物質であるテイショバクチンを産生することが判明し、これは耐性の発現が検出されることなく、人間の健康に関連するいくつかの耐性細菌種を阻害します5,18。テイショバクチンは数十年ぶりに発見された新しいクラスの抗生物質であり、この技術が新薬の発見と大きなプレート数の異常を克服するための有望なルートであることを示しています19。本明細書では、Berdyらによる以前の論文に基づく、使いやすさと汚染防止9のために最適化された修正されたiChipが提示される。
プロトコル
1. 培地の調製と滅菌
- 1 Lの水に0.1 gの馬鈴薯デンプン、1 gのカサミノ酸、0.125 gのカゼイン消化物、および15 gの細菌学的または工業用寒天からなるコハク酸最小塩培地(SMS)を調製し、滅菌します。
- 培地と100 mLの分子グレードの水を、20分間、121°Cの液体サイクルでオートクレーブ滅菌します。
注:寒天細胞混合物と接触する他のすべてのアイテムが無菌状態で購入されていることを確認してください(ピペットチップや遠心チューブなど)、それ以外の場合はオートクレーブもかけてください。
2.変更されたiChip構造
- 5 mm寒天パンチツールを使用して、4つの96ウェルプレートのウェルの底を取り外します。
- 0.05 μm のポリカーボネートメンブレンを 7.6 cm x 11 cm の長方形にカットします。これは、96 ウェルプレートの底部の寸法に等しくなります。
- シリコーンシーラントを使用して、0.05 μmのポリカーボネートメンブレンを96ウェルプレートの底に接着し、接着剤がウェルをシールしますが、ウェルの開口部を完全に覆うことはありません。少なくとも24時間、または接着剤の指示に従って乾燥させます。
注:シーラントは、防水性があり、毒性がなく、100%シリコーンでなければなりません。ほとんどの水族館のシーラントはうまく機能します。
3. 細胞懸濁液の調製
- 4本の15 mL遠心分離チューブA〜Dおよび4本の50 mL遠心分離チューブE〜Hを標識し、それぞれに4.5 mLの滅菌水を加えます。
- 50 mLの遠心分離チューブに1 gの土壌を計量し、滅菌水10 mLを加え、10分間ボルテックスします。
- 土壌懸濁液を10分間沈殿させます。
- 土壌からの細胞を含む上清0.5mLをチューブAにピペットで移し、よく混ぜます。
- チューブA中の細胞懸濁液0.5mLをチューブBに加え、十分に混合します。0.5mLの溶液BをチューブCに移し、十分に混合します。残りのすべての遠心分離チューブについてこれを繰り返し、8つの遠心分離チューブ全体で一連の10倍希釈を完了します。
- チューブHから0.5mLを取り出して、すべてのチューブに同じ容量にします。.
注:これらの濃度は、地元の土壌に最適化されています。この手順を初めて試みる場合は、適切なサンプル濃度を見つけるために、より広い範囲の希釈を実行します。
4. iChipの接種方法
- iChipsを95%エタノールに少なくとも15分間完全に沈めます。
- エタノールからプレートを取り出し、滅菌ペーパータオルの上に置きます。エタノールを蒸発させながら、層流フードでUV滅菌器を15分間オンにして、さらに滅菌します。
- 滅菌済みSMS培地360μLをプレートの1列目にピペットで注入し、コントロールウェルとして使用します。
- チューブEの細胞懸濁液に50°Cに冷却したSMS45 mLを加え、細胞懸濁液と寒天をよく混ぜ合わせます。
- ステップ4.4の寒天細胞混合物360μLを96ウェルプレートの他のすべてのウェルにピペットで移します。
注:寒天は急速に固まるため、マルチチャンネルピペットをお勧めします。一度混合した寒天細胞混合物を再加熱することは、微生物を殺すリスクがあるため、お勧めできません。 - 培地をセットしたら、プレートの上部をPCRプレートカバーで密封します。
- チューブF-Hでステップ3.3から3.6を繰り返し、濃度が10倍異なる合計4つのiChipを充填します。
注:メディアは、必要に応じてキャップを緩めて30秒間電子レンジにかけるか、プレート間の60°Cの温水浴に保持して、それぞれで溶融したままにすることができます。
5. インキュベーション
- 手順3.2で使用した土の約1インチが入った箱にプレートを置き、膜を下にしてカバーを置きます。
- 蓋をした容器内のプレートを、25°Cの暗所でインキュベートします。
注意: 1週間後、変更されたiChipの内容を調べて、セットアップ中に汚染が発生しなかったことを確認します。汚染は、プレートのすべてのウェルで単一または少数の微生物が異常増殖することによって示されます。 - 6週間後、iChipsを調べます。コロニーの分離には、ウェルの25%未満で増殖を含むプレートを使用してください。
6. コロニーの分離
- 改質したiChipsを滅菌水で3回すすぎ、すべての土壌微粒子を取り除きます。
- プレートの上部と側面を95%エタノールで拭き、半透膜のある面を避けます。
- 滅菌ブレードを使用して、コロニーを含む1つのウェルの周りのプレートカバーを切断します。
- 滅菌ストリーキングツールを使用して、コロニーを穿孔し、4象限法を使用して100 x 15 mmプレートのSMS寒天にストリークします。
- 井戸ごとに1つのコロニーを成長させているiChips内の他のすべてのコロニーについて繰り返します。
注:コロニーが小さい場合は、プレートカバーがウェルの周りで取り外されない限り、プレートを土壌に戻し、継代培養するのに十分な大きさになるまでインキュベートすることができます。 - SMSプレートを25°Cでインキュベートし、成長をモニターします。
- コロニーを調べて、無菌培養物が分離されていることを確認します。
7. 微生物の同定
- 回収された分離株のゲノムを配列決定します20。
- JSpeciesWSまたは別のゲノムベースの同定プラットフォームを使用してゲノムを評価し、GenomesDB内の既知の微生物に対する各分離株の類似性を判断します。
- コミュニティが種識別のために受け入れたしきい値を下回る分離株を、推定的に新しいものとして分類します。
結果
プロトコルの視覚的な概要を 図1に示します。修正されたiChip実験が成功すると、少なくとも1つのプレートでウェルの25%未満で成長が含まれ、コントロールウェルでは成長が起こりません。成長を伴うこの数の井戸は、一般に、単一のコロニーが井戸で分離されることを保証します。さらに、コロニーの数、またはコロニーを含むウェルの数は、一連の希釈から調製された後続のプレートごとに10倍に減少するはずです。代表的な結果を 図 2 に示します。ここで、プレート7と8は、ウェルの25%未満で成長を含み、コロニーを含むウェルの割合は表1に示すように各プレートで減少するため、成功したiChipです 。 試験のすべてのプレートにウェルごとに複数のコロニーが含まれている場合、またはどのウェルにもコロニーがない場合、それは否定的な結果を構成します。
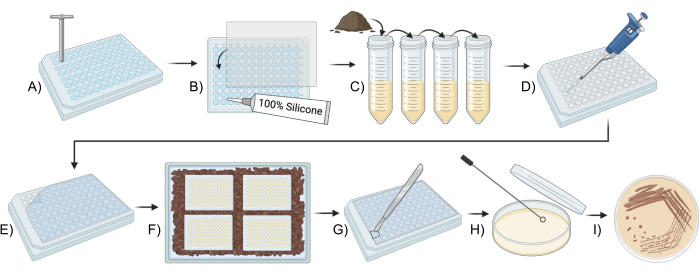
図1:改良型iChipを使用して土壌微生物を培養するための概略図(A、B)プレートの構造(ステップ2)。(C)接種材料の調製(ステップ3)。(D、E) iChip セットアップ (ステップ 4)。(f)iChipのインキュベーション(ステップ5)。(G-I)分離株の従来の培地への移し替え(ステップ6)。この図の拡大版を表示するには、ここをクリックしてください。
| アイチップ | 寒天懸濁液中の土壌濃度 | コロニーのある井戸 | コロニー形成ユニット |
| E | 5.0 x 10-6 g/mL | すべての | 数え切れないほどたくさんあります |
| F | 5.0 x 10-7 g/mL | すべての | 数え切れないほどたくさんあります |
| G | 5.0 x 10-8 g/mL | 22 | 22 |
| H | 5.0 x 10-9 g/mL | 6 | 6 |
表1:成功したiChip実験の代表的なデータで、コロニーを含むウェルの数は、連続して希釈するごとに減少します。
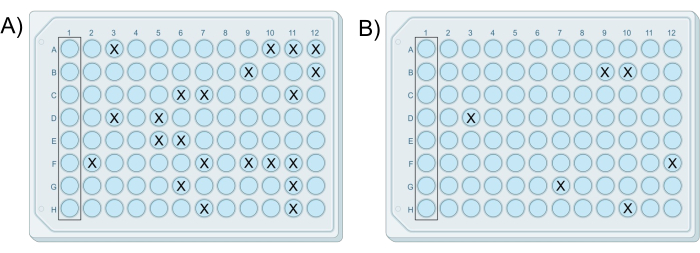
図2:2つの成功した改変iChipの例示的な結果。 iChipは、(A)細胞寒天混合物Gから調製したプレートには22コロニーが含まれ、(B)細胞寒天混合物Hから調製したプレートには6つのコロニーが含まれ、混合物間の10倍の希釈差と一致するという同じ実験から得られたものです。 この図の拡大版を表示するには、ここをクリックしてください。
図3A-Gに示すように、コロニーを肉眼または顕微鏡で視覚化できます。コロニーは、図3Bに見られるように寒天の表面に成長しているかもしれないし、図3Fのコロニーのように寒天に埋め込まれているかもしれない。図3Gのように、成功した改変iChipの同じウェルに2つのコロニーがある場合があります。場合によっては、コロニーが十分に離れているため、別々に針で穴を開けて継代培養することができます。しかし、図3Gのものは、無菌性微生物として容易に培養できるとは考えにくい。iChipから従来のSMSメディアに転送されたコロニーの例を図4に示します。
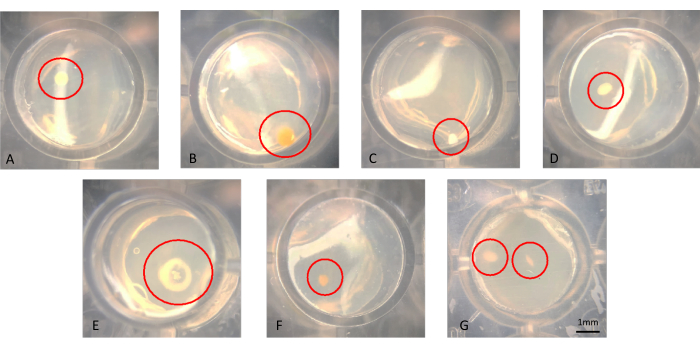
図3:インキュベーション6週間後にiChipで成長するコロニーの代表的な顕微鏡図(A-F)継代培養されるべき単一のコロニーが観察され、(G)2つのコロニーが近接して観察され、これらは無菌培養としては得られにくい。この図の拡大版を表示するには、ここをクリックしてください。
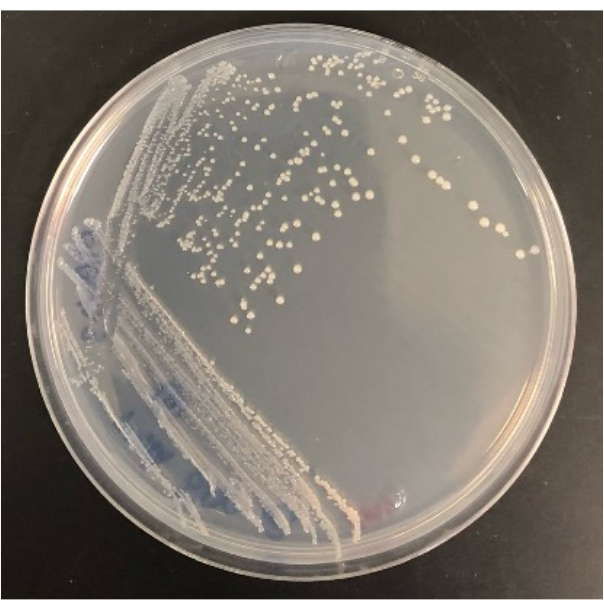
図4:改変されたiChipから分離された細菌の代表的な結果。この図の拡大版を見るには、ここをクリックしてください。
改変されたiChipから従来の寒天プレート上にコロニーを分離する能力も、実験の成功を決定します。改変されたiChipで増殖するすべてのコロニーが、従来のプレートへの転写後も生き残るわけではありません。iChipでは、コロニーは成長因子と土壌栄養素にさらされ、栽培性が向上します。一部の微生物にとって、家畜化の期間では、従来の条件で培養可能にするには不十分です。iChipからプレートへのコロニーの回収率は実験ごとに異なりますが、この方法では容易に培養可能な微生物も培養できるため、プレートで成長するものもあります。例えば、 図2 の改変iChipには合計28個のコロニーが含まれており、継代後に寒天プレート上で成長したコロニーの数は16個でしたが、12個は移植後も生き残れませんでした。分離物は、 図4に示すように、フルサイズの寒天プレートで培養すると、同定してさらなる研究に使用できます。
改変iChip実験が成功すると、既知の微生物と種レベルで異なる微生物が分離されるはずです。各分離株と既知の微生物との類似性の程度は、単離されたDNAを既知の微生物のゲノムと比較することによって見つけられます。各分離株に最も近い関連微生物のテトラ Z スコアは、類似性の割合を表します。 図5 は、単離された12の細菌のうち、4つは新種以上、2つはおそらく新株、6つは既知の微生物であることを示しています。いくつかの新規種または株の回収は、以前に発表されたiChip実験と一致しており、この改変されたiChip構造が新規種11の回収の増加を妨げないことを示している。
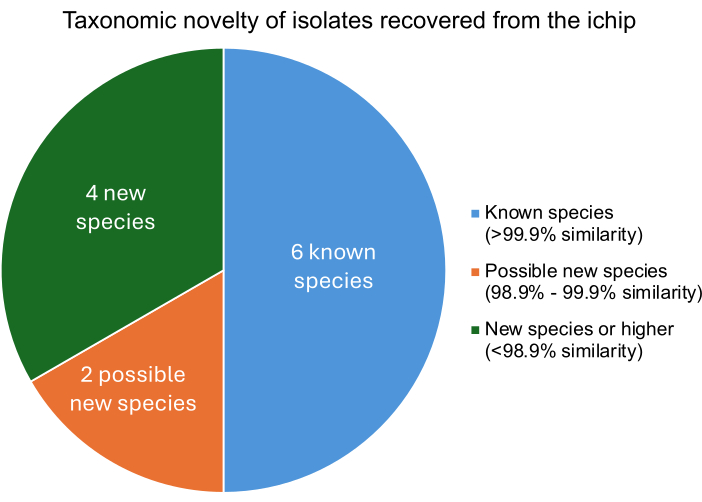
図5:iChip実験から回収された分離株の分類学的分類レベル。 類似性の割合は、既知の細菌に対する分離株の類似性を説明するテトラZスコアに基づいて決定されました。このスコアは、>99.9%の類似性が種の一致を示し、98.9%-99.9%の類似性が新種の可能性があることを示し、<98.9%の類似性が新種以上を示します。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
近年、ゲノムマイニングやサイレント生合成経路の調査など、数多くの方法が新しい生理活性化学物質の発見を促進しています21,22。しかし、このような方法を用いて発見されたNPは、多くの場合、既知の化合物と高い構造的類似性を示します。これまで培養されていなかった微生物にアクセスできるようになると、化学的多様性が広がり、微生物耐性との闘いに役立つ新しい作用機序を持つNPが解き放たれます。iChip単離技術は、新規微生物の培養性を向上させることが示されており、新規NPの発見のための微生物のライブラリーを構築するために使用できます。
iChipの最も初期の概念化は2002年に発表され、半透膜で密封された寒天細胞混合物を含む単一の金属リングで構成され、insitu23でインキュベートされました。その後、2010年に多くのウェルを持つ小さなチップを搭載するために改良が加えられ11、その後、2017年に影響力のあるNature Protocols誌に掲載された9の論文で、一般的な実験材料から構築された安価なiChipが開発されました。この出版物では、Natureプロトコルにいくつかの変更が加えられ、その実用性と使いやすさが向上しました。Natureのプロトコルでは、チップの両面に半透膜を接着しますが、この方法では片面に接着プレートカバーを使用します。iChipがロードされた後にシリコーン接着剤を使用してメンブレンを取り付けると、問題が発生する可能性があります。私たちの知る限り、すべての無毒のシリコーン接着剤は、固まるときに酢酸を放出し、細胞の生存率に影響を与える可能性があります24。また、このビデオで概説されているプロトコルは、PCRプレートカバーを使用して充填されたプレートをシールすることにより、接種後の取り扱い量を大幅に削減し、無菌性をさらに確保し、セットアップ時間を短縮します。この実験から得られた複数の新規分離株の同定に基づくと、単一の半透過性メンブレンのみを使用すると、2つの半透過性メンブレンで構築されたiChipsで報告されている新規生物の発見率の増加が抑制されます。ただし、影響を定量化するには、より大きなサンプルサイズでのさらなる実験が必要になります。
別の方法の変更は、ウェルの25%未満で増殖を含む改変されたiChipからのみ継代培養することです。プレートのウェルの大部分でコロニーが成長する場合、簡単には見えない場合でも、複数の微生物を含むウェルが存在する可能性があります。メソッド開発中に、複数のコロニーを含むウェルから継代培養する場合、アキセン培養物を得ることはほとんど不可能であることがわかりました。非無素血培養物の単離は、生物活性アッセイおよび同定の観点から、下流に重大な問題を提示します。したがって、簡単にするために、ウェルの一部でコロニーが成長している改変iChipのみを継代培養することをお勧めします。
iChip法で発生する可能性のある最も重要な問題は、ウェルまたはiChip全体が他の微生物に汚染されることです。汚染は、コントロールウェルで増殖するコロニー、または改変されたiChipの複数のウェルまたは全領域で増殖する同じ微生物によって示されます。汚染源は、セットアップ中の材料の不適切な滅菌、または不適切な無菌技術が原因である可能性があります。このような場合は、使用するすべての材料が方法に示されているようにオートクレーブ滅菌またはエタノール滅菌されていることを確認し、寒天細胞混合物以外の非滅菌アイテムと改変iChipとの間に接触が発生しないようにしてください。単一の微生物の異常増殖が複数の改変されたiChipウェルの底部または上部で観察された場合、それはウェルとメンブレンの間の不完全なシールの結果である可能性が最も高いです。この場合、使用する接着剤がエタノールで劣化しない100%シリコーンであることを確認し、建設中に半透膜とPCRプレートカバーが各ウェルの周囲に完全に密閉されていることを確認してください。
現在のプロトコルで使用されている希釈シリーズは、1000倍の範囲の希釈が含まれているため、保存期間が1週間未満のほとんどの土壌タイプで適切な希釈を提供するはずです。ただし、土壌の種類によって微生物の数には大きなばらつきがあります。コントロールウェルに増殖が含まれていないが、4つの改変iChipすべての各ウェルで複数のコロニーが増殖している場合、使用された細胞濃度は十分に低くありませんでした。希釈シリーズは、改変されたiChipのセットアップに使用した細胞-培地混合物中の細胞濃度が低くなるように改変する必要があります。同様に、これらのプレートのいずれにも成長が観察されない場合は、細胞 - 培地混合物に使用される濃度を増やす必要があります。あるいは、使用した培地の温度が微生物の生存には高すぎた可能性があります。このような場合、培地は固化せずにできるだけ冷却してから、希釈した細胞懸濁液に添加する必要があります。
この技術は、大きなプレート数の異常を克服するための重要な進歩です。しかし、従来の培養技術の不適当性によって、改変されたiChipから従来の寒天プレートへの移動を生き残らない微生物の数によって示されるように、それは依然として制限されています。以前の出版物では、iChipでの複数回の継代培養とインキュベーションにより、微生物の培養性がさらに向上することが報告されています。iChipの寒天上での栽培化時間と土壌成長因子への曝露が長くなると、寒天だけでコロニーが成長する可能性が高くなります。しかし、このアプローチは、単一のiChipインキュベーション25,26よりも新規微生物の可能性が高くなるとは報告されていません。
新規微生物の培養能力を高めるために、他にも多くの戦術が模索され、修正されています。例えば、iChipデバイス全体を半透性材料で作ることは、隣接するウェル27における微生物の共培養を容易にするために提案されている。そうは言っても、この出版物で概説されている構造の利点は、1つのプレートを構築するコストが約12ドル(96ウェルプレートあたり4ドル、メンブレンあたり8ドル、PCRカバーあたり2ドル)に相当する低コストであることです。さらに、そのシンプルな構造は、説明どおりに使用すると単純なツールになり、カスタマイズの多くの可能性を提供します。このプロトコルでは、細菌に選択的な培地を使用しますが、理論的には、細菌抑制培地を使用して真菌を標的にしたり、低栄養寒天培地を使用して微生物を胞子形成したりするなど、使用する培地を変更することにより、目的の微生物集団を標的とするように実験設定を調整することができます。
開示事項
著者は、利益相反がないと宣言しています。
謝辞
このプロジェクトを可能にしたのは、カナダ農業・農業食品省を通じて提供されたJ-baseの資金援助(J-001757、J-001842)に感謝します。このプロトコルのビデオコンテンツを撮影してくれたBrett van Heyningenに感謝します。また、この出版物に記載されている実験で使用された土壌サンプルを収集してくれたRon Mattersに感謝します。
資料
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
参考文献
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved