Method Article
בידוד מיקרואורגניזמים בקרקע באמצעות טכנולוגיית iChip
In This Article
Summary
טכניקת iChip עושה שימוש במכשיר בידוד זול ופשוט באתרו המגביר את שיעורי גילוי המיקרואורגניזמים החדשים מהאדמה. ניתן להשתמש במיקרואורגניזמים חדשים למחקר נוסף הנוגע למיקרוביום הקרקע או לגילוי מוצרים טבעיים, בין היתר.
Abstract
טכניקת הבידוד של iChip משתמשת במכשיר בידוד באתרו המגביר את יכולת הטיפוח של מיקרואורגניזמים שלא היו ניתנים לעיבוד בעבר. מיקרואורגניזמים הם מקור חשוב לכימיה חדשה ומולקולות ביו-אקטיביות פוטנציאליות. עם זאת, רק 1% מהמיקרואורגניזמים הסביבתיים ניתנים לתרבית בשיטות מעבדה קונבנציונליות. עם העלייה בעמידות לאנטיביוטיקה, מציאת תרופות חדשות למלחמה בזיהומים ומחלות היא בעלת חשיבות עליונה, ושיטה קריטית למציאת מולקולות חדשות היא גילוי מיקרואורגניזמים חדשים. על ידי דגירה של מושבות של מיקרואורגניזמים באדמה בבארות של צלחת בת 96 בארות, אטומות בקרום חדיר למחצה ומודגרות על גבי האדמה, החיידקים נמצאים במגע עם מים וגורמי גדילה מהאדמה, מה שמאפשר בידוד של מיקרובים חדשים בסביבה מעבדתית. לאחר תקופה של ביות ב-iChip, מיקרואורגניזמים יכולים להיות תת-תרבית על מדיה קונבנציונלית ולהשתמש בהם למחקר נוסף. מכשיר זה הוא בעל ערך לגילוי מולקולות ביו-אקטיביות ולמחקר מיקרוביום בקרקע ושימש בעבר בשני היישומים.
Introduction
חיידקים סביבתיים הם מקור עשיר למוצרים טבעיים (NPs)1. מטבוליטים אלה אינם חיוניים להישרדותם של המיקרואורגניזמים אלא מיוצרים כדי להקל על ההתיישבות שלהם על ידי תחרות על מיקרואורגניזמים אחרים בסביבתם2. האבולוציה כיווננה את המבנים הכימיים ואת הפעילויות של NPs, והפכה אותם לחומרים אנטי-מיקרוביאליים יעילים. דוגמה כזו היא דפטומיצין, אנטיביוטיקה שאושרה על ידי ה-FDA בשנת 20033. בשנים האחרונות, עמידות חיידקים גדלה בשכיחות ובחומרה, ותרופות חדשות כמו דפטומיצין עומדות כטיפולי קו אחרון נגד זיהומים שפיתחו עמידות לאנטיביוטיקה ישנהיותר. פיתוח אנטיביוטיקה חדשה ותרופות אחרות חיוני לשמירה על זיהומים ומחלות נפוצות ניתנים לטיפול.
למרות שלעתים קרובות NPs מייצרים מובילים טובים לתרופות, תעשיית התרופות פנתה לשיטות סינתטיות לגילוי תרופות לאחר תור הזהב של האנטיביוטיקה בשנות ה-50 עד שנות ה-60. עד שנות ה-70, אותם מיקרואורגניזמים ומטבוליטים אנטי-מיקרוביאליים המשיכו להיות מבודדים שוב ושוב, עם פחות ופחות מועמדים לתרופות חדשותשהתגלו 1,6. המיקרוביום של האדמה מכיל מגוון מיקרוביאלי גדול, אולם רק מספר קטן של מיקרואורגניזמים יכולים להיות מבודדים בתנאי מעבדה קונבנציונליים. הרוב המכריע של המיקרואורגניזמים בקרקע של מיקרואורגניזמים שזוהו על ידי גנומיקה אינם נצפים בתרבית בשיטות תרבות קונבנציונליות, מה שמוביל לכך שהנושא נטבע "אנומליה גדולה של ספירת הצלחות"7. המיקרובים הבלתי ניתנים לתרבות האלה נקראים חומר אפל מיקרוביאלי מכיוון שהם ידועים כקיימים, אך לא ניתן לחקור אותם במבחנה עד שניתן לבודד אותם כתרביות טהורות. מיקרואורגניזמים בלתי ניתנים לתרבות אלה יכולים ככל הנראה לייצר שפע של מועמדים לתרופות, ויש לכך חשיבות רבה בעידן העמידות המיקרוביאלית.
טכניקת iChip היא שיטה אחת שניתן להשתמש בה כדי להגביר את ההתאוששות של מיקרואורגניזמים חדשים מהסביבה 8,9. טכנולוגיה זו מסייעת בהדמיית הסביבה הטבעית של חיידקים במהלך הדגירה, ובכך מגדילה את יכולת הגידול של מיקרואורגניזמים שאחרת אינם יכולים לשרוד בתנאי מעבדה קונבנציונליים10. iChips מותאמים כבר פותחו ושימשו לבידוד מיקרואורגניזמים ממקורות רבים ושונים, כגון אדמה, משקעים, סביבות ימיות ומעיים של בעלי חיים 11,12,13,14,15,16. אולי המקרה המשפיע ביותר של השימוש בטכנולוגיה זו היה על ידי NovoBiotic Pharmaceuticals, שם התגלה חיידק חדש, Eleftheria terrae,17. נמצא כי מיקרואורגניזם זה מייצר Teixobactin, אנטיביוטיקה מסוג חדש, המעכבת מספר מיני חיידקים עמידים הרלוונטיים לבריאות האדם ללא התפתחות עמידות מזוהה 5,18. זו הייתה תגלית משפיעה מכיוון ש-Teixobactin היא הסוג החדש הראשון של אנטיביוטיקה שהתגלה מזה עשרות שנים והיא סימן לכך שטכניקה זו היא דרך מבטיחה לגילוי תרופות חדשות ולהתגברות על האנומליה הגדולה של ספירת הצלחות19. כאן, מוצג iChip שונה המבוסס על פרסום קודם של Berdy et al. אשר עבר אופטימיזציה לנוחות השימוש ומניעת זיהום9.
Protocol
1. הכנת מדיה ועיקור
- הכן ועיקר מדיה מלחים מינימלית סוקסינט (SMS), המורכבת מ 0.1 גרם עמילן תפוחי אדמה, 1 גרם חומצות קסאמינו, 0.125 גרם עיכול קזאין ו -15 גרם אגר בקטריולוגי או טכני ב -1 ליטר מים.
- חיטוי המדיה ו-100 מ"ל של מים בדרגה מולקולרית באמצעות מחזור נוזל של 20 דקות, 121 מעלות צלזיוס.
הערה: ודא שכל שאר הפריטים שיבואו במגע עם תערובת תאי האגר נרכשים סטריליים, כולל קצות פיפטה וצינורות צנטריפוגה, אחרת, חיטוי גם אותם.
2. בניית iChip שונה
- הסר את תחתיות הבארות בארבע צלחות של 96 בארות באמצעות כלי ניקוב אגר 5 מ"מ.
- חותכים ממברנות פוליקרבונט 0.05 מיקרומטר למלבנים בגודל 7.6 ס"מ על 11 ס"מ, או שווה למידות החלק התחתון של צלחת 96 הבארות.
- בעזרת איטום סיליקון, הדביקו ממברנות פוליקרבונט של 0.05 מיקרומטר לתחתית לוחות 96 הבארות, וודא שהדבק אוטם את הבארות, אך אינו מכסה לחלוטין את פתחי הבארות. יש להניח לייבוש למשך 24 שעות לפחות או לפי ההוראות שעל הדבק.
הערה: חומר האיטום חייב להיות עמיד למים, לא רעיל ו-100% סיליקון. רוב חומרי האיטום לאקווריום יעבדו היטב.
3. הכנת תרחיפי תאים
- סמן ארבעה צינורות צנטריפוגה של 15 מ"ל A-D וארבעה צינורות צנטריפוגה של 50 מ"ל E-H והוסף 4.5 מ"ל מים סטריליים לכל אחד.
- מודדים 1 גרם אדמה לצינור צנטריפוגה של 50 מ"ל, מוסיפים 10 מ"ל מים סטריליים ומערבולת למשך 10 דקות.
- תן למתלה האדמה להתיישב במשך 10 דקות.
- פיפטה 0.5 מ"ל של תאים המכיל סופרנטנט מהאדמה לתוך צינור A ומערבבים היטב.
- הוסף 0.5 מ"ל מתלה התאים בצינור A לצינור B וערבב היטב. מעבירים 0.5 מ"ל תמיסה B לצינור C ומערבבים היטב. חזור על פעולה זו עבור כל צינורות הצנטריפוגה הנותרים, והשלים סדרה של דילולים פי 10 על פני שמונת צינורות הצנטריפוגה.
- הסר 0.5 מ"ל מצינור H כדי לקבל נפח שווה בכל הצינורות.
הערה: ריכוזים אלה מותאמים לקרקעות מקומיות. כאשר מנסים הליך זה בפעם הראשונה, בצע מגוון גדול יותר של דילולים כדי למצוא את ריכוז הדגימה המתאים.
4. חיסון iChip שונה
- טבלו לחלוטין את ה-iChips באתנול 95% למשך 15 דקות לפחות.
- מוציאים את הצלחות מהאתנול ומניחים אותן על מגבת נייר סטרילית. תוך כדי מתן אפשרות לאתנול להתאדות, הפעל את מעקר ה-UV במכסה הזרימה הלמינרי למשך 15 דקות כדי לעקר אותם עוד יותר.
- פיפטה 360 מיקרוליטר של מדיית SMS סטרילית לתוך העמודה הראשונה של הצלחת כדי לשמש כבארות בקרה.
- הוסף 45 מ"ל של SMS מקורר ל-50 מעלות צלזיוס למתלה התא בצינור E וערבב היטב כדי לשלב את מתלה התא והאגר.
- פיפטה 360 מיקרוליטר של תערובת תאי אגר משלב 4.4 לכל הבארות האחרות של צלחת 96 הבארות.
הערה: פיפטה רב-ערוצית מומלצת מכיוון שאגר מתמצק במהירות. לא מומלץ לחמם מחדש את תערובת תאי האגר לאחר ערבוב בגלל הסיכון להרוג את המיקרואורגניזמים. - לאחר הגדרת המדיה, אטום את החלק העליון של הלוחות עם מכסה לוחית PCR.
- חזור על שלבים 3.3 עד 3.6 עם צינורות F-H כדי למלא בסך הכל ארבעה iChips עם הבדלים של פי עשרה בריכוזים.
הערה: ניתן לחמם את המדיה במיקרוגל למשך 30 שניות עם מכסה משוחרר בעת הצורך או לשמור באמבט מים חמים של 60 מעלות צלזיוס בין הצלחות כדי להבטיח שהיא תישאר מותכת עבור כל אחת מהן.
5. דגירה
- הניחו את הצלחות בקופסה המכילה כסנטימטר אחד מהאדמה ששימשה בשלב 3.2, כשצד הממברנה כלפי מטה והניחו את המכסה.
- דגרו את הצלחות במיכל המכוסה, במקום חשוך בטמפרטורה של 25 מעלות צלזיוס.
הערה: לאחר שבוע, בדוק את התוכן של ה-iChips ששונו כדי לוודא שלא התרחש זיהום במהלך ההתקנה. זיהום מסומן על ידי צמיחת יתר של מיקרואורגניזמים בודדים או מעטים בכל בארות הצלחת. - לאחר 6 שבועות, בדוק את ה-iChips. השתמש בצלחות המכילות גידול בפחות מ -25% מהבארות לבידוד המושבה.
6. בידוד מושבה
- שטפו את ה-iChips שהשתנו פי 3 במים סטריליים כדי להסיר את כל חלקיקי האדמה.
- נגב את החלק העליון והדפנות של הצלחת עם 95% אתנול, והימנע מהצד עם הממברנה החדירה למחצה.
- בעזרת להב סטרילי, חותכים את מכסה הצלחת סביב באר אחת המכילה מושבה.
- בעזרת כלי פסים סטרילי, חודרים את המושבה ופסים על אגר SMS בלוחות של 100 על 15 מ"מ בשיטת ארבעת הרבעים.
- חזור על הפעולה עבור כל המושבות האחרות ב-iChips שגדלות מושבה אחת לכל באר.
הערה: עבור מושבות זעירות, ניתן להחזיר את הצלחת לאדמה ולדגור אותה עד שהיא גדולה מספיק לתת-תרבות כל עוד מכסה הצלחת לא הוסר סביב הבאר. - דגרו את לוחות ה-SMS ב-25 מעלות צלזיוס ועקבו אחר הצמיחה.
- בדוק מושבות כדי לוודא שתרביות אקסניות בודדו.
7. זיהוי מיקרואורגניזמים
- רצף את הגנום של המבודדים ששוחזרו20.
- הערך את הגנומים באמצעות JSpeciesWS או פלטפורמת זיהוי מבוססת גנום אחרת כדי לקבוע את מידת הדמיון של כל מבודד למיקרואורגניזמים ידועים ב-GenomesDB.
- סווג מבודדים מתחת לסף המקובל בקהילה לזיהוי מינים כחדשים לכאורה.
תוצאות
סקירה ויזואלית של הפרוטוקול מוצגת באיור 1. ניסוי iChip מותאם מוצלח מביא לפחות צלחת אחת המכילה גידול בפחות מ-25% מהבארות, ללא גידול בבארות הבקרה. מספר זה של בארות עם צמיחה מבטיח בדרך כלל שמושבות בודדות מבודדות בבארות. בנוסף, מספר המושבות, או מספר הבארות המכילות מושבות, אמור לרדת פי עשרה עם כל צלחת עוקבת שהוכנה מסדרת הדילול. תוצאות מייצגות מוצגות באיור 2. כאשר לוחות 7 ו-8 הם iChips מוצלחים מכיוון שהם מכילים גידול בפחות מ-25% מהבארות, ואחוז הבארות המכילות מושבות יורד עם כל לוח, כפי שמוצג בטבלה 1. אם כל הלוחות בניסוי מכילים יותר ממושבה אחת לבאר או ללא מושבות בבארות כלשהן, זו תוצאה שלילית.
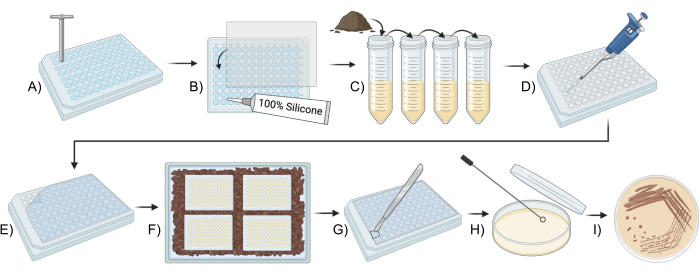
איור 1: סקירה סכמטית לשימוש ב-iChip מותאם לתרבית מיקרואורגניזמים באדמה. (A,B) בניית הלוח (שלב 2). (C) הכנת חיסון (שלב 3). (D,E) הגדרת iChip (שלב 4). (F) דגירה של ה-iChip (שלב 5). (ג-י) העברת המבודדים למדיה קונבנציונלית (שלב 6). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
| איצ'יפ | ריכוז אדמה בתרחיף אגר | בארות עם מושבות | יחידות יוצרות מושבות |
| E | 5.0 x 10-6 גרם/מ"ל | כל | יותר מדי מכדי לספור |
| F | 5.0 x 10-7 גרם/מ"ל | כל | יותר מדי מכדי לספור |
| G | 5.0 x 10-8 גרם/מ"ל | 22 | 22 |
| H | 5.0 x 10-9 גרם/מ"ל | 6 | 6 |
טבלה 1: נתונים מייצגים מניסוי מוצלח של iChip שבו מספר הבארות המכילות מושבות יורד עם כל דילול בסדרה.
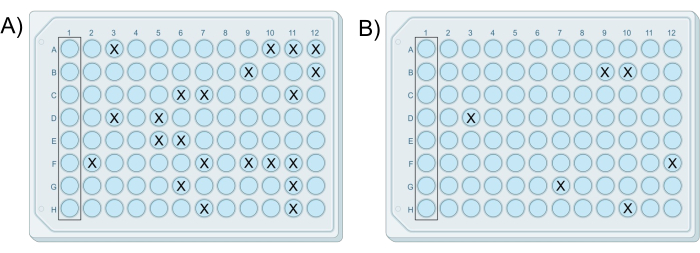
איור 2: תוצאות לדוגמה של שני iChips ששונו בהצלחה. ה-iChips היו מאותו ניסוי שבו (A) הצלחת שהוכנה מתערובת תאים-אגר G מכילה 22 מושבות, ו-(B) הצלחת שהוכנה מתערובת תא-אגר H מכילה שש מושבות, בהתאם להפרש הדילול של פי 10 בין התערובות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
אפשר לדמיין מושבות בעין בלתי או במיקרוסקופיה, כפי שמוצג באיור 3A-G. מושבות עשויות לגדול על פני השטח של האגר, כפי שניתן לראות באיור 3B או משובצות באגר, כמו למשל המושבה באיור 3F. ייתכנו מקרים שבהם ישנן שתי מושבות באותה באר ב-iChip מותאם מוצלח, כמו באיור 3G. במקרים מסוימים, המושבות רחוקות מספיק זו מזו כדי שניתן יהיה לנקב אותן במחט בנפרד ולהפוך אותן לתת-תרבותיות; עם זאת, לא סביר שניתן יהיה לגדל בקלות את אלה באיור 3G כמיקרואורגניזמים אקסניים. דוגמאות למושבות שהועברו מה-iChip למדיית SMS קונבנציונלית מוצגות באיור 4.
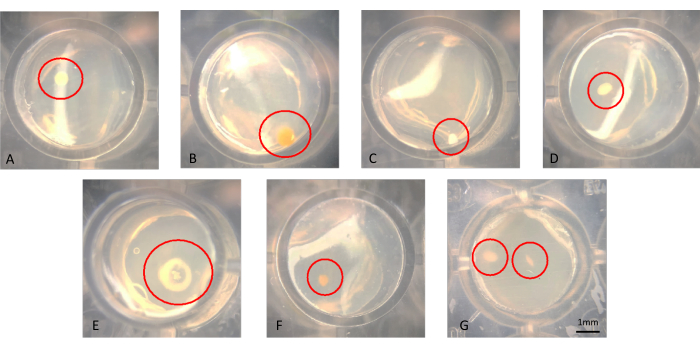
איור 3: תצוגה מיקרוסקופית מייצגת של מושבות הגדלות ב-iChip לאחר 6 שבועות של דגירה. (A-F) נצפות מושבות בודדות, שמהן יש לתת-תרבית, וב-(G) נצפות שתי מושבות בסמיכות, שקשה להשיג כתרביות אקסניות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
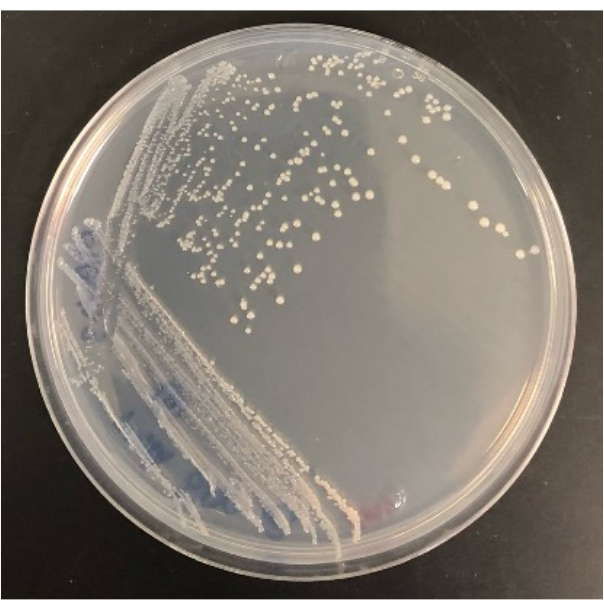
איור 4: תוצאה מייצגת של חיידק מבודד מה-iChip המותאם לאחר שגדל על לוחית אגר SMS. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
היכולת לבודד מושבות מה-iChip המותאם ללוחות אגר קונבנציונליים קובעת גם את הצלחת הניסוי. לא כל המושבות הגדלות ב-iChip המותאם ישרדו את המעבר לצלחות קונבנציונליות. ב-iChip, מושבות חשופות לגורמי גדילה וחומרי הזנה בקרקע, מה שמגביר את יכולת הטיפוח שלהן. עבור מיקרואורגניזמים מסוימים, תקופת ביות אינה מספיקה כדי להפוך אותם לניתנים לתרבות בתנאים קונבנציונליים. קצב ההתאוששות של מושבות מ-iChip לצלחות ישתנה מניסוי לניסוי, אך חלקן צריכות לגדול בצלחות מכיוון ששיטה זו תגדל גם מיקרואורגניזמים הניתנים לעיבוד בקלות. לדוגמה, ה-iChips המותאם באיור 2 הכיל 28 מושבות בסך הכל, ומספר המושבות שגדלו על לוחות אגר לאחר תת-תרבות היה 16, בעוד ש-12 לא שרדו את ההעברה. ניתן לזהות מבודדים ולהשתמש בהם למחקר נוסף לאחר תרבית על צלחות אגר בגודל מלא, כפי שמוצג באיור 4.
ניסוי iChip מותאם מוצלח אמור לגרום לבידוד של מיקרואורגניזמים השונים ממיקרואורגניזמים ידועים ברמת המין. מידת הדמיון של כל מבודד למיקרואורגניזמים ידועים נמצאת על ידי השוואת ה-DNA המבודד לגנומים של מיקרואורגניזמים ידועים. ציוני הטטרה Z של המיקרואורגניזמים הקרובים ביותר לכל מבודד מתארים את אחוז הדמיון. איור 5 מראה שמתוך שנים עשר החיידקים שבודדו, ארבעה היו מינים חדשים ומעלה, שניים היו ככל הנראה זנים חדשים, ושישה היו מיקרואורגניזמים ידועים. ההתאוששות של מספר מינים או זנים חדשים עולה בקנה אחד עם ניסויי iChip שפורסמו בעבר, מה שמצביע על כך שמבנה iChip שונה זה אינו מעכב את הגידול בהתאוששות של מינים חדשים11.
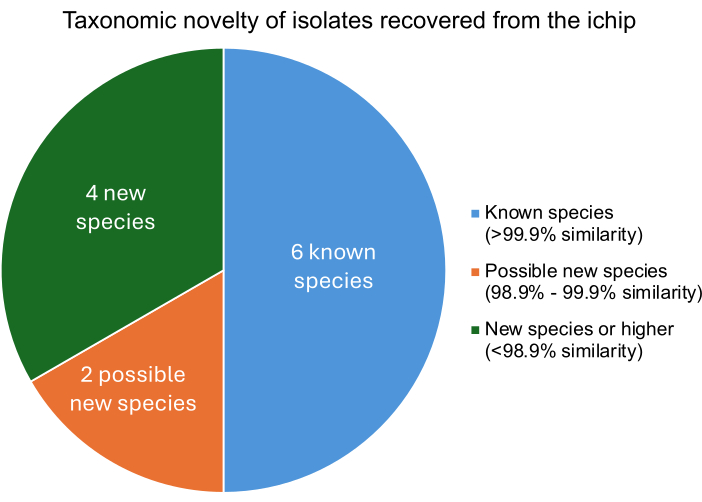
איור 5: רמת הסיווג הטקסונומית של מבודדים ששוחזרו מניסוי iChip. אחוז הדמיון נקבע על סמך ציוני טטרה Z, המתארים את הדמיון של המבודדים לחיידקים ידועים כאשר דמיון של >99.9% מצביע על התאמה בין מינים, דמיון של 98.9%-99.9% מצביע על מין חדש אפשרי, ודמיון של <98.9% מצביע על מין חדש ומעלה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
שיטות רבות, כגון כריית גנום ובחינת מסלולים ביוסינתטיים שקטים, הקלו על גילוי כימיקלים ביו-אקטיביים חדשים בשנים האחרונות21,22. עם זאת, NPs שהתגלו בשיטות כאלה מציגים לעתים קרובות דמיון מבני גבוה לתרכובות ידועות. השגת גישה למיקרואורגניזמים שלא תורבתו בעבר תפתח מגוון כימי גדול יותר ו-NPs עם דרכי פעולה חדשות שיכולות לסייע טוב יותר במאבק בעמידות מיקרוביאלית. הוכח כי טכניקת בידוד iChip מגבירה את יכולת הטיפוח של מיקרואורגניזמים חדשים, שניתן להשתמש בהם לבניית ספריות של מיקרואורגניזמים לגילוי NPs חדשים.
ההמשגה המוקדמת ביותר של iChip פורסמה בשנת 2002, שהורכבה מטבעות מתכת בודדות המכילות תערובת תאי אגר אטומה בממברנות חדירות למחצה ומודגרת באתרה23. מאוחר יותר הוא חזר על עצמו ב-2010 כדי להכיל שבב קטן עם בארות רבות11, ואחריו פותח iChip זול שנבנה מחומרי מעבדה נפוצים בפרסום משפיע של Nature Protocols ב-20179. מספר שינויים נעשו בפרוטוקול Nature בפרסום זה כדי לשפר את המעשיות וקלות השימוש בו. פרוטוקול Nature כולל הדבקת קרום חדיר למחצה לשני צידי השבב בהשוואה לשיטה זו, המשתמשת בכיסוי צלחת דבק בצד אחד. בעיות עלולות להתעורר בעת שימוש בדבק סיליקון לחיבור הממברנה לאחר טעינת ה-iChip. למיטב ידיעתנו, כל דבקי הסיליקון הלא רעילים פולטים חומצה אצטית כשהם מתייצבים, מה שעלול להשפיע על כדאיות התא24. הפרוטוקול המתואר בסרטון זה גם מפחית משמעותית את כמות הטיפול הנדרשת לאחר החיסון על ידי שימוש בכיסוי לוחית ה-PCR כדי לאטום את הצלחת הממולאה, מה שמבטיח עוד יותר סטריליות ומפחית את זמן ההגדרה. בהתבסס על זיהוי של מספר מבודדים חדשים מניסוי זה, השימוש רק בממברנה חדירה למחצה אחת אכן מעכב את העלייה בשיעורי גילוי אורגניזמים חדשים שדווחו עבור iChips שנבנו עם שני ממברנות חדירות למחצה. עם זאת, יידרשו ניסויים נוספים עם גודל מדגם גדול יותר כדי לכמת את ההשפעה.
שינוי שיטה נוסף הוא רק תת-תרבות מה-iChips ששונו המכילים גידול בפחות מ-25% מהבארות. אם מושבות גדלות ברוב הבארות של צלחת, סביר להניח שיש כמה בארות המכילות מיקרואורגניזמים מרובים, גם אם לא ניתן לראות אותם בקלות. במהלך פיתוח השיטה נמצא כי במידה רבה בלתי אפשרי להשיג תרביות אקסניות בעת תת-תרבות מבארות המכילות יותר ממושבה אחת. הבידוד של תרביות שאינן אקסניות מציג בעיות משמעותיות במורד הזרם מבחינת בדיקות וזיהוי ביו-אקטיביות. לפיכך, לשם הפשטות, מומלץ לתת-תרבית רק iChips מותאמים עם מושבות הגדלות בחלק מהבארות.
הנושא המשמעותי ביותר שיכול להתעורר בשיטות iChip הוא זיהום בארות או iChips שלמים במיקרואורגניזמים אחרים. זיהום מסומן על ידי מושבות הגדלות בבארות הבקרה, או אותו מיקרואורגניזם הגדל על פני מספר בארות או אזור שלם של ה-iChip המותאם. מקור הזיהום יכול לנבוע מעיקור לקוי של חומרים במהלך ההקמה, או טכניקה אספטית לא נכונה. במקרים כאלה, ודא שכל החומרים המשמשים הם חיטוי או אתנול מעוקרים כפי שמצוין בשיטה, וודא שלא מתרחש מגע בין פריטים לא סטריליים לבין ה-iChip ששונה מלבד תערובת תאי האגר. אם נצפתה צמיחת יתר של מיקרואורגניזם בודד בתחתית או בחלק העליון של מספר בארות iChip ששונו, סביר להניח שזו תוצאה של איטום לא שלם בין הבארות לממברנה. במקרה זה, ודא שהדבק המשמש הוא 100% סיליקון, שאינו מתכלה באתנול, וודא שהממברנה החדירה למחצה וכיסוי צלחת ה-PCR אטומים לחלוטין סביב כל באר במהלך הבנייה.
סדרת הדילול המשמשת בפרוטוקול הנוכחי אמורה לספק דילול הולם לרוב סוגי הקרקע עם תקופת אחסון של פחות משבוע מכיוון שהיא כוללת טווח של פי אלף של דילול. עם זאת, יכולה להיות שונות משמעותית בספירת המיקרואורגניזמים בין סוגי האדמה. אם בארות הבקרה אינן מכילות צמיחה אך מושבות מרובות צומחות בכל באר מכל ארבעת ה-iChips שעברו שינוי, ריכוזי התאים שבהם השתמשו לא היו נמוכים מספיק. יש לשנות את סדרת הדילול כך שתגיע לריכוזי תאים נמוכים יותר בתערובות המדיה התאית המשמשות להגדרת ה-iChips ששונו. באופן דומה, אם לא נצפתה צמיחה באף אחת מהצלחות הללו, יש להגדיל את הריכוזים המשמשים בתערובות המדיה התאית. לחלופין, יתכן שהטמפרטורה של המדיה בה נעשה שימוש הייתה גבוהה מדי להישרדותם של מיקרואורגניזמים. במקרה כזה, יש לאפשר למדיה להתקרר ככל האפשר מבלי להתמצק לפני הוספתה לתרחיף התא המדולל.
הטכנולוגיה היא התקדמות חשובה לקראת התגברות על האנומליה הגדולה של ספירת הלוחות. עם זאת, הוא עדיין מוגבל על ידי חוסר ההתאמה של טכניקות תרבות קונבנציונליות, כפי שמעיד מספר המיקרואורגניזמים שאינם שורדים את המעבר מה-iChip השונה ללוחות אגר קונבנציונליים. פרסומים קודמים דיווחו כי סבבים מרובים של תת-תרבות ודגירה ב-iChips מגדילים עוד יותר את יכולת הטיפוח של המיקרואורגניזמים. זמן הביות הממושך והחשיפה לגורמי צמיחת קרקע בזמן האגר ב-iChip מגדיל את הסיכוי של מושבה לגדול על אגר בלבד. עם זאת, גישה זו לא דווחה כמניבה סבירות גבוהה יותר למיקרואורגניזמים חדשים מאשר דגירה בודדת של iChip25,26.
טקטיקות רבות אחרות נחקרות ומשתנות כדי להגביר את יכולת הגידול של מיקרואורגניזמים חדשים. לדוגמא, הכנת מכשיר iChip שלם מחומר חדיר למחצה הוצעה כדי להקל על תרבות משותפת של מיקרואורגניזמים בבארות שכנות27. עם זאת, יתרון הבנייה המתואר בפרסום זה הוא העלות הנמוכה שלה כאשר עלות בניית לוח אחד שווה לכ-12 דולר (4 דולר לצלחת 96 בארות, 8 דולר לממברנה, 2 דולר לכיסוי PCR). יתר על כן, המבנה הפשוט שלו הופך אותו לכלי לא מסובך בשימוש כמתואר ומספק אפשרויות רבות להתאמה אישית. למרות שפרוטוקול זה משתמש במדיום סלקטיבי עבור חיידקים, ניתן תיאורטית לכוון את מערך הניסוי כך שיתמקד באוכלוסיית חיידקים רצויה על ידי שינוי המדיום בו נעשה שימוש, כגון שימוש במדיה מדכאת חיידקים כדי למקד פטריות, או אגר דל חומרים מזינים עבור מיקרואורגניזמים נבגים.
Disclosures
המחברים הצהירו שאין להם ניגודי אינטרסים.
Acknowledgements
אנו אסירי תודה על המימון הבסיסי J (J-001757, J-001842) שניתן באמצעות החקלאות והחקלאות הקנדית, שאפשר את הפרויקט הזה. אנו מודים לברט ואן היינינגן על צילום תוכן הווידאו עבור פרוטוקול זה. אנו גם רוצים להודות ל-Ron Matters על איסוף דגימות אדמה ששימשו בניסויים המתוארים בפרסום זה.
Materials
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
References
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved