Method Article
Isolamento de microrganismos do solo usando a tecnologia iChip
Neste Artigo
Resumo
A técnica iChip faz uso de um dispositivo de isolamento in-situ simples e barato que aumenta as taxas de descoberta de novos microrganismos do solo. Novos microrganismos podem ser usados para estudos adicionais relativos ao microbioma do solo ou descoberta de produtos naturais, entre outras aplicações.
Resumo
A técnica de isolamento iChip usa um dispositivo de isolamento in-situ que aumenta a capacidade de cultivo de microrganismos anteriormente não cultiváveis. Os microrganismos são uma fonte importante de novos produtos químicos e moléculas potencialmente bioativas. No entanto, apenas 1% dos microrganismos ambientais podem ser cultivados usando métodos laboratoriais convencionais. Com o aumento da resistência antimicrobiana, encontrar novos medicamentos para combater infecções e doenças é de suma importância, e um método crítico para encontrar novas moléculas é a descoberta de novos microrganismos. Ao incubar colônias de microrganismos do solo nos poços de uma placa de 96 poços, selados com uma membrana semipermeável e incubados no topo do solo, os micróbios estão em contato com a água e os fatores de crescimento do solo, permitindo o isolamento de novos micróbios em um ambiente de laboratório. Após um período de domesticação em um iChip, os microrganismos podem ser subcultivados em meios convencionais e usados para estudos posteriores. Este dispositivo é valioso para a descoberta de moléculas bioativas e pesquisa do microbioma do solo e já foi usado anteriormente em ambas as aplicações.
Introdução
As bactérias ambientais são uma rica fonte de produtos naturais (NPs)1. Esses metabólitos não são vitais para a sobrevivência dos microrganismos, mas são produzidos para facilitar sua colonização, superando outros microrganismos em seu entorno2. A evolução ajustou as estruturas químicas e as atividades dos NPs, tornando-os agentes antimicrobianos eficazes. Um exemplo é a daptomicina, um antibiótico aprovado pelo FDA em 20033. Nos últimos anos, a resistência microbiana aumentou em ocorrência e gravidade, e novos medicamentos como a daptomicina são tratamentos de última linha contra infecções que desenvolveram resistência a antibióticos mais antigos4. O desenvolvimento de novos antibióticos e outros medicamentos é essencial para manter as infecções e doenças comuns tratáveis.
Embora os NPs muitas vezes sejam bons medicamentos, a indústria farmacêutica voltou-se para métodos sintéticos para a descoberta de medicamentos após a era de ouro dos antibióticos nas décadas de 1950 a 19605. Na década de 1970, os mesmos microrganismos e metabólitos antimicrobianos continuaram a ser reisolados repetidamente, com cada vez menos novos candidatos a medicamentos sendo descobertos 1,6. O microbioma do solo contém grande diversidade microbiana, mas apenas um pequeno número de microrganismos pode ser isolado em condições convencionais de laboratório. A grande maioria dos microrganismos do solo detectados pela genômica não são observados quando cultivados usando métodos convencionais de cultura, levando a que a questão seja cunhada como "a grande anomalia da contagem de placas"7. Esses micróbios não cultiváveis foram chamados de matéria escura microbiana, pois são conhecidos, mas não podem ser estudados in vitro até que possam ser isolados como culturas puras. Esses microrganismos não cultiváveis provavelmente podem produzir uma grande variedade de candidatos a medicamentos, o que é de grande importância na era da resistência microbiana.
A técnica iChip é um método que pode ser usado para aumentar a recuperação de novos microrganismos do ambiente 8,9. Essa tecnologia ajuda a simular o ambiente natural das bactérias durante a incubação, aumentando assim a capacidade de cultivo de microrganismos que, de outra forma, não sobreviveriam às condições convencionaisde laboratório 10. Os iChips modificados já foram desenvolvidos e usados para isolar microrganismos de muitas fontes diferentes, como solo, sedimentos, ambientes marinhos e intestinos de animais 11,12,13,14,15,16. Talvez o caso mais impactante do uso dessa tecnologia tenha sido pela NovoBiotic Pharmaceuticals, onde uma nova bactéria, Eleftheria terrae, foi descoberta17. Descobriu-se que esse microrganismo produz Teixobactina, um antibiótico de uma nova classe, que inibe várias espécies bacterianas resistentes pertinentes à saúde humana sem detectar o desenvolvimento de resistência 5,18. Esta foi uma descoberta impactante, pois a teixobactina é a primeira nova classe de antibióticos descoberta em décadas e é um sinal de que essa técnica é uma rota promissora para descobertas de novos medicamentos e superação da grande anomalia da contagem de placas19. Aqui, é apresentado um iChip modificado com base em uma publicação anterior de Berdy et al., que foi otimizado para facilidade de uso e prevenção de contaminação9.
Protocolo
1. Preparação e esterilização de meios
- Prepare e esterilize o meio de sais mínimos de succinato (SMS), que consiste em 0,1 g de amido de batata, 1 g de casaminoácidos, 0,125 g de digestão de caseína e 15 g de ágar bacteriológico ou técnico em 1 L de água.
- Esterilize em autoclave o meio e 100 mL de água de grau molecular usando um ciclo líquido de 20 min, 121 °C.
NOTA: Certifique-se de que todos os outros itens que entrarão em contato com a mistura de células de ágar sejam adquiridos estéreis, incluindo pontas de pipeta e tubos de centrífuga, caso contrário, autoclave-os também.
2. Construção modificada do iChip
- Remova o fundo dos poços em quatro placas de 96 poços usando uma ferramenta de punção de ágar de 5 mm.
- Corte as membranas de policarbonato de 0,05 μm em retângulos de 7,6 cm x 11 cm, ou igual às dimensões da parte inferior da placa de 96 poços.
- Usando um selante de silicone, cole membranas de policarbonato de 0,05 μm no fundo das placas de 96 poços, garantindo que o adesivo vede os poços, mas não cubra totalmente as aberturas dos poços. Deixe secar por pelo menos 24 h ou seguindo as instruções do adesivo.
NOTA: O selante deve ser à prova d'água, não tóxico e 100% silicone. A maioria dos selantes de aquário funcionará bem.
3. Preparação de suspensões celulares
- Rotule quatro tubos de centrífuga de 15 mL AD e quatro tubos de centrífuga de 50 mL E-H e adicione 4.5 mL de água estéril a cada um.
- Meça 1 g de solo em um tubo de centrífuga de 50 mL, adicione 10 mL de água estéril e vortex por 10 min.
- Deixe a suspensão do solo repousar por 10 min.
- Pipetar 0,5 ml do sobrenadante contendo células do solo para o tubo A e misturar bem.
- Adicione 0,5 mL da suspensão celular no tubo A ao tubo B e misture bem. Transfira 0,5 ml da solução B para o tubo C e misture bem. Repita isso para todos os tubos de centrífuga restantes, completando uma série de diluições de 10 vezes nos oito tubos de centrífuga.
- Remova 0,5 mL do tubo H para ter o mesmo volume em todos os tubos.
NOTA: Essas concentrações são otimizadas para solos locais. Ao tentar este procedimento pela primeira vez, efectue uma gama maior de diluições para encontrar a concentração de amostra adequada.
4. Inoculação iChip modificada
- Mergulhe completamente os iChips em etanol a 95% por pelo menos 15 min.
- Retire as placas do etanol e coloque-as sobre papel toalha estéril. Enquanto permite que o etanol evapore, ligue o esterilizador UV na capela de fluxo laminar por 15 min para esterilizá-los ainda mais.
- Pipete 360 μL de meio SMS estéril na primeira coluna da placa para atuar como poços de controle.
- Adicionar 45 ml de SMS arrefecido a 50 °C à suspensão celular no tubo E e misturar bem para combinar a suspensão celular e o ágar.
- Pipetar 360 μL da mistura de ágar-célula da etapa 4.4 em todos os outros poços da placa de 96 poços.
NOTA: Uma pipeta multicanal é recomendada, pois o ágar-ágar solidifica rapidamente. Não é aconselhável reaquecer a mistura de células de ágar uma vez misturada devido ao risco de matar os microrganismos. - Assim que a mídia estiver definida, sele a parte superior das placas com uma tampa de placa PCR.
- Repita as etapas 3.3 a 3.6 com tubos FH para preencher um total de quatro iChips com diferenças de dez vezes nas concentrações.
NOTA: A mídia pode ser colocada no micro-ondas por 30 s com uma tampa solta quando necessário ou mantida em banho-maria a 60 °C entre as placas para garantir que permaneça derretida para cada uma.
5. Incubação
- Coloque as placas em uma caixa contendo aproximadamente uma polegada do solo usado na etapa 3.2, com a membrana voltada para baixo e coloque a cobertura.
- Incubar as placas no recipiente coberto, num local escuro a 25 °C.
NOTA: Após uma semana, examine o conteúdo dos iChips modificados para garantir que não ocorreu contaminação durante a configuração. A contaminação é indicada pelo crescimento excessivo de um ou poucos microrganismos em todos os poços de uma placa. - Após 6 semanas, examine os iChips. Use as placas contendo crescimento em menos de 25% dos poços para isolamento de colônias.
6. Isolamento de colônias
- Enxágue os iChips modificados 3x com água estéril para remover todas as partículas de sujeira.
- Limpe a parte superior e as laterais da placa com etanol a 95%, evitando o lado com a membrana semipermeável.
- Usando uma lâmina estéril, corte a tampa da placa ao redor de um poço contendo uma colônia.
- Usando uma ferramenta de estrias estéreis, perfure a colônia e listre em ágar SMS em placas de 100 x 15 mm usando o método de quatro quadrantes.
- Repita para todas as outras colônias nos iChips que estão crescendo uma colônia por poço.
NOTA: Para colônias minúsculas, a placa pode ser colocada de volta no solo e incubada até ficar grande o suficiente para subcultura, desde que a cobertura da placa não tenha sido removida ao redor do poço. - Incubar as placas SMS a 25 °C e monitorizar o crescimento.
- Examine as colônias para garantir que as culturas axênicas tenham sido isoladas.
7. Identificação de microrganismos
- Sequenciar os genomas dos isolados recuperados20.
- Avalie os genomas usando JSpeciesWS ou outra plataforma de identificação baseada em genoma para determinar o grau de semelhança de cada isolado com microrganismos conhecidos no GenomesDB.
- Categorize os isolados abaixo dos limites aceitos pela comunidade para identificação de espécies como supostamente novos.
Resultados
Uma visão geral do protocolo é mostrada na Figura 1. Um experimento iChip modificado bem-sucedido resulta em pelo menos uma placa contendo crescimento em menos de 25% dos poços, sem que ocorra crescimento nos poços de controle. Esse número de poços com crescimento geralmente garante que colônias únicas sejam isoladas em poços. Além disso, o número de colônias, ou o número de poços contendo colônias, deve diminuir dez vezes com cada placa subsequente preparada a partir da série de diluições. Os resultados representativos são mostrados na Figura 2. onde as placas 7 e 8 são iChips bem-sucedidos, pois contêm crescimento em menos de 25% dos poços, e a porcentagem de poços contendo colônias diminui a cada placa, conforme mostrado na Tabela 1. Se todas as placas em um teste contiverem mais de uma colônia por poço ou nenhuma colônia em nenhum poço, isso constitui um resultado negativo.
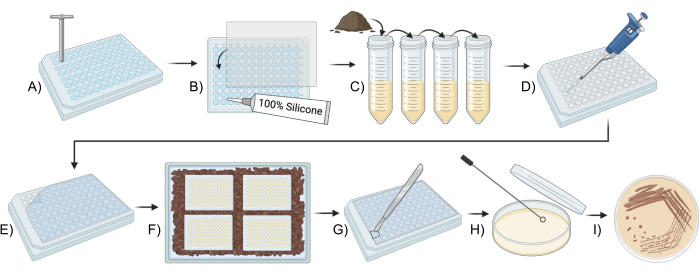
Figura 1: Visão geral esquemática do uso de um iChip modificado para cultivar micróbios do solo. (A,B) Construção da placa (Etapa 2). (C) Preparação do inóculo (Etapa 3). (D,E) Configuração do iChip (Etapa 4). (F) Incubação do iChip (Etapa 5). (G-I) Transferência dos isolados para meios convencionais (Etapa 6). Clique aqui para ver uma versão maior desta figura.
| Ichip | Concentração de solo em suspensão de ágar | Poços com colônias | Unidades Formadoras de Colônias |
| E | 5,0 x 10-6 g/mL | Todo | Muitos para contar |
| F | 5,0 x 10-7 g/mL | Todo | Muitos para contar |
| G | 5,0 x 10-8 g/mL | 22 | 22 |
| H | 5,0 x 10-9 g/mL | 6 | 6 |
Tabela 1: Dados representativos de um experimento iChip bem-sucedido em que o número de poços contendo colônias diminui a cada diluição em série.
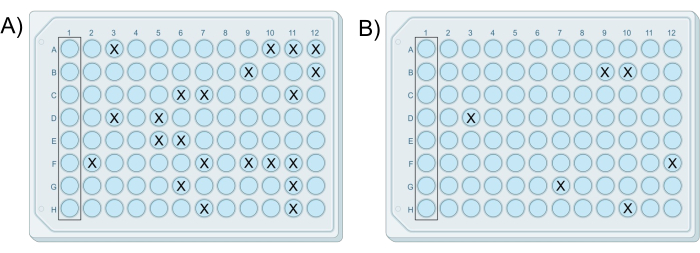
Figura 2: Resultados exemplares de dois iChips modificados com sucesso. Os iChips foram do mesmo experimento em que (A) a placa preparada a partir da mistura célula-ágar G contém 22 colônias e (B) a placa preparada a partir da mistura célula-ágar H contém seis colônias, consistente com a diferença de diluição de 10 vezes entre as misturas. Clique aqui para ver uma versão maior desta figura.
Pode-se visualizar colônias a olho nu ou microscopia, como mostrado na Figura 3A-G. As colônias podem estar crescendo na superfície do ágar, como visto na Figura 3B ou embutidas no ágar, como a colônia na Figura 3F. Pode haver casos em que existem duas colônias no mesmo poço em um iChip modificado bem-sucedido, como na Figura 3G. Em alguns casos, as colônias estão distantes o suficiente para que possam ser perfuradas com uma agulha separadamente e subcultivadas; no entanto, é improvável que aqueles na Figura 3G possam ser facilmente cultivados como microrganismos axênicos. Exemplos de colônias transferidas do iChip para mídia SMS convencional são mostrados na Figura 4.
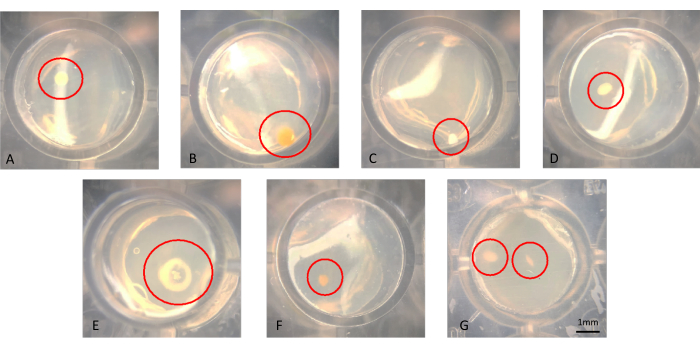
Figura 3: Visão microscópica representativa das colônias crescendo em um iChip após 6 semanas de incubação. (AF) Colônias únicas são observadas, que devem ser subcultivadas, e em (G) duas colônias são observadas em estreita proximidade, que são difíceis de obter como culturas axênicas. Clique aqui para ver uma versão maior desta figura.
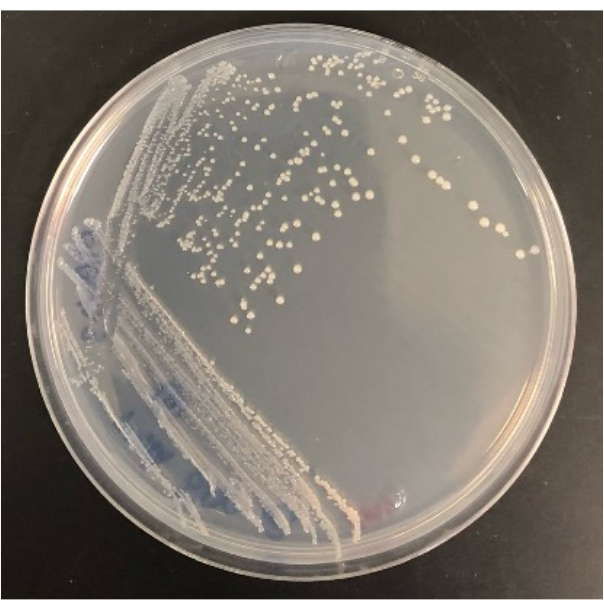
Figura 4: Resultado representativo de uma bactéria isolada do iChip modificado uma vez crescendo em uma placa de ágar SMS. Clique aqui para ver uma versão maior desta figura.
A capacidade de isolar colônias do iChip modificado em placas de ágar convencionais também determina o sucesso do experimento. Nem todas as colônias que crescem no iChip modificado sobreviverão à transferência para placas convencionais. Em um iChip, as colônias são expostas a fatores de crescimento e nutrientes do solo, aumentando sua capacidade de cultivo. Para alguns microrganismos, um período de domesticação não é suficiente para torná-los cultiváveis em condições convencionais. A taxa de recuperação de colônias de um iChip para placas varia de experimento para experimento, mas algumas devem crescer em placas, pois esse método também cultiva microrganismos facilmente cultiváveis. Por exemplo, os iChips modificados na Figura 2 continham 28 colônias no total, e o número de colônias que cresceram em placas de ágar após a subcultura foi de 16, enquanto 12 não sobreviveram à transferência. Os isolados podem ser identificados e usados para estudos posteriores, uma vez cultivados em placas de ágar de tamanho normal, conforme mostrado na Figura 4.
Um experimento iChip modificado bem-sucedido deve resultar no isolamento de microrganismos que diferem de microrganismos conhecidos no nível da espécie. O grau de similaridade de cada isolado com microrganismos conhecidos é encontrado comparando-se o DNA isolado com os genomas de microrganismos conhecidos. Os escores tetra Z dos microrganismos relacionados mais próximos de cada isolado descrevem a porcentagem de similaridade. A Figura 5 mostra que das doze bactérias isoladas, quatro eram novas espécies ou mais, duas provavelmente eram novas cepas e seis eram microrganismos conhecidos. A recuperação de várias novas espécies ou cepas é consistente com experimentos iChip publicados anteriormente, indicando que esta construção modificada do iChip não impede o aumento na recuperação de novas espécies11.
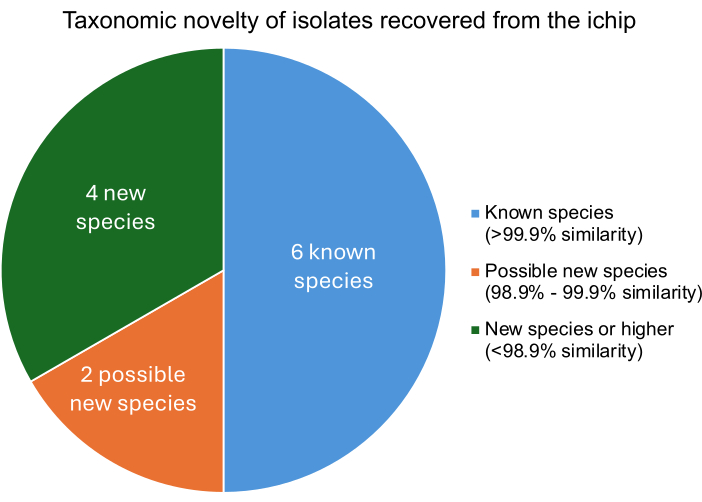
Figura 5: O nível de classificação taxonômica dos isolados recuperados de um experimento iChip. A similaridade percentual foi determinada com base nos escores tetra Z, que descrevem a semelhança dos isolados com bactérias conhecidas, onde >99,9% de similaridade indica uma correspondência de espécie, 98,9%-99,9% de similaridade indica uma possível nova espécie e <98,9% de similaridade indica uma nova espécie ou superior. Clique aqui para ver uma versão maior desta figura.
Discussão
Numerosos métodos, como mineração de genoma e exame de vias biossintéticas silenciosas, facilitaram a descoberta de novos produtos químicos bioativos nos últimos anos21,22. No entanto, NPs descobertos usando tais métodos geralmente exibem alta semelhança estrutural com compostos conhecidos. Obter acesso a microrganismos anteriormente não cultivados desbloqueará maior diversidade química e NPs com novos modos de ação que podem ajudar melhor no combate à resistência microbiana. A técnica de isolamento iChip demonstrou aumentar a cultivabilidade de novos microrganismos, que podem ser usados para construir bibliotecas de microrganismos para a descoberta de novos NPs.
A conceituação mais antiga de um iChip foi publicada em 2002, que consistia em anéis metálicos únicos contendo uma mistura de células de ágar selada com membranas semipermeáveis e incubada in situ23. Mais tarde, foi iterado em 2010 para conter um pequeno chip com muitos poços11, seguido pelo desenvolvimento de um iChip barato construído a partir de materiais comuns de laboratório em uma influente publicação da Nature Protocols em 20179. Várias modificações foram feitas no protocolo da Nature nesta publicação para melhorar sua praticidade e facilidade de uso. O protocolo Nature envolve a colagem de uma membrana semipermeável em ambos os lados de um chip em comparação com esse método, que usa uma tampa de placa adesiva em um lado. Podem surgir problemas ao usar cola de silicone para prender a membrana após o carregamento do iChip. Até onde sabemos, todos os adesivos de silicone não tóxicos emitem ácido acético à medida que endurecem, o que pode afetar a viabilidade celular24. O protocolo descrito neste vídeo também reduz significativamente a quantidade de manuseio necessária após a inoculação, usando a tampa da placa PCR para selar a placa cheia, garantindo ainda mais a esterilidade e reduzindo o tempo de configuração. Com base na identificação de vários novos isolados deste experimento, o uso de apenas uma única membrana semipermeável inibe o aumento nas taxas de descoberta de novos organismos que foi relatado para iChips construídos com duas membranas semipermeáveis. No entanto, mais experimentos com um tamanho de amostra maior seriam necessários para quantificar o impacto.
Outra modificação do método é apenas subcultura dos iChips modificados que contêm crescimento em menos de 25% dos poços. Se as colônias crescem na maioria dos poços de uma placa, provavelmente existem alguns poços que contêm vários microorganismos, mesmo que não sejam facilmente visíveis. Durante o desenvolvimento do método, verificou-se que era praticamente impossível obter culturas axênicas ao subcultivar de poços contendo mais de uma colônia. O isolamento de culturas não axênicas apresenta problemas significativos a jusante em termos de ensaios e identificação de bioatividade. Assim, para simplificar, apenas iChips modificados com colônias crescendo em uma parte dos poços são recomendados para serem subcultivados.
O problema mais significativo que pode surgir com os métodos iChip é a contaminação de poços ou iChips inteiros com outros microrganismos. A contaminação é indicada por colônias que crescem nos poços de controle, ou o mesmo microrganismo que cresce em vários poços ou em uma área inteira do iChip modificado. A fonte de contaminação pode ser devido à esterilização inadequada dos materiais durante a configuração ou técnica asséptica inadequada. Nesses casos, certifique-se de que todos os materiais usados sejam esterilizados em autoclave ou etanol conforme indicado no método e certifique-se de que não ocorra contato entre quaisquer itens não estéreis e o iChip modificado que não seja a mistura de células de ágar. Se um crescimento excessivo de um único microrganismo for observado na parte inferior ou superior de vários poços iChip modificados, é mais provável que seja o resultado de uma vedação incompleta entre os poços e a membrana. Nesse caso, certifique-se de que o adesivo usado seja 100% silicone, que não se degrada em etanol, e certifique-se de que a membrana semipermeável e a cobertura da placa de PCR estejam completamente vedadas ao redor de cada poço durante a construção.
A série de diluições utilizada no protocolo atual deve proporcionar uma diluição adequada para a maioria dos tipos de solo com um período de armazenamento inferior a uma semana, uma vez que inclui uma gama de diluições mil vezes. No entanto, pode haver variação significativa na contagem de microrganismos entre os tipos de solo. Se os poços de controle não contiverem crescimento, mas várias colônias estiverem crescendo em cada poço de todos os quatro iChips modificados, as concentrações de células usadas não foram baixas o suficiente. A série de diluições deve ser modificada para atingir concentrações celulares mais baixas nas misturas de meios celulares utilizadas para configurar os iChips modificados. Do mesmo modo, se não se observar qualquer crescimento em qualquer uma destas placas, as concentrações utilizadas nas misturas células-meios devem ser aumentadas. Alternativamente, é possível que a temperatura do meio utilizado tenha sido muito alta para a sobrevivência dos microrganismos. Nesse caso, o meio deve esfriar o máximo possível sem solidificar antes de ser adicionado à suspensão de células diluídas.
A tecnologia é um avanço importante para superar a grande anomalia da contagem de placas. No entanto, ainda é limitado pela inadequação das técnicas convencionais de cultivo, conforme indicado pelo número de microrganismos que não sobrevivem à transferência do iChip modificado para placas de ágar convencionais. Publicações anteriores relataram que várias rodadas de subcultura e incubação em iChips aumentam ainda mais a cultivabilidade dos microrganismos. O tempo de domesticação prolongado e a exposição a fatores de crescimento do solo durante o uso de ágar no iChip aumentam as chances de uma colônia crescer apenas em ágar. No entanto, não foi relatado que essa abordagem produza uma probabilidade maior de novos microrganismos do que uma única incubação de iChip25,26.
Muitas outras táticas estão sendo exploradas e modificadas para aumentar a cultivabilidade de novos microrganismos. Por exemplo, a fabricação de um dispositivo iChip inteiro a partir de um material semipermeável foi proposta para facilitar a co-cultura de microrganismos em poços vizinhos27. Dito isto, uma vantagem da construção descrita nesta publicação é seu baixo custo, com o custo de construção de uma placa equivalendo a aproximadamente US$ 12 (US$ 4 por placa de 96 poços, US$ 8 por membrana, US$ 2 por cobertura de PCR). Além disso, sua construção simples o torna uma ferramenta descomplicada quando usada conforme descrito e oferece muitas possibilidades de personalização. Embora este protocolo use um meio seletivo para bactérias, a configuração experimental pode, teoricamente, ser ajustada para atingir uma população microbiana desejada, modificando o meio usado, como o uso de meios supressores de bactérias para atingir fungos ou ágar com baixo teor de nutrientes para esporulação de microrganismos.
Divulgações
Os autores declararam não ter conflitos de interesse.
Agradecimentos
Reconhecemos com gratidão o financiamento J-base (J-001757, J-001842) fornecido pela Agriculture and Agri-Food Canada, que tornou este projeto possível. Agradecemos a Brett van Heyningen por filmar o conteúdo do vídeo para este protocolo. Também gostaríamos de agradecer a Ron Matters pela coleta de amostras de solo usadas nos experimentos descritos nesta publicação.
Materiais
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
Referências
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados