Method Article
Isolamento dei microrganismi del suolo con la tecnologia iChip
In questo articolo
Riepilogo
La tecnica iChip si avvale di un dispositivo di isolamento in situ semplice ed economico che aumenta i tassi di scoperta di nuovi microrganismi dal suolo. Nuovi microrganismi possono essere utilizzati per ulteriori studi relativi al microbioma del suolo o alla scoperta di prodotti naturali, tra le altre applicazioni.
Abstract
La tecnica di isolamento iChip utilizza un dispositivo di isolamento in situ che aumenta la coltivabilità di microrganismi precedentemente non coltivabili. I microrganismi sono un'importante fonte di nuove sostanze chimiche e molecole potenzialmente bioattive. Tuttavia, solo l'1% dei microrganismi ambientali può essere coltivato con metodi di laboratorio convenzionali. Con l'aumento della resistenza antimicrobica, trovare nuovi farmaci per combattere infezioni e malattie è di primaria importanza, e un metodo fondamentale per trovare nuove molecole è la scoperta di nuovi microrganismi. Incubando colonie di microrganismi del suolo nei pozzetti di una piastra da 96 pozzetti, sigillata con una membrana semipermeabile e incubata sopra il suolo, i microbi sono in contatto con l'acqua e i fattori di crescita del suolo, consentendo l'isolamento di nuovi microbi in un ambiente di laboratorio. Dopo un periodo di addomesticamento in un iChip, i microrganismi possono potenzialmente essere subcoltivati su terreni convenzionali e utilizzati per ulteriori studi. Questo dispositivo è prezioso per la scoperta di molecole bioattive e la ricerca sul microbioma del suolo ed è stato utilizzato in precedenza in entrambe le applicazioni.
Introduzione
I batteri ambientali sono una ricca fonte di prodotti naturali (NP)1. Questi metaboliti non sono vitali per la sopravvivenza dei microrganismi, ma sono invece prodotti per facilitare la loro colonizzazione superando gli altri microrganismi nel loro ambiente2. L'evoluzione ha messo a punto le strutture chimiche e le attività delle NP, rendendole efficaci agenti antimicrobici. Un esempio di questo tipo è la daptomicina, un antibiotico approvato dalla FDA nel 20033. Negli ultimi anni, la resistenza microbica è aumentata in termini di incidenza e gravità e nuovi farmaci come la daptomicina rappresentano trattamenti di ultima linea contro le infezioni che hanno sviluppato resistenza agli antibiotici più vecchi4. Lo sviluppo di nuovi antibiotici e altri farmaci è essenziale per mantenere curabili le infezioni e le malattie comuni.
Sebbene le NP siano spesso buone per i farmaci, l'industria farmaceutica si è rivolta a metodi sintetici per la scoperta di farmaci dopo l'età d'oro degli antibiotici negli anni '50 e '605. Negli anni '70, gli stessi microrganismi e metaboliti antimicrobici continuavano ad essere reisolati ripetutamente, con la scoperta di sempre meno nuovi farmaci candidati 1,6. Il microbioma del suolo contiene una grande diversità microbica, ma solo un piccolo numero di microrganismi può essere isolato in condizioni di laboratorio convenzionali. La stragrande maggioranza dei microrganismi del suolo rilevati dalla genomica non viene osservata quando coltivata con metodi di coltura convenzionali, il che ha portato a definire la questione "l'anomalia del grande conteggio delle piastre"7. Questi microbi incoltivabili sono stati chiamati materia oscura microbica in quanto sono noti per esistere, ma non possono essere studiati in vitro fino a quando non possono essere isolati come colture pure. Questi microrganismi non coltivabili possono probabilmente produrre una grande quantità di farmaci candidati, il che è di grande importanza nell'era della resistenza microbica.
La tecnica iChip è un metodo che può essere utilizzato per aumentare il recupero di nuovi microrganismi dall'ambiente 8,9. Questa tecnologia aiuta a simulare l'ambiente naturale dei batteri durante l'incubazione, aumentando così la coltivabilità di microrganismi che altrimenti non potrebbero sopravvivere alle condizioni di laboratorio convenzionali10. Gli iChip modificati sono già stati sviluppati e utilizzati per isolare microrganismi da molte fonti diverse, come il suolo, i sedimenti, gli ambienti marini e l'intestino degli animali 11,12,13,14,15,16. Forse il caso più impattante dell'uso di questa tecnologia è stato quello di NovoBiotic Pharmaceuticals, dove èstato scoperto un nuovo batterio, l'Eleftheria terrae. È stato scoperto che questo microrganismo produce Teixobactina, un antibiotico di una nuova classe, che inibisce diverse specie batteriche resistenti pertinenti alla salute umana senza rilevare lo sviluppo di resistenza 5,18. Questa è stata una scoperta di grande impatto in quanto la teixobactina è la prima nuova classe di antibiotici scoperta da decenni ed è un segno che questa tecnica è un percorso promettente per la scoperta di nuovi farmaci e il superamento dell'anomalia del grande numero di piastre19. In questo articolo, viene presentato un iChip modificato basato su una precedente pubblicazione di Berdy et al., ottimizzato per la facilità d'uso e la prevenzione della contaminazione9.
Protocollo
1. Preparazione dei terreni e sterilizzazione
- Preparare e sterilizzare il terreno di coltura con sali minimi di succinato (SMS), che consiste in 0,1 g di fecola di patate, 1 g di casamino acidi, 0,125 g di caseina digestato e 15 g di agar batteriologico o tecnico in 1 L di acqua.
- Sterilizzare in autoclave il terreno e 100 ml di acqua di grado molecolare utilizzando un ciclo liquido di 20 minuti, 121 °C.
NOTA: Assicurarsi che tutti gli altri elementi che entreranno in contatto con la miscela di cellule di agar siano acquistati sterili, compresi i puntali delle pipette e le provette da centrifuga, altrimenti sterilizzarli in autoclave.
2. Costruzione iChip modificata
- Rimuovere il fondo dei pozzetti in quattro piastre da 96 pozzetti utilizzando un punzone per agar da 5 mm.
- Tagliare membrane in policarbonato da 0,05 μm in rettangoli di 7,6 cm x 11 cm, o uguali alle dimensioni del fondo della piastra a 96 pozzetti.
- Utilizzando un sigillante siliconico, far aderire membrane in policarbonato da 0,05 μm sul fondo delle piastre a 96 pozzetti, assicurandosi che l'adesivo sigilli i pozzetti, ma non copra completamente le aperture dei pozzetti. Lasciare asciugare per almeno 24 ore o seguendo le istruzioni riportate sull'adesivo.
NOTA: Il sigillante deve essere impermeabile, atossico e 100% silicone. La maggior parte dei sigillanti per acquari funzionerà bene.
3. Preparazione delle sospensioni cellulari
- Etichettare quattro provette da centrifuga da 15 mL A-D e quattro provette da centrifuga da 50 mL E-H e aggiungere 4,5 mL di acqua sterile a ciascuna.
- Misurare 1 g di terreno in una provetta da centrifuga da 50 ml, aggiungere 10 ml di acqua sterile e agitare per 10 minuti.
- Lasciare riposare la sospensione del terreno per 10 min.
- Pipettare 0,5 mL di surnatante contenente cellule dal terreno nella provetta A e mescolare accuratamente.
- Aggiungere 0,5 mL della sospensione cellulare nella provetta A alla provetta B e mescolare accuratamente. Trasferire 0,5 mL di soluzione B nella provetta C e mescolare accuratamente. Ripetere l'operazione per tutte le provette da centrifuga rimanenti, completando una serie di diluizioni di 10 volte nelle otto provette da centrifuga.
- Rimuovere 0,5 mL dalla provetta H per avere un volume uguale in tutte le provette.
NOTA: Queste concentrazioni sono ottimizzate per i terreni locali. Quando si tenta questa procedura per la prima volta, eseguire una gamma più ampia di diluizioni per trovare la concentrazione del campione appropriata.
4. Inoculazione di iChip modificata
- Immergere completamente gli iChips in etanolo al 95% per almeno 15 minuti.
- Togliete le piastre dall'etanolo e mettetele su un tovagliolo di carta sterile. Lasciando evaporare l'etanolo, accendere lo sterilizzatore UV nella cappa a flusso laminare per 15 minuti per sterilizzarlo ulteriormente.
- Pipettare 360 μl di terreno SMS sterile nella prima colonna della piastra per fungere da pozzetti di controllo.
- Aggiungere 45 mL di SMS raffreddato a 50 °C alla sospensione cellulare nella provetta E e mescolare accuratamente per combinare la sospensione cellulare e l'agar.
- Pipettare 360 μl della miscela di cellule di agar della fase 4.4 in tutti gli altri pozzetti della piastra a 96 pozzetti.
NOTA: Si consiglia una pipetta multicanale poiché l'agar si solidifica rapidamente. Si sconsiglia di riscaldare la miscela di cellule di agar una volta miscelata a causa del rischio di uccidere i microrganismi. - Una volta che il terreno è solidificato, sigillare la parte superiore delle piastre con un coperchio per piastre PCR.
- Ripetere i passaggi da 3.3 a 3.6 con le provette F-H per riempire un totale di quattro iChip con differenze di concentrazione di dieci volte.
NOTA: Il supporto può essere cotto nel microonde per 30 s con un tappo allentato quando necessario o conservato in un bagno di acqua calda a 60 °C tra le piastre per garantire che rimanga fuso per ciascuna.
5. Incubazione
- Posizionare le piastre in una scatola contenente circa un pollice del terreno utilizzato nel passaggio 3.2, con la membrana rivolta verso il basso e posizionare il coperchio.
- Incubare le piastre nel contenitore coperto, in un luogo buio a 25 °C.
NOTA: Dopo una settimana, esaminare il contenuto degli iChip modificati per assicurarsi che non si siano verificate contaminazioni durante la configurazione. La contaminazione è indicata dalla crescita eccessiva di uno o pochi microrganismi in tutti i pozzetti di una piastra. - Dopo 6 settimane, esamina gli iChip. Utilizzare le piastre contenenti la crescita in meno del 25% dei pozzetti per l'isolamento delle colonie.
6. Isolamento delle colonie
- Sciacquare gli iChip modificati 3 volte con acqua sterile per rimuovere tutte le particelle di terreno.
- Pulire la parte superiore e i lati della piastra con etanolo al 95%, evitando il lato con la membrana semipermeabile.
- Usando una lama sterile, tagliare il coperchio della piastra attorno a un pozzetto contenente una colonia.
- Utilizzando uno strumento sterile per striature, forare la colonia e striare su agar SMS in piastre da 100 x 15 mm utilizzando il metodo a quattro quadranti.
- Ripeti per tutte le altre colonie negli iChip che stanno crescendo una colonia per pozzetto.
NOTA: Per le colonie minuscole, la piastra può essere riposizionata sul terreno e incubata fino a quando non è abbastanza grande da poter essere sottocoltivata, purché il coperchio della piastra non sia stato rimosso intorno al pozzetto. - Incubare le piastre SMS a 25 °C e monitorare la crescita.
- Esamina le colonie per assicurarti che le colture axeniche siano state isolate.
7. Identificazione dei microrganismi
- Sequenziare i genomi degli isolati recuperati20.
- Valutare i genomi utilizzando JSpeciesWS o un'altra piattaforma di identificazione basata sul genoma per determinare il grado di somiglianza di ciascun isolato con microrganismi noti in GenomesDB.
- Classificare gli isolati al di sotto delle soglie accettate dalla comunità per l'identificazione delle specie come putativamente nuovi.
Risultati
Una panoramica visiva del protocollo è mostrata nella Figura 1. Un esperimento di iChip modificato di successo ha come risultato che almeno una piastra contiene una crescita in meno del 25% dei pozzetti, senza che si verifichi alcuna crescita nei pozzetti di controllo. Questo numero di pozzi in crescita garantisce generalmente che le singole colonie siano isolate nei pozzi. Inoltre, il numero di colonie, o il numero di pozzetti contenenti colonie, dovrebbe diminuire di dieci volte con ogni successiva piastra preparata dalla serie di diluizioni. I risultati rappresentativi sono mostrati nella Figura 2. dove le piastre 7 e 8 hanno successo iChip in quanto contengono crescita in meno del 25% dei pozzetti e la percentuale di pozzetti contenenti colonie diminuisce con ciascuna piastra, come mostrato nella Tabella 1. Se tutte le piastre in una prova contengono più di una colonia per pozzetto o nessuna colonia in nessun pozzetto, ciò costituisce un risultato negativo.
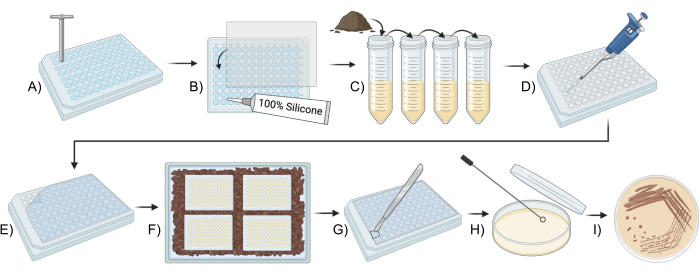
Figura 1: Panoramica schematica per l'utilizzo di un iChip modificato per la coltura di microbi del suolo. (A,B) Costruzione della piastra (Passaggio 2). (C) Preparazione dell'inoculo (Fase 3). (D,E) Configurazione dell'iChip (Passaggio 4). (F) Incubazione dell'iChip (Fase 5). (G-I) Trasferimento degli isolati in terreni convenzionali (Fase 6). Clicca qui per visualizzare una versione più grande di questa figura.
| Ichip | Concentrazione del suolo in sospensione di agar | Pozzi con colonie | Unità formanti colonie |
| E | 5,0 x 10-6 g/mL | Tutto | Troppi per essere contati |
| F | 5,0 x 10-7 g/mL | Tutto | Troppi per essere contati |
| G | 5,0 x 10-8 g/mL | 22 | 22 |
| H | 5,0 x 10-9 g/mL | 6 | 6 |
Tabella 1: Dati rappresentativi di un esperimento iChip di successo in cui il numero di pozzetti contenenti colonie diminuisce ad ogni diluizione in serie.
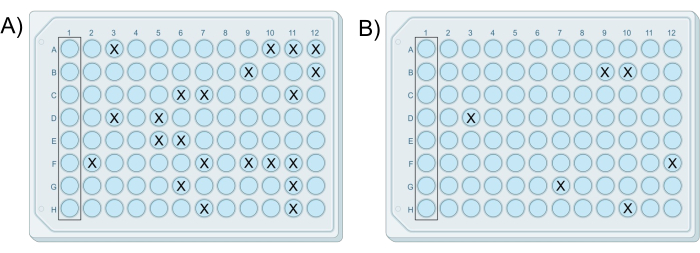
Figura 2: Risultati esemplificativi di due iChip modificati riusciti. Gli iChip provenivano dallo stesso esperimento in cui (A) la piastra preparata dalla miscela di cellule-agar G contiene 22 colonie e (B) la piastra preparata dalla miscela cellule-agar H contiene sei colonie, coerenti con la differenza di diluizione di 10 volte tra le miscele. Clicca qui per visualizzare una versione più grande di questa figura.
Si possono visualizzare le colonie ad occhio nudo o al microscopio, come mostrato nella Figura 3A-G. Le colonie possono crescere sulla superficie dell'agar, come si vede nella Figura 3B o incorporate nell'agar, come la colonia nella Figura 3F. Ci possono essere casi in cui ci sono due colonie nello stesso pozzetto in un iChip modificato riuscito, come nella Figura 3G. In alcuni casi, le colonie sono abbastanza distanti tra loro da poter essere forate con un ago separatamente e sottocoltivate; tuttavia, è improbabile che quelli nella Figura 3G possano essere facilmente coltivati come microrganismi axenici. Esempi di colonie trasferite dall'iChip su supporti SMS convenzionali sono mostrati nella Figura 4.
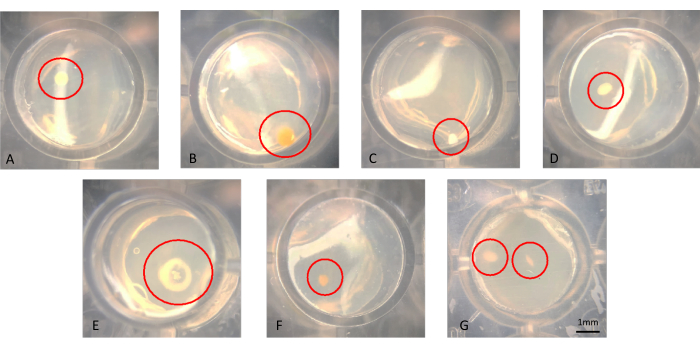
Figura 3: Vista microscopica rappresentativa delle colonie che crescono in un iChip dopo 6 settimane di incubazione. (A-F) Si osservano singole colonie, che dovrebbero essere sottocoltivate, e in (G) si osservano due colonie in stretta vicinanza, che sono difficili da ottenere come colture axeniche. Clicca qui per visualizzare una versione più grande di questa figura.
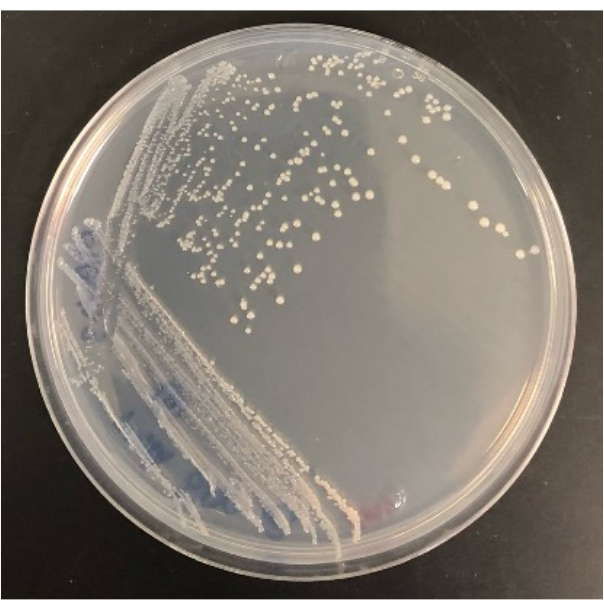
Figura 4: Risultato rappresentativo di un batterio isolato dall'iChip modificato una volta cresciuto su una piastra di agar SMS. Fare clic qui per visualizzare una versione più grande di questa figura.
Anche la capacità di isolare le colonie dall'iChip modificato su piastre di agar convenzionali determina il successo dell'esperimento. Non tutte le colonie che crescono nell'iChip modificato sopravviveranno al trasferimento su piastre convenzionali. In un iChip, le colonie sono esposte a fattori di crescita e nutrienti del suolo, aumentando la loro coltivabilità. Per alcuni microrganismi, un periodo di addomesticamento non è sufficiente per renderli coltivabili in condizioni convenzionali. Il tasso di recupero delle colonie da un iChip a piastre varia da esperimento a esperimento, ma alcune dovrebbero crescere in piastre poiché questo metodo coltiverà anche microrganismi facilmente coltivabili. Ad esempio, gli iChip modificati nella Figura 2 contenevano 28 colonie in totale e il numero di colonie che sono cresciute su piastre di agar dopo la sottocoltura è stato di 16, mentre 12 non sono sopravvissute al trasferimento. Gli isolati possono essere identificati e utilizzati per ulteriori studi una volta coltivati su piastre di agar di dimensioni standard, come mostrato nella Figura 4.
Un esperimento iChip modificato di successo dovrebbe portare all'isolamento di microrganismi che differiscono dai microrganismi noti a livello di specie. Il grado di somiglianza di ciascun isolato con microrganismi noti si trova confrontando il DNA isolato con i genomi di microrganismi noti. I punteggi tetra Z dei microrganismi imparentati più vicini a ciascun isolato descrivono la percentuale di somiglianza. La Figura 5 mostra che dei dodici batteri isolati, quattro erano specie nuove o superiori, due erano probabilmente nuovi ceppi e sei erano microrganismi noti. Il recupero di diverse nuove specie o ceppi è coerente con gli esperimenti iChip precedentemente pubblicati, indicando che questa costruzione iChip modificata non impedisce l'aumento del recupero di nuove specie11.
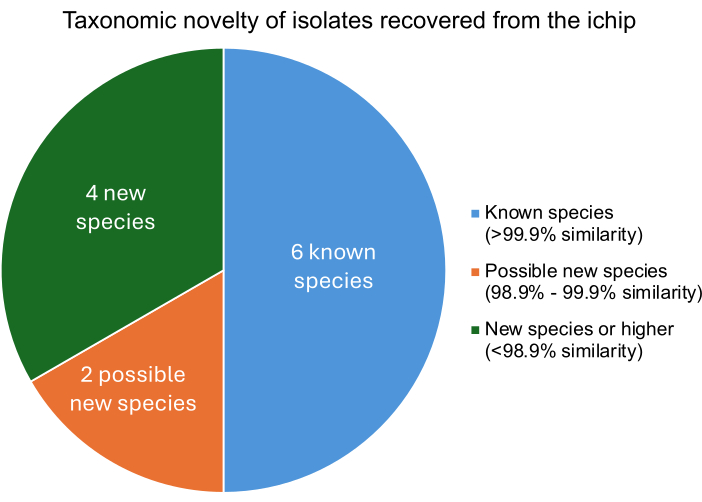
Figura 5: Il livello di classificazione tassonomica degli isolati recuperati da un esperimento iChip. La percentuale di somiglianza è stata determinata in base ai punteggi tetra Z, che descrivono la somiglianza degli isolati con i batteri noti, dove la somiglianza del >99,9% indica una corrispondenza di specie, la somiglianza del 98,9%-99,9% indica una possibile nuova specie e la somiglianza del <98,9% indica una nuova specie o superiore. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Numerosi metodi, come l'estrazione del genoma e l'esame di percorsi biosintetici silenti, hanno facilitato la scoperta di nuove sostanze chimiche bioattive negli ultimi anni21,22. Tuttavia, le NP scoperte utilizzando tali metodi mostrano spesso un'elevata somiglianza strutturale con composti noti. Ottenere l'accesso a microrganismi precedentemente non coltivati sbloccherà una maggiore diversità chimica e NP con nuove modalità d'azione che possono aiutare meglio a combattere la resistenza microbica. La tecnica di isolamento iChip ha dimostrato di aumentare la coltivabilità di nuovi microrganismi, che possono essere utilizzati per costruire librerie di microrganismi per la scoperta di nuove NP.
La prima concettualizzazione di un iChip è stata pubblicata nel 2002, che consisteva in singoli anelli metallici contenenti una miscela di celle di agar sigillate con membrane semipermeabili e incubate in situ23. Successivamente è stato iterato nel 2010 per contenere un piccolo chip con molti pozzetti11, seguito dallo sviluppo di un iChip economico costruito con materiali di laboratorio comuni in un'influente pubblicazione di Nature Protocols nel 20179. In questa pubblicazione sono state apportate diverse modifiche al protocollo Nature per migliorarne la praticità e la facilità d'uso. Il protocollo Nature prevede l'incollaggio di una membrana semipermeabile su entrambi i lati di un chip rispetto a questo metodo, che utilizza una copertura adesiva su un lato. Possono sorgere problemi quando si utilizza la colla siliconica per fissare la membrana dopo che l'iChip è stato caricato. Per quanto ne sappiamo, tutti gli adesivi siliconici non tossici emettono acido acetico durante la presa, il che può influire sulla vitalità cellulare24. Il protocollo descritto in questo video riduce anche significativamente la quantità di manipolazione necessaria dopo l'inoculazione utilizzando il coperchio della piastra PCR per sigillare la piastra riempita, garantendo ulteriormente la sterilità e riducendo il tempo di configurazione. Sulla base dell'identificazione di più nuovi isolati da questo esperimento, l'uso di una sola membrana semipermeabile inibisce l'aumento dei tassi di scoperta di nuovi organismi che è stato riportato per gli iChip costruiti con due membrane semipermeabili. Tuttavia, sarebbero necessari ulteriori esperimenti con un campione di dimensioni maggiori per quantificare l'impatto.
Un'altra modifica del metodo consiste nel sottocoltivare solo gli iChip modificati che contengono una crescita in meno del 25% dei pozzi. Se le colonie crescono nella maggior parte dei pozzetti di una piastra, è probabile che ci siano alcuni pozzetti che contengono più microrganismi, anche se non facilmente visibili. Durante lo sviluppo del metodo si è scoperto che era in gran parte impossibile ottenere colture axeniche quando si subcoltivava da pozzi contenenti più di una colonia. L'isolamento di colture non assoniche presenta problemi significativi a valle in termini di saggi di bioattività e identificazione. Pertanto, per semplicità, si consiglia di sottocoltivare solo gli iChip modificati con colonie che crescono in una parte dei pozzetti.
Il problema più significativo che può sorgere con i metodi iChip è la contaminazione di pozzetti o interi iChip con altri microrganismi. La contaminazione è indicata da colonie che crescono nei pozzetti di controllo o dallo stesso microrganismo che cresce in diversi pozzetti o in un'intera area dell'iChip modificato. La fonte di contaminazione potrebbe essere dovuta a una sterilizzazione inadeguata dei materiali durante la configurazione o a una tecnica asettica impropria. In tali casi, assicurarsi che tutti i materiali utilizzati siano sterilizzati in autoclave o etanolo come indicato nel metodo e assicurarsi che non si verifichi alcun contatto tra gli articoli non sterili e l'iChip modificato diverso dalla miscela di agar-cellule. Se si osserva una crescita eccessiva di un singolo microrganismo nella parte inferiore o superiore di più pozzetti iChip modificati, è molto probabilmente il risultato di una tenuta incompleta tra i pozzetti e la membrana. In questo caso, assicurarsi che l'adesivo utilizzato sia silicone al 100%, che non si degrada in etanolo, e assicurarsi che la membrana semipermeabile e il coperchio della piastra PCR siano completamente sigillati attorno a ciascun pozzetto durante la costruzione.
La serie di diluizioni utilizzata nell'attuale protocollo dovrebbe fornire una diluizione adeguata per la maggior parte dei tipi di terreno con un periodo di stoccaggio inferiore a una settimana, in quanto include un intervallo di diluizioni di mille volte. Tuttavia, ci possono essere variazioni significative nel numero di microrganismi tra i tipi di suolo. Se i pozzetti di controllo non contengono crescita ma crescono più colonie in ciascun pozzetto di tutti e quattro gli iChip modificati, le concentrazioni cellulari utilizzate non erano sufficientemente basse. La serie di diluizioni deve essere modificata per raggiungere concentrazioni cellulari più basse nelle miscele di terreni cellulari utilizzate per impostare gli iChip modificati. Analogamente, se non si osserva alcuna crescita in nessuna di queste piastre, le concentrazioni utilizzate nelle miscele di terreni cellulari devono essere aumentate. In alternativa, è possibile che la temperatura del mezzo utilizzato fosse troppo alta per la sopravvivenza dei microrganismi. In tal caso, il terreno deve essere lasciato raffreddare il più possibile senza solidificarsi prima di essere aggiunto alla sospensione cellulare diluita.
La tecnologia è un importante passo avanti verso il superamento dell'anomalia del grande numero di targhe. Tuttavia, è ancora limitato dall'inadeguatezza delle tecniche di coltura convenzionali, come indicato dal numero di microrganismi che non sopravvivono al trasferimento dall'iChip modificato alle piastre di agar convenzionali. Pubblicazioni precedenti hanno riportato che più cicli di sottocoltura e incubazione in iChips aumentano ulteriormente la coltivabilità dei microrganismi. Il tempo di addomesticamento prolungato e l'esposizione ai fattori di crescita del suolo durante l'assunzione di agar nell'iChip aumentano le possibilità di crescita di una colonia solo con agar. Tuttavia, non è stato riportato che questo approccio produca una maggiore probabilità di nuovi microrganismi rispetto a una singola incubazione di iChip 25,26.
Molte altre tattiche sono in fase di esplorazione e modifica per aumentare la coltivabilità di nuovi microrganismi. Ad esempio, è stato proposto di realizzare un intero dispositivo iChip con un materiale semipermeabile per facilitare la co-coltura di microrganismi nei pozzetti vicini27. Detto questo, un vantaggio della costruzione delineato in questa pubblicazione è il suo basso costo, con il costo di costruzione di una piastra pari a circa $ 12 ($ 4 per piastra da 96 pozzetti, $ 8 per membrana, $ 2 per copertura PCR). Inoltre, la sua costruzione semplice lo rende uno strumento semplice se utilizzato come descritto e offre molte possibilità di personalizzazione. Sebbene questo protocollo utilizzi un mezzo selettivo per i batteri, la configurazione sperimentale può teoricamente essere regolata per mirare a una popolazione microbica desiderata modificando il mezzo utilizzato, ad esempio utilizzando terreni che sopprimono i batteri per colpire i funghi o agar a basso contenuto di nutrienti per i microrganismi sporulanti.
Divulgazioni
Gli autori hanno dichiarato di non avere conflitti di interesse.
Riconoscimenti
Riconosciamo con gratitudine il finanziamento J-base (J-001757, J-001842) fornito attraverso Agriculture and Agri-Food Canada, che ha reso possibile questo progetto. Ringraziamo Brett van Heyningen per aver girato il contenuto video per questo protocollo. Vorremmo anche ringraziare Ron Matters per aver raccolto campioni di terreno utilizzati negli esperimenti descritti in questa pubblicazione.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
Riferimenti
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon