Method Article
Isolement des micro-organismes du sol à l’aide de la technologie iChip
Dans cet article
Résumé
La technique iChip utilise un dispositif d’isolement in situ simple et peu coûteux qui augmente les taux de découverte de nouveaux micro-organismes dans le sol. De nouveaux micro-organismes peuvent être utilisés pour des études plus approfondies relatives au microbiome du sol ou à la découverte de produits naturels, entre autres applications.
Résumé
La technique d’isolement iChip utilise un dispositif d’isolement in situ qui augmente la cultivabilité de micro-organismes auparavant incultivables. Les micro-organismes sont une source importante de nouvelles chimies et de molécules potentiellement bioactives. Cependant, seulement 1 % des micro-organismes environnementaux peuvent être cultivés à l’aide de méthodes de laboratoire conventionnelles. Avec l’augmentation de la résistance aux antimicrobiens, la découverte de nouveaux médicaments pour lutter contre les infections et les maladies est de la plus haute importance, et la découverte de nouveaux micro-organismes est une méthode essentielle pour trouver de nouvelles molécules. En incubant des colonies de micro-organismes du sol dans les puits d’une plaque de 96 puits, scellés par une membrane semi-perméable et incubés sur le sol, les microbes sont en contact avec l’eau et les facteurs de croissance du sol, ce qui permet d’isoler de nouveaux microbes en laboratoire. Après une période de domestication dans une iChip, les micro-organismes peuvent potentiellement être sous-cultivés sur des milieux conventionnels et utilisés pour une étude plus approfondie. Ce dispositif est précieux pour la découverte de molécules bioactives et la recherche sur le microbiome du sol et a déjà été utilisé dans les deux applications.
Introduction
Les bactéries environnementales sont une riche source de produits naturels (NP)1. Ces métabolites ne sont pas vitaux à la survie des micro-organismes, mais sont plutôt produits pour faciliter leur colonisation en supplantant les autres micro-organismes de leur environnement2. L’évolution a affiné les structures chimiques et les activités des NP, ce qui en fait des agents antimicrobiens efficaces. C’est le cas de la daptomycine, un antibiotique approuvé par la FDA en 20033. Au cours des dernières années, l’apparition et la gravité de la résistance microbienne ont augmenté, et de nouveaux médicaments comme la daptomycine constituent des traitements de dernière intention contre les infections qui ont développé une résistance aux antibiotiques plusanciens4. La mise au point de nouveaux antibiotiques et d’autres médicaments est essentielle pour que les infections et les maladies courantes puissent être traitées.
Bien que les NP soient souvent de bonnes pistes de médicaments, l’industrie pharmaceutique s’est tournée vers les méthodes synthétiques pour la découverte de médicaments après l’âge d’or des antibiotiques dans les années 1950 à 19605. Dans les années 1970, les mêmes micro-organismes et métabolites antimicrobiens ont continué à être réisolés à plusieurs reprises, et de moins en moins de nouveaux médicaments candidats ont été découverts 1,6. Le microbiome du sol contient une grande diversité microbienne, mais seul un petit nombre de micro-organismes peuvent être isolés dans des conditions de laboratoire conventionnelles. La grande majorité des micro-organismes du sol détectés par la génomique ne sont pas observés lorsqu’ils sont cultivés à l’aide de méthodes de culture conventionnelles, ce qui a conduit à l’invention de la « grande anomalie du comptage sur plaque »7. Ces microbes incultivables ont été appelés matière noire microbienne car ils sont connus, mais ne peuvent pas être étudiés in vitro tant qu’ils ne peuvent pas être isolés en tant que cultures pures. Ces micro-organismes incultivables peuvent probablement produire une multitude de candidats médicaments, ce qui est d’une grande importance à l’ère de la résistance microbienne.
La technique iChip est une méthode qui peut être utilisée pour augmenter la récupération de nouveaux micro-organismes de l’environnement 8,9. Cette technologie permet de simuler l’environnement naturel des bactéries pendant l’incubation, augmentant ainsi la cultivabilité des micro-organismes qui, autrement, ne pourraient pas survivre dans les conditions de laboratoire conventionnelles10. Des iChips modifiées ont déjà été développées et utilisées pour isoler des micro-organismes provenant de nombreuses sources différentes, telles que le sol, les sédiments, les environnements marins et les intestins d’animaux 11,12,13,14,15,16. Le cas le plus marquant de l’utilisation de cette technologie a peut-être été celui de NovoBiotic Pharmaceuticals, où une nouvelle bactérie, Eleftheria terrae, a été découverte17. On a constaté que ce micro-organisme produisait de la teixobactine, un antibiotique d’une nouvelle classe, qui inhibe plusieurs espèces bactériennes résistantes pertinentes pour la santé humaine sans développement de résistance détecté 5,18. Il s’agit d’une découverte percutante, car la teixobactine est la première nouvelle classe d’antibiotiques découverte depuis des décennies, et c’est un signe que cette technique est une voie prometteuse pour la découverte de nouveaux médicaments et le dépassement de la grande anomalie de numérationsur plaque 19. Ici, une iChip modifiée basée sur une publication précédente de Berdy et al. est présentée, qui a été optimisée pour la facilité d’utilisation et la prévention de la contamination9.
Protocole
1. Préparation et stérilisation des milieux
- Préparez et stérilisez un milieu de sels minimaux de succinate (SMS), qui se compose de 0,1 g de fécule de pomme de terre, 1 g d’acides casaminés, 0,125 g de digestion de caséine et 15 g de gélose bactériologique ou technique dans 1 L d’eau.
- Stérilisez à l’autoclave le milieu et 100 ml d’eau de qualité moléculaire en utilisant un cycle liquide de 20 minutes à 121 °C.
REMARQUE : Assurez-vous que tous les autres articles qui entreront en contact avec le mélange agar-cellule sont achetés stériles, y compris les pointes de pipette et les tubes à centrifuger, sinon autoclavez-les également.
2. Construction iChip modifiée
- Retirez le fond des puits en quatre plaques de 96 puits à l’aide d’un poinçonneur de gélose de 5 mm.
- Coupez des membranes en polycarbonate de 0,05 μm en rectangles de 7,6 cm x 11 cm, soit égaux aux dimensions du fond de la plaque à 96 puits.
- À l’aide d’un mastic silicone, collez des membranes en polycarbonate de 0,05 μm au fond des plaques de 96 puits, en veillant à ce que l’adhésif scelle les puits, mais ne recouvre pas entièrement les ouvertures des puits. Laissez sécher pendant au moins 24 h ou en suivant les instructions sur l’adhésif.
REMARQUE : Le scellant doit être imperméable, non toxique et 100 % silicone. La plupart des scellants d’aquarium fonctionneront bien.
3. Préparation des suspensions cellulaires
- Étiquetez quatre tubes à centrifuger de 15 mL A-D et quatre tubes à centrifuger de 50 mL E-H et ajoutez 4,5 mL d’eau stérile à chacun.
- Mesurez 1 g de sol dans un tube à centrifuger de 50 ml, ajoutez 10 ml d’eau stérile et agitez pendant 10 minutes.
- Laissez la suspension du sol reposer pendant 10 min.
- Pipeter 0,5 mL de cellules contenant du surnageant du sol dans le tube A et bien mélanger.
- Ajouter 0,5 mL de suspension cellulaire dans le tube A dans le tube B et bien mélanger. Transvasez 0,5 mL de solution B dans le tube C et mélangez soigneusement. Répétez cette opération pour tous les tubes de centrifugation restants, en complétant une série de dilutions 10 fois sur les huit tubes de centrifugation.
- Retirer 0,5 mL du tube H pour avoir un volume égal dans tous les tubes.
REMARQUE : Ces concentrations sont optimisées pour les sols locaux. Lorsque vous tentez cette procédure pour la première fois, effectuez une plus grande gamme de dilutions pour trouver la concentration appropriée de l’échantillon.
4. Inoculation iChip modifiée
- Immergez complètement les iChips dans de l’éthanol à 95 % pendant au moins 15 min.
- Retirez les plaques de l’éthanol et placez-les sur une serviette en papier stérile. Tout en laissant l’éthanol s’évaporer, allumez le stérilisateur UV dans la hotte à flux laminaire pendant 15 minutes pour les stériliser davantage.
- Pipette 360 μL de milieu SMS stérile dans la première colonne de la plaque pour servir de puits de contrôle.
- Ajouter 45 mL de SMS refroidi à 50 °C dans la suspension cellulaire du tube E et bien mélanger pour combiner la suspension cellulaire et la gélose.
- Pipeter 360 μL du mélange agar-cellule de l’étape 4.4 dans tous les autres puits de la plaque à 96 puits.
REMARQUE : Une pipette multicanaux est recommandée car la gélose se solidifie rapidement. Il n’est pas conseillé de réchauffer le mélange agar-cellule une fois mélangé en raison du risque de tuer les micro-organismes. - Une fois le support pris, scellez le haut des plaques avec un couvercle de plaque PCR.
- Répétez les étapes 3.3 à 3.6 avec les tubes F-H pour remplir un total de quatre iChips avec des différences de concentrations dix fois supérieures.
REMARQUE : Le support peut être passé au micro-ondes pendant 30 secondes avec un capuchon desserré si nécessaire ou conservé dans un bain d’eau chaude à 60 °C entre les plaques pour s’assurer qu’il reste fondu pour chacune.
5. Incubation
- Placez les plaques dans une boîte contenant environ un pouce de terre utilisée à l’étape 3.2, côté membrane vers le bas et placez le couvercle.
- Incuber les plaques dans le récipient couvert, dans un endroit sombre à 25 °C.
REMARQUE : Après une semaine, examinez le contenu des iChips modifiés pour vous assurer qu’aucune contamination ne s’est produite lors de la configuration. La contamination est indiquée par la prolifération d’un seul ou de quelques micro-organismes dans tous les puits d’une plaque. - Après 6 semaines, examinez les iChips. Utilisez les plaques contenant la croissance dans moins de 25 % des puits pour isoler la colonie.
6. Isolement de la colonie
- Rincez l’iChips 3x modifié avec de l’eau stérile pour éliminer toutes les particules de saleté.
- Essuyez le haut et les côtés de la plaque avec de l’éthanol à 95 %, en évitant le côté avec la membrane semi-perméable.
- À l’aide d’une lame stérile, coupez le couvercle de la plaque autour d’un puits contenant une colonie.
- À l’aide d’un outil de stries stériles, percer la colonie et les traînées sur la gélose SMS en plaques de 100 x 15 mm en utilisant la méthode des quatre quadrants.
- Répétez l’opération pour toutes les autres colonies dans les iChips qui cultivent une colonie par puits.
REMARQUE : Pour les colonies minuscules, la plaque peut être remise sur le sol et incubée jusqu’à ce qu’elle soit suffisamment grande pour la sous-culture, à condition que le couvercle de la plaque n’ait pas été enlevé autour du puits. - Incuber les plaques SMS à 25 °C et surveiller la croissance.
- Examinez les colonies pour vous assurer que les cultures axéniques ont été isolées.
7. Identification des micro-organismes
- Séquencer les génomes des isolats récupérés20.
- Évaluez les génomes à l’aide de JSpeciesWS ou d’une autre plateforme d’identification basée sur le génome pour déterminer le degré de similitude de chaque isolat avec des micro-organismes connus dans GenomesDB.
- Catégoriser les isolats en dessous des seuils acceptés par la communauté pour l’identification des espèces comme étant présumés nouveaux.
Résultats
La figure 1 présente une vue d’ensemble visuelle du protocole. Une expérience iChip modifiée réussie aboutit à ce qu’au moins une plaque contienne de la croissance dans moins de 25 % des puits, sans croissance dans les puits témoins. Ce nombre de puits avec croissance garantit généralement que les colonies individuelles sont isolées dans les puits. De plus, le nombre de colonies, ou le nombre de puits contenant des colonies, devrait être décuplé à chaque plaque suivante préparée à partir de la série de dilutions. Des résultats représentatifs sont présentés à la figure 2. où les plaques 7 et 8 sont des iChips réussies car elles contiennent de la croissance dans moins de 25 % des puits, et le pourcentage de puits contenant des colonies diminue avec chaque plaque, comme le montre le tableau 1. Si toutes les plaques d’un essai contiennent plus d’une colonie par puits ou aucune colonie dans un puits, cela constitue un résultat négatif.
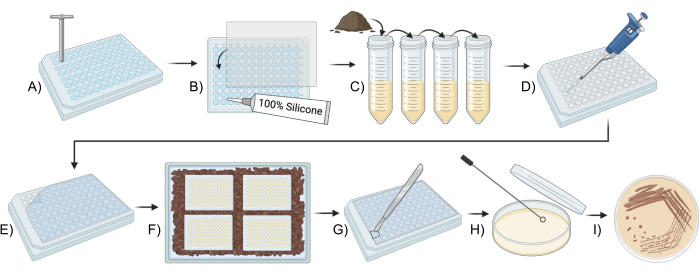
Figure 1 : Vue d’ensemble schématique de l’utilisation d’une iChip modifiée pour la culture des microbes du sol. (A,B) Construction de la plaque (étape 2). (C) Préparation de l’inoculum (étape 3). (D,E) Configuration de l’iChip (étape 4). f) Incubation de l’iChip (étape 5). (G-I) Transfert des isolats dans un milieu conventionnel (étape 6). Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Ichip | Concentration du sol en suspension de gélose | Puits avec colonies | Unités formant colonies |
| E | 5,0 x 10-6 g/mL | Tout | Trop nombreux pour être comptés |
| F | 5,0 x 10-7 g/mL | Tout | Trop nombreux pour être comptés |
| G | 5,0 x 10-8 g/mL | 22 | 22 |
| H | 5,0 x 10-9 g/mL | 6 | 6 |
Tableau 1 : Données représentatives d’une expérience iChip réussie où le nombre de puits contenant des colonies diminue à chaque dilution en série.
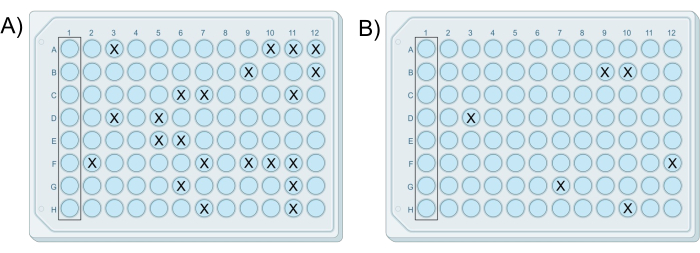
Figure 2 : Résultats exemplaires de deux iChips modifiées réussies. Les iChips provenaient de la même expérience dans laquelle (A) la plaque préparée à partir du mélange cellule-gélose G contient 22 colonies, et (B) la plaque préparée à partir du mélange cellule-gélose H contient six colonies, ce qui correspond à la différence de dilution de 10 fois entre les mélanges. Veuillez cliquer ici pour voir une version agrandie de cette figure.
On peut visualiser les colonies à l’œil nu ou au microscope, comme le montre la figure 3A-G. Les colonies peuvent se développer à la surface de la gélose, comme on le voit sur la figure 3B, ou intégrées dans la gélose, comme la colonie de la figure 3F. Il peut y avoir des cas où il y a deux colonies dans le même puits dans une iChip modifiée réussie, comme dans la figure 3G. Dans certains cas, les colonies sont suffisamment éloignées les unes des autres pour qu’elles puissent être percées avec une aiguille séparément et sous-cultivées ; cependant, il est peu probable que ceux de la figure 3G puissent être facilement cultivés en tant que microorganismes axéniques. La figure 4 montre des exemples de colonies transférées de l’iChip sur des supports SMS conventionnels.
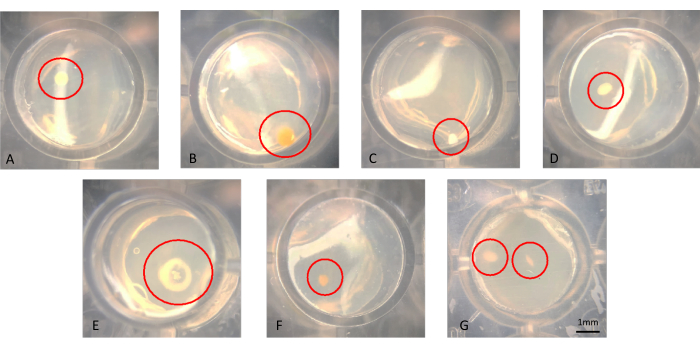
Figure 3 : Vue microscopique représentative des colonies qui se développent dans une iChip après 6 semaines d’incubation. (A-F) Des colonies uniques sont observées, à partir desquelles elles doivent être sous-cultivées, et dans (G) deux colonies sont observées à proximité, qui sont difficiles à obtenir en tant que cultures axéniques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
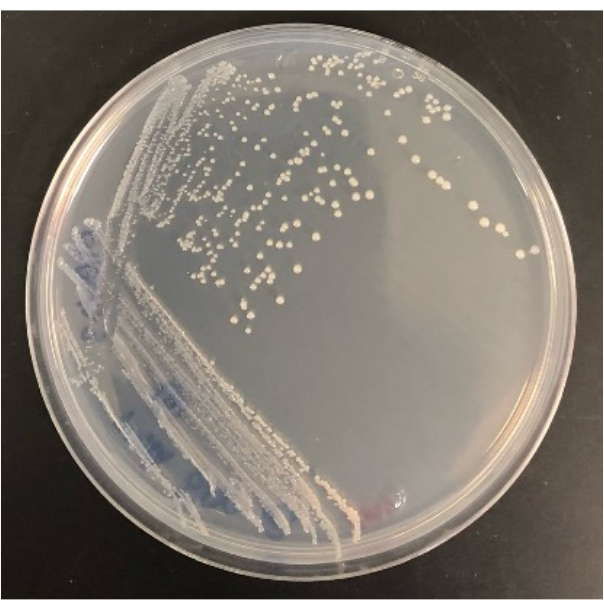
Figure 4 : Résultat représentatif d’une bactérie isolée de l’iChip modifiée une fois qu’elle s’est développée sur une plaque de gélose SMS. Veuillez cliquer ici pour voir une version agrandie de cette figure.
La capacité d’isoler les colonies de l’iChip modifiée sur des plaques de gélose conventionnelles détermine également le succès de l’expérience. Toutes les colonies qui poussent dans l’iChip modifiée ne survivront pas au transfert vers des plaques conventionnelles. Dans un iChip, les colonies sont exposées à des facteurs de croissance et à des nutriments du sol, ce qui augmente leur cultivabilité. Pour certains micro-organismes, une période de domestication n’est pas suffisante pour les rendre cultivables dans des conditions conventionnelles. Le taux de récupération des colonies d’une iChip aux plaques variera d’une expérience à l’autre, mais certaines devraient se développer dans des plaques car cette méthode permettra également de cultiver des micro-organismes facilement cultivables. Par exemple, les iChips modifiés de la figure 2 contenaient 28 colonies au total, et le nombre de colonies qui ont poussé sur des plaques de gélose après la sous-culture était de 16, tandis que 12 n’ont pas survécu au transfert. Les isolats peuvent être identifiés et utilisés pour une étude plus approfondie une fois cultivés sur des plaques de gélose de taille normale, comme le montre la figure 4.
Une expérience iChip modifiée réussie devrait permettre d’isoler des micro-organismes qui diffèrent des micro-organismes connus au niveau de l’espèce. Le degré de similitude de chaque isolat avec des micro-organismes connus est déterminé en comparant l’ADN isolé aux génomes de micro-organismes connus. Les scores tétra Z des micro-organismes les plus proches de chaque isolat décrivent le pourcentage de similitude. La figure 5 montre que sur les douze bactéries isolées, quatre étaient de nouvelles espèces ou d’une espèce supérieure, deux étaient probablement de nouvelles souches et six étaient des micro-organismes connus. Le rétablissement de plusieurs espèces ou souches nouvelles est conforme aux expériences iChip publiées précédemment, ce qui indique que cette construction modifiée de l’iChip n’entrave pas l’augmentation du rétablissement des espèces nouvelles11.
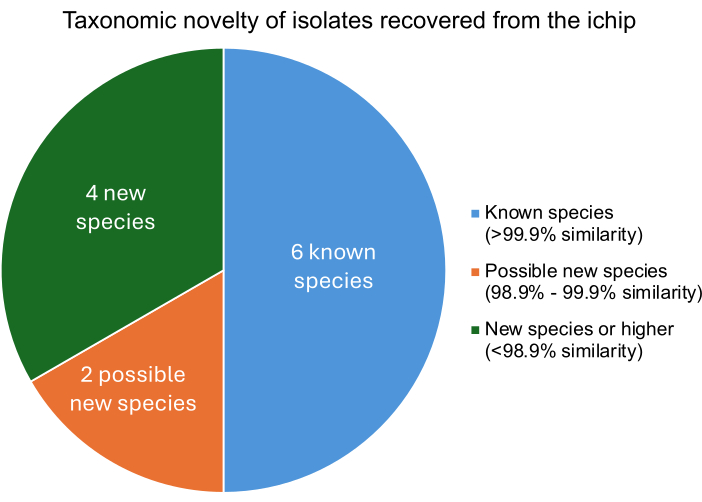
Figure 5 : Le niveau de classification taxonomique des isolats récupérés dans une expérience iChip. Le pourcentage de similitude a été déterminé à partir des scores tétra Z, qui décrivent la similitude des isolats avec des bactéries connues, où >99,9 % de similitude indique une correspondance d’espèce, 98,9 %-99,9 % de similitude indique une nouvelle espèce possible et <98,9 % de similitude indique une nouvelle espèce ou plus. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
De nombreuses méthodes, telles que l’exploration du génome et l’examen des voies de biosynthèse silencieuses, ont facilité la découverte de nouveaux produits chimiques bioactifs au cours des dernières années21,22. Cependant, les NP découverts à l’aide de ces méthodes présentent souvent une grande similitude structurelle avec des composés connus. L’accès à des micro-organismes auparavant non cultivés permettra d’obtenir une plus grande diversité chimique et des NP avec de nouveaux modes d’action qui peuvent mieux aider à lutter contre la résistance microbienne. Il a été démontré que la technique d’isolement iChip augmente la cultivabilité de nouveaux micro-organismes, qui peuvent être utilisés pour construire des bibliothèques de micro-organismes pour la découverte de nouvelles NP.
La première conceptualisation d’une iChip a été publiée en 2002, qui consistait en des anneaux métalliques uniques contenant un mélange de cellules de gélose scellées avec des membranes semi-perméables et incubées in situ23. Il a ensuite été itéré en 2010 pour contenir une petite puce avec de nombreux puits11, suivi par le développement d’une iChip peu coûteuse construite à partir de matériaux de laboratoire courants dans une publication influente de Nature Protocols en 20179. Plusieurs modifications ont été apportées au protocole Nature dans cette publication afin d’en améliorer l’aspect pratique et la facilité d’utilisation. Le protocole Nature consiste à coller une membrane semi-perméable des deux côtés d’une puce par rapport à cette méthode, qui utilise un couvercle de plaque adhésive sur un côté. Des problèmes peuvent survenir lors de l’utilisation de colle silicone pour fixer la membrane après le chargement de l’iChip. À notre connaissance, tous les adhésifs à base de silicone non toxiques émettent de l’acide acétique en se fixant, ce qui peut avoir un impact sur la viabilité cellulaire24. Le protocole décrit dans cette vidéo réduit également considérablement la quantité de manipulation nécessaire après l’inoculation en utilisant le couvercle de la plaque PCR pour sceller la plaque remplie, garantissant ainsi la stérilité et réduisant le temps de préparation. D’après l’identification de plusieurs nouveaux isolats dans le cadre de cette expérience, l’utilisation d’une seule membrane semi-perméable inhibe l’augmentation des taux de découverte de nouveaux organismes qui a été signalée pour les iChips construites avec deux membranes semi-perméables. Cependant, d’autres expériences avec un échantillon de plus grande taille seraient nécessaires pour quantifier l’impact.
Une autre modification de la méthode consiste à ne sous-cultiver que des iChips modifiés qui contiennent de la croissance dans moins de 25 % des puits. Si des colonies se développent dans la majorité des puits d’une plaque, il y a probablement des puits qui contiennent plusieurs micro-organismes, même s’ils ne sont pas facilement visibles. Au cours de la mise au point de la méthode, il a été constaté qu’il était en grande partie impossible d’obtenir des cultures axéniques lors de la sous-culture à partir de puits contenant plus d’une colonie. L’isolement des cultures non axéniques pose des problèmes importants en aval en termes de dosages de bioactivité et d’identification. Ainsi, pour simplifier, il est recommandé de ne sous-cultiver que des iChips modifiés avec des colonies poussant dans une partie des puits.
Le problème le plus important qui peut survenir avec les méthodes iChip est la contamination de puits ou d’iChips entiers par d’autres micro-organismes. La contamination est indiquée par des colonies qui poussent dans les puits témoins, ou par le même micro-organisme qui se développe dans plusieurs puits ou dans une zone entière de l’iChip modifié. La source de contamination pourrait être due à une stérilisation inadéquate des matériaux lors de la mise en place ou à une technique d’asepsie inappropriée. Dans de tels cas, assurez-vous que tous les matériaux utilisés sont stérilisés à l’autoclave ou à l’éthanol comme indiqué dans la méthode, et assurez-vous qu’aucun contact ne se produit entre les articles non stériles et l’iChip modifié autre que le mélange agar-cellule. Si une prolifération d’un seul micro-organisme est observée au fond ou au sommet de plusieurs puits iChip modifiés, c’est probablement le résultat d’une étanchéité incomplète entre les puits et la membrane. Dans ce cas, assurez-vous que l’adhésif utilisé est 100 % silicone, qui ne se dégrade pas dans l’éthanol, et assurez-vous que la membrane semi-perméable et le couvercle de la plaque PCR sont complètement scellés autour de chaque puits pendant la construction.
La série de dilutions utilisée dans le protocole actuel devrait fournir une dilution adéquate pour la plupart des types de sols avec une période d’entreposage de moins d’une semaine, car elle comprend une gamme de dilutions mille fois supérieure. Cependant, il peut y avoir des variations importantes dans le nombre de micro-organismes entre les types de sol. Si les puits témoins ne contiennent pas de croissance, mais que plusieurs colonies se développent dans chaque puits des quatre iChips modifiés, les concentrations cellulaires utilisées n’étaient pas assez faibles. La série de dilution doit être modifiée pour atteindre des concentrations cellulaires plus faibles dans les mélanges cellules-milieux utilisés pour configurer les iChips modifiés. De même, si aucune croissance n’est observée dans l’une de ces plaques, les concentrations utilisées dans les mélanges cellules-milieux doivent être augmentées. Alternativement, il est possible que la température du milieu utilisé ait été trop élevée pour la survie des micro-organismes. Dans ce cas, il faut laisser refroidir le milieu autant que possible sans se solidifier avant d’être ajouté à la suspension cellulaire diluée.
La technologie est une avancée importante pour surmonter la grande anomalie du comptage sur plaque. Cependant, elle reste limitée par l’inadéquation des techniques de culture conventionnelles, comme l’indique le nombre de micro-organismes qui ne survivent pas au transfert de l’iChip modifié aux plaques de gélose conventionnelles. Des publications antérieures ont rapporté que plusieurs cycles de sous-culture et d’incubation dans des iChips augmentent encore la cultivabilité des micro-organismes. Le temps de domestication prolongé et l’exposition aux facteurs de croissance du sol lors de l’utilisation de la gélose dans l’iChip augmentent les chances qu’une colonie se développe uniquement sur la gélose. Cependant, il n’a pas été signalé que cette approche produisait une probabilité plus élevée de micro-organismes nouveaux qu’une seule incubation d’iChip25,26.
De nombreuses autres tactiques sont explorées et modifiées pour augmenter la cultivabilité de nouveaux micro-organismes. Par exemple, la fabrication d’un dispositif iChip entier à partir d’un matériau semi-perméable a été proposée pour faciliter la co-culture de micro-organismes dans les puits voisins27. Cela dit, l’un des avantages de la construction décrits dans cette publication est son faible coût, le coût de construction d’une plaque équivalant à environ 12 $ (4 $ par plaque de 96 puits, 8 $ par membrane, 2 $ par couverture PCR). De plus, sa construction simple en fait un outil simple lorsqu’il est utilisé comme décrit et offre de nombreuses possibilités de personnalisation. Bien que ce protocole utilise un milieu sélectif pour les bactéries, le dispositif expérimental peut théoriquement être ajusté pour cibler une population microbienne souhaitée en modifiant le milieu utilisé, par exemple en utilisant des milieux suppresseurs de bactéries pour cibler les champignons, ou une gélose à faible teneur en nutriments pour les micro-organismes sporulants.
Déclarations de divulgation
Les auteurs ont déclaré n’avoir aucun conflit d’intérêts.
Remerciements
Nous sommes reconnaissants du financement des services J (J-001757, J-001842) fourni par Agriculture et Agroalimentaire Canada, qui a rendu ce projet possible. Nous remercions Brett van Heyningen d’avoir filmé le contenu vidéo de ce protocole. Nous tenons également à remercier Ron Matters d’avoir collecté des échantillons de sol utilisés dans les expériences décrites dans cette publication.
matériels
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
Références
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon