Method Article
iChip 기술을 사용한 토양 미생물 분리
요약
iChip 기술은 토양에서 새로운 미생물 발견 속도를 높이는 저렴하고 간단한 현장 격리 장치를 사용합니다. 새로운 미생물은 다른 응용 분야 중에서도 토양 미생물 군집 또는 천연 제품 발견과 관련된 추가 연구에 사용할 수 있습니다.
초록
iChip 분리 기술은 이전에는 배양할 수 없었던 미생물의 배양 가능성을 높이는 현장 분리 장치를 사용합니다. 미생물은 새로운 화학 물질과 잠재적으로 생체 활성 분자의 중요한 공급원입니다. 그러나 기존 실험실 방법으로는 환경 미생물의 1%만 배양할 수 있습니다. 항생제 내성이 증가함에 따라 감염 및 질병을 퇴치하기 위한 신약을 찾는 것이 가장 중요하며, 새로운 분자를 찾기 위한 중요한 방법은 새로운 미생물을 발견하는 것입니다. 반투과성 멤브레인으로 밀봉된 96웰 플레이트의 우물에서 토양 미생물 군체를 배양하고 토양 위에서 배양함으로써 미생물은 토양의 물 및 성장 인자와 접촉하여 실험실 환경에서 새로운 미생물을 격리할 수 있습니다. iChip에서 일정 기간 가축화 된 후, 미생물은 잠재적으로 기존 배지에 하대 배양되어 추가 연구에 사용될 수 있습니다. 이 장치는 생체 활성 분자 발견 및 토양 마이크로바이옴 연구에 유용하며 이전에 두 응용 분야 모두에서 사용되었습니다.
서문
환경 박테리아는 천연 제품(NP)의 풍부한 공급원입니다.1. 이러한 대사 산물은 미생물의 생존에 필수적이지 않으며, 대신 주변의 다른 미생물을 능가하여 미생물의 군집화를 촉진하기 위해 생성됩니다2. 진화는 NP의 화학 구조와 활동을 미세 조정하여 효과적인 항균제가 되었습니다. 이러한 예로는 2003 년 FDA에서 승인 한 항생제 인 daptomycin이 있습니다3. 최근 몇 년 동안 미생물 내성의 발생 빈도와 중증도가 증가했으며, 답토마이신(daptomycin)과 같은 신약은 오래된 항생제에 내성이 생긴 감염에 대한 최후의 치료제로 자리매김하고 있다4. 새로운 항생제 및 기타 약물의 개발은 일반적인 감염 및 질병을 치료할 수 있도록 하는 데 필수적입니다.
NP는 종종 좋은 약물 리드를 만들지만, 제약 산업은 1950년대에서 1960년대 사이의 항생제의 황금기 이후 약물 발견을 위해 합성 방법으로 전환했습니다5. 1970년대까지 동일한 미생물과 항균 대사 산물은 계속해서 반복적으로 재분리되었고, 발견되는 신약 후보 물질은 점점 더 적어졌습니다 1,6. 토양 마이크로바이옴에는 매우 다양한 미생물이 포함되어 있지만 기존 실험실 조건에서는 소수의 미생물만 분리할 수 있습니다. 유전체학에 의해 검출된 미생물의 토양 미생물 중 대다수는 기존의 배양 방법을 사용하여 배양할 때 관찰되지 않기 때문에 "대판수 변칙(the great plate count anomaly)"7이라는 문제가 생겨났습니다. 이 배양할 수 없는 미생물은 존재하는 것으로 알려져 있기 때문에 미생물 암흑 물질이라고 불렸지만 순수한 배양으로 분리될 때까지 체외에서 연구할 수 없습니다. 이러한 배양할 수 없는 미생물은 풍부한 약물 후보를 생산할 수 있으며, 이는 미생물 내성 시대에 매우 중요합니다.
iChip 기술은 환경에서 새로운 미생물의 회수를 증가시키는 데 사용할 수 있는 한 가지 방법입니다 8,9. 이 기술은 배양 중 박테리아의 자연 환경을 시뮬레이션하는 데 도움이 되며, 이를 통해 기존 실험실 조건에서 살아남을 수 없는 미생물의 배양 가능성을 높입니다10. 변형된 iChip은 이미 개발되어 토양, 퇴적물, 해양 환경 및 동물의 창자와 같은 다양한 출처에서 미생물을 분리하는 데 사용되었습니다 11,12,13,14,15,16. 이 기술을 사용한 가장 영향력 있는 사례는 NovoBiotic Pharmaceuticals에서 새로운 박테리아인 Eleftheria terrae를 발견한 사례일 것입니다17. 이 미생물은 새로운 종류의 항생제인 테익소박틴(Teixobactin)을 생산하는 것으로 밝혀졌는데, 이는 내성 발달을 감지하지 않고 인체 건강과 관련된 여러 내성 박테리아 종을 억제합니다 5,18. 테익소박틴(Teixobactin)은 수십 년 만에 발견된 최초의 새로운 종류의 항생제이기 때문에 이는 영향력 있는 발견이었으며, 이 기술이 새로운 약물 발견을 위한 유망한 경로이며 플레이트 수 이상현상 19을 극복하고 있다는 신호입니다. 여기에서는 Berdy et al.의 이전 간행물을 기반으로 한 수정된 iChip이 제시되며, 이는 사용 편의성 및 오염 방지를 위해 최적화되었습니다9.
프로토콜
1. 배지 준비 및 살균
- 물 1L에 감자 전분 0.1g, 카사미노산 1g, 카제인 다이제스트 0.125g, 세균 또는 기술 한천 15g으로 구성된 숙시네이트 최소 염 매체(SMS)를 준비하고 살균합니다.
- 20분, 121°C의 액체 사이클을 사용하여 배지와 100mL의 분자 등급 물을 오토클레이브 멸균합니다.
알림: 한천-세포 혼합물과 접촉할 다른 모든 품목(피펫 팁 및 원심분리기 튜브 포함)을 멸균 상태로 구입하고, 그렇지 않으면 오토클레이브해야 합니다.
2. 수정된 iChip 구조
- 96mm 한천 펀치 도구를 사용하여 5개의 웰 플레이트에서 웰 바닥을 제거합니다.
- 0.05μm 폴리카보네이트 멤브레인을 7.6cm x 11cm 직사각형 또는 96웰 플레이트 바닥 치수와 동일하게 자릅니다.
- 실리콘 실런트를 사용하여 0.05μm 폴리카보네이트 멤브레인을 96웰 플레이트의 바닥에 접착하여 접착제가 웰을 밀봉하지만 웰의 개구부를 완전히 덮지는 않도록 합니다. 최소 24시간 동안 건조시키거나 접착제의 지시에 따라 건조시키십시오.
알림: 실란트는 방수, 무독성 및 100% 실리콘이어야 합니다. 대부분의 수족관 실란트는 잘 작동합니다.
3. 세포 현탁액의 준비
- 4개의 15mL 원심분리 튜브 A-D와 4개의 50mL 원심분리 튜브 E-H에 라벨을 붙이고 각각에 4.5mL의 멸균수를 추가합니다.
- 50mL 원심분리기 튜브에 흙 1g을 넣고 멸균수 10mL를 넣고 10분 동안 소용돌이칩니다.
- 토양 현탁액을 10분 동안 가라앉힙니다.
- 토양의 세포를 포함하는 상등액 0.5mL를 튜브 A에 피펫팅하고 철저히 혼합합니다.
- 튜브 A의 세포 현탁액 0.5mL를 튜브 B에 넣고 완전히 혼합합니다. 용액 B 0.5mL를 튜브 C에 옮기고 완전히 혼합합니다. 나머지 모든 원심분리기 튜브에 대해 이 작업을 반복하여 8개의 원심분리기 튜브에 걸쳐 일련의 10배 희석을 완료합니다.
- 튜브 H에서 0.5mL를 제거하여 모든 튜브에서 동일한 부피를 유지합니다.
참고: 이러한 농도는 지역 토양에 최적화되어 있습니다. 이 절차를 처음 시도하는 경우 적절한 시료 농도를 찾기 위해 더 넓은 범위의 희석을 수행하십시오.
4. 변형 된 iChip 접종
- iChip을 95% 에탄올에 최소 15분 동안 완전히 담그십시오.
- 에탄올에서 플레이트를 제거하고 멸균 종이 타월에 올려 놓습니다. 에탄올이 증발하도록 하는 동안 층류 후드의 UV 살균기를 15분 동안 켜서 추가로 살균합니다.
- 360 μL의 멸균 SMS 매체를 플레이트의 첫 번째 컬럼에 피펫팅하여 대조 웰 역할을 합니다.
- 50°C로 냉각된 SMS 45mL를 튜브 E의 세포 현탁액에 첨가하고 완전히 혼합하여 세포 현탁액과 한천을 결합합니다.
- 단계 4.4의 한천-세포 혼합물을 360μL를 96웰 플레이트의 다른 모든 웰로 피펫팅합니다.
참고: 한천이 빠르게 응고되므로 멀티채널 피펫을 사용하는 것이 좋습니다. 한 번 혼합된 한천-세포 혼합물은 미생물을 죽일 위험이 있으므로 다시 가열하지 않는 것이 좋습니다. - 미디어가 설정되면 PCR 플레이트 커버로 플레이트 상단을 밀봉합니다.
- F-H 튜브로 3.3-3.6단계를 반복하여 농도가 10배 차이나는 총 4개의 iChip을 채웁니다.
알림: 미디어는 필요할 때 느슨한 캡으로 30초 동안 전자레인지에 돌리거나 플레이트 사이의 60°C 뜨거운 수조에 보관하여 각각이 용융된 상태로 유지되도록 할 수 있습니다.
5. 배양
- 3.2단계에서 사용된 약 1인치의 흙이 들어 있는 상자에 멤브레인 면이 아래를 향하게 하여 플레이트를 놓고 덮개를 놓습니다.
- 뚜껑이 있는 용기에 담긴 플레이트를 25°C의 어두운 곳에서 배양합니다.
알림: 일주일 후 수정된 iChip의 내용물을 검사하여 설정 중 오염이 발생하지 않았는지 확인하십시오. 오염은 플레이트의 모든 웰에서 단일 또는 소수의 미생물이 과도하게 증식하는 것으로 표시됩니다. - 6 주 후에 iChip을 검사하십시오. 콜로니 격리를 위해 25% 미만의 웰에서 성장을 포함하는 플레이트를 사용하십시오.
6. 식민지 격리
- 개조된 iChips 3x를 멸균수로 헹구어 모든 토양 미립자를 제거합니다.
- 반투과성 멤브레인이 있는 면을 피하고 95% 에탄올로 플레이트의 상단과 측면을 닦습니다.
- 멸균 칼날을 사용하여 군체가 들어있는 하나의 우물 주위의 판 덮개를 자릅니다.
- 멸균 줄무늬 도구를 사용하여 4사분면 방법을 사용하여 100 x 15mm 플레이트의 SMS 한천에 집락과 줄무늬를 뚫습니다.
- 웰당 하나의 콜로니를 성장시키는 iChip의 다른 모든 콜로니에 대해 반복합니다.
참고: 작은 군집의 경우, 플레이트 덮개가 우물 주변에서 제거되지 않는 한 플레이트를 토양에 다시 놓고 하위 배양할 수 있을 만큼 충분히 커질 때까지 배양할 수 있습니다. - SMS 플레이트를 25°C에서 배양하고 성장을 모니터링합니다.
- 군체를 조사하여 도끼 배양이 격리되었는지 확인하십시오.
7. 미생물 동정
- 회수된 분리물의 게놈 염기서열분석 20.
- JSpeciesWS 또는 다른 게놈 기반 식별 플랫폼을 사용하여 게놈을 평가하여 GenomesDB의 알려진 미생물에 대한 각 분리물의 유사성 정도를 결정합니다.
- 종 식별을 위해 공동체가 허용하는 임계값 이하로 격리된 것을 새로운 것으로 추정되는 것으로 분류합니다.
결과
프로토콜의 시각적 개요는 그림 1에 나와 있습니다. 성공적인 변형 iChip 실험은 적어도 하나의 플레이트가 웰의 25% 미만에서 성장을 포함하며 제어 웰에서는 성장이 발생하지 않습니다. 성장이 있는 이 수의 우물은 일반적으로 단일 군체가 우물에서 격리되도록 합니다. 또한, 군체의 수 또는 군체를 포함하는 우물의 수는 일련의 희석액에서 준비된 각 후속 플레이트와 함께 10배로 감소해야 합니다. 대표적인 결과가 그림 2에 나와 있습니다. 여기서 플레이트 7과 8은 우물의 25% 미만에서 성장을 포함하기 때문에 성공적인 iChip이며, 콜로니를 포함하는 우물의 비율은 표 1 과 같이 각 플레이트마다 감소합니다. 임상시험의 모든 플레이트에 웰당 하나 이상의 콜로니가 포함되어 있거나 웰에 콜로니가 없는 경우 부정적인 결과를 구성합니다.
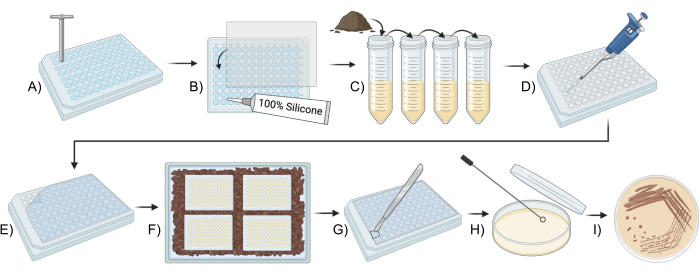
그림 1: 변형된 iChip을 사용하여 토양 미생물을 배양하기 위한 개략도. (A,B) 플레이트의 구성(2단계). (C) 접종물 준비(3단계). (D,E) iChip 설정(4단계). (F) iChip의 배양(5단계). (지아이) 분리물을 기존 매체로 옮깁니다(6단계). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 아이칩 | 한천 현탁액의 토양 농도 | 식민지가 있는 우물 | 식민지 형성 단위 |
| E | 5.0 x 10-6 g / mL | 모두 | 셀 수 없을 정도로 많습니다. |
| F | 5.0 x 10-7g /mL | 모두 | 셀 수 없을 정도로 많습니다. |
| G | 5.0 x 10-8g /mL | 22 | 22 |
| H | 5.0 x 10-9g /mL | 6 | 6 |
표 1: 성공적인 iChip 실험의 대표 데이터로, 콜로니를 포함하는 우물의 수가 직렬로 희석될 때마다 감소합니다.
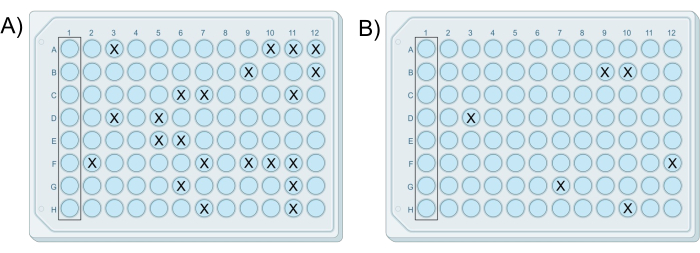
그림 2: 두 개의 성공적인 수정된 iChip의 예시적인 결과. iChips는 (A) 세포-한천 혼합물 G로 제조된 플레이트에는 22개의 콜로니가 포함되어 있고 (B) 세포-한천 혼합물 H로 제조된 플레이트에는 혼합물 간의 10배 희석 차이와 일치하는 6개의 콜로니가 포함되어 있는 동일한 실험에서 수행되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
그림 3A-G와 같이 육안 또는 현미경으로 군체를 시각화할 수 있습니다. 군체는 그림 3B에서 볼 수 있듯이 한천의 표면에서 자라거나 그림 3F의 군체와 같이 한천에 내장될 수 있습니다. 도 3G와 같이 성공적으로 수정된 iChip의 동일한 웰에 두 개의 콜로니가 있는 경우가 있을 수 있습니다. 어떤 경우에는 군체가 충분히 멀리 떨어져 있어 따로 바늘로 뚫고 하반배양할 수 있습니다. 그러나 그림 3G의 물질이 축류 미생물로 쉽게 배양될 수 있을 것 같지는 않습니다. iChip에서 종래의 SMS 매체로 전송되는 콜로니의 예가 그림 4에 나타나 있다.
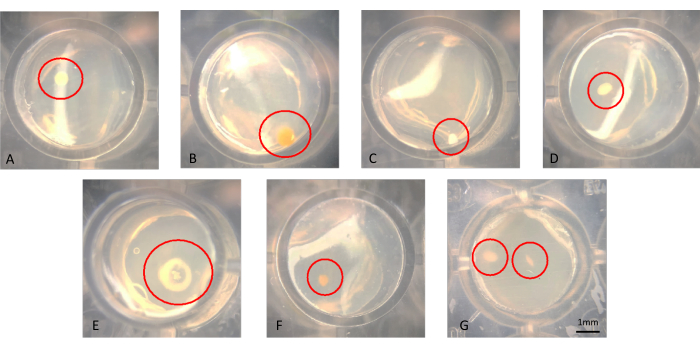
그림 3: 배양 6주 후 iChip에서 자라는 콜로니의 대표적인 현미경 이미지. (A-F) 단일 콜로니가 관찰되며, 이는 하반배양되어야 하며, (G)에서는 두 개의 콜로니가 근접하게 관찰되는데, 이는 축배양으로 얻기 어렵습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
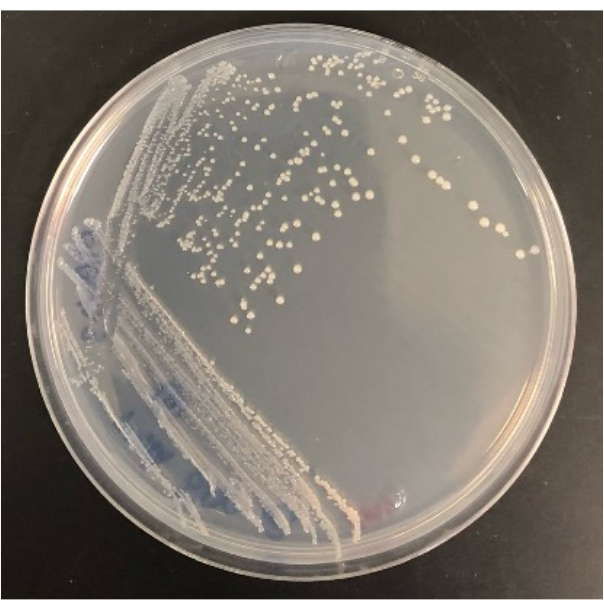
그림 4: SMS 한천 플레이트에서 자란 변형된 iChip에서 분리된 박테리아의 대표적인 결과.이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
변형된 iChip에서 콜로니를 기존 한천 플레이트로 분리하는 능력도 실험의 성공을 결정합니다. 변형된 iChip에서 자라는 모든 콜로니가 기존 플레이트로의 이동에서 살아남는 것은 아닙니다. iChip에서 군체는 성장 인자와 토양 영양분에 노출되어 재배 가능성을 높입니다. 일부 미생물의 경우, 가축화 기간만으로는 기존 조건에서 배양할 수 없습니다. iChip에서 플레이트로의 콜로니 회수율은 실험마다 다르지만, 이 방법은 쉽게 배양할 수 있는 미생물도 배양할 수 있으므로 일부는 플레이트에서 성장해야 합니다. 예를 들어, 그림 2 의 변형된 iChip에는 총 28개의 콜로니가 포함되어 있으며, 하위배양 후 한천 플레이트에서 자란 콜로니의 수는 16개인 반면 12개는 이전에서 살아남지 못했습니다. 그림 4와 같이 전체 크기의 한천 플레이트에서 배양된 후 분리물을 식별하여 추가 연구에 사용할 수 있습니다.
성공적인 변형 iChip 실험은 종 수준에서 알려진 미생물과 다른 미생물을 분리하는 결과를 가져와야 합니다. 알려진 미생물에 대한 각 분리물의 유사성 정도는 분리된 DNA를 알려진 미생물의 게놈과 비교하여 발견됩니다. 각 분리물에 가장 가까운 관련 미생물의 테트라 Z 점수는 유사성 백분율을 설명합니다. 그림 5 는 분리된 12개의 박테리아 중 4개는 새로운 종 이상이고, 2개는 새로운 균주일 가능성이 있으며, 6개는 알려진 미생물임을 보여줍니다. 몇 가지 새로운 종 또는 균주의 회수는 이전에 발표된 iChip 실험과 일치하며, 이는 이러한 변형된 iChip 구조가 새로운 종11의 회수 증가를 방해하지 않음을 나타냅니다.
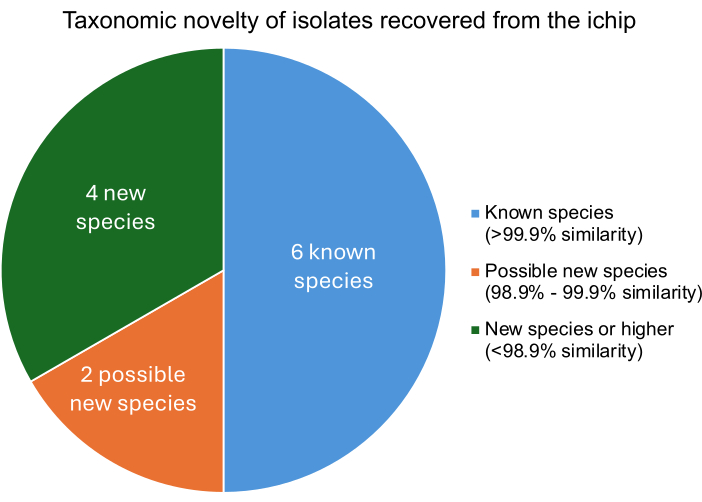
그림 5: iChip 실험에서 회수된 분리물의 분류학적 분류 수준. 유사성 비율은 테트라 Z 점수를 기반으로 결정되었으며, 이는 알려진 박테리아에 대한 분리 유사성을 설명하며, >99.9% 유사성은 종 일치를 나타내고, 98.9%-99.9% 유사성은 가능한 새로운 종을 나타내고, <98.9% 유사성은 새로운 종 이상을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
토론
게놈 채굴 및 조용한 생합성 경로 검사와 같은 수많은 방법은 최근 몇 년 동안 새로운 생리 활성 화학 물질의 발견을 촉진했습니다21,22. 그러나 이러한 방법을 사용하여 발견된 NP는 종종 알려진 화합물과 높은 구조적 유사성을 나타냅니다. 이전에 배양되지 않은 미생물에 접근하면 더 큰 화학적 다양성과 미생물 내성을 퇴치하는 데 더 도움이 될 수 있는 새로운 작용 모드를 가진 NP가 잠금 해제됩니다. iChip 분리 기술은 새로운 미생물의 배양 가능성을 증가시키는 것으로 나타났으며, 이는 새로운 NP의 발견을 위한 미생물 라이브러리를 구축하는 데 사용할 수 있습니다.
iChip의 가장 초기의 개념화는 2002년에 발표되었으며, 이는 반투과성 멤브레인으로 밀봉되고 in situ23에서 배양된 한천 세포 혼합물을 포함하는 단일 금속 링으로 구성되었습니다. 이후 2010년에 많은 웰11이 있는 작은 칩을 포함하기 위해 반복되었으며, 2017년 영향력 있는 Nature Protocols 간행물에서 일반적인 실험실 재료로 구성된 저렴한 iChip이 개발되었습니다9. 이 간행물에서 Nature 프로토콜은 실용성과 사용 편의성을 개선하기 위해 몇 가지 수정이 이루어졌습니다. Nature 프로토콜은 한쪽 면에 접착판 덮개를 사용하는 이 방법과 비교하여 칩의 양면에 반투과성 멤브레인을 붙이는 것입니다. iChip이 로드된 후 실리콘 접착제를 사용하여 멤브레인을 부착할 때 문제가 발생할 수 있습니다. 우리가 아는 한, 모든 무독성 실리콘 접착제는 굳어지는 대로 아세트산을 방출하며, 이는 세포 생존력에 영향을 미칠 수 있습니다24. 이 비디오에 설명된 프로토콜은 또한 PCR 플레이트 커버를 사용하여 채워진 플레이트를 밀봉함으로써 접종 후 필요한 취급량을 크게 줄여 무균성을 더욱 보장하고 설정 시간을 단축합니다. 이 실험에서 여러 개의 새로운 분리체를 식별한 것을 기반으로 단일 반투과성 멤브레인만 사용하면 두 개의 반투과성 멤브레인으로 구성된 iChip에 대해 보고된 새로운 유기체 발견 속도의 증가를 억제할 수 있습니다. 그러나 영향을 정량화하기 위해서는 더 큰 표본 크기를 사용한 추가 실험이 필요할 것입니다.
또 다른 방법 수정은 웰의 25% 미만에서 성장을 포함하는 변형된 iChip에서만 하반배양하는 것입니다. 군체가 플레이트의 대부분의 우물에서 자라면 쉽게 볼 수 없더라도 여러 미생물을 포함하는 일부 우물이 있을 수 있습니다. 방법 개발 과정에서 하나 이상의 식민지를 포함하는 우물에서 하반배양할 때 축삭 배양을 얻는 것이 크게 불가능하다는 것이 밝혀졌습니다. 비축성 배양액의 분리는 생체 활성 분석 및 식별 측면에서 다운스트림에 중요한 문제를 제기합니다. 따라서, 단순화를 위해, 웰의 일부에서 자라는 콜로니가 있는 변형된 iChips만 하대배양하는 것이 좋습니다.
iChip 방법에서 발생할 수 있는 가장 중요한 문제는 우물 또는 전체 iChip이 다른 미생물로 오염되는 것입니다. 오염은 대조군 우물에서 자라는 군체 또는 여러 우물 또는 변형된 iChip의 전체 영역에서 자라는 동일한 미생물로 표시됩니다. 오염원은 설정 중 재료의 부적절한 멸균 또는 부적절한 무균 기술로 인한 것일 수 있습니다. 이러한 경우, 사용된 모든 재료가 방법에 표시된 대로 오토클레이브 또는 에탄올이 멸균되었는지 확인하고, 한천-세포 혼합물 이외의 비멸균 품목과 변형된 iChip 사이에 접촉이 발생하지 않도록 합니다. 여러 개의 변형된 iChip 웰의 하단 또는 상단에서 단일 미생물의 과잉 증식이 관찰되는 경우, 이는 웰과 멤브레인 사이의 불완전한 밀봉의 결과일 가능성이 큽니다. 이 경우 사용된 접착제가 에탄올에서 분해되지 않는 100% 실리콘인지 확인하고 시공 중 반투막과 PCR 플레이트 덮개가 각 웰 주위를 완전히 밀봉했는지 확인하십시오.
현재 프로토콜에 사용된 희석 시리즈는 1,000배 범위의 희석액을 포함하기 때문에 보관 기간이 1주일 미만인 대부분의 토양 유형에 적절한 희석을 제공해야 합니다. 그러나 토양 유형에 따라 미생물 수에 상당한 차이가 있을 수 있습니다. 대조군 웰에는 성장이 없지만 4개의 변형된 iChip 모두의 각 웰에서 여러 콜로니가 자라고 있는 경우 사용된 세포 농도가 충분히 낮지 않습니다. 희석 시리즈는 변형된 iChip을 설정하는 데 사용되는 세포-매체 혼합물에서 더 낮은 세포 농도에 도달하도록 수정해야 합니다. 유사하게, 이러한 플레이트에서 성장이 관찰되지 않으면 세포-배지 혼합물에 사용되는 농도를 증가시켜야 합니다. 또는 사용된 매체의 온도가 미생물의 생존에 너무 높았을 가능성이 있습니다. 이러한 경우, 배지는 희석된 세포 현탁액에 첨가되기 전에 응고되지 않고 가능한 한 많이 냉각되도록 해야 합니다.
이 기술은 엄청난 접시 수 이상 현상을 극복하기 위한 중요한 발전입니다. 그러나, 변형된 iChip에서 기존 한천 플레이트로의 이동에서 살아남지 못하는 미생물의 수에서 알 수 있듯이 기존 배양 기술의 부적합성에 의해 여전히 제한됩니다. 이전 간행물에서는 iChips에서 여러 차례의 하위 배양 및 배양이 미생물의 배양 가능성을 더욱 증가시킨다고보고했습니다. iChip의 한천에 있는 동안 길어진 가축화 시간과 토양 성장 인자에 대한 노출은 한천에서만 군체가 자랄 가능성을 높입니다. 그러나 이 접근법은 단일 iChip 배양보다 새로운 미생물의 가능성이 더 높은 것으로 보고되지 않았습니다25,26.
새로운 미생물의 배양 가능성을 높이기 위해 다른 많은 전술이 연구되고 수정되고 있습니다. 예를 들어, 반투과성 물질로부터 전체 iChip 장치를 만드는 것은 이웃 우물(27)에서 미생물의 공동 배양을 용이하게 하기 위해 제안되었다. 즉, 이 간행물에 요약된 구성의 장점은 플레이트 하나를 만드는 데 드는 비용이 약 $12(96웰 플레이트당 $4, 멤브레인당 $8, PCR 커버당 $2)에 해당하는 저렴한 비용입니다. 또한 간단한 구조로 인해 설명된 대로 사용할 때 복잡하지 않은 도구가 되며 사용자 정의를 위한 많은 가능성을 제공합니다. 이 프로토콜은 박테리아에 대해 선택적인 배지를 사용하지만, 이론적으로 실험 설정은 곰팡이를 표적으로 하기 위해 박테리아 억제 배지를 사용하거나 미생물을 포자화하기 위해 영양이 적은 한천을 사용하는 것과 같이 사용된 배지를 수정하여 원하는 미생물 집단을 표적으로 하도록 조정할 수 있습니다.
공개
저자는 이해 상충이 없다고 선언했습니다.
감사의 말
이 프로젝트를 가능하게 한 캐나다 농업농식품부(Agriculture and Agri-Food Canada)를 통해 제공된 J-base 자금(J-001757, J-001842)에 감사드립니다. 이 프로토콜의 비디오 콘텐츠를 촬영해 주신 Brett van Heyningen에게 감사드립니다. 또한 이 간행물에 설명된 실험에 사용된 토양 샘플을 수집한 Ron Matters에 감사드립니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.1-20 µL pipette tips | VWR | 76322-158 | Pack of 768 |
| 0.2 mL PCR tubes | ThermoFisher Scientific | AB0337 | Case of 1000 |
| 0.5-5 mL single channel pipette | VWR | CA11020-004 | |
| 1 L glass bottle | Millipore Sigma | CLS13951L | Must be autoclaveable. |
| 100 x 15 mm Petri plates | VWR | 25384-342 | Case of 500 |
| 100% Silicone sealant | Marineland | 31003 | |
| 1000 µL multichannel pipette tips | ThermoFisher Scientific | 9401113 | Case of 960 |
| 100-1000 µL pipette tips | VWR | 76322-164 | Pack of 768 |
| 100-1000 µL single channel pipette | VWR | 76169-240 | |
| 100-1200 µL multichannel pippette | ThermoFisher Scientific | 46300800 | Must have 360 µL volume capacity. |
| 1-10 µL single channel pipette | VWR | 76169-232 | |
| 1-5 mL pipette tips | VWR | CA11020-008 | Pack of 500 |
| 15 mL sterile centrifuge tubes | VWR | CA21008-918 | Case of 500 |
| 16s rRNA-F primer (AGAGTTTGATCCTGGCTCAG) – 10mM | Integrated DNA Technologies | 51-01-19-06 | 10 µg |
| 16s rRNA-R primer (ACGGCTACCTTGTTACGACTT) – 10mM | Integrated DNA Technologies | 51-01-19-07 | 10 µg |
| 4 mm cork borer | VWR | 470121-860 | |
| 50 mL sterile centrifuge tubes | VWR | CA21008-940 | Case of 500 |
| 95% ethanol | Thermo Fisher Scientific | A412-500 | 500 mL |
| 96-well plate | VWR | 10062-900 | Case of 100 |
| Autoclave | Cole-Parmer | UZ-01850-34 | 8 L, 115 VAC |
| Bacteriological agar | ThermoFisher Scientific | 443570010 | 1 kg |
| bin | Thomas Scientific | 1216H91 | 5 bins per pack |
| Casamino acids | ThermoFisher Scientific | 223120 | 500 g |
| Casein digest | ThermoFisher Scientific | 211610 | 500 g |
| Electrofluoresis grade agarose | Thermo Fisher Scientific | J66501.30 | 250 g |
| iBright FL1500 Imaging System | ThermoFisher Scientific | A44115 | |
| Laminar flow hood | CleanTech | 1000-6-A | |
| Minion Nanopore Sequencer | Oxford Nanopore Technologies | MinIon Mk1C | |
| NanoDrop One/One Microvolume UV-Vis Spectrophotometer | ThermoFisher Scientific | ND-ONE-W | |
| Nuclease Free Water | Thermo Fisher Scientific | AM9937 | 10 x 50 mL |
| Nucleobond HMW DNA kit | Takara | 740160.2 | |
| Paper towel | VWR | 89402-824 | |
| Phusion Green Hot Start II High-Fidelity PCR Master Mix | Thermo Fisher Scientific | F566L | 500 Reactions |
| Potato starch | ThermoFisher Scientific | 419690025 | 2.5 kg |
| QIAGEN CLC Genomics Workbench Software or similar | Qiagen | ||
| Rapid Barcoding 24 Kit | Oxford Nanopore Technologies | SQK-RBK114.24 | |
| SimpliAmp thermal cycler | Applied Biosystems | A24811 | |
| Sterile Inoculation loops with needle | VVWR | 76534-512 | Case of 1000 |
| Sterile surgical blade | VWR | 76457-444 | |
| SYBR Safe, or simmilar | ThermoFisher Scientific | S33101 | |
| UltraPure Agarose | ThermoFisher Scientific | 16500-500 | |
| Vortex | VWR | 76549-928 | Must accomadate 15 and 50 mL centrifuge tubes |
| VWR Stereo Zoom Trinocular Microscope | VWR | 89404-476 | |
| Whatman Nuclepore Track-Etched Membranes | Millipore Sigma | WHA113502 | L x W 8 in. x 10 in., pore size 0.03 μm |
참고문헌
- Newman, D. J., Cragg, G. M. Natural products as sources of new drugs over the nearly four decades from 01/1981 to 09/2019. J Nat Prod. 83 (3), 770-803 (2020).
- Fouillaud, M., Dufosse, L. Microbial secondary metabolism and biotechnology. Microorganisms. 10 (1), 123 (2022).
- Rizzetto, G., et al. Role of daptomycin in cutaneous wound healing: A narrative review. Antibiotics. 11 (7), 944 (2022).
- Miethke, M., et al. Towards the sustainable discovery and development of new antibiotics. Nat Rev Chem. 5 (10), 726-749 (2021).
- Gunjal, V., Thakare, R., Chopra, S., Reddy, D. S. Teixobactin: A paving stone toward a new class of antibiotics. J Med Chem. 63, 12171-12195 (2020).
- Atanasov, A. G., Zotchev, S. B., Dirsch, V. M., Supuran, C. T. Natural products in drug discovery: Advances and opportunities. Nat Rev Drug Discov. 20 (3), 200-216 (2021).
- Epstein, S. S. The phenomenon of microbial uncultivability. Curr Opin Microbiol. 16 (5), 636-642 (2013).
- Wright, G. An irresistible newcomer. Nature. 517, 422-444 (2015).
- Berdy, B., Spoering, A., Ling, L., Epstein, S. In situ cultivation of previously uncultivable microorganisms using the ichip. Nat Protoc. 12 (10), 2232-2242 (2017).
- Jung, D., et al. Triggering growth via growth initiation factors in nature: A putative mechanism for in situ cultivation of previously uncultivated microorganisms. Front Microbiol. 12, 537194 (2021).
- Nichols, D., et al. Use of ichip for high-throughput in situ cultivation of "uncultivable" microbial species. Appl Environ Microbiol. 76 (8), 2445-2450 (2010).
- Megaw, J., Kelly, S. A., Thompson, T. P., Skvortsov, T., Gilmore, B. F. Profiling the microbial community of a triassic halite deposit in Northern Ireland: An environment with significant potential for biodiscovery. FEMS Microbiol Lett. 366 (22), fnz242 (2019).
- Vitorino, I., et al. Novel and conventional isolation techniques to obtain planctomycetes from marine environments. Microorganisms. 9 (10), 2078 (2021).
- Moote, P., Polo, R. O., Uwiera, R. R. E., Inglis, G. D. Comparison of strategies for isolation anaerobic bacteria from the porcine intestine. Appl Environ Microbiol. 87 (9), e00088-e00121 (2021).
- Vitorino, I. R., et al. Rhodopirellula aestuarii sp. Nov., a novel member of the genus rhodopirellula isolated from brackish sediments collected in the tagus river estuary, Portugal. Syst Appl Microbiol. 45 (6), 126360 (2022).
- Ding, H., et al. Hanstruepera marina sp. Nov. and Hanstruepera flava sp. Nov., two novel species in the family flavobacteriaceae isolated by a modified in situ cultivation technique from marine sediment. Front Microbiol. 13, 957397 (2022).
- Ling, L. L., et al. A new antibiotic kills pathogens without detectable resistance. Nature. 517 (7535), 455-459 (2015).
- Qi, Y. K., et al. Discovery, synthesis, and optimization of teixobactin, a novel antibiotic without detectable bacterial resistance. J Pept Sci. 28 (11), e3428 (2022).
- Piddock, L. J. Teixobactin, the first of a new class of antibiotics discovered by ichip technology. J of Antimicrob Chemother. 70 (10), 2679-2680 (2015).
- Gong, L., Wong, C. H., Idol, J., Ngan, C. Y., Wei, C. L. Ultra-long read sequencing for whole genomic DNA analysis. J Vis Exp. (145), e58954 (2019).
- Covington, B. C., Xu, F., Seyedsayamdost, M. R. A natural product chemist's guide to unlocking silent biosynthetic gene clusters. Annu Rev Biochem. 90, 763-788 (2021).
- Lee, N., et al. Mini review: Genome mining approaches for the identification of secondary metabolite biosynthetic gene clusters in Streptomyces. Comput Mol Biol. 18, 1548-1556 (2020).
- Kaeverlein, T., Lewis, K., Epstein, S. S. Isolating "uncultivable" microorganisms in pure culture in a simulated natural environment. Science. 296 (5570), 1127-1129 (2002).
- Trcek, J., Mira, N. P., Jarboe, L. R. Adaptation and tolerance of bacteria against acetic acid. Appl Microbiol Biotechnol. 99 (15), 6215-6229 (2015).
- Kurm, V., Van Der Putten, W. H., Hol, W. H. G. Cultivation-success of rare soil bacteria is not influenced by incubation time and growth medium. PLOS One. 14 (1), e0210073 (2019).
- Buerger, S., et al. Microbial scout hypothesis and microbial discovery. Appl Environ Microbiol. 78 (9), 3229-3233 (2012).
- Lodhi, A. F., Zhang, Y., Adil, M., Deng, Y. Antibiotic discovery: Combining isolation chip (ichip) technology and co-culture technique. Appl Microbiol Biotechnol. 102 (17), 7333-7341 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유