Method Article
Xénogreffe ortho- et ectopique de poisson zèbre de mélanome oculaire pour récapituler la tumeur primaire et le développement de métastases expérimentales
Dans cet article
Résumé
Ici, nous présentons un protocole pour établir des modèles polyvalents de xénogreffes de poisson-zèbre orthotopique et ectopique pour le mélanome oculaire afin d’évaluer la cinétique de croissance de la tumeur primaire, la dissémination, l’extravasation et la formation de métastases périvasculaires à distance et l’effet de l’inhibition chimique sur celles-ci.
Résumé
Il n’existe actuellement aucun modèle animal pour le mélanome oculaire métastatique. Le manque de modèles de maladies métastatiques a grandement entravé la recherche et le développement de nouvelles stratégies pour le traitement du mélanome oculaire métastatique. Dans ce protocole, nous décrivons un moyen rapide et efficace de générer des modèles embryonnaires de poisson-zèbre pour le stade primaire et disséminé du mélanome oculaire, en utilisant respectivement l’orthotopie rétro-orbitaire et la greffe intravasculaire de cellules ectopiques. En combinant ces deux stratégies de greffe différentes, nous pouvons récapituler l’étiologie du cancer dans sa totalité, progressant d’une croissance tumorale primitive et localisée sous l’œil à la formation de métastases périvasculaires dans la queue. Ces modèles nous permettent de modifier rapidement et facilement les cellules cancéreuses avant l’implantation avec un marquage spécifique, une interférence génétique ou chimique ; et de traiter les hôtes greffés avec des inhibiteurs (petits moléculaires) pour atténuer le développement tumoral.
Ici, nous décrivons la génération et la quantification de la greffe orthotopique et ectopique de mélanomes oculaires (mélanome conjonctival et uvéal) à l’aide de lignées cellulaires stables marquées par fluorescence. Ce protocole est également applicable à la greffe de cellules primaires dérivées de la biopsie du patient et du matériel dérivé du patient/PDX (manuscrit en préparation). Dans les heures qui suivent la greffe, la migration et la prolifération cellulaires peuvent être visualisées et quantifiées. Les deux foyers tumoraux sont facilement disponibles pour l’imagerie avec la microscopie à épifluorescence et la microscopie confocale. À l’aide de ces modèles, nous pouvons confirmer ou infirmer l’activité des stratégies d’inhibition chimique ou génétique dans les 8 jours suivant le début de l’expérience, ce qui permet non seulement un dépistage très efficace sur des lignées cellulaires stables, mais permet également un dépistage dirigé par le patient pour les approches de médecine de précision.
Introduction
La dissémination métastatique est considérée comme la principale cause de décès du mélanome oculaire ; À l’heure actuelle, il n’existe pas de régime de traitement viable pour le mélanome oculaire disséminé 1,2. De plus, il n’existe aucun modèle animal disponible pour le mélanome oculaire qui reflète la maladie métastatique. Pour combler cette lacune, nous avons généré deux modèles distincts de poisson-zèbre qui récapitulent soit la formation tumorale primaire, soit les premiers stades de la dissémination métastatique, permettant ainsi l’étude de ces processus normalement difficiles à étudier 3. Les modèles de micro-métastases permettent d’analyser les dernières phases de la propagation métastatique, y compris le homing, la colonisation et l’extravasation. Des interventions génétiques ou chimiques à ce stade et au-delà pourraient potentiellement fournir une prise puissante dans le traitement du mélanome oculaire métastatique.
L’utilisation des larves de poisson-zèbre comme receveur de xénogreffons et d’allogreffes est soutenue par les forces intrinsèques de cette espèce, telles que sa transparence optique aux premiers stades de son développement (ou tout son cycle de vie pour les mutants casper 4), sa fécondité élevée et sa fécondation ex utero 5. Une homologie transcriptionnelle élevée chez les vertébrés garantit la conservation des mécanismes de signalisation de base entre le poisson zèbre et les humains et donc une traduisibilité potentielle élevée des résultats 6, bien que les approches génétiques soient parfois gâchées ou compliquées en raison de la duplication du génome téléostéen 7. Les développements récents ont souligné l’importance des modèles de xénogreffes de poisson-zèbre en tant qu'« avatars » précliniques de la maladie humaine8, produisant ainsi une multitude de modèles de thérapie anticancéreuse personnalisés pour l’évaluation préclinique des stratégies de traitement à partir d’une seule expérience de poisson-zèbre 9.
Compte tenu du manque de modèles animaux et du manque concordant d’options de traitement pour le mélanome oculaire métastatique, nos modèles fournissent une plate-forme translationnelle rapide et facile pour dépister à la fois les altérations génétiques (intrinsèques aux cellules cancéreuses) ou développer des stratégies d’intervention chimique dans un cadre préclinique. Dans le même modèle, nous pouvons visualiser et mesurer la cinétique de croissance des cellules cancéreuses, le taux de greffe/potentiel métastatique et le retour cellulaire au niveau de l’animal entier en utilisant un grossissement de faible niveau dans un microscope stéréofluorescent, et effectuer des mesures similaires en utilisant une analyse microscopique confocale à grossissement moyen ou élevé pour disséquer différentes étapes de la progression du mélanome oculaire à une résolution subcellulaire de 10.
Nous décrivons ici des protocoles complets et détaillés pour : la génération de cellules cancéreuses marquées par fluorescence à l’aide d’une transduction lentivirale hautement optimisée11 ; greffes intraveineuses et rétro-orbitaires (RO) ultérieures de ces cellules dans des larves de poisson-zèbre 2 jours après la fécondation (DPF) pour générer des modèles ectopiques et orthotopiques respectivement ; suivi de l’acquisition et de l’analyse des données. Ces méthodes, bien que complètes pour les applications décrites dans le présent document, peuvent être modifiées pour greffer des cellules dans la cavité cérébrale postérieure, le foie et l’espace périvitellin lorsque nécessaire (uniquement en changeant le site d’injection ou le moment de l’injection)12,13.
En tant que preuve de concept, nous avons développé les résultats de Pontes et al. 2018, où nous avons montré une dose et une réponse spécifique à la mutation cellulaire intrinsèque des lignées cellulaires de mélanome conjonctival dans le modèle de poisson zèbre 14. Nous avons approfondi ces résultats en montrant l’efficacité de l’inhibiteur spécifique de la mutation BRAF V600E, le vemurafénib, dans des modèles de mélanome conjonctival métastatique et primaire.
Protocole
Toutes les expériences sur les animaux ont été approuvées par le Comité d’expérimentation animale (Dier Experimenten Commission, D.E.C.) sous licence AVD1060020172410. Tous les animaux ont été entretenus conformément aux directives locales à l’aide de protocoles standard (www.ZFIN.org).
1. Préparation
- Réactifs
- Préparez l’eau de l’œuf : 0,6 mg/L de sel marin de concentration finale.
- Préparez 5 mg/mL de tricaïne 25x : Mélangez 5 g de poudre de tricaïne (éthyl-3-aminobenzoate de méthanesulfonate ou MS-222), 900 mL d’eau déminéralisée et 21 mL de 1 M de Tris (pH 9). Ajuster au pH 7 et remplir jusqu’à 1 L. La tricaïne peut être conservée à 4 °C à court terme (jusqu’à six mois) ou à température ambiante pendant un mois à température ambiante à l’abri de la lumière du soleil.
- Préparer de l’agarose à 1,5 % (p/v) dans de l’eau d’œuf : 1,5 g dans 100 mL de DPBS. Micro-ondes pour dissoudre.
- Préparer de l’agarose à bas point de fusion à 1 % (p/v) dans de l’eau d’œuf : 1,5 g dans 100 mL de DPBS. Micro-ondes pour dissoudre.
- Préparez une pâte PVP40 à 2 % (p/v) dans du DPBS : 1 g de PVP40 dans 50 ml de DPBS. Vortex et incubation à 37 °C pour faciliter la dissolution. Conserver à température ambiante.
- Utilisez le DMSO. Il est souvent utilisé comme solvant dans les traitements médicamenteux et doit être stocké à une température de 2 à 8 °C dans l’obscurité.
- Utilisez TrypLE, un substitut synthétique de la trypsine qui est moins dommageable pour les cellules et permet la dispersion en douceur des cellules fortement adhérentes.
- Préparez la solution saline tamponnée au phosphate (DPBS) de Dulbecco sans Mg2+ et Ca2+ pour laver les cellules. L’absence de Ca2+ altère l’adhésion cellule-cellule par le biais des cadhérines.
- Préparez des plasmides lentiviraux : psPAX2 (plasmide #12260) et pMD2.G (plasmide #12259) offerts par Didier Trono et soit un GFP (plasmide #106172) soit tdTomato (plasmide #106173) codant pour le plasmide de transfert (Addgene).
- Utilisation LipodD293 : réactif de transfection hautement efficace HEK293T optimisé.
- Plat d’agarose
REMARQUE : Lorsque vous utilisez des plats qui ont été conservés pendant une longue période, assurez-vous d’ajouter un petit volume d’eau d’œuf dans les plats avant de commencer l’injection (cela empêchera le poisson de se dessécher trop rapidement).- Préparez des plats enrobés d’agarose à 1,5 % (p/v) (agarose dissoute dans l’eau d’œuf).
- Utiliser immédiatement ou conserver à 4 °C en position inversée.
2. Aiguilles
REMARQUE : Assurez-vous que les capillaires ont été calibrés sur le filament utilisé. Lors de la commutation du filament ou du capillaire, déterminez la valeur de rampe des capillaires sur le filament utilisé (voir le manuel de l’extracteur d’aiguille).
- Un capillaire en verre donnera deux aiguilles de micro-injection. Avant de fabriquer des aiguilles, vérifiez l’intégrité structurelle du filament (filament de boîte de 2,5 mm) de l’extracteur d’aiguille.
- Assurez-vous que le filament et le capillaire sont calibrés pour obtenir la valeur de rampe correspondante. Lorsque l’intégrité structurelle des filaments est compromise (c’est-à-dire inégale, trous, fondue, etc.), changez le filament.
- Utilisez le programme suivant (Aiguille #99, Chaleur=rampe+15, traction=95, vitesse=60, temps=90). Rangez les aiguilles dans une boîte de Pétri désignée (contenant de l’argile ou du ruban adhésif pour coller les aiguilles)
3. Génération de particules lentivirales
REMARQUE : Pour éviter une perte de temps et de ressources, un contrôle rapide de la tumorigénicité peut être effectué avant la transduction lentivirale. Ceci est fait pour s’assurer que la lignée cellulaire à utiliser est suffisamment tumorigène dans le modèle de poisson zèbre, à cette fin, les cellules peuvent être colorées avec un CMdiI (ou traceur analogue) comme décrit dans Liverani et al. 2017 15.
- Plaquez les cellules HEK 293t un jour avant la transfection pour obtenir une confluence d’environ 70 % (ce qui est systématiquement fait en divisant un ballon plein dans le ballon de culture du même volume à une dilution de 1:3 un jour avant).
- Le jour de la transfection, co-transfectez les plasmides d’emballage requis psPAX2 et pMD2.G exprimant l’enveloppe virale du plasmide avec un GFP (plasmide #106172) ou tdTomato (plasmide #106173) codant pour le plasmide de transfert. La quantité exacte de plasmide utilisée est spécifiée dans le tableau 1.
REMARQUE : psPAX2 et pMD2.G ont été offerts par Didier Trono (plasmide Addgene #12260 et #12259, respectivement).
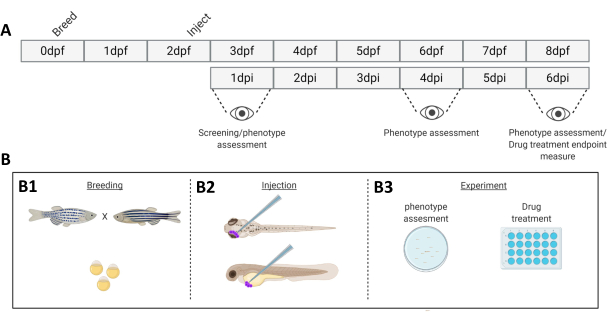
Graphique 1. Représentation schématique du système de greffe de poisson-zèbre décrit. A) La chronologie de l’approche, avec l’élevage du poisson-zèbre au jour 0 (B1). Les poissons sont récoltés le matin après le croisement du poisson (jour 1). Après 48 à 54 heures, les poissons ont en grande partie éclos (muant leur chorion) et les poissons sont injectés (rétro-orbitale ou systémique, B2) après avoir nettoyé l’eau des débris du chorion (jour 2). Les larves sont ensuite tamisées à l’aide d’un microscope stéréofluorescent et toutes les larves présentant des phénotypes indésirables sont éliminées (jour 3). Selon l’objectif de l’expérience, soit les larves sont imagées au fil du temps (B3, cinétique de greffe, imagerie à 1, 4 et 6 jours après l’injection (dpi)) ou les poissons sont randomisés et répartis dans des groupes expérimentaux, traités avec des médicaments et comparés à un témoin véhiculaire (dépistage de drogues, imagerie à 6 dpi). Veuillez cliquer ici pour voir une version agrandie de cette figure.
- Mélangez tous les plasmides dans 500 μL de milieu exempt de sérum, pour permettre un mélange complet de tous les plasmides. Ajouter 32 μL de réactif LipoD293 à 500 μL de DMEM sans sérum, et agiter au vortex pour mélanger complètement. Mélangez soigneusement les deux volumes. Laissez les plasmides et le lipoD293 se complexer pendant 20 minutes.
- Ajouter goutte à goutte dans une fiole de culture de 2 cellules de75 cm contenant 70 % de cellules de HEK293T confluentes contenant 9 mL de milieu de culture complet. Ajouter le mélange de transfection directement sur la couche cellulaire à l’aide d’une pipette sérologique (fiole en orientation horizontale).
- Remplacer le milieu par 20 ml de DMEM complet frais 16 heures après la transfection. Récoltez le surnageant 72 heures après la transfection. Aliquote du surnageant viral dans des aliquotes de 1 mL et conserver à -80 °C. Le surnageant lentiviral est stable à -80 °C pendant au moins 1 an.
4. Transduction lentivirale
- Avant la transduction lentivirale, établissez une courbe d’élimination lorsque vous utilisez une construction lentivirale sélectionnable.
- Pour la courbe de destruction, plaquez la lignée cellulaire à transduire, dans une plaque à 12 puits (confluence environ 10-20 %). Ajouter une courbe de dose du sélectif (concentrations approximatives pour les courbes de destruction : puromycine 0,5-10 μg/mL, blasticidine 1-20 μg/mL, généticine (G418) 100-2000 μg/mL, hygromycine 100-2000 μg/mL).
- Changez de milieu tous les trois jours pour assurer une concentration stable du sélectif choisi.
- Ajouter 1 mL de surnageant lentiviral à 9 mL de milieu de culture, contenant une concentration finale de 8 μg/mL de polybrène sur 20 à 40 % de cellules confluentes. Les volumes peuvent être réduits, tout en conservant ce rapport surnageant/moyen.
- 16 à 24 heures après la transduction, changez le support. Si nécessaire, répétez l’étape précédente pour améliorer la pénétrance phénotypique (vérifiez la fluorescence pour décider si une autre transduction est nécessaire).
- 48 heures après la transduction, sélectionner les cellules à l’aide de l’antibiotique correspondant au marqueur de résistance incorporé dans la cassette lentivirale. La concentration à utiliser pour la sélection de la population de cellules transduites devrait tuer la population de type sauvage dans les 7 jours suivant l’application du sélectif (c.-à-d. permettre aux cellules transduites de dépasser la population de type sauvage).
- Appliquer un surnageant viral dans différentes multiplicités d’infection (MOI) pour s’assurer que la transduction et les lésions génétiques induites par le génome cellulaire n’affectent pas négativement la viabilité cellulaire ou la tumorigénicité.
5. Élevage du poisson-zèbre
- Le jour 0, 2 jours avant la greffe de cellules cancéreuses, accouplez le poisson-zèbre adulte en « croisement familial » à température ambiante (Figure 1).
- Retirez l’aquarium de poisson-zèbre du système de logement (maintenu à 28,5 °C).
- Séparez les poissons en petits groupes de reproduction dans un rapport de 1:1 mâle : femelle, avec 10 poissons par grappe. Placez les poissons dans de petits bassins d’élevage, dans de l’eau puisée dans le système de logement, au-dessus d’une grille inclinée (inclinée, pour imiter les bas-fonds dans lesquels le poisson-zèbre se reproduirait naturellement).
REMARQUE : Induit par la baisse de la température de 28,5 °C à la température ambiante (25 °C) et l’entrée dans la prochaine phase de lumière du cycle obscurité/lumière, le poisson frayera. - Par la suite, retirez les adultes et transférez-les dans leur réservoir d’habitation.
- Récupérez les œufs et lavez-les à l’eau des œufs à l’aide d’une passoire. Répartissez les œufs à environ 75-100 par plat et maintenez-les à 28,5°C.
- Environ 6 heures après le prélèvement, nettoyez la vaisselle des embryons morts ou malformés.
- Le lendemain matin, remplacez l’eau de l’œuf et nettoyez à nouveau la vaisselle des embryons morts.
6. Cellules de récolte
REMARQUE : Une bonne préparation des cellules est essentielle à la procédure d’implantation, l’utilisation d’une quantité superflue de cellules permet un traitement en aval plus facile. La troisième étape de centrifugation est critique, car cela ne vous laissera que la pastille de cellule, le PBS restant collé sur les côtés du tube de centrifugation dépasse largement le volume final de remise en suspension.
- Préchauffez tous les milieux et solutions utilisés en culture cellulaire dans un bain-marie à 37 °C avant utilisation.
- Ajouter 2 mL de TryplE par flacon de2 cultures de 75 cm ou 1 mL par flacon de25 cm et incuber jusqu’à ce que toutes les cellules soient arrondies. Pour la plupart des lignées cellulaires, 2 à 5 minutes devraient suffire. Pour les cellules hautement épithéliales ou les cellules fibroblastiques, 5 à 10 minutes devraient permettre un bon détachement (une trypsinisation insuffisante entravera les processus en aval et facilitera l’agrégation cellulaire lors de l’implantation).
- Tapotez doucement le côté de la fiole pour déloger les cellules restantes.
- Additionnez le volume de culture d’origine du milieu complet. Pipeter de haut en bas doucement mais soigneusement avec une pipette sérologique pour cisailler les agglomérations cellulaires en suspension unicellulaire. Ne générez pas de mousse au cours de ce processus car la mousse est indicative d’un cisaillement mécanique des cellules.
- Transvaser dans un tube stérile de 15 mL et centrifuger pendant 5 minutes à 200 x g à température ambiante. Aspirer le surnageant et ajouter 1 mL de PBS stérile. Remettez soigneusement et soigneusement les cellules en suspension à l’aide d’une pipette stérile de 1000 μL.
- Retirer la suspension cellulaire de 20 μL pour le comptage et transférer la suspension cellulaire restante dans la centrifugeuse. Centrifuger pendant 4 minutes à 200 x g à température ambiante.
- ÉTAPE CRITIQUE : Retirer tout le PBS, centrifuger pendant 30 s à 200 x g à température ambiante, et retirer le PBS restant.
- Diluer les cellules à 250 cellules/nL dans du polyvinylpyrrolidon 40 à 2 % (PVP40, 2 % (p/v) dans le DPBS) comme suit :
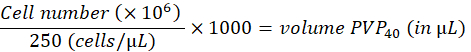
(par exemple,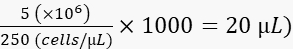
- Remettre soigneusement les cellules en suspension, tout en évitant la formation de bulles d’air (les cellules peuvent être conservées pendant au moins 2 heures en PVP40 à 2 % sans perte de potentiel tumorigène).
7. Modélisation de la xénogreffe
Toutes les expériences doivent être effectuées dans le respect des réglementations locales en matière de bien-être animal.
Selon l’application, deux variantes principales de la conception expérimentale sont classées : une évaluation du phénotype (7.1 l’étape de pré-criblage) et deuxièmement, 7.2 un criblage où les cellules ont été modifiées avant la greffe ou 7.3 où les embryons sont traités avec un inhibiteur chimique.
- Présélection et détermination du potentiel tumorigène
- Greffer des larves de poisson-zèbre d’intérêt (WT, transgénique ou ligne rapporteure) à 2 dpf avec un nombre variable de cellules fluorescentes (c’est-à-dire 200, 400, 600 ±100).
- Criblez les larves 16 à 24 heures après l’injection pour éliminer les valeurs aberrantes (nombre de cellules en circulation extrêmement élevé ou faible pour le modèle ectopique, ou cellules à l’intérieur de la tête pour le modèle orthotopique) et retirer les poissons mal greffés. Indiquer le nombre de larves par groupe expérimental pour l’analyse de groupe par rapport à l’analyse cinétique des mêmes larves.
- Surveiller les larves de poisson-zèbre à intervalles réguliers (1, 2, 4, 6 jours après l’injection (ppp)) et imager 20 individus (comme décrit aux étapes 9 et 10), à partir d’un bassin de ±50 larves.
- Surveiller le phénotype général et la progression de la maladie, puis quantifier avec ImageJ (mesure de la densité intégrée du signal fluorophore dans les cellules cancéreuses).
- Tracez les données pour visualiser la cinétique de croissance des cellules cancéreuses chez le poisson-zèbre (Figure 3).
- Modifier a priori les cellules (knock down ou knock out d’un gène d’intérêt) et les greffer dans le poisson zèbre.
- Greffez le poisson et éliminez tous les phénotypes indésirables (par condition).
- Imagez les individus à 1 dpi (20 larves par groupe). Les individus peuvent être imagés à des intervalles définis (1, 2, 4 et 6 dpi).
- À 6 dpi après l’imagerie, euthanasier le poisson en le surdosant avec de la tricaïne (10 fois plus élevé à 0,4 mg/mL) et le jeter sur du papier absorbant tapissant un entonnoir.
- Traitez le poisson avec des médicaments après la greffe.
- Avant l’application du médicament sur le poisson-zèbre greffé, déterminez la dose maximale tolérée (DMT) sur le poisson-zèbre (titrez de 10 μM à 0,150 nM, en utilisant le volume le plus élevé de solvant comme contrôle négatif), nous avons fixé la DMT comme la concentration où >80 % des individus survivent à l’ensemble du traitement.
- Un jour après l’injection, supprimez les phénotypes indésirables.
- Répartissez au hasard les poissons en groupes (36-48 individus/condition) et maintenez-les dans une assiette de 24 puits avec 6 larves par puits dans 1 mL d’eau d’œuf.
- Appliquez les médicaments 24 heures après la greffe. Comme contrôle, utilisez la même quantité de solvant (DMSO, EtOH, etc.) au volume le plus élevé appliqué pour un groupe expérimental.
- Commencer le traitement médicamenteux à la dose maximale tolérée. Changez l’eau de l’œuf contenant du médicament tous les deux jours. Retirez l’eau des œufs et les larves mortes aussi complètement que possible à chaque changement.
8. L’injection
REMARQUE : Utilisez un contrôleur d’impulsions pneumatique couplé à une conduite d’air comprimé, fournissant une pression supérieure à 100 psi. Cela permet d’avoir une pression suffisante pour injecter (≈20 psi) et pour éjecter d’éventuels agrégats cellulaires (≈100 psi). La pression et le temps de démarrage doivent être d’environ 200 ms à 20 psi. Si l’un ou l’autre doit être diminué de plus de 50 % au début de l’injection, soit que la suspension cellulaire est trop fluide (concentration cellulaire ou PVP40 trop faible), soit que l’ouverture de l’aiguille est trop grande.
- Retirez délicatement une aiguille capillaire de son récipient. Casser l’aiguille pour former une ouverture de ø20 μm, à l’aide d’une pince de fin horloger.
- Remettez soigneusement et soigneusement les cellules en suspension à l’aide d’une pointe de pipette de 20 μL. Pipeter la suspension de la cellule dans l’aiguille capillaire en verre ouverte à l’aide d’une longue pointe (microchargeur). Chargez l’aiguille dans le micromanipulateur.
- Placer ~20 à 40 larves anesthésiées dans 0,04 mg/mL de tricaïne sur une boîte d’agarose à l’aide d’une pipette de transfert. Enlever l’excès d’humidité pour immobiliser les larves à l’aide d’une pipette de transfert. Les larves seront principalement orientées de manière latérale en raison de la présence d’un sac vitellin encore relativement grand.
- Injecter aux larves environ 200, 400 et 600 cellules via le Canal de Cuvier (doC) pour le modèle anti-topique.
- De même, injecter les larves par voie rétro-orbitaire (RO). Pour obtenir le modèle orthotopique (injection de 100 ±50 cellules), modifiez la longueur de l’impulsion pneumatique sur la picopompe (à partir de ~20 psi, 200 ms et ajustez en conséquence). Pendant l’injection, assurez-vous que les larves ne se dessèchent pas. Assurez-vous que toutes les larves (ou la plupart) sont injectées.
- Rincer les larves injectées avec de l’eau fraîche pour œufs et les transférer dans une boîte de Pétri propre étiquetée (contenant jusqu’à 150 individus par boîte). Répétez ce processus jusqu’à ce qu’une quantité suffisante de larves soit injectée.
- Après la greffe, maintenez le poisson à 34 °C dans un incubateur humidifié, où 34 °C est la température la plus élevée facilement tolérée par le poisson-zèbre et permet une greffe efficace de cellules cancéreuses de mammifères.
REMARQUE : En général, avec l’injection de lignées cellulaires uniques dans doC et OI, nous avons observé une mort approximative due à des dommages mécaniques de <5 % (les dommages mécaniques tuent les larves entre 1 et 16 heures après l’injection).
9. Dépistage
- À l’aide d’un microscope à stéréofluorescence, dépister le phénotype approprié chez le poisson 1 heure après l’implantation en comparant les cellules modifiées a priori (ou 1 jour après l’implantation, lors du dépistage des médicaments, avant l’affectation aléatoire dans les groupes de traitement).
- Les larves implantées par le biais du doC doivent avoir des cellules dans la queue entre 1 heure et 16 heures après l’implantation. Retirez tous les autres poissons, y compris les poissons présentant une anomalie, de la flaque injectée.
REMARQUE : Les larves implantées rétro-orbitaire ne doivent avoir des cellules que dans l’interstitium derrière l’œil, les larves qui ont des cellules réparties dans toute la tête ou le corps sont retirées de la piscine. - Nettoyer les larves ayant fait l’objet d’un dépistage positif et les répartir au hasard dans des groupes expérimentaux.
- Après la greffe, maintenez le poisson à 34 °C dans un incubateur humidifié et surveillez-le quotidiennement. La dissémination hématogène des cellules implantées par le doC est presque instantanée, tandis que la propagation métastatique des cellules implantées dans la cavité RO se propagera après 2 à 4 jours.
10. Imagerie épifluorescente de larves de poisson-zèbre
- Anesthésier les larves de poisson-zèbre avec 0,2 mg/mL de tricaïne, soit en ajoutant de la tricaïne à l’eau du poisson, soit en déplaçant une sous-population de poissons de la boîte d’entretien vers une boîte contenant 0,2 mg/mL de tricaïne.
- Gardez le poisson-zèbre dans un plat avec de la tricaïne jusqu’à ce qu’il reste immobile, jusqu’à ce que la stimulation de la ligne latérale n’induise pas de comportement de vol.
- Transférer le poisson dans une boîte de Pétri recouverte d’agarose, environ 10 par plat. Retirez la majorité de l’eau en soulevant doucement une extrémité du plat (permettant à l’eau de s’accumuler doucement dans l’extrémité inférieure de la boîte de Pétri). Si cela est fait avec soin, tous les poissons s’aligneront, la queue tournée vers le bas.
- Imagez tous les poissons du haut vers le bas du plat. Lavez ensuite le poisson avec de l’eau d’œuf dans un plat sans tricaïne.
- Répétez l’opération jusqu’à ce qu’un nombre suffisant d’individus soient imagés.
- Ensuite, transférez les larves à 34 °C ou réformez-les (à 6 dpi) par surdosage de tricaïne (c.-à-d. 0,5 mg/mL, en incubant pendant 10 min, avant de les jeter sur du papier absorbant tapissant un entonnoir).
11. Imagerie confocale de larves de poisson-zèbre (greffées)
- Anesthésier le poisson-zèbre avec 0,2 mg/mL de tricaïne comme décrit précédemment.
- Placez une parabole confocale à fond de verre sous un stéréomicroscope et faites la mise au point sur le fond de la parabole. Transférez 5 à 10 larves dans une boîte confocale à fond de verre. Retirez autant d’eau que possible.
- Recouvrez les larves d’agarose à 42 °C, 1 % d’agarose à faible point de fusion, dissoute dans de l’eau d’œuf. Assurez-vous que l’agarose a refroidi à au moins 42 °C avant utilisation ; Des températures plus élevées peuvent endommager ou tuer les larves.
- À l’aide du stéréomicroscope, orientez rapidement mais doucement les larves en les poussant vers le bas, à l’aide d’une pointe de micro-chargeur réduite. Si une orientation ventrale est nécessaire, maintenez les larves en place avec la pince d’une pince d’horloger (sans toucher l’embryon).
- Tandis que les ensembles d’agarose ajustent finement l’orientation des larves. Laissez les larves prendre complètement avant de les transférer au microscope confocal.
12. Réglage du microscope confocal
- Activez les lignes laser d’excitation vertes (488 nm) et rouges (564 nm). Placez la parabole confocale dans le support du microscope confocal. À l’aide de l’épifluorescence, déplacez le faisceau de lumière pour qu’il fusionne avec le premier poisson (réglage x et y). À travers l’oculaire, réglez le foyer pour qu’il coïncide avec le centre des larves (réglage z).
- Réglez le gain de 700 sur les deux canaux fluorescents, 1-5 % de puissance laser. Augmentez la puissance du laser et diminuez le décalage jusqu’à une plage dynamique complète approximative. Ne sursaturez pas le signal, mais améliorez le signal pour qu’il n’affiche que quelques pixels saturés.
- Lors de la capture d’un point, placez le début et la fin des larves le long d’un axe ( x ou y), s’ils sont placés le long d’un axe, un embryon entier peut être imagé en 1 x 4 segments et peut être post-traité en une seule image à l’aide d’ImageJ.
- Après l’imagerie, retirez les larves de l’agarose en la déchirant doucement autour des larves incrustées à l’aide d’une pince d’horloger. Sinon, euthanasiez les larves en les surdosant avec de la tricaïne non diluée, en recouvrant l’agarose d’une couche de tricaïne et en l’incubant pendant 10 minutes.
13. Analyse des données
- Ouvrez les différents ensembles de données dans ImageJ/Fiji (c’est-à-dire contrôle, médicament A, médicament B, médicament A+B) séparément, en commençant par le contrôle du véhicule.
- Ouvrez la macro d’analyse (script annoté disponible) (http://doi.org/10.5281/zenodo.4290225).
- En bref, l’analyse macro fait ce qui suit : concatène toutes les images ouvertes (une condition) ; divise les images en canaux séparés constituant l’image ; ferme tous les canaux accessoires (en quittant le canal des cellules cancéreuses) ; exécute un algorithme de seuillage, sur l’ensemble de la séquence concaténée ; mesure la densité intégrée de chaque image individuelle ; et enregistre les mesures sous forme de feuille Excel dans le dossier racine.
- Exécutez l’analyse de macro dans toutes les conditions.
- Combinez les mesures (en général au moins n=2*20) et supprimez les valeurs aberrantes (Q-test dans le Graph pad Prism v8).
- Normaliser les mesures soit au contrôle du solvant, soit au jour 1 (en fonction du type d’expérience, le premier pour une expérience d’inhibition de médicament et le second pour une expérience de cinétique de croissance). Exprimer les mesures sous forme de charge de cellules cancéreuses normalisée (axe y ) au fil du temps ou de condition (axe x ), comme le montrent respectivement la figure 3 et la figure 4.
Résultats
Nous avons fourni des instructions étape par étape pour une approche rapide et facile pour passer d’une nouvelle lignée cellulaire à son analyse. Nous commençons par la surexpression d’un traceur fluorescent à l’aide d’une cassette de surexpression lentivirale (étapes 3 et 4). Ceci est suivi d’une préparation cellulaire pour assurer le moins de volume mort possible lors de l’injection, permettant d’injecter un nombre élevé de cellules à la fois dans l’espace doC et rétro-orbital (étapes 6 et 7). Par la suite, nous effectuons l’acquisition de données à semi-haut débit à l’aide de la microscopie stéréo-fluorescente et de la microscopie confocale à plus fort grossissement pour l’analyse qualitative de la dissémination des cellules cancéreuses du corps entier (Figure 2 et étapes 10, 11 et 12). Il faut être prudent lors de l’acquisition des données, car pour assurer la reproductibilité de l’imagerie microscopique stéréo et confocale, les paramètres génériques et la normalisation sont définis (étapes 11 et 12). L’analyse des données est abordée (à l’aide d’imageJ/Fiji) 16, ainsi que la normalisation à l’aide des macros imageJ (étape 13).
À l’étape 3, nous avons mentionné le marquage transitoire des cellules (cancéreuses) pour effectuer un pré-criblage rapide afin d’évaluer le potentiel tumorigène d’une nouvelle lignée cellulaire cancéreuse. Une mise en garde importante est que, bien qu’elle soit facile à utiliser et qu’elle dure longtemps, la coloration transitoire décrite ici a la possibilité de former des artefacts (c’est-à-dire qu’il faut veiller à ce que les fragments cellulaires puissent être distingués des cellules entières, comme cela a été largement fait par Fior et ses collègues 9). D’après notre expérience, la formation de ces artefacts est directement liée à l’extrême stabilité de la coloration et à la luminosité (même après la mort cellulaire), où les fragments cellulaires sont dispersés et absorbés par les cellules immunitaires, qui pourraient par la suite être faussement conclues comme dérivant de métastases actives.
Dans les deux modèles décrits, la greffe systémique par le doC et la greffe localisée dans l’espace rétro-orbitaire, le dépistage approfondi des larves un jour après l’injection est d’une importance capitale. Comme le montre la figure 2B , toutes les larves qui présentent un déplacement mécanique des cellules greffées dans la zone de la tête (au-delà du site rétro-orbitaire) dans le modèle rétro-orbitaire et des cellules dans le sac vitellin, ou qui présentent un œdème dans le bassin injecté de docC, doivent être retirées. Tous les phénotypes sélectionnés négativement sont représentés sous forme de points confocaux à haute résolution dans la figure 2, mais peuvent être facilement vus et éliminés par observation stéréomicroscope.
Au fil du temps, les cellules migreront et proliféreront. Pour le modèle rétro-orbitaire, nous avons observé une infiltration dans les tissus voisins pour CRMM1, mais nous avons observé moins de prolifération pour CRMM2. Nous avons observé de manière frappante des métastases à distance entre 2 et 4 dpi chez certains individus (20 %), où nous avons mesuré une différence significative à 6 dpi, comme le montre la figure 4. Pour les deux lignées cellulaires, nous avons testé le potentiel prolifératif lorsqu’elles sont injectées dans les deux sites. Pour CRMM1, il y a eu une augmentation significative (p<0,0001) du nombre de cellules cancéreuses pour ou aux sites d’injection, lorsqu’elle est représentée par une charge de cellules tumorales normalisée, se normalisant jusqu’au premier jour pour chaque modèle (augmentation de 7,8 fois, ±3,2 pour le modèle RO et une augmentation de 15 fois ±8,8 pour le modèle doC). Le CRMM2 n’a pas montré de croissance significative lorsqu’il a été normalisé au premier jour pour chaque modèle individuel (augmentation de 2,4 fois, augmentation de ±1,9 et 2,3 fois, ±1,14 pour l’OI et le doC). Il a été constaté que CRMM1 prolifère facilement dans le tissu rétro-orbitaire et le tissu hématopoïétique caudal après la greffe. La lignée cellulaire CRMM2 était moins proliférative dans les deux modèles, mais il est intéressant de noter qu’elle s’est avérée capable de métastases à distance lorsqu’elle est injectée dans l’espace rétro-orbitaire, comme le montrent les figures3B et C.
Après avoir dépisté les larves injectées à 1 dpi et assigné au hasard les individus à des groupes de traitement ou de contrôle, les poissons ont été traités pendant 6 jours, en changeant l’eau contenant du Vemurafenib (cet inhibiteur peut facilement être échangé contre tout autre composé antitumoral titré). Nous avons choisi de développer le modèle de dissémination du mélanome conjonctival hématogène précédemment publié en greffant CRMM114, en testant l’efficacité du Vemurafenib sur CRMM1 orthotopique. CRMM1 a montré une forte réduction significative du groupe endogreffé ectopique traité au Vemurafenib (P<0,0001) et une réponse rabougrie mais significative pour le modèle greffé orthotopiquement (p<0,05), comme le montre la figure 4.
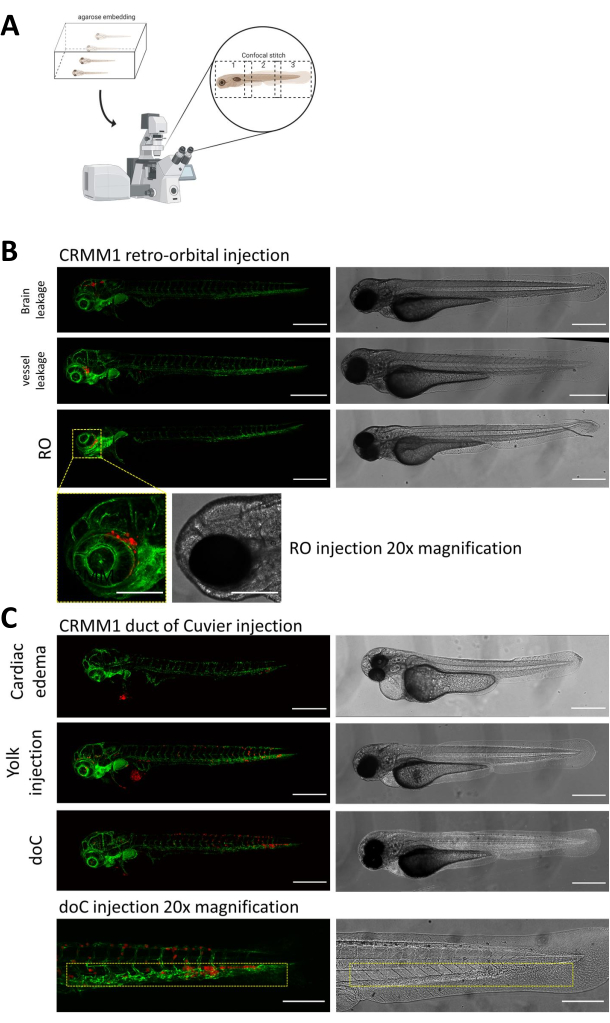
Graphique 2. Évaluation phénotypique et dépistage après injection. A) Représentation schématique de la génération de points confocaux de xénogreffe de poisson-zèbre, produisant des images homogènes à haute résolution après intégration de la projection confocale ultérieure. Ici, les xénogreffes de poisson-zèbre sont noyées dans de l’agarose à faible point de fusion à 1 % et montées sur une boîte confocale à fond de verre (comme décrit à l’étape 11.3). B) Tous les résultats possibles de la greffe rétro-orbitaire et du canal de Cuvier sont affichés injectés dans un poisson zèbre rapporteur de vaisseaux sanguins fluorescents verts (TG :fli :GFP), avec des cellules colorées par lentiviral sur l’expression de tdTomato). Nous désignons la greffe correcte à 1 dpi (panel RO) et les phénotypes indésirables (à la fois fuite cérébrale et fuite des vaisseaux sanguins). Les deux dernières populations doivent être éliminées pour s’assurer qu’elles ne confondent pas les résultats expérimentaux en aval. C) Les phénotypes indésirables pour la greffe hématogène par le canal de Cuvier (doC) sont les contours où les larves œdémateuses cardiaques (œdème cardiaque) et les larves avec des cellules fuyant dans le sac vitellin (injection de vitellin) doivent être retirées pour éviter toute interférence avec les mesures en aval. Les larves correctement injectées sont réparties dans des groupes expérimentaux tels que décrits à l’étape 7.1. (Toutes les images ont été acquises à 1 dpi, à l’aide d’un microscope confocal, barres d’échelle de 200 μm. Les cases jaunes indiquent les sites métastatiques pour les greffes d’OI et de doc, la région de la tête et le tissu hématopoïétique caudal, respectivement). Veuillez cliquer ici pour voir une version agrandie de cette figure.
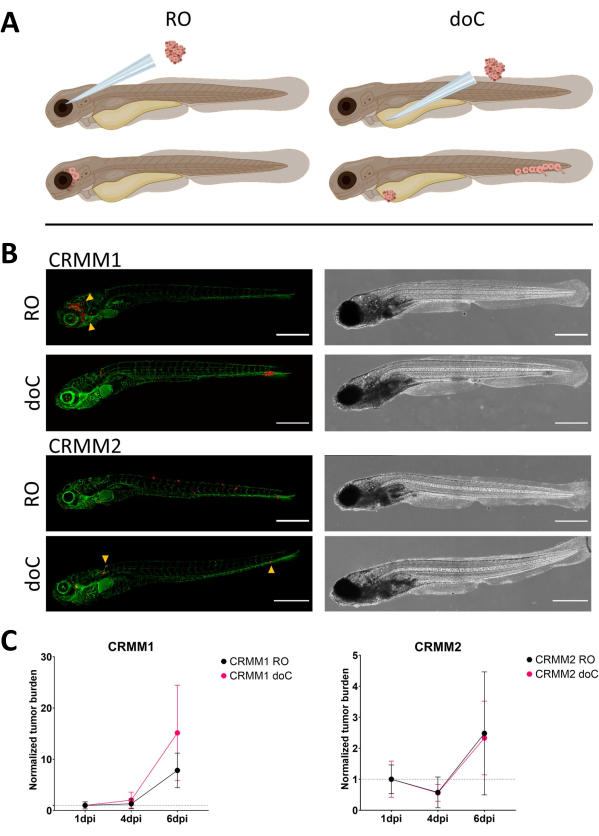
Graphique 3. L’analyse comparative des lignées cellulaires de mélanome conjonctival CRMM1 et CRMM2 montre une capacité métastatique et de croissance différentielle. A) Représentation schématique des modèles d’injection, du modèle rétro-orbitaire (RO) et du modèle de greffe hématogène (doC) les poissons utilisés sont des rapporteurs de vaisseaux sanguins verts TG(fli :GFP), avec des cellules surexprimant tdTomato indiquées en rouge. B) Phénotypes représentatifs de poissons greffés avec CRMM1 et CRMM2, CRMM1 présente une prise de greffe efficace (RO et doC) et une invasion à petite échelle dans le tissu entourant le site de greffe de l’OI (RO, pointes de flèches jaunes). CRMM2 présente une efficacité de greffe remarquablement plus faible pour les deux modèles de greffe, mais présente des métastases à distance lorsqu’il est injecté rétro-orbitaire (comme le montre l’OI, indiquée par les pointes de flèches). (Toutes les images ont été acquises à 6 dpi, microscope confocal, barres d’échelle de 200 μm. Les pointes de flèches jaunes indiquent les sites métastatiques pour les greffes d’OI et de docC, la région de la tête et le tissu hématopoïétique caudal, respectivement). C) Tracés de la greffe cinétique pour CRMM1 et CRMM2, en comparant les deux modèles de greffe au jour 1 (normalisation au jour 1), il y a une augmentation significative (p<0,0001) de la charge tumorale normalisée pour la lignée cellulaire CRMM1 (entre 1 dpi et 6 dpi) où il y a une tendance à la hausse (non significative) pour CRMM2. CRMM1 révèle une différence significative entre la croissance de l’OI et celle du doc, où le modèle doC montre un taux d’expansion tumorale plus élevé (environ 2 fois plus élevé pour les larves greffées de doC). Les graphiques affichent la moyenne et l’erreur type de la moyenne (SEM). Tous les groupes ont été normalisés à 1 dpi pour chaque condition individuelle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
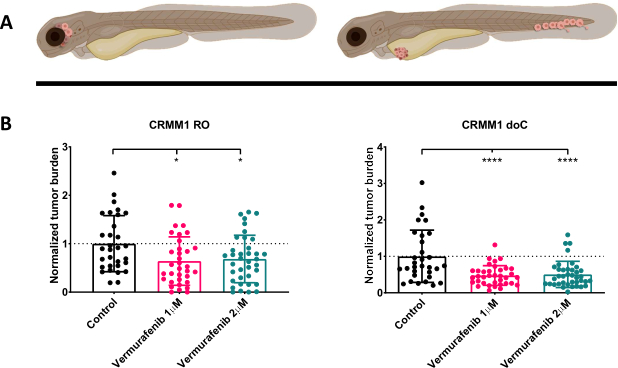
Graphique 4. L’inhibiteur de BRAF V600E, le Vemurafenib, inhibe de manière significative les larves de mélanome conjonctival greffées de l’OI et du doC. A) Représentation schématique des phénotypes du poisson-zèbre, modèles RO et doC. B) Les larves greffées RO et doC, injectées avec la lignée cellulaire de mélanome conjonctival CRMM1, présentent une réduction significative de la charge tumorale normalisée (p<0,05 et P<0,001 respectivement). Les modèles de poisson-zèbre greffé d’un ddC indiquent une réponse médicamenteuse améliorée et une relation indépendante de la dose avec l’inhibition des médicaments, indiquant une possible saturation de l’inhibition). Les graphiques montrent la moyenne et l’erreur-type de la moyenne (SEM), tous les groupes ont été normalisés pour contrôler chaque lignée cellulaire individuelle. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Réactif | Volume |
| psPAX2 | 1,71 pmol (12,14 μg) |
| pMD2.G | 0,94 pmol (3,66 μg) |
| Plasmide de transfert* | 1,64 pmol (calculer volume exact) |
Tableau 1.
Discussion
Ici, nous avons défini une approche méticuleuse pour modéliser le mélanome oculaire primaire et métastatique dans les xénogreffes de poisson-zèbre. En combinant à la fois un modèle d’injection orthotopique localisée et un modèle d’injection ectopique systémique, nous avons récapitulé l’étiologie de la cancérogenèse d’un cancer pour lequel aucun modèle animal n’était disponible auparavant. La transparence inhérente à la larve précoce du poisson-zèbre permet de suivre les cellules cancéreuses marquées par fluorescence à l’échelle de l’animal entier, assurant ainsi une visualisation facile des sites métastatiques potentiels17. De plus, l’analyse microscopique confocale à fort grossissement nous permet de suivre les cellules à une résolution subcellulairede 10.
Nous avons fourni des instructions étape par étape pour une approche rapide et facile pour passer d’une nouvelle lignée cellulaire à l’établissement de la xénogreffe et à son analyse. Nous commençons par la surexpression d’un traceur fluorescent à l’aide d’une cassette de surexpression lentivirale (étapes 3 et 4) suivie d’une préparation cellulaire pour assurer le moins de volume mort possible lors de l’injection. Cela permet d’injecter un grand nombre de cellules à la fois dans l’espace doC et dans l’espace rétro-orbital (étapes 7 et 8). Ensuite, nous effectuons l’acquisition de données à semi-haut débit à l’aide de la microscopie stéréofluorescente et de la microscopie confocale à grossissement plus élevé pour l’analyse qualitative de la dissémination des cellules cancéreuses du corps entier (figure 2 et étapes 9 et 10). Il faut être prudent lors de l’acquisition des données, car pour assurer la reproductibilité de l’imagerie microscopique stéréo et confocale, les paramètres génériques et la normalisation sont définis (étapes 11 et 12). L’analyse des données est abordée (à l’aide d’imageJ/Fiji) 16, ainsi que la normalisation à l’aide des macros ImageJ (étape 13).
À l’étape 3, nous mentionnons le marquage transitoire des cellules (cancéreuses) pour effectuer un pré-dépistage rapide afin d’évaluer le potentiel tumorigène d’une nouvelle lignée cellulaire cancéreuse. Une mise en garde importante est que, bien qu’elle soit facile à utiliser et qu’elle ait une longue durée de vie, la coloration transitoire décrite ici a la possibilité de former des artefacts (par exemple, il faut veiller à ce que les fragments de cellules puissent être distingués des cellules entières, comme cela a été largement fait par Fior et ses collègues 9). D’après notre expérience, la formation de ces artefacts est directement liée à l’extrême stabilité de la coloration et à la luminosité (même après la mort cellulaire), où les fragments cellulaires sont dispersés et absorbés par les cellules immunitaires, qui pourraient par la suite être faussement conclues comme dérivant de métastases actives.
À l’aide de ces modèles, nous avons simulé le développement d’une tumeur primaire en confinant physiquement les cellules greffées dans l’interstice rétro-orbitaire. Un dépistage approfondi ultérieur 1 jour après la greffe garantit que les cellules trouvées à un site éloigné plus tard dans l’expérience ont activement métastasé (intravasées et disséminées, pour finalement extravaser à la niche métastatique). La greffe par la doC, la veine cardinale commune embryonnaire, permet une implantation facile et hautement reproductible de grandes quantités de cellules (à un surplus de 600 cellules correctement concentrées), contournant efficacement les étapes primaires de la cascade métastatique (intravasation) et nous permettant de nous concentrer sur les étapes ultérieures de la cascade métastatique (adhésion, extravasation et excroissance). Bien qu’ils soient des outils puissants lorsqu’ils sont utilisés correctement, les deux modèles doivent être surveillés de près au cours du premier jour suivant la greffe pour s’assurer qu’aucune conclusion faussement positive n’est tirée au cours des dernières étapes de l’expérience.
Conformément aux publications précédentes, nous avons montré que les lignes de mélanome conjonctival forment facilement des colonies métastatiques après dissémination dans le système de circulation sanguine du poisson-zèbre14. Ici, nous rapportons l’élargissement du répertoire de greffe avec l’injection rétro-orbitaire comme modèle orthotopique, et la métastase active ultérieure au tissu hématopoïétique caudal de la lignée cellulaire CRMM2. Par la suite, nous rapportons l’efficacité de l’inhibiteur spécifique de BRAF V600E, Vemurafenib, également sur la forme primaire de mélanome conjonctival lorsqu’il est modélisé chez les larves de poisson-zèbre.
En utilisant les méthodes susmentionnées, un chercheur expérimenté est capable de générer plus de centaines de larves greffées par jour (environ 200 par heure) de l’un ou l’autre modèle proposé. Dans un délai de deux semaines, un médicament peut être à la fois titré pour la dose maximale tolérée et testé sur un modèle de xénogreffe établi. Du début à la fin, l’utilisation d’une lignée cellulaire non transduite, jusqu’à l’obtention d’un profil de sensibilité aux médicaments dans le modèle de poisson zèbre peut être obtenue en un mois (étant donné que la lignée cellulaire injectée est tumorigène dans le modèle de poisson zèbre). Entre nos mains, aussi peu que 20 larves par expérience et deux répétitions biologiques ont produit de manière reproductible une inhibition robuste du médicament, lorsque deux expériences individuelles entrent en conflit (ou ne produisent pas d’inhibition de croissance statistiquement significative), une troisième répétition biologique peut être effectuée.
Grâce à des ajustements mineurs, ces modèles nous ont permis d’adapter rapidement ces stratégies d’implantation pour le glioblastome (injection dans la cavité cérébrale postérieure), le cancer du sein (injection doC) et l’ostéosarcome (doC) entre autres 18,19,20,21. Ces modèles peuvent ensuite être utilisés à la fois pour la recherche fondamentale et le criblage préclinique de médicaments uniques et de stratégies médicamenteuses combinatoires. Récemment, nous avons décrit différents régimes d’administration de médicaments et leur photo-activation à l’aide de ces modèles 13.
Déclarations de divulgation
Aucun.
Remerciements
Ces travaux ont été soutenus par un financement du programme de recherche et d’innovation Horizon 2020 de l’Union européenne dans le cadre de la convention de subvention n° 667787 (projet UM Cure 2020, www.umcure2020.org). Le Conseil chinois des bourses d’études est aimablement reconnaissant pour l’octroi d’une bourse de doctorat à J.Y.
matériels
| Name | Company | Catalog Number | Comments |
| 2.5mm box filament | Science products | FB255B | for pulling micro injection needles using a Sutter P97 or P1000 |
| 3mL transfer pipettes | Merck | Z350796 | for transfer and selection of zebrafish embryos |
| Agarose | Milipore | 2120 | 1.5% (w/v) in eggwater, 1.5 g in 100 mL DPBS, microwave to dissolve, for injecting and stereofluorescence imaging of zebrafish larvae |
| Capillaries: borosilicate glass outer | World precision instruments | BF100-78-10 | Borosilicate glass capillaries used for needle preparation |
| DMSO | Sigma | D8418 | Often used as solvent in drug treatments, should be stored at 2-8°C the dark. |
| DPBS | Thermo Fischer Scientific | 14190144 | Dulbecco’s phosphate buffered saline, without Mg2+ and Ca2+ for washing the cells, lack of Ca2+ impairs cell-cell adhesion through cadherins and prevents cell aggregation during injection |
| Egg water | Instant ocean | SS15-10 | 0.6 mg/L final concentration sea salt in demineralized water |
| GFP encoding lentiviral transfer plasmid | Addgene | Plasmid #106172 | Generated in Snaar lab, available at Addgene |
| Hek293T | ATCC | CRL-3216 | Stable cell line for generating lentiviral particles, contains SV40-T antigen required for the generation of lentiviral particles |
| Leica sp8 confocal | Leica | Leica TCS SP8 | automated stage confocal microscope with 405/488/514/635nm lasers |
| LipodD293 | Signagen | SL100668 | Highly efficient HEK293t optimized transfection reagent |
| Low-melting agarose | Milipore | 2070 | 1% (w/v) in eggwater 1.5 g in 100 mL DPBS, microwave to dissolve, for embedding zebrafish larvae for confocal imaging |
| Micro loader tips | Fischer scientific | 10289651 | flexible microloader tips |
| Micro manipulator | World precision instruments | M3301R | x/y/z manual micro manipulator for microinjection |
| Needle puller: P-97 or P-1000 | Sutter | P-97 | needle puller used for generating standardized micro engraftment needles |
| Nr.5 watchmakers forceps | VWR | HAMMHSC818-11 | fine watchmakers forceps used for breaking back needles |
| Picopump | World precision instruments | SYS-PV820 | pulse controller supplying pressure for microinjection |
| pMD2.G | Addgene | plasmid #12259 | Gifted by Didier Trono, 2nd generation lentiviral virulence plasmid |
| psPAX2 | Addgene | plasmid #12260 | Gifted by Didier Trono, 2nd generation lentiviral packaging plasmid |
| PVP40 | Sigma-Aldrich | PVP40 | Polyvinylpyrrolidone average mol wt 40,000) PVP40 2% (w/v) in DPBS, 1 g PVP40 in 50 mL DPBS. Vortex and incubate at 37°C to facilitate dissolving. Store at room temperature. |
| tdTomato encoding lentiviral transfer plasmid | Addgene | Plasmid #106173 | Generated in Snaar lab, available at Addgene |
| transmitted light stereo microscope | Leica | leica M50 with (MDG33 base) | leica transmitted light microscope with mirror adjustable illumination. |
| Tricaine | Sigma-Aldrich | E10521 | Ethyl 3-aminobenzoate methanesulfonate or MS-222 |
| TryplE | Thermo Fischer Scientific | 12604-01 | Synthetic trypsine replacement, less damaging to the cells and allows for the gentle dispersion of strongly adherent cells. (Thermo- |
| willco dish | WillCo wells | GWST-5040 | 50mm glass bottom dishes, allow for the embedding of up to 20 zebrafish larvae, enabling the imaging of multiple conditions in one dish due to its large optical glass surfac |
Références
- Yang, J., Manson, D. K., Marr, B. P., Carvajal, R. D. Treatment of uveal melanoma: where are we now. Therapeutic Advances in Medical Oncology. 10, (2018).
- Wong, J. R., Nanji, A. A., Galor, A., Karp, C. L. Management of conjunctival malignant melanoma: A review and update. Expert Review of Ophthalmology. 9, 185-204 (2014).
- Nguyen, D. X., Bos, P. D., Massagué, J. Metastasis: from dissemination to organ-specific colonization. Nature Reviews Cancer. 9, 274-284 (2009).
- White, R. M., et al. Transparent Adult Zebrafish as a Tool for In Vivo Transplantation Analysis. Cell Stem Cell. 2, 183-189 (2008).
- Zon, L. I., Peterson, R. T. In vivo drug discovery in the zebrafish. Nature Reviews Drug Discovery. 4, 35-44 (2005).
- Howe, K., et al. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496, 498-503 (2013).
- Palmblad, M., et al. Parallel deep transcriptome and proteome analysis of zebrafish larvae. BMC Research Notes. 6, 428 (2013).
- Yan, C., et al. Visualizing Engrafted Human Cancer and Therapy Responses in Immunodeficient Zebrafish. Cell. 177, 1903-1914 (2019).
- Fior, R., et al. Single-cell functional and chemosensitive profiling of combinatorial colorectal therapy in zebrafish xenografts. Proceedings of the National Academy of Sciences of the United States of America. 114, 8234-8243 (2017).
- Campbell, P. D., Chao, J. A., Singer, R. H., Marlow, F. L. Dynamic visualization of transcription and RNA subcellular localization in zebrafish. Development. 142, 1368-1374 (2015).
- Campeau, E., et al. A Versatile Viral System for Expression and Depletion of Proteins in Mammalian Cells. PLoS One. 4, 6529 (2009).
- vander Helm, D., et al. Mesenchymal stromal cells prevent progression of liver fibrosis in a novel zebrafish embryo model. Scientific Reports. 8, 16005 (2018).
- Chen, Q., et al. TLD1433 photosensitizer inhibits conjunctival melanoma cells in zebrafish ectopic and orthotopic tumour models. Cancers. 12, (2020).
- Pontes, K. C. d. e. S., et al. Evaluation of ( fli:GFP ) Casper Zebrafish Embryos as a Model for Human Conjunctival Melanoma. Investigative Opthalmology & Visual Science. 58, 6065 (2017).
- Liverani, C., et al. Innovative approaches to establish and characterize primary cultures: an ex vivo 3D system and the zebrafish model. Biology Open. 6, 133-140 (2017).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9, 676-682 (2012).
- White, R. M., et al. Transparent Adult Zebrafish as a Tool for In Vivo Transplantation Analysis. Cell Stem Cell. 2, 183-189 (2008).
- Mercatali, L., et al. Development of a patient-derived xenograft (PDX) of breast cancer bone metastasis in a Zebrafish model. International Journal of Molecular Sciences. 17, (2016).
- Tulotta, C., et al. Imaging cancer angiogenesis and metastasis in a zebrafish embryo model. Advances in Experimental Medicine and Biology. 916, 239-263 (2016).
- Paauwe, M., et al. Endoglin expression on cancer-associated fibroblasts regulates invasion and stimulates colorectal cancer metastasis. Clinical Cancer Research. 24, 6331-6344 (2018).
- Cao, J., et al. Overexpression of EZH2 in conjunctival melanoma offers a new therapeutic target. Journal of Pathology. 245, (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon