Method Article
Reconstitution de cortex minimembranaires d’actine sur des bicouches lipidiques soutenues
Dans cet article
Résumé
Ce protocole décrit la formation de bicouches lipidiques soutenues et l’ajout de filaments cytosquelettiques et de protéines motrices pour étudier la dynamique de réseaux cytosquelettiques reconstitués et attachés à la membrane à l’aide de la microscopie à fluorescence.
Résumé
La surface d’une cellule vivante fournit une plate-forme active polyvalente pour de nombreux processus cellulaires, qui découlent de l’interaction de la membrane plasmique avec le cortex d’actine sous-jacent. Au cours des dernières décennies, des systèmes minimaux reconstitués basés sur des bicouches lipidiques soutenues en combinaison avec des réseaux de filaments d’actine se sont révélés très utiles pour démêler les mécanismes de base et les conséquences des réseaux d’actine attachés à la membrane, ainsi que pour étudier les fonctions des protéines individuelles associées à la membrane. Ici, nous décrivons comment reconstituer de tels systèmes composites actifs in vitro qui consistent en des bicouches lipidiques soutenues par un fluide couplées via des protéines de liaison à l’actine associées à la membrane à des filaments d’actine dynamiques et des moteurs de myosine qui peuvent être facilement observés par microscopie à fluorescence par réflexion interne totale. Une conception à chambre ouverte permet d’assembler le système étape par étape et de contrôler systématiquement de nombreux paramètres tels que la concentration de protéines de liaison, la concentration d’actine, la longueur du filament d’actine, le rapport actine / myosine, ainsi que les niveaux d’ATP. Enfin, nous discutons de la façon de contrôler la qualité du système, de la façon de détecter et de résoudre les problèmes courants et de certaines limites de ce système par rapport à la surface de la cellule vivante.
Introduction
La membrane plasmique d’une cellule animale vivante interagit constamment avec le cytosquelette d’actine adjacent, et ensemble ils forment un matériau composite actif qui remplit une multitude de fonctions cellulaires 1,2. Pour étudier les processus à cette interface membrane lipidique-actine, la reconstitution des réseaux cytosquelettiques au-dessus des bicouches lipidiques (SLB) prises en charge s’est avérée très utile. Cette approche systémique minimale permet le contrôle précis des composants du réseau du cytosquelette et de la composition lipidique. Par rapport aux membranes lipidiques autonomes des vésicules unilamellaires géantes, la géométrie plane des SLB permet une utilisation efficace des techniques de microscopie de pointe telles que la super-résolution3,4, la fluorescence par réflexion interne totale (TIRF)5,6,7 ou la diffusion interférométrique 8 étudier l’organisation spatiale et la dynamique des réseaux cytosquelettiques. TIRF fournit le contraste le plus élevé pour les composants marqués par fluorescence, car le signal des molécules marquées non liées dans la solution contribuant au signal de fond est minime.
Nous décrivons ici un protocole de base pour la formation de réseaux d’actomyosine attachés à des bicouches lipidiques supportées, qui sont largement utilisés dans le domaine pour étudier la physique des réseaux actifs quasi-2D 9,10,11 et leur effet sur l’organisation membranaire3,5,12,13,14,15,16 (Figure 1 ). Cette approche ne se limite pas aux réseaux à base d’actine, mais peut également être facilement adaptée pour explorer des microtubules, des filaments intermédiaires ou des réseaux composites de nature mixte et pour étudier une variété d’interactions entre les protéines membranaires lipidiques et les composants du cytosquelette à l’aide de méthodes de microscopie sensibles à la surface.
Pour garder ce protocole ciblé, nous avons exclu une description détaillée de la purification et du marquage des protéines d’actine et de myosine ou des détails sur la façon de régler et de contrôler la contractilité et l’organisation des réseaux d’actomyosine. Il convient de se référer à d’autres protocoles publiés parallèlement à celui-ci dans la collection de méthodes JoVE, In Vitro Reconstitution of Cytoskeleton Networks for Biomaterials, Biophysics and Active Matter Research17.

Figure 1 : Schéma du système composite actif actine-membrane in vitro. Créé avec Biorender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Réactifs et équipements
- Préparez de nouveaux tampons comme indiqué dans le tableau 1. Utilisez de l’eau désionisée ultrapure avec une résistivité de 18,2 MΩ·cm à 25 °C. Stérilisez tous les tampons en les faisant passer à travers des filtres de 0,22 μm sous vide. Dégazer les tampons utilisés pour la chromatographie sur colonne.
- Purifier l’actine des muscles squelettiques comme décrit précédemment18,19. Ajouter 20% de glycérol à la solution finale purifiée de G-actine et faire des aliquotes de 500 μL (pour le marquage ou les expériences en vrac) et 10 μL (pour les expériences individuelles) volume. Congeler les aliquotes en trempant les tubes dans de l’azote liquide pendant 30 s, puis les conserver à −80 °C pendant 18 mois maximum.
REMARQUE: Alternativement, l’actine purifiée ou la poudre d’acétone peut être achetée dans le commerce. - Étiqueter la G-actine du muscle squelettique purifié avec tout colorant maléimide fluorescent tel que décrit précédemment5. Déterminer la concentration et le degré de marquage de la protéine par spectrophotométrie en utilisant A290 nm corrigé pour l’actine (εactine = 26 600 M-1 cm-1) et A λmax du colorant. Faire des aliquotes de 10 μL et congeler en trempant les tubes dans de l’azote liquide pendant 30 s et conserver à −80 °C jusqu’à 18 mois.
REMARQUE: L’étiquetage avec des esters NHS conjugués à la lysine créera de l’actine non fonctionnelle et devrait être évité. - Purifiez la myosine II des muscles squelettiques en suivant le protocole20. Exécutez SDS-PAGE en utilisant 10% de gel de polyacrylamide suivi d’une coloration Coomassie pour déterminer le niveau de pureté de la protéine21. Conservez la myosine-II des muscles squelettiques purifiés à -20 °C sous forme liquide dans un tampon de myosine II contenant 50 % de glycérol.
REMARQUE: La myosine II stockée peut être utilisée jusqu’à 2 ans. - Étiqueter la myosine-II purifiée avec tout colorant maléimide fluorescent tel que décrit précédemment5. Évitez d’étiqueter les moteurs de myosine avec des colorants NHS-esters. Déterminer la concentration et le degré de marquage par spectrophotométrie en utilisant A280 nm corrigés de myosine II et Aλmax du colorant. Conservez la myosine II recyclée (foncée ou étiquetée) à 4 °C et utilisez-la dans les 6 semaines.
- Purification de la protéine de coiffage
- Obtenir la protéine de coiffage murin en suivant un protocole antérieur22. Exécutez SDS-PAGE en utilisant un gel de polyacrylamide à 10% suivi d’une coloration Coomassie pour déterminer le niveau de pureté de la protéine. Mesurer la concentration à l’aide de A280 nm de protéine de coiffage (εCP = 99 530 M-1 cm-1).
- Ajouter 20% de glycérol à la solution protéique et faire 5 μL aliquotes dans des tubes PCR de 200 μL. Plongez les tubes dans de l’azote liquide et conservez-les à -80 °C jusqu’à 2 ans.
REMARQUE: L’activité de la protéine de coiffage est vérifiée en polymérisant des quantités fixes de G-actine fluorescente en présence de différentes quantités de protéines de coiffage. Les filaments sont ensuite imagés au microscope et leur distribution de longueur est quantifiée. Plus la concentration relative de protéine de coiffage est élevée, plus les distributions de filaments d’actine sont courtes. Voir Köster et al.5.
- Exprimer une protéine de liaison membrane-actine fluorescente, par exemple, pour ce protocole, utiliser 10xHis-YFP-EzrinABD (HYE), l’exprimer dans Bl21DE3* Escherichia Coli et purifier comme décrit précédemment23. Déterminer la concentration de la protéine par spectrophotométrie.
- Conserver la protéine dans de petites aliquotes dans un tampon de chromatographie par filtration sur gel (ou tout autre tampon approprié) contenant 20 % de glycérol à −80 °C. Dans ces conditions, la protéine est stable pendant plus de 2 ans.
REMARQUE: Le choix de la protéine de liaison actine-membrane et du marqueur fluorescent dépend du type de question que l’on aborde. Un large éventail de stratégies de liaison lipidique ont été développées au cours des dernières années, notamment les protéines marquées à l’histidine24, la biotine-streptavidine 25 et l’ADN simple brin26. - Préparation des vésicules multi-lamellaires (MLV)
REMARQUE: Le flux de travail des MLV aux bicouches lipidiques prises en charge est illustré à la figure 2.- Placer 5 à 10 flacons en verre ambré dans un bécher en verre de 200 ml. Remplissez le bécher avec une solution de nettoyage à 2%, juste assez pour immerger les flacons en verre. Soniquez-les au bain-marie pendant 30 min à pleine impulsion et à 65 °C.
- Sortez les flacons de la solution et rincez-les abondamment à l’eau distillée. Placer les flacons dans un bécher en verre contenant 2 N NaOH et soniquer pendant 20 min. Aucun chauffage n’est nécessaire pendant cette étape.
- Sortez les flacons de la solution de NaOH et rincez abondamment à l’eau distillée. Sécher les flacons à l’intérieur d’un four à air chaud réglé à 65 °C pendant 2 h ou plus.
- Conservez les flacons nettoyés dans un bécher propre scellé avec un film transparent jusqu’à 6 semaines.
ATTENTION : Effectuez les étapes suivantes à l’intérieur d’une hotte chimique. Manipulez le chloroforme et les solutions lipidiques avec des seringues en verre Hamilton étanches aux gaz pour éviter la contamination par le plastique. - Rincer plusieurs fois les seringues Hamilton et quelques flacons en verre ambré avec du chloroforme pur. Prélever la poudre lipidique stockée dans des ampoules de verre du congélateur à −20 °C et ajouter des volumes adéquats de chloroforme pour dissoudre les poudres lipidiques à des concentrations de 10 à 25 mg/mL.
- Transférer la solution de l’ampoule dans un flacon en verre ambré fraîchement nettoyé et étiqueter. Effectuez cette étape sur la glace pour réduire l’évaporation du chloroforme.
- Préparer une solution mère DOPC à une concentration de 10-25 mg/mL et DGS-NTA-Ni2+ avec une concentration de 1-10 mg/mL.
- Pour faire un mélange lipidique fonctionnel, prenez un flacon en verre propre et rincez-le 2x avec du chloroforme. Ajouter 300 μL de chloroforme pur au flacon pour servir de base pour un meilleur mélange des composants. Cela n’affectera pas les concentrations finales des lipides car tout le chloroforme sera séché dans les prochaines étapes.
- Ajouter les volumes mesurés de solutions lipidiques mères dans le flacon pour obtenir les mélanges lipidiques de travail souhaités. La concentration lipidique cible dans le tampon de réhydratation lipidique est de 4 mM. Sécher le mélange lipidique sous un courant lent de gazN2 à l’intérieur de la hotte chimique à température ambiante. Cette étape peut prendre jusqu’à 30 minutes pour chaque flacon.
- Une fois que tout le solvant a séché, dessécher sous vide le film lipidique pendant >2 h à température ambiante pour éliminer toute trace de chloroforme laissée. Resuspendre le mélange lipidique desséché dans un tampon de réhydratation lipidique pour une concentration lipidique finale de 4 mM.
- Incuber pendant 5-10 min pour permettre la réhydratation des lipides. Vortex la solution lipidique pendant environ 30 s pour former des MLV.
- Fabriquer des aliquotes de 0,5 à 1 mL des MLV dans des tubes microcentrifugés de 1,5 mL. Plonger les tubes dans de l’azote liquide, sceller avec un film transparent et conserver à −20 °C (jusqu’à 6 semaines).
REMARQUE : Les concentrations du stock de lipides sont choisies de manière à permettre des volumes suffisamment importants pour permettre un pipetage fiable à l’aide des seringues Hamilton. Si les volumes nécessaires à la fabrication du stock sont trop importants pour dissoudre la poudre lipidique séchée, procéder à de multiples dilutions du stock pour assurer un mélange reproductible de divers lipides.
- Préparation de petites vésicules unilamellaires (VUS)
- Retirer une partie aliquote des MLV de l’entreposage à −20 °C et la décongeler à température ambiante. Congeler les vésicules en plongeant le tube de microcentrifugation dans de l’azote liquide pendant 15-30 s et le mettre immédiatement dans un bain-marie réglé à 45 °C jusqu’à ce que la solution ait complètement décongelé (1-2 min). Répétez le cycle de gel-dégel ci-dessus 10x-15x jusqu’à ce que la solution semble moins trouble.
REMARQUE: Réglez la température du bain-marie plus élevée que la température de transition du mélange lipidique en cours de décongélation pour permettre un mélange lipidique uniforme. - Équilibrer une mini-extrudeuse à base de seringue équipée d’une membrane filtrante en polycarbonate de taille de pores de 80 nm avec tampon de réhydratation SUV. Assurez-vous qu’il n’y a pas de fuite ou de bulles dans le système. Alors que la méthode d’extrusion donne des VUS monodispersés avec un minimum de dommages lipidiques, les mélanges lipidiques avec une charge négative peuvent adhérer à la membrane en polycarbonate.
- Faites passer doucement la solution lipidique décongelée à travers l’extrudeuse prééquilibrée d’un côté à l’autre, puis inversement. Répétez le cycle 5x-10x jusqu’à ce que la solution lipidique devienne visiblement claire, indiquant la formation de SUV avec ~100 nm de diamètre.
- Centrifuger la suspension extrudée (ou soniquer la solution en pointe; voir note ci-dessous) à 15 000 x g pendant 60 min à 4 °C pour enduire les débris lipidiques. Recueillir les 80% supérieurs de la solution sans déranger la pastille et sans créer de bulles. Transférer le surnageant contenant les VUS dans un tube microcentrifuge neuf et le conserver sur de la glace jusqu’à 6 jours.
REMARQUE: Une alternative à la centrifugation est la sonication de pointe effectuée comme suit. Allumez un sonicateur microtip et définissez les paramètres suivants : Amplitude = 30% du maximum, ON temps = 10 s, OFF time = 60 s. Nettoyez l’extrémité du micro-sonicateur avec de l’eau désionisée suivie de 2 N NaOH, du chloroforme et de l’eau désionisée. Trempez la pointe du sonicateur dans chacune de ces solutions et sonicez pendant 1-2 cycles en utilisant les paramètres ci-dessus. Trempez l’extrémité propre dans la solution de vésicules congelées et soniquez pendant 3 à 6 cycles sur la glace jusqu’à ce que la solution devienne claire. - Après la centrifugation, vérifier les signes d’une dégradation élevée des lipides ou d’une irruse lipidique échouée comme la formation d’une fine pellicule blanchâtre et / ou d’une pastille clairement visible. Dans ces cas, ne continuez pas et répétez les étapes de préparation du VUS.
REMARQUE: La durée de conservation des VUS peut différer pour différents mélanges lipidiques. Les SUV en DOPC: DGS-NTA-Ni2+ sont stables jusqu’à 6 jours pour les besoins de ces expériences. Vous trouverez des conseils pour résoudre les problèmes courants dans le tableau 2.
- Retirer une partie aliquote des MLV de l’entreposage à −20 °C et la décongeler à température ambiante. Congeler les vésicules en plongeant le tube de microcentrifugation dans de l’azote liquide pendant 15-30 s et le mettre immédiatement dans un bain-marie réglé à 45 °C jusqu’à ce que la solution ait complètement décongelé (1-2 min). Répétez le cycle de gel-dégel ci-dessus 10x-15x jusqu’à ce que la solution semble moins trouble.
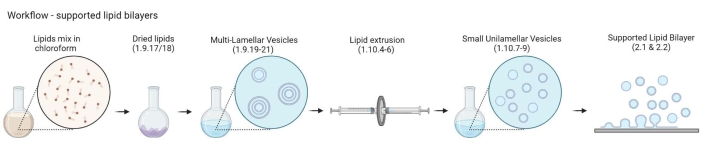
Figure 2 : Schéma montrant le flux de travail depuis la préparation des vésicules multilamellaires et des petites vésicules unilamellaires jusqu’à la formation de bicouches lipidiques soutenues. Créé avec Biorender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Reconstitution de réseaux d’actine attachés à la membrane
- Préparation des chambres d’échantillonnage
- Prenez 3 à 5 lamelles rectangulaires en verre et placez-les dans un pot Coplin. Allumez le sonicateur de bain et réglez la température à 65 °C. Remplissez le pot Coplin avec une solution de nettoyage à 2% pour immerger complètement les lamelles de couverture et placez-le dans le sonicateur pendant 30 minutes en mode pleine impulsion.
- Utilisez des pinces émoussées enduites de PTFE pour retirer les lamelles de couvercle une par une du pot. Rincez-les abondamment à l’eau distillée et placez-les dans un autre pot Coplin rempli de 2 N NaOH.
- Sonicer les lamelles de couverture pendant 20 min en mode pleine impulsion. Retirez les lamelles de couverture une par une, rincez abondamment à l’eau distillée et placez-les dans un autre pot Coplin rempli d’eau distillée.
REMARQUE: Si vous le souhaitez, soniquez les lamelles de couverture dans de l’eau distillée pendant 20 minutes, puis rincez-les à nouveau à l’eau distillée. - Immédiatement avant de commencer l’expérience, prendre le pot contenant les lamelles de couverture dans une hotte chimique équipée d’une alimentation en gazN2 .
- Optimiser la pression d’air du flux de gaz N2 par essais et erreurs afin qu’il soit juste suffisant pour déplacer l’eau de la surface de la glissière de recouvrement sans la casser. Aligner le flux de gazN2 parallèle au plan de glissement de couverture pour réduire la possibilité de casser le couvercle.
- Utilisez des gants et des pinces pour retirer les lamelles de couvercle une par une du pot afin de les sécher sous le jet N2 . Séchez les deux côtés de chaque lamelle de couverture et placez-les sur une grille en plastique propre avec un couvercle. Placez la boîte avec les lamelles de couverture dans un dessiccateur pour éviter tout contact avec des particules de poussière dans l’air.
NOTE: Les lamelles de couverture séchées N 2 peuvent être stockées dans un dessiccateur où elles peuvent rester hydrophiles jusqu’à2 jours. Cette stratégie peut être utile lorsque de nombreuses bicouches sont nécessaires pour l’expérience ou si l’expérience dure plus de 8 heures. - Prenez des tubes PCR autoclavés et découpez leurs couvercles et leurs moitiés coniques inférieures avec une lame chirurgicale tranchante. Prenez les tubes cylindriques demi-coupés un par un, appliquez un adhésif durcissable aux UV sur le bord lisse de chaque tube coupé et placez-le inversé sur un couvercle fraîchement nettoyé de sorte que le bord repose à plat sur le couvercle.
- Ne déplacez pas le cylindre latéralement une fois qu’il est positionné sur la glissière de couverture pour vous assurer que la colle ne se répand pas dans l’espace central de la chambre. Les lamelles rectangulaires peuvent accueillir confortablement jusqu’à trois chambres de réaction, et les chambres rondes ne peuvent en accueillir qu’une seule au centre (Figure 1).
- Placez les lamelles de recouvrement du palier de chambre à l’intérieur d’un nettoyeur à ozone UV avec une alimentation en O2 et un aspirateur (ou utilisez un illuminateur UV). Allumez la lumière UV et allumez pendant 3-5 minutes pour permettre à l’adhésif de polymériser. Effectuez un éclairage plus long (10-15 min) pour améliorer l’hydrophilie du verre de couverture et, par conséquent, la qualité de la bicouche lipidique.
- Rangez les chambres d’échantillonnage sèches éclairées par UV jusqu’à 8 heures dans de petites boîtes en plastique (telles que des boîtes rectangulaires vides) enveloppées dans un film transparent pour réduire le contact avec les particules de poussière dans l’air.
REMARQUE: Un flux constantd’O2 en présence de lumière UV forme des radicaux d’ozone et d’oxygène qui peuvent éliminer les impuretés organiques de la surface de la lamelle de couverture. Un vide empêchera la fuite d’ozone toxique qui se forme pendant le processus. - Retirez les lamelles de recouvrement et testez les chambres pour détecter les fuites en les remplissant d’eau distillée. Chaque chambre peut contenir jusqu’à ~150 μL d’échantillon. Jetez les chambres qui fuient.
REMARQUE: Une autre option de nettoyage excellente et sûre est le nettoyant plasma. Les réglages de temps et de puissance dépendent du modèle, mais assurez-vous de ne pas trop traiter les lames de verre avec du plasma, car cela entraînerait une réduction de la mobilité des lipides. Le traitement de surface peut affecter la mobilité des lipides27, comme cela a été observé avec un traitement prolongé avec la solution nettoyante (>45 min) ou NaOH (>30 min).
- Préparation des bicouches lipidiques soutenues
- Lavez chaque chambre avec un tampon de formation SLB (ou 1x PBS) pour éliminer tout contaminant de surface, en laissant 100 μL de tampon à la fin. Marquez le niveau du tampon à 100 μL avec un marqueur permanent pour suivre de manière reproductible les changements de volume.
- Ajouter 2 μL de CaCl2 0,1 M dans la chambre. Cela améliore l’adsorption des vésicules à la surface du verre, améliorant la formation de bicouches à l’étape suivante. Ajouter 8 μL de la solution SUV (à partir de l’étape 1.10.) dans chaque chambre et incuber pendant 15 min à 25 °C.
NOTE: Le volume du mélange SUV à ajouter peut être estimé en calculant le nombre total de lipides (avec une surface moyenne de 0,72 nm2) qui sont nécessaires pour couvrir complètement la zone hydrophile exposée du puits avec deux couches lipidiques. - Lavez les vésicules non liées avec un tampon de motilité de l’actine (1x KMEH). Tout d’abord, retirez 50 μL du tampon de formation SLB, ne laissant que 50 μL dans la chambre d’échantillonnage. Deuxièmement, ajoutez 100 μL de 1x KMEH dans la chambre. Mélanger délicatement puis retirer 100 μL du tampon sans toucher le fond.
REMARQUE: Il est important d’être doux pendant le lavage. Assurez-vous que l’embout de la pipette ne touche pas le fond de la chambre. Gardez la pipette inclinée pour diriger le flux de tampon vers la paroi de la chambre et non directement vers la bicouche, car un écoulement direct peut perturber la bicouche. Veillez à ne pas introduire de bulles d’air pendant le pipetage, car l’air peut atteindre la bicouche lipidique et causer des défauts. - Répétez les lavages 10x en ajoutant 100 μL de 1x KMEH et en enlevant 100 μL.
- Ajouter 10 μL de caséine β 1 mg/mL à la bicouche, mélanger doucement et incuber pendant 5 à 10 min. La caséine β bloque les régions de la lamelle de couverture où la bicouche ne s’est pas formée. Laver la caséine β 3x avec 1x KMEH comme décrit à l’étape 2.2.3. et ramener le niveau tampon à la marque de 100 μL.
- Ajout d’un agent de liaison membrane-actine
- Pendant l’incubation de la β-caséine (étape 2.2.5.), retirez une partie aliquote de la protéine de liaison membrane-actine à −80 °C, décongelez-la rapidement à 37 °C, puis conservez-la sur la glace. Diluer l’aliquote avec un tampon de dilution protéique à une concentration de 1 μM.
- Ajouter la protéine de liaison à une concentration finale définie (généralement 5-20 nM) et mélanger doucement. Pour assurer un équilibre rapide de la protéine dans la chambre, ajoutez des volumes supérieurs à 20 μL en prémélangeant la protéine de liaison avec 1x KMEH.
- Incuber pendant 40 min à température ambiante. Lavez 3x avec 1x tampon KMEH pour éliminer la protéine HSE non liée (comme à l’étape 2.2.3.). Ramener le niveau tampon dans chaque chambre à la marque de 100 μL. L’échantillon est maintenant prêt pour l’imagerie.
- Évaluation de la qualité de la bicouche lipidique
Remarque : Il s’agit d’une étape facultative qui ne doit pas être effectuée à chaque fois. Nous recommandons que cette évaluation soit effectuée chaque fois que de nouveaux VUS sont fabriqués à partir de stocks MLV congelés.- Allumez le microscope, les lasers d’excitation et les caméras de détection. Assurez-vous que le laser est aligné, que l’objectif est nettoyé et que le logiciel est prêt à acquérir des images.
- Mettez de l’huile sur l’objectif 100x, montez l’échantillon sur la platine du microscope et concentrez l’objectif sur la bicouche. Assurez-vous que la position du laser est telle qu’il subisse une réflexion interne totale sur l’échantillon. Utilisez un laser d’excitation de 488 nm pour vérifier la distribution de l’intensité de fluorescence du 10xHis-YFP-EzrinABD lié à la bicouche.
NOTE: Les bicouches de bonne qualité montrent une distribution uniforme et à grande échelle de l’intensité de fluorescence. Les mauvaises bicouches montrent des taches fluorescentes intenses et inégales. - Pour déterminer l’intégrité de la bicouche, effectuez un test FRAP.
- Sélectionnez une région d’intérêt sur la bicouche et enregistrez quelques images du champ de vision en utilisant des conditions d’imagerie qui fournissent un rapport signal sur bruit de 5:1 ou plus. Mettez l’enregistrement en pause et fermez le diaphragme de champ du microscope TIRF pour concentrer un faisceau laser concentré sur une petite région circulaire de la bicouche afin de blanchir localement les fluorophores.
- Allumez le laser à sa sortie maximale pour photoblanchir la petite région pendant 3-10 s, puis éteignez le laser. Rouvrez le diaphragme de champ à son rayon d’origine, réajustez l’état d’imagerie aux paramètres (pré-blanchiment) et reprenez immédiatement pour enregistrer la récupération du signal fluorescent dans le champ de vision.
- Vérifiez si la bicouche est fluide. Les bonnes bicouches avec une diffusion latérale normale se rétablissent rapidement, tandis que les mauvaises bicouches se rétablissent lentement ou ne récupèrent pas du tout (Figure 3). Si la bicouche ne se rétablit pas, consultez la section de dépannage et redémarrez. Enregistrez les images en tant que fichiers TIFF 16 bits. Pour une estimation quantitative du coefficient de diffusion, consultez l’étape 3. sous.
- Polymérisation de l’actine fluorescente
REMARQUE: Pour gagner du temps, commencez à polymériser l’actine pendant le temps d’incubation de la protéine HSE se liant à la bicouche (étape 2.3.) ou pendant l’évaluation de la qualité de la bicouche (étape 2.4.).- Mélangez la G-actine non marquée et marquée par fluorescence dans un rapport molaire de 10:1 et complétez-la avec du G-Buffer de sorte que la concentration de G-actine soit de 20 μM. La concentration à laquelle l’actine est finalement polymérisée sera de 1/4 de cette valeur. Ajouter 1/10 de tampon ME 10x au mélange pour obtenir une solution 1x et incuber pendant 2 min. Cette étape remplace les ions Ca 2+ liés à la G-actine par des ions Mg2+. Assurez-vous que le volume final est en multiples de 10 μL.
- Ajouter la quantité désirée de protéine de capping comme suit. Décongelez rapidement un flacon de bouillon de protéines à 37 °C, puis conservez-le sur la glace. Diluer avec du tampon G de telle sorte que la concentration de la protéine de coiffage soit maintenant deux fois la concentration finale souhaitée dans le mélange de polymérisation. Ajouter un volume égal de la solution de protéine de coiffage diluée au mélange d’actine à partir de l’étape 2.5.1.
- Enfin, ajoutez un volume égal de tampon cible 2x frais au mélange réactionnel. Le volume final de la solution doit être quatre fois le volume du mélange d’actine à la fin de l’étape 2.5.2. S’assurer que la concentration finale de KMEH est de 1x, d’ATP est de 1 mM, de BSA est de 1 mg/mL et de G-actine est de 5 μM.
Incuber dans l’obscurité à 25 °C pendant 45-60 min pour permettre la polymérisation.
NOTE: C’est ce qu’on appelle la stratégie de tampon cible, dans laquelle un volume de Mg 2+ G-actine (étape 2.5.1.) est mélangé avec un volume de mélange de protéines de coiffage (étape 2.5.2.) et deux volumes de tampon cible2x (étape 2.5.3.). Cela facilite l’augmentation ou la réduction de la quantité d’actine et la modification de la concentration relative de la protéine de coiffage (ou de tout autre modulateur de l’actine; Graphique 4).
- Ajout de filaments d’actine fluorescents
- Coupez quelques pointes de 200 μL avec une lame tranchante ou des ciseaux pour les rendre émoussées. Extraire doucement à la pipette le volume requis d’actine polymérisée de 5 μM (à partir de l’étape 2.5.3.) à l’aide d’une pointe de pipette émoussée (pour éviter le cisaillement des filaments d’actine) et l’ajouter à un tube PCR autoclavé propre.
- Ajouter 1x KMEH au tube pour faire le volume >20 μL et mélanger doucement pour éviter le cisaillement de la F-actine. De la chambre d’échantillonnage montée, retirez un volume égal du tampon.
- Ajouter la solution d’actine polymérisée dans la chambre et pipeter doucement de haut en bas 3x sans toucher la bicouche au fond. Cela permet aux filaments d’actine de se répartir uniformément sur la bicouche. Montez l’échantillon sur le microscope TIRF (voir les étapes 2.4.1 et 2.4.2.).
- On peut enregistrer le processus de liaison de la F-actine à la bicouche. Incuber pendant 20-30 min. Enregistrez quelques images de différents champs de vision après que l’ajout de F-actine ait atteint un état stable. Observer le changement dans l’organisation spatiale de 10xHis-YFP-EzrinABD avant (homogène) et après l’organisation de l’actine.
REMARQUE: HYE est uniformément réparti sur la bicouche lipidique en l’absence d’actine. Lors de l’ajout de filaments d’actine, HYE colocalise avec F-actine. L’étendue de la colocalisation dépend de l’affinité de liaison à l’actine de la protéine de liaison; plus l’affinité est forte, plus la colocalisation est élevée et plus la mobilité latérale de la protéine de liaison est lente (Figure 5).
- Ajout de myosine II
- Après 30 minutes d’incubation d’actine, remontez l’échantillon sur le microscope (s’il n’a pas été monté). Vérifiez le signal dans les canaux de la protéine de liaison et de l’actine F. Ajustez les conditions d’imagerie si nécessaire.
- Sélectionnez une bonne région avec un signal de protéine de liaison uniforme et des filaments d’actine uniformément dispersés et aucun artefact pour un enregistrement en accéléré de longue durée. Enregistrez 10 à 15 images à 0,1-0,2 Hz avant l’ajout de myosine et mettez l’enregistrement en pause. Extraire à la pipette le volume requis de myosine-II musculaire recyclée du flacon brut à l’aide d’une pointe de pipette émoussée (pour empêcher le cisaillement des filaments de myosine) et l’ajouter à un tube PCR autoclavé propre.
- Ajouter immédiatement 1x KMEH dans le tube pour obtenir un volume de >20 μL et mélanger doucement. On peut également ajouter de l’ATP, un mélange régénérant de l’ATP, des agents photostabilisants, etc. au cours de cette étape. Retirez soigneusement un volume égal du tampon de la chambre d’échantillonnage montée sans la déranger.
- Ajouter délicatement la solution de myosine dans la chambre d’échantillonnage. Ne pas pipeter de haut en bas car cela perturberait les filaments liés à la surface. Reprenez immédiatement l’enregistrement accéléré et observez le système alors qu’il évolue de l’état pré-myosine aux flux contractiles d’acto-myosine alimentés par l’ATP et à la formation d’aster à un état de brouillage appauvri en ATP (voir les résultats représentatifs).
- Prenez des images d’arrière-plan pour tous les canaux à l’aide d’un échantillon de mémoire tampon uniquement. Enregistrez toutes les images en tant que fichiers .tiff 16 bits. Consultez le tableau 2 pour obtenir des conseils sur la résolution des problèmes courants.

Figure 3 : Évaluation de la qualité des bicouches avec dosage rapide du FRAP. Les bicouches lipidiques (SLB) prises en charge préparées à partir de lipides DOPC et Ni-NTA (98:2 mol%) sont recouvertes de HYE (linker membrane-actine marqué 10xHis-YFP). Une fois la protéine non liée éliminée, la bicouche fluorescente est imagée au microscope TIRF. Une petite région sur la bicouche est photoblanchie avec une puissance laser élevée, et la récupération de la fluorescence est enregistrée. (A) Une bonne bicouche récupère toujours rapidement, avec un coefficient de diffusion attendu de 1-1,5μm2/s pour la composition lipidique utilisée dans ce cas. (B) Les bicouches défectueuses se rétablissent très lentement ou ne se rétablissent pas du tout. (C) Images représentatives de bicouches défectueuses : (C-i) une bicouche avec des trous, (C-ii) une bicouche avec de grandes plaques lipidiques immobiles, et (C-iii) une bicouche avec de petits points immobiles. Veuillez cliquer ici pour voir une version agrandie de cette figure.
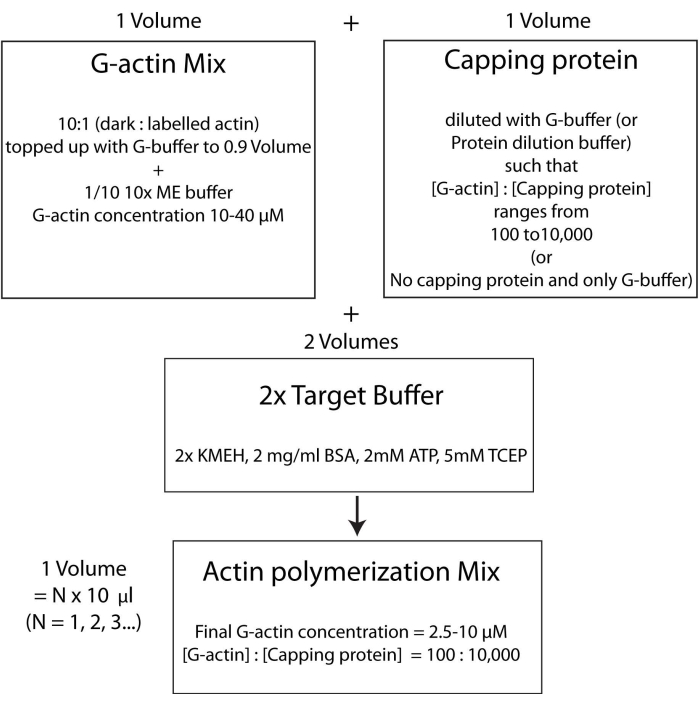
Figure 4 : Schéma montrant comment polymériser l’actine à l’aide de la méthode du tampon cible. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 5 : Organisation spatiale de HYE lors de la liaison à la F-actine. Instantanés TIRF montrant l’organisation spatiale de HYE avant et après l’ajout de filaments d’actine (marqués avec Atto-635 maleimide). L’organisation HYE est homogène avant l’ajout de F-actine et devient colocalisée et coalignée le long des filaments d’actine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
3. Analyse des données
- À l’aide du logiciel Fiji (https://imagej.net), soustrayez l’arrière-plan des images de protéines de liaison (à partir de l’étape 2.4.). Mesurer les valeurs d’intensité moyenne à partir du point blanchi et d’une région de référence.
- Normaliser les traces temporelles entre le point blanchi et la région de référence et l’intensité de leurs valeurs d’intensité respectives avant l’eau de Javel. Divisez chaque point temporel dans les valeurs de région blanchie normalisée par les points temporels respectifs dans le tracé temporel de la région de référence normalisée. Corriger le tracé temporel normalisé résultant pour le bruit de fond et pour toute fluctuation systématique d’intensité lors de l’acquisition (photoblanchiment global, z-drift, etc.).
- Utiliser une méthode manuellesans ajustement 28 pour estimer le coefficient de diffusion des protéines attachées bicouches. En bref, la moitié du temps du profil de récupération, τ1/2, peut être calculée en regardant le moment où le profil de récupération normalisé atteint la moitié de son état d’équilibre:
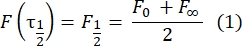
Ici, F 0 est l’intensité moyenne dans la région blanchie dans la première image après le photoblanchiment, et F∞ est la valeur à long terme de la récupération de la bicouche. - Estimer le rayon efficace d’eau de Javel, re, un paramètre qui corrige la diffusion pendant le photoblanchiment, à partir d’un balayage linéaire du profil de la tache post-eau de Javel29. La demi-largeur à la moitié minimale de ce balayage linéaire passant par le centre de la tache d’eau de Javel, r 1/2, se rapporte à re comme suit:
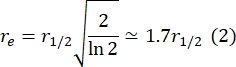
Le τ1/2 calculé à l’étape 2.8.3., r e calculé à l’étape 2.8.4., et le rayon d’eau de Javel initialement défini, rn, sont utilisés pour calculer le coefficient de diffusion (D) à l’aide de la formule suivante: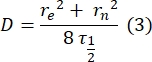
- Utiliser une méthode manuellesans ajustement 28 pour estimer le coefficient de diffusion des protéines attachées bicouches. En bref, la moitié du temps du profil de récupération, τ1/2, peut être calculée en regardant le moment où le profil de récupération normalisé atteint la moitié de son état d’équilibre:
- Analyse de l’image des asters d’actomyosine
- En utilisant Fidji, soustrayez l’arrière-plan de toutes les images enregistrées dans tous les canaux. Corrigez les images pour tout motif d’éclairage ou d’interférence non uniforme à l’aide de la correction de champ plat.
REMARQUE: On peut utiliser des lames en plastique coloré, qui sont de bons échantillons plats pour effectuer de telles corrections. Pour la protéine de liaison et les filaments d’actine sur une bicouche plane, on peut également utiliser la projection moyenne de plusieurs images pré-myosine pour créer des cartes de correction d’éclairage spécifiques au canal.- Pour le canal HYE, illustré ici, prenez une projection d’intensité moyenne de plusieurs images HYE (enregistrées à partir de différentes régions de la bicouche lipidique avant l’ajout de myosine). Appliquez un filtre gaussien approprié (σ = 50 pixels à 80 pixels) à la projection moyenne (à partir d’images pré-myosine ou de tout échantillon plat standard).
- Convertissez l’image filtrée en image 32 bits. Divisez toutes les valeurs de pixels par la moyenne de l’image entière. Cela donnera une carte de correction normalisée pour le canal HYE. Divisez toutes les images du canal HYE avec cette carte pour une correction en champ plat. Créez des cartes de correction pour d’autres canaux en utilisant la même stratégie.
- Corriger le photoblanchiment à l’aide d’une méthode de rapport exponentiel ou simple (selon le profil de décroissance de l’intensité) aux Fidji.
- Pour corriger tout désalignement temporel x-y (mouvement de translation), fusionnez tous les canaux corrigés par l’eau de Javel en une seule Hyperstack. À l’aide du plug-in Hyperstack-Reg aux Fidji, appliquez une transformation Corps rigide ou Traduction.
- Enfin, divisez l’Hyperstack aligné en canaux individuels et enregistrez-les séparément en tant que piles TIFF 16 bits pour une analyse plus approfondie.
- En utilisant Fidji, soustrayez l’arrière-plan de toutes les images enregistrées dans tous les canaux. Corrigez les images pour tout motif d’éclairage ou d’interférence non uniforme à l’aide de la correction de champ plat.
Résultats
Pour la représentation, voici un profil post-blanchiment typique de la 1ère image après photoblanchiment (image à t = 0 s sur la figure 3A) et son ajustement à la fonction suivante28 (voir Figure 6A) :

La valeur de r e (23,94 μm) calculée par l’ajustement à cette courbe est très similaire à la valeur r e calculée à l’étape 2.8.4. (23,24 μm). Ici, K est un paramètre de profondeur d’eau de Javel qui peut être directement estimé à partir de F0 (décrit à l’étape 2.8.4.). De même, la figure 6B montre le profil de récupération et son ajustement à la fonction28 suivante :
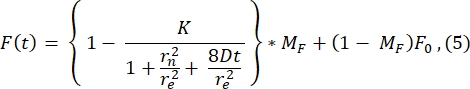
Nous constatons que la valeur ajustée du coefficient de diffusion est de 1,34 μm 2/s, une valeur qui correspond étroitement à la valeur de 1,39 μm 2/s calculée par la formule de l’étape2.8.4. Ici, MF représente la fraction mobile de la bicouche lipidique qui représente la fraction de la population blanchie qui se rétablit. La mobilité des molécules ancrées dans les lipides dépend bien sûr de la composition lipidique et de son état physique (phase liquide ou gel). Pour nos expériences utilisant des membranes lipidiques à base de DOPC, la mobilité doit être de >1 μm2/s, et la fraction mobile ne doit pas être inférieure à 0,9 pour indiquer une bonne bicouche lipidique. Nous recommandons l’utilisation de la méthode sans ajustement manuel pour un test rapide de la qualité et de la mobilité de la bicouche. La méthode d’ajustement peut être utile lors de l’automatisation de l’analyse de nombreuses courbes FRAP. De plus, si l’on veut effectuer une expérience FRAP plus sophistiquée pour caractériser systématiquement la diffusion dans le système, nous recommandons au lecteur de cette revue de Lorén et al.30 pour plus de détails sur les modèles d’ajustement et les pièges potentiels dans la conception expérimentale.

Figure 6 : Quantification du coefficient de diffusion des bicouches lipidiques. (A) Profil linéaire de la première image après photoblanchiment (t = 0 s sur la figure 3A) et son ajustement à l’équation 4 pour calculer le rayon effectif d’eau de Javel. (B) Le profil de récupération de la région blanchie et son adéquation à l’équation 5 pour calculer le coefficient de diffusion et la fraction mobile. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Un résultat typique des expériences décrites ci-dessus montrant l’assemblage dynamique et l’organisation d’un réseau d’acto-myosine lié à une bicouche lipidique supportée imagée par microscopie TIRF est illustré à la figure 7 et à la vidéo supplémentaire S1.
La figure 7 montre un montage d’image de la protéine de liaison, de la F-actine et de la myosine-II.
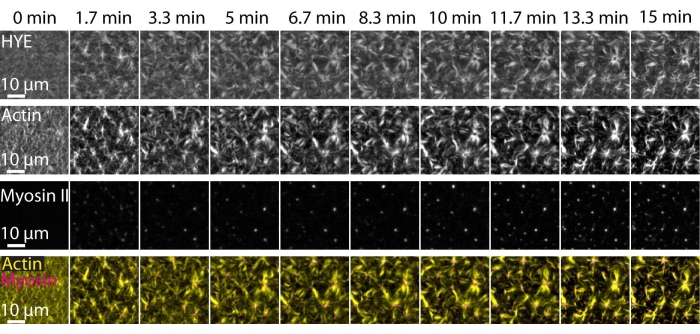
Figure 7 : Les flux d’actomyosine contractile entraînent le regroupement local de la protéine de liaison membrane-actine HYE. Instantanés TIRF de HYE (marqué YFP), de filaments d’actine (marqués avec Atto-635 maleimide) et de filaments de myosine II (marqués avec Atto-565 maleimide) lors de l’ajout de myosine II à un SLB contenant HYE et F-actine. Le temps est indiqué en haut : 0 min est immédiatement avant que les myofilaments fluorescents ne commencent à apparaître dans le champ FRBR. HYE et F-actine sont répartis de manière homogène sur la bicouche lipidique avant l’ajout de myosine (0 min). L’activité de la myosine induit des flux d’actomyosine contractile, qui émergent en structures de type aster à l’état d’équilibre (15 min), entraînant un regroupement local du composant membranaire couplé (HYE). La rangée inférieure est une fusion d’images d’actine (jaune) et de myosine II (magenta) montrant l’organisation de l’actine et de la myosine à différents moments. Les images utilisées dans la réalisation de ces montages ont été corrigées aux Fidji pour le signal de fond, les motifs d’intensité non uniformes et les mouvements de translation. Barre d’échelle = 10 μm. Pour plus de détails, voir Vidéo supplémentaire S1. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom de la mémoire tampon | Composition | |
| Tampon de réhydratation lipidique | 50 mM HEPES, 150 mM NaCl, 5% saccharose, pH 7,5 | |
| Tampon SLB Formation | 50 mM HEPES, 150 mM NaCl, pH 5-6 | |
| Tampon de stockage SLB | 50 mM HEPES, 150 mM NaCl, pH 7,2 | |
| Tampon de dilution des protéines | 20 mM HEPES, 100 mM KCl, 1mM TCEP ou DTT, pH 7,2 | |
| Tampon échangeur d’ions 1X ME ou Actin | 50 mM MgCl2, 0,2 mM EGTA, 10 mM HEPES, pH 7,2 (conserver à 4°C) | |
| 1X tampon de polymérisation KMEH ou Actine | 50 mM KCl, 1 mM MgCl2, 1 mM EGTA, 50 mM HEPES, pH 7,2 | |
| 100 mM de stock ATP | 100 mM de sel disodique ATP, 50 mM de Tris, 50 mM de NaCl, 5 mM de MgCl2, 2 mM d’EGTA, pH 7,5 (à conserver à -20°C) | |
| 2x Tampon cible | 2x KMEH, 2 mg/ml de BSA, 2mM d’ATP, 5mM de PTCE (conservés à 4°C) | |
| G-buffer | Tris 2 mM, CaCl2 0,1 mM, 0,2 mM ATP, 0,5 mM de PTCE, NaN3 à 0,04 %, pH 8 (à conserver à 4 °C) | |
| Tampon myosine II | 500 mM KCl, 1 mM EDTA, 10-20 mM Hepes, pH 7,0 | |
| Tampon de chromatographie par filtration sur gel | 50 mM Tris-HCl, 150-300 mM NaCl, 5 mM TCEP, 0,1% Tween-20, pH 7,5 | |
| Protéine de bouchage Tampon de stockage | 10 mM Tris· Cl, 50 mM NaCl, 1 mM de PTCE, pH 7,5, glycérol à 20 % | |
Tableau 1 : Liste des compositions tampons utilisées dans le présent protocole.
| Problèmes courants et leur dépannage | Problème | Cause | Solutions possibles | ||||||
| 1 | La bicouche lipidique ne montre aucune diffusion | La cause la plus probable de ce problème est le verre de couverture sale qui peut se produire lorsque la solution de nettoyage est vieillie ou que le chauffage n’a pas eu lieu pendant la sonication du bain. Ces bicouches ont un aspect « vésiculaire » car les vésicules éclatées collent au verre de couverture mais ne fusionnent pas les unes avec les autres. L’utilisation de VUM de plus de 6 semaines ou de VUS de plus de 6 jours, ou l’ajout de faibles quantités de VUS peut également entraîner la formation de bicouches vésiculeuses. | Utilisez une solution de nettoyage fraîche. Assurez-vous que le chauffage est allumé et que la température se situe entre 45 et 65 °C. Utilisez des mélanges de lipides frais. (L’utilisation d’une sonde lipidique fluorescente par rapport à une sonde protéique fluorescente peut parfois se manifester différemment. Par exemple, si la bicouche présente des défauts de sous-diffraction et que l’étape de passivation de surface est sautée (ou ne fonctionne pas), la sonde lipidique montrera une distribution d’intensité uniforme, mais la sonde protéique fluorescente peut afficher des taches fluorescentes brillantes.) | ||||||
| 2 | La bicouche lipidique a des taches brillantes | Une longue incubation de VUS pour la formation de bicouches peut créer une bicouche lipidique qui diffuse globalement, mais avec des taches lumineuses occasionnelles. Ces patchs peuvent être des bicouches multicouches qui peuvent attirer de grandes quantités de sondes fluorescentes. | 15-20 min d’incubation avec des SUV suffisent. Assurez-vous que la sonde ne s’agrège pas : un spin dur rapide de la protéine linker (300 x g pendant 15 min à 4 °C) peut éliminer les agrégats | ||||||
| 3 | La bicouche lipidique a des trous sombres | Cela se produit lorsque la bicouche est fabriquée à partir de vieux VUS et imagée pendant des heures prolongées (> 4 heures après la formation), ou lorsque le pH de la solution change radicalement en raison d’une imagerie prolongée (par exemple, dans l’état ATP élevé et en présence de certains piégeurs d’oxygène), ou lorsque la surface est sur-passivée avec de la bêta-caséine (ajouter trop de bêta-caséine pendant plus de 10-15 minutes et ou ne pas la laver). | Utilisez des lipides frais. Réduisez la fréquence d’images de l’imagerie ou le temps d’éclairage laser effectif. Utilisez des tampons avec une capacité de mise en mémoire tampon plus élevée. | ||||||
| 4 | La bicouche lipidique montre une diffusion lente | Les bicouches lipidiques avec un pourcentage élevé de cholestérol, des lipides saturés longs ou des lipides chargés diffusent plus lentement. | Dans de tels cas, préparez votre échantillon à haute température. On peut également utiliser une composition lipidique simple et testée comme témoin avec des compositions lipidiques complexes et non testées. Assurez-vous que le verre est propre. | ||||||
| 5 | L’actine ne polymérise pas | Le tampon cible est ancien, le stock de G-actine est trop vieux, l’ancienne et la nouvelle G-actine ont été copolymérisées. | Assurez-vous que le Ca 2+ est remplacé par Mg2+ avant polymérisation (en utilisant un tampon ME). Utilisez du bouillon d’ATP-Mg2+ frais. Utilisez de la G-actine fraîchement recyclée. Assurez-vous que la concentration de F-actine (en termes de G-actine) ajoutée à la bicouche est supérieure à 0,2 μM. Pour des concentrations plus faibles, utilisez de la F-actine stabilisée à la phalloïdine. | ||||||
| 6 | L’actine ne se lie pas à la bicouche | L’agent de liaison membrane-actine n’est pas ajouté ou ajouté à une très faible concentration, ce qui peut être déduit de la fluorescence de la protéine de liaison. Si la fluorescence est décente, l’agent de liaison membrane-actine a perdu sa capacité de liaison à l’actine. De plus, si la protéine de liaison est non spécifiquement liée à la surface du verre (lorsque la bicouche est mauvaise), elle pourrait ne pas recruter de filaments d’actine. | Assurez-vous que la bicouche diffuse. Utilisez une protéine de liaison fraîche | ||||||
| 7 | Le signal fluorescent F-actine est faible | Le rapport entre l’actine marquée et l’actine foncée est trop faible. L’actine marquée ou l’actine non marquée est trop ancienne et elles ne copolymérisent pas l’une avec l’autre. | Recyclez à nouveau l’actine et recommencez la ploymérisation avec de l’actine fraîchement recyclée. Les photodommages peuvent détruire ou dépolymériser la F-actine; Si possible, utilisez des colorants rouges ou rouge lointain pour l’actine (et la myosine). | ||||||
| 8 | La myosine ne montre pas de contractilité | on peut observer qu’après l’ajout d’ATP au système infusé de myosine, il n’y a pas de contractilité de l’acto-myosine. | Vérifiez si la concentration ou le niveau de pureté de la myosine est bon. Utilisez de la myosine fraîchement recyclée (utilisez dans les 6 semaines suivant le recyclage). L’ajout d’ATP frais au mélange de myosine peut aider. Tampons de dégazage et utilisation de piégeurs d’oxygène, etc. peut réduire les photodommageables des moteurs. D’autres informations peuvent être trouvées dans les protocoles de Plastino et al. ou Stam et al. de la même collection de méthodes | ||||||
| 9 | Le verre de couverture n’est pas hydrophile | Le verre de couverture n’est pas nettoyé correctement. | Un verre de couverture hydrophillique propre est crucial pour la formation de bicouche lipidique. Une lecture visuelle utile de l’hydrophilie du verre de couverture après le protocole de nettoyage consiste à observer le mouillage du verre par l’eau. Ajoutez un petit volume d’eau à une lamelle de couverture plate. L’eau restera sous la forme d’une gouttelette ronde si la lamelle de couverture n’est pas nettoyée correctement. Cependant, le même volume d’eau va s’étaler et former une fine couche, sur un verre hydrophile traité. Ce comportement mouillant de l’eau sur la surface du verre de couverture peut être utilisé pour déterminer si les étapes de nettoyage avec la solution de nettoyage / NaOH ont fonctionné. | ||||||
Tableau 2 : Guide de dépannage résumant les problèmes courants et les solutions correspondantes.
Vidéo supplémentaire S1 : Les flux contractiles d’actomyosine entraînent le regroupement local de la protéine de liaison membrane-actine HYE. Timelapse TIRF de HYE (marqué YFP), de filaments d’actine (marqués avec Atto-635 maleimide) et de filaments de myosine II (marqués avec Atto-565 maleimide) lors de l’ajout de myosine II à un SLB contenant HYE et F-actine. Le temps est indiqué en haut : 0 min est immédiatement avant que les myofilaments fluorescents ne commencent à apparaître dans le champ FRBR. Barre d’échelle = 10 μm. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Ce protocole présente une plate-forme polyvalente et un point de départ pour concevoir des expériences visant à étudier l’interface membrane-cortex des cellules. Les étapes critiques sont la préparation de lames de verre propres, l’utilisation de lipides frais pour une formation efficace de SUV (les deux affectant la qualité des SLB), et l’utilisation de protéines de myosine II fraîchement recyclées pour la réorganisation dynamique du filament d’actine. Lors de l’imagerie de la dynamique sur une longue période, il est très important d’incorporer un système de piégeage de l’oxygène (par exemple, l’acide protocatéchuique et la protocatéchuate 3 4-dioxygénase 5,31).
La conception à chambre ouverte permet l’ajout séquentiel de composants à un système existant sans induire de flux lipidiques. Cela peut être un avantage important par rapport aux approches à chambre fermée couramment utilisées ou au travail utilisant des protéines encapsulées dans les liposomes36. Les effets contraires tels que la déformation membranaire induite par les protéines ne peuvent pas être étudiés avec des bicouches lipidiques adsorbées par du verre.
Les bicouches lipidiques peuvent être formées avec une large gamme de compositions lipidiques. Elle commence par l’adsorption des vésicules lipidiques à la surface du verre hydrophile, suivie soit d’une rupture spontanée des vésicules due aux interactions surface-vésicule et vésicule-vésicule directe, soit par les vésicules adsorbées atteignant une couverture critique, après quoi une petite fraction des vésicules se rompt, formant des bords actifs, ce qui conduit finalement à la formation de bicouche32 . Outre le verre, divers substrats peuvent être utilisés pour former des bicouches lipidiques supportées, telles que le mica (par exemple, pour la microscopie à force atomique), les substrats mous (par exemple, le poly-di-méthyl-siloxane), les coussins polymères33,34,35, couvrant entre les trous des grilles de microscopie électronique 14. Les bicouches d’interface gouttelettes sont une autre méthode intéressante pour créer des bicouches lipidiques stableset autonomes 36. L’inclusion de réseaux d’acto-myosine dans des vésicules ou des émulsions est une méthode très puissante pour étudier ce système minimal dans une géométrie cellulaire37,38, et qui est décrite en détail ailleurs 39.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Ce travail a été soutenu par le fonds de recherche AXA et le programme de biomédecine quantitative Warwick-Wellcome (Wellcome ISSF, RMRCB0058) pour DVK, NCBS-TIFR pour AB et ST, et la bourse Wellcome-DBT Margdarshi (IA/M/15/1/502018) pour SM. DVK tient également à remercier la Biophysical Society d’avoir permis l’événement de réseautage virtuel « Challenges in understanding multi-component cytoskeletal networks from the molecular to the meso-scale », qui a contribué à la création de cette collection de protocoles.
matériels
| Name | Company | Catalog Number | Comments |
| 1,2 dipalmitoyl-sn-glycero-3-phosphoethanolamine-N- (lissamine rhodamine B sulfonyl) | Avanti Polar Lipids | 810158 | 16:0 RhoPE |
| 1,2-dioleoyl-sn-glycero-3- [(N-(5-amino-1 carboxypentyl) iminodiacetic acid) succinyl] (nickel salt) | Avanti Polar Lipids | 790404 | DGS-NTA-Ni2+ |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850375 | DOPC |
| 1,2-dipalmitoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850355 | DPPC |
| Amber glass vials | ThermoFisher | B7990-2A | |
| ATP disodium salt | Sigma Aldrich | A26209 | |
| Attofluor cell chamber | ThermoFisher | A7816 | |
| Bath sonicator | GT Sonic | 1860QTS | |
| beta-casein | Sigma Aldrich | C6905 | |
| CaCl2 | ThermoFisher | 12135 | |
| chloroform | Sigma Aldrich | 650471 | alternatively from Electron Microscopy Sciences, 50980296 |
| Cover slips, #1, 25 mm diameter, Gold Seal | Harvard Apparatus | 64-0705B | |
| Cover slips, #1, 40x22 mm, Gold Seal | ThermoFisher | 48404-031 | |
| EDTA | ThermoFisher | G12635 | |
| EGTA | Himedia | MB130 | |
| Gas tight glass syringes, with removebla needle, blunt, volumes 10 µL, 100 µL, 500 µL | Hammilton | 1700 series | |
| Hellmanex III | Hellma Analytics | Z805939 | cleaning solution |
| HEPES | Himedia | RM380 | |
| KaH2PO4 | ThermoFisher | G13405 | |
| KCl | ThermoFisher | G13305 | |
| KOH | ThermoFisher | G26708 | |
| Lipid extruder | Avanti Polar Lipids | 61000-1EA | |
| MgCl2 | ThermoFisher | G15535 | |
| Microtip sonicator | Sonics | VC750 | 3 mm Tip diameter |
| Na2CO3 | ThermoFisher | G15955 | |
| NaCl | Himedia | GRM853 | |
| NaH2PO4 | ThermoFisher | G15825 | |
| NaOH | ThermoFisher | G27815 | |
| Nikon Ti Eclipse TIRF microscope | Nikon | With a TIRF unit connected through a polarization-conserving optical fibre to an Agilent monolithic laser combiner MLC400 with multiple laser lines with a 100X, 1.45 NA Nikon Oil Objective with two 512 x 512-pixel EMCCD cameras (Photometrics Evolve 512) with a 100X, 1.45 NA Nikon Oil Objective with two 512 x 512-pixel EMCCD cameras (Photometrics Evolve 512) | |
| NOA88 | Norland Products | 8801 | |
| PTFE Coated Tweezer Style #2A | Structure Probe | 0S2AT-XD | |
| Refrigerated microcentrifuge | Eppendorf | 5424R | |
| Sucrose | ThermoFisher | G15925 | |
| UV-illuminator | Novascan | PSD PRO-UV | needs vacuum and O2 supply |
Références
- Köster, D. V., Mayor, S. Cortical actin and the plasma membrane: Inextricably intertwined. Current Opinion in Cell Biology. 38, 81-89 (2016).
- Rao, M., Mayor, S. Active organization of membrane constituents in living cells. Current Opinion in Cell Biology. 29, 126-132 (2014).
- Honigmann, A., et al. A lipid bound actin meshwork organizes liquid phase separation in model membranes. eLife. 3, 01671 (2014).
- Das, A., et al. Stratification relieves constraints from steric hindrance in the generation of compact actomyosin asters at the membrane cortex. Science Advances. 6 (11), (2020).
- Köster, D. V., et al. Actomyosin dynamics drive local membrane component organization in an in vitro active composite layer. Proceedings of the National Academy of Sciences. 113 (12), 1645-1654 (2016).
- Vogel, S. K., Heinemann, F., Chwastek, G., Schwille, P. The design of MACs (minimal actin cortices). Cytoskeleton. 70 (11), 706-717 (2013).
- Murrell, M., Thoresen, T., Gardel, M. Reconstitution of contractile actomyosin arrays. Methods in Enzymology. 540, 265-282 (2014).
- Mosby, L. S., et al. Myosin II filament dynamics in actin networks revealed with interferometric scattering microscopy. Biophysical Journal. 118 (8), 1946-1957 (2020).
- Seara, D. S., et al. Entropy production rate is maximized in non-contractile actomyosin. Nature Communications. 9 (1), 4948 (2018).
- Linsmeier, I., et al. Disordered actomyosin networks are sufficient to produce cooperative and telescopic contractility. Nature Communications. 7, 12615 (2016).
- Vogel, S. K., Petrasek, Z., Heinemann, F., Schwille, P. Myosin motors fragment and compact membrane-bound actin filaments. eLife. 2, 00116 (2013).
- Ditlev, J. A., et al. A composition-dependent molecular clutch between T cell signaling condensates and actin. eLife. 8, 42695 (2019).
- Banjade, S., Rosen, M. K. Phase transitions of multivalent proteins can promote clustering of membrane receptors. eLife. 3, 04123 (2014).
- Heinemann, F., Vogel, S. K., Schwille, P. Lateral membrane diffusion modulated by a minimal actin cortex. Biophysical Journal. 104 (7), 1465-1475 (2013).
- Vogel, S. K., Greiss, F., Khmelinskaia, A., Schwille, P. Control of lipid domain organization by a biomimetic contractile actomyosin cortex. eLife. 6, 24350 (2017).
- Block, S. Brownian motion at lipid membranes: A comparison of hydrodynamic models describing and experiments quantifying diffusion within lipid bilayers. Biomolecules. 8 (2), 30 (2018).
- JoVE, JoVE Methods Collection. In vitro reconstitution of cytoskeleton networks for biomaterials, biophysics and active matter research. Journal of Visualized Experiments. , (2022).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Enzymology. 85, 164-181 (1982).
- Spudich, J. A., Watt, S. The regulation of rabbit skeletal muscle contraction. I. Biochemical studies of the interaction of the tropomyosin-troponin complex with actin and the proteolytic fragments of myosin. Journal of Biological Chemistry. 246 (15), 4866-4871 (1971).
- Pollard, T. D. Myosin purification and characterization. Methods in Cell Biology. 24, 331-371 (1982).
- JoVE Science Education Database. Separating protein with SDS-PAGE. Journal of Visualized Experiments. , (2022).
- Bieling, P., et al. WH2 and proline-rich domains of WASP-family proteins collaborate to accelerate actin filament elongation. The EMBO Journal. 37 (1), 102-121 (2018).
- Shrivastava, R., Köster, D., Kalme, S., Mayor, S., Neerathilingam, M. Tailor-made ezrin actin binding domain to probe its interaction with actin in-vitro. PLoS One. 10 (4), 0123428 (2015).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5, 5412 (2014).
- Selden, N. S., et al. Chemically programmed cell adhesion with membrane-anchored oligonucleotides. Journal of the American Chemical Society. 134 (2), 765-768 (2012).
- Seu, K. J., et al. Effect of surface treatment on diffusion and domain formation in supported lipid bilayers. Biophysical Journal. 92 (7), 2445-2450 (2007).
- Kang, M., Day, C. A., Kenworthy, A. K., DiBenedetto, E. Simplified equation to extract diffusion coefficients from confocal FRAP data. Traffic. 13 (12), 1589-1600 (2012).
- Kang, M., Day, C. A., Kenworthy, A. K. A novel computational framework for D(t) from Fluorescence Recovery after Photobleaching data reveals various anomalous diffusion types in live cell membranes. Traffic. 20 (11), 867-880 (2019).
- Lorén, N., et al. Fluorescence recovery after photobleaching in material and life sciences: Putting theory into practice. Quarterly Reviews of Biophysics. 48 (3), 323-387 (2015).
- Alvarado, J., Sheinman, M., Sharma, A., MacKintosh, F. C., Koenderink, G. H. Molecular motors robustly drive active gels to a critically connected state. Nature Physics. 9 (7), 1-7 (2013).
- Andrecka, J., Spillane, K. M., Ortega-Arroyo, J., Kukura, P. Direct observation and control of Supported lipid bilayer formation with interferometric scattering microscopy. ACS Nano. 7 (12), 10662-10670 (2013).
- Lin, W. -. C., et al. Supported membrane formation, characterization, functionalization, and patterning for application in biological science and technology. Current Protocols in Chemical Biology. 2 (4), 235-269 (2010).
- Richter, R. P., Bérat, R., Brisson, A. R. Formation of solid-supported lipid bilayers: An integrated view. Langmuir. 22 (8), 3497-3505 (2006).
- Sapuri-Butti, A. R., Butti, R. C., Parikh, A. N. Characterization of supported membranes on topographically patterned polymeric elastomers and their applications to microcontact printing. Langmuir. 23 (25), 12645-12654 (2007).
- Leptihn, S., et al. Constructing droplet interface bilayers from the contact of aqueous droplets in oil. Nature Protocols. 8 (6), 1048-1057 (2013).
- Bashirzadeh, Y., Moghimianavval, H., Liu, A. P. Encapsulated actomyosin patterns drive cell-like membrane shape changes. iScience. 25 (5), 104236 (2022).
- Bashirzadeh, Y., Wubshet, N., Litschel, T., Schwille, P., Liu, A. P. Rapid encapsulation of reconstituted cytoskeleton inside giant unilamellar vesicles. Journal of Visualized Experiments. (177), e63332 (2021).
- Murrell, M., Chen, S., Sun, Z. G. In vitro reconstitution of actin cytoskeleton inside giant unilamellar vesicles. Journal of Visualized Experiments. , (2022).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon