Method Article
Reconstituição de Córtices de Actina Mínima Amarrados à Membrana em Bicamadas Lipídicas Suportadas
Neste Artigo
Resumo
Este protocolo descreve a formação de bicamadas lipídicas suportadas e a adição de filamentos citoesqueléticos e proteínas motoras para estudar a dinâmica de redes citoesqueléticas reconstituídas e ligadas à membrana usando microscopia de fluorescência.
Resumo
A superfície de uma célula viva fornece uma plataforma ativa versátil para numerosos processos celulares, que surgem da interação da membrana plasmática com o córtex de actina subjacente. Nas últimas décadas, sistemas mínimos reconstituídos baseados em bicamadas lipídicas suportadas em combinação com redes de filamentos de actina provaram ser muito instrumentais no desvendamento de mecanismos básicos e consequências de redes de actina amarradas à membrana, bem como no estudo das funções de proteínas individuais associadas à membrana. Aqui, descrevemos como reconstituir tais sistemas compostos ativos in vitro que consistem em bicamadas lipídicas suportadas por fluidos acopladas via proteínas de ligação à actina associadas à membrana a filamentos dinâmicos de actina e motores de miosina que podem ser prontamente observados através de microscopia de fluorescência de reflexão interna total. Um projeto de câmara aberta permite montar o sistema de maneira passo a passo e controlar sistematicamente muitos parâmetros, como concentração de proteína ligadora, concentração de actina, comprimento do filamento de actina, relação actina/miosina, bem como níveis de ATP. Finalmente, discutimos como controlar a qualidade do sistema, como detectar e solucionar problemas comuns e algumas limitações desse sistema em comparação com a superfície celular viva.
Introdução
A membrana plasmática de uma célula animal viva interage constantemente com o citoesqueleto de actina adjacente e, juntas, formam um material compósito ativo que cumpre uma infinidade de funções celulares 1,2. Para estudar os processos nesta interface membrana lipídica-actina, a reconstituição de redes citoesqueléticas em cima de bicamadas lipídicas suportadas (SLBs) provou ser muito útil. Esta abordagem de sistema mínimo permite o controle preciso dos componentes da rede do citoesqueleto e da composição lipídica. Em comparação com as membranas lipídicas independentes das vesículas unilamelares gigantes, a geometria planar dos SLBs permite o uso eficiente de técnicas de microscopia de última geração, como super-resolução3,4, fluorescência de reflexão interna total (TIRF)5,6,7 ou espalhamento interferométrico 8 estudar a organização espacial e a dinâmica das redes citoesqueléticas. O TIRF fornece o maior contraste para componentes marcados fluorescentemente, uma vez que o sinal de moléculas marcadas não ligadas na solução que contribuem para o sinal de fundo é mínimo.
Aqui, descrevemos um protocolo básico para a formação de redes de actomiosina amarradas a bicamadas lipídicas suportadas, que são amplamente utilizadas em campo para estudar a física de redes ativas, quase-2D 9,10,11 e seu efeito na organização da membrana 3,5,12,13,14,15,16 (Figura 1 ). Esta abordagem não se limita a redes baseadas em actina, mas também pode ser adaptada facilmente para explorar microtúbulos, filamentos intermediários ou redes compostas de natureza mista e para estudar uma variedade de interações entre proteínas da membrana lipídica e componentes citoesqueléticos usando métodos de microscopia sensíveis à superfície.
Para manter este protocolo focado, excluímos uma descrição detalhada da purificação e marcação das proteínas actina e miosina ou detalhes sobre como sintonizar e controlar a contratilidade e organização das redes de actomiosina. Deve-se referir a outros protocolos que são publicados ao lado deste na JoVE Methods Collection, In Vitro Reconstitution of Cytoskeleton Networks for Biomaterials, Biophysics and Active Matter Research17.

Figura 1: Esquema do sistema composto ativo de membrana de actina in vitro . Criado com Biorender. Por favor, clique aqui para ver uma versão maior desta figura.
Protocolo
1. Reagentes e equipamento
- Prepare buffers frescos conforme listado na Tabela 1. Use água ultrapura, deionizada, com uma resistividade de 18,2 MΩ·cm a 25 °C. Esterilize todos os buffers passando-os através de filtros de 0,22 μm sob vácuo. Desgaseifique os tampões utilizados para cromatografia em coluna.
- Purificar a actina do músculo esquelético conforme descrito anteriormente18,19. Adicionar glicerol a 20% à solução final de acetina G purificada e fazer alíquotas de 500 μL (para marcação ou experiências a granel) e 10 μL (para experiências individuais) de volume. Congele as alíquotas mergulhando os tubos em nitrogênio líquido por 30 s e, em seguida, armazene-os a -80 °C por até 18 meses.
NOTA: Alternativamente, actina purificada ou acetona em pó pode ser comprado comercialmente. - Rotule a G-actina purificada do músculo esquelético com quaisquer corantes fluorescentes de maleimida, conforme descrito anteriormente5. Determinar a concentração e o grau de marcação da proteína por espectrofotometria usando A290nm corrigido para actina (εactina = 26.600 M-1 cm-1) e A λmax do corante. Faça alíquotas de 10 μL e congele rapidamente mergulhando os tubos em nitrogênio líquido por 30 s e armazene a -80 °C por até 18 meses.
NOTA: A rotulagem com ésteres NHS conjugantes de lisina criará actina não funcional e deve ser evitada. - Purificar a miosina II do músculo esquelético seguindo o protocolo20. Execute SDS-PAGE usando gel de poliacrilamida a 10% seguido de coloração Coomassie para determinar o nível de pureza da proteína21. Armazenar miosina II do músculo esquelético purificado a -20 °C na forma líquida em tampão de miosina II com 50% de glicerol.
NOTA: A miosina II armazenada pode ser usada por até 2 anos. - Rotule a miosina-II purificada com quaisquer corantes fluorescentes de maleimida, conforme descrito anteriormente5. Evite rotular os motores de miosina com corantes NHS-ésteres. Determinar a concentração e o grau de marcação por espectrofotometria usando A280nm corrigidos de miosina II e Aλmax do corante. Conservar a miosina II reciclada (escura ou rotulada) a 4 °C e utilizar no prazo de 6 semanas.
- Purificação da proteína de cobertura
- Obter proteína de tamponamento murino seguindo um protocolo anterior22. Execute SDS-PAGE usando gel de poliacrilamida a 10% seguido de coloração Coomassie para determinar o nível de pureza da proteína. Meça a concentração usando A280nm de proteína de encapsulamento (εCP = 99.530 M-1 cm-1).
- Adicione 20% de glicerol à solução proteica e faça alíquotas de 5 μL em tubos de PCR de 200 μL. Mergulhe os tubos em nitrogênio líquido e armazene-os a -80 °C por até 2 anos.
NOTA: A atividade da proteína de encapsulamento é verificada pela polimerização de quantidades fixas de G-actina fluorescente na presença de diferentes quantidades de proteína de encapsulamento. Os filamentos são então fotografados sob um microscópio, e sua distribuição de comprimento é quantificada. Quanto maior a concentração relativa de proteína de encapsulamento, menores as distribuições do filamento de actina. Ver Köster et al.5.
- Expresse uma proteína ligadora de membrana fluorescente de actina, por exemplo, para este protocolo use 10xHis-YFP-EzrinABD (HYE), expresse-a em Bl21DE3* Escherichia Coli e purifique conforme descrito anteriormente23. Determinar a concentração da proteína por espectrofotometria.
- Armazenar a proteína em pequenas alíquotas em tampão de cromatografia de filtração em gel (ou qualquer outro tampão apropriado) com glicerol a 20% a -80 °C. Nestas condições, a proteína é estável por mais de 2 anos.
NOTA: A escolha da proteína ligadora actina-membrana e do marcador fluorescente depende do tipo de questão que se está a abordar. Uma ampla gama de estratégias de ligação lipídica foi desenvolvida nos últimos anos, incluindo proteínas24 marcadas com histidina, estreptavidina25 de biotina e DNA de fita simples26. - Preparação de vesículas multilamelares (MLVs)
NOTA: O fluxo de trabalho de MLVs para bicamadas lipídicas suportadas é representado na Figura 2.- Coloque 5-10 frascos de vidro âmbar em um copo de vidro de 200 mL. Encha o copo com uma solução de limpeza a 2%, apenas o suficiente para submergir os frascos para injetáveis de vidro. Sonicate-os em banho-maria por 30 min a pulso cheio e 65 °C.
- Retire os frascos para injetáveis da solução e lave-os cuidadosamente com água destilada. Coloque os frascos para injetáveis num copo de vidro contendo 2 N NaOH e sonicate durante 20 min. Nenhum aquecimento é necessário durante esta etapa.
- Retire os frascos para injetáveis da solução de NaOH e enxágue bem com água destilada. Secar os frascos para injetáveis no interior de um forno de ar quente regulado a 65 °C durante 2 h ou mais.
- Armazene os frascos limpos em um copo limpo selado com filme transparente por até 6 semanas.
CUIDADO: Execute as seguintes etapas dentro de um exaustor químico. Manuseie o clorofórmio e as soluções lipídicas com seringas de vidro Hamilton à prova de gás para evitar a contaminação por plástico. - Lave as seringas Hamilton e alguns frascos para injetáveis de vidro âmbar várias vezes com clorofórmio puro. Pegue o pó lipídico armazenado em ampolas de vidro do congelador de -20 °C e adicione volumes adequados de clorofórmio para dissolver os pós lipídicos a concentrações de 10-25 mg/mL.
- Transfira a solução da ampola para um frasco para injetáveis de vidro âmbar acabado de limpar e rotule-a adequadamente. Execute este passo no gelo para reduzir a evaporação do clorofórmio.
- Faça uma solução-mãe de DOPC com uma concentração de 10-25 mg/mL e DGS-NTA-Ni2+ com uma concentração de 1-10 mg/mL.
- Para fazer uma mistura lipídica funcional, pegue um frasco de vidro limpo e enxágue-o 2x com clorofórmio. Adicione 300 μL de clorofórmio puro ao frasco para injetáveis para servir de base para uma melhor mistura dos componentes. Isso não afetará as concentrações finais dos lipídios, pois todo o clorofórmio será seco nas próximas etapas.
- Adicione volumes medidos de soluções-estoque de lipídios ao frasco para injetáveis para fazer as misturas lipídicas de trabalho desejadas. A concentração lipídica alvo no tampão de reidratação lipídica é de 4 mM. Seque a mistura lipídica sob um fluxo lento de gás N2 dentro do exaustor químico à temperatura ambiente. Este passo pode demorar até 30 minutos para cada frasco para injetáveis.
- Depois de todo o solvente ter secado, desidratar a vácuo o filme lipídico por >2 h à temperatura ambiente para remover quaisquer vestígios de clorofórmio restantes. Ressuspender a mistura lipídica desidratada em tampão de reidratação lipídica para uma concentração lipídica final de 4 mM.
- Incubar por 5-10 min para permitir a reidratação dos lipídios. Vórtice a solução lipídica por cerca de 30 s para formar MLVs.
- Faça alíquotas de 0,5-1 mL dos MLVs em tubos de microcentrífuga de 1,5 mL. Mergulhe os tubos em nitrogênio líquido, sele com um filme transparente e armazene a -20 °C (por até 6 semanas).
NOTA: As concentrações de estoque lipídico são escolhidas para permitir volumes suficientemente grandes que permitam uma pipetagem confiável usando as seringas de Hamilton. Se os volumes necessários para fazer o estoque forem muito grandes para dissolver o pó lipídico seco, faça várias diluições do estoque para garantir a mistura reprodutível de vários lipídios.
- Preparação de pequenas vesículas unilamelares (SUVs)
- Retire uma alíquota de MLVs do armazenamento de -20 °C e descongele-a à temperatura ambiente. Congele rapidamente as vesículas mergulhando o tubo de microcentrífuga em nitrogênio líquido por 15-30 s e coloque-o imediatamente em banho-maria a 45 °C até que a solução tenha descongelado completamente (1-2 min). Repita o ciclo de congelamento-descongelamento acima 10x-15x até que a solução pareça menos turva.
NOTA: Defina a temperatura do banho-maria mais elevada do que a temperatura de transição da mistura lipídica que está a ser descongelada para permitir uma mistura lipídica uniforme. - Equilibre uma mini extrusora à base de seringa equipada com uma membrana filtrante de policarbonato de 80 nm de tamanho de poro com tampão de reidratação SUV. Certifique-se de que não há vazamento ou bolhas no sistema. Enquanto o método de extrusão produz SUVs monodispersos com o mínimo de dano lipídico, as misturas lipídicas com carga negativa podem aderir à membrana de policarbonato.
- Passe suavemente a solução lipídica descongelada através da extrusora pré-equilibrada de um lado para o outro e depois para trás. Repita o ciclo 5x-10x até que a solução lipídica fique visivelmente clara, indicando a formação de SUVs com ~100 nm de diâmetro.
- Centrifugar a suspensão extrudada (ou tip-sonicate a solução; ver nota abaixo) a 15.000 x g durante 60 min a 4 °C para expelir os detritos lipídicos. Recolha os 80% superiores da solução sem perturbar o pellet e sem criar bolhas. Transfira o sobrenadante contendo os SUVs para um tubo de microcentrífuga fresco e armazene no gelo por até 6 dias.
NOTA: Uma alternativa à centrifugação é a sonicação da ponta realizada da seguinte forma. Ligue um sonicator de microponta e defina as seguintes configurações: Amplitude = 30% do máximo, ON time = 10 s, OFF time = 60 s. Limpe a ponta do micro-sonicador com água deionizada seguida de 2 N NaOH, clorofórmio e novamente água deionizada. Mergulhe a ponta do sonicador em cada uma dessas soluções e sonicate por 1-2 ciclos usando as configurações acima. Mergulhe a ponta limpa na solução de vesícula descongelada e sonicate por 3-6 ciclos no gelo até que a solução fique clara. - Após a centrifugação, verifique se há sinais de alta degradação lipídica ou uma falha na extrusão lipídica como a formação de um fino filme esbranquiçado e / ou um pellet claramente visível. Nestes casos, não prossiga e repita os passos de preparação do SUV novamente.
NOTA: A vida útil dos SUVs pode diferir para diferentes misturas lipídicas. SUVs feitos de DOPC: DGS-NTA-Ni2+ são estáveis por até 6 dias para os fins desses experimentos. Dicas para resolver problemas comuns podem ser encontradas na Tabela 2.
- Retire uma alíquota de MLVs do armazenamento de -20 °C e descongele-a à temperatura ambiente. Congele rapidamente as vesículas mergulhando o tubo de microcentrífuga em nitrogênio líquido por 15-30 s e coloque-o imediatamente em banho-maria a 45 °C até que a solução tenha descongelado completamente (1-2 min). Repita o ciclo de congelamento-descongelamento acima 10x-15x até que a solução pareça menos turva.
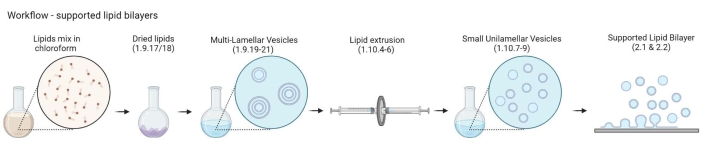
Figura 2: Esquema mostrando o fluxo de trabalho desde a preparação de vesículas multilamelares e pequenas vesículas unilamelares até a formação de bicamadas lipídicas suportadas. Criado com Biorender. Por favor, clique aqui para ver uma versão maior desta figura.
2. Reconstituição de redes de actina ligadas à membrana
- Preparação de câmaras de amostra
- Pegue 3-5 tampas de vidro retangulares e coloque-as dentro de um frasco Coplin. Ligue o sonicator de banho e ajuste a temperatura para 65 °C. Encha o frasco Coplin com solução de limpeza a 2% para submergir totalmente as tampas e coloque-o no sonicator por 30 minutos no modo de pulso total.
- Use pinças revestidas de PTFE contundentes para remover as tampas uma a uma do frasco. Lave-os bem com água destilada e coloque-os em outro frasco Coplin cheio de 2 N NaOH.
- Sonicate as tampas por 20 minutos no modo de pulso cheio. Retire as tampas uma a uma, enxágue bem com água destilada e coloque em outro frasco Coplin cheio de água destilada.
NOTA: Opcionalmente, sonicate as folhas de cobertura em água destilada por 20 minutos e, em seguida, enxágue-as novamente com água destilada. - Imediatamente antes de iniciar o experimento, pegue o frasco contendo as tampas em um exaustor químico equipado com uma fonte de gás N2 .
- Otimize a pressão do ar do fluxo de gás N2 por tentativa e erro para que seja apenas o suficiente para deslocar a água da superfície de deslizamento da cobertura sem quebrá-la. Alinhe o fluxo de gás N2 paralelamente ao plano de deslizamento de cobertura para reduzir a possibilidade de quebrar o deslizamento de cobertura.
- Use luvas e fórceps para remover as tampas uma a uma do frasco para secá-las sob o fluxo N2 . Seque ambos os lados de cada tampa e coloque-os em uma grade de plástico limpo com uma tampa. Coloque a caixa com as tampas em um exsicador para evitar o contato com partículas de poeira no ar.
NOTA: As folhas de cobertura secas N 2 podem ser armazenadas em um exsicador onde podem permanecer hidrofílicas por até2 dias. Essa estratégia pode ser útil quando muitas bicamadas são necessárias para o experimento ou se o experimento leva mais de 8 h. - Pegue tubos de PCR autoclavados e corte suas tampas e metades cônicas inferiores com uma lâmina cirúrgica afiada. Pegue os tubos cilíndricos semi-cortados um por um, aplique adesivo curável UV na borda lisa de cada tubo de corte e coloque-o invertido em uma tampa recém-limpa, de modo que a borda fique plana na tampa.
- Não mova o cilindro lateralmente depois de posicionado na tampa para garantir que a cola não derrame para o espaço central da câmara. As coberturas retangulares podem acomodar confortavelmente até três câmaras de reação, e as redondas podem acomodar apenas uma no centro (Figura 1).
- Coloque as tampas do rolamento da câmara dentro de um limpador de ozônio UV com uma fonte de O2 e vácuo (ou use um iluminador UV). Ligue a luz UV e acenda por 3-5 min para permitir que o adesivo polimerize. Realize uma iluminação mais longa (10-15 min) para melhorar a hidrofilicidade do vidro de cobertura e, portanto, a qualidade da bicamada lipídica.
- Armazene as câmaras de amostra secas iluminadas por UV por até 8 h dentro de pequenas caixas plásticas (como caixas de deslizamento de cobertura retangulares vazias) envoltas em filme transparente para reduzir o contato com partículas de poeira no ar.
NOTA: Um fluxo constante de O2 na presença de luz UV forma radicais de ozônio e oxigênio que podem remover impurezas orgânicas da superfície da tampa. Um vácuo evitará o vazamento de ozônio tóxico que é formado durante o processo. - Retire as tampas e teste as câmaras quanto a vazamentos, enchendo-as com água destilada. Cada câmara pode conter até ~150 μL de amostra. Descarte as câmaras com vazamento.
NOTA: Outra ótima e segura opção de limpeza é o limpador de plasma. As configurações de tempo e potência dependem do modelo, mas certifique-se de não sobretratar as lâminas de vidro com plasma, pois isso resultará em uma redução da mobilidade lipídica. O tratamento superficial pode afetar a mobilidade dos lipídios27, como tem sido observado com o tratamento prolongado com a solução de limpeza (>45 min) ou NaOH (>30 min).
- Preparação de bicamadas lipídicas suportadas
- Lave cada câmara com tampão de formação SLB (ou 1x PBS) para remover quaisquer contaminantes de superfície, deixando 100 μL de tampão no final. Marque o nível do buffer a 100 μL com um marcador permanente para rastrear de forma reprodutível as mudanças no volume.
- Adicionar 2 μL de CaCl2 0,1 M à câmara. Isso melhora a adsorção das vesículas à superfície do vidro, aumentando a formação de bicamadas na próxima etapa. Adicionar 8 μL da solução SUV (a partir da fase 1.10) a cada câmara e incubar durante 15 minutos a 25 °C.
NOTA: O volume da mistura SUV a ser adicionada pode ser estimado calculando-se o número total de lipídios (com uma área média de 0,72 nm2) que são necessários para cobrir completamente a área hidrofílica exposta do poço com duas camadas lipídicas. - Lave as vesículas não ligadas com tampão de motilidade de actina (1x KMEH). Primeiro, remova 50 μL do tampão de formação SLB, deixando apenas 50 μL na câmara de amostragem. Em segundo lugar, adicione 100 μL de 1x KMEH à câmara. Misture suavemente e, em seguida, retire 100 μL do tampão sem tocar no fundo.
NOTA: É importante ser suave durante a lavagem. Certifique-se de que a ponta da pipeta não toque na parte inferior da câmara. Mantenha a pipeta inclinada para direcionar o fluxo de buffer para a parede da câmara e não diretamente para a bicamada, pois um fluxo direto pode interromper a bicamada. Tenha cuidado para não introduzir bolhas de ar durante a pipetação, pois o ar pode atingir a bicamada lipídica e causar defeitos nela. - Repita as lavagens 10x adicionando 100 μL de 1x KMEH e removendo 100 μL.
- Adicione 10 μL de 1 mg/mL de β-caseína à bicamada, misture suavemente e incube por 5-10 min. β-caseína bloqueia as regiões na tampa onde a bicamada não se formou. Lavar β-caseína 3x com 1x KMEH, conforme descrito na etapa 2.2.3. e traga o nível do buffer de volta para a marca de 100 μL.
- Adição de ligador membrana-actina
- Durante a incubação β-caseína (etapa 2.2.5.), retire uma alíquota da proteína ligadora membrana-actina de -80 °C, descongele-a rapidamente a 37 °C e mantenha-a no gelo. Diluir a alíquota com tampão de diluição proteica até uma concentração de 1 μM.
- Adicione a proteína ligadora a uma concentração final definida (tipicamente 5-20 nM) e misture suavemente. Para garantir um rápido equilíbrio da proteína na câmara, adicione volumes maiores que 20 μL pré-misturando a proteína ligadora com 1x KMEH.
- Incubar durante 40 min à temperatura ambiente. Lave 3x com 1x tampão KMEH para remover a proteína HSE não ligada (como na etapa 2.2.3.). Traga o nível de tampão em cada câmara de volta à marca de 100 μL. A amostra agora está pronta para geração de imagens.
- Avaliação da qualidade da bicamada lipídica
Observação : esta é uma etapa opcional que não precisa ser executada todas as vezes. Recomendamos que esta avaliação seja realizada sempre que os SUVs frescos forem feitos a partir de estoques de MLV congelados.- Ligue o microscópio, os lasers de excitação e as câmeras de detecção. Certifique-se de que o laser está alinhado, a objetiva está limpa e o software está pronto para adquirir imagens.
- Coloque óleo na objetiva de 100x, monte a amostra no estágio do microscópio e concentre a objetiva na bicamada. Certifique-se de que a posição do laser seja tal que sofra uma reflexão interna total na amostra. Use um laser de excitação de 488 nm para verificar a distribuição de intensidade de fluorescência do 10xHis-YFP-EzrinABD ligado a duas camadas.
NOTA: Bicamadas de boa qualidade mostram uma distribuição uniforme e em larga escala da intensidade de fluorescência. Bicamadas ruins mostram manchas fluorescentes intensas e irregulares. - Para determinar a integridade da bicamada, execute um ensaio FRAP.
- Selecione uma região de interesse na bicamada e grave algumas imagens do campo de visão usando condições de imagem que forneçam uma relação sinal-ruído de 5:1 ou superior. Pause a gravação e feche o diafragma de campo do microscópio TIRF para focar um feixe de laser concentrado em uma pequena região circular da bicamada para branquear localmente os fluoróforos.
- Ligue o laser até sua saída máxima para fotobranquear a pequena região por 3-10 s e, em seguida, desligue o laser. Reabra o diafragma de campo ao seu raio original, reajuste a condição de imagem de volta às configurações (pré-alvejante) e retome imediatamente para registrar a recuperação do sinal fluorescente no campo de visão.
- Verifique se a bicamada é fluida. As boas bicamadas com difusão lateral normal se recuperam rapidamente, enquanto as más bicamadas se recuperam lentamente ou não se recuperam (Figura 3). Se a bicamada não estiver se recuperando, verifique a seção de solução de problemas e reinicie. Salve as imagens como arquivos TIFF de 16 bits. Para uma estimativa quantitativa do coeficiente de difusão, verifique a etapa 3. abaixo.
- Polimerização de actina fluorescente
NOTA: Para poupar tempo, iniciar a polimerização da actina durante o tempo de incubação da ligação da proteína HSE à bicamada (passo 2.3.) ou durante a avaliação da qualidade da bicamada (passo 2.4.).- Misture a G-actina não marcada e fluorescentemente marcada em uma proporção molar de 10:1 e cubra com G-Buffer para que a concentração de G-actina seja de 20 μM. A concentração na qual a actina é finalmente polimerizada será de 1/4 deste valor. Adicione 1/10 de tampão 10x ME à mistura para uma solução 1x e incube por 2 min. Esta etapa substitui os íons Ca 2+ ligados à actina G poríons Mg2+. Certifique-se de que o volume final esteja em múltiplos de 10 μL.
- Adicione a quantidade desejada de proteína de cobertura da seguinte forma. Descongele rapidamente um frasco para injetáveis de proteína de cobertura a 37 °C e, em seguida, mantenha-o no gelo. Diluir com tampão G de tal forma que a concentração de proteína de encapsulamento agora seja o dobro da concentração final desejada na mistura de polimerização. Adicionar um volume igual da solução de proteína de cobertura diluída à mistura de actina a partir da fase 2.5.1.
- Finalmente, adicione um volume igual de buffer alvo fresco de 2x à mistura de reação. O volume final da solução deve ser quatro vezes superior ao volume da mistura de actina no final da fase 2.5.2. Certifique-se de que a concentração final de KMEH é de 1x, de ATP é de 1 mM, de BSA é de 1 mg/mL e de G-actina é de 5 μM.
Incubar no escuro a 25 °C por 45-60 min para permitir que a polimerização aconteça.
NOTA: Isso é chamado de estratégia de tampão de destino, na qual um volume de Mg 2+ G-actina (etapa 2.5.1.) é misturado com um volume de mistura de proteínas de encapsulamento (etapa 2.5.2.) e dois volumes de tampão de destino2x (etapa 2.5.3.). Isso torna mais fácil aumentar ou diminuir a quantidade de actina e alterar a concentração relativa de proteína de cobertura (ou qualquer outro modulador de actina; Figura 4).
- Adição de filamentos fluorescentes de actina
- Corte algumas pontas de 200 μL com uma lâmina afiada ou tesoura para torná-las sem pontas. Pipetar suavemente o volume necessário de actina polimerizada de 5 μM (a partir do passo 2.5.3.) com uma ponta de pipeta de extremidade romba (para evitar a cisalhamento dos filamentos de actina) e adicioná-lo a um tubo de PCR autoclavado limpo.
- Adicione 1x KMEH ao tubo para tornar o volume >20 μL e misture suavemente para evitar o cisalhamento da actina F. Da câmara de amostra montada, remova um volume igual do buffer.
- Adicione a solução de actina polimerizada à câmara e pipete suavemente para cima e para baixo 3x sem tocar na bicamada na parte inferior. Isso permite que os filamentos de actina se distribuam uniformemente na bicamada. Monte a amostra no microscópio TIRF (ver etapas 2.4.1. e 2.4.2.).
- Pode-se registrar o processo de ligação da actina F à bicamada. Incubar por 20-30 min. Grave algumas imagens de diferentes campos de visão após a adição de actina F ter atingido um estado estacionário. Observe a mudança na organização espacial de 10xHis-YFP-EzrinABD antes (homogêneo) e após a organização da actina.
NOTA: HYE é uniformemente distribuído sobre a bicamada lipídica na ausência de actina. Após a adição de filamentos de actina, HYE colocaliza-se com F-actina. A extensão da colocalização depende da afinidade de ligação à actina da proteína ligadora; quanto maior a afinidade, maior a colocalização e mais lenta a mobilidade lateral da proteína ligadora (Figura 5).
- Adição de miosina II
- Após 30 minutos de incubação da actina, monte a amostra de volta no microscópio (se ela estiver desmontada). Verifique o sinal na proteína ligadora e nos canais de actina F. Ajuste as condições de imagem, se necessário.
- Selecione uma boa região com sinal uniforme de proteína ligadora e filamentos de actina uniformemente espalhados e sem artefatos para um longo registro de lapso de tempo. Grave 10-15 quadros a 0,1-0,2 Hz antes da adição de miosina e pause a gravação. Pipetar o volume necessário de miosina muscular reciclada II do frasco para injetáveis de reserva com uma ponta de pipeta de extremidade romba (para evitar a cisalhamento dos filamentos de miosina) e adicionar a um tubo de PCR autoclavado limpo.
- Adicione imediatamente 1x KMEH ao tubo para tornar o volume >20 μL e misture suavemente. Pode-se também adicionar ATP, mistura regeneradora de ATP, agentes foto-estabilizadores, etc. durante esta etapa. Remova cuidadosamente um volume igual do tampão da câmara de amostra montada sem perturbá-la.
- Adicione suavemente a solução de miosina à câmara de amostra. Não pipete para cima e para baixo, pois isso perturbará os filamentos ligados à superfície. Retome imediatamente o registro de lapso de tempo e observe o sistema à medida que ele evolui do estado pré-miosina para fluxos contráteis de actomiosina alimentados por ATP e formação de aster para um estado emperrado empobrecido de ATP (ver resultados representativos).
- Tire imagens de fundo para todos os canais usando apenas uma amostra de buffer. Salve todas as imagens como arquivos de .tiff de 16 bits. Consulte a Tabela 2 para obter dicas para resolver problemas comuns.

Figura 3: Avaliação da qualidade das bicamadas com ensaio rápido de FRAP. As bicamadas lipídicas suportadas (SLBs) preparadas a partir de lipídios DOPC e Ni-NTA (98:2 mol%) são revestidas com HYE (ligador de membrana-actina marcado com 10xHis-YFP). Depois que a proteína não ligada é lavada, a bicamada fluorescente é fotografada sob um microscópio TIRF. Uma pequena região na bicamada é fotobranqueada com alta potência do laser, e a recuperação da fluorescência é registrada. (A) Uma boa bicamada sempre se recupera rapidamente, com um coeficiente de difusão esperado de 1-1,5 μm2/s para a composição lipídica utilizada neste caso. (B) Os bilayers ruins se recuperam muito lentamente ou não se recuperam. (C) Imagens representativas de bicamadas ruins: (C-i) uma bicamada com buracos, (C-ii) uma bicamada com manchas lipídicas grandes e imóveis e (C-iii) uma bicamada com pontos pequenos e imóveis. Por favor, clique aqui para ver uma versão maior desta figura.
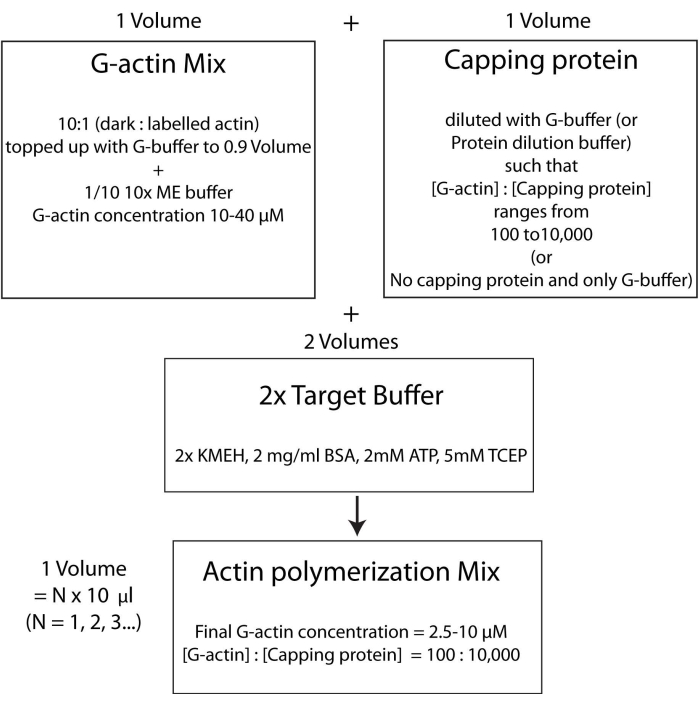
Figura 4: Esquema mostrando como polimerizar a actina usando o método do tampão de destino. Clique aqui para ver uma versão maior desta figura.

Figura 5: Organização espacial de HYE após a ligação à actina F. Instantâneos TIRF mostrando a organização espacial do HYE antes e após a adição de filamentos de actina (marcados com maleimida de Atto-635). A organização HYE é homogênea antes da adição de F-actina e torna-se colocalizada e co-alinhada ao longo dos filamentos de actina. Por favor, clique aqui para ver uma versão maior desta figura.
3. Análise dos dados
- Usando o software Fiji (https://imagej.net), subtraia o plano de fundo das imagens da proteína do vinculador (da etapa 2.4.). Meça os valores médios de intensidade do ponto branqueado e de uma região de referência.
- Normalizar os traços temporais do ponto branqueado e da região de referência até a intensidade de seus respectivos valores de intensidade pré-alvejante. Divida cada ponto de tempo nos valores da região branqueada normalizada pelos respectivos pontos de tempo no traço de tempo da região de referência normalizada. Corrija o traço de tempo normalizado resultante para o fundo e para quaisquer flutuações sistemáticas na intensidade durante a aquisição (fotobranqueamento global, deriva z, etc.).
- Utilizar um método manual e livre de encaixe28 para estimar o coeficiente de difusão das proteínas amarradas em duas camadas. Resumidamente, o intervalo de tempo do perfil de recuperação, τ1/2, pode ser calculado observando o momento em que o perfil de recuperação normalizado atinge metade de seu estado estacionário:
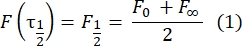
Aqui, F 0 é a intensidade média na região branqueada no primeiro quadro após o fotobranqueamento, e F∞ é o valor de estado estacionário de longo prazo da recuperação da bicamada. - Estimar o raio efetivo da água sanitária, re, parâmetro que corrige a difusão durante o fotobranqueamento, a partir de uma varredura de linha do perfil do ponto pós-alvejante29. A meia largura a meio mínimo desta varredura de linha que passa pelo centro do ponto de lixívia, r 1/2, refere-se a re da seguinte forma:
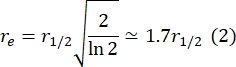
O τ1/2 calculado na etapa 2.8.3., r e calculado na etapa 2.8.4., e o raio de lixívia originalmente definido, rn, são usados para calcular o coeficiente de difusão (D) usando a seguinte fórmula: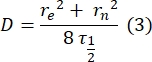
- Utilizar um método manual e livre de encaixe28 para estimar o coeficiente de difusão das proteínas amarradas em duas camadas. Resumidamente, o intervalo de tempo do perfil de recuperação, τ1/2, pode ser calculado observando o momento em que o perfil de recuperação normalizado atinge metade de seu estado estacionário:
- Análise de imagens de actomiosina asters
- Usando Fiji, subtraia o fundo de todas as imagens gravadas em todos os canais. Corrija as imagens para qualquer padrão de iluminação ou interferência não uniforme usando a correção de campo plano.
NOTA: Pode-se usar lâminas de plástico coloridas, que são boas amostras planas para fazer tais correções. Para os filamentos de proteína e actina ligantes em uma bicamada plana, pode-se também usar a projeção média de múltiplas imagens pré-miosina para criar mapas de correção de iluminação específicos do canal.- Para o canal HYE, mostrado aqui, faça uma projeção de intensidade média de várias imagens HYE (gravadas de diferentes regiões da bicamada lipídica antes da adição de miosina). Aplique um filtro gaussiano apropriado (σ = 50 pixels a 80 pixels) à projeção média (de imagens pré-miosina ou qualquer amostra plana padrão).
- Converta a imagem filtrada em uma imagem de 32 bits. Divida todos os valores de pixel pela média da imagem inteira. Isso fornecerá um mapa de correção normalizado para o canal HYE. Divida todas as imagens no canal HYE com este mapa para correção de campo plano. Crie mapas de correção para outros canais usando a mesma estratégia.
- Correto para fotobranqueamento usando um método de razão exponencial ou simples (dependendo do perfil de decaimento de intensidade) em Fiji.
- Para corrigir qualquer desalinhamento temporal x-y (movimento de translação), mescle todos os canais corrigidos por fotobranqueamento em um único Hyperstack. Usando o plugin Hyperstack-Reg em Fiji, aplique uma transformação de Corpo Rígido ou Tradução.
- Por fim, divida o Hyperstack alinhado em canais individuais e salve-os separadamente como pilhas TIFF de 16 bits para análise posterior.
- Usando Fiji, subtraia o fundo de todas as imagens gravadas em todos os canais. Corrija as imagens para qualquer padrão de iluminação ou interferência não uniforme usando a correção de campo plano.
Resultados
Para representação, aqui é mostrado um perfil típico de pós-bláspero da 1ª imagem após o fotobranqueamento (imagem em t = 0 s na Figura 3A) e seu ajuste à seguinte função28 (ver Figura 6A):

O valor de r e (23,94 μm) calculado pelo ajuste a esta curva é muito semelhante ao r e calculado no passo 2.8.4. (23,24 μm). Aqui, K é um parâmetro de profundidade de água sanitária que pode ser estimado diretamente a partir de F0 (descrito na etapa 2.8.4.). Da mesma forma, a Figura 6B mostra o perfil de recuperação e seu ajuste à seguinte função28:
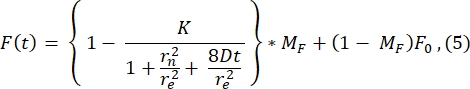
Encontramos o valor ajustado do coeficiente de difusão como sendo 1,34 μm 2/s, um valor que concorda estreitamente com o valor de 1,39 μm 2/s que é calculado pela fórmula na etapa2.8.4. Aqui, MF representa a fração móvel da bicamada lipídica que representa a fração da população branqueada que se recupera de volta. A mobilidade das moléculas ancoradas em lipídios depende, é claro, da composição lipídica e de seu estado físico (fase líquida ou gel). Para nossos experimentos usando membranas lipídicas à base de DOPC, a mobilidade deve ser >1 μm2/s, e a fração móvel não deve ser inferior a 0,9 para indicar uma boa bicamada lipídica. Recomendamos o uso do método manual sem encaixe para um teste rápido da qualidade e mobilidade da bicamada. O método de ajuste pode ser útil ao automatizar a análise para muitas curvas FRAP. Além disso, se alguém quiser realizar um experimento FRAP mais sofisticado para caracterizar sistematicamente a difusão no sistema, recomendamos ao leitor esta revisão de Lorén et al.30 para obter mais detalhes sobre modelos de ajuste e possíveis armadilhas no delineamento experimental.

Figura 6: Quantificação do coeficiente de difusão das bicamadas lipídicas. (A) Perfil da linha da primeira imagem após o fotobranqueamento (t = 0 s na Figura 3A) e seu ajuste à equação 4 para calcular o raio efetivo de alvejante. (B) O perfil de recuperação da região branqueada e seu ajuste à equação 5 para cálculo do coeficiente de difusão e fração móvel. Por favor, clique aqui para ver uma versão maior desta figura.
Um resultado típico dos experimentos descritos acima, mostrando a montagem dinâmica e a organização de uma rede de acto-miosina ligada em uma bicamada lipídica suportada por microscopia TIRF, é representado na Figura 7 e no Vídeo Suplementar S1.
A Figura 7 mostra uma montagem de imagem da proteína ligadora, F-actina e miosina-II.
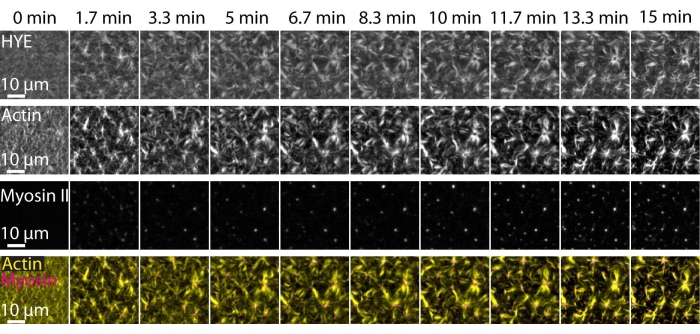
Figura 7: Os fluxos contráteis de actomiosina conduzem ao agrupamento local da proteína ligadora membrana-actina HYE. Instantâneos TIRF de HYE (marcados com YFP), filamentos de actina (marcados com maleimida de Atto-635) e filamentos de miosina II (marcados com maleimida de Atto-565) após a adição de miosina II a um SLB contendo HYE e F-actina. O tempo é indicado no topo: 0 min é imediatamente antes que os miofilamentos fluorescentes comecem a aparecer no campo TIRF. HYE e F-actina são distribuídos homogeneamente sobre a bicamada lipídica antes da adição de miosina (0 min). A atividade da miosina induz fluxos contráteis de actomiosina, que emergem em estruturas semelhantes a aster no estado estacionário (15 min), levando ao agrupamento local do componente de membrana acoplado (HYE). A linha mais baixa é uma fusão de imagens de actina (amarelo) e miosina II (magenta) mostrando a organização da actina e miosina em diferentes pontos de tempo. As imagens usadas na confecção dessas montagens foram corrigidas em Fiji para sinal de fundo, padrões de intensidade não uniformes e movimento de translação. Barra de escala = 10 μm. Para obter detalhes, consulte Vídeo suplementar S1. Por favor, clique aqui para ver uma versão maior desta figura.
| Nome do buffer | Composição | |
| Tampão de Reidratação Lipídica | 50 mM HEPES, 150 mM NaCl, 5% de sacarose, pH 7,5 | |
| Buffer de formação SLB | 50 mM HEPES, 150 mM NaCl, pH 5-6 | |
| Buffer de armazenamento SLB | 50 mM HEPES, 150 mM NaCl, pH 7,2 | |
| Tampão de diluição de proteínas | 20 mM HEPES, 100 mM KCl, 1mM TCEP ou TDT, pH 7,2 | |
| 1X ME ou buffer de troca iônica Actin | 50 mM MgCl2, 0,2 mM EGTA, 10 mM HEPES, pH 7,2 (armazenar a 4°C) | |
| 1X KMEH ou tampão de polimerização Actin | 50 mM KCl, 1 mM MgCl2, 1 mM EGTA, 50 mM HEPES, pH 7,2 | |
| Estoque de ATP de 100 mM | Sal dissódico ATP de 100 mM, Tris de 50 mM, NaCl de 50 mM, MgCl2 de 5 mM, EGTA de 2 mM, pH 7,5 (armazenar a -20°C) | |
| 2x buffer de destino | 2x KMEH, 2 mg/ml BSA, 2mM ATP, 5mM TCEP (armazenado a 4°C) | |
| G-buffer | 2 mM Tris, 0,1 mM CaCl2, 0,2 mM ATP, 0,5 mM TCEP, 0,04 % NaN3, pH 8 (armazenar a 4°C) | |
| Tampão de miosina II | 500 mM KCl, 1 mM EDTA, 10-20 mM Hepes, pH 7,0 | |
| Tampão de cromatografia por filtração em gel | 50 mM Tris-HCl, 150-300 mM NaCl, 5 mM TCEP, 0,1% Tween-20, pH 7,5 | |
| Tampando o tampão de armazenamento de proteína | Tris de 10 mM· Cl, 50 mM NaCl, 1 mM TCEP, pH 7,5, 20% de glicerol | |
Tabela 1: Lista de composições de buffer usadas neste protocolo.
| Problemas comuns e sua solução de problemas | Problema | Causa | Soluções possíveis | ||||||
| 1 | A bicamada lipídica não apresenta difusão | A causa mais provável para este problema é o vidro de cobertura sujo que pode acontecer quando a solução de limpeza é envelhecida ou o aquecimento não ocorreu durante a sonicação do banho. Essas bicamadas têm uma aparência "vesicular" porque as vesículas estouradas aderem ao vidro da tampa, mas não se fundem umas com as outras. Usar MLVs com mais de 6 semanas ou SUVs com mais de 6 dias, ou adicionar baixas quantidades de SUVs também pode levar à formação de bicamadas vesiculares. | Use uma solução de limpeza fresca. Certifique-se de que o aquecedor está ligado e a temperatura está entre 45-65°C. Use misturas lipídicas frescas. (O uso de uma sonda lipídica fluorescente versus uma sonda de proteína fluorescente às vezes pode se manifestar de forma diferente. Por exemplo, se a bicamada tiver defeitos de subdifração e a etapa de passivação superficial for ignorada (ou não funcionar), a sonda lipídica mostrará uma distribuição de intensidade uniforme, mas a sonda de proteína fluorescente poderá exibir pontos fluorescentes brilhantes.) | ||||||
| 2 | A bicamada lipídica tem manchas brilhantes | A incubação longa de SUVs para a formação de bicamadas pode criar uma bicamada lipídica que é difusora em geral, mas com manchas brilhantes ocasionais. Essas manchas podem ser bicamadas de várias camadas que podem atrair grandes quantidades de sonda fluorescente. | 15-20 min de incubação com SUVs é suficiente. Certifique-se de que a sonda não está se agregando: um giro rápido e duro da proteína do ligador (300 x g por 15 min a 4 °C) pode remover os agregados | ||||||
| 3 | A bicamada lipídica tem buracos escuros | Isso acontece quando a bicamada é feita de SUVs antigos e fotografada por horas prolongadas (> 4 horas após a formação), ou o pH da solução muda drasticamente devido a imagens prolongadas (por exemplo, no estado ATP alto e na presença de certos sequestradores de oxigênio), ou quando a superfície é passivada em excesso com beta-caseína (adicionando muita beta-caseína por mais de 10-15 minutos e ou não lavando-a). | Use lipídios frescos. Reduza a taxa de quadros de imagem ou o tempo efetivo de iluminação a laser. Use buffers com maior capacidade de buffering. | ||||||
| 4 | Bicamada lipídica apresenta difusão lenta | Bicamadas lipídicas com alta porcentagem de colesterol, lipídios saturados longos ou lipídios carregados difundem-se mais lentamente. | Nesses casos, prepare sua amostra a uma temperatura alta. Pode-se também usar uma composição lipídica simples e testada como controle, juntamente com composições lipídicas complexas e não testadas. Certifique-se de que o vidro está limpo. | ||||||
| 5 | A actina não polimeriza | O tampão alvo é antigo, o estoque de G-actina é muito antigo, o antigo e o novo G-actina foram copolimerizados. | Certifique-se de que o Ca 2+ é substituído por Mg2+ antes da polimerização (usando o tampão ME). Use ATP-Mg2+ fresco estoque. Use G-actina recém-reciclada. Certifique-se de que a concentração de F-actina (em termos de G-actina) adicionada à bicamada é superior a 0,2 μM. Para concentrações mais baixas, use faloidina estabilizada F-actina. | ||||||
| 6 | A actina não se liga à bicamada | O ligador membrana-actina não é adicionado ou adicionado em concentração muito baixa – isso pode ser inferido a partir da fluorescência da proteína ligadora. Se a fluorescência for decente, o ligador membrana-actina perdeu a capacidade de ligação à actina. Além disso, se a proteína ligadora não estiver especificamente ligada à superfície do vidro (quando a bicamada é ruim), ela pode não recrutar filamentos de actina. | Certifique-se de que a bicamada está se difundindo. Use proteína de ligação fresca | ||||||
| 7 | O sinal fluorescente de acetina F é fraco | A proporção de actina rotulada para actina escura é muito baixa. Ou a actina rotulada ou a actina não marcada é muito antiga e eles não estão copolimerizando uns com os outros. | Recicle a actina novamente e tente novamente a eloirização com actina recém-reciclada. O fotodano pode destruir ou despolimerizar a F-actina; se possível, use corantes vermelhos ou vermelhos para actina (e miosina). | ||||||
| 8 | A miosina não mostra contratilidade | pode-se observar que, após a adição de ATP ao sistema infundido com miosina, não há contratilidade da actomiosina. | Verifique se a concentração de miosina ou o nível de pureza é bom. Use miosina recém-reciclada (use dentro de 6 semanas após a reciclagem). Adicionar ATP fresco à mistura de miosina pode ajudar. Desgaseificação de buffers e uso de sequestradores de oxigênio, etc. pode reduzir o fotodano dos motores. Mais informações podem ser encontradas nos protocolos de Plastino et al. ou Stam et al. da mesma coleção de métodos | ||||||
| 9 | Coverglass não é hidrofílico | O vidro da tampa não é limpo corretamente. | O vidro de cobertura hidrofílico limpo é crucial para a formação de bicamadas lipídicas. Uma leitura útil e visual da hidrofilicidade do vidro de cobertura após o protocolo de limpeza é observar a umectação do copo pela água. Adicione um pequeno volume de água a uma tampa plana. A água permanecerá na forma de uma gota redonda se a tampa não for limpa corretamente. No entanto, o mesmo volume de água se espalhará e formará uma camada fina, em uma coberta hidrofílica tratada. Este comportamento de umectação da água na superfície do vidro de cobertura pode ser usado para verificar se as etapas de limpeza com a solução de limpeza / NaOH funcionaram. | ||||||
Tabela 2: Guia de solução de problemas resumindo problemas comuns e soluções correspondentes.
Vídeo Suplementar S1: Os fluxos de actomiosina contrátil conduzem o agrupamento local da proteína ligadora membrana-actina HYE. Timelapse TIRF de HYE (marcado com YFP), filamentos de actina (marcados com maleimida de Atto-635) e filamentos de miosina II (marcados com maleimida de Atto-565) após a adição de miosina II a um SLB contendo HYE e F-actina. O tempo é indicado no topo: 0 min é imediatamente antes que os miofilamentos fluorescentes comecem a aparecer no campo TIRF. Barra de escala = 10 μm. Clique aqui para baixar este arquivo.
Discussão
Este protocolo apresenta uma plataforma versátil e um ponto de partida para projetar experimentos para estudar a interface membrana-córtex das células. As etapas críticas são a preparação de lâminas de vidro limpas, usando lipídios frescos para a formação eficiente de SUV (ambos afetando a qualidade dos SLBs) e o uso de proteínas de miosina II recém-recicladas para a reorganização dinâmica do filamento de actina. Ao fazer a dinâmica da imagem por um longo período de tempo, é muito importante incorporar um sistema de sequestro de oxigênio (por exemplo, ácido protocatecúico e protocatecuato 3 4-dioxigenase 5,31).
O projeto de câmara aberta permite a adição sequencial de componentes a um sistema existente sem induzir fluxos lipídicos. Isso pode ser uma vantagem importante sobre as abordagens de câmara fechada comumente usadas ou trabalhar usando proteínas encapsuladas dentro de lipossomas36. Efeitos contrários, como a deformação da membrana induzida por proteínas, não podem ser estudados com bicamadas lipídicas adsorvidas em vidro.
As bicamadas lipídicas podem ser formadas com uma ampla gama de composições lipídicas. Inicia-se com a adsorção das vesículas lipídicas à superfície do vidro hidrofílico, seguida de ruptura espontânea da vesícula devido às interações superfície-vesícula e vesícula-vesícula direta ou das vesículas adsorvidas atingindo uma cobertura crítica após a qual uma pequena fração das vesículas se rompe, formando bordas ativas, o que eventualmente leva à formação de duas camadas32 . Além do vidro, vários substratos podem ser utilizados para formar bicamadas lipídicas suportadas, como Mica (por exemplo, para microscopia de força atômica), substratos moles (por exemplo, poli-di-metil-siloxano), almofadas poliméricas33,34,35, abrangendo entre orifícios de grades de microscopia eletrônica 14. As bicamadas de interface de gotículas são outro método interessante para criar bicamadas lipídicas estáveise independentes 36. A inclusão de redes de acto-miosina em vesículas ou emulsões é um método muito poderoso para estudar esse sistema mínimo em uma geometria semelhante a uma célula37,38, e que é descrito em detalhes em outro lugar 39.
Divulgações
Os autores não têm conflitos de interesse a declarar.
Agradecimentos
Este trabalho foi apoiado pelo fundo de pesquisa AXA e pelo Programa de Biomedicina Quantitativa Warwick-Wellcome (Wellcome ISSF, RMRCB0058) para DVK, NCBS-TIFR para AB e ST, e a bolsa Wellcome-DBT Margdarshi (IA / M / 15 / 1 / 502018) para SM. A DSK DSK também gostaria de agradecer à Sociedade Biofísica por permitir o evento de rede virtual "Desafios na compreensão de redes citoesqueléticas multicomponentes da molecular à meso-escala", o que contribuiu para a criação dessa coleção de protocolos.
Materiais
| Name | Company | Catalog Number | Comments |
| 1,2 dipalmitoyl-sn-glycero-3-phosphoethanolamine-N- (lissamine rhodamine B sulfonyl) | Avanti Polar Lipids | 810158 | 16:0 RhoPE |
| 1,2-dioleoyl-sn-glycero-3- [(N-(5-amino-1 carboxypentyl) iminodiacetic acid) succinyl] (nickel salt) | Avanti Polar Lipids | 790404 | DGS-NTA-Ni2+ |
| 1,2-dioleoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850375 | DOPC |
| 1,2-dipalmitoyl-sn-glycero-3-phosphocholine | Avanti Polar Lipids | 850355 | DPPC |
| Amber glass vials | ThermoFisher | B7990-2A | |
| ATP disodium salt | Sigma Aldrich | A26209 | |
| Attofluor cell chamber | ThermoFisher | A7816 | |
| Bath sonicator | GT Sonic | 1860QTS | |
| beta-casein | Sigma Aldrich | C6905 | |
| CaCl2 | ThermoFisher | 12135 | |
| chloroform | Sigma Aldrich | 650471 | alternatively from Electron Microscopy Sciences, 50980296 |
| Cover slips, #1, 25 mm diameter, Gold Seal | Harvard Apparatus | 64-0705B | |
| Cover slips, #1, 40x22 mm, Gold Seal | ThermoFisher | 48404-031 | |
| EDTA | ThermoFisher | G12635 | |
| EGTA | Himedia | MB130 | |
| Gas tight glass syringes, with removebla needle, blunt, volumes 10 µL, 100 µL, 500 µL | Hammilton | 1700 series | |
| Hellmanex III | Hellma Analytics | Z805939 | cleaning solution |
| HEPES | Himedia | RM380 | |
| KaH2PO4 | ThermoFisher | G13405 | |
| KCl | ThermoFisher | G13305 | |
| KOH | ThermoFisher | G26708 | |
| Lipid extruder | Avanti Polar Lipids | 61000-1EA | |
| MgCl2 | ThermoFisher | G15535 | |
| Microtip sonicator | Sonics | VC750 | 3 mm Tip diameter |
| Na2CO3 | ThermoFisher | G15955 | |
| NaCl | Himedia | GRM853 | |
| NaH2PO4 | ThermoFisher | G15825 | |
| NaOH | ThermoFisher | G27815 | |
| Nikon Ti Eclipse TIRF microscope | Nikon | With a TIRF unit connected through a polarization-conserving optical fibre to an Agilent monolithic laser combiner MLC400 with multiple laser lines with a 100X, 1.45 NA Nikon Oil Objective with two 512 x 512-pixel EMCCD cameras (Photometrics Evolve 512) with a 100X, 1.45 NA Nikon Oil Objective with two 512 x 512-pixel EMCCD cameras (Photometrics Evolve 512) | |
| NOA88 | Norland Products | 8801 | |
| PTFE Coated Tweezer Style #2A | Structure Probe | 0S2AT-XD | |
| Refrigerated microcentrifuge | Eppendorf | 5424R | |
| Sucrose | ThermoFisher | G15925 | |
| UV-illuminator | Novascan | PSD PRO-UV | needs vacuum and O2 supply |
Referências
- Köster, D. V., Mayor, S. Cortical actin and the plasma membrane: Inextricably intertwined. Current Opinion in Cell Biology. 38, 81-89 (2016).
- Rao, M., Mayor, S. Active organization of membrane constituents in living cells. Current Opinion in Cell Biology. 29, 126-132 (2014).
- Honigmann, A., et al. A lipid bound actin meshwork organizes liquid phase separation in model membranes. eLife. 3, 01671(2014).
- Das, A., et al. Stratification relieves constraints from steric hindrance in the generation of compact actomyosin asters at the membrane cortex. Science Advances. 6 (11), (2020).
- Köster, D. V., et al. Actomyosin dynamics drive local membrane component organization in an in vitro active composite layer. Proceedings of the National Academy of Sciences. 113 (12), 1645-1654 (2016).
- Vogel, S. K., Heinemann, F., Chwastek, G., Schwille, P. The design of MACs (minimal actin cortices). Cytoskeleton. 70 (11), 706-717 (2013).
- Murrell, M., Thoresen, T., Gardel, M. Reconstitution of contractile actomyosin arrays. Methods in Enzymology. 540, 265-282 (2014).
- Mosby, L. S., et al. Myosin II filament dynamics in actin networks revealed with interferometric scattering microscopy. Biophysical Journal. 118 (8), 1946-1957 (2020).
- Seara, D. S., et al. Entropy production rate is maximized in non-contractile actomyosin. Nature Communications. 9 (1), 4948(2018).
- Linsmeier, I., et al. Disordered actomyosin networks are sufficient to produce cooperative and telescopic contractility. Nature Communications. 7, 12615(2016).
- Vogel, S. K., Petrasek, Z., Heinemann, F., Schwille, P. Myosin motors fragment and compact membrane-bound actin filaments. eLife. 2, 00116(2013).
- Ditlev, J. A., et al. A composition-dependent molecular clutch between T cell signaling condensates and actin. eLife. 8, 42695(2019).
- Banjade, S., Rosen, M. K. Phase transitions of multivalent proteins can promote clustering of membrane receptors. eLife. 3, 04123(2014).
- Heinemann, F., Vogel, S. K., Schwille, P. Lateral membrane diffusion modulated by a minimal actin cortex. Biophysical Journal. 104 (7), 1465-1475 (2013).
- Vogel, S. K., Greiss, F., Khmelinskaia, A., Schwille, P. Control of lipid domain organization by a biomimetic contractile actomyosin cortex. eLife. 6, 24350(2017).
- Block, S. Brownian motion at lipid membranes: A comparison of hydrodynamic models describing and experiments quantifying diffusion within lipid bilayers. Biomolecules. 8 (2), 30(2018).
- JoVE, JoVE. JoVE Methods Collection. In vitro reconstitution of cytoskeleton networks for biomaterials, biophysics and active matter research. Journal of Visualized Experiments. , Cambridge, MA. (2022).
- Pardee, J. D., Spudich, J. A. Purification of muscle actin. Methods in Enzymology. 85, 164-181 (1982).
- Spudich, J. A., Watt, S. The regulation of rabbit skeletal muscle contraction. I. Biochemical studies of the interaction of the tropomyosin-troponin complex with actin and the proteolytic fragments of myosin. Journal of Biological Chemistry. 246 (15), 4866-4871 (1971).
- Pollard, T. D. Myosin purification and characterization. Methods in Cell Biology. 24, 331-371 (1982).
- JoVE Science Education Database. Separating protein with SDS-PAGE. Journal of Visualized Experiments. , Cambridge, MA. (2022).
- Bieling, P., et al. WH2 and proline-rich domains of WASP-family proteins collaborate to accelerate actin filament elongation. The EMBO Journal. 37 (1), 102-121 (2018).
- Shrivastava, R., Köster, D., Kalme, S., Mayor, S., Neerathilingam, M. Tailor-made ezrin actin binding domain to probe its interaction with actin in-vitro. PLoS One. 10 (4), 0123428(2015).
- Nye, J. A., Groves, J. T. Kinetic control of histidine-tagged protein surface density on supported lipid bilayers. Langmuir. 24 (8), 4145-4149 (2008).
- Honigmann, A., et al. Scanning STED-FCS reveals spatiotemporal heterogeneity of lipid interaction in the plasma membrane of living cells. Nature Communications. 5, 5412(2014).
- Selden, N. S., et al. Chemically programmed cell adhesion with membrane-anchored oligonucleotides. Journal of the American Chemical Society. 134 (2), 765-768 (2012).
- Seu, K. J., et al. Effect of surface treatment on diffusion and domain formation in supported lipid bilayers. Biophysical Journal. 92 (7), 2445-2450 (2007).
- Kang, M., Day, C. A., Kenworthy, A. K., DiBenedetto, E. Simplified equation to extract diffusion coefficients from confocal FRAP data. Traffic. 13 (12), 1589-1600 (2012).
- Kang, M., Day, C. A., Kenworthy, A. K. A novel computational framework for D(t) from Fluorescence Recovery after Photobleaching data reveals various anomalous diffusion types in live cell membranes. Traffic. 20 (11), 867-880 (2019).
- Lorén, N., et al. Fluorescence recovery after photobleaching in material and life sciences: Putting theory into practice. Quarterly Reviews of Biophysics. 48 (3), 323-387 (2015).
- Alvarado, J., Sheinman, M., Sharma, A., MacKintosh, F. C., Koenderink, G. H. Molecular motors robustly drive active gels to a critically connected state. Nature Physics. 9 (7), 1-7 (2013).
- Andrecka, J., Spillane, K. M., Ortega-Arroyo, J., Kukura, P. Direct observation and control of Supported lipid bilayer formation with interferometric scattering microscopy. ACS Nano. 7 (12), 10662-10670 (2013).
- Lin, W. -C., et al. Supported membrane formation, characterization, functionalization, and patterning for application in biological science and technology. Current Protocols in Chemical Biology. 2 (4), 235-269 (2010).
- Richter, R. P., Bérat, R., Brisson, A. R. Formation of solid-supported lipid bilayers: An integrated view. Langmuir. 22 (8), 3497-3505 (2006).
- Sapuri-Butti, A. R., Butti, R. C., Parikh, A. N. Characterization of supported membranes on topographically patterned polymeric elastomers and their applications to microcontact printing. Langmuir. 23 (25), 12645-12654 (2007).
- Leptihn, S., et al. Constructing droplet interface bilayers from the contact of aqueous droplets in oil. Nature Protocols. 8 (6), 1048-1057 (2013).
- Bashirzadeh, Y., Moghimianavval, H., Liu, A. P. Encapsulated actomyosin patterns drive cell-like membrane shape changes. iScience. 25 (5), 104236(2022).
- Bashirzadeh, Y., Wubshet, N., Litschel, T., Schwille, P., Liu, A. P. Rapid encapsulation of reconstituted cytoskeleton inside giant unilamellar vesicles. Journal of Visualized Experiments. (177), e63332(2021).
- Murrell, M., Chen, S., Sun, Z. G. In vitro reconstitution of actin cytoskeleton inside giant unilamellar vesicles. Journal of Visualized Experiments. , (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados