Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Protocole de dosage de cisaillement pour la détermination des propriétés d’un matériau unicellulaire
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ce protocole décrit la quantification des propriétés mécaniques des lignées cellulaires cancéreuses et non cancéreuses in vitro. Les différences conservées dans la mécanique des cellules cancéreuses et normales peuvent agir comme un biomarqueur qui peut avoir des implications dans le pronostic et le diagnostic.
Résumé
La biomécanique irrégulière est une caractéristique de la biologie du cancer qui fait l’objet d’études approfondies. Les propriétés mécaniques d’une cellule sont similaires à celles d’un matériau. La résistance d’une cellule au stress et à la tension, son temps de relaxation et son élasticité sont autant de propriétés qui peuvent être dérivées et comparées à d’autres types de cellules. La quantification des propriétés mécaniques des cellules cancéreuses (malignes) par rapport aux cellules normales (non malignes) permet aux chercheurs de découvrir davantage les fondements biophysiques de cette maladie. Bien que les propriétés mécaniques des cellules cancéreuses soient connues pour différer systématiquement des propriétés mécaniques des cellules normales, il n’existe pas de procédure expérimentale standard pour déduire ces propriétés à partir de cellules en culture.
Cet article décrit une procédure pour quantifier les propriétés mécaniques de cellules individuelles in vitro à l’aide d’un test de cisaillement fluide. Le principe derrière ce test consiste à appliquer une contrainte de cisaillement fluide sur une seule cellule et à surveiller optiquement la déformation cellulaire résultante au fil du temps. Les propriétés mécaniques des cellules sont ensuite caractérisées à l’aide de l’analyse de corrélation d’images numériques (DIC) et de l’ajustement d’un modèle viscoélastique approprié aux données expérimentales générées par l’analyse DIC. Dans l’ensemble, le protocole décrit ici vise à fournir une méthode plus efficace et ciblée pour le diagnostic des cancers difficiles à traiter.
Introduction
L’étude des différences biophysiques entre les cellules cancéreuses et non cancéreuses ouvre de nouvelles possibilités diagnostiques et thérapeutiques1. Comprendre comment les différences en biomécanique/mécanobiologie contribuent à la progression tumorale et à la résistance au traitement révélera de nouvelles voies pour la thérapie ciblée et le diagnostic précoce2.
Bien que l’on sache que les propriétés mécaniques des cellules cancéreuses diffèrent de celles des cellules normales (p. ex. viscoélasticité de la membrane plasmique et de l’enveloppe nucléaire)3,4,5, il manque des méthodes robustes et reproductibles pour mesurer ces propriétés dans les cellules vivantes6. La méthode de dosage de cisaillement est utilisée pour quantifier les propriétés mécaniques des cellules en soumettant des cellules individuelles à une contrainte de cisaillement fluide et en analysant leurs réponses individuelles et leur résistance à la contrainte appliquée 3,4,5,7,8,9. Bien que plusieurs méthodes et techniques aient été utilisées pour caractériser les propriétés mécaniques de cellules individuelles, celles-ci ont tendance à affecter les propriétés des matériaux cellulaires en i) perforant/endommageant la membrane cellulaire en raison de la profondeur d’indentation, des géométries complexes de l’extrémité ou du raidissement du substrat associé à la microscopie à force atomique (AFM)10,11, ii) induisant des photodommages cellulaires pendant le piégeage optique 12, 13, ou iii) induisant des états de stress complexes associés à l’aspiration de micropipettes14,15. Ces effets externes sont associés à des incertitudes importantes dans la précision des mesures de viscoélasticité cellulaire 6,16,17.
Pour remédier à ces limitations, la méthode d’essai de cisaillement décrite ici fournit une approche simple et hautement contrôlable pour simuler le flux physiologique dans le corps sans affecter les propriétés du matériel cellulaire dans le processus. Les contraintes de cisaillement des fluides dans ce test représentent les contraintes mécaniques subies par les cellules du corps, soit par les fluides dans l’interstitium tumoral, soit dans le sang pendant la circulation18,19,20. En outre, ces stress fluides favorisent divers comportements malins dans les cellules cancéreuses, y compris la progression, la migration, les métastases et la mort cellulaire 19,21,22,23 qui varient entre les cellules tumorigènes et non tumorigènes. De plus, les caractéristiques mécaniques altérées des cellules cancéreuses (c’est-à-dire qu’elles sont souvent plus « molles » que les cellules normales trouvées dans le même organe) leur permettent de persister dans des microenvironnements tumoraux hostiles, d’envahir les tissus normaux environnants et de métastaser vers des sites éloignés24,25,26. En créant un environnement pseudo-biologique où les cellules subissent des niveaux physiologiques de stress de cisaillement des fluides, un processus physiologiquement pertinent et non destructeur pour la cellule est obtenu. Les réponses cellulaires à ces contraintes de cisaillement de fluide appliquées nous permettent de caractériser les propriétés mécaniques des cellules.
Cet article fournit un protocole d’essai de cisaillement pour l’étude approfondie des propriétés mécaniques et du comportement des cellules cancéreuses et non cancéreuses soumises à une contrainte de cisaillement appliquée. Les cellules répondent aux forces extérieures de manière élastique et visqueuse et peuvent donc être idéalisées comme un matériau viscoélastique3. Cette technique est classée en: (i) culture cellulaire de cellules individuelles dispersées, (ii) application contrôlée de contrainte de cisaillement fluide, (iii) imagerie in situ et observation du comportement cellulaire (y compris la résistance au stress et à la déformation), (iv) analyse de la déformation des cellules pour déterminer l’étendue de la déformation, et (v) caractérisation des propriétés viscoélastiques des cellules individuelles. En interrogeant ces propriétés mécaniques et ces comportements, la mécanobiologie cellulaire complexe peut être distillée en données quantifiables. Un protocole décrivant cette méthode permet le catalogage et la comparaison entre divers types de cellules malignes et non malignes. La quantification de ces différences a le potentiel d’établir des biomarqueurs diagnostiques et thérapeutiques.
Protocole
1. Préparation pour le test de cisaillement unicellulaire
- Culture cellulaire
- Ensemencer environ 50 000 cellules simples en suspension dans une boîte de Petri de 35 mm x 10 mm contenant 2 ml de milieux de culture.
REMARQUE : Vortex des cellules en suspension avant l’ensemencement pour briser les agrégats cellulaires. - Incuber les cellules à 37 °C et laisser entre 10 et 48 h pour l’attachement cellulaire et la formation complète des protéines cytosquelettiques.
REMARQUE: Tenez compte de la durée de l’attachement cellulaire, ainsi que des taux de prolifération et de croissance, pour assurer une croissance et une fixation cellulaires adéquates tout en évitant l’agrégation cellulaire. Ces paramètres varient selon le type de cellule.
- Ensemencer environ 50 000 cellules simples en suspension dans une boîte de Petri de 35 mm x 10 mm contenant 2 ml de milieux de culture.
2. Expérience d’essai de cisaillement
- Préparation du fluide d’écoulement visqueux de l’essai de cisaillement
- Pour obtenir un fluide légèrement visqueux (0,015-0,02 Pa·s), mesurer et ajouter 0,05 % en poids de méthylcellulose non toxique et non allergène (4 Pa·s) dans le milieu de culture.
- Pour assurer un mélange homogène, préchauffer le milieu de culture de base pendant ~10-20 min à une température de ~60-70 °C avec un agitateur magnétique/plaque chauffante. Tout en remuant continuellement le média, ajouter doucement la méthylcellulose de manière à ce qu’elle se disperse rapidement, afin d’éviter la coagulation des particules de méthylcellulose. Laissez ce processus se poursuivre pendant ~15-24 h pour assurer une solution claire de milieu + cellulose.
REMARQUE: Évitez de chauffer excessivement la solution. - Pour mesurer la viscosité du fluide d’écoulement, tester ~0,5-1 mL du fluide d’écoulement représentatif à l’aide d’un rhéomètre. À partir de la lecture, déterminez la viscosité du fluide et utilisez cette valeur pour représenter la viscosité du milieu fluide de cisaillement (μ) afin de calculer la contrainte de cisaillement à l’aide de l’équation (2).
- Installation de l’appareil de cisaillement
- Installer le système de dosage de cisaillement de seringues doubles (60 ml ou 100 ml) connectées à une pompe à seringue programmable pour la perfusion et le retrait du milieu de culture visqueux (figure 1).
- Fixez les deux seringues à la chambre d’écoulement via des tubes de 1/16 de pouce et des connecteurs de tube.
- Fixez un joint en caoutchouc pour assurer un écoulement contrôlé et uniforme sur des cellules individuelles le long du trajet d’écoulement (Figure 1). Le joint en caoutchouc est disponible en différentes tailles selon le profil d’écoulement à atteindre (laminaire ou turbulent) et la zone d’observation souhaitée (par exemple, longueur de 22,5 mm, largeur de 2,5 mm et hauteur de 0,254 mm) (figure 1).
- Programmez la pompe pour qu’elle perfuse et retire un certain volume de liquide (p. ex. 60 mL) à une vitesse désignée (p. ex. 1 mL/min) et choisissez les seringues correspondantes (p. ex. 60 mL).
REMARQUE : Tenez compte des préréglages de volume maximal de perfusion et de retrait pour éviter le bourrage ou le mauvais fonctionnement. Utiliser l’équation (2) pour le calcul du taux de cisaillement de la pompe requis (en supposant que la contrainte et la viscosité requises sont connues).
- Configuration pré-cisaillement
- Remplissez la seringue avec le fluide visqueux préparé.
- Fixez la seringue, remplie de 60 ou 100 mL (ou au besoin) de fluide visqueux, et une seringue vide de 60 mL à leurs emplacements respectifs sur la pompe à seringue programmable. Via des connecteurs de tubes et de tubes, connectez les deux seringues à la chambre d’écoulement.
- Pour assurer une identification facile des cellules individuelles et une connexion fixée entre la chambre d’écoulement et la boîte de Pétri, fixez le joint en caoutchouc à la chambre d’écoulement.
- Aspirer les milieux de culture cellulaire de la boîte de Petri contenant les cellules d’intérêt.
- Laver les cellules mortes et les cellules faiblement attachées à l’aide d’une solution saline tamponnée au phosphate (PBS).
- Aspirez le PBS.
- Insérez et fixez la chambre d’écoulement et le joint en caoutchouc (~34 mm x 9 mm) sur la boîte de Petri (35 mm x 10 mm) contenant les cellules attachées.
- Placez la chambre d’écoulement microfluidique + cellules ajustée sur une boîte cultivée sur un microscope inversé, avec un objectif de microscope suffisamment élevé pour obtenir des images de haute qualité avec des valeurs de pixels élevées (généralement entre un grossissement de 40x et 63x ) et un moniteur d’affichage.
- Sélectionnez l’option d’image en direct (time-lapse sur certains logiciels) à partir du logiciel de microscope sur le moniteur d’affichage. Assurez-vous que le logiciel de microscope sur le PC a une fonctionnalité t (time-lapse) ou peut prendre des enregistrements vidéo.
- Concentrez l’objectif du microscope, en assurant un contraste adéquat et des bords cellulaires distincts. Ceci est nécessaire pour l’analyse de l’image post-cisaillement. Déplacez l’étage du microscope pour vous assurer que les cellules sont clairement visibles sur le moniteur d’affichage et sont des images en direct.
- Sélectionnez une cellule ou plusieurs cellules distinctes dans le trajet d’imagerie/d’écoulement de la chambre d’écoulement ajustée + boîte de Petri (la zone/le chemin créé par l’installation du joint à la chambre d’écoulement).
- Cisaillement et imagerie
- Pour maintenir un débit uniforme continu, choisir des taux de perfusion et de retrait similaires et assurer un débit laminaire du fluide, habituellement entre 1 mL/min et 5 mL/min. Pour les régimes à faible flux laminaire, assurer un nombre de Reynold de Re < 100.
- Cliquez sur Exécuter sur la pompe de cisaillement pour injecter et retirer le fluide de cisaillement (milieux visqueux préparés) à un débit continu. Assurez-vous qu’il n’y a pas de bulles pendant la perfusion de liquide, car cela pourrait introduire un stress externe non comptabilisé sur les cellules.
- Commencez à enregistrer une vidéo en cliquant sur Enregistrer sur le logiciel du microscope avant que le fluide de cisaillement infusé n’entre en contact avec la ou les cellules d’intérêt sous le microscope.
- Continuer à enregistrer pendant 7 minutes, ou pendant la durée souhaitée de l’exposition au stress, ou jusqu’à ce que la ou les cellules d’intérêt cisaillent le fond de la boîte. Cliquez sur Arrêter l’enregistrement sur le logiciel du microscope lorsque l’exécution est terminée comme vous le souhaitez.
- Enregistrez l’enregistrement et extrayez-le en tant que fichiers .tiff. De préférence, extrayez les images à 1 image par seconde pour faciliter l’analyse.
3. Traitement des données
- Procédure de corrélation d’images numériques (analyse d’images)
- Si l’enregistrement du microscope a été extrait sous forme de fichier vidéo, convertissez-le en images (de préférence .tiff format de fichier).
- Importez les images dérivées de l’enregistrement du test de cisaillement dans le logiciel Davis 10.1.2 (logiciel DIC) pour suivre le mouvement des structures naturellement structurées de la cellule en localisant chaque bloc de pixels (sous-ensemble) de l’image de référence dans les nouvelles images respectives (images déformées) (Figure 2).
- Pour une corrélation optimisée, utilisez une taille de sous-ensemble de 31 x 31 pixels, une taille de pas (distance de déformation de chaque sous-ensemble) de 20 pixels et la somme de l’option de piste différentielle , qui suit la déformation d’une nouvelle image par rapport à la dernière image. Le résultat de cette corrélation est un diagramme de temps de déformation (Figure 3) qui peut être exporté sous forme de fichier .csv pour une analyse plus approfondie dans MATLAB.
- Cartographiez la région d’intérêt pour une cellule choisie. Sélectionnez des points arbitraires dans la cellule mappée pour suivre la déformation. Pour une forme irrégulière, telle que la cellule, utilisez un masque polygonal pour cartographier la géométrie de la cellule.
- Après la cartographie, choisissez des points spécifiques de la cellule (noyau ou cytoplasme) à analyser en cliquant sur Ajouter une jauge de contrainte et en dessinant des jauges de contrainte individuelles à des points situés à l’intérieur de la limite cellulaire définie.
- Cliquez sur Exécuter pour commencer le traitement de la déformation et obtenir des données de déformation en fonction du temps.
- Double-cliquez (ou cliquez avec le bouton droit) sur le tracé de temps de contrainte généré et sélectionnez Exporter les données en tant que feuille de calcul.
4. Caractérisation des propriétés mécaniques
- Caractérisation des propriétés viscoélastiques
- Enregistrez le fichier .csv contenant les données de temps de contrainte du logiciel DIC dans un dossier séparé pour faciliter la lisibilité par MATLAB.
- Exécutez MATLAB et cliquez sur l’onglet éditeur pour ouvrir une page d’éditeur afin d’écrire un code qui lit la feuille de calcul, cellule par cellule.
- Modifiez le chemin MATLAB (le chemin du dossier qui accède au fichier qui vous intéresse) pour accéder au dossier contenant les données à analyser, par exemple, Utilisateurs/Nom d’utilisateur/Bureau/données.
- Sur la page de l’éditeur MATLAB, accédez aux données de la feuille de calcul à l’aide du code personnalisé. Par exemple : a1= xlsread('data','run1','A4:A183'), où a1 représente l’identificateur, xlsread est la fonction MATLAB qui lit le fichier .csv (dans ce cas, sous forme de feuille de calcul), data est le nom du fichier, run1 est le nom de la feuille et A4:A183 est la plage de données d’intérêt dans la cellule A des données de feuille de calcul à analyser. Pour un ajustement complet, analysez x et y (temps et déformation, respectivement). Par exemple:
a1=xlsread('data','run1','A4:A183');
b1=xlsread('données','run1','B4:B183');
a1 = x (temps) et b1 = y (déformation). - Dans MATLAB, cliquez sur Applications | Installateur de courbe | Équation personnalisée. Effacez l’équation personnalisée représentative et entrez l’équation du modèle viscoélastique [équation (1)], où ε représente la variable x et t représente la variable y.
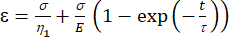 (1)
(1)
Ici, ε représente la déformation, σ représente la contrainte de cisaillement, η1 représente la viscosité, E représente l’élasticité, t représente le temps et τ représente le temps de relaxation, qui caractérise le temps maximum nécessaire pour que la cellule retrouve sa forme initiale après une déformation primaire. Il est exprimé par τ =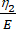 , où η2 est le terme de viscosité secondaire pour le deuxième tableau de bord (Figure 4).
, où η2 est le terme de viscosité secondaire pour le deuxième tableau de bord (Figure 4). - Réaffectez de nouvelles variables aux paramètres viscoélastiques dans l’interface d’équation personnalisée. (ε, η1, E, σ, t et τ =
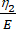 ) représenteront (x, a, b, K, y et c), respectivement. Ici, x et y sont respectivement les variables indépendantes et dépendantes. La contrainte de cisaillement (σ) peut être déterminée à l’aide de l’équation (2):
) représenteront (x, a, b, K, y et c), respectivement. Ici, x et y sont respectivement les variables indépendantes et dépendantes. La contrainte de cisaillement (σ) peut être déterminée à l’aide de l’équation (2):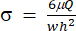 (2)
(2)
Ici, μ représente la viscosité du fluide de cisaillement, Q est le débit de la pompe et w et h sont la largeur et la hauteur du canal d’écoulement montrées à la figure 1C, respectivement. - Cliquez sur Sélectionner les données pour sélectionner l’heure (a1) et la souche (b1) pour chaque ensemble de données.
- Assurez-vous que l’option Ajustement automatique est cochée. Cela exécute l’ajustement automatiquement lorsque les données (x et y) sont sélectionnées.
- Sélectionnez les méthodes d’ajustement pour resserrer les conditions aux limites. Cliquez sur Options avancées dans la catégorie Méthodes et sélectionnez Moindres carrés non linéaires. Sous Robuste, sélectionnez Désactivé, puis sous Algorithme, sélectionnez Trust-Region. Laissez tous les autres paramètres tels quels.
- Les nouvelles variables post-ajustement (a, b et c) représentent respectivement les propriétés viscoélastiques, la viscosité, l’élasticité et le temps de relaxation (η1, E, EQUAT) de la cellule (Figure 5).
- Recherchez une valeur R-carré élevée de l’ajustement (R2 > 80%) pour vous assurer que la sortie de données peut être considérée comme un véritable ajustement du modèle viscoélastique (Figure 3)
Résultats
Le protocole d’essai de cisaillement associé à une analyse de déformation à l’aide de CIVD et d’un modèle viscoélastique permet de quantifier les propriétés mécaniques d’une seule cellule in vitro. Cette méthode a été testée sur des lignées cellulaires humaines et murines, y compris des cellules mammaires humaines normales (MCF-10A)3,4,9, des cellules de cancer du sein triple négatif moins métastatiques (MDA-MB-468)3, des cellules cancéreuses du sein triple négat...
Discussion
La méthode de dosage par cisaillement, qui comprend la mise en place d’un environnement pseudo-mécanobiologique pour simuler l’interaction des cellules avec le microenvironnement mécanique environnant et leurs réponses aux contraintes mécaniques, a produit un catalogue de propriétés mécaniques cellulaires, dont les modèles montrent des atypies physiques conservées parmi les lignées cellulaires cancéreuses 3,4,5,7,8
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents à divulguer.
Remerciements
Les auteurs remercient les chercheurs précédents du groupe Soboyejo du Worcester Polytechnic Institute qui ont été les premiers à mettre au point cette technique: les Drs Yifang Cao, Jingjie Hu et Vanessa Uzonwanne. Ce travail a été soutenu par le National Cancer Institute (NIH / NCI K22 CA258410 to M.D.). Les figurines ont été créées avec BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| CELL CULTURE | |||
| .25% Trypsin, 2.21 mM EDTA, 1x[-] sodium bicarbonate | Corning | 25-053-ci | For cellular detachment from substrate in cell culture |
| 15 mL centrifuge tubes | Falcon by Corning | 05-527-90 | |
| 35 mm Petri dishes | Corning | 430165 | |
| 50 mL centrifuge tubes | Falcon by Corning | 14-432-22 | |
| centrifuge | any | For sterile cell culture | |
| Dulbecco's Modification of Eagle's Medium (DMEM) 1x | Corning | 10-013-cv | Or any other media for culturing cells. DMEM was used for culturing U87 cells |
| gloves | any | For sterile cell culture | |
| Heracell Vios 160i CO2 Incubator | Thermo Scientific | 51033770 | For Incubation during cell culture |
| Hood | any | For sterile cell culture | |
| micropipette | any | For sterile cell culture | |
| micropipette tips | any | For sterile cell culture | |
| Microscope | Leica/any | For sterile cell culture | |
| Phosphate Buffered Saline without calcium and magnesium PBS, 1x | Corning | 21-040-CM | |
| pipetman | any | For sterile cell culture | |
| pipette tips | any | For sterile cell culture | |
| Precision GP 10 liquid incubator | Thermo Scientific | TSGP02 | |
| T25 flask | Corning | 430639 | |
| T75 flask | Corning | 430641U | |
| SHEAR ASSAY | |||
| 100 mL beaker | any | For creating DMEM + methyl cellulose viscous shear media | |
| DMEM | Corning | ||
| Flow chamber + rubber gasket | Glycotech | 31-001 | Circular Flow chamber Kit ( for 35 mm tissue culture dishes) |
| Hybrid Rheometer | HR-2 Discovery Hybrid Rheometer | For determination of shear fluid viscosity | |
| magnetic stir bar | any | For creating DMEM + methyl cellulose viscous shear media | |
| magnetic stir plate | any | For creating DMEM + methyl cellulose viscous shear media | |
| methyl cellulose | any | To increase viscosity of DMEM in flow media | |
| Syringe Pump | KD Scientific Geminin 88 plus | 788088 | For programming fluid infusion and withdrawal |
| syringes, tubing, and connectors | For shear apparatus setup | ||
| SOFTWARE | |||
| ABAQUS software | Simulia | ||
| Digitial Image Correlation software | LaVision, Germany | DAVIS 10.1.2 | |
| Imaging software | Leica/any microscope software | ||
| MATLAB | MATLAB | MATLAB_R2020B |
Références
- Sethi, S., Ali, S., Philip, P. A., Sarkar, F. H. Clinical advances in molecular biomarkers for cancer diagnosis and therapy. International Journal of Molecular Sciences. 14 (7), 14771-14784 (2013).
- Runel, G., Lopez-Ramirez, N., Chlasta, J., Masse, I. Biomechanical properties of cancer cells. Cells. 10 (4), 887 (2021).
- Hu, J., Zhou, Y., Obayemi, J. D., Du, J., Soboyejo, W. O. An investigation of the viscoelastic properties and the actin cytoskeletal structure of triple negative breast cancer cells. Journal of the Mechanical Behavior of Biomedical Materials. 86, 1-13 (2018).
- Onwudiwe, K., et al. Investigation of creep properties and the cytoskeletal structures of non-tumorigenic breast cells and triple-negative breast cancer cells. Journal of Biomedical Materials Research. Part A. 110 (5), 1004-1020 (2022).
- Ani, C. J., et al. A shear assay study of single normal/breast cancer cell deformation and detachment from poly-di-methyl-siloxane (PDMS) surfaces. Journal of the Mechanical Behavior of Biomedical Materials. 91, 76-90 (2019).
- Suresh, S. Biomechanics and biophysics of cancer cells. Acta Biomaterialia. 3 (4), 413-438 (2007).
- Cao, Y., et al. Investigation of the viscoelasticity of human osteosarcoma cells using a shear assay method. Journal of Materials Research. 21 (8), 1922-1930 (2006).
- Cao, Y. On the measurement of human osteosarcoma cell elastic modulus using shear assay experiments. Journal of Materials Science. Materials in Medicine. 18 (1), 103-109 (2007).
- Onwudiwe, K., et al. Actin cytoskeletal structure and the statistical variations of the mechanical properties of non-tumorigenic breast and triple-negative breast cancer cells. Journal of the Mechanical Behavior of Biomedical Materials. 119, 104505 (2021).
- Kirmizis, D., Logothetidis, S. Atomic force microscopy probing in the measurement of cell mechanics. International Journal of Nanomedicine. 5, 137-145 (2010).
- Haase, K., Pelling, A. E. Investigating cell mechanics with atomic force microscopy. Journal of the Royal Society. Interface. 12 (104), 20140970 (2015).
- Zhang, H., Liu, K. K. Optical tweezers for single cells. Journal of the Royal Society. Interface. 5 (24), 671-690 (2008).
- Peterman, E. J. G., Gittes, F., Schmidt, C. F. Laser-induced heating in optical traps. Biophysical Journal. 84, 1308-1316 (2003).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33 (1), 15-22 (2000).
- Evans, E., Yeung, A. Apparent viscosity and corticcal tension of blood granulocytes determined by micropipet aspiration. Biophysical Journal. 56 (1), 151-160 (1989).
- Van Vliet, K. J., Bao, G., Suresh, S. The biomechanics toolbox: experimental approaches for living cells and biomolecules. Acta Materialia. 51 (19), 5881-5905 (2003).
- Moeendarbary, E., Harris, A. R. Cell mechanics: principles, practices, and prospects. Wiley Interdisciplinary Reviews. Systems Biology and Medicine. 6 (5), 371-388 (2014).
- Choi, H. Y., et al. Hydrodynamic shear stress promotes epithelial-mesenchymal transition by downregulating ERK and GSK3beta activities. Breast Cancer Research. 21 (1), 6 (2019).
- Northcott, J. M., Dean, I. S., Mouw, J. K., Weaver, V. M. Feeling stress: The mechanics of cancer progression and aggression. Frontiers in Cell and Developmental Biology. 6, 17 (2018).
- Onwudiwe, K., Najera, J., Siri, S., Datta, M. Do tumor mechanical stresses promote cancer immune escape. Cells. 11 (23), 3840 (2022).
- Heldin, C. H., Rubin, K., Pietras, K., Ostman, A. High interstitial fluid pressure - an obstacle in cancer therapy. Nature Reviews. Cancer. 4 (10), 806-813 (2004).
- Krog, B. L., Henry, M. D. Biomechanics of the circulating tumor cell microenvironment. Advances in Experimental Medicine and Biology. 1092, 209-233 (2018).
- Moose, D. L., et al. Cancer cells resist mechanical destruction in circulation via RhoA/actomyosin-dependent mechano-adaptation. Cell Reports. 30 (11), 3864-3874 (2020).
- Mao, B. H., Nguyen Thi, K. M., Tang, M. J., Kamm, R. D., Tu, T. Y. The interface stiffness and topographic feature dictate interfacial invasiveness of cancer spheroids. Biofabrication. 15 (1), (2023).
- Kashani, A. S., Packirisamy, M. Cancer cells optimize elasticity for efficient migration. Royal Society Open Science. 7 (10), 200747 (2020).
- Riehl, B. D., Kim, E., Bouzid, T., Lim, J. Y. The role of microenvironmental cues and mechanical loading milieus in breast cancer cell progression and metastasis. Frontiers in Bioengineering and Biotechnology. 8, 608526 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon