È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Protocollo di saggio di taglio per la determinazione delle proprietà dei materiali a singola cellula
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo delinea la quantificazione delle proprietà meccaniche di linee cellulari cancerose e non cancerose in vitro. Le differenze conservate nella meccanica delle cellule cancerose e normali possono agire come un biomarcatore che può avere implicazioni nella prognosi e nella diagnosi.
Abstract
La biomeccanica irregolare è un segno distintivo della biologia del cancro soggetta a studi approfonditi. Le proprietà meccaniche di una cella sono simili a quelle di un materiale. La resistenza di una cellula allo stress e alla tensione, il suo tempo di rilassamento e la sua elasticità sono tutte proprietà che possono essere derivate e confrontate con altri tipi di cellule. Quantificare le proprietà meccaniche delle cellule cancerose (maligne) rispetto alle cellule normali (non maligne) consente ai ricercatori di scoprire ulteriormente i fondamenti biofisici di questa malattia. Mentre le proprietà meccaniche delle cellule tumorali sono note per differire costantemente dalle proprietà meccaniche delle cellule normali, manca una procedura sperimentale standard per dedurre queste proprietà dalle cellule in coltura.
Questo articolo delinea una procedura per quantificare le proprietà meccaniche di singole cellule in vitro utilizzando un saggio a taglio fluido. Il principio alla base di questo test comporta l'applicazione dello sforzo di taglio del fluido su una singola cellula e il monitoraggio ottico della deformazione cellulare risultante nel tempo. Le proprietà meccaniche delle celle vengono successivamente caratterizzate utilizzando l'analisi di correlazione delle immagini digitali (DIC) e adattando un modello viscoelastico appropriato ai dati sperimentali generati dall'analisi DIC. Nel complesso, il protocollo qui delineato mira a fornire un metodo più efficace e mirato per la diagnosi di tumori difficili da trattare.
Introduzione
Lo studio delle differenze biofisiche tra cellule cancerose e non cancerose consente nuove opportunità diagnostiche e terapeutiche1. Comprendere come le differenze nella biomeccanica / meccanobiologia contribuiscono alla progressione del tumore e alla resistenza al trattamento rivelerà nuove strade per la terapia mirata e la diagnosi precoce2.
Mentre è noto che le proprietà meccaniche delle cellule tumorali differiscono dalle cellule normali (ad esempio, viscoelasticità della membrana plasmatica e dell'involucro nucleare)3,4,5, mancano metodi robusti e riproducibili per misurare queste proprietà nelle cellule vive6. Il metodo del saggio di taglio viene utilizzato per quantificare le proprietà meccaniche delle cellule sottoponendo singole cellule a stress di taglio del fluido e analizzando le loro risposte individuali e la resistenza allo stress applicato 3,4,5,7,8,9. Sebbene siano stati utilizzati diversi metodi e tecniche per caratterizzare le proprietà meccaniche delle singole cellule, questi tendono a influenzare le proprietà del materiale cellulare i) perforando / danneggiando la membrana cellulare a causa della profondità di indentazione, geometrie complesse della punta o irrigidimento del substrato associato alla microscopia a forza atomica (AFM) 10,11, ii) inducendo fotodanno cellulare durante l'intrappolamento ottico 12, 13, o iii) inducendo stati di sollecitazione complessi associati all'aspirazione di micropipette14,15. Questi effetti esterni sono associati a significative incertezze nell'accuratezza delle misurazioni della viscoelasticità cellulare 6,16,17.
Per affrontare queste limitazioni, il metodo di analisi del taglio qui descritto fornisce un approccio altamente controllabile e semplice per simulare il flusso fisiologico nel corpo senza influire sulle proprietà del materiale cellulare nel processo. Le sollecitazioni di taglio dei fluidi in questo test rappresentano le sollecitazioni meccaniche sperimentate dalle cellule del corpo dai fluidi all'interno dell'interstizio tumorale o nel sangue durante la circolazione18,19,20. Inoltre, questi stress fluidi promuovono vari comportamenti maligni nelle cellule tumorali, tra cui progressione, migrazione, metastasi e morte cellulare 19,21,22,23 che variano tra cellule tumorigeniche e non tumorigeniche. Inoltre, le caratteristiche meccaniche alterate delle cellule tumorali (cioè, sono spesso "più morbide" delle cellule normali che si trovano all'interno dello stesso organo) consentono loro di persistere in microambienti tumorali ostili, invadere i tessuti normali circostanti e metastatizzare in siti distanti24,25,26. Creando un ambiente pseudo-biologico in cui le cellule sperimentano livelli fisiologici di stress da taglio dei fluidi, si ottiene un processo fisiologicamente rilevante e non distruttivo per la cellula. Le risposte cellulari a queste sollecitazioni di taglio dei fluidi applicate ci permettono di caratterizzare le proprietà meccaniche delle cellule.
Questo documento fornisce un protocollo di saggio di taglio per lo studio approfondito delle proprietà meccaniche e del comportamento delle cellule cancerose e non cancerose sotto sforzo di taglio applicato. Le cellule rispondono alle forze esterne in modo elastico e viscoso e possono quindi essere idealizzate come materiale viscoelastico3. Questa tecnica è classificata in: (i) coltura cellulare di singole cellule disperse, (ii) applicazione controllata dello stress di taglio del fluido, (iii) imaging in situ e osservazione del comportamento cellulare (compresa la resistenza allo stress e alla deformazione), (iv) analisi della deformazione delle cellule per determinare l'entità della deformazione e (v) caratterizzazione delle proprietà viscoelastiche delle singole cellule. Interrogando queste proprietà e comportamenti meccanici, la meccanobiologia cellulare complessa può essere distillata in dati quantificabili. Un protocollo che delinea questo metodo consente la catalogazione e il confronto tra vari tipi di cellule maligne e non maligne. Quantificare queste differenze ha il potenziale per stabilire biomarcatori diagnostici e terapeutici.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Preparazione per il saggio di taglio a cellula singola
- Coltura cellulare
- Seminare circa 50.000 cellule singole sospese in una capsula di Petri di 35 mm x 10 mm contenente 2 ml di terreno di coltura.
NOTA: Vortice le celle sospese prima della semina per rompere gli aggregati cellulari. - Incubare le cellule a 37 °C e consentire tra 10 e 48 ore per l'attaccamento cellulare e la formazione completa della proteina citoscheletrica.
NOTA: Considerare la durata dell'attaccamento cellulare, così come i tassi di proliferazione e crescita, per garantire un'adeguata crescita cellulare e attaccamento evitando l'aggregazione cellulare. Questi parametri variano a seconda del tipo di cella.
- Seminare circa 50.000 cellule singole sospese in una capsula di Petri di 35 mm x 10 mm contenente 2 ml di terreno di coltura.
2. Esperimento di saggio di taglio
- Preparazione del saggio di taglio mezzi a flusso viscoso
- Per garantire un flusso leggermente viscoso (0,015-0,02 Pa·s), misurare e aggiungere lo 0,05% in peso di metilcellulosa non tossica e anallergica (4 Pa·s) ai terreni di coltura.
- Per garantire una miscela omogenea, preriscaldare il terreno di coltura di base per ~10-20 minuti ad una temperatura di ~60-70 °C con un agitatore magnetico/piastra calda. Mescolando continuamente il mezzo, aggiungere delicatamente la metilcellulosa in modo che si disperda rapidamente, per evitare la coagulazione delle particelle di metilcellulosa. Lasciare che questo processo continui per ~ 15-24 ore per garantire una soluzione chiara di media + cellulosa.
NOTA: Evitare un eccessivo riscaldamento della soluzione. - Per misurare la viscosità del fluido di flusso, testare ~ 0,5-1 mL del mezzo di flusso rappresentativo utilizzando un reometro. Dalla lettura, determinare la viscosità del fluido e utilizzare questo valore per rappresentare la viscosità del mezzo fluido di taglio (μ) per calcolare lo sforzo di taglio utilizzando l'equazione (2).
- Configurazione dell'apparato di taglio
- Impostare il sistema di dosaggio a taglio di siringhe doppie (60 ml o 100 ml) collegate a una pompa a siringa programmabile per l'infusione e il prelievo dei terreni di coltura viscosi (Figura 1).
- Collegare entrambe le siringhe alla camera di flusso tramite tubi da 1/16 di pollice e connettori per tubi.
- Fissare una guarnizione in gomma per fornire un flusso controllato e uniforme su singole celle lungo il percorso del flusso (Figura 1). La guarnizione in gomma è disponibile in diverse dimensioni a seconda del profilo di flusso da raggiungere (laminare o turbolento) e dell'area di osservazione desiderata (ad esempio, lunghezza di 22,5 mm, larghezza di 2,5 mm e altezza di 0,254 mm) (Figura 1).
- Programmare la pompa per infondere e prelevare un certo volume di fluido (ad esempio, 60 ml) ad una velocità designata (ad esempio, 1 ml / min) e selezionare le siringhe corrispondenti (ad esempio, 60 ml).
NOTA: Tenere conto dei preset del volume massimo di infusione e prelievo per evitare inceppamenti o malfunzionamenti. Utilizzare l'equazione (2) per il calcolo della velocità di taglio della pompa richiesta (supponendo che le sollecitazioni e la viscosità richieste siano note).
- Configurazione pre-taglio
- Riempire la siringa con il mezzo di flusso viscoso preparato.
- Collegare la siringa, riempita con 60 o 100 ml (o secondo necessità) di fluido di flusso viscoso, e una siringa vuota da 60 mL nelle rispettive posizioni sulla pompa a siringa programmabile. Tramite tubi e connettori per tubi, collegare entrambe le siringhe alla camera di flusso.
- Per garantire una facile identificazione delle singole celle e una connessione fissata tra la camera di flusso e la capsula di Petri, collegare la guarnizione in gomma alla camera di flusso.
- Aspirare il terreno di coltura cellulare dalla capsula di Petri contenente le cellule di interesse.
- Lavare via le cellule morte e le cellule attaccate liberamente usando soluzione salina tamponata fosfato (PBS).
- Aspirare il PBS.
- Inserire e fissare la camera di flusso e la guarnizione in gomma (~34 mm x 9 mm) sulla capsula di Petri (35 mm x 10 mm) contenente le celle collegate.
- Posizionare la camera di flusso microfluidica + le cellule montate su un piatto coltivato su un microscopio invertito, con un obiettivo del microscopio abbastanza alto da ottenere immagini di alta qualità con valori di pixel elevati (di solito tra 40x e 63x ingrandimento) e un monitor del display.
- Selezionare l'opzione immagine live (time-lapse su alcuni software) dal software del microscopio sul monitor del display. Assicurarsi che il software del microscopio sul PC abbia una funzionalità t (time-lapse) o che possa effettuare registrazioni video.
- Focalizzare l'obiettivo del microscopio, garantendo un contrasto adeguato e bordi cellulari distinti. Questo è necessario per l'analisi dell'immagine post-taglio. Spostare lo stadio del microscopio per assicurarsi che le cellule siano chiaramente visibili sul monitor del display e siano immagini dal vivo.
- Selezionare una cella o più celle distinte all'interno del percorso di imaging/flusso della camera di flusso montata + piastra di Petri (l'area/percorso creato dal montaggio della guarnizione alla camera di flusso).
- Cesoia e imaging
- Per mantenere un flusso uniforme continuo, selezionare velocità di infusione e prelievo simili e garantire un flusso laminare del fluido, di solito tra 1 ml / min e 5 ml / min. Per regimi a basso flusso laminare, assicurarsi che un numero di Re di Reynold < 100.
- Fare clic su Esegui sulla pompa di taglio per iniettare e prelevare il fluido di taglio (mezzi viscosi preparati) a una velocità continua. Assicurarsi che non ci siano bolle durante l'infusione di liquidi, poiché ciò potrebbe introdurre stress esterno non giustificato sulle cellule.
- Iniziare a registrare un video facendo clic su Registra sul software del microscopio prima che il fluido di taglio infuso entri in contatto con le cellule di interesse al microscopio.
- Continuare a registrare per 7 minuti, o per la durata desiderata dell'esposizione allo stress, o fino a quando la cella o le celle di interesse tagliano il fondo del piatto. Fare clic su Interrompi registrazione sul software del microscopio quando l'esecuzione è completata come desiderato.
- Salva la registrazione ed estrai come file .tiff. Preferibilmente, estrai le immagini a 1 fotogramma al secondo per una facile analisi.
3. Trattamento dei dati
- Procedura di correlazione digitale delle immagini (analisi delle immagini)
- Se la registrazione dal microscopio è stata estratta come file video, convertirlo in fotogrammi immagine (preferibilmente .tiff formato di file).
- Importare le immagini derivate dalla registrazione del saggio di taglio nel software Davis 10.1.2 (software DIC) per tracciare il movimento delle strutture modellate naturalmente della cella localizzando ciascun blocco di pixel (sottoinsieme) dell'immagine di riferimento nelle rispettive nuove immagini (immagini deformate) (Figura 2).
- Per una correlazione ottimizzata, utilizzare una dimensione del sottoinsieme di 31 x 31 pixel, una dimensione del passo (distanza di deformazione di ciascun sottoinsieme) di 20 pixel e la somma dell'opzione traccia differenziale , che tiene traccia della deformazione di una nuova immagine rispetto all'ultima immagine. Il risultato di questa correlazione è un grafico strain-time (Figura 3) che può essere esportato come file .csv per ulteriori analisi in MATLAB.
- Mappa la regione di interesse per una singola cella scelta. Selezionare punti arbitrari all'interno della cella mappata per tenere traccia della deformazione. Per una forma irregolare, ad esempio la cella, usate una maschera poligonale per mappare la geometria della cella.
- Dopo la mappatura, scegliere punti specifici sulla cellula (nucleo o citoplasma) da analizzare facendo clic su aggiungi estensimetro e disegnando singoli estensimetri in punti all'interno del confine cellulare definito.
- Fare clic su Esegui per iniziare l'elaborazione della deformazione e ottenere i dati di deformazione rispetto al tempo.
- Fare doppio clic (o fare clic con il pulsante destro del mouse) sul grafico del tempo di deformazione generato e selezionare Esporta dati come foglio di calcolo.
4. Caratterizzazione delle proprietà meccaniche
- Caratterizzazione delle proprietà viscoelastiche
- Salvare il file .csv contenente i dati del tempo di deformazione dal software DIC in una cartella separata per una facile leggibilità da parte di MATLAB.
- Esegui MATLAB e fai clic sulla scheda editor per aprire una pagina dell'editor per scrivere un codice che legge il foglio di calcolo, cella per cella.
- Modificare il percorso MATLAB (il percorso della cartella che accede al file di interesse) per accedere alla cartella contenente i dati da analizzare, ad esempio Utenti/Username/Desktop/data.
- Nella pagina dell'editor MATLAB, accedi ai dati del foglio di calcolo utilizzando il codice personalizzato. Ad esempio: a1= xlsread('data','run1','A4:A183'), dove a1 rappresenta l'identificatore, xlsread è la funzione MATLAB che legge il file .csv (in questo caso, come foglio di calcolo), data è il nome del file, run1 è il nome del foglio e A4:A183 è l'intervallo di dati di interesse nella cella A dei dati del foglio di calcolo da analizzare. Per un adattamento completo, analizza x e y (tempo e sforzo, rispettivamente). Per esempio:
a1=xlsread('data','run1','A4:A183');
b1=xlsread('data','run1','B4:B183');
a1 = x (tempo) e b1 = y (deformazione). - In MATLAB, fai clic su App | Curve Fitter Equazione personalizzata. Deselezionate l'equazione personalizzata rappresentativa e immettete l'equazione del modello viscoelastico [equazione (1)], dove ε rappresenta la variabile x e t rappresenta la variabile y.
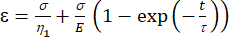 (1)
(1)
Qui, ε rappresenta la deformazione, σ rappresenta lo sforzo di taglio, η1 rappresenta la viscosità, E rappresenta l'elasticità, t rappresenta il tempo e τ rappresenta il tempo di rilassamento, che caratterizza il tempo massimo richiesto alla cella per tornare alla sua forma originale dopo la deformazione primaria. È espresso come τ =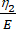 , dove η2 è il termine di viscosità secondario per il secondo dashpot (Figura 4).
, dove η2 è il termine di viscosità secondario per il secondo dashpot (Figura 4). - Riassegnare nuove variabili ai parametri viscoelastici all'interno dell'interfaccia dell'equazione personalizzata. (ε, η1, E, σ, t e τ =
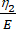 ) rappresenterà (x, a, b, K, y e c), rispettivamente. Qui, x e y sono rispettivamente le variabili indipendenti e dipendenti. Lo sforzo di taglio (σ) può essere determinato usando l'equazione (2):
) rappresenterà (x, a, b, K, y e c), rispettivamente. Qui, x e y sono rispettivamente le variabili indipendenti e dipendenti. Lo sforzo di taglio (σ) può essere determinato usando l'equazione (2):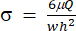 (2)
(2)
Qui, μ rappresenta la viscosità del fluido di taglio, Q è la portata della pompa e w e h sono rispettivamente la larghezza e l'altezza del canale di flusso mostrate nella Figura 1C. - Fare clic su Seleziona dati per selezionare il tempo (a1) e la deformazione (b1) per ciascun set di dati.
- Assicurarsi che l'opzione Auto Fit box sia selezionata. In questo modo viene eseguito automaticamente l'adattamento quando vengono selezionati i dati (x e y).
- Selezionare i metodi di adattamento per stringere le condizioni al contorno. Fare clic su Opzioni avanzate nella categoria Metodi e selezionare Minimi quadrati non lineari. In Robusto selezionare Disattivato e in Algoritmo selezionare Area trust. Lascia tutti gli altri parametri così come sono.
- Le nuove variabili post-fitting (a, b e c) rappresentano rispettivamente le proprietà viscoelastiche, la viscosità, l'elasticità e il tempo di rilassamento (η1, E, EQUAT) della cella (Figura 5).
- Cercare un valore R-quadrato elevato dell'adattamento (R2 > 80%) per garantire che l'output dei dati possa essere considerato un vero adattamento del modello viscoelastico (Figura 3)
Access restricted. Please log in or start a trial to view this content.
Risultati
Il protocollo di saggio a taglio accoppiato con l'analisi della deformazione utilizzando DIC e un modello viscoelastico è riuscito a quantificare le proprietà meccaniche di una singola cella in vitro. Questo metodo è stato testato su linee cellulari umane e murine, tra cui cellule mammarie umane normali (MCF-10A)3,4,9, cellule di carcinoma mammario triplo negativo meno metastatiche (MDA-MB-468)3, cellule di carcinoma mammario triplo negativo (MDA-MB-231)3, cellule di osteosarcoma umano...
Access restricted. Please log in or start a trial to view this content.
Discussione
Il metodo di saggio di taglio, che include la creazione di un ambiente pseudo-meccanobiologico per simulare l'interazione delle cellule con il microambiente meccanico circostante e le loro risposte alle sollecitazioni meccaniche, ha prodotto un catalogo di proprietà meccaniche cellulari, i cui modelli mostrano atipie fisiche conservate tra le linee cellulari cancerose 3,4,5,7,8
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti da rivelare.
Riconoscimenti
Gli autori ringraziano i precedenti ricercatori del gruppo Soboyejo del Worcester Polytechnic Institute che per primi hanno aperto la strada a questa tecnica: i dottori Yifang Cao, Jingjie Hu e Vanessa Uzonwanne. Questo lavoro è stato sostenuto dal National Cancer Institute (NIH / NCI K22 CA258410 a M.D.). Le figure sono state create con BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| CELL CULTURE | |||
| .25% Trypsin, 2.21 mM EDTA, 1x[-] sodium bicarbonate | Corning | 25-053-ci | For cellular detachment from substrate in cell culture |
| 15 mL Centrifuge tubes | Falcon by Corning | 05-527-90 | |
| 35 mm Petri dishes | Corning | 430165 | |
| 50 mL Centrifuge tubes | Falcon by Corning | 14-432-22 | |
| Centrifuge | any | For sterile cell culture | |
| Dulbecco's Modification of Eagle's Medium (DMEM) 1x | Corning | 10-013-cv | Or any other media for culturing cells. DMEM was used for culturing U87 cells |
| Gloves | any | For sterile cell culture | |
| Heracell Vios 160i CO2 Incubator | Thermo Scientific | 51033770 | For Incubation during cell culture |
| Hood | any | For sterile cell culture | |
| Micropipette | any | For sterile cell culture | |
| Micropipette tips | any | For sterile cell culture | |
| Microscope | Leica/any | For sterile cell culture | |
| Phosphate Buffered Saline without calcium and magnesium PBS, 1x | Corning | 21-040-CM | |
| Pipetman | any | For sterile cell culture | |
| Pipette tips | any | For sterile cell culture | |
| Precision GP 10 liquid incubator | Thermo Scientific | TSGP02 | |
| T25 Flask | Corning | 430639 | |
| T75 Flask | Corning | 430641U | |
| SHEAR ASSAY | |||
| 100 mL beaker | any | For creating DMEM + methyl cellulose viscous shear media | |
| DMEM | Corning | ||
| Flow chamber + rubber gasket | Glycotech | 31-001 | Circular Flow chamber Kit ( for 35 mm tissue culture dishes) |
| Hybrid Rheometer | HR-2 Discovery Hybrid Rheometer | For determination of shear fluid viscosity | |
| Magnetic stir bar | any | For creating DMEM + methyl cellulose viscous shear media | |
| Magnetic stir plate | any | For creating DMEM + methyl cellulose viscous shear media | |
| Methyl cellulose | any | To increase viscosity of DMEM in flow media | |
| Syringe Pump | KD Scientific Geminin 88 plus | 788088 | For programming fluid infusion and withdrawal |
| Syringes, tubing, and connectors | For shear apparatus setup | ||
| SOFTWARE | |||
| ABAQUS software | Simulia | ||
| Digitial Image Correlation software | LaVision, Germany | DAVIS 10.1.2 | |
| Imaging software | Leica/any microscope software | ||
| MATLAB | MATLAB | MATLAB_R2020B |
Riferimenti
- Sethi, S., Ali, S., Philip, P. A., Sarkar, F. H. Clinical advances in molecular biomarkers for cancer diagnosis and therapy. International Journal of Molecular Sciences. 14 (7), 14771-14784 (2013).
- Runel, G., Lopez-Ramirez, N., Chlasta, J., Masse, I. Biomechanical properties of cancer cells. Cells. 10 (4), 887(2021).
- Hu, J., Zhou, Y., Obayemi, J. D., Du, J., Soboyejo, W. O. An investigation of the viscoelastic properties and the actin cytoskeletal structure of triple negative breast cancer cells. Journal of the Mechanical Behavior of Biomedical Materials. 86, 1-13 (2018).
- Onwudiwe, K., et al. Investigation of creep properties and the cytoskeletal structures of non-tumorigenic breast cells and triple-negative breast cancer cells. Journal of Biomedical Materials Research. Part A. 110 (5), 1004-1020 (2022).
- Ani, C. J., et al. A shear assay study of single normal/breast cancer cell deformation and detachment from poly-di-methyl-siloxane (PDMS) surfaces. Journal of the Mechanical Behavior of Biomedical Materials. 91, 76-90 (2019).
- Suresh, S. Biomechanics and biophysics of cancer cells. Acta Biomaterialia. 3 (4), 413-438 (2007).
- Cao, Y., et al. Investigation of the viscoelasticity of human osteosarcoma cells using a shear assay method. Journal of Materials Research. 21 (8), 1922-1930 (2006).
- Cao, Y. On the measurement of human osteosarcoma cell elastic modulus using shear assay experiments. Journal of Materials Science. Materials in Medicine. 18 (1), 103-109 (2007).
- Onwudiwe, K., et al. Actin cytoskeletal structure and the statistical variations of the mechanical properties of non-tumorigenic breast and triple-negative breast cancer cells. Journal of the Mechanical Behavior of Biomedical Materials. 119, 104505(2021).
- Kirmizis, D., Logothetidis, S. Atomic force microscopy probing in the measurement of cell mechanics. International Journal of Nanomedicine. 5, 137-145 (2010).
- Haase, K., Pelling, A. E. Investigating cell mechanics with atomic force microscopy. Journal of the Royal Society. Interface. 12 (104), 20140970(2015).
- Zhang, H., Liu, K. K. Optical tweezers for single cells. Journal of the Royal Society. Interface. 5 (24), 671-690 (2008).
- Peterman, E. J. G., Gittes, F., Schmidt, C. F. Laser-induced heating in optical traps. Biophysical Journal. 84, 1308-1316 (2003).
- Hochmuth, R. M. Micropipette aspiration of living cells. Journal of Biomechanics. 33 (1), 15-22 (2000).
- Evans, E., Yeung, A. Apparent viscosity and corticcal tension of blood granulocytes determined by micropipet aspiration. Biophysical Journal. 56 (1), 151-160 (1989).
- Van Vliet, K. J., Bao, G., Suresh, S. The biomechanics toolbox: experimental approaches for living cells and biomolecules. Acta Materialia. 51 (19), 5881-5905 (2003).
- Moeendarbary, E., Harris, A. R. Cell mechanics: principles, practices, and prospects. Wiley Interdisciplinary Reviews. Systems Biology and Medicine. 6 (5), 371-388 (2014).
- Choi, H. Y., et al. Hydrodynamic shear stress promotes epithelial-mesenchymal transition by downregulating ERK and GSK3beta activities. Breast Cancer Research. 21 (1), 6(2019).
- Northcott, J. M., Dean, I. S., Mouw, J. K., Weaver, V. M. Feeling stress: The mechanics of cancer progression and aggression. Frontiers in Cell and Developmental Biology. 6, 17(2018).
- Onwudiwe, K., Najera, J., Siri, S., Datta, M. Do tumor mechanical stresses promote cancer immune escape. Cells. 11 (23), 3840(2022).
- Heldin, C. H., Rubin, K., Pietras, K., Ostman, A. High interstitial fluid pressure - an obstacle in cancer therapy. Nature Reviews. Cancer. 4 (10), 806-813 (2004).
- Krog, B. L., Henry, M. D. Biomechanics of the circulating tumor cell microenvironment. Advances in Experimental Medicine and Biology. 1092, 209-233 (2018).
- Moose, D. L., et al. Cancer cells resist mechanical destruction in circulation via RhoA/actomyosin-dependent mechano-adaptation. Cell Reports. 30 (11), 3864-3874 (2020).
- Mao, B. H., Nguyen Thi, K. M., Tang, M. J., Kamm, R. D., Tu, T. Y. The interface stiffness and topographic feature dictate interfacial invasiveness of cancer spheroids. Biofabrication. 15 (1), (2023).
- Kashani, A. S., Packirisamy, M. Cancer cells optimize elasticity for efficient migration. Royal Society Open Science. 7 (10), 200747(2020).
- Riehl, B. D., Kim, E., Bouzid, T., Lim, J. Y. The role of microenvironmental cues and mechanical loading milieus in breast cancer cell progression and metastasis. Frontiers in Bioengineering and Biotechnology. 8, 608526(2021).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon