Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Methods Article
Génération de lignées cellulaires knock-out Protein-E CENP-E-/- associées au centromère à l’aide du système CRISPR/Cas9
Dans cet article
Résumé
Cet article rapporte la construction de cellules knock-out de la protéine E associée au centromère (CENP-E) à l’aide du système CRISPR/Cas9 et de trois stratégies de criblage basées sur le phénotype. Nous avons utilisé la lignée cellulaire knock-out CENP-E pour établir une nouvelle approche visant à valider la spécificité et la toxicité des inhibiteurs de CENP-E, ce qui est utile pour le développement de médicaments et la recherche biologique.
Résumé
Le système CRISPR (clustered regularly interspaced short palindromic repeats)/Cas9 est apparu comme un outil puissant pour une édition de gènes précise et efficace dans une variété d’organismes. La protéine E associée au centromère (CENP-E) est une kinésine dirigée vers l’extrémité positive nécessaire à la capture des kinétochores-microtubules, à l’alignement des chromosomes et au point de contrôle de l’assemblage du fuseau. Bien que les fonctions cellulaires des protéines CENP-E aient été bien étudiées, il a été difficile d’étudier les fonctions directes des protéines CENP-E à l’aide de protocoles traditionnels, car l’ablation de CENP-E conduit généralement à l’activation du point de contrôle de l’assemblage du fuseau, à l’arrêt du cycle cellulaire et à la mort cellulaire. Dans cette étude, nous avons complètement éliminé le gène CENP-E dans les cellules HeLa humaines et réussi à générer les cellules CENP-E-/- HeLa à l’aide du système CRISPR/Cas9.
Trois stratégies de criblage optimisées basées sur le phénotype ont été établies, y compris le criblage des colonies cellulaires, les phénotypes d’alignement des chromosomes et les intensités fluorescentes des protéines CENP-E, qui améliorent efficacement l’efficacité du criblage et le taux de réussite expérimentale des cellules knock-out CENP-E . Il est important de noter que la délétion CENP-E entraîne un désalignement des chromosomes, la localisation anormale des protéines sérine/thréonine kinase B (BubR1) du point de contrôle mitotique BUB1 et des défauts mitotiques. De plus, nous avons utilisé le modèle de cellules HeLa knock-out CENP-E pour développer une méthode d’identification des inhibiteurs spécifiques de CENP-E.
Dans cette étude, une approche utile pour valider la spécificité et la toxicité des inhibiteurs de CENP-E a été établie. De plus, cet article présente les protocoles d’édition du génome CENP-E à l’aide du système CRISPR/Cas9, qui pourrait être un outil puissant pour étudier les mécanismes de CENP-E dans la division cellulaire. De plus, la lignée cellulaire knock-out CENP-E contribuerait à la découverte et à la validation des inhibiteurs de CENP-E, qui ont des implications importantes pour le développement de médicaments antitumoraux, les études des mécanismes de division cellulaire en biologie cellulaire et les applications cliniques.
Introduction
L’édition du génome par ingénierie médie les modifications ciblées des gènes dans une variété de cellules et d’organismes. Chez les eucaryotes, la mutagénèse spécifique au site peut être introduite par l’application de nucléases spécifiques à la séquence qui stimulent la recombinaison homologue de l’ADNcible 1. Ces dernières années, plusieurs technologies d’édition du génome, notamment les nucléases à doigts de zinc (ZFN)2,3, les nucléases effectrices de type activateur de transcription (TALEN)4,5 et les méganucléases à tête chercheuse 6,7, ont été conçues pour cliver les génomes à des sites spécifiques, mais ces approches nécessitent une ingénierie complexe des protéines et des procédures expérimentales redondantes. Des études ont montré que le système CRISPR/Cas (procaryotes de type II) est une technologie efficace d’édition de gènes, qui médie spécifiquement le clivage de l’ADN guidé par l’ARN et spécifique au site dans une grande variété de cellules et d’espèces 8,9,10,11. La technologie d’inactivation des gènes CRISPR/Cas9 a révolutionné les domaines de la biologie fondamentale, de la biotechnologie et de la médecine12.
Les bactéries et la plupart des archées ont développé un système immunitaire adaptatif basé sur l’ARN qui utilise les protéines CRISPR et Cas pour identifier et détruire les virus et les plasmides13. Streptococcus pyogenes L’endonucléase Cas9 (SpCas9) contient le résolvase de jonction de Holliday (RuvC) de type RuvC et le domaine His-Asn-His (HNH), qui peuvent servir efficacement de médiateurs pour les cassures double brin (DSB) spécifiques à la séquence en fournissant un ARN monoguide synthétique (ARNsg) contenant des ARN CRISPR (ARNcr) et un ARNcr trans-activateur (ARNtracr)14,15,16. Les DSB peuvent être réparées par la voie de jonction d’extrémité non homologue (NHEJ) ou de réparation dirigée par homologie (HDR), qui introduit de multiples mutations, y compris des insertions, des délétions ou des substitutions de nucléotides uniques sans cicatrice, dans les cellules de mammifères 1,8. La voie NHEJ sujette aux erreurs et la voie HDR haute fidélité peuvent être utilisées pour médier l’inactivation génique par des insertions ou des suppressions, ce qui peut provoquer des mutations par décalage de cadre et des codons d’arrêt prématurés10.
La kinésine-7 CENP-E est nécessaire à la fixation des kinétochores-microtubules et à l’alignement des chromosomes pendant la division cellulaire 17,18,19. La micro-injection d’anticorps20,21, la déplétion de l’ARNsi 22,23, l’inhibition chimique 24,25,26 et la délétion génétique 27,28,29 de CENP-E entraînent un désalignement chromosomique, l’activation du point de contrôle de l’assemblage du fuseau et des défauts mitotiques, ce qui entraîne une aneuploïdie et une instabilité chromosomique 19,30. Chez la souris, la délétion de CENP-E entraîne un développement anormal et une létalité embryonnaire aux tout premiers stades du développement 27,29,31. La délétion génétique de CENP-E entraîne généralement un désalignement chromosomique et la mort cellulaire 26,27,29, ce qui constitue un obstacle à l’étude des fonctions et des mécanismes des protéines CENP-E.
Une étude récente a établi une lignée cellulaire conditionnelle de knock-out CENP-E à l’aide d’une méthode d’édition de gènes CRISPR/Cas9 inductible par l’auxine32, qui permet une dégradation rapide des protéines CENP-E dans un temps relativement court33. Cependant, à ce jour, des lignées cellulaires stables de CENP-E knock-out n’ont pas été établies, ce qui constitue un défi technique non résolu en biologie de CENP-E. Compte tenu de la robustesse génétique34, des réponses de compensation génétique 35,36,37 et des environnements intracellulaires complexes, comme les conséquences directes de la délétion complète de CENP-E peuvent être complexes et imprévisibles, il est important d’établir des lignées cellulaires knock-out CENP-E pour l’étude des mécanismes d’alignement des chromosomes, du point de contrôle de l’assemblage du fuseau et des voies de signalisation en aval.
La découverte et les applications des inhibiteurs de CENP-E sont importantes pour le traitement du cancer. À ce jour, sept types d’inhibiteurs de CENP-E ont été trouvés et synthétisés, notamment GSK923295 et ses dérivés24,25, PF-277138,39, dérivés d’échafaudage imidazo[1,2-a]pyridine40,41, composé A42,43, syntelin 44,45, UA6278446 et dérivés de benzo[d]pyrrolo[2,1-b]thiazole47. Parmi ces inhibiteurs, GSK923295 est un inhibiteur allostérique et efficace de CENP-E qui se lie au domaine moteur de CENP-E et inhibe l’activité de l’ATPase stimulée par les microtubules CENP-E avec un Ki de 3,2 ± 0,2 nM24,25. Cependant, par rapport aux effets inhibiteurs de la GSK923295 sur les cellules cancéreuses en culture, les effets thérapeutiques de la GSK923295 chez les patients cancéreux cliniques ne sont pas idéaux48,49, ce qui a également soulevé des inquiétudes quant à la spécificité de GSK923295 pour CENP-E. De plus, la spécificité et les effets secondaires d’autres inhibiteurs de CENP-E sur les protéines CENP-E sont des questions clés dans la recherche sur le cancer.
Dans cette étude, nous avons complètement éliminé le gène CENP-E dans les cellules HeLa à l’aide du système CRISPR/Cas9. Trois stratégies de criblage optimisées basées sur le phénotype ont été établies, y compris le criblage des colonies cellulaires, les phénotypes d’alignement des chromosomes et les intensités fluorescentes des protéines CENP-E, afin d’améliorer l’efficacité du criblage et le taux de réussite de l’édition du gène CENP-E. De plus, les lignées cellulaires knock-out CENP-E peuvent être utilisées pour tester la spécificité des composés candidats pour CENP-E.
Protocole
1. Construction des vecteurs d’inactivation du gène CRISPR/Cas9
- Sélectionner la séquence d’ADN génomique cible sur le gène CENP-E humain (numéro d’enregistrement GenBank. NM_001286734.2) et concevoir l’ARNsg à l’aide d’un outil de conception CRISPR en ligne (http://crispor.tefor.net/).
- Entrez une seule séquence génomique, sélectionnez le génome de « Homo sapiens-human-UCSC Dec 2013 (hg38 analysis set) + single nucleotide polymorphisms (SNPs) : dbSNP148 », et sélectionnez le motif adjacent protospacer « 20 bp-NGG-spCas9 ». Choisissez deux séquences guides, dont sgRNA-1, 5'-CGGCCGCACTCGCACGCAGA-3' et sgRNA-2 5'-TTCTTTAGAGACGCGGGCTC-3', en fonction des scores de spécificité50, de l’efficacité prédite10 et des hors cibles minimales.
- Ordonner et synthétiser les oligonucléotides d’ADN simple brin (ssODNs). Diluer les ssODN dans ddH2O jusqu’à une concentration finale de 100 μM. Pour phosphoryler et recuire les oligonucléotides d’ARNsg, ajoutez 1 μL d’oligo direct, 1 μL d’oligo inverse, 0,5 μL de polynucléotide kinase T4, 1 μL de tampon polynucléotide kinase T4 et 6,5 μL d’eau exempte de RNase dans un tube d’amplification en chaîne par polymérase (PCR), et incubez le tube à 37 °C pendant 30 min, 95 °C pendant 5 min ; puis, baissez à 25 °C à 5 °C min-1.
- Digérer le plasmide pX458 à l’aide de l’enzyme de restriction Bbs
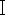 . Ajouter 1 μg du plasmide pX458, 2 μL de tampon 10x G, 1 μL de Bbs
. Ajouter 1 μg du plasmide pX458, 2 μL de tampon 10x G, 1 μL de Bbs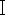 , et ajouter de la ddH2O sans RNase à 20 μL dans un tube à centrifuger de 1,5 mL. Incuber le tube à 37 °C pendant 2 h. Ensuite, purifiez le plasmide linéaire à l’aide du kit d’extraction de gel d’ADN en colonne selon les protocoles du fabricant.
, et ajouter de la ddH2O sans RNase à 20 μL dans un tube à centrifuger de 1,5 mL. Incuber le tube à 37 °C pendant 2 h. Ensuite, purifiez le plasmide linéaire à l’aide du kit d’extraction de gel d’ADN en colonne selon les protocoles du fabricant. - Lister les oligonucléotides de l’ARNsg dans le plasmide pX458 (plasmide pSpCas9(BB)-2A-protéine fluorescente verte (GFP), Addgene ID. 48138). Ajouter 1 μL d’oligonucléotides recuits, 25 ng du plasmide linéaire, 1 μL de tampon de ligature d’ADN 10x T4, 0,5 μL d’ADN ligase T4 (350 U/μL) et compléter à 10 μL avec ddH2O dans un tube à centrifuger de 1,5 mL. Incuber la solution de ligature à 16 °C pendant 2 h.
- Incuber 10 μL du plasmide construit avec les cellules DH5α compétentes sur de la glace pendant 30 min, un choc thermique à 42 °C pendant 45 s, et les placer immédiatement sur de la glace pendant 5 min. Ajouter 500 μL de milieu Luria-Bertani (LB) et ensemencer les cellules sur une plaque LB contenant de l’ampicilline (100 μg/mL). Incuber à 37 °C pendant 16 h et cribler les clones résistants.
- Sélectionnez 5 à 10 colonies individuelles avec une pointe de pipette stérilisée et transférez-les dans une culture de 1 mL de milieu LB avec de l’ampicilline (100 μg/mL). Incuber la culture à 37 °C et agiter à 180 tr/min pendant 12 à 16 h.
- Isolez l’ADN plasmidique à l’aide d’un kit d’extraction plasmidique selon les protocoles du fabricant. Valider l’ADN plasmidique de chaque colonie par séquençage de Sanger à partir du site promoteur U6 à l’aide de l’amorce U6-Fwd 5'-GAGGGCCTATTTCCCATGATTCC-3'.
- Extrayez et purifiez les plasmides à l’aide du kit d’ADN plasmidique sans endo selon les protocoles du fabricant.
2. Transfection, isolement et criblage des cellules HeLa knock-out CENP-E
- Cultiver des cellules HeLa dans un milieu d’aigle modifié Dulbecco (DMEM) complétées par 10 % de sérum de veau fœtal et 1 % de pénicilline/streptomycine dans un incubateur humidifié à 37 °C avec 5 % de CO2.
- Incuber les cellules HeLa à l’aide de la trypsine/éthylène diamine tétraacétique (EDTA) à 0,25 % à 37 °C pendant 2 à 3 min, dissocier les cellules par pipetage doux, puis ensemencer les cellules dans de nouvelles plaques à un rapport de dilution de 1 :4 pour le passage cellulaire.
- Ensemencer les cellules dans une plaque à 12 puits et cultiver à 60-70% de confluence avant la transfection. Transfecter les plasmides pX458-sgRNA dans des cellules HeLa à l’aide des réactifs référencés selon les protocoles du fabricant (voir le tableau des matériaux).
- Mélangez délicatement 1 μg de plasmide d’ARNsg pX458-validé et 50 μL de milieu sérique réduit dans le tube A. Ensuite, mélangez délicatement 2 μL de réactif de transfection et 50 μL de milieu sérique réduit dans le tube B. Incuber les tubes A et B séparément à température ambiante pendant 5 min.
- Mélanger doucement les tubes A et B et incuber à température ambiante pendant 5 min. Ajouter le mélange dans une assiette à 12 puits, incuber les cellules pendant 6 h et remplacer le milieu par un milieu DMEM frais.
- Examinez les cellules HeLa après 24 h ou 48 h à l’aide d’un microscope à fluorescence pour vérifier l’efficacité de la transfection. Après transfection pendant 48 h, dissocier complètement les cellules HeLa transfectées à l’aide de trypsine/EDTA à 0,25 % à 37 °C pendant 3 à 5 min, compter le nombre de cellules à l’aide d’une chambre de Neubauer ou d’un compteur de cellules automatisé, et mettre les cellules en plaques distinctes de 96 puits pour chaque population transfectée selon des méthodes de dilution en série10.
- Remettez les cellules dans un incubateur humidifié, puis cultivez-les pendant 1 à 2 semaines supplémentaires. Changez le milieu de culture une fois tous les 3 jours. Pour le criblage et la validation phénotypiques de l’inactivation de CENP-E , dissocier et dilater les cellules dans des plaques de 24 ou 12 puits pendant 5 à 7 jours.
- Criblez les cellules mutantes avec des diamètres de colonie cellulaire plus petits à l’aide d’un microscope inversé équipé de lentilles d’objectif 10x et 20x.
REMARQUE : Les mutations CENP-E entraînent généralement une augmentation significative du nombre de cellules en division dans les colonies cellulaires, ce qui peut être l’un des indicateurs clés pour le dépistage des cellules mutantes CENP-E . - Récoltez les cellules pour l’extraction de l’ADN à l’aide du kit d’extraction d’ADN génomique animal en colonne selon les protocoles du fabricant. Configurez les réactions de PCR comme suit : 0,25 μL de Taq polymérase, 5 μL de tampon 10x (Mg2+ plus), 4 μL de mélanges dNTP, 500 ng de matrice d’ADN, 1 μL d’oligo direct, 1 μL d’oligo inverse, et ajustez ddH2O à 50 μL. Utilisez les paramètres de programme PCR suivants pour l’amplification génique : 98 °C, 10 s ; 98 °C, 10 s, 55 °C, 30 s, 72 °C, 60 s pendant 33 cycles ; 72 °C, 10 min, puis maintenir à 4 °C.
NOTE : Les amorces spécifiques pour le clonage du locus cible sont énumérées comme suit : cible CENP-E F1, 5'-GAGGGTCCTGGCCATTTTCTCTG-3' ; Cible CENP-E R1, 5'-AGATCTCCGATCCTCCCCTCTCTC-3' ; Cible CENP-E F2, 5'-TGGTAACTGCATTTTGGTGTTCTAC-3' ; Cible R2 du CENP-E , 5'-CCTGTTGCAACGTGAGGGAAG-3'. - Lister l’ADN cible dans le vecteur pMD18-T selon les protocoles du fabricant. Transfectez le plasmide ligaturé dans des cellules DH5α compétentes et cultivez-les pour la sélection des clones.
- Effectuez un séquençage de Sanger pour déterminer les types d’inactivation de CENP-E . Isolez l’ADN plasmidique à l’aide d’un kit d’extraction plasmidique selon les protocoles du fabricant. Effectuez le séquençage de Sanger de l’ADN plasmidique de chaque colonie à l’aide des amorces directe et inverse M13. amorce M13F, 5'-TGTAAAACGACGGCCAGT-3' ; Amorce M13R, 5'-CAGGAAACAGCTATGACC-3'.
- Ensemencer les cellules HeLa de type sauvage et les cellules mutantes CENP-E sur des lamelles de verre de 12 mm dans une plaque de 24 puits, respectivement. Retirer le milieu DMEM complet et fixer les cellules dans une solution fixatrice de paraformaldéhyde/solution saline tamponnée au phosphate (PBS) à 4 % à température ambiante pendant 10 min.
- Colorer les noyaux avec la solution de 4',6-diamidino-2-phénylindole (DAPI) à température ambiante pendant 5 min. Criblez et validez les cellules mutantes CENP-E en fonction du phénotype de l’alignement des chromosomes à l’aide d’un microscope à fluorescence équipé d’un objectif Plan Fluor 40x/Numerical Aperture (NA) 0,75.
- Criblage et validation des cellules knock-out CENP-E par coloration par immunofluorescence et analyse (voir rubrique 3) des protéines CENP-E.
3. Coloration par immunofluorescence et microscopie confocale à haute résolution
- Fixer les cellules avec une solution fixatrice de paraformaldéhyde/PBS à 4 % à température ambiante pendant 10 min et les immerger pendant 2 x 5 min dans 1 PBS.
REMARQUE : Assurez-vous que les cellules sont en bon état et que les cellules sont collectées et fixées doucement pour éviter le détachement cellulaire en métaphase. - Perméabiliser les cellules avec 0,25% de Triton X-100/PBS à température ambiante pendant 10 min et les immerger dans 1x PBS pendant 2 x 5 min.
- Bloquer les cellules avec 1 % de BSA/PBST (0,1 % de Tween 20 dans PBS) à température ambiante pendant 1 h. Diluer les anticorps primaires avec 1 % de BSA/PBST et incuber les échantillons à 4 °C pendant 12 h.
- Jeter les solutions d’anticorps primaires, rincer les cellules 3 x 5 min avec 1x PBS et incuber les cellules avec des anticorps secondaires dilués à température ambiante pendant 2 h.
- Jeter les anticorps secondaires et rincer les cellules dans 1x PBS pendant 3 x 5 min.
- Teindre les noyaux avec du DAPI à température ambiante pendant 5 min. Montez les diapositives avec le support de montage. Scellez les lames avec du vernis à ongles.
- Observez et enregistrez les images de fluorescence à l’aide d’un microscope confocal à balayage équipé d’un objectif 63x/NA 1.40.
4. Préparation des chromosomes et analyse du caryotype
- Cultiver les cellules HeLa de type sauvage et CENP-E-/- dans un milieu DMEM complet complété par 10 % de sérum bovin fœtal et 1 % de pénicilline/streptomycine pendant 24 h, puis incuber les cellules HeLa de type sauvage et CENP-E-/- avec 300 nM de colchicine pendant 5 h.
- Incuber les cellules avec 0,25 % de trypsine/EDTA à 37 °C pendant 3 min et recueillir les cellules dans des tubes à centrifuger de 1,5 ml.
- Centrifuger les cellules à 1 000 × g pendant 5 minutes à température ambiante, jeter les surnageants, ajouter 1,2 mL de solution de KCl à 0,075 mol/L et incuber à 37 °C pendant 20 min.
- Centrifuger les cellules à 1 000 × g pendant 5 min à température ambiante, jeter les surnageants, ajouter 0,2 mL de solution fixatrice (méthanol : acide acétique glacial = 3 :1) pour la préfixation, et mélanger doucement pendant 1 min.
- Centrifuger les cellules à 1 000 × g pendant 5 min à température ambiante, recueillir les pastilles, ajouter 1,5 mL de solution fixatrice à température ambiante pendant 10 min, puis centrifuger à 1 000 × g pendant 5 min à température ambiante.
- Jeter les surnageants, ajouter 0,6 mL de solutions fixatrices et préparer une suspension cellulaire. Ajoutez 3 à 5 gouttes de suspensions cellulaires d’une hauteur de 35 à 40 cm sur les toboggans glaciaires.
REMARQUE : Maintenez la hauteur de libération des suspensions de cellules sur les glissières à 35-40 cm ; sinon, les chromosomes risquent d’être trop dispersés ou non séparés les uns des autres. - Séchez immédiatement les lames avec une lampe à alcool, colorez les échantillons avec une solution colorante Giemsa à 10 % pendant 7 min, rincez les lames à l’eau courante pendant 2 min et observez les échantillons à l’aide d’un microscope optique équipé d’un objectif Plan Fluor 40x/NA 0,75.
5. Essai de formation de colonies cellulaires
- Préparez les suspensions cellulaires dans les plaques à 6 puits à une densité cellulaire de 1 000 à 2 000 cellules/puits. Secouez doucement les plaques pour répartir uniformément les cellules.
- Placez les plaques à 6 puits dans un incubateur de CO2 et incubez pendant 2 à 3 semaines à 37 °C avec 5 % de CO2. Changez de milieu de culture une fois tous les 5 jours. Enregistrez les images à l’aide d’un microscope inversé équipé d’objectifs 10x et 20x. Récoltez les cellules lorsque les colonies sont visibles (des centaines de cellules dans un clone).
- Pour étudier les effets de la GSK923295, cultivez les cellules dans les plaques à 6 puits pendant 24 h et ajoutez 2 mL de GSK923295 aux concentrations finales de 10, 25, 50, 100 et 200 nM.
REMARQUE : Ne déplacez pas la boîte de culture au début de la formation des clones pour éviter la perte de cellules, qui peut former de nouveaux clones et affecter le criblage des cellules transfectées. - Jeter le milieu de culture et fixer les cellules avec 1 % de PFA à température ambiante pendant 10 min. Teindre les colonies avec une solution de coloration violet cristallin à 0,1 % à température ambiante pendant 15 min. Rincez les échantillons avec 1x PBS trois fois. Enregistrez les images des colonies cellulaires et quantifiez les diamètres de chaque colonie à l’aide du logiciel ImageJ. Choisissez l’outil Sélection de ligne droite , cliquez sur Analyser, choisissez Mesurer et enregistrez la longueur.
- Rincer les colonies avec 1 mL de solution d’acide acétique à 10 % pendant 5 min. Transférer 200 μL des solutions surnageantes dans une plaque de 96 puits et mesurer les valeurs d’absorbance à l’aide d’un spectrophotomètre à microplaques àA 600 nm.
6. Essai de viabilité cellulaire
- Ensemencer les cellules dans les plaques de 24 puits et cultiver les cellules pendant 48 h à une densité de 80 à 90 %.
- Incuber les cellules avec 100 μL de trypsine/EDTA à 0,25 % à 37 °C pendant 3 min.
- Mélanger 400 μL de PBS avec les cellules à l’aide d’un pistolet à pipette. Transférer 100 μL de suspensions cellulaires dans la plaque à 96 puits.
- Ajouter 20 μL de solution de MTS dans chaque puits avec une concentration finale de 317 μg/mL et mélanger doucement selon les protocoles du fabricant. Incuber les échantillons à 37 °C pendant 1 à 3 h dans un incubateur de CO2 .
- Enregistrer les valeurs d’absorbance de chaque puits au pic d’absorbance deA 490 nm à l’aide d’un spectrophotomètre à microplaques. Utilisez une longueur d’onde de référence de A630 nm pour soustraire le bruit de fond apporté par les débris cellulaires et l’absorbance non spécifique (viabilité cellulaire = A490 nm - A630 nm).
Résultats
Les cellules CENP-E-/- HeLa ont été générées avec succès à l’aide du système CRISPR/Cas9 (Figure 1). La chronologie et les étapes expérimentales critiques de cette méthode sont illustrées à la figure 1. Tout d’abord, nous avons conçu et synthétisé les sgRNA spécifiques de CENP-E, recuit et ligaturé les sgRNA dans le plasmide pX458, transfecté le plasmide dans des cellules HeLa et les avons cultivés pendant 48 ...
Discussion
La kinésine-7 CENP-E est un régulateur clé de l’alignement des chromosomes et du point de contrôle de l’assemblage du fuseau pendant la division cellulaire 17,19,20. La délétion génétique de CENP-E entraîne généralement l’activation du point de contrôle de l’assemblage du fuseau, l’arrêt du cycle cellulaire et la mort cellulaire 27,29,51,52.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous remercions tous les membres du laboratoire de cytosquelette de l’Université médicale du Fujian pour leurs discussions utiles. Nous remercions Jun-Jin Lin, Zhi-Hong Huang, Ling Lin, Li-Li Pang, Lin-Ying Zhou, Xi Lin et Min-Xia Wu du Centre de services technologiques publics de l’Université médicale du Fujian pour leur assistance technique. Nous remercions Si-Yi Zheng, Ying Lin et Qi Ke du Centre d’enseignement expérimental des sciences médicales fondamentales de l’Université médicale du Fujian pour leur soutien. Cette étude a été soutenue par les subventions suivantes : Fondation nationale des sciences naturelles de Chine (numéro de subvention 82001608 et 82101678), Fondation des sciences naturelles de la province du Fujian, Chine (numéro de subvention 2019J05071), Fonds conjoints pour l’innovation de la science et de la technologie, province du Fujian, Chine (numéro de subvention 2021Y9160), et Projet de financement de démarrage de la recherche scientifique sur les talents de haut niveau de l’Université médicale du Fujian (numéro de subvention XRCZX2017025).
matériels
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | |
| 1.5 mL centrifuge tube | Axygen | MCT-150-C | |
| 24-well plate | Corning | 3524 | |
| 4S Gelred, 10,000x in water | Sangon Biotech (Shanghai) | A616697 | |
| 50 mL centrifuge tube | Corning | 430828 | |
| 6 cm Petri dish | Corning | 430166 | |
| 95% ethanol | Sinopharm Chemical Reagent | 10009164 | |
| 96-well plate | Corning | 3599 | |
| Acetic acid | Sinopharm Chemical Reagent | 10000218 | Dissolve in H2O to prepare a 10% working solution. |
| Agarose | Sangon Biotech (Shanghai) | A620014 | |
| Alexa Fluor 488-labeled Goat Anti-Mouse IgG(H+L) | Beyotime | A0428 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Alexa Fluor 488-labeled Goat Anti-Rabbit IgG(H+L) | Beyotime | A0423 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Alexa Fluor 555-labeled Donkey Anti-Mouse IgG(H+L) | Beyotime | A0460 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Anhydrous ethanol | Sinopharm Chemical Reagent | 100092690 | |
| Anti-BubR1 rabbit monoclonal antibody | Abcam | ab254326 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution |
| Anti-CENP-B mouse monoclonal antibody | Santa Cruz Biotechnology | sc-376392 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:50 dilution. |
| Anti-CENP-E rabbit monoclonal antibody | Abcam | ab133583 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution. |
| Anti-fade mounting medium | Beyotime | P0131 | Slowing down the quenching of fluorescent signals. |
| Anti-α-tubulin mouse monoclonal antibody | Abcam | ab7291 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution. |
| Biotek Epoch Microplate Spectrophotometer | Biotek Instruments | Biotek Epoch | |
| Bovine Serum Albumin (BSA) | Sinopharm Chemical Reagent | 69003435 | |
| BpiI (BbsI) | Thermo Fisher Scientific | ER1011 | |
| CellTiter 96 aqueous one solution cell proliferation assay | Promega | G3580 | |
| Centrifuge | Eppendorf | 5424BK745380 | |
| Colchicine | Sinopharm Chemical Reagent | 61001563 | |
| Confocal scanning microscope | Leica | Leica TCS SP8 | |
| Coverslip | CITOTEST | 80344-1220 | |
| DAPI | Beyotime | C1006 | |
| DH5α competent cells | Sangon Biotech (Shanghai) | B528413 | |
| DL2000 DNA marker | TaKaRa | 3427A | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | C11995500BT | |
Endo-free plasmid mini kit  | Omega | D6950 | |
| Ezup Column Animal Genomic DNA Purification Kit | Sangon Biotech (Shanghai) | B518251 | |
| Fetal bovine serum | Zhejiang Tianhang Biotechnology | 11011-8611 | |
| Gentian violet | Sinopharm Chemical Reagent | 71019944 | Dissolve in PBS to prepare 0.1% gentian violet/PBS. |
| Giemsa staining solution | Sinopharm Chemical Reagent | 71020260 | |
| GraphPad Prism version 8.0 software | GraphPad | www.graphpad.com | Statistical analysis. |
| GSK923295 | MedChemExpress | HY-10299 | |
| HeLa cell line | ATCC | CCL-2 | |
| Humidified incubator | Heal Force | HF90/HF240 | |
| Image J software | National Institutes of Health | https://imagej.nih.gov/ij/ | Image processing and analysis. |
| Inverted microscope | Nanjing Jiangnan Novel Optics | XD-202 | |
| LB agar powder | Sangon Biotech (Shanghai) | A507003 | |
| Lipo6000 transfection reagent | Beyotime | C0526 | |
| Nikon Ti-S2 microscope | Nikon | Ti-S2 | |
| Opti-MEM reduced serum medium | Gibco | 31985070 | |
| Paraformaldehyde | Sinopharm Chemical Reagent | 80096618 | Dissolve in PBS to prepare 4% paraformaldehyde/PBS. |
| Penicillin-streptomycin solution | HyClone | SV30010 | |
| SanPrep column DNA gel extraction kit | Sangon Biotech (Shanghai) | B518131 | |
| SanPrep column plasmid mini-preps kit | Sangon Biotech (Shanghai) | B518191 | |
| T4 DNA ligase | TaKaRa | 2011A | |
| T4 polynucleotide kinase | TaKaRa | 2021A | |
| TaKaRa Ex Taq | TaKaRa | RR001A | |
| Triton X-100 | Sinopharm Chemical Reagent | 30188928 | Dissolve in PBS to prepare 0.25% Triton X-100/PBS. |
| Tween 20 | Sinopharm Chemical Reagent | 30189328 | Dissolve in PBS to prepare 0.1% Tween 20/PBS. |
Références
- Jiang, W., Bikard, D., Cox, D., Zhang, F., Marraffini, L. A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology. 31 (3), 233-239 (2013).
- Bibikova, M., Beumer, K., Trautman, J. K., Carroll, D. Enhancing gene targeting with designed zinc finger nucleases. Science. 300 (5620), 764 (2003).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Wood, A. J., et al. Targeted genome editing across species using ZFNs and TALENs. Science. 333 (6040), 307 (2011).
- Stoddard, B. L. Homing endonuclease structure and function. Quarterly Reviews of Biophysics. 38 (1), 49-95 (2005).
- Pâques, F., Duchateau, P. Meganucleases and DNA double-strand break-induced recombination: perspectives for gene therapy. Current Gene Therapy. 7 (1), 49-66 (2007).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Shmakov, S., et al. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell. 60 (3), 385-397 (2015).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Jia, N., Patel, D. J. Structure-based functional mechanisms and biotechnology applications of anti-CRISPR proteins. Nature Reviews Molecular Cell Biology. 22 (8), 563-579 (2021).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jackson, S. A., et al. CRISPR-Cas: Adapting to change. Science. 356 (6333), eaal5056 (2017).
- Wood, K. W., Sakowicz, R., Goldstein, L. S., Cleveland, D. W. CENP-E is a plus end-directed kinetochore motor required for metaphase chromosome alignment. Cell. 91 (3), 357-366 (1997).
- Craske, B., Welburn, J. P. I. Leaving no-one behind: how CENP-E facilitates chromosome alignment. Essays in Biochemistry. 64 (2), 313-324 (2020).
- Yu, K. W., Zhong, N., Xiao, Y., She, Z. Y. Mechanisms of kinesin-7 CENP-E in kinetochore-microtubule capture and chromosome alignment during cell division. Biology of the Cell. 111 (6), 143-160 (2019).
- Schaar, B. T., Chan, G. K., Maddox, P., Salmon, E. D., Yen, T. J. CENP-E function at kinetochores is essential for chromosome alignment. Journal of Cell Biology. 139 (6), 1373-1382 (1997).
- McEwen, B. F., et al. CENP-E is essential for reliable bioriented spindle attachment, but chromosome alignment can be achieved via redundant mechanisms in mammalian cells. Molecular Biology of the Cell. 12 (9), 2776-2789 (2001).
- Yao, X., Abrieu, A., Zheng, Y., Sullivan, K. F., Cleveland, D. W. CENP-E forms a link between attachment of spindle microtubules to kinetochores and the mitotic checkpoint. Nature Cell Biology. 2 (8), 484-491 (2000).
- Kim, Y., Heuser, J. E., Waterman, C. M., Cleveland, D. W. CENP-E combines a slow, processive motor and a flexible coiled coil to produce an essential motile kinetochore tether. Journal of Cell Biology. 181 (3), 411-419 (2008).
- Qian, X., et al. Discovery of the first potent and selective inhibitor of Centromere-Associated Protein E: GSK923295. ACS Medicinal Chemistry Letters. 1 (1), 30-34 (2010).
- Wood, K. W., et al. Antitumor activity of an allosteric inhibitor of centromere-associated protein-E. Proceedings of the National Academy of Sciences of the United States of America. 107 (13), 5839-5844 (2010).
- Pisa, R., Phua, D. Y. Z., Kapoor, T. M. Distinct mechanisms of resistance to a CENP-E inhibitor emerge in near-haploid and diploid cancer cells. Cell Chemical Biology. 27 (7), 850.e6-857.e6 (2020).
- Putkey, F. R., et al. Unstable kinetochore-microtubule capture and chromosomal instability following deletion of CENP-E. Developmental Cell. 3 (3), 351-365 (2002).
- Weaver, B. A., Silk, A. D., Montagna, C., Verdier-Pinard, P., Cleveland, D. W. Aneuploidy acts both oncogenically and as a tumor suppressor. Cancer Cell. 11 (1), 25-36 (2007).
- Silk, A. D., et al. Chromosome missegregation rate predicts whether aneuploidy will promote or suppress tumors. Proceedings of the National Academy of Sciences of the United States of America. 110 (44), E4134-E4141 (2013).
- Guo, Y., Kim, C., Ahmad, S., Zhang, J., Mao, Y. CENP-E-dependent BubR1 autophosphorylation enhances chromosome alignment and the mitotic checkpoint. Journal of Cell Biology. 198 (2), 205-217 (2012).
- Weaver, B. A., Cleveland, D. W. Aneuploidy: instigator and inhibitor of tumorigenesis. Cancer Research. 67 (21), 10103-10105 (2007).
- Nishimura, K., Fukagawa, T. An efficient method to generate conditional knockout cell lines for essential genes by combination of auxin-inducible degron tag and CRISPR/Cas9. Chromosome Research. 25 (3-4), 253-260 (2017).
- Owa, M., Dynlacht, B. A non-canonical function for Centromere-associated protein-E controls centrosome integrity and orientation of cell division. Communications Biology. 4 (1), 358 (2021).
- Barabási, A. L., Oltvai, Z. N. Network biology: understanding the cell's functional organization. Nature Reviews Genetics. 5 (2), 101-113 (2004).
- Rossi, A., et al. Genetic compensation induced by deleterious mutations but not gene knockdowns. Nature. 524 (7564), 230-233 (2015).
- Ma, Z., et al. PTC-bearing mRNA elicits a genetic compensation response via Upf3a and COMPASS components. Nature. 568 (7751), 259-263 (2019).
- El-Brolosy, M. A., et al. Genetic compensation triggered by mutant mRNA degradation. Nature. 568 (7751), 193-197 (2019).
- Kung, P. P., et al. Chemogenetic evaluation of the mitotic kinesin CENP-E reveals a critical role in triple-negative breast cancer. Molecular Cancer Therapeutics. 13 (8), 2104-2115 (2014).
- Kim, J. H., et al. Development of a novel HAC-based "gain of signal" quantitative assay for measuring chromosome instability (CIN) in cancer cells. Oncotarget. 7 (12), 14841-14856 (2016).
- Hirayama, T., et al. Synthetic studies of centromere-associated protein-E (CENP-E) inhibitors: 1. Exploration of fused bicyclic core scaffolds using electrostatic potential map. Bioorganic & Medicinal Chemistry. 21 (17), 5488-5502 (2013).
- Hirayama, T., et al. Synthetic studies on Centromere-associated protein-E (CENP-E) inhibitors: 2. Application of electrostatic potential map (EPM) and structure-based modeling to Imidazo[1,2-a]pyridine derivatives as anti-tumor agents. Journal of Medicinal Chemistry. 58 (20), 8036-8053 (2015).
- Ohashi, A., et al. A novel time-dependent CENP-E inhibitor with potent antitumor activity. PLoS One. 10 (12), e0144675 (2015).
- Ohashi, A., et al. Aneuploidy generates proteotoxic stress and DNA damage concurrently with p53-mediated post-mitotic apoptosis in SAC-impaired cells. Nature Communications. 6, 7668 (2015).
- Ding, X., et al. Probing CENP-E function in chromosome dynamics using small molecule inhibitor syntelin. Cell Research. 20 (12), 1386-1389 (2010).
- Liu, X., et al. Phase separation drives decision making in cell division. Journal of Biological Chemistry. 295 (39), 13419-13431 (2020).
- Henderson, M. C., et al. UA62784, a novel inhibitor of centromere protein E kinesin-like protein. Molecular Cancer Therapeutics. 8 (1), 36-44 (2009).
- Yamane, M., et al. Identification of benzo[d]pyrrolo[2,1-b]thiazole derivatives as CENP-E inhibitors. Biochemical and biophysical research communications. 519 (3), 505-511 (2019).
- Lock, R. B., et al. Initial testing of the CENP-E inhibitor GSK923295A by the pediatric preclinical testing program. Pediatric Blood & Cancer. 58 (6), 916-923 (2012).
- Chung, V., et al. First-time-in-human study of GSK923295, a novel antimitotic inhibitor of centromere-associated protein E (CENP-E), in patients with refractory cancer. Cancer Chemotherapy and Pharmacology. 69 (3), 733-741 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature biotechnology. 31 (9), 827-832 (2013).
- Weaver, B. A., Cleveland, D. W. Decoding the links between mitosis, cancer, and chemotherapy: The mitotic checkpoint, adaptation, and cell death. Cancer Cell. 8 (1), 7-12 (2005).
- Manchado, E., Guillamot, M., Malumbres, M. Killing cells by targeting mitosis. Cell Death and Differentiation. 19 (3), 369-377 (2012).
- Dang, Y., et al. Optimizing sgRNA structure to improve CRISPR-Cas9 knockout efficiency. Genome Biology. 16, 280 (2015).
- Ratz, M., Testa, I., Hell, S. W., Jakobs, S. CRISPR/Cas9-mediated endogenous protein tagging for RESOLFT super-resolution microscopy of living human cells. Scientific Reports. 5, 9592 (2015).
- Koch, B., et al. Generation and validation of homozygous fluorescent knock-in cells using CRISPR-Cas9 genome editing. Nature Protocols. 13 (6), 1465-1487 (2018).
- Yan, Q., et al. Multiplex CRISPR/Cas9-based genome engineering enhanced by Drosha-mediated sgRNA-shRNA structure. Scientific Reports. 6, 38970 (2016).
- Cheng, Y., et al. CRISPR/Cas9-mediated chicken TBK1 gene knockout and its essential role in STING-mediated IFN-β induction in chicken cells. Frontiers in Immunology. 9, 3010 (2019).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Zeng, W., Guo, L., Xu, S., Chen, J., Zhou, J. High-throughput screening technology in industrial biotechnology. Trends in Biotechnology. 38 (8), 888-906 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nature Biotechnology. 33 (2), 187-197 (2015).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Ablain, J., Durand, E. M., Yang, S., Zhou, Y., Zon, L. I. A CRISPR/Cas9 vector system for tissue-specific gene disruption in zebrafish. Developmental Cell. 32 (6), 756-764 (2015).
- Pickar-Oliver, A., Gersbach, C. A. The next generation of CRISPR-Cas technologies and applications. Nature Reviews Molecular Cell Biology. 20 (8), 490-507 (2019).
- Nalawansha, D. A., Gomes, I. D., Wambua, M. K., Pflum, M. K. H. HDAC inhibitor-induced mitotic arrest is mediated by Eg5/KIF11 acetylation. Cell Chemical Biology. 24 (4), 481.e5-492.e5 (2017).
- Kavalapure, R. S., et al. Design, synthesis, and molecular docking study of some 2-((7-chloroquinolin-4-yl) amino) benzohydrazide Schiff bases as potential Eg5 inhibitory agents. Bioorganic Chemistry. 116, 105381 (2021).
- Calligaris, D., Lafitte, D. Chemical inhibitors: the challenge of finding the right target. Chemistry & Biology. 18 (5), 555-557 (2011).
- Łomzik, M., et al. Metal-dependent cytotoxic and kinesin spindle protein inhibitory activity of Ru, Os, Rh, and Ir half-sandwich complexes of Ispinesib-derived ligands. Inorganic Chemistry. 59 (20), 14879-14890 (2020).
- Ferro, L. S., et al. Structural and functional insight into regulation of kinesin-1 by microtubule-associated protein MAP7. Science (New York, N.Y.). 375 (6578), 326-331 (2022).
- Atherton, J., et al. The mechanism of kinesin inhibition by kinesin-binding protein. eLife. 9, e61481 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon