È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione di linee cellulari knockout CENP-E-/- associate al centromero della proteina E utilizzando il sistema CRISPR/Cas9
In questo articolo
Riepilogo
Questo articolo riporta la costruzione di cellule knockout della proteina E associata al centromero (CENP-E) utilizzando il sistema CRISPR/Cas9 e tre strategie di screening basate sul fenotipo. Abbiamo utilizzato la linea cellulare knockout CENP-E per stabilire un nuovo approccio per convalidare la specificità e la tossicità degli inibitori CENP-E, utile per lo sviluppo di farmaci e la ricerca biologica.
Abstract
Il sistema CRISPR (clustered regularly interspaced short palindromic repeats)/Cas9 è emerso come un potente strumento per l'editing genetico preciso ed efficiente in una varietà di organismi. La proteina-E ASSOCIATA AL CENTROMERO (CENP-E) è una chinesina diretta all'estremità positiva necessaria per la cattura del cinetocoro-microtubuli, l'allineamento cromosomico e il checkpoint dell'assemblaggio del fuso. Sebbene le funzioni cellulari delle proteine CENP-E siano state ben studiate, è stato difficile studiare le funzioni dirette delle proteine CENP-E utilizzando i protocolli tradizionali perché l'ablazione di CENP-E di solito porta all'attivazione del checkpoint dell'assemblaggio del fuso, all'arresto del ciclo cellulare e alla morte cellulare. In questo studio, abbiamo completamente eliminato il gene CENP-E nelle cellule HeLa umane e generato con successo le cellule CENP-E-/- HeLa utilizzando il sistema CRISPR/Cas9.
Sono state stabilite tre strategie di screening ottimizzate basate sul fenotipo, tra cui lo screening delle colonie cellulari, i fenotipi di allineamento cromosomico e le intensità fluorescenti delle proteine CENP-E, che migliorano efficacemente l'efficienza dello screening e il tasso di successo sperimentale delle cellule knockout CENP-E . È importante sottolineare che la delezione di CENP-E provoca il disallineamento cromosomico, la posizione anomala delle proteine serina/treonina chinasi B (BubR1) del checkpoint mitotico BUB1 e difetti mitotici. Inoltre, abbiamo utilizzato il modello di cellula HeLa knockout di CENP-E per sviluppare un metodo di identificazione per inibitori specifici di CENP-E.
In questo studio è stato stabilito un approccio utile per convalidare la specificità e la tossicità degli inibitori di CENP-E. Inoltre, questo articolo presenta i protocolli di editing genetico CENP-E utilizzando il sistema CRISPR/Cas9, che potrebbe essere un potente strumento per studiare i meccanismi di CENP-E nella divisione cellulare. Inoltre, la linea cellulare knockout di CENP-E contribuirebbe alla scoperta e alla convalida degli inibitori di CENP-E, che hanno importanti implicazioni per lo sviluppo di farmaci antitumorali, studi sui meccanismi di divisione cellulare nella biologia cellulare e applicazioni cliniche.
Introduzione
L'editing genomico ingegnerizzato media le modificazioni mirate dei geni in una varietà di cellule e organismi. Negli eucarioti, la mutagenesi sito-specifica può essere introdotta dall'applicazione di nucleasi sequenza-specifiche che stimolano la ricombinazione omologa del DNAbersaglio 1. Negli ultimi anni, diverse tecnologie di editing del genoma, tra cui le nucleasi a dita di zinco (ZFNs)2,3, le nucleasi effettrici simili all'attivatore di trascrizione (TALEN)4,5 e le meganucleasi homing 6,7, sono state progettate per scindere i genomi in siti specifici, ma questi approcci richiedono una complessa ingegneria proteica e procedure sperimentali ridondanti. Gli studi hanno dimostrato che il sistema CRISPR (Regular Interspaced Short Palindromic Repeats) procariotico di tipo II è una tecnologia di editing genetico efficiente, che media specificamente la scissione del DNA guidata dall'RNA e sito-specifica in un'ampia varietà di cellule e specie 8,9,10,11. La tecnologia di knockout genico CRISPR/Cas9 ha rivoluzionato i campi della biologia di base, della biotecnologia e della medicina12.
I batteri e la maggior parte degli archaea hanno sviluppato un sistema immunitario adattativo basato sull'RNA che utilizza le proteine CRISPR e Cas per identificare e distruggere virus e plasmidi13. Streptococcus pyogenes L'endonucleasi Cas9 (SpCas9) contiene la risoluvasi della giunzione di Holliday simile a RuvC (RuvC) e il dominio His-Asn-His (HNH), che possono mediare in modo efficiente le rotture a doppio filamento (DSB) specifiche della sequenza fornendo un RNA sintetico a guida singola (sgRNA) contenente RNA CRISPR (crRNA) e crRNA transattivante (tracrRNA)14,15,16 . I DSB possono essere riparati attraverso la via di giunzione non omologa (NHEJ) o di riparazione diretta all'omologia (HDR), che introduce mutazioni multiple, tra cui inserzioni, delezioni o sostituzioni a singolo nucleotide senza cicatrici, nelle cellule di mammifero 1,8. Sia l'NHEJ soggetto a errori che la via HDR ad alta fedeltà possono essere utilizzati per mediare il knockout genico attraverso inserzioni o delezioni, che possono causare mutazioni frameshift e codoni di stop prematuri10.
La chinesina-7 CENP-E è necessaria per l'attacco del cinetocoro-microtubulo e l'allineamento cromosomico durante la divisione cellulare 17,18,19. La microiniezione di anticorpi20,21, la deplezione di siRNA22,23, l'inibizione chimica 24,25,26 e la delezione genetica 27,28,29 di CENP-E portano al disallineamento cromosomico, all'attivazione del checkpoint dell'assemblaggio del fuso e ai difetti mitotici, che si traducono in aneuploidia e instabilità cromosomica 19,30. Nei topi, la delezione di CENP-E provoca uno sviluppo anomalo e la letalità embrionale nelle primissime fasi dello sviluppo 27,29,31. La delezione genetica di CENP-E di solito porta al disallineamento cromosomico e alla morte cellulare 26,27,29, che è un ostacolo nello studio delle funzioni e dei meccanismi delle proteine CENP-E.
Uno studio recente ha stabilito una linea cellulare knockout condizionale di CENP-E utilizzando un metodo di editing genetico CRISPR/Cas9 inducibile dall'auxina32, che consente una rapida degradazione delle proteine CENP-E in un tempo relativamente breve33. Tuttavia, ad oggi, non sono state stabilite linee cellulari knockout stabili per CENP-E, il che rappresenta una sfida tecnica irrisolta nella biologia del CENP-E. Considerando la robustezza genetica34, le risposte di compensazione genetica 35,36,37 e gli ambienti intracellulari complessi, poiché le conseguenze dirette della delezione completa di CENP-E possono essere complesse e imprevedibili, è importante stabilire linee cellulari knockout CENP-E per lo studio dei meccanismi di allineamento cromosomico, checkpoint dell'assemblaggio del fuso e vie di segnalazione a valle.
La scoperta e le applicazioni degli inibitori di CENP-E sono importanti per il trattamento del cancro. Ad oggi, sono stati trovati e sintetizzati sette tipi di inibitori di CENP-E, tra cui GSK923295 e i suoi derivati24,25, PF-277138,39, derivati dell'impalcatura imidazo[1,2-a]piridina40,41, composto-A42,43, sintelina44,45, UA6278446 e derivati benzo[d]pirrolo[2,1-b]tiazolo47. Tra questi inibitori, GSK923295 è un inibitore allosterico ed efficiente di CENP-E che si lega al dominio motorio di CENP-E e inibisce l'attività dell'ATPasi stimolata dai microtubuli di CENP-E con un Ki di 3,2 ± 0,2 nM24,25. Tuttavia, rispetto agli effetti inibitori della GSK923295 sulle cellule tumorali in coltura, gli effetti terapeutici della GSK923295 nei pazienti oncologici clinici non sono ideali48,49, il che ha anche sollevato preoccupazioni sulla specificità della GSK923295 per CENP-E. Inoltre, la specificità e gli effetti collaterali di altri inibitori di CENP-E sulle proteine CENP-E sono questioni chiave nella ricerca sul cancro.
In questo studio, abbiamo completamente eliminato il gene CENP-E nelle cellule HeLa utilizzando il sistema CRISPR/Cas9. Sono state stabilite tre strategie di screening ottimizzate basate sul fenotipo, tra cui lo screening delle colonie cellulari, i fenotipi di allineamento cromosomico e le intensità fluorescenti delle proteine CENP-E, per migliorare l'efficienza dello screening e il tasso di successo dell'editing genetico CENP-E. Inoltre, le linee cellulari knockout di CENP-E possono essere utilizzate per testare la specificità dei composti candidati per CENP-E.
Protocollo
1. Costruzione dei vettori di knockout del gene CRISPR/Cas9
- Selezionare la sequenza di DNA genomico bersaglio sul gene umano CENP-E (GenBank Accession No. NM_001286734.2) e progettare l'sgRNA utilizzando uno strumento di progettazione CRISPR online (http://crispor.tefor.net/).
- Inserire una singola sequenza genomica, selezionare il genoma di "Homo sapiens-human-UCSC Dec 2013 (hg38 analysis set) + polimorfismi a singolo nucleotide (SNPs): dbSNP148", e selezionare il motivo adiacente al protospaziatore "20 bp-NGG-spCas9". Scegliere due sequenze guida, tra cui sgRNA-1, 5'-CGGCCGCACTCGCACGCAGA-3' e sgRNA-2 5'-TTCTTTAGAGACGCGGGCTC-3', in base ai punteggi di specificità50, all'efficienza prevista10 e agli off-target minimi.
- Ordinare e sintetizzare gli oligonucleotidi di DNA a singolo filamento (ssODN). Diluire gli ssODN in ddH2O fino a una concentrazione finale di 100 μM. Per fosforilare e ricuocere gli oligo sgRNA, aggiungere 1 μL di oligo diretto, 1 μL di oligo inverso, 0,5 μL di polinucleotide chinasi T4, 1 μL di tampone polinucleotide chinasico T4 e 6,5 μL di acqua priva di RNasi in una provetta di reazione a catena della polimerasi (PCR) e incubare la provetta a 37 °C per 30 min, 95 °C per 5 min; quindi, scendere a 25 °C a 5 °C min-1.
- Digerire il plasmide pX458 utilizzando l'enzima di restrizione Bbs
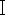 . Aggiungere 1 μg del plasmide pX458, 2 μL di tampone 10x G, 1 μL di Bbs
. Aggiungere 1 μg del plasmide pX458, 2 μL di tampone 10x G, 1 μL di Bbs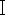 e aggiungere ddH2O privo di RNasi a 20 μL in una provetta da centrifuga da 1,5 mL. Incubare la provetta a 37 °C per 2 ore. Quindi, purificare il plasmide lineare utilizzando il kit di estrazione del gel di DNA della colonna secondo i protocolli del produttore.
e aggiungere ddH2O privo di RNasi a 20 μL in una provetta da centrifuga da 1,5 mL. Incubare la provetta a 37 °C per 2 ore. Quindi, purificare il plasmide lineare utilizzando il kit di estrazione del gel di DNA della colonna secondo i protocolli del produttore. - Legare gli oligo sgRNA nel plasmide pX458 (plasmide pSpCas9(BB)-2A-green fluorescent protein (GFP), Addgene ID. 48138). Aggiungere 1 μL di oligonucleotidi ricotti, 25 ng del plasmide lineare, 1 μL di tampone di legatura del DNA T4 10x, 0,5 μL di DNA ligasi T4 (350 U/μL) e portare a 10 μL con ddH2O in una provetta da centrifuga da 1,5 mL. Incubare la soluzione di legatura a 16 °C per 2 ore.
- Incubare 10 μL del plasmide costruito con le cellule DH5α competenti su ghiaccio per 30 minuti, shock termico a 42 °C per 45 secondi e posizionarle immediatamente su ghiaccio per 5 minuti. Aggiungere 500 μL di terreno Luria-Bertani (LB) e seminare le cellule su una piastra LB contenente ampicillina (100 μg/mL). Incubare a 37 °C per 16 ore e vagliare i cloni resistenti.
- Selezionare 5-10 colonie singole con un puntale per pipetta sterilizzato e trasferirle in una coltura da 1 mL di terreno LB con ampicillina (100 μg/mL). Incubare la coltura a 37 °C e agitarla a 180 rpm per 12-16 h.
- Isolare il DNA plasmidico utilizzando un kit di estrazione plasmidica secondo i protocolli del produttore. Validare il DNA plasmidico di ciascuna colonia mediante sequenziamento Sanger dal sito del promotore U6 utilizzando il primer U6-Fwd 5'-GAGGGCCTATTTCCCATGATTCC-3'.
- Estrarre e purificare i plasmidi utilizzando il kit di DNA plasmidico endo-free secondo i protocolli del produttore.
2. Trasfezione, isolamento e screening delle cellule HeLa knockout di CENP-E
- Colture di cellule HeLa in terreno di coltura Dulbecco Modified Eagle's Medium (DMEM) integrate con il 10% di siero fetale bovino e l'1% di penicillina/streptomicina in incubatore umidificato a 37 °C con il 5% di CO2 .
- Incubare le cellule HeLa utilizzando acido tetraacetico (EDTA) allo 0,25% di tripsina/etilendiammina a 37 °C per 2-3 minuti, dissociare le cellule pipettando delicatamente, quindi seminare le cellule in nuove piastre con un rapporto di diluizione di 1:4 per il passaggio cellulare.
- Seminare le cellule in una piastra a 12 pozzetti e coltivare al 60-70% di confluenza prima della trasfezione. Trasfettare i plasmidi pX458-sgRNA in cellule HeLa utilizzando i reagenti di riferimento secondo i protocolli del produttore (vedere la tabella dei materiali).
- Miscelare delicatamente 1 μg di plasmide pX458-sgRNA convalidato e 50 μL di terreno sierico ridotto nella provetta A. Quindi, mescolare delicatamente 2 μL del reagente di trasfezione e 50 μL di terreno sierico ridotto nella provetta B. Incubare separatamente le provette A e B a temperatura ambiente per 5 minuti.
- Mescolare delicatamente sia la provetta A che la provetta B e incubare a temperatura ambiente per 5 minuti. Aggiungere la miscela in una piastra da 12 pozzetti, incubare le cellule per 6 ore e sostituire il terreno con un terreno DMEM fresco.
- Esaminare le cellule HeLa dopo 24 o 48 ore utilizzando un microscopio a fluorescenza per verificarne l'efficienza di trasfezione. Dopo la trasfezione per 48 ore, dissociare completamente le cellule HeLa trasfettate utilizzando tripsina/EDTA allo 0,25% a 37 °C per 3-5 minuti, contare il numero di cellule utilizzando una camera di Neubauer o un contatore di cellule automatizzato e piastre le cellule in tre piastre separate da 96 pozzetti per ciascuna popolazione trasfettata secondo i metodi di diluizione seriale10.
- Rimettere le cellule in un incubatore umidificato e poi coltivarle per altre 1-2 settimane. Cambiare il terreno di coltura una volta ogni 3 giorni. Per lo screening basato sul fenotipo e la convalida del knockout di CENP-E , dissociare ed espandere le cellule in piastre da 24 o 12 pozzetti per 5-7 giorni.
- Eseguire lo screening delle cellule mutanti con diametri di colonie cellulari più piccoli utilizzando un microscopio invertito dotato di obiettivi 10x e 20x.
NOTA: Le mutazioni di CENP-E di solito provocano un aumento significativo del numero di cellule in divisione nelle colonie cellulari, che può essere uno degli indicatori chiave per lo screening delle cellule mutanti di CENP-E . - Raccogliere le cellule per l'estrazione del DNA utilizzando il kit di estrazione del DNA genomico animale su colonna secondo i protocolli del produttore. Impostare le reazioni PCR come segue: 0,25 μL di Taq polimerasi, 5 μL di tampone 10x (Mg2+ plus), 4 μL di miscele di dNTP, 500 ng di DNA stampo, 1 μL di oligo diretto, 1 μL di oligo inverso e regolare ddH2O a 50 μL. Utilizzare le seguenti impostazioni del programma PCR per l'amplificazione genica: 98 °C, 10 s; 98 °C, 10 s, 55 °C, 30 s, 72 °C, 60 s per 33 cicli; 72 °C, 10 min, quindi mantenere a 4 °C.
NOTA: I primer specifici per la clonazione del locus bersaglio sono elencati come segue: CENP-E target F1, 5'-GAGGGTCCTGGCCATTTTCCTG-3'; Obiettivo CENP-E R1, 5'-AGATCTCCGATCCTCCCCTGTC-3'; Bersaglio CENP-E F2, 5'-TGGTAACTGCATTTTGGTGTTCTAC-3'; Obiettivo CENP-E R2, 5'-CCTGTTGCAACGTGAGGGAAG-3'. - Legare il DNA bersaglio nel vettore pMD18-T secondo i protocolli del produttore. Trasfettare il plasmide legato in cellule DH5α competenti e in coltura per la selezione dei cloni.
- Eseguire il sequenziamento Sanger per determinare i tipi di knockout CENP-E . Isolare il DNA plasmidico utilizzando un kit di estrazione plasmidica secondo i protocolli del produttore. Eseguire il sequenziamento Sanger del DNA plasmidico di ciascuna colonia utilizzando i primer M13 forward e reverse. Innesco M13F, 5'-TGTAAAACGACGGCCAGT-3'; Innesco M13R, 5'-CAGGAAACAGCTATGACC-3'.
- Seminare le cellule HeLa wild-type e mutanti CENP-E su vetrini coprioggetti da 12 mm in una piastra da 24 pozzetti, rispettivamente. Rimuovere il terreno DMEM completo e fissare le cellule in una soluzione fissativa salina tamponata con paraformaldeide/fosfato al 4% (PBS) a temperatura ambiente per 10 minuti.
- Colorare i nuclei con la soluzione di 4',6-diamidino-2-fenilindolo (DAPI) a temperatura ambiente per 5 minuti. Eseguire lo screening e convalidare le cellule mutanti CENP-E in base al fenotipo di allineamento cromosomico utilizzando un microscopio a fluorescenza dotato di un obiettivo Plan Fluor 40x/Apertura numerica (NA) 0,75.
- Eseguire lo screening e convalidare le cellule knockout di CENP-E utilizzando la colorazione e l'analisi in immunofluorescenza (vedere paragrafo 3) delle proteine CENP-E.
3. Colorazione in immunofluorescenza e microscopia confocale ad alta risoluzione
- Fissare le cellule con una soluzione fissativa di paraformaldeide/PBS al 4% a temperatura ambiente per 10 minuti e immergerle per 2 x 5 minuti in 1x PBS.
NOTA: Assicurarsi che le cellule siano in buone condizioni e che le cellule siano raccolte e fissate delicatamente per evitare il distacco delle cellule in metafase. - Permeabilizzare le cellule con Triton X-100/PBS allo 0,25% a temperatura ambiente per 10 minuti e immergerle in 1x PBS per 2 x 5 minuti.
- Bloccare le celle con l'1% di BSA/PBST (0,1% Tween 20 in PBS) a temperatura ambiente per 1 ora. Diluire gli anticorpi primari con BSA/PBST all'1% e incubare i campioni a 4 °C per 12 ore.
- Scartare le soluzioni di anticorpi primari, sciacquare le cellule 3 x 5 minuti con 1x PBS e incubare le cellule con anticorpi secondari diluiti a temperatura ambiente per 2 ore.
- Eliminare gli anticorpi secondari e sciacquare le cellule in 1x PBS per 3 x 5 min.
- Colorare i nuclei con DAPI a temperatura ambiente per 5 min. Montare le guide con il mezzo di montaggio. Sigillare i vetrini con lo smalto.
- Osservare e registrare le immagini di fluorescenza utilizzando un microscopio confocale a scansione dotato di un obiettivo 63x/NA 1.40.
4. Preparazione cromosomica e analisi del cariotipo
- Coltura delle cellule wild-type e CENP-E-/- HeLa in terreno DMEM completo integrato con il 10% di siero fetale bovino e l'1% di penicillina/streptomicina per 24 ore e quindi incubare le cellule wild-type e CENP-E-/- HeLa con colchicina 300 nM per 5 ore.
- Incubare le cellule con tripsina/EDTA allo 0,25% a 37 °C per 3 minuti e raccogliere le cellule in provette da centrifuga da 1,5 mL.
- Centrifugare le cellule a 1.000 × g per 5 minuti a temperatura ambiente, scartare i surnatanti, aggiungere 1,2 mL di soluzione KCl 0,075 mol/L e incubare a 37 °C per 20 minuti.
- Centrifugare le cellule a 1.000 × g per 5 minuti a temperatura ambiente, scartare i surnatanti, aggiungere 0,2 mL della soluzione fissativa (metanolo: acido acetico glaciale = 3:1) per la prefissazione e mescolare delicatamente per 1 minuto.
- Centrifugare le cellule a 1.000 × g per 5 minuti a temperatura ambiente, raccogliere i pellet, aggiungere 1,5 mL della soluzione fissativa a temperatura ambiente per 10 minuti, quindi centrifugare a 1.000 × g per 5 minuti a temperatura ambiente.
- Scartare i surnatanti, aggiungere 0,6 mL di soluzioni fissative e preparare una sospensione cellulare. Aggiungere 3-5 gocce di sospensione cellulare da un'altezza di 35-40 cm sugli scivoli di ghiaccio.
NOTA: Mantenere l'altezza per il rilascio delle sospensioni cellulari sui vetrini a 35-40 cm; in caso contrario, i cromosomi potrebbero essere troppo dispersi o non separati l'uno dall'altro. - Asciugare immediatamente i vetrini con una lampada ad alcool, colorare i campioni con la soluzione colorante Giemsa al 10% per 7 minuti, sciacquare i vetrini con acqua corrente per 2 minuti e osservare i campioni utilizzando un microscopio ottico dotato di obiettivo Plan Fluor 40x/NA 0,75.
5. Saggio di formazione di colonie cellulari
- Preparare le sospensioni cellulari nelle piastre a 6 pozzetti con una densità cellulare di 1.000-2.000 cellule/pozzetto. Agitare delicatamente le piastre per distribuire uniformemente le cellule.
- Mettere le piastre a 6 pozzetti in un incubatore di CO2 e incubare per 2-3 settimane a 37 °C con il 5% di CO2 . Cambiare il terreno di coltura una volta ogni 5 giorni. Registra le immagini utilizzando un microscopio invertito dotato di obiettivi 10x e 20x. Raccogli le cellule quando le colonie sono visibili (centinaia di cellule all'interno di un clone).
- Per studiare gli effetti della GSK923295, coltivare le cellule nelle piastre a 6 pozzetti per 24 ore e aggiungere 2 ml di GSK923295 alle concentrazioni finali di 10, 25, 50, 100 e 200 nM.
NOTA: Non spostare la piastra di coltura nella fase iniziale della formazione del clone per evitare la perdita di cellule, che può formare nuovi cloni e influenzare lo screening delle cellule trasfettate. - Scartare il terreno di coltura e fissare le cellule con PFA all'1% a temperatura ambiente per 10 minuti. Colorare le colonie con una soluzione colorante allo 0,1% di crystal violet a temperatura ambiente per 15 min. Risciacquare i campioni con 1x PBS tre volte. Registra le immagini delle colonie cellulari e quantifica i diametri di ciascuna colonia utilizzando il software ImageJ. Scegliete lo strumento di selezione Linea retta , fate clic su Analizza, scegliete Misura e registrate la lunghezza.
- Sciacquare le colonie con 1 mL di soluzione di acido acetico al 10% per 5 min. Trasferire 200 μL delle soluzioni surnatanti in una piastra a 96 pozzetti e misurare i valori di assorbanza utilizzando uno spettrofotometro per micropiastre a A600 nm.
6. Saggio di vitalità cellulare
- Seminare le cellule nelle piastre a 24 pozzetti e coltivare le cellule per 48 ore all'80-90% di densità.
- Incubare le cellule con 100 μL di tripsina/EDTA allo 0,25% a 37 °C per 3 min.
- Miscelare 400 μL di PBS con le cellule con una pistola a pipetta. Trasferire 100 μL delle sospensioni cellulari nella piastra a 96 pozzetti.
- Aggiungere 20 μL di soluzione MTS in ciascun pozzetto con una concentrazione finale di 317 μg/mL e mescolare delicatamente secondo i protocolli del produttore. Incubare i campioni a 37 °C per 1-3 ore in un incubatore a CO2 .
- Registrare i valori di assorbanza di ciascun pozzetto al picco di assorbanza di A490 nm utilizzando uno spettrofotometro per micropiastre. Utilizzare una lunghezza d'onda di riferimento di A630 nm per sottrarre il fondo fornito dai detriti cellulari e dall'assorbanza non specifica (Vitalità cellulare = A490 nm- A630 nm).
Risultati
Le cellule CENP-E-/- HeLa sono state generate con successo utilizzando il sistema CRISPR/Cas9 (Figura 1). La tempistica e le fasi sperimentali critiche di questo metodo sono mostrate nella Figura 1. In primo luogo, abbiamo progettato e sintetizzato gli sgRNA specifici per CENP-E, ricotto e legato gli sgRNA nel plasmide pX458, trasfettato il plasmide in cellule HeLa e coltivate per 48 ore. Le cellule trasfettate sono state dissociate ...
Discussione
Kinesin-7 CENP-E è un regolatore chiave nell'allineamento cromosomico e nel checkpoint dell'assemblaggio del fuso durante la divisione cellulare 17,19,20. La delezione genetica di CENP-E di solito provoca l'attivazione del checkpoint dell'assemblaggio del fuso, l'arresto del ciclo cellulare e la morte cellulare 27,29,51,52.<...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Ringraziamo tutti i membri del Laboratorio di Citoscheletro dell'Università di Medicina del Fujian per le utili discussioni. Ringraziamo Jun-Jin Lin, Zhi-Hong Huang, Ling Lin, Li-Li Pang, Lin-Ying Zhou, Xi Lin e Min-Xia Wu del Public Technology Service Center, Fujian Medical University per la loro assistenza tecnica. Ringraziamo Si-Yi Zheng, Ying Lin e Qi Ke del Centro di Insegnamento Sperimentale di Scienze Mediche di Base presso l'Università di Medicina del Fujian per il loro supporto. Questo studio è stato sostenuto dalle seguenti sovvenzioni: National Natural Science Foundation of China (numero di sovvenzione 82001608 e 82101678), Natural Science Foundation della provincia del Fujian, Cina (numero di sovvenzione 2019J05071), i fondi congiunti per l'innovazione della scienza e della tecnologia, la provincia del Fujian, Cina (numero di sovvenzione 2021Y9160) e il progetto di finanziamento per la ricerca scientifica di talenti di alto livello della Fujian Medical University (numero di sovvenzione XRCZX2017025).
Materiali
| Name | Company | Catalog Number | Comments |
| 0.25% Trypsin-EDTA | Gibco | 25200056 | |
| 1.5 mL centrifuge tube | Axygen | MCT-150-C | |
| 24-well plate | Corning | 3524 | |
| 4S Gelred, 10,000x in water | Sangon Biotech (Shanghai) | A616697 | |
| 50 mL centrifuge tube | Corning | 430828 | |
| 6 cm Petri dish | Corning | 430166 | |
| 95% ethanol | Sinopharm Chemical Reagent | 10009164 | |
| 96-well plate | Corning | 3599 | |
| Acetic acid | Sinopharm Chemical Reagent | 10000218 | Dissolve in H2O to prepare a 10% working solution. |
| Agarose | Sangon Biotech (Shanghai) | A620014 | |
| Alexa Fluor 488-labeled Goat Anti-Mouse IgG(H+L) | Beyotime | A0428 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Alexa Fluor 488-labeled Goat Anti-Rabbit IgG(H+L) | Beyotime | A0423 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Alexa Fluor 555-labeled Donkey Anti-Mouse IgG(H+L) | Beyotime | A0460 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:500 dilution. |
| Anhydrous ethanol | Sinopharm Chemical Reagent | 100092690 | |
| Anti-BubR1 rabbit monoclonal antibody | Abcam | ab254326 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution |
| Anti-CENP-B mouse monoclonal antibody | Santa Cruz Biotechnology | sc-376392 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:50 dilution. |
| Anti-CENP-E rabbit monoclonal antibody | Abcam | ab133583 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution. |
| Anti-fade mounting medium | Beyotime | P0131 | Slowing down the quenching of fluorescent signals. |
| Anti-α-tubulin mouse monoclonal antibody | Abcam | ab7291 | For immunofluorescence. Dissolve in 1% BSA/PBST. 1:100 dilution. |
| Biotek Epoch Microplate Spectrophotometer | Biotek Instruments | Biotek Epoch | |
| Bovine Serum Albumin (BSA) | Sinopharm Chemical Reagent | 69003435 | |
| BpiI (BbsI) | Thermo Fisher Scientific | ER1011 | |
| CellTiter 96 aqueous one solution cell proliferation assay | Promega | G3580 | |
| Centrifuge | Eppendorf | 5424BK745380 | |
| Colchicine | Sinopharm Chemical Reagent | 61001563 | |
| Confocal scanning microscope | Leica | Leica TCS SP8 | |
| Coverslip | CITOTEST | 80344-1220 | |
| DAPI | Beyotime | C1006 | |
| DH5α competent cells | Sangon Biotech (Shanghai) | B528413 | |
| DL2000 DNA marker | TaKaRa | 3427A | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | C11995500BT | |
Endo-free plasmid mini kit  | Omega | D6950 | |
| Ezup Column Animal Genomic DNA Purification Kit | Sangon Biotech (Shanghai) | B518251 | |
| Fetal bovine serum | Zhejiang Tianhang Biotechnology | 11011-8611 | |
| Gentian violet | Sinopharm Chemical Reagent | 71019944 | Dissolve in PBS to prepare 0.1% gentian violet/PBS. |
| Giemsa staining solution | Sinopharm Chemical Reagent | 71020260 | |
| GraphPad Prism version 8.0 software | GraphPad | www.graphpad.com | Statistical analysis. |
| GSK923295 | MedChemExpress | HY-10299 | |
| HeLa cell line | ATCC | CCL-2 | |
| Humidified incubator | Heal Force | HF90/HF240 | |
| Image J software | National Institutes of Health | https://imagej.nih.gov/ij/ | Image processing and analysis. |
| Inverted microscope | Nanjing Jiangnan Novel Optics | XD-202 | |
| LB agar powder | Sangon Biotech (Shanghai) | A507003 | |
| Lipo6000 transfection reagent | Beyotime | C0526 | |
| Nikon Ti-S2 microscope | Nikon | Ti-S2 | |
| Opti-MEM reduced serum medium | Gibco | 31985070 | |
| Paraformaldehyde | Sinopharm Chemical Reagent | 80096618 | Dissolve in PBS to prepare 4% paraformaldehyde/PBS. |
| Penicillin-streptomycin solution | HyClone | SV30010 | |
| SanPrep column DNA gel extraction kit | Sangon Biotech (Shanghai) | B518131 | |
| SanPrep column plasmid mini-preps kit | Sangon Biotech (Shanghai) | B518191 | |
| T4 DNA ligase | TaKaRa | 2011A | |
| T4 polynucleotide kinase | TaKaRa | 2021A | |
| TaKaRa Ex Taq | TaKaRa | RR001A | |
| Triton X-100 | Sinopharm Chemical Reagent | 30188928 | Dissolve in PBS to prepare 0.25% Triton X-100/PBS. |
| Tween 20 | Sinopharm Chemical Reagent | 30189328 | Dissolve in PBS to prepare 0.1% Tween 20/PBS. |
Riferimenti
- Jiang, W., Bikard, D., Cox, D., Zhang, F., Marraffini, L. A. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology. 31 (3), 233-239 (2013).
- Bibikova, M., Beumer, K., Trautman, J. K., Carroll, D. Enhancing gene targeting with designed zinc finger nucleases. Science. 300 (5620), 764 (2003).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nature Reviews Genetics. 11 (9), 636-646 (2010).
- Joung, J. K., Sander, J. D. TALENs: a widely applicable technology for targeted genome editing. Nature Reviews Molecular Cell Biology. 14 (1), 49-55 (2013).
- Wood, A. J., et al. Targeted genome editing across species using ZFNs and TALENs. Science. 333 (6040), 307 (2011).
- Stoddard, B. L. Homing endonuclease structure and function. Quarterly Reviews of Biophysics. 38 (1), 49-95 (2005).
- Pâques, F., Duchateau, P. Meganucleases and DNA double-strand break-induced recombination: perspectives for gene therapy. Current Gene Therapy. 7 (1), 49-66 (2007).
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Shmakov, S., et al. Discovery and functional characterization of diverse class 2 CRISPR-Cas systems. Molecular Cell. 60 (3), 385-397 (2015).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nature Protocols. 8 (11), 2281-2308 (2013).
- Jia, N., Patel, D. J. Structure-based functional mechanisms and biotechnology applications of anti-CRISPR proteins. Nature Reviews Molecular Cell Biology. 22 (8), 563-579 (2021).
- Hsu, P. D., Lander, E. S., Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell. 157 (6), 1262-1278 (2014).
- Horvath, P., Barrangou, R. CRISPR/Cas, the immune system of bacteria and archaea. Science. 327 (5962), 167-170 (2010).
- Gasiunas, G., Barrangou, R., Horvath, P., Siksnys, V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proceedings of the National Academy of Sciences of the United States of America. 109 (39), E2579-E2586 (2012).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jackson, S. A., et al. CRISPR-Cas: Adapting to change. Science. 356 (6333), eaal5056 (2017).
- Wood, K. W., Sakowicz, R., Goldstein, L. S., Cleveland, D. W. CENP-E is a plus end-directed kinetochore motor required for metaphase chromosome alignment. Cell. 91 (3), 357-366 (1997).
- Craske, B., Welburn, J. P. I. Leaving no-one behind: how CENP-E facilitates chromosome alignment. Essays in Biochemistry. 64 (2), 313-324 (2020).
- Yu, K. W., Zhong, N., Xiao, Y., She, Z. Y. Mechanisms of kinesin-7 CENP-E in kinetochore-microtubule capture and chromosome alignment during cell division. Biology of the Cell. 111 (6), 143-160 (2019).
- Schaar, B. T., Chan, G. K., Maddox, P., Salmon, E. D., Yen, T. J. CENP-E function at kinetochores is essential for chromosome alignment. Journal of Cell Biology. 139 (6), 1373-1382 (1997).
- McEwen, B. F., et al. CENP-E is essential for reliable bioriented spindle attachment, but chromosome alignment can be achieved via redundant mechanisms in mammalian cells. Molecular Biology of the Cell. 12 (9), 2776-2789 (2001).
- Yao, X., Abrieu, A., Zheng, Y., Sullivan, K. F., Cleveland, D. W. CENP-E forms a link between attachment of spindle microtubules to kinetochores and the mitotic checkpoint. Nature Cell Biology. 2 (8), 484-491 (2000).
- Kim, Y., Heuser, J. E., Waterman, C. M., Cleveland, D. W. CENP-E combines a slow, processive motor and a flexible coiled coil to produce an essential motile kinetochore tether. Journal of Cell Biology. 181 (3), 411-419 (2008).
- Qian, X., et al. Discovery of the first potent and selective inhibitor of Centromere-Associated Protein E: GSK923295. ACS Medicinal Chemistry Letters. 1 (1), 30-34 (2010).
- Wood, K. W., et al. Antitumor activity of an allosteric inhibitor of centromere-associated protein-E. Proceedings of the National Academy of Sciences of the United States of America. 107 (13), 5839-5844 (2010).
- Pisa, R., Phua, D. Y. Z., Kapoor, T. M. Distinct mechanisms of resistance to a CENP-E inhibitor emerge in near-haploid and diploid cancer cells. Cell Chemical Biology. 27 (7), 850.e6-857.e6 (2020).
- Putkey, F. R., et al. Unstable kinetochore-microtubule capture and chromosomal instability following deletion of CENP-E. Developmental Cell. 3 (3), 351-365 (2002).
- Weaver, B. A., Silk, A. D., Montagna, C., Verdier-Pinard, P., Cleveland, D. W. Aneuploidy acts both oncogenically and as a tumor suppressor. Cancer Cell. 11 (1), 25-36 (2007).
- Silk, A. D., et al. Chromosome missegregation rate predicts whether aneuploidy will promote or suppress tumors. Proceedings of the National Academy of Sciences of the United States of America. 110 (44), E4134-E4141 (2013).
- Guo, Y., Kim, C., Ahmad, S., Zhang, J., Mao, Y. CENP-E-dependent BubR1 autophosphorylation enhances chromosome alignment and the mitotic checkpoint. Journal of Cell Biology. 198 (2), 205-217 (2012).
- Weaver, B. A., Cleveland, D. W. Aneuploidy: instigator and inhibitor of tumorigenesis. Cancer Research. 67 (21), 10103-10105 (2007).
- Nishimura, K., Fukagawa, T. An efficient method to generate conditional knockout cell lines for essential genes by combination of auxin-inducible degron tag and CRISPR/Cas9. Chromosome Research. 25 (3-4), 253-260 (2017).
- Owa, M., Dynlacht, B. A non-canonical function for Centromere-associated protein-E controls centrosome integrity and orientation of cell division. Communications Biology. 4 (1), 358 (2021).
- Barabási, A. L., Oltvai, Z. N. Network biology: understanding the cell's functional organization. Nature Reviews Genetics. 5 (2), 101-113 (2004).
- Rossi, A., et al. Genetic compensation induced by deleterious mutations but not gene knockdowns. Nature. 524 (7564), 230-233 (2015).
- Ma, Z., et al. PTC-bearing mRNA elicits a genetic compensation response via Upf3a and COMPASS components. Nature. 568 (7751), 259-263 (2019).
- El-Brolosy, M. A., et al. Genetic compensation triggered by mutant mRNA degradation. Nature. 568 (7751), 193-197 (2019).
- Kung, P. P., et al. Chemogenetic evaluation of the mitotic kinesin CENP-E reveals a critical role in triple-negative breast cancer. Molecular Cancer Therapeutics. 13 (8), 2104-2115 (2014).
- Kim, J. H., et al. Development of a novel HAC-based "gain of signal" quantitative assay for measuring chromosome instability (CIN) in cancer cells. Oncotarget. 7 (12), 14841-14856 (2016).
- Hirayama, T., et al. Synthetic studies of centromere-associated protein-E (CENP-E) inhibitors: 1. Exploration of fused bicyclic core scaffolds using electrostatic potential map. Bioorganic & Medicinal Chemistry. 21 (17), 5488-5502 (2013).
- Hirayama, T., et al. Synthetic studies on Centromere-associated protein-E (CENP-E) inhibitors: 2. Application of electrostatic potential map (EPM) and structure-based modeling to Imidazo[1,2-a]pyridine derivatives as anti-tumor agents. Journal of Medicinal Chemistry. 58 (20), 8036-8053 (2015).
- Ohashi, A., et al. A novel time-dependent CENP-E inhibitor with potent antitumor activity. PLoS One. 10 (12), e0144675 (2015).
- Ohashi, A., et al. Aneuploidy generates proteotoxic stress and DNA damage concurrently with p53-mediated post-mitotic apoptosis in SAC-impaired cells. Nature Communications. 6, 7668 (2015).
- Ding, X., et al. Probing CENP-E function in chromosome dynamics using small molecule inhibitor syntelin. Cell Research. 20 (12), 1386-1389 (2010).
- Liu, X., et al. Phase separation drives decision making in cell division. Journal of Biological Chemistry. 295 (39), 13419-13431 (2020).
- Henderson, M. C., et al. UA62784, a novel inhibitor of centromere protein E kinesin-like protein. Molecular Cancer Therapeutics. 8 (1), 36-44 (2009).
- Yamane, M., et al. Identification of benzo[d]pyrrolo[2,1-b]thiazole derivatives as CENP-E inhibitors. Biochemical and biophysical research communications. 519 (3), 505-511 (2019).
- Lock, R. B., et al. Initial testing of the CENP-E inhibitor GSK923295A by the pediatric preclinical testing program. Pediatric Blood & Cancer. 58 (6), 916-923 (2012).
- Chung, V., et al. First-time-in-human study of GSK923295, a novel antimitotic inhibitor of centromere-associated protein E (CENP-E), in patients with refractory cancer. Cancer Chemotherapy and Pharmacology. 69 (3), 733-741 (2012).
- Hsu, P. D., et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nature biotechnology. 31 (9), 827-832 (2013).
- Weaver, B. A., Cleveland, D. W. Decoding the links between mitosis, cancer, and chemotherapy: The mitotic checkpoint, adaptation, and cell death. Cancer Cell. 8 (1), 7-12 (2005).
- Manchado, E., Guillamot, M., Malumbres, M. Killing cells by targeting mitosis. Cell Death and Differentiation. 19 (3), 369-377 (2012).
- Dang, Y., et al. Optimizing sgRNA structure to improve CRISPR-Cas9 knockout efficiency. Genome Biology. 16, 280 (2015).
- Ratz, M., Testa, I., Hell, S. W., Jakobs, S. CRISPR/Cas9-mediated endogenous protein tagging for RESOLFT super-resolution microscopy of living human cells. Scientific Reports. 5, 9592 (2015).
- Koch, B., et al. Generation and validation of homozygous fluorescent knock-in cells using CRISPR-Cas9 genome editing. Nature Protocols. 13 (6), 1465-1487 (2018).
- Yan, Q., et al. Multiplex CRISPR/Cas9-based genome engineering enhanced by Drosha-mediated sgRNA-shRNA structure. Scientific Reports. 6, 38970 (2016).
- Cheng, Y., et al. CRISPR/Cas9-mediated chicken TBK1 gene knockout and its essential role in STING-mediated IFN-β induction in chicken cells. Frontiers in Immunology. 9, 3010 (2019).
- Zhou, Y., et al. High-throughput screening of a CRISPR/Cas9 library for functional genomics in human cells. Nature. 509 (7501), 487-491 (2014).
- Zeng, W., Guo, L., Xu, S., Chen, J., Zhou, J. High-throughput screening technology in industrial biotechnology. Trends in Biotechnology. 38 (8), 888-906 (2020).
- Tsai, S. Q., et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nature Biotechnology. 33 (2), 187-197 (2015).
- Kim, D., et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nature Biotechnology. 34 (8), 863-868 (2016).
- Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A., Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature. 533 (7603), 420-424 (2016).
- Nishimasu, H., et al. Engineered CRISPR-Cas9 nuclease with expanded targeting space. Science. 361 (6408), 1259-1262 (2018).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Ablain, J., Durand, E. M., Yang, S., Zhou, Y., Zon, L. I. A CRISPR/Cas9 vector system for tissue-specific gene disruption in zebrafish. Developmental Cell. 32 (6), 756-764 (2015).
- Pickar-Oliver, A., Gersbach, C. A. The next generation of CRISPR-Cas technologies and applications. Nature Reviews Molecular Cell Biology. 20 (8), 490-507 (2019).
- Nalawansha, D. A., Gomes, I. D., Wambua, M. K., Pflum, M. K. H. HDAC inhibitor-induced mitotic arrest is mediated by Eg5/KIF11 acetylation. Cell Chemical Biology. 24 (4), 481.e5-492.e5 (2017).
- Kavalapure, R. S., et al. Design, synthesis, and molecular docking study of some 2-((7-chloroquinolin-4-yl) amino) benzohydrazide Schiff bases as potential Eg5 inhibitory agents. Bioorganic Chemistry. 116, 105381 (2021).
- Calligaris, D., Lafitte, D. Chemical inhibitors: the challenge of finding the right target. Chemistry & Biology. 18 (5), 555-557 (2011).
- Łomzik, M., et al. Metal-dependent cytotoxic and kinesin spindle protein inhibitory activity of Ru, Os, Rh, and Ir half-sandwich complexes of Ispinesib-derived ligands. Inorganic Chemistry. 59 (20), 14879-14890 (2020).
- Ferro, L. S., et al. Structural and functional insight into regulation of kinesin-1 by microtubule-associated protein MAP7. Science (New York, N.Y.). 375 (6578), 326-331 (2022).
- Atherton, J., et al. The mechanism of kinesin inhibition by kinesin-binding protein. eLife. 9, e61481 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon