Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Interaction entre le bactériophage T4 et E . coli dans l’intestin murin : un modèle prototypique pour l’étude de la dynamique hôte-bactériophage in vivo
Dans cet article
Résumé
Les bactériophages (phages), des virus qui infectent les bactéries, font partie intégrante du microbiome intestinal. Bien que ces habitants symbiotiques déterminent la forme physique bactérienne et la dynamique des populations, on comprend peu de choses sur leur impact sur l’homéostasie intestinale et les maladies. Ce protocole étudie des phages T4 isolés dans un modèle murin, adaptable à d’autres paires phage-bactérie.
Résumé
Les bactériophages (phages) sont des virus qui infectent les bactéries avec une spécificité au niveau de l’espèce et de la souche et sont les entités biologiques les plus abondantes dans tous les écosystèmes connus. Au sein des communautés bactériennes, telles que celles que l’on trouve dans le microbiote intestinal, les phages sont impliqués dans la régulation de la dynamique des populations de microbiote et dans l’évolution bactérienne. La recherche sur les phages a connu un regain d’intérêt au cours de la dernière décennie, en partie en raison des capacités de destruction spécifiques à l’hôte des phages lytiques, qui offrent un outil prometteur pour contrer la menace croissante des bactéries résistantes aux antimicrobiens. De plus, des études récentes démontrant que les phages adhèrent au mucus intestinal suggèrent qu’ils pourraient avoir un rôle protecteur dans la prévention de l’invasion bactérienne dans l’épithélium sous-jacent. Il est important de noter que, comme les microbiomes bactériens, les phagéomes perturbés ont été associés à une aggravation des résultats dans des maladies telles que les maladies inflammatoires de l’intestin. Des études antérieures ont démontré que les phages peuvent moduler le microbiome des animaux et des humains grâce à des greffes de filtrat fécal, ce qui est bénéfique pour la santé de l’hôte. Cette récente vague de recherche s’accompagne de la nécessité d’établir et de normaliser des protocoles pour étudier les phages dans le contexte du microbiome intestinal. Ce protocole fournit un ensemble de procédures pour étudier les phages T4 isolés et leur hôte bactérien, Escherichia coli, dans le contexte du tractus gastro-intestinal murin. Les méthodes décrites ici décrivent comment partir d’un lysat de phage, l’administrer à des souris et évaluer les effets sur l’hôte bactérien et les niveaux de phages. Ce protocole peut être modifié et appliqué à d’autres paires phage-bactérie et fournit un point de départ pour étudier la dynamique hôte-phage in vivo.
Introduction
Les bactériophages, ou phages, sont des virus qui infectent et tuent les bactéries avec une spécificité au niveau de l’espèce et de la souche1. Les phages jouent un rôle important dans les communautés bactériennes complexes telles que le microbiote intestinal, où ils ont été impliqués dans la régulation de la dynamique des populations et la conduite de la fitness bactérienne2. Au cours de la dernière décennie, il y a eu un regain d’intérêt pour la recherche sur les phages en raison de l’augmentation des agents pathogènes résistants aux antimicrobiens3 et du potentiel de la phagothérapie comme stratégie de traitement alternative. Ces dernières années, les cocktails de phages lytiques ont été utilisés par voie intraveineuse avec un certain succès dans les infections septiques bactériennes graves et résistantes aux antibiotiques chez l’homme 3,4. La phagothérapie orale a également été proposée comme alternative potentielle aux antibiotiques pour traiter les infections intestinales et l’inflammation. De plus, les phages ont été impliqués dans le succès des greffes de filtrat fécal (FFT), qui sont des préparations de microbiote fécal filtrées pour éliminer les bactéries, dans le traitement de l’infection récurrente à Clostridioides difficile (ICDr)5,6, des troubles inflammatoires de l’intestin (MICI)7,8 et de l’entérocolite nécrosante chez les porcs prématurés9. Compte tenu de ces résultats, il est important de prendre en compte les interactions entre les phages et le microbiote intestinal, et entre les phages et l’hôte mammifère, car l’ajout de nouveaux phages dans une communauté préexistante peut avoir des effets indirects sur la communauté dans son ensemble, et pas seulement sur ses bactéries cibles 2,10.
L’étude in vitro des interactions des phages avec leurs bactéries cibles s’est avérée utile pour comprendre les mécanismes et les impacts des interactions phages et bactéries dans l’intestin. Dans ce contexte, il a été démontré que les phages T4 spécifiques d’Escherichia coli de l’ordre des Caudovirales ont besoin de domaines de type immunoglobuline (Ig) situés dans des protéines de capside externe (Hoc) hautement antigéniques à la surface du virion pour adhérer au mucus intestinal11. De plus, des tests transwell ont montré que les phages T4 sont capables d’interagir avec les cultures de cellules épithéliales et de se déplacer à travers les couches cellulaires par macropinocytose12,13. Ces résultats soutiennent l’hypothèse selon laquelle les phages peuvent interagir avec leur hôte métazoaire, même s’ils sont incapables d’infecter les cellules eucaryotes. Ces modèles, bien qu’utiles, ne disposent pas de toute la gamme d’interactions complexes qui se produisent dans un écosystème intestinal et qui sont nécessaires à une exploration complète de l’interaction tripartite entre les phages, les bactéries et l’hôte métazoaire.
Les modèles murins sont un outil important pour étudier les phages dans des environnements complexes. Une application souhaitable de l’administration de phages est une stratégie alternative pour traiter les infections résistantes aux antimicrobiens ou les agents pathogènes associés aux maladies inflammatoires chroniques, y compris les MICI. Cependant, la littérature émergente suggère que le comportement des phages in vitro ne représente pas pleinement les fonctions in vivo. Buttimer et al.14 ont démontré qu’un cocktail de phages était capable d’épuiser les bactéries ciblées dans un consortium simplifié de microbiote humain in vitro, mais ne pouvait pas être répliqué in vivo chez des souris gnotobiotiques colonisées par le même consortium bactéries-phages. De plus, dans un microbiome de souris conventionnel, le phage T7 a entraîné un épuisement sélectif de ses bactéries intestinales cibles, bien qu’une récupération progressive ait été observée au fil du temps, indiquant une résistance évoluée15. D’autres études ont démontré la coexistence de phages administrés par voie orale et de leurs souches bactériennes cibles in vivo 2,16. En effet, au-delà de la coexistence phage/bactérie, l’administration de phages a entraîné des changements généralisés dans la composition et la fonction globales de la communauté du microbiote 2,16. Ceci est pertinent dans les contextes pathologiques, car plusieurs études ont trouvé des associations entre l’augmentation de l’abondance relative des Caudovirales et des MICI 7,8,17 qui étaient indépendantes des changements dans l’abondance bactérienne7. On ne sait pas encore s’il s’agit d’un facteur ou d’une conséquence de la pathogenèse de la maladie.
L’objectif historique de l’étude des phages a été la relation entre un phage et sa bactérie cible. Cependant, il est également important de prendre en compte les interactions potentielles entre le phage et la muqueuse, l’épithélium et le système immunitaire de l’hôte métazoaire. Ces interactions jouent toutes un rôle important dans la réponse globale à l’infection intestinale par les phages. Pour le démontrer, des phages ont été étudiés sur des souris sans germes (GF) afin d’élucider leur impact sur le système immunitaire sans interférence du microbiote8. Dans ce système, les acides nucléiques des phages ont été détectés par des récepteurs de type Toll (TLR) situés dans les endosomes des cellules immunitaires phagocytaires (macrophages et cellules dendritiques). Cela a activé la signalisation en aval et stimulé la production d’interféron (IFN)-γ8 ou d’IFNs de type I18 dépendant des lymphocytes T. De plus, Fluckiger et al.19 ont impliqué les lymphocytes T CD8+ mémoires dans la reconnaissance des antigènes codés par des phages (prophages), ce qui a entraîné une réactivité croisée des lymphocytes T avec les antigènes tumoraux, ce qui a entraîné une réduction de la charge tumorale. Enfin, la production d’anticorps spécifiques aux phages a été documentée dans des études sur des souris où des phages ont été délivrés à des modèles animaux de manière continue par l’eau potable 8,20, ou par gavage oral répété pendant plusieurs mois20, démontrant la capacité des protéines des phages à promouvoir des réponses immunitaires humorales. Bien que ces modes d’inoculation des phages permettent un amorçage optimal et continu du système immunitaire, ils peuvent ne pas représenter les interactions naturelles entre les phages et l’environnement intestinal, ni la cinétique de la phagothérapie appliquée par voie orale. Jusqu’à présent, un nombre limité d’études ont examiné les interactions d’un phage avec une seule espèce bactérienne dans des modèles murins monocolonisés21. Cependant, les souris monocolonisées se sont avérées essentielles pour déchiffrer les effets spécifiques des microbes des espèces individuelles sur le tractus gastro-intestinal (GI) et le développement immunitaire 22,23,24, et elles peuvent encore s’avérer utiles pour comprendre les interactions tripartites entre les phages, leurs bactéries cibles et l’hôte métazoaire.
Il est passionnant de constater qu’il reste encore beaucoup à apprendre sur les interactions entre les phages intestinaux et les bactéries commensales intestinales, ainsi que sur les interactions qui se produisent entre l’hôte métazoaire et les phages qui y résident. Ce protocole fournit un ensemble de procédures pour étudier le phage T4 isolé et son homologue bactérien, E. coli (K-12, BW25113), à l’aide d’un modèle murin gnotobiotique. Ces procédures standardisées fournissent également une base pour optimiser d’autres dyades phage/bactérie en adaptant les paramètres de croissance aux paires d’intérêt. Les méthodes décrites ici décrivent : (1) la préparation de lysats de phages et de véhicules T4 pour le gavage oral de souris ; (2) Administration orale de phage T4 à des souris gnotobiotiques monocolonisées par E. coli ; (3) Surveillance des niveaux de phages T4 dans les excréments et les tissus de souris au fil du temps.
Pour les résultats représentatifs présentés ici, des lysats de phages T4 purifiés ont été propagés à partir de stocks de banques de phages maintenus par le laboratoire Rohwer. La méthode Phage-on-Tap pour la propagation du phage T4 a été adaptée25, comme indiqué dans ce protocole. La méthode permet d’obtenir des stocks de phages à titre élevé et à faible teneur en endotoxines en trois jours. En utilisant cette approche, 10 mL de ≥ 1010 unités formant des plaques (UFP)/mL de phage T4 avec < 0,5 unité d’endotoxine (UE)/mL ont été collectés régulièrement. Les teneurs en endotoxines recommandées pour l’administration orale ou intraveineuse chez la souris sont respectivement de ≤ 20 UE/ml et ≤ de 5 UE/kg/h (ou 0,1 UE administrée en 1 heure pour une souris de 20 g), ce qui en fait une méthode appropriée de préparation de phages pour l’inoculation in vivo . Tous les stocks de phages ont été stockés à 4 °C dans un tampon de magnésium salin (SM) (recette fournie à l’étape 1.1.5.1). E. coli a été cultivé dans des milieux LB. Pour diverses paires phage-bactérie, divers milieux de culture et conditions de croissance peuvent être adaptés à partir de ce protocole. Les phages peuvent également provenir de l’environnement, comme les eaux usées, l’eau de mer, le sol et le contenu intestinal, et peuvent être isolés et purifiés selon Sambrook et Russell26 avant la préparation en utilisant les conditions de croissance et de propagation appropriées pour chaque paire phage-hôte d’intérêt25. Les phages peuvent également être obtenus auprès de sources commerciales (voir le tableau des matériaux) ou de banques de phages.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les expériences ont été menées conformément aux lignes directrices établies par les protocoles approuvés par le Comité de protection des animaux et le Comité de biosécurité de l’Université de la Colombie-Britannique (A23-0113, B19-0038). Les souris ont été hébergées à l’Université de la Colombie-Britannique dans des conditions exemptes d’agents pathogènes au Center for Disease Modelling. Les souris C57BL/6 ont été élevées dans l’installation dans un isolateur à film flexible stérile, fourni avec un régime stérile pour souris, de l’eau, de la litière et du matériel de nidification. Les souris ont été maintenues sur un cycle jour/nuit de 12 heures. Les souris expérimentales, mâles et femelles, ont été appariées selon l’âge dans chaque expérience, allant de 6 à 12 semaines et pesant de 15 à 30 g pour toutes les expériences.
1. Préparation de lysats de phages et de véhicules pour gavage oral chez la souris
- Purification des phages et génération de stock
REMARQUE : Dans cette étude, les phages T4 sont cultivés et titrés en plaquant sur une pelouse de bactéries en utilisant la méthode de la gélose unique, décrite ci-dessous. La méthode de la double gélose a été décrite précédemment25,27et peut également être utilisé avec une efficacité similaire. La méthode de la gélose à couche unique a été choisie pour ce protocole car elle offre une meilleure visibilité de la plaque28.- Cultiver E. coli dans 5 mL de milieu stérile LB dans des tubes de culture stériles en polystyrène ou en verre (avec bouchons). Incuber les tubes à 37 °C en agitant à 200 tr/min pendant la nuit, jusqu’à ce que la phase stationnaire soit atteinte.
- Préparez la gélose molle dans un flacon en verre en autocavant LB avec 0,5 % de gélose et en refroidissant à 50 °C ou moins (tout en restant liquide) avant la supplémentation. Préparez suffisamment de gélose molle pour 15 ml par assiette.
- Compléter la gélose molle avec du MgSO4 et du CaCl2 jusqu’à une concentration finale de 1 mM pour chacune. Ajouter 100 μL de culture d’E. coli pendant la nuit pour chaque 3 mL de gélose molle. Remuez doucement à l’aide d’une barre d’agitation magnétique pour homogénéiser les suppléments et la culture dans la gélose molle.
REMARQUE : L’uniformité du volume et de la densité d’E. coli qui est ajouté à la préparation de gélose molle est essentielle (décrite dans la section des résultats représentatifs). - Ajouter 15 mL de gélose molle + E. coli par boîte de Pétri (15 cm de diamètre) à l’aide d’une pipette sérologique. Laissez les assiettes prendre à température ambiante pour une utilisation le jour même.
REMARQUE : Les assiettes prendront environ 20 minutes avec les couvercles ouverts. La gélose restera molle mais ne se déplacera pas lorsque les plaques sont inversées. - Préparez 8 à 10 dilutions en série du phage T4 source dans des facteurs de 10 en diluant dans un tampon SM. Placer 5 μL de chaque dilution sur des plaques. Laisser sécher, retourner et incuber les plaques à 37 °C pendant la nuit.
- Pour préparer un tampon de phage salin de magnésium (SM), dans 1 L de H2O désionisé, dissoudre 100 mM de NaCl, 8 mM de MgSO4·7H2O et 50 mM de Tris-HCl (pH 7,4). Autoclave et conserve à température ambiante.
REMARQUE : Le lendemain, E. coli devrait avoir poussé dans une pelouse tout au long de la gélose LB molle. Des plaques, ou zones de croissance dégagées visibles dans la pelouse d’E. coli , apparaîtront là où les hôtes infectés ont tué les E . coli par les phages. Chaque plaque représente une unité de formation de plaque (pfu).
- Pour préparer un tampon de phage salin de magnésium (SM), dans 1 L de H2O désionisé, dissoudre 100 mM de NaCl, 8 mM de MgSO4·7H2O et 50 mM de Tris-HCl (pH 7,4). Autoclave et conserve à température ambiante.
- Prélevez une seule plaque de la plaque de gélose souple en poussant une pointe de pipette stérile au centre de la plaque, créant un bouchon de gélose à l’extrémité de la pointe. Remettre le bouchon de la plaque en suspension dans 1 mL de tampon SM dans un tube de microcentrifugation de 1,7 mL. Agiter le tube à vitesse maximale pendant 1 min pour mélanger.
- Centrifuger le tube à 4000 x g pendant 5 min à température ambiante pour éliminer les débris du lysat. Transférez le surnageant dans un nouveau tube de microcentrifugation.
- Pour propager le phage isolé, répétez les étapes 1.1.1 à 1.1.3. Ajouter 100 μL du phage isolé à une aliquote de 15 mL de gélose molle contenant E. coli dans un tube conique. Retournez le tube trois fois pour mélanger et versez la gélose dans une boîte de Pétri.
- Laisser sécher les assiettes avant de les retourner et de les incuber toute la nuit à 37 °C. Préparez une plaque de contrôle de pelouse sans phage pour confirmer la viabilité d’E. coli .
REMARQUE : Après une nuit d’incubation, la plaque témoin doit avoir une pelouse d’E. coli tandis que la plaque contenant des phages doit être complètement lysée. - Pour extraire le phage de la plaque, ajouter 5 mL de tampon SM à la surface de la plaque débarrassée des phages et agiter à 70 tr/min sur une bascule pendant 15 min à température ambiante.
- Recueillir le tampon de la plaque dans un tube à centrifuger conique de 50 mL et centrifuger à 4000 x g pendant 5 min à température ambiante pour granuler les débris de la plaque de gélose.
- Prélever le lysat stock de phages T4 dans un nouveau tube et le conserver à 4 °C jusqu’au titrage et à la propagation.
REMARQUE : Les phages T4 sont stables pendant plus de 10 ans25 ; cependant, la stabilité dépendra du type de phage et des conditions d’entreposage. Le titre de phage varie également avec le temps de stockage. Les phages sont sensibles au gel. Par conséquent, la cryoconservation est recommandée dans l’azote liquide et en évitant les cycles de gel-dégel car cela peut endommager les phages, réduisant ainsi le titrede phage 25. - Titrer le lysat de stock de phages T4 pour déterminer la concentration en pfu/mL, comme décrit précédemment 25,29,30. Un stock de phages T4 à titre élevé contiendra 10à 8 UFP/ml ou plus.
- Préparation de lysats de phages expérimentaux
- Culture E . coli dans 5 mL de milieu LB dans des tubes de culture stériles en polystyrène ou en verre (avec bouchons). Incuber les tubes pendant la nuit à 37 °C en agitant à 200 tr/min, jusqu’à ce que la phase stationnaire soit atteinte.
- Sous-culture : culture d’E. coli pendant la nuit 1:50 dans un milieu de 100 mL LB dans une fiole conique en verre. Incuber à 37 °C en agitant à 200 tr/min jusqu’à ce que les bactéries aient atteint la phase exponentielle précoce à moyenne (environ 1,5 h).
REMARQUE : Une courbe de croissance initiale peut être effectuée pour déterminer la densité optique à 600 nm (OD600) à laquelle la culture bactérienne est en phase exponentielle. La culture dans un tube conique en verre est suggérée, car des preuves récentes ont montré que les phages sont capables d’adhérer à des plastiques tels que le polypropylène31. - Ajouter 100 μL du stock de phages T4 à titre élevé de l’étape 1.1 dans la sous-culture d’E. coli et incuber à 37 °C en agitant pendant 3 h, ou jusqu’à ce que le nouveau lysat ne soit plus trouble. Prélever le lysat de phage T4 dans des tubes coniques de 50 mL et passer directement aux étapes de nettoyage ou, si nécessaire, conserver à 4 °C jusqu’au lendemain.
REMARQUE : Une plus grande taille de burst de phage (indiquant un nombre moyen plus élevé de virions libérés par cycle de réplication) nécessite une période d’incubation plus longue pour la sous-culture bactérienne à l’étape 1.2.2. Cela permet d’augmenter la densité bactérienne avant de se doper des stocks de phages. - Centrifuger les lysats à 4000 x g pendant 20 min à température ambiante pour granuler les bactéries et les débris cellulaires restants. Stériliser le surnageant obtenu à l’aide d’un filtre en nylon de 0,22 μm et transférer le filtrat dans de nouveaux tubes à centrifuger coniques de 50 mL.
- Ajoutez 0,1 volume de chloroforme à chaque volume de lysat de phage T4 filtré pour tuer les bactéries restantes et empêcher la croissance bactérienne. Agiter brièvement pour mélanger et incuber à température ambiante pendant 10 min.
REMARQUE : Les phages enveloppés de lipides sont sensibles au chloroforme, ce qui peut réduire le titrede 25 des phages. Ignorez cette étape si nécessaire.
ATTENTION : Le chloroforme est un solvant organique toxique qui est dangereux lorsqu’il est inhalé, ingéré ou absorbé par la peau. Utilisez une hotte et un équipement de protection individuelle (EPI) approprié lorsque vous travaillez avec du chloroforme. Utilisez des pipettes sérologiques en verre plutôt qu’en polystyrène lorsque vous travaillez avec du chloroforme, car il n’est pas compatible avec la plupart des plastiques. Le chloroforme peut être placé dans des tubes à centrifuger coniques en polypropylène pour les étapes 1.2.5 à 1.2.6, mais ne doit pas être stocké à long terme dans des tubes en plastique. - Centrifuger le lysat à 4000 x g pendant 5 min à température ambiante pour séparer le chloroforme du lysat. À l’aide d’une pipette sérologique, transférez soigneusement la couche supérieure de lysat dans un nouveau tube conique de 50 mL sans perturber la couche de chloroforme sous-jacente. Jeter les déchets de chloroforme dans le contenant approprié pour les déchets liquides dangereux. Conserver les lysats à 4 °C jusqu’à concentration et échange de tampon le lendemain.
- Concentrer les lysats de phages dans un dispositif de filtration centrifuge de 100 kDa (voir le tableau des matériaux) en ajoutant 13 mL de lysat de phages dans le réservoir supérieur de l’appareil et en centrifugeant à 4000 x g pendant 5 minutes, ou jusqu’à ce que la majeure partie du lysat ait traversé le filtre dans le réservoir inférieur.
REMARQUE : Essayez d’avoir environ 2 ml de lysat à la fin du temps d’essorage. À mesure que la concentration de phages augmente dans le filtre avec l’ajout de lysat, les temps d’essorage augmenteront probablement. Les dispositifs de filtration centrifuge ont une butée physique pour les empêcher de tourner à sec32. Ne laissez pas la membrane filtrante sécher si une utilisation continue est prévue (c’est-à-dire en retirant tout le liquide du réservoir supérieur). Les temps d’essorage peuvent varier en fonction du type de phage et du titre. - À l’aide d’une pipette P200 ou P1000, pipeter doucement les ~2 mL de lysat restants de haut en bas dans le réservoir supérieur pour déboucher la membrane filtrante après chaque essorage. Jeter le filtrat du réservoir inférieur dans un récipient à déchets, en laissant le phage concentré dans le réservoir supérieur.
- Répéter les étapes 1.2.7 à 1.2.8 jusqu’à ce que tout le volume de lysat de phage ait été passé à travers le dispositif de filtration, avec ~2 mL de phage concentré retenu dans le réservoir supérieur après chaque essorage.
- Déboucher la membrane filtrante après le dernier essorage en pipetant le lysat restant (~2 mL) dans le réservoir supérieur de haut en bas. Laver le phage (échange de tampon) en ajoutant 12 mL de tampon SM dans le réservoir supérieur et centrifuger à 4000 x g pendant 10 min, ou jusqu’à ce que la majeure partie du tampon soit passée à travers le filtre.
- Jetez le filtrat et répétez l’étape de lavage (étape 1.2.10). Remettre en suspension les 2 mL restants de lysat dans le tampon SM jusqu’à un volume final de 10 mL (ou moins), pour un stockage à long terme. Transférez le lysat dans un tube à centrifuger conique de 50 mL et conservez-le à 4 °C jusqu’à ce que les endotoxines soient éliminées.
REMARQUE : Il est facultatif de titrer le phage à ce stade pour s’assurer que le lysat de phage a été concentré (> 108 pfu/mL) et pour déterminer la perte de phage pendant le processus d’élimination des endotoxines. - Éliminer les endotoxines contaminantes du lysat de phage T4 en ajoutant 0,4 volume de 1-octanol (voir le tableau des matériaux) au volume total de lysat.
REMARQUE : Les endotoxines sont éliminées car elles sont hautement immunostimulantes, et leur présence peut donc conduire à l’induction d’une réponse immunitaire innée indépendante des phages25.
ATTENTION : Le 1-octanol est un composé aromatique, organique et inflammable avec une forte odeur et est un irritant pour les yeux. Portez l’EPI approprié lorsque vous travaillez avec du 1-octanol. Effectuez tous les travaux dans une hotte pour éviter l’inhalation de vapeur. Utilisez un film d’étanchéité pour éviter les fuites de tubes lorsque vous travaillez à l’extérieur de la hotte. - Scellez le couvercle du tube à centrifuger conique avec un film d’étanchéité pour éviter les fuites. Agiter à 120 tr/min sur une plate-forme ou un agitateur à bascule à température ambiante pendant 1 h, suivi d’une incubation à 4 °C pendant 1,5 h, sans agiter.
- Centrifuger à 4000 x g pendant 10 min pour séparer le lysat éliminé des endotoxines du 1-octanol. La couche de 1-octanol flottera au-dessus du lysat. Utilisez une pipette P1000 pour prélever soigneusement la plus grande quantité possible de 1-octanol et jetez-la dans le récipient approprié pour les déchets liquides dangereux/inflammables.
- À l’aide d’une aiguille de 18 g et d’une seringue de 10 ml, recueillir le lysat de phage sous la couche de 1-octanol restante, en prenant soin de ne pas prélever la couche de 1-octanol. Conserver le lysat à 4 °C jusqu’à ce qu’il soit sous vide rapide.
- Transférer 1 mL d’aliquotes de lysat de phage T4 dans des tubes de microcentrifugation stériles de 1,5 mL. Accélérer le vide avec les couvercles ouverts à 4000 x g à température ambiante pour évaporer le 1-octanol résiduel du lysat. Vide rapide pendant 3 h ou jusqu’à ce que le volume du lysat de phage ait été réduit de 30 %25. Conserver à 4 °C jusqu’au titrage.
- Titrer le lysat de phage pour déterminer la concentration en pfu/mL. Diluez le lysat de phage T4 résultant dans un tampon SM à la concentration souhaitée et ré-titer pour confirmer.
- Quantifier les endotoxines présentes dans le lysat de phage T4 à l’aide d’une trousse de quantification des endotoxines chromogènes selon les instructions du fabricant (voir le tableau des matières). Comparez les niveaux d’endotoxines dans le lysat de phage final avec un échantillon de pré-élimination des endotoxines, des témoins de véhicules, un tampon et de l’eau potable de souris.
REMARQUE : Le kit de quantification des endotoxines chromogènes est inactivé par le 1-octanol25. Effectuer des étapes de vide rapide pour éliminer le 1-octanol (étape 1.2.16) avant de quantifier les endotoxines. La teneur en endotoxines de l’eau distillée est estimée à 20 UE/ml25 ; cependant, la teneur en endotoxines de l’eau potable des souris peut varier d’une installation à l’autre. Si les concentrations d’endotoxines dans le lysat de phage dépassent la quantité présente dans l’eau potable, envisager de répéter les étapes d’élimination des endotoxines (1.2.12-1.2.16). - Conserver les lysats de phages T4 concentrés et exempts d’endotoxines dans un tampon SM à 4 °C.
- Préparation d’un lysat bactérien sans phages pour la commande de véhicules
REMARQUE : Un lysat bactérien sans phage peut être préparé pour contrôler tout effet dû aux contaminants bactériens (p. ex., endotoxine) générés lors de la production de lysat de phage à l’étape 1.2, qui pourraient avoir une incidence sur les résultats expérimentaux lorsqu’il est inoculé à la souris.- À partir d’une culture nocturne d’E. coli, sous-culture de E . coli 1:50 dans 100 mL de milieu LB. Sans ajouter de phage, continuer à cultiver la bactérie en agitant à 37 °C pendant 3 h, ou en respectant le temps d’incubation du lysat de phage produit à l’étape 1.2.
- Transférer la culture d’E. coli dans des tubes coniques de 50 mL et lyser les cellules à l’aide d’une sonde sonicatrice (voir le tableau des matériaux) sur de la glace à 30 kHz pendant des impulsions de 30 s (3x) pour lyser manuellement les cellules bactériennes.
- Suivez le reste du protocole selon l’étape 1.2, en commençant par l’étape 1.2.4, y compris toutes les étapes de nettoyage, de lavage et d’élimination des endotoxines.
REMARQUE : Lors de la concentration à l’aide du dispositif de filtration centrifuge, le lysat du véhicule s’écoulera à travers le filtre plus rapidement que le lysat de phage, en raison de l’absence de phage. Par conséquent, raccourcissez les temps de centrifugation de 5 minutes à 2-5 minutes pour vous assurer que la membrane ultrafiltrante ne se dessèche pas lors d’une centrifugation prolongée. - Titrez le lysat du véhicule pour confirmer qu’il ne contient pas de phage.
- Utilisez une trousse de quantification des endotoxines chromogènes selon les instructions du fabricant pour mesurer les niveaux d’endotoxines dans le lysat du véhicule. Diluer le lysat de véhicule dans un tampon SM pour correspondre aux niveaux d’endotoxines dans le lysat de phage.
- Contrôles expérimentaux alternatifs : préparation de lysat de phages inactivé par la chaleur
REMARQUE : Une alternative au lysat bactérien sans phage est un lysat de phage inactivé par la chaleur. L’inactivation thermique dissocie les virions des phages, tandis que les endotoxines résiduelles telles que les lipopolysaccharides (LPS) sont thermostables 8,33. Cette méthode a été utilisée par Gogokhia et al.8 pour déterminer si des virions de phages intacts et viables étaient nécessaires pour l’activation immunitaire. Les chercheurs sont encouragés à tester les deux méthodes (sans phages ou inactivées par la chaleur) et à déterminer quel contrôle est le plus approprié pour leurs besoins expérimentaux. Quel que soit le choix choisi, il est important de reconnaître qu’un contrôle tampon est peu susceptible d’être approprié en raison de la manipulation importante nécessaire pour concentrer et nettoyer un stock de phages.- Transférez le volume requis de lysat de phages T4 nettoyé, purifié et dilué de l’étape 1.2 (100 μL par souris) dans des tubes de microcentrifugation stériles munis de verrous de couvercle.
- Chauffer les lysats sur un bloc chauffant à 95 °C pendant 15 min16.
- FACULTATIF : Si l’élimination des acides nucléiques phage/bactériens est nécessaire, effectuer un traitement à la DNase I et à la RNase A conformément à Jakočiūnė et Moodley34. Brièvement:
- Ajouter 50 μL de tampon DNase I 10x, 1 μL de DNase I (1 U/μL) et 1 μL de RNase A (10 mg/mL) (voir le tableau des matières) à 450 μL de lysat de phage inactivé par la chaleur.
- Incuber les lysats à 37 °C pendant 1,5 h dans un bloc thermique, sans agiter.
- Inactiver la DNase I et la RNase A en ajoutant 20 μL 0,5 M d’acide éthylènediaminetétraacétique (EDTA)34.
- Maintenir les lysats à température ambiante pendant 10 minutes pour refroidir et combiner les lysats si plusieurs tubes sont utilisés. Conserver à 4 °C jusqu’au titrage.
- Effectuez un titre de phage pour confirmer qu’aucun phage T4 viable n’est présent dans le lysat. Conserver les lysats inactivés par la chaleur à 4 °C.
- Utilisez une trousse de quantification des endotoxines chromogènes selon les instructions du fabricant pour mesurer les niveaux d’endotoxines dans le lysat inactivé par la chaleur. Diluer le lysat inactivé par la chaleur dans un tampon SM en fonction des niveaux d’endotoxines présents dans le lysat de phage apparié.
2. Administration et surveillance du phage T4 chez les souris monocolonisées par E. coli
- Monocolonisation de souris par E. coli
- Préparez E. coli pour l’administration à des souris GF8 en cultivant E . coli prélevé sur une seule colonie dans un milieu LB pendant la nuit.
- Dans des conditions strictes et stériles35, administrer 200 μL de culture d’E. coli à des souris GF logées dans des isolateurs vinyliques stériles ou des cages hermétiques de bioexclusion par gavage oral. Si vous utilisez des bactéries aérotolérantes, les souris peuvent également être colonisées par l’application de 200 μL de culture bactérienne sur le dos de chaque souris.
REMARQUE : La dose d’inoculation dépendra de la souche, car différentes bactéries présentent une capacité de survie au pH différent36,37. Zucoloto et al.35 recommandent l’inoculation avec 1 x 108 ufc par animal. Les résultats représentatifs présentés ici ont été générés à partir de souris inoculées avec une culture de nuit (ufc n’a pas été déterminé). Des expériences pilotes peuvent être réalisées pour déterminer une dose qui entraîne une colonisation fiable de la souche de souris d’intérêt. Le volume de bactéries à administrer par gavage oral peut être affecté par l’âge et le poids des souris. Consultez le protocole d’éthique animale de laboratoire ou d’établissement pour connaître les doses maximales autorisées. Le volume suggéré ici est basé sur une tolérance de gavage de 10 % du poids corporel de la souris (par exemple, 200 μL maximum pour une souris de 20 g). Certaines espèces bactériennes ne tolèrent pas l’acidité de l’estomac et ne parviennent pas à coloniser l’intestin. Envisager d’abord un gavage avec 100 μL de 1 M de NaHCO3 pour neutraliser les acides gastriques2. Si vous utilisez un anaérobie strict pour coloniser, le microbe devra être cultivé dans des conditions anaérobies et transféré à l’animalerie gnotobiotique dans des récipients hermétiques préparés individuellement (1 pour chaque souris). Effectuez chaque gavage rapidement une fois le récipient ouvert pour limiter la perte de viabilité microbienne due à l’exposition à l’oxygène. - Surveiller les souris pour détecter les effets néfastes sur la santé.
REMARQUE : En cas d’effets néfastes sur la santé, se référer aux normes établies par l’établissement de soins aux animaux concerné. Les effets indésirables possibles sur la santé comprennent, sans s’y limiter : (1) Aspiration de liquide de gavage : les symptômes comprennent l’expulsion de bulles par le nez, la respiration « bouche ouverte » / halètement. (2) Perforation de l’œsophage : les symptômes comprennent une santé rapidement malade, une courbure, une léthargie, entraînant la mort de l’animal dans les 24 heures. (3) Inflammation de l’œsophage due à des gavages répétés : les symptômes incluent une difficulté à insérer l’aiguille du gavage. (4) Diarrhée due à des altérations du microbiome. - Confirmer la colonisation bactérienne dans les boulettes fécales par culture au moins une fois par semaine et/ou par séquençage de l’ARNr 16S35.
REMARQUE : Si vous colonisez des reproducteurs dans un isolateur gnotobiotique pour la production de souris expérimentales, planifiez au moins 9 semaines à l’avance la génération de souris expérimentales de première génération de progéniture (F1) âgées de 6 semaines. Alternativement, les souris adultes GF peuvent être monocolonisées. Dans cette approche, il est recommandé d’attendre 7 semaines après la colonisation avant l’inoculation des phages, car c’est le temps nécessaire à la muqueuse intestinale pour se stabiliser après l’introduction d’un microbiote complexe chez les souris GF38.
- Inoculation orale du phage T4 chez des souris monocolonisées par E. coli
- Diluer les lysats de phages T4 et les témoins de véhicules à une concentration prédéterminée dans le tampon MS. Selon Hsu et al.2, diluer les lysats de phages pour permettre l’administration de 2 x 106 pfu par souris2.
REMARQUE : Une concentration plus ou moins élevée de phage peut être utilisée en fonction de la stabilité du phage in vivo. Des doses de 2 x 102, 2 x 104 et 2 x 106 pfu de phage T4 par souris ont entraîné une colonisation stable et à long terme dans l’intestin qui ne semblait pas dépendre de la dose (voir la section des résultats représentatifs). La cinétique des phages-bactéries doit donc être testée in vivo avant les expériences à grande échelle. - Dans des conditions stériles et gnotobiotiques, gaver chaque souris avec 100 μL de NaHCO3 1 M autoclavé pour neutraliser les acides gastriques. Attendre 10 min, puis gavage avec 100 μL de lysat de phage T4 ou contrôle du véhicule.
- Surveiller les souris pour détecter les effets néfastes sur la santé.
- Diluer les lysats de phages T4 et les témoins de véhicules à une concentration prédéterminée dans le tampon MS. Selon Hsu et al.2, diluer les lysats de phages pour permettre l’administration de 2 x 106 pfu par souris2.
3. Surveillance des niveaux de phages T4 in vivo
REMARQUE : Une fois que les souris ont été inoculées avec des phages, la concentration des phages et des bactéries cibles peut être mesurée dans des échantillons de matières fécales ou de tissus. Cela fournit des informations sur la cinétique de l’infection par les phages et la dynamique de colonisation des deux organismes.
- Placage ponctuel du phage T4 pour déterminer la concentration dans les boulettes fécales
- Prélevez les boulettes fécales de chaque souris dans des tubes à microcentrifuger stériles et pré-pesés pour mesurer les niveaux de phages T4 et d’E. coli . Conserver les tubes sur de la glace jusqu’à ce qu’ils soient placés.
REMARQUE : Placez les échantillons sur de la glace entre le prélèvement et le placage pour ralentir la croissance des bactéries aérotolérantes. Pendant plusieurs heures, il peut encore y avoir une croissance de bactéries aérobies, ou une mort de bactéries anaérobies due à l’exposition à l’oxygène, ce qui pourrait entraîner un nombre asymétrique de bactéries. Par conséquent, le placage bactérien et phagique doit être effectué dès que possible après le prélèvement de l’échantillon. Les bactéries anaérobies obligatoires ne tolèrent pas l’exposition à l’oxygène. Pour préserver l’échantillon, prélevez les échantillons dans des tubes scellés et transférez les tubes dans une chambre anaérobie dès que possible après le prélèvement. Si aucune croissance n’est détectée dans les échantillons fécaux, envisagez des alternatives telles que la qPCR de l’ARNr 16S pour la quantification bactérienne. - Enregistrer le poids final de chaque tube et calculer le poids de l’échantillon en soustrayant le poids initial du tube. Cela sera utilisé pour normaliser la concentration de phages T4 et d’E. coli au poids de l’échantillon (pfu/g ou ufc/g, respectivement).
- Ajouter 1 mL de tampon SM stérile dans chaque tube et agiter complètement à vitesse maximale (>1 min) pour homogénéiser les granules fécales. Si l’échantillon est inférieur à 15 mg, un plus petit volume de tampon SM peut être ajouté. Enregistrer le volume de tampon MS ajouté à chaque échantillon pour calculer l’ufc/g ou l’ufp/g de l’échantillon.
- Préparer une série de 8 dilutions en série (ou plus selon la concentration attendue dans le lysage) de 20 μL de chaque échantillon dans 180 μL de tampon SM, par facteur de 10. Vortex chaque échantillon homogénéisé brièvement pour mélanger avant de l’ajouter au premier tube/puits. Pipeter pour mélanger entre chaque ajout et changer les pointes entre chaque dilution pour éviter de gonfler le nombre de phages ou de bactéries via le transfert d’échantillon.
REMARQUE : Si les fibres et les débris présents dans l’échantillon entravent le pipetage, ajoutez un tampon supplémentaire à la boue fécale pour diluer davantage l’échantillon de base. - Placer 5 μL de chaque dilution sur des plaques de gélose molle LB contenant E. coli (pour les tests sur plaques de phages) ou des plaques de gélose LB (gélose à 1,5 %, pour les tests de colonies bactériennes) pour déterminer la concentration de phages T4 et d’E . coli dans chaque échantillon. Pour plus de précision, repérez chaque échantillon en trois exemplaires.
NOTA : Si vous préparez des dilutions en série dans une plaque à 96 puits, une pipette multicanaux P20 à 8 canaux peut être utilisée pour pipeter chaque colonne d’échantillon dilué en série sur des plaques. Changez les pointes entre chaque dilution, même si vous passez de la plus diluée à la plus concentrée, car le phage peut adhérer aux parois de la pointe de la pipette et modifier la quantité de phage ajoutée à chaque nouveau puits31. - Laissez sécher chaque endroit avant de retourner la plaque et de la placer dans l’incubateur. Incuber toute la nuit à 37 °C.
- Pour chaque échantillon, sélectionnez la dilution à laquelle il y a 3 à 30 plaques dénombrables par endroit. Comptez et notez le nombre de plaques à l’endroit et la dilution utilisée.
- Calculer le pfu/g de l’échantillon en divisant le nombre de plaques par le volume plaqué à chaque endroit pour obtenir le pfu/μL. Multiplier ce chiffre par le facteur de dilution et par le volume de tampon MS ajouté à chaque échantillon pour obtenir le pfu/échantillon. Enfin, divisez par le poids de l’échantillon pour obtenir30 pfu/g.

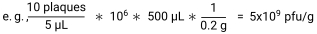
- Prélevez les boulettes fécales de chaque souris dans des tubes à microcentrifuger stériles et pré-pesés pour mesurer les niveaux de phages T4 et d’E. coli . Conserver les tubes sur de la glace jusqu’à ce qu’ils soient placés.
- Échantillonnage de tissus aux paramètres expérimentaux
- À chaque moment choisi, euthanasier les souris conformément au protocole d’éthique animale approuvé par l’établissement et recueillir le contenu cæcal, le contenu de l’intestin grêle et du gros intestin et tout tissu d’intérêt dans des tubes à microcentrifuger stériles et prépesés de 2 mL à fond rond.
- Enregistrer les poids finaux de chaque tube pour calculer le poids de l’échantillon. Conservez les mouchoirs et le contenu cæcal sur de la glace jusqu’au jour même du placage.
- Ajouter un tampon SM stérile dans chaque tube et noter les volumes ajoutés.
- Pour le contenu cæcal et intestinal, échantillonner soigneusement les vortex (>1 min) conformément à l’étape 3.1.3.
- Pour les échantillons de tissus, ajoutez une bille métallique stérile dans chaque tube. Homogénéiser les tissus à l’aide d’un lyseur tissulaire (voir tableau des matériaux) à 20 Hz pendant 5 min ou jusqu’à ce que les tissus soient dissociés et que la suspension soit homogène.
REMARQUE : Si les échantillons ne s’homogénéisent pas bien, envisagez d’augmenter le volume de tampon SM ajouté, d’augmenter le temps d’homogénéisation ou d’augmenter la fréquence d’homogénéisation à 30 Hz. Un homogénéisateur de tissus peut également être utilisé pour dissocier les tissus tout en permettant la récupération des bactéries et des phages39,40. Les homogénéisateurs fonctionnent à des fréquences plus élevées que les lyseurs tissulaires tout en préservant l’intégrité des bactéries. - Préparer des dilutions en série de chaque échantillon par facteur de 10 et effectuer un placage ponctuel pour déterminer les concentrations de phages E. coli et T4 dans chaque échantillon, comme décrit à l’étape 3.1.4-3.1.841.
Access restricted. Please log in or start a trial to view this content.
Résultats
Pour étudier les interactions entre la dyade phage/E. coli T4 dans l’intestin murin, le phage T4 et les lysats de véhicule ont été préparés, nettoyés et purifiés (Figure 1A). Les lysats de phages T4 ont été titrés par test sur plaque et dilués à 2 x 107 pfu/mL (2 x 106 pfu/souris) dans un tampon SM. Les lysats de véhicule ont également été titrés pour confirmer l’absence de présence de phages viables et dilués dans le même volume de tam...
Access restricted. Please log in or start a trial to view this content.
Discussion
L’étude des phages dans le microbiome présente un défi important par rapport à leurs homologues bactériens. Plus précisément, les phages ne contiennent pas de marqueur phylogénétique conservé commun à tous les phages apparenté aux sous-unités ribosomales 16S et 18S qui permettent de faciliter le séquençage et l’identification des espèces procaryotes et eucaryotes, respectivement42. Cependant, avec les progrès des approches de séquençage de nouvelle génération, y compris l?...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Les auteurs reconnaissent que les terres sur lesquelles ils ont effectué cette recherche sont le territoire traditionnel, ancestral et non cédé de la nation xwməθkwəy̓əm (Musqueam). La terre sur laquelle il est situé a toujours été un lieu d’apprentissage pour le peuple Musqueam, qui depuis des millénaires transmet sa culture, son histoire et ses traditions d’une génération à l’autre sur ce site. Nous encourageons les autres à en apprendre davantage sur les terres autochtones dans lesquelles ils vivent et travaillent à https://native-land.ca. Les auteurs remercient le Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG) pour le soutien des bourses d’études supérieures du Canada - maîtrise (N.P.), de la bourse de stagiaire Michael Smith Health Research BC (RT-2023-3174, à MH), du Programme de subventions à la découverte du Conseil de recherches en sciences naturelles et en génie du Canada (CRSNG) (RGPIN-2019-04591 à C.T., RGPIN-2016-04282 à la CDO), de l’Institut canadien de recherches avancées / L’humain et le microbiome (FL-001253 Appt 3362, à C.T.), la bourse de boursier de la Fondation Michael-Smith pour la recherche en santé (18239, à C.T.), les Instituts de recherche en santé du Canada (PJT-159458 à la CDO) et la Fondation canadienne pour l’innovation (34673 à la CDO et 38277 à CT). Nous sommes reconnaissants du soutien technique du Centre de modélisation des maladies de l’Université de la Colombie-Britannique et d’ubcFLOW, qui est soutenu par l’initiative de résilience biologique GREx de l’Université de la Colombie-Britannique, ainsi que des membres des laboratoires Osborne et Tropini pour les discussions critiques et l’évaluation du manuscrit. Les figures 1A et 2A ont été créées à l’aide de Biorender.com.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| 1-octanol (99%) | Thermofisher | CAAAA15977-AP | |
| 50 ml PES Steriflip Sterile Disposable Vacuum Filter Units | Millipore Sigma | SCGP00525 | |
| Agarose (Low-EEO/Multi-Purpose/Molecular Biology Grade) | Fisher BioReagents | BP160-500 | |
| Amicon® 100kDa Ultra-15 centrifugal filter device, Ultracel-100 | Millipore Sigma | UFC910008 | |
| BD Microtainer® Tubes, SST | BD Medical | 365967 | |
| Bioexclusion airtight cages (ISO cages) | Techiplast | 1245ISOCAGE | |
| C1000 Touch™ Thermal Cycler with 96-Well Fast Reaction Module | BioRad | 1851196 | |
| Calcium Chloride Dihydrate (White Crystals to Powder) | Fisher BioReagents | BP510-500 | |
| Cap Locks For 1.5ML Tube 100/pk | Andwin Scientific | 16812612 | |
| Chloroform (Ethanol as Preservative/Certified ACS) | Fisher | C298-500 | |
| Copper coated steel beads (4.5 mm) | Crosman Corporation | 0767 | |
| DNeasy Blood & Tissue Kit (50) | Thermo Scientific | 69504 | |
| DreamTaq Green PCR Master Mix (2X) | Thermo Scientific | K1081 | |
| Ethylenediaminetetraacetic acid (EDTA) disodium salt solution, for molecular biology, 0.5 M in H2O | Sigma Aldrich | E7889 | |
| Fisher BioReagents™ Agar, Powder / Flakes, Fisher BioReagents™ | Fisher Bioreagents | BP1423-500 | |
| Fisher BioReagents™ Microbiology Media: LB Broth (Powder) - Lennox | Fisher Bioreagents | BP1427-500 | |
| GeneRuler 100 bp DNA Ladder | Thermo Scientific | SM0241 | |
| Green FastMix® qPCR mix, 1250 rxns | QuantaBio | 95072-012 | |
| HEPA filters for isocage lids, AUTOCLAVABLE H14 FILTERS FOR ISO LINE- IRRADIATED | Techiplast | UISOHEPAXTBOX-300 | |
| Magnesium sulfate heptahydrate | Fisher BioReagents | BP213-1 | |
| MaxQ 6000 Incubated Shaker | Thermo Scientific | 8354-30-0009 | |
| Microbiology Media: LB Broth (Powder) - Lennox | Fisher BioReagents | BP1427-500 | |
| Microcentrifuge Tubes with Locking Snap Cap, 2ml | Fisher | 14-666-315 | |
| Parafilm sealing film | Bemis | PM-996 | |
| Phage stocks | Carolina Biological Supply | n/a | |
| PicoLab® Mouse Diet 20 EXT | LabDiet | 5R58 | |
| Pierce™ Chromogenic Endotoxin Quant Kit | Thermo Scientific | A39552S | |
| RNase A (17,500 U) | Qiagen | 19101 | |
| RNase-free DNase Set | Qiagen | 79254 | |
| Sodium Bicarbonate (Fine White Powder) | Fisher Chemical | BP328-500 | |
| Sodium Chloride (Crystalline/Certified ACS) | Fisher Chemical | S271 | |
| Sonicator (probe model CL-18; power source model FB50) | Fisher scentific | n/a | |
| Sterile flexible film isolator | Class Biologically Clean | n/a | |
| SYBR™ Safe DNA Gel Stain | Invitrogen | S33102 | |
| T100 Thermal Cycler | BioRad | 1861096 | |
| T4 phage primer, forward (CCACACATAGCGCGAGTATAA) | IDT | n/a | |
| T4 phage primer, forward (GAAACTCGGTCAGGCTATCAA) | IDT | n/a | |
| TissueLyser II | Qiagen | 85300 | |
| Tris-HCl, 1M Solution, pH 8.0, Molecular Biology Grade, Ultrapure | Thermo Scientific | AAJ22638AE | |
| Water, (DNASE, RNASE free) | Fisher BioReagents | BP2484100 |
Références
- Rohwer, F., Segall, A. M. A century of phage lessons. Nature. 528 (7580), 46-47 (2015).
- Hsu, B. B., et al. Dynamic modulation of the gut microbiota and metabolome by bacteriophages in a mouse model. Cell Host & Microbe. 25 (6), 803-814.e5 (2019).
- Gordillo Altamirano, F. L., Barr, J. J. Phage Therapy in the postantibiotic era. Clinical Microbiology Reviews. 32 (2), (2019).
- Schooley, R. T., et al. Development and use of personalized bacteriophage-based therapeutic cocktails to treat a patient with a disseminated resistant Acinetobacter baumannii infection. Antimicrobial Agents and Chemotherapy. 61 (10), (2017).
- Ott, S. J., et al. Efficacy of Sterile Fecal Filtrate Transfer for Treating Patients With Clostridium difficile Infection. Gastroenterology. 152 (4), 799-811.e7 (2017).
- Zuo, T., et al. Bacteriophage transfer during faecal microbiota transplantation in Clostridium difficile infection is associated with treatment outcome. Gut. 67 (4), 634-643 (2018).
- Norman, J. M., et al. Disease-specific alterations in the enteric virome in inflammatory bowel disease. Cell. 160 (3), 447-460 (2015).
- Gogokhia, L., et al. Expansion of bacteriophages is linked to aggravated intestinal inflammation and colitis. Cell Host & Microbe. 25 (2), 285-299.e8 (2019).
- Brunse, A., et al. Fecal filtrate transplantation protects against necrotizing enterocolitis. The ISME Journal. 16 (3), 686-694 (2022).
- Duerkop, B. A., Clements, C. V., Rollins, D., Rodrigues, J. L. M., Hooper, L. V. A composite bacteriophage alters colonization by an intestinal commensal bacterium. Proceedings of the National Academy of Sciences of the United States of America. 109 (43), 17621-17626 (2012).
- Barr, J. J., et al. Bacteriophage adhering to mucus provide a non-host-derived immunity. Proceedings of the National Academy of Sciences of the United States of America. 110 (26), 10771-10776 (2013).
- Nguyen, S., et al. Bacteriophage Transcytosis provides a mechanism to cross epithelial cell layers. mBio. 8 (6), (2017).
- Bichet, M. C., et al. Bacteriophage uptake by mammalian cell layers represents a potential sink that may impact phage therapy. iScience. 24 (4), 102287(2021).
- Buttimer, C., et al. Impact of a phage cocktail targeting Escherichia coli and Enterococcus faecalis as members of a gut bacterial consortium in vitro and in vivo. Frontiers in Microbiology. 13, 936083(2022).
- Li, Y., et al. Bacteriophages allow selective depletion of gut bacteria to produce a targeted-bacterium-depleted mouse model. Cell Reports Methods. 2 (11), 100324(2022).
- Reyes, A., Wu, M., McNulty, N. P., Rohwer, F. L., Gordon, J. I. Gnotobiotic mouse model of phage-bacterial host dynamics in the human gut. Proceedings of the National Academy of Sciences of the United States of America. 110 (50), 20236-20241 (2013).
- Federici, S., et al. Targeted suppression of human IBD-associated gut microbiota commensals by phage consortia for treatment of intestinal inflammation. Cell. 185 (16), 2879-2898.e4 (2022).
- Sweere, J. M., et al. Bacteriophage trigger antiviral immunity and prevent clearance of bacterial infection. Science (New York, N.Y.). 363 (6434), (2019).
- Fluckiger, A., et al. Cross-reactivity between tumor MHC class I-restricted antigens and an enterococcal bacteriophage. Science (New York, N.Y.). 369 (6506), 936-942 (2020).
- Majewska, J., et al. Induction of Phage-Specific Antibodies by Two Therapeutic Staphylococcal Bacteriophages Administered per os. Frontiers in Immunology. 10, 2607(2019).
- Weiss, M., et al. In vivo replication of T4 and T7 bacteriophages in germ-free mice colonized with Escherichia coli. Virology. 393 (1), 16-23 (2009).
- Thomson, C. A., Morgan, S. C., Ohland, C., McCoy, K. D. From germ-free to wild: modulating microbiome complexity to understand mucosal immunology. Mucosal Immunology. 15 (6), 1085-1094 (2022).
- Al-Asmakh, M., Zadjali, F. Use of germ-free animal models in microbiota-related research. Journal of Microbiology and Biotechnology. 25 (10), 1583-1588 (2015).
- Ivanov, I. I., et al. Induction of intestinal Th17 cells by segmented filamentous bacteria. Cell. 139 (3), 485-498 (2009).
- Bonilla, N., et al. Phage on tap-a quick and efficient protocol for the preparation of bacteriophage laboratory stocks. PeerJ. 4, e2261(2016).
- Sambrook, J., Russell, D. W. Molecular Cloning: A Laboratory Manual. 1, Cold Spring Harbor Laboratory Press. New York, NY. (2001).
- Kropinski, A. M., Mazzocco, A., Waddell, T. E., Lingohr, E., Johnson, R. P. Enumeration of bacteriophages by double agar overlay plaque assay. Methods in Molecular Biology (Clifton, N.J). 501, 69-76 (2009).
- Manikantha, B., Karthika, R., Murugadas, V., Vishnuvinayagam, S., Rao, B. M. Comparison of the single agar and double agar layer methods for enumeration of bacteriophages. Fishery Technology. 59, 60-63 (2022).
- Sanders, E. R. Aseptic laboratory techniques: plating methods. Journal of Visualized Experiments. 63, e3064(2012).
- Louten, J. Chapter 7 - Detection and diagnosis of viral infections. Essential Human Virology. , 111-132 (2016).
- Richter, Ł, et al. Adsorption of bacteriophages on polypropylene labware affects the reproducibility of phage research. Scientific Reports. 11 (1), 7387(2021).
- Merck KGaA. Amicon Ultra-15 Centrifugal Filter Devices. , Darmstadt, Germany. https://www.emdmillipore.com/CA/en/product/Amicon-Ultra-15-Centrifugal-Filter-Unit,MM_NF-UFC901024#documentation (2018).
- Hecker, W., Witthauer, D., Staerk, A. Validation of dry heat inactivation of bacterial endotoxins. PDA Journal of Pharmaceutical Science and Technology. 48 (4), 197-204 (1994).
- Jakočiūnė, D., Moodley, A. A Rapid bacteriophage DNA extraction method. Methods and Protocols. 1 (3), 27(2018).
- Zucoloto, A. Z., Yu, I. L., McCoy, K. D., McDonald, B. Generation, maintenance, and monitoring of gnotobiotic mice. STAR Protocols. 2 (2), 100536(2021).
- Ng, K. M., et al. Single-strain behavior predicts responses to environmental pH and osmolality in the gut microbiota. mBio. 14 (4), e0075323(2023).
- McCallum, G., Tropini, C. The gut microbiota and its biogeography. Nature Reviews. Microbiology. , (2023).
- Bergstrom, K., Xia, L. The barrier and beyond: Roles of intestinal mucus and mucin-type O-glycosylation in resistance and tolerance defense strategies guiding host-microbe symbiosis. Gut Microbes. 14 (1), 2052699(2022).
- Askar, M., Ashraf, W., Scammell, B., Bayston, R. Comparison of different human tissue processing methods for maximization of bacterial recovery. European Journal of Clinical Microbiology & Infectious Diseases. 38 (1), 149-155 (2019).
- Redanz, S., Podbielski, A., Warnke, P. Improved microbiological diagnostic due to utilization of a high-throughput homogenizer for routine tissue processing. Diagnostic Microbiology and Infectious Disease. 82 (3), 189-193 (2015).
- Bhinder, G., et al. The Citrobacter rodentium mouse model: studying pathogen and host contributions to infectious colitis. Journal of Visualized Experiments. 72, e50222(2013).
- Reyes, A., Semenkovich, N. P., Whiteson, K., Rohwer, F., Gordon, J. I. Going viral: next-generation sequencing applied to phage populations in the human gut. Nature Reviews Microbiology. 10 (9), 607-617 (2012).
- Camarillo-Guerrero, L. F., Almeida, A., Rangel-Pineros, G., Finn, R. D., Lawley, T. D. Massive expansion of human gut bacteriophage diversity. Cell. 184 (4), 1098-1109.e9 (2021).
- Reyes, A., et al. Viruses in the faecal microbiota of monozygotic twins and their mothers. Nature. 466 (7304), 334-338 (2010).
- Bach, M. S., et al. Filamentous bacteriophage delays healing of Pseudomonas-infected wounds. Cell Reports. Medicine. 3 (6), 100656(2022).
- Filyk, H. A., Osborne, L. C. The multibiome: The intestinal ecosystem's influence on immune homeostasis, health, and disease. EBioMedicine. 13, 46-54 (2016).
- Rohwer, F., Merry, Y., Maughan, H., Hisakawa, N. Heather Life in Our Phage World: A Centennial Field Guide to the Earth's Most Diverse Inhabitants. Wholon. , San Diego. (2014).
- Glonti, T., Pirnay, J. P. In Vitro techniques and measurements of phage characteristics that are important for phage therapy success. Viruses. 14 (7), 1490(2022).
- Fraser, J. S., Yu, Z., Maxwell, K. L., Davidson, A. R. Ig-like domains on bacteriophages: a tale of promiscuity and deceit. Journal of Molecular Biology. 359 (2), 496-507 (2006).
- Li, H., et al. The outer mucus layer hosts a distinct intestinal microbial niche. Nature Communications. 6, 8292(2015).
- Bergström, A., et al. Nature of bacterial colonization influences transcription of mucin genes in mice during the first week of life. BMC Research Notes. 5, 402(2012).
- Adams, M. H. Bacteriophages. , Interscience Publishers. https://books.google.co.uk/books?id=3wVrAAAAMAAJ (1959).
- Kutter, E., Sulakvelidze, A. Bacteriophages: Biology and Applications. , CRC Press. https://books.google.co.uk/books?id=flHOvAEACAAJ (2004).
- Bao, H., et al. Dysbiosis and intestinal inflammation caused by Salmonella Typhimurium in mice can be alleviated by preadministration of a lytic phage. Microbiological Research. 260, 127020(2022).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon