Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Imagerie calcique en direct de monocouches organoïdes intestinaux humains infectés par un virus à l’aide d’indicateurs de calcium codés génétiquement
Dans cet article
Résumé
Ce protocole décrit une approche pour effectuer l’imagerie calcique dans les organoïdes intestinaux humains infectés par le virus et propose une approche d’analyse.
Résumé
La signalisation calcique est un régulateur intégral de presque tous les tissus. Dans l’épithélium intestinal, le calcium est impliqué dans la régulation de l’activité sécrétoire, de la dynamique de l’actine, des réponses inflammatoires, de la prolifération des cellules souches et de nombreuses autres fonctions cellulaires non caractérisées. En tant que tel, la cartographie de la dynamique de signalisation du calcium dans l’épithélium intestinal peut fournir un aperçu des processus cellulaires homéostatiques et dévoiler des réponses uniques à divers stimuli. Les organoïdes intestinaux humains (HIO) sont un modèle à haut débit, dérivé de l’homme, pour étudier l’épithélium intestinal et représentent donc un système utile pour étudier la dynamique du calcium. Cet article décrit un protocole permettant de transduire de manière stable les HIO avec des indicateurs calciques génétiquement codés (GECI), d’effectuer une microscopie à fluorescence en direct et d’analyser les données d’imagerie pour caractériser de manière significative les signaux calciques. À titre d’exemple représentatif, des HIO tridimensionnels ont été transduits avec des lentivirus pour exprimer de manière stable GCaMP6s, un GECI cytosolique à base de protéines fluorescentes vertes. Les HIO modifiés ont ensuite été dispersés dans une suspension unicellulaire et ensemencés sous forme de monocouches. Après la différenciation, les monocouches de HIO ont été infectées par le rotavirus et/ou traitées avec des médicaments connus pour stimuler une réponse calcique. Un microscope à épifluorescence équipé d’une chambre d’imagerie en direct humidifiée et à température contrôlée a permis d’imager à long terme des monocouches infectées ou traitées par des médicaments. Après l’imagerie, les images acquises ont été analysées à l’aide du logiciel d’analyse gratuit ImageJ. Dans l’ensemble, ces travaux établissent un pipeline adaptable pour caractériser la signalisation cellulaire dans les DOI.
Introduction
Le calcium est un deuxième messager largement conservé qui joue un rôle essentiel dans la régulation de la physiologie cellulaire1. Compte tenu de sa forte charge, de sa petite taille et de sa grande solubilité dans des conditions physiologiques, le calcium est un manipulateur idéal de la conformation des protéines. Cela fait du calcium un moyen puissant de transduire les signaux électrochimiques en altérations enzymatiques, transcriptionnelles ou post-transcriptionnelles. Les gradients stricts de concentration de calcium à travers le réticulum endoplasmique (RE) et les membranes plasmiques créent une force motrice élevée qui permet des changements rapides de la concentration de calcium cytosolique. De multiples mécanismes, y compris la mise en mémoire tampon et le transport actif, maintiennent étroitement ce gradient. Bien que nécessaire aux fonctions cellulaires normales, cet entretien est coûteux en énergie, ce qui le rend particulièrement sensible aux états de stress 2.
En tant que tel, la dérégulation du calcium dans le cytosol est un signal quasi universel de nombreux types de stress cellulaire. Les perturbations métaboliques, les toxines, les agents pathogènes, les dommages mécaniques et les perturbations génétiques peuvent tous perturber la signalisation du calcium. Quel que soit le stimulus, au niveau de la cellule entière, des augmentations soutenues et incontrôlées du calcium cytosolique peuvent favoriser l’apoptose et éventuellement la nécrose 3,4. Cependant, les altérations des taux de calcium cytosolique de plus faible amplitude ou de fréquence plus élevée ont des effets variables2. De même, les résultats des fluctuations calciques peuvent différer en fonction du microdomaine spatial dans lequel elles se produisent5. La surveillance des niveaux de calcium peut donc donner un aperçu des processus de signalisation dynamiques, mais cela nécessite un échantillonnage avec une résolution temporelle et spatiale relativement élevée.
Les indicateurs calciques génétiquement codés (GECI) sont des outils puissants pour l’échantillonnage continu dans les systèmes de cellules vivantes6. Certains des GECI les plus largement utilisés sont des protéines fluorescentes sensibles au calcium à base de GFP, connues sous le nom de GCaMPs7. Le GCaMP canonique est une fusion de trois domaines protéiques distincts : une GFP permutée circulairement (cpGFP), la calmoduline et M136. Le domaine de la calmoduline subit un changement de conformation lors de la liaison du calcium, ce qui permet son interaction avec M13. L’interaction calmoduline-M13 induit un changement conformationnel de la cpGFP qui augmente son émission fluorescente lors de l’excitation. En tant que tel, une augmentation de la concentration de calcium est corrélée à une augmentation de l’intensité de fluorescence GCaMP. Ces capteurs peuvent être cytosoliques ou ciblés sur des organites spécifiques8.
Comme la plupart des tissus, le calcium régule diverses fonctions au sein de l’épithélium gastro-intestinal. L’épithélium intestinal fait partie intégrante de l’absorption des nutriments et des liquides, mais doit également former une barrière étanche et une interface immunitaire pour éviter l’invasion d’agents pathogènes ou les agressions toxiques. Les voies dépendantes du calcium influencent presque toutes ces fonctions vitales 9,10,11. Cependant, la signalisation calcique dans l’épithélium intestinal reste une frontière sous-explorée avec un potentiel prometteur en tant que cible thérapeutique. Alors que la surveillance de la dynamique du calcium dans l’épithélium intestinal in vivo continue de présenter des défis, les organoïdes intestinaux humains (DOI) offrent un système ex vivo adaptable pour l’expérimentation12. Les HIO sont des sphéroïdes tridimensionnels (3D) dérivés de cellules souches intestinales humaines et, lors de la différenciation, récapitulent une grande partie de la diversité cellulaire de l’épithélium intestinal natif12.
Ce protocole décrit des méthodes complètes pour concevoir des OIS qui expriment des GECI, puis préparer des OIS modifiés sous forme de monocouches pour l’imagerie du calcium sur cellules vivantes. Il propose l’infection virale comme exemple d’une manipulation pathologique qui perturbe la signalisation calcique et fournit une approche analytique pour quantifier ces changements.
Protocole
Tous les organoïdes intestinaux humains (HIO) utilisés dans ce protocole et les expériences représentatives ont été dérivés de tissus humains obtenus et conservés par le Texas Medical Center Digestive Diseases Enteroid Core. Tous les échantillons ont été prélevés conformément à un protocole approuvé par le comité d’examen institutionnel du Baylor College of Medicine.
1. Préparation des matériaux et des réactifs
- Pour l’entretien des organoïdes, prélever des plaques à 24 puits traitées par culture cellulaire, une matrice membranaire basale (BMM), des tubes coniques de 15 ml et des tubes coniques de 1,5 ml.
- Pour préparer un milieu complet sans facteurs de croissance (CMGF-), ajouter à 500 mL de DMEM F12 avancé 5 mL de 1M HEPES, 5 mL d’antibiotique-antimycosique 100x, 5 mL de supplément de glutamine 100x.
- Pour préparer les milieux contenant du Wnt, de la R-spondine et de la Noggin (WRNE), mélanger à parts égales des milieux conditionnés au CMGF et au Wnt, ajouter un milieu conditionné à la Noggin, 10 % en volume, un milieu conditionné à la R-spondine, 20 % en volume, 50 ng/mL de facteur de croissance épidermique humain, 10 mM de nicotinamide, 10 nM [Leu15]-Gastrine I, 500 nM A-83-01, 10 μM SB202190, 1 supplément de B27, 1 supplément de N2 et 1 mM de N-acétylcystéine.
- Pour la transduction lentivirale, préparer du sérum de veau fœtal (FBS), 0,05 % de trypsine-EDTA dans 1 solution saline tamponnée au phosphate (PBS), CMGF- avec 10 % de FBS, stérile 1x PBS, Polybrène, 10 μM Y-27632, Lentivirus, Wnt WRNE élevé + 10 μM Y-27632
- Pour générer des monocouches organoïdes, préparer une lame de culture cellulaire à 10 puits à fond de verre, FBS, collagène IV (1 mg/mL dans (di)H2O désionisé), matrice membranaire basale, 0,5 mM EDTA dans 1x PBS, 5 mM EDTA dans 1x PBS, tampon de dissociation enzymatique, CMGF- avec 10% FBS, WRNE + 10 μM Y-27632.
- Pour l’infection virale des monocouches organoïdes, préparez de la trypsine, du rotavirus, du CMGF-, de l’aiguille 25G, du PBS stérile 1x et du milieu de différenciation sans rouge de phénol.
- Pour préparer un milieu de différenciation sans rouge de phénol, prenez 500 mL de milieu de culture cellulaire sans rouge de phénol, ajoutez 5 mL d’acides aminés non essentiels 100x MEM, 5 mL de 100x L-Glutamine, 5 mL de pyruvate de sodium 100 nM et 7,5 mL de 1M HEPES.
- Pour la coloration par immunofluorescence des organoïdes, préparer 4 % de formaldéhyde (16 % de formaldéhyde dilué dans 1x PBS), Triton X-100 (0,1% Triton X-100 dans 1x PBS), Albumine sérique bovine (3% d’albumine sérique bovine dans 1x PBS), solution NH4Cl (50 mM), DAPI (1 μg/mL de solution DAPI dans 1x PBS).
2. Ingénierie d’organoïdes pour exprimer des capteurs de calcium codés génétiquement
NOTA : Ce protocole décrit les étapes à suivre pour transduire un seul puits d’organoïdes intestinaux humains tridimensionnels plaqués dans 30 μL de matrice membranaire basale (BMM) sur une plaque à 24 puits13. La plupart des lignes contiendront environ 400 000 cellules par puits. Un deuxième puits, non transduit, devrait être inclus comme témoin. Conservez tous les réactifs et les suspensions cellulaires sur de la glace.
- De 2 à 5 jours après le dernier passage, retirer le milieu d’entretien WRNE de deux puits d’OIR. Remplacez-le par 300 μL de trypsine-EDTA à 0,05 % par puits et pipetez doucement de haut en bas 5 fois pour détacher le BMM de la plaque. Placer dans un incubateur à 37 °C pendant 4 min.
- Ajouter 500 μL de CMGF- + 10% FBS par puits. Enduisez une pointe de pipette à faible liant de 1 ml avec FBS en pipetant 1 mL de FBS de haut en bas 2x. FBS peut être réutilisé sur plusieurs pointes. À l’aide de l’embout préenduit, pipetez les HIO de haut en bas 10x.
- Transférez le contenu de chaque puits dans son propre tube de microcentrifugation pré-enrobé de 1,5 ml. Rincez chaque puits avec 500 μL supplémentaires de CMGF- et ajoutez le nettoyant dans le tube correspondant.
- Centrifuger les tubes à 100 x g dans une centrifugeuse à godets oscillants pendant 5 min à 4 °C. Éliminez le surnageant et tout résidu de BMM.
- Remettre en suspension dans 1 mL de 1x PBS. Divisez chaque tube en deux tubes de microcentrifugation pour un total de 4 tubes. Centrifuger les tubes à 100 x g pendant 5 min à 4 °C. Enlever le surnageant.
- Remettre chaque tube en suspension une fois de plus dans 1x PBS et centrifuger à 100 x g pendant 5 min à 4 °C.
- Préparer 400 μL de milieu de transduction et de milieu témoin (tableau 1). Remettre en suspension 2 tubes dans 200 μL du milieu témoin et 2 tubes dans 200 μL du milieu de transduction.
- Incuber dans un incubateur de culture cellulaire à 37 °C pendant 24 h. Remettre en suspension en pipetant périodiquement de haut en bas avec une pointe enduite (p. ex., 2 h après la transduction (hpt), 12 hpt, 18 hpt) pour favoriser une transduction uniforme.
- Après 24 h, centrifuger les tubes à 100 x g pendant 5 min à 4 °C. Enlever le surnageant.
- Remettre le granulé en suspension dans 500 μL de 1x PBS pour le laver. Centrifuger à 100 x g pendant 5 min à 4 °C. Enlever le surnageant.
- À l’aide d’un embout de pipette glacé de 200 μL, remettre en suspension chacun des 4 granulés dans 30 μL de BMM. Pipeter doucement de haut en bas pour disperser uniformément.
- Placez le contenu de chaque tube dans son propre puits sur une plaque de 24 puits. Incuber pendant 10 min à 37 °C pour permettre à BMM de se solidifier avant d’ajouter 500 μL de HighWnt WRNE + 10μM Y-27632.
- Laisser croître les HIO transduits pendant 1 semaine, en rafraîchissant le milieu (HighWnt WRNE + 10 μM Y-27632) tous les deux jours. Après 1 semaine, vérifier l’expression de l’indicateur fluorescent par microscopie. Si le signal est fort, commencez la sélection du médicament. Si le signal est faible, répétez la transduction comme décrit ci-dessus.
- Une fois la ligne établie, vérifier le fonctionnement de l’indicateur de fluorescence par traitement agoniste. L’ADP 100 nM est un agoniste fiable pour la validation GCaMP. Testez les organoïdes 3D pour la validation initiale, comme décrit ci-dessous.
- Après un passage, plaquez un puits (ou plusieurs) d’organoïdes dans BMM sur une plaque inférieure d’imagerie séparée. Plaquer les organoïdes à environ 1/3 de la densité normale pour éviter un chevauchement excessif lors de l’imagerie. Ne continuez pas à faire passer ces DOI après un traitement agoniste, car il est difficile d’assurer la stérilité.
- Prévoyez 2 à 3 jours pour que les DOI se rétablissent après le passage. Avant l’imagerie, passez le milieu à un milieu de différenciation sans rouge de phénol.
- À l’aide d’un microscope à fluorescence, mettez en place un cycle de 3 minutes avec des images acquises toutes les 5 s en utilisant une excitation de 488 nm et un jeu de filtres FITC/GFP. Après 30 s d’imagerie, ajoutez 100 nM ADP ou le contrôle du véhicule. Continuez à imager jusqu’à ce que le signal revienne à la ligne de base, ~2 min. Une augmentation transitoire de ~2 fois de la fluorescence GCaMP avec le traitement ADP indique une transduction réussie et une fonction de biocapteur. Pour une estimation plus précise de l’efficacité de la transduction, répétez le test d’agoniste avec imagerie en utilisant des monocouches générées par le processus décrit dans la partie 3.
3. Préparation des monocouches HIO pour l’imagerie par fluorescence en direct
- Enduisez tous les puits d’une lame de chambre inférieure d’imagerie à 10 puits avec du collagène IV. Pour ce faire, mélangez 34 μL de 1 mg/mL de collagène IV avec 960 μL d’eau déminéralisée stérile. Ajouter 95 μL de solution IV de collagène dilué dans chaque puits et incuber à 37 °C pendant 0,5 à 2 h.
- Retirer le milieu d’entretien WRNE de 4 puits d’OIE 3D. Les DOI devraient s’écouler de 5 à 7 jours à compter de leur dernier passage.
REMARQUE : 1 plaque de 10 puits nécessite généralement ~1,25 x 106 cellules. Cela nécessite généralement 2 à 4 puits d’HIO 3D plaqués dans 30 μL de BMM chacun, mais varient en fonction de la densité. - Ajouter 500 μL de 1x PBS + 0,5 mM EDTA par puits. À l’aide d’un embout pré-enduit de 1 ml, pipeter doucement de haut en bas pour détacher le BMM de la plaque. Transférez la suspension dans un tube conique pré-enduit de 15 mL, en combinant des puits similaires dans le même tube.
- Rincez chaque puits avec 500 μL supplémentaires de PBS + 0,5 mM d’EDTA. Centrifuger à 300 x g pendant 5 min à 4 °C. Retirez le surnageant et le BMM résiduel.
- À l’aide d’un embout pré-enduit, remettre en suspension le reste de la pastille dans 3 mL de PBS + 5 mM d’EDTA (notez que c’est 10x plus d’EDTA que le premier lavage).
- Centrifuger à 300 x g pendant 5 min à 4 °C. Retirez le surnageant et le BMM résiduel. Remettre la pastille en suspension dans 2 mL de tampon de dissociation enzymatique.
- Incuber dans un bain-marie à 37 °C pendant 5 min. Ajouter 3 mL de CMGF- + 10 % FBS et pipeter doucement pour mélanger.
- Centrifuger à 300 x g pendant 5 min à 4 °C. Retirez le surnageant. Ajouter 1 mL de CMGF-.
- Pipeter vigoureusement de haut en bas avec un embout pré-enduit 80x-100x pour décomposer mécaniquement les HIO en cellules individuelles. Centrifuger à 300 x g pendant 5 min à 4 °C. Enlever le surnageant.
- Remettre en suspension 1 mL de WRNE + 10 μM Y-27632. Obtenir un nombre de cellules pour la suspension de 1 mL.
- Diluer la suspension cellulaire avec WRNE + 10μM Y-27632 pour obtenir une concentration de 1,25 x 105 cellules/100 μL (1,25 x 106 cellules/mL).
- À l’aide d’une pipette de 200 μL, retirez la solution de collagène de la plaque préparée à l’étape 1, car le collagène s’est maintenant déposé au fond du puits. Évitez de toucher le fond du puits avec la pointe de la pipette.
- À l’aide d’une pointe de pipette pré-enduite de 200 μL, ajouter 100 μL de la solution cellulaire de l’étape 3.11 (1,25 x 105 cellules) par puits.
- Incuber pendant 24 h dans un incubateur de culture cellulaire à 37 °C. Après 24 h, retirer le milieu de culture de tous les puits et le remplacer par 100 μL de milieu de différenciation par puits.
REMARQUE : À ce stade, les cellules devraient adhérer à la plaque. Notez que la monocouche ne sera probablement pas confluente ou entièrement plane. - Replacez la lame dans l’incubateur de culture cellulaire à 37 °C. Rafraîchir le milieu de différenciation toutes les 24 h jusqu’à ce que la monocouche soit confluente. Cela nécessite généralement 3 à 5 jours à partir du placage. Après ce point, les monocouches sont prêtes pour les applications en aval.
4. Infection virale des monocouches HIO
- Préparez l’inoculum du virus. Si nécessaire, la trypsine active le stock de virus. Dans le cas du rotavirus, ajouter 10 μg/mL de trypsine de Worthington aux stocks de virus et incuber à 37 °C pendant 1 h.
- Diluez le stock de virus activé en utilisant l’une des deux méthodes suivantes.
- Si vous visez le nombre maximal de cellules infectées en utilisant une seule souche virale, mélangez 50 μL de souche virale activée avec 50 μL de CMGF-.
- Si vous comparez plusieurs souches, préparez les inoculums pour obtenir des multiplicités d’infection équivalentes. Utilisez la formule suivante pour déterminer le volume de stock de virus par puits nécessaire pour le moment d’inertie souhaité. Ajouter CMGF- au volume de stock de virus calculé pour obtenir un volume final de 100 μL par puits.
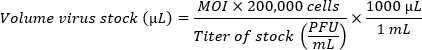
REMARQUE : Une seule monocouche HIO plaquée dans un puits d’un diamètre de 5,46 mm (plaque à 96 puits) contient environ 200 000 cellules. En raison de la faible efficacité de l’infection dans les monocouches, un moment d’inertie élevé (100 à 1 million de particules virales par cellule) est préférable. Le moment d’inertie optimal doit être déterminé empiriquement.
- Infectez les monocouches en retirant délicatement le milieu de la monocouche HIO à l’aide d’une pipette de 200 ml. Remplacer par 100 mL d’inoculum viral ou d’inoculum simulé.
- Étant donné que la plupart des souches de rotavirus se propagent dans des cellules MA104, utiliser un lysat de cellules MA104 non infectées pour l’inoculum simulé. Utiliser un volume équivalent au volume de stock de virus utilisé pour les puits infectés et ajouter CMGF- pour un volume final de 100 mL par puits.
- Pour les virus qui infectent les cellules par voie basolatérale, comme le rotavirus14, utilisez une aiguille 25G pour marquer la monocouche. Il est plus facile de faire une seule entaille sur toute la longueur de la monocouche, du bas vers le haut du puits. Appuyez doucement l’aiguille dans la monocouche en biseau vers le haut, dans le sens opposé au mouvement, et faites-la glisser sur toute la longueur de la monocouche pour créer une cicatrice (Figure 2A).
- Incuber dans un incubateur à 37 °C pendant 2 h. Éliminez le virus et simulez l’inoculum. Lavez les monocouches une fois avec 1x PBS.
- Ajouter 100 μL de milieu de différenciation sans rouge de phénol. Placez la lame dans un incubateur à 37 °C jusqu’à ce qu’elle soit prête à être photographiée. La fenêtre d’imagerie optimale variera en fonction de la cinétique de l’infection virale. Dans le cas du rotavirus, commencer l’imagerie 6 à 8 h après l’infection.
- Diluez le stock de virus activé en utilisant l’une des deux méthodes suivantes.
5. Imagerie Ca2+ des monocouches infectées
- Préchauffez l’incubateur à 37 °C. Faire fonctionner le CO2 humidifié à 0,02 L/min. Placez la lame contenant les monocouches infectées dans la chambre de l’incubateur et scellez le couvercle.
- À l’aide de l’éclairage à fond clair (BF) et d’un objectif 20x, sélectionnez les coordonnées X et Y des champs de vision de la monocouche qui sera imagée. Il est préférable de sélectionner les points en tenant compte de la région marquée, car la plupart des cellules infectées se trouveront près de la rayure.
- Optimisez les paramètres d’imagerie et acquérez une image à l’aide d’une excitation de 488 nm. Pour minimiser la phototoxicité, réglez la source lumineuse à 50 % de puissance avec un temps d’exposition de 50 ms. Les paramètres d’acquisition appropriés peuvent varier et doivent être optimisés en acquérant plusieurs acquisitions. Assurez-vous qu’aucun pixel n’est saturé. Ajustez le temps d’exposition et la puissance lumineuse selon vos besoins pour vous assurer que toute la plage dynamique du capteur de calcium fluorescent peut être détectée.
- Si vous utilisez d’autres fluorophores (par exemple, des virus marqués par fluorescence), répétez l’optimisation sur ces canaux. Configurez la boucle d’imagerie et acquérez une image de 488 nm à chaque coordonnée X, Y sélectionnée dans une boucle de 1 min. Imagez cette boucle en continu. Lors de l’utilisation d’un virus marqué par fluorescence, acquérez une image sur le canal approprié toutes les 10e boucle (c’est-à-dire 1 image/10 min).
- Collectez des images pendant ~18 h. Si vous utilisez des virus sans marqueurs fluorescents, fixez les monocouches et effectuez une coloration par immunofluorescence pour identifier les cellules infectées. Effectuez cette opération après avoir terminé l’exécution de l’imagerie comme décrit ci-dessous.
- Retirez le support d’imagerie et remplacez-le par 100 μL de formaldéhyde à 4 % dans 1x PBS. Incuber à température ambiante pendant 30 min.
- Retirer le fixateur et tremper avec 100 μL de 50mM NH4Cl pendant 10 min. Retirer NH4Cl. Perméabiliser les monocouches avec 100 μL de Triton X-100 à 0,1% dans 1x PBS pendant 1 h à température ambiante ou toute la nuit à 4 °C.
- Supprimer le tampon de perméabilisation. Bloquer avec 100 μL d’albumine sérique bovine à 3 % dans 1x PBS pendant 1 h.
- Retirez la solution bloquante. Ajouter les anticorps primaires dilués à la concentration recommandée/optimisée dans 1x PBS. Ajouter 100 μL de solution d’anticorps par puits.
- Incuber toute la nuit à 4 °C en berçant doucement. Retirez les anticorps primaires. Laver 3x avec 1x PBS.
- Ajouter des anticorps secondaires conjugués à la fluorescence dilués à la concentration recommandée/optimisée dans 1x PBS. Ajouter 100 μL par puits. Incuber pendant 2 h à température ambiante.
REMARQUE : FPBase15 est une excellente ressource pour vous aider à sélectionner des secondaires qui éviteront les débordements lors du multiplexage. La plupart des produits secondaires sont efficaces à une dilution de 1 :1000 dans 1x PBS. - Retirez les anticorps secondaires. Préparez une solution DAPI de 1 μg/mL dans 1x PBS et ajoutez 100 μL par puits. Incuber pendant 20 min. Retirer le DAPI. Laver 3x avec 1x PBS.
- Conservez les monocouches fixes dans 100 μL de PBS 1x pour l’imagerie. Replacez la lame sur la platine du microscope, rechargez les coordonnées X et Y de l’exécution d’imagerie en direct et imagez chaque multipoint sur le canal qui correspond à l’anticorps secondaire qui a été utilisé pour la coloration. Référencez les images de l’exécution d’imagerie en direct pour vous assurer que les mêmes points sont capturés.
6. Quantification des ondes calciques intercellulaires
- Assurez-vous que Fidji est Just ImageJ (FIJI) est installé avec les plug-ins suivants : Bio-Formats (doit être préinstallé dans FIJI mais ne sera pas dans ImageJ) ; Livre de recettes : Pour l’installer, à partir du menu FIJI, accédez à Aide > mises à jour > Gérer les sites de mise à jour. Consultez le livre de recettes. Redémarrez FIJI.
- Divisez le fichier de données de l’exécution d’imagerie en direct en plusieurs fichiers, en séparant chaque coordonnée X, Y. Dans Nikon NES Elements, sélectionnez Fichier > Importer/Exporter > Fractionner les multipoints. Sélectionnez un dossier à exporter et un préfixe approprié.
- Ouvrez FIDJI. Chargez un seul fichier à partir des multipoints fractionnés. Si vous utilisez une image multicouche, divisez les couches. Sélectionnez la fenêtre avec les images du canal 488 nm (GCaMP).
- Exécutez la fonction DeltaF vers le haut pour déterminer la variation de la valeur de chaque pixel d’un point temporel à l’autre. Pour ce faire, sélectionnez Cookbook > T-functions > Delta F Up.
- Faites défiler la pile jusqu’à ce qu’une image avec une onde de calcium soit trouvée. Avec l’onde calcique en vue, définissez un seuil en cliquant sur Image > Ajuster > seuil. Ajustez la limite inférieure pour minimiser la quantité de signal qui ne fait pas partie de l’onde qui dépasse le seuil. Pour les images 16 bits acquises à l’aide des paramètres décrits ci-dessus, commencez par un seuil inférieur de 600 et ajustez-le si nécessaire.
- Exécutez l’analyseur de particules pour segmenter les ondes en cliquant sur Analyser > Analyser les particules. Définissez la taille minimale de l’onde en μm2 en ajustant la plage. Il est défini sur 0-infinity à la ligne de base. Pour les images de cellules MA104 acquises à l’aide d’un objectif 20x, commencez par augmenter la limite inférieure à 10 000 μm2 et ajustez au besoin.
- Cochez les cases Afficher les résultats et Effacer les résultats. Dans le menu déroulant « Afficher », sélectionnez Contours, puis cliquez sur OK. Cela devrait se traduire par 2 sorties : Une fenêtre, Résultats, répertoriera chaque onde détectée, la zone de l’onde et ses valeurs d’intensité moyenne, minimale et maximale (en fonction du delta F). L’autre fenêtre, intitulée Dessin de [nom du fichier], comprendra une nouvelle pile avec les contours des vagues segmentées. Utilisez-le pour vérifier la détection des vagues. Ajustez les valeurs seuils et la plage de tailles si nécessaire, et vérifiez à nouveau les appels d’onde.
- Pour le traitement par lots, utilisez le script de macro inclus (Fichier de codage supplémentaire 1). Pour l’utiliser, suivez les étapes décrites ci-dessous.
- Divisez les points multiples en fichiers uniques comme décrit à l’étape 6.2. Ouvrez FIDJI. Sélectionnez Traiter > Batch > Macro. Dans la zone « entrée », fournissez la carte au dossier contenant les fichiers multipoints fractionnés. Laissez la case « sortie » vide.
- Collez le script du fichier de codage supplémentaire 1 dans la zone. Ajustez le seuil d’intensité et le seuil de taille dans le script pour refléter les paramètres optimisés des étapes 6.5 et 6.6.
- Cliquez sur Traiter. En fonction du nombre de multipoints et de la vitesse de traitement de l’ordinateur, le traitement de toutes les images peut prendre 30 minutes ou plus. Une fois terminé, les données seront écrites dans un nouveau fichier de feuille de calcul nommé « Renommez-moi après l’écriture » sur le bureau. Le fichier de sortie doit contenir, pour chaque multipoint, un nombre de vagues et des mesures de vagues (surface, intensité moyenne, intensité minimale et maximale et densité d’intensité).
Résultats
La figure 1A montre un dôme BMM contenant des organoïdes intestinaux humains en 3 dimensions qui ont été transduits pour exprimer de manière stable les GCaMP6. La figure 1B montre la même lignée d’organoïde replaqué en monocouche à 24, 48 et 72 h après le semis. Pour valider la fonction des GCaMP6s, la monocouche a été imagée par microscopie à fluorescence toutes les 2 s pendant 4 min, et 100 nM d’ADP ont été ajoutés au milieu après ~20 s....
Discussion
Les altérations des taux cytosoliques de Ca2+ peuvent être à la fois une cause et un effet de pathologies au sein de l’épithélium 10,16,17. L’augmentation du calcium cytosolique peut entraîner directement la sécrétion via l’activation du canal chlorure dépendant du calcium TMEM16A18,19. L’activation de TMEM16A en réponse au Ca2+ pe...
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents à divulguer.
Remerciements
Ces travaux ont été financés par des subventions R01DK115507 et R01AI158683 (PI : J. M. Hyser) des National Institutes of Health (NIH). Le soutien aux stagiaires a été fourni par des subventions des NIH F30DK131828 (PI : J.T. Gebert), F31DK132942 (PI : F. J. Scribano) et F32DK130288 (PI : K.A. Engevik). Nous tenons à remercier le Texas Medical Center Digestive Diseases Enteroid Core pour avoir fourni le milieu d’entretien des organoïdes.
matériels
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
Références
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon