É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de cálcio vivo de monocamadas organoides intestinais humanas infectadas por vírus usando indicadores de cálcio codificados geneticamente
Neste Artigo
Resumo
Este protocolo descreve uma abordagem para a realização de imagens de cálcio em organoides intestinais humanos infectados por vírus e oferece uma abordagem para análise.
Resumo
A sinalização de cálcio é um regulador integral de quase todos os tecidos. Dentro do epitélio intestinal, o cálcio está envolvido na regulação da atividade secretora, dinâmica da actina, respostas inflamatórias, proliferação de células-tronco e muitas outras funções celulares não caracterizadas. Como tal, o mapeamento da dinâmica de sinalização do cálcio dentro do epitélio intestinal pode fornecer informações sobre os processos celulares homeostáticos e revelar respostas únicas a vários estímulos. Os organoides intestinais humanos (HIOs) são um modelo de alto rendimento, derivado do ser humano, para estudar o epitélio intestinal e, portanto, representam um sistema útil para investigar a dinâmica do cálcio. Este artigo descreve um protocolo para transduzir HIOs de forma estável com indicadores de cálcio codificados geneticamente (GECIs), realizar microscopia de fluorescência ao vivo e analisar dados de imagem para caracterizar significativamente os sinais de cálcio. Como um exemplo representativo, HIOs 3-dimensionais foram transduzidos com lentivírus para expressar de forma estável GCaMP6s, um GECI citosólico baseado em proteína fluorescente verde. Os HIOs projetados foram então dispersos em uma suspensão de célula única e semeados como monocamadas. Após a diferenciação, as monocamadas de HIO foram infectadas com rotavírus e/ou tratadas com drogas conhecidas por estimular uma resposta de cálcio. Um microscópio de epifluorescência equipado com uma câmara de imagem viva umidificada e com temperatura controlada permitiu a obtenção de imagens de longo prazo de monocamadas infectadas ou tratadas com drogas. Após a aquisição de imagem, as imagens adquiridas foram analisadas usando o software de análise disponível gratuitamente, ImageJ. De modo geral, este trabalho estabelece um pipeline adaptável para caracterizar a sinalização celular em HIOs.
Introdução
O cálcio é um segundo mensageiro amplamente conservado que desempenha um papel crítico na regulação da fisiologia celular1. Dada a sua forte carga, tamanho pequeno e alta solubilidade em condições fisiológicas, o cálcio é um manipulador ideal da conformação proteica. Isso torna o cálcio um meio poderoso para transduzir sinais eletroquímicos em alterações enzimáticas, transcricionais ou pós-transcricionais. Os rígidos gradientes de concentração de cálcio através do retículo endoplasmático (RE) e membranas plasmáticas criam uma alta força motriz que permite mudanças rápidas na concentração citosólica de cálcio. Vários mecanismos, incluindo buffering e transporte ativo, mantêm firmemente esse gradiente. Embora necessária para as funções celulares normais, essa manutenção é energeticamente cara, tornando-a particularmente suscetível em estados de estresse 2.
Como tal, a desregulação do cálcio dentro do citosol é um sinal quase universal de muitos tipos de estresse celular. Distúrbios metabólicos, toxinas, patógenos, danos mecânicos e perturbações genéticas podem interromper a sinalização do cálcio. Independentemente do estímulo, em nível de células inteiras, elevações sustentadas e descontroladas do cálcio citosólico podem promover apoptose e, eventualmente, necrose 3,4. Alterações nos níveis citosólicos de cálcio de menor amplitude ou maior frequência, entretanto, têm efeitos variáveis2. Da mesma forma, os resultados das flutuações do cálcio podem diferir com base no microdomínio espacial em que ocorrem5. O monitoramento dos níveis de cálcio pode, portanto, oferecer informações sobre processos dinâmicos de sinalização, mas isso requer amostragem com resolução temporal e espacial relativamente alta.
Indicadores de cálcio codificados geneticamente (GECIs) são ferramentas poderosas para amostragem contínua em sistemas de células vivas6. Algumas das GECIs mais utilizadas são as proteínas fluorescentes responsivas ao cálcio baseadas em GFP, conhecidas como GCaMPs7. A GCaMP canônica é uma fusão de três domínios proteicos distintos: uma GFP circularmente permutada (cpGFP), calmodulina e M136. O domínio da calmodulina sofre uma mudança de conformação ao se ligar ao cálcio, permitindo sua interação com M13. A interação calmodulina-M13 induz uma mudança conformacional na cpGFP que aumenta sua emissão fluorescente após excitação. Como tal, um aumento na concentração de cálcio correlaciona-se com um aumento na intensidade de fluorescência do GCaMP. Esses sensores podem ser citosólicos ou direcionados para organelasespecíficas8.
Semelhante à maioria dos tecidos, o cálcio regula uma variedade de funções dentro do epitélio gastrointestinal. O epitélio intestinal é essencial para a absorção de nutrientes e fluidos, mas também deve formar uma barreira apertada e interface imunológica para evitar a invasão de patógenos ou insultos tóxicos. As vias dependentes de cálcio influenciam quase todas essas funções vitais 9,10,11. No entanto, a sinalização do cálcio dentro do epitélio intestinal permanece uma fronteira pouco explorada com potencial promissor como alvo terapêutico. Enquanto o monitoramento da dinâmica do cálcio no epitélio intestinal in vivo continua a apresentar desafios, os organoides intestinais humanos (HIOs) oferecem um sistema ex vivo adaptável para experimentação12. As HIOs são esferoides tridimensionais (3D) derivadas de células-tronco intestinais humanas e, ao serem diferenciadas, recapitulam grande parte da diversidade celular do epitélio intestinal nativo12.
Este protocolo descreve métodos abrangentes para projetar HIOs que expressam GECIs e, em seguida, preparar HIOs projetados como monocamadas para imagens de cálcio de células vivas. Ele oferece a infecção viral como um exemplo de manipulação patológica que interrompe a sinalização de cálcio e fornece uma abordagem analítica para quantificar essas alterações.
Protocolo
Todos os organoides intestinais humanos (HIOs) utilizados neste protocolo e os experimentos representativos foram derivados de tecido humano obtido e mantido pelo Texas Medical Center Digestive Diseases Enteroid Core. Todas as amostras foram coletadas de acordo com um protocolo aprovado pelo Comitê de Ética em Pesquisa da Baylor College of Medicine.
1. Preparação de materiais e reagentes
- Para manutenção dos organoides, coletar placas de 24 poços tratadas com cultura celular, matriz de membrana basal (BMM), tubos cônicos de 15 mL e tubos cônicos de 1,5 mL.
- Para preparar meios completos sem fatores de crescimento (CMGF-), a 500 mL de DMEM F12 avançado adicionar 5 mL de 1M HEPES, 5 mL de 100x antibiótico-antimicótico, 5 mL de suplemento de glutamina 100x.
- Para preparar os meios contendo Wnt, R-spondina e Noggin (WRNE), misture partes iguais de meios condicionados com CMGF e Wnt, adicione meio condicionado com Noggin, 10% em volume, meio condicionado com R-espondina, 20% em volume, fator de crescimento epidérmico humano de 50 ng/mL, nicotinamida 10 mM, [Leu15]-Gastrina I 10 nM, A-83-01 nM, SB202190 de 10 μM, 1x suplemento B27, 1x suplemento de N2 e 1 mM de N-acetilcisteína.
- Para transdução lentiviral, preparar Soro Fetal Bovino (SFB), Tripsina-EDTA 0,05% em solução salina tamponada com fosfato (PBS) 1x, CMGF- com 10% FBS, Estéril 1x PBS, Polibreno, 10 μM Y-27632, Lentivírus, WRNE Alto Wnt + 10 μM Y-27632
- Para gerar monocamadas organoides, preparar lâmina de cultura celular de 10 poços com fundo de vidro, FBS, Colágeno IV (1 mg/mL em (di)H2O desionizado), matriz de membrana basal, EDTA 0,5 mM em 1x PBS, EDTA 5 mM em 1x PBS, tampão de dissociação enzimática, CMGF- com 10% FBS, WRNE + 10 μM Y-27632.
- Para infecção viral de monocamadas organoides, preparar tripsina, estoque de rotavírus, CMGF-, agulha 25G, Sterile 1x PBS e meios de diferenciação livres de fenol vermelho.
- Para preparar o meio de diferenciação livre de vermelho fenólico, tomar 500 mL de meio de cultura de células livres de vermelho fenol, adicionar 5 mL de aminoácidos não essenciais 100x MEM, 5 mL de 100x L-Glutamina, 5 mL de piruvato de sódio 100 nM e 7,5 mL de HEPES 1M.
- Para coloração por imunofluorescência de organoides, preparar formaldeído a 4% (formaldeído a 16% diluído em 1x PBS), Triton X-100 (Triton X-100 a 0,1% em 1x PBS), albumina de soro bovino (albumina de soro bovino a 3% em 1x PBS), solução de NH4Cl (50 mM), DAPI (1 μg/mL de solução DAPI em 1x PBS).
2. Engenharia de organoides para expressar sensores de cálcio codificados geneticamente
NOTA: Este protocolo descreve os passos para transduzir um único poço de organoides intestinais humanos tridimensionais plaqueados em 30 μL de Matriz de Membrana Basal (BMM) em uma placa de 24 poços13. A maioria das linhas conterá cerca de 400.000 células por poço. Um segundo poço, não transduzido, deve ser incluído como controle. Mantenha todos os reagentes e suspensões celulares no gelo.
- Após 2-5 dias da última passagem, remova o meio WRNE de manutenção de dois poços de HIOs. Substitua por 300 μL de 0,05% de tripsina-EDTA por poço e pipete suavemente para cima e para baixo 5x para separar o BMM da placa. Colocar numa incubadora a 37 °C durante 4 min.
- Adicionar 500 μL de CMGF- + 10% FBS por poço. Pré-revestir uma ponta de pipeta de baixa ligação de 1 mL com FBS pipetando 1 mL de FBS para cima e para baixo 2x. O FBS pode ser reutilizado em várias pontas. Com a ponta pré-revestida, pipetar os HIOs para cima e para baixo 10x.
- Transfira o conteúdo de cada poço para seu próprio tubo de microcentrífuga pré-revestido de 1,5 mL. Enxaguar cada poço com mais 500 μL de CMGF- e adicionar a lavagem ao respectivo tubo.
- Centrifugar os tubos a 100 x g em uma centrífuga de balde oscilante por 5 min a 4 °C. Remova o sobrenadante e qualquer BMM residual.
- Ressuspender em 1 mL de PBS 1x. Divida cada tubo em dois tubos de microcentrífuga para 4 tubos totais. Centrifugar os tubos a 100 x g durante 5 minutos a 4 °C. Remover sobrenadante.
- Ressuspender cada tubo uma vez adicional em 1x PBS e centrifugar a 100 x g por 5 min a 4 °C.
- Preparar 400 μL de meio de transdução e meio controle (Tabela 1). Ressuspender 2 tubos em 200 μL do meio controle e 2 tubos em 200 μL do meio de transdução.
- Incubar em estufa de cultura celular a 37 °C durante 24 horas. Ressuspenda pipetando para cima e para baixo com uma ponta revestida periodicamente (por exemplo, 2 h pós-transdução (hpt), 12 hpt, 18 hpt) para incentivar a transdução uniforme.
- Após 24 h, tubos de centrifugação a 100 x g por 5 min a 4 °C. Remover sobrenadante.
- Ressuspenda o pellet em 500 μL de 1x PBS para lavar. Centrifugar a 100 x g durante 5 min a 4 °C. Remover sobrenadante.
- Usando uma ponta de pipeta gelada de 200 μL, ressuspenda cada um dos 4 pellets em 30 μL de BMM. Pipetar suavemente para cima e para baixo para dispersar uniformemente.
- Coloque o conteúdo de cada tubo em seu próprio poço em uma placa de 24 poços. Incubar durante 10 min a 37 °C para permitir que o BMM se solidifique antes de adicionar 500 μL de HighWnt WRNE + 10μM Y-27632.
- Permitir que os HIOs transduzidos cresçam por 1 semana, atualizando a mídia (HighWnt WRNE + 10 μM Y-27632) a cada dois dias. Após 1 semana, verificar a expressão do indicador fluorescente por microscopia. Se o sinal for forte, comece a seleção de drogas. Se o sinal for fraco, repita a transdução conforme descrito acima.
- Uma vez estabelecida a linha, verifique a função do indicador de fluorescência via tratamento agonista. O ADP 100nM é um agonista confiável para validação do GCaMP. Teste organoides 3D para validação inicial, conforme descrito abaixo.
- Após uma passagem, pranche um poço (ou múltiplo) de organoides em BMM em uma placa de fundo de imagem separada. Placa os organoides em cerca de 1/3 da densidade normal para evitar sobreposição excessiva durante a imagem. Não continue a passar por esses HIOs após o tratamento com agonistas, pois é difícil garantir a esterilidade.
- Aguarde 2-3 dias para que os HIOs se recuperem após a passagem. Antes da aquisição de imagens, mude o meio para o meio de diferenciação livre de fenol vermelho.
- Usando um microscópio fluorescente, montou uma corrida de 3 minutos com imagens adquiridas a cada 5 s usando excitação de 488 nm e um conjunto de filtros FITC/GFP. Após 30 s de imagem, adicione 100 nM ADP ou controle do veículo. Continue a criação de imagens até que o sinal retorne à linha de base próxima, ~2 min. Um aumento transitório de ~2 vezes na fluorescência do GCaMP com tratamento com ADP indica transdução bem-sucedida e função do biossensor. Para uma estimativa mais precisa da eficiência da transdução, repita o teste agonista com imagens usando monocamadas geradas através do processo descrito na parte 3.
3. Preparação de monocamadas HIO para imagens de fluorescência ao vivo
- Revestir todos os poços de uma lâmina de câmara inferior de imagem de 10 poços com Colágeno IV. Para isso, misture 34 μL de 1 mg/mL de Colágeno IV com 960 μL de água deionizada estéril. Adicionar 95 μL de solução de colágeno IV diluída a cada poço e incubar a 37 °C por 0,5-2 h.
- Remova o meio de manutenção WRNE de 4 poços de HIOs 3D. HIOs devem ser 5-7 dias a partir de sua última passagem.
NOTA: 1 placa de 10 poços normalmente requer ~1,25 x 106 células. Isso normalmente requer 2-4 poços de HIOs 3D banhados em 30 μL de BMM cada, mas varia de acordo com a densidade. - Adicionar 500 μL de 1x PBS + 0,5 mM EDTA por poço. Com uma ponta pré-revestida de 1 mL, pipete para cima e para baixo suavemente para desprender o BMM da placa. Transfira a suspensão para um tubo cônico pré-revestido de 15 mL, combinando como poços no mesmo tubo.
- Enxaguar cada poço com 500 μL adicionais de PBS + 0,5 mM EDTA. Centrifugar a 300 x g durante 5 min a 4 °C. Remova o sobrenadante e o BMM residual.
- Usando uma ponta pré-revestida, ressuspenda o pellet restante em 3 mL de PBS + 5 mM EDTA (note que isso é 10x mais EDTA do que a primeira lavagem).
- Centrifugar a 300 x g durante 5 min a 4 °C. Remova o sobrenadante e o BMM residual. Ressuspender o pellet em 2 mL de tampão de dissociação enzimática.
- Incubar em esferas a 37 °C/banho-maria durante 5 min. Adicione 3 mL de CMGF- + 10% FBS e pipetar suavemente para misturar.
- Centrifugar a 300 x g durante 5 min a 4 °C. Remova o sobrenadante. Adicionar 1 mL de CMGF-.
- Pipete vigorosamente para cima e para baixo com uma ponta pré-revestida 80x-100x para quebrar mecanicamente HIOs em células únicas. Centrifugar a 300 x g durante 5 min a 4 °C. Remover sobrenadante.
- Ressuspender em 1 mL de WRNE + 10 μM Y-27632. Obter uma contagem de células para a suspensão de 1 mL.
- Diluir a suspensão celular com WRNE + 10μM Y-27632 para atingir uma concentração de 1,25 x 105 células/100 μL (1,25 x 106 células/mL).
- Usando uma pipeta de 200 μL, remova a solução de colágeno da placa preparada na etapa 1, pois o colágeno já se instalou no fundo do poço. Evite tocar o fundo do poço com a ponta da pipeta.
- Usando uma ponteira de pipeta pré-revestida de 200 μL, adicionar 100 μL da solução celular da etapa 3.11 (1,25 x 105 células) por poço.
- Incubar durante 24 h numa estufa de cultura celular a 37 °C. Após 24 h, retirar o meio de cultura de todos os poços e substituí-lo por 100 μL de meio de diferenciação por poço.
OBS: Neste ponto, as células devem estar aderidas à placa. Observe que a monocamada provavelmente não será confluente ou totalmente plana. - Coloque a lâmina de volta na incubadora de cultura de células a 37 °C. Atualizar o meio de diferenciação a cada 24 h até que a monocamada seja confluente. Isso geralmente requer 3-5 dias de chapeamento. Após esse ponto, as monocamadas estão prontas para aplicações downstream.
4. Infecção viral das monocamadas HIO
- Prepare o inóculo do vírus. Se necessário, a tripsina ativa o estoque de vírus. Para rotavírus, adicionar 10 μg/mL de tripsina de Worthington aos estoques de vírus e incubar a 37 °C por 1 h.
- Dilua o estoque de vírus ativado usando um dos dois métodos a seguir.
- Se almejar o número máximo de células infectadas usando apenas uma cepa viral, misture 50 μL do estoque viral ativado com 50 μL de CMGF-.
- Se comparar múltiplas cepas, prepare os inóculos para alcançar multiplicidades equivalentes de infecção (MOIs). Use a fórmula a seguir para determinar o volume de estoque de vírus por poço necessário para o MOI desejado. Adicione CMGF- ao volume de estoque de vírus calculado para atingir um volume final de 100 μL por poço.
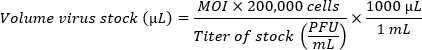
NOTA: Uma única monocamada HIO banhada em um poço com um diâmetro de 5,46 mm (placa de 96 poços) contém cerca de 200.000 células. Devido à baixa eficiência da infecção em monocamadas, um alto MOI (100-1 milhão de partículas virais por célula) é preferível. O MOI ótimo deve ser determinado empiricamente.
- Infecte as monocamadas removendo suavemente o meio da monocamada HIO usando uma pipeta de 200 mL. Substitua por 100 mL de inóculo viral ou inóculo simulado.
- Como a maioria dos estoques de rotavírus é propagada em células MA104, use um lisado celular MA104 não infectado para o inóculo simulado. Use um volume equivalente ao volume de estoque de vírus usado para os poços infectados e adicione CMGF- para um volume final de 100 mL por poço.
- Para vírus que infectam células basolateralmente, como o rotavírus14, use uma agulha 25G para pontuar a monocamada. É mais fácil fazer uma única pontuação em todo o comprimento da monocamada, da base ao topo do poço. Pressione suavemente a agulha na monocamada em um ângulo com o bisel para cima, oposto à direção do movimento, e arraste-a por todo o comprimento da monocamada para criar uma cicatriz (Figura 2A).
- Incubar numa incubadora a 37 °C durante 2 h. Remova o vírus e simule o inóculo. Lave as monocamadas uma vez com 1x PBS.
- Adicionar 100 μL de meio de diferenciação livre de fenol vermelho. Coloque o slide em uma incubadora de 37 °C até que esteja pronto para a imagem. A janela de imagem ideal variará com base na cinética da infecção viral. Para rotavírus, comece a imagem em 6-8 h após a infecção.
- Dilua o estoque de vírus ativado usando um dos dois métodos a seguir.
5. Ca2+ Imagem de monocamadas infectadas
- Pré-aqueça a incubadora de topo de estágio a 37 °C. Executar CO2 umidificado a 0,02 L/min. Coloque a lâmina com as monocamadas infectadas na câmara da incubadora e sele a tampa.
- Usando iluminação de campo brilhante (BF) e uma objetiva de 20x, selecione as coordenadas X e Y dos campos de visão dentro da monocamada que será fotografada. É melhor selecionar pontos com a região marcada em vista, pois a maioria das células infectadas estará perto do arranhão.
- Otimize os parâmetros de imagem e adquira uma imagem usando excitação de 488 nm. Para minimizar a fototoxicidade, ajuste a fonte de luz para 50% de potência com um tempo de exposição de 50 ms. Os parâmetros de aquisição apropriados podem variar e devem ser otimizados através da aquisição múltipla de aquisições. Verifique se nenhum pixel está saturado. Ajuste o tempo de exposição e a potência da luz conforme necessário para garantir que toda a faixa dinâmica do sensor de cálcio fluorescente possa ser detectada.
- Se estiver usando outros fluoróforos (por exemplo, vírus marcados fluorescentemente), repita a otimização nesses canais. Configure o loop de imagem e adquira uma imagem de 488 nm em cada coordenada X, Y selecionada em um loop de 1 min. Imagine esse loop continuamente. Ao usar um vírus marcado fluorescentemente, adquira uma imagem no canal apropriado a cada 10º loop (ou seja, 1 imagem/10 min).
- Colete imagens por ~18 h. Se estiver usando vírus sem etiquetas fluorescentes, fixe monocamadas e realize coloração de imunofluorescência para identificar células infectadas. Execute isso depois de concluir a execução da imagem, conforme descrito abaixo.
- Remova o meio de imagem e substitua por 100 μL de formaldeído a 4% em PBS 1x. Incubar à temperatura ambiente durante 30 min.
- Remova o fixador e tempere com 100 μL de 50mM NH4Cl por 10 min. Remova NH4Cl. Permeabilize as monocamadas com 100 μL de Triton X-100 a 0,1% em 1x PBS por 1 h à temperatura ambiente ou durante a noite a 4 °C.
- Remova o tampão de permeabilização. Bloquear com 100 μL de albumina de soro bovino a 3% em PBS 1x por 1 h.
- Remova a solução de bloqueio. Adicionar anticorpos primários diluídos na concentração recomendada/otimizada em 1x PBS. Adicionar 100 μL de solução de anticorpos por poço.
- Incubar durante a noite a 4 °C com balanço suave. Remover anticorpos primários. Lave 3x com 1x PBS.
- Adicionar anticorpos secundários conjugados a fluorescência diluídos na concentração recomendada/otimizada em 1x PBS. Adicionar 100 μL por poço. Incubar durante 2 h à temperatura ambiente.
NOTA: FPBase15 é um ótimo recurso para ajudar a selecionar secundários que evitarão sangria durante a multiplexação. A maioria dos secundários é eficaz em uma diluição de 1:1000 em 1x PBS. - Remover anticorpos secundários. Fazer uma solução DAPI de 1 μg/mL em 1x PBS e adicionar 100 μL por poço. Incubar por 20 min. Lave 3x com 1x PBS.
- Mantenha as monocamadas fixas em 100 μL de 1x PBS para obtenção de imagens. Coloque a lâmina de volta no palco do microscópio, recarregue as coordenadas X e Y da imagem ao vivo e obtenha imagens de cada multiponto no canal que corresponde ao anticorpo secundário que foi usado para coloração. Faça referência às imagens da execução de imagens ao vivo para garantir que os mesmos pontos sejam capturados.
6. Quantificação das ondas de cálcio intercelulares
- Certifique-se de que Fiji é Just ImageJ (FIJI) está instalado com os seguintes plug-ins: Bio-Formats (deve ser pré-instalado em FIJI, mas não estará no ImageJ); Livro de receitas: Para instalar, no menu FIJI, vá para Ajuda > atualizações > Gerenciar sites de atualização. Confira o livro de receitas. Reinicie o FIJI.
- Divida o arquivo de dados da execução de imagens ao vivo em vários arquivos, separando cada coordenada X, Y. No Nikon NES Elements, selecione Arquivo > Importar/Exportar > Dividir Multipontos. Selecione uma pasta para exportação e um prefixo apropriado.
- Abra FIJI. Carregue um único arquivo a partir dos multipontos divididos. Se estiver usando uma imagem multicanal, divida os canais. Selecione a janela com as imagens do canal de 488 nm (GCaMP).
- Execute a função DeltaF Up para determinar a alteração em cada valor de pixel de um ponto de tempo para o próximo. Para fazer isso, selecione Livro de receitas > funções T > Delta F Up.
- Percorra a pilha até encontrar uma imagem com uma onda de cálcio. Com a onda de cálcio em exibição, defina um limite clicando em Imagem > Ajustar > Limite. Ajuste o limite inferior para minimizar a quantidade de sinal que não faz parte da onda que a faz passar do limite. Para imagens de 16 bits adquiridas usando os parâmetros descritos acima, comece com um limite inferior de 600 e ajuste conforme necessário.
- Execute o analisador de partículas para segmentar ondas clicando em Analisar > Analisar partículas. Defina o tamanho mínimo da onda em μm2 ajustando o intervalo. Ele é definido como 0-infinito na linha de base. Para imagens de células MA104 adquiridas usando uma objetiva de 20x, comece aumentando o limite inferior para 10.000 μm2 e ajuste conforme apropriado.
- Marque as caixas Exibir resultados e Limpar resultados. No menu suspenso "Mostrar", selecione Contornos e clique em OK. Isso deve resultar em 2 saídas: Uma janela, Resultados, listará cada onda detectada, a área da onda e seus valores de intensidade média, mínima e máxima (com base no delta F). A outra janela, intitulada Desenho de [nome do arquivo], incluirá uma nova pilha com os contornos de ondas segmentadas. Use isso para verificar a detecção de onda. Ajuste os valores de limite e o intervalo de tamanho, se necessário, e verifique novamente as chamadas de onda.
- Para processamento em lote, use o script de macro incluído (Arquivo de codificação suplementar 1). Para utilizar isso, siga as etapas descritas abaixo.
- Divida vários pontos em arquivos únicos, conforme descrito na etapa 6.2. Abra FIJI. Selecione Processo > Lote > Macro. Na caixa "entrada", forneça o mapa para a pasta que contém os arquivos multiponto divididos. Deixe a caixa "saída" vazia.
- Cole o script do Arquivo de Codificação Suplementar 1 na caixa. Ajuste o limite de intensidade e o limite de tamanho no script para refletir os parâmetros otimizados das etapas 6.5 e 6.6.
- Clique em Processar. Dependendo do número de multipontos e da velocidade de processamento do computador, pode levar 30 minutos ou mais para processar todas as imagens. Depois de concluídos, os dados serão gravados em um novo arquivo de planilha chamado "Renomear-me depois de escrever" na área de trabalho. O arquivo de saída deve conter, para cada multiponto, uma contagem de ondas e métricas de onda (área, intensidade média, intensidade mínima e máxima e densidade de intensidade).
Resultados
A Figura 1A mostra uma cúpula de BMM contendo organoides intestinais humanos tridimensionais que foram transduzidos para expressar GCaMP6s de forma estável. A Figura 1B mostra a mesma linha de organoide replaqueada como monocamada 24, 48 e 72 h pós-semeadura. Para validar a função das GCaMP6s, a monocamada foi imageada por microscopia de fluorescência a cada 2 s por 4 min, e 100 nM de ADP foram adicionados ao meio após ~20 s. O ADP provoca a liberação d...
Discussão
Alterações nos níveis citosólicos de Ca2+ podem ser causa e efeito de patologias do epitélio 10,16,17. O aumento do cálcio citosólico pode direcionar diretamente a secreção via ativação do canal de cloreto cálcio-dependente TMEM16A18,19. A ativação do TMEM16A em resposta ao Ca2+ permite o efluxo apical de cloreto, estabelecendo um grad...
Divulgações
Os autores não têm interesses financeiros concorrentes a divulgar.
Agradecimentos
Este trabalho foi apoiado por subsídios R01DK115507 e R01AI158683 (PI: J. M. Hyser) do National Institutes of Health (NIH). O apoio aos estagiários foi fornecido pelos F30DK131828 (PI: J.T. Gebert), F31DK132942 (PI: F. J. Scribano) e F32DK130288 (PI: K.A. Engevik). Gostaríamos de agradecer ao Texas Medical Center Digestive Diseases Enteroid Core por fornecer o meio de manutenção de organoides.
Materiais
| Name | Company | Catalog Number | Comments |
| Advanced DMEM F12 | Gibco | 12634028 | |
| [Leu15]-Gastrin I | Sigma-Aldrich | G9145 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 0.05% Trypsin EDTA | Gibco | 25300054 | |
| 1.5mL microcentrifuge tubes | Fisherbrand | 5408137 | |
| 15mL conical tubes | Thermofisher Scientific | 0553859A | |
| 16% formaldehyde | Thermofisher Scientific | 28906 | |
| 1M HEPES | Gibco | 15630080 | |
| 1M HEPES | Gibco | 15630080 | |
| 1X PBS | Corning | 21-040-CV | |
| 25 gauge needle | Thermofisher Scientific | 1482113D | |
| A-83-01 | Tocris | 2939 | |
| ADP | Sigma-Aldrich | A2754 | |
| Advanced DMEM F12 | Gibco | 12634028 | |
| Antibiotic-antimycocytic | Gibco | 15240062 | |
| Antibiotic-antimycotic | Gibco | 15240062 | |
| B27 Supplement | Gibco | 17504-044 | |
| Bovine serum albumin | FisherScientific | BP1600100 | |
| CellView Cell Culture Slide, PS, 75/25 MM, Glass Bottom, 10 compartments | Greiner | 543979 | |
| Collagen IV | Sigma Aldrich | C5533 | |
| DAPI | Thermofisher Scientific | D1306 | |
| EDTA | Corning | 46-034-CI | |
| Fetal bovine serum | Corning | 35010CV | |
| Fetal bovine serum | Corning | 35010CV | |
| Fluorobrite | Gibco | A1896701 | |
| GlutaMAX | Gibco | 35050079 | |
| GlutaMAX | Gibco | 35050079 | |
| Human epidermal growth factor | ProteinTech | HZ-1326 | |
| Lentivirus | VectorBuilder | (variable) | |
| Matrigel | BD Biosceicen | 356231/CB40230C | |
| N2 Supplement | Gibco | 17502-048 | |
| N-acetylcysteine | Sigma-Aldrich | A9165-5G | |
| NH4Cl | Sigma-Aldrich | A9434 | |
| Nicotinamide | Sigma-Aldrich | N0636 | |
| Nunc Cell Culture Treated 24-well Plates | Thermofisher Scientific | 142475 | |
| Polybrene | MilliporeSigma | TR1003G | |
| SB202190 | Sigma-Aldrich | S70767 | |
| Triton X-100 | Fisher BioReagents | BP151100 | |
| TrypLE Express Enzyme, no phenol red | Thermofisher Scientific | 12604013 | |
| Trypsin | Worthington Biochemical | NC9811754 | |
| Y-27632 | Tocris | 1254 |
Referências
- Bootman, M. D., Bultynck, G. Fundamentals of cellular calcium signaling: A primer. Cold Spring Harb Perspect Biol. 12 (1), a038802 (2020).
- Clapham, D. E. Calcium signaling. Cell. 131 (6), 1047-1058 (2007).
- Danese, A., et al. Cell death as a result of calcium signaling modulation: A cancer-centric prospective. Biochim Biophys Acta Mol Cell Res. 1868 (8), 119061 (2021).
- Harr, M. W., Distelhorst, C. W. Apoptosis and autophagy: Decoding calcium signals that mediate life or death. Cold Spring Harb Perspect Biol. 2 (10), a005579 (2010).
- Barak, P., Parekh, A. B. Signaling through Ca2+ microdomains from store-operated CRAC channels. Cold Spring Harb Perspect Biol. 12 (7), a035097 (2020).
- Nakai, J., Ohkura, M., Imoto, K. A high signal-to-noise Ca(2+) probe composed of a single green fluorescent protein. Nat Biotechnol. 19 (2), 137-141 (2001).
- Erofeev, A. I., Vinokurov, E. K., Vlasova, O. L., Bezprozvanny, I. B. GCaMP, a family of single-fluorophore genetically encoded calcium indicators. J Evol Biochem Phys. 59 (4), 1195-1214 (2023).
- Suzuki, J., Kanemaru, K., Iino, M. Genetically encoded fluorescent indicators for organellar calcium imaging. Biophys J. 111 (6), 1119-1131 (2016).
- Nászai, M., Cordero, J. B. Intestinal stem cells: Got calcium. Curr Biol. 26 (3), R117-R119 (2016).
- Barrett, K. E. Calcium-mediated chloride secretion in the intestinal epithelium: Significance and regulation. Curr Top Membr. 53, 257-282 (2002).
- Xu, J., et al. Calcium-sensing receptor regulates intestinal dipeptide absorption via Ca2+ signaling and IKCa activation. Physiol Rep. 8 (1), e14337 (2020).
- Clevers, H. Modeling development and disease with organoids. Cell. 165 (7), 1586-1597 (2016).
- Lin, S. C., Haga, K., Zeng, X. L., Estes, M. K. Generation of CRISPR–Cas9-mediated genetic knockout human intestinal tissue–derived enteroid lines by lentivirus transduction and single-cell cloning. Nat Protoc. 17 (4), 1004-127 (2022).
- Crawford, S. E., Ramani, S., Blutt, S. E., Estes, M. K. Organoids to dissect gastrointestinal virus-host interactions: What have we learned. Viruses. 13 (6), 999 (2021).
- Lambert, T. J. FPbase: a community-editable fluorescent protein database. Nat Methods. 16 (4), 277-278 (2019).
- Lai, Y., et al. Inhibition of calcium-triggered secretion by hydrocarbon-stapled peptides. Nature. 603 (7903), 949-956 (2022).
- Chang-Graham, A. L., et al. Rotavirus induces intercellular calcium waves through ADP signaling. Science. 370 (6519), eabc3621 (2020).
- Lee, B., et al. Anoctamin 1/TMEM16A controls intestinal Cl− secretion induced by carbachol and cholera toxin. Exp Mol Med. 51 (8), 1-14 (2019).
- Saha, T., et al. Intestinal TMEM16A control luminal chloride secretion in a NHERF1 dependent manner. Biochem Biophys Rep. 25, 100912 (2021).
- Mroz, M. S., Keely, S. J. Epidermal growth factor chronically upregulates Ca2+-dependent Cl− conductance and TMEM16A expression in intestinal epithelial cells. J Physiol. 590 (8), 1907-1920 (2012).
- Sui, J., et al. Dual role of Ca2+-activated Cl− channel transmembrane member 16A in lipopolysaccharide-induced intestinal epithelial barrier dysfunction in vitro. Cell Death Dis. 11 (5), 404 (2020).
- Bellono, N. W., et al. Enterochromaffin cells are gut chemosensors that couple to sensory neural pathways. Cell. 170 (1), 185-198.e16 (2017).
- Paradis, T., Bègue, H., Basmaciyan, L., Dalle, F., Bon, F. Tight junctions as a key for pathogens invasion in intestinal epithelial cells. Int J Mol Sci. 22 (5), 2506 (2021).
- Samak, G., et al. Calcium/Ask1/MKK7/JNK2/c-Src signalling cascade mediates disruption of intestinal epithelial tight junctions by dextran sulfate sodium. Biochem J. 465 (3), 503-515 (2015).
- Deng, H., Gerencser, A. A., Jasper, H. Signal integration by Ca2+ regulates intestinal stem cell activity. Nature. 528 (7581), 212-217 (2015).
- Saurav, S., Tanwar, J., Ahuja, K., Motiani, R. K. Dysregulation of host cell calcium signaling during viral infections: Emerging paradigm with high clinical relevance. Mol Aspects Med. 81, 101004 (2021).
- Chang-Graham, A. L., et al. Rotavirus calcium dysregulation manifests as dynamic calcium signaling in the cytoplasm and endoplasmic reticulum. Sci Rep. 9 (1), 10822 (2019).
- Hyser, J. M., Collinson-Pautz, M. R., Utama, B., Estes, M. K. Rotavirus disrupts calcium homeostasis by NSP4 viroporin activity. mBio. 1 (5), e00265-e00310 (2010).
- Pham, T., Perry, J. L., Dosey, T. L., Delcour, A. H., Hyser, J. M. The Rotavirus NSP4 viroporin domain is a calcium-conducting ion channel. Sci Rep. 7, 43487 (2017).
- Crawford, S. E., Hyser, J. M., Utama, B., Estes, M. K. Autophagy hijacked through viroporin-activated calcium/calmodulin-dependent kinase kinase-β signaling is required for rotavirus replication. Proc Natl Acad Sci U S A. 109 (50), E3405-E3413 (2012).
- Crawford, S. E., Criglar, J. M., Liu, Z., Broughman, J. R., Estes, M. K. COPII vesicle transport is required for Rotavirus NSP4 interaction with the autophagy protein LC3 II and trafficking to viroplasms. J Virol. 94 (1), e01341 (2019).
- Pando, V., Iša, P., Arias, C. F., Ló Pez, S. Influence of calcium on the early steps of Rotavirus infection. Virology. 295 (1), 190-200 (2002).
- Hyser, J. M., Estes, M. K. Pathophysiological consequences of calcium-conducting viroporins. Annu Rev Virol. 2 (1), 473-496 (2015).
- Strtak, A. C., et al. Recovirus NS1-2 has viroporin activity that induces aberrant cellular calcium signaling to facilitate virus replication. mSphere. 4 (5), e00506-e00519 (2019).
- In, J. G., Foulke-Abel, J., Clarke, E., Kovbasnjuk, O. Human colonoid monolayers to study interactions between pathogens, commensals, and host intestinal epithelium. J Vis Exp. (146), 59357 (2019).
- Hirota, A., AlMusawi, S., Nateri, A. S., Ordóñez-Morán, P., Imajo, M. Biomaterials for intestinal organoid technology and personalized disease modeling. Acta Biomater. 132, 272-287 (2021).
- Cevallos Porta, D., López, S., Arias, C. F., Isa, P. Polarized rotavirus entry and release from differentiated small intestinal cells. Virology. 499, 65-71 (2016).
- Mirabelli, C., et al. Human Norovirus efficiently replicates in differentiated 3D-human intestinal enteroids. J Virol. 96 (22), e0085522 (2022).
- Icha, J., Weber, M., Waters, J. C., Norden, C. Phototoxicity in live fluorescence microscopy, and how to avoid it. Bioessays. 39 (8), 28749075 (2017).
- Li, J., et al. Engineering of NEMO as calcium indicators with large dynamics and high sensitivity. Nat Methods. 20 (6), 918-924 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados