Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Électroporation à base de substrat poreux avec surveillance de l’impédance électrique transépithéliale
Dans cet article
Résumé
L’électroporation de substrat poreux (PSEP) associe une distribution constante et à haut débit à une viabilité élevée des cellules. L’introduction de mesures d’impédance électrique transépithéliale (TEEI) donne un aperçu des processus intermédiaires du PSEP et permet une administration sans étiquette. Cet article traite d’une méthode permettant d’effectuer simultanément des expériences d’administration PSEP et une analyse de mesure TEEI.
Résumé
L’électroporation sur substrat poreux (PSEP) est une méthode émergente d’électroporation qui offre un débit élevé et une livraison constante. Comme de nombreux autres types d’administration intracellulaire, le PSEP s’appuie fortement sur des marqueurs fluorescents et la microscopie fluorescente pour déterminer la réussite de l’administration. Pour mieux comprendre les étapes intermédiaires du processus d’électroporation, une plateforme PSEP avec surveillance intégrée de l’impédance électrique transépithéliale (TEEI) a été développée. Les cellules sont cultivées dans des inserts disponibles dans le commerce avec des membranes poreuses. Après une période d’incubation de 12 h pour permettre la formation d’une monocouche cellulaire entièrement confluente, les inserts sont placés dans un milieu de transfection situé dans les puits du dispositif PSEP. Les monocouches cellulaires sont ensuite soumises à une forme d’onde définie par l’utilisateur, et l’efficacité de distribution est confirmée par la microscopie fluorescente. Ce flux de travail peut être considérablement amélioré grâce à des mesures TEEI entre la microscopie pulsée et la microscopie fluorescente pour collecter des données supplémentaires sur le processus PSEP, et ces données TEEI supplémentaires sont corrélées avec des paramètres de livraison tels que l’efficacité et la viabilité de la livraison. Cet article décrit un protocole permettant d’effectuer des mesures PSEP avec TEEI.
Introduction
L’électroporation est une technique dans laquelle les cellules sont exposées à un champ électrique, créant des pores temporaires dans la membrane cellulaire à travers lesquels les marchandises, y compris les protéines, l’ARN et l’ADN, peuvent passer 1,2. La version la plus utilisée est l’électroporation en vrac (BEP). La PEB est réalisée en remplissant une cuvette avec un électrolyte contenant des millions de cellules, en exposant l’électrolyte à une haute tension et en permettant à la cargaison de pénétrer dans les cellules par diffusion ou endocytose1. Les BEP présentent de nombreux avantages, notamment un débit élevé et de nombreux systèmes disponibles dans le commerce. Cependant, il existe des limites à la livraison du BEP. Le positionnement incohérent des cellules par rapport aux électrodes et le blindage du champ électrique par rapport aux cellules adjacentes entraînent une variabilité significative de l’exposition au champ électrique pendant les PEB3 et 4. La haute tension requise pour le BEP a également un impact négatif significatif sur la viabilité des cellules5. Depuis sa création en 20116, il y a eu un intérêt croissant pour une méthode d’électroporation appelée électroporation sur substrat poreux (PSEP), bien qu’elle soit parfois désignée par d’autres noms, notamment l’électroporation localisée et la nano- ou micro-électroporation 1,7,8. Contrairement à la suspension cellulaire de BEP, la PSEP est réalisée sur des cellules qui adhèrent à un substrat poreux. Non seulement un état d’adhérence est préféré pour la majorité des lignées cellulaires humaines9, mais les pores du substrat se concentrent également sur le courant électrique, localisant le potentiel électrique transmembranaire (TMP) dans des régions spécifiques de la membrane cellulaire10,11. Cette localisation permet une réduction significative de la tension appliquée, diminuant les dommages et augmentant la viabilité des cellules. Cette combinaison d’effets aide à contrôler le développement des pores de la membrane cellulaire, ce qui se traduit par une administration plus cohérente et plus efficace 1,5,12.
Une étude récente a présenté un dispositif PSEP doté d’un réseau d’électrodes plaquées or à six puits pour maintenir des inserts de membrane poreuse disponibles dans le commerce13 (figures 1A, B), une pratique qui a été introduite pour la première fois par Vindis et al.14. L’appareil peut appliquer des impulsions et mesurer l’impédance électrique à travers la monocouche de cellule, connue sous le nom d’impédance électrique transépithéliale (TEEI), en temps réel13. L’interface utilisateur de l’appareil permet un contrôle total de la forme d’onde et de la polarité de l’électroporation. Il est important de noter que les mesures d’impédance en temps réel peuvent être utilisées pour prédire les résultats de l’administration sans avoir besoin de réactifs coûteux ou de marqueurs fluorescents, un concept connu sous le nom d’administration sans marquage15.
La plate-forme PSEP se compose de deux principaux composants électriques personnalisés : le corps principal de l’appareil, qui abrite le générateur d’impulsions et l’équipement de mesure TEEI, et le réseau d’électrodes, où les substrats poreux sont insérés et où l’électroporation se produit. Des diagrammes pour tous les composants électroniques personnalisés et imprimés en 3D peuvent être trouvés sur GitHub : https://github.com/YangLabUNL/PSEP-TEEI. En plus de l’électronique personnalisée, un ordinateur est également nécessaire au bon fonctionnement de la plate-forme. Le logiciel personnalisé nécessite MATLAB (version 2021a ou ultérieure) pour s’exécuter, et Microsoft Excel pour stocker et accéder aux données à des fins d’analyse. Le programme contrôle l’électronique personnalisée et fournit l’interface utilisateur graphique (GUI) pour ajuster les paramètres. Ces programmes ont également été mis à disposition sur GitHub : https://github.com/YangLabUNL/PSEP-TEEI.
Les données préliminaires suggèrent que ce processus est possible pour différents types de cellules adhérentes (Figure 1C), mais cet article ne traitera que de la préparation des cellules A431 à l’aide de paramètres qui se sont avérés optimaux pour cette lignée cellulaire par Brooks et al.13. De plus, comme la cargaison d’iodure de propidium (PI) est cytotoxique, deux expériences sont réalisées, la première avec un milieu de transfection PI à haute concentration pour quantifier l’efficacité de l’administration, et la seconde avec uniquement des milieux de culture cellulaire pour mesurer l’EIEO sur des échelles de temps plus longues. Ces expériences utilisent des formes d’onde d’électroporation identiques, ce qui permet de corréler les résultats (Figure 1D).
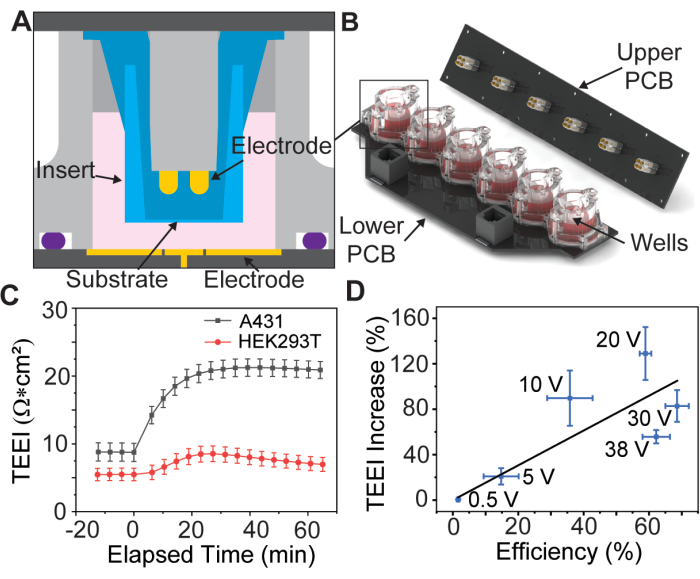
Figure 1 : Schéma d’assemblage du réseau d’électrodes et données de base. (A) Modèle CAO d’un insert à l’intérieur d’un puits du réseau d’électrodes. (B) Modèle CAO du réseau d’électrodes. (C) Augmentation de l’impédance due au PSEP pour certaines lignées cellulaires, n = 3 par lignée cellulaire. Barre d’erreur : erreur type de la moyenne. (D) Efficacité de la livraison vs. TEEI augmenter les données de corrélation. L’efficacité de livraison a été calculée en divisant le nombre de cellules étiquetées dans les images PI et calcein des expériences de livraison par le nombre total de cellules identifiées avec Hoechst. Le nombre de cellules a été déterminé à l’aide d’un pipeline CellProfiler personnalisé, n = 6 par tension. Barre d’erreur : erreur type (axes x et y) de la moyenne. Cette figure est reproduite à partir de Brooks et al.13 avec autorisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
Les détails des réactifs et de l’équipement utilisés dans l’étude sont énumérés dans la table des matériaux.
1. Préparation des réactifs et culture cellulaire
- Préparez le milieu de culture cellulaire en ajoutant 50 ml de sérum fœtal bovin (FBS) et 5 ml de pénicilline-streptomycine dans un contenant de 500 ml de milieu Eagle modifié de Dulbecco (DMEM). Produire onze aliquotes de 50 mL pour réduire le risque de contamination et réfrigérer à 4 °C.
- Créer 1 mL de 25 μg/mL de fibronectine plasmatique humaine dans une solution mère de solution saline tamponnée au phosphate (PBS) selon les instructions du fabricant.
- Créer 15 mL d’iodure de propidium à 0,1 mg/mL dans une solution mère de DMEM pour permettre des expériences avec des concentrations de cargaison variables.
- Cultiver des cellules A431 dans une fiole T75 contenant 12 mL du milieu de culture cellulaire préparé. Les cellules ont été passées tous les 1 à 2 jours pour maintenir une confluence de 50 %.
2. Préparation de l’échantillon
- Enrobage de fibronectine
- Sélectionnez douze inserts et deux plaques à 24 puits. Placez les inserts dans une plaque à puits, en créant deux rangées de six. Mettez la deuxième assiette de côté pour plus tard.
- Créez 1 300 μL de solution de fibronectine de 1 μg/mL en mélangeant 52 μL de solution mère de fibronectine et 1 248 μL de PBS dans un tube de 1,5 mL.
- Répartissez 100 μL de la solution de fibronectine dans chaque insert. Incuber les inserts dans la plaque du puits à 37 °C pendant 3 h.
- Ajuster la concentration cellulaire pour optimiser la densité cellulaire
- Environ 1 h avant la fin de l’incubation de la fibronectine, retirez le flacon T75 de cellules A431 de l’incubateur pour l’extraction des cellules.
- Retirer le support dans le ballon à l’aide d’un aspirateur et laver avec 5 mL de PBS. Retirez le PBS de la même manière et ajoutez 3 ml de trypsine. Incuber pendant 3-4 min avant de tapoter le côté de la fiole pour détacher complètement les cellules.
- Ajouter 6 mL de milieu de culture cellulaire dans le ballon, en mélangeant vigoureusement avec une pipette pour détacher les cellules restantes, et transférer le contenu dans un tube à centrifuger de 15 mL. Centrifugeuse à 100 x g et 20 °C pendant 5 min.
- Retirez le milieu de culture cellulaire et la trypsine du tube de centrifugation à l’aide d’un aspirateur, en prenant soin de ne pas perturber la pastille cellulaire. Ajoutez 1 mL de milieu dans le tube à centrifuger et pipetez d’avant en arrière (sans produire de bulles) pour briser la pastille de la cellule et remettre les cellules en suspension.
- Pipeter 10 μL de suspension cellulaire, 40 μL de milieu de culture cellulaire et 50 μL de colorant bleu trypan dans un tube de 200 μL, à l’aide d’une pipette pour bien mélanger.
- Prélever 10 μL du mélange de colorant et l’injecter dans un hémocytomètre. Comptez les cellules en utilisant la dilution de 10 % du mélange de colorants pour estimer le nombre total de cellules dans le tube à centrifuger de 15 ml.
REMARQUE : Pour ce protocole, supposons une concentration de 5 000 000 cellules/mL. - Multipliez la densité de semis souhaitée par la surface de la membrane de l’insert, divisez par le nombre de cellules/mL dans la suspension et multipliez par 1 000 pour calculer les microlitres de suspension de cellules nécessaires par insert.
- Pour déterminer la quantité totale de suspension cellulaire requise, multipliez ce chiffre par 10 (pour vous assurer qu’il y a suffisamment de cellules pour 9 échantillons, car 3 des 12 inserts seront des témoins sans cellules) et arrondissez au nombre entier le plus proche. Dans ce cas, un total de 135 μL de suspension cellulaire est nécessaire pour cette expérience.
- Créez 2 000 μL de solution cellulaire ajustée en mélangeant les 135 μL précédemment calculés de la suspension cellulaire avec 1 865 μL de milieu de culture cellulaire dans un tube à centrifuger séparé de 15 mL.
- Cellules d’ensemencement
- Retirez l’excès de fibronectine de chaque insert une fois l’incubation de fibronectine terminée.
- Lavez les inserts deux fois en ajoutant 100 μL d’eau distillée stérile dans chaque insert. Retirez l’eau en suivant le même ordre que celui dans lequel elle a été ajoutée pour assurer un temps de lavage constant entre les inserts.
- Lavez à nouveau l’insert en ajoutant 100 μL de milieu de culture cellulaire dans chaque insert. Retirez le support en suivant le même ordre qu’il a été ajouté pour assurer un temps de lavage constant entre les inserts.
- Inserts d’échantillons de cellules
- Ensemencer les cellules en pipetant 200 μL de solution cellulaire ajustée dans chaque insert. Pour assurer une confluence constante entre les inserts, mélangez la solution cellulaire dans le tube à centrifuger avant la distribution et mélangez à nouveau dans chaque insert après la distribution.
- Inserts de contrôle négatif
- Pipeter 200 μL de milieu de culture cellulaire dans chaque insert. Pour rester cohérent avec les inserts d’échantillons cellulaires, utilisez la pipette pour mélanger les milieux de culture cellulaire à l’intérieur de chaque insert.
- Étiquetage et incubation
- Tracez une ligne divisant la deuxième plaque de puits en deux colonnes de trois puits de large (pour les conditions exécutées en trois exemplaires) à l’aide d’un marqueur permanent. Séparez chaque colonne en rangées. Étiquetez chaque région de la grille avec les paramètres appropriés.
- Ajoutez 1 ml de milieu de culture cellulaire dans chaque puits pour recevoir un encart pour l’expérience. Transférez les inserts de la plaque du puits de préparation à leur emplacement approprié dans la plaque du puits d’expérience étiquetée et incubez à 37 °C pendant au moins 12 h.
3. Procédure expérimentale
- Expérience d’accouchement
- Pipeter 1,5 mL de la solution d’IP à 0,1 mg/mL dans chaque puits du réseau d’électrodes. Placez un insert dans chaque puits du réseau d’électrodes, en insérant les pieds de l’insert dans les rainures d’alignement de sorte que l’insert affleure la surface supérieure du puits (Figure 1A,B).
- Vissez la carte de circuit imprimé (PCB) de l’électrode supérieure sur le dessus des puits du réseau d’électrodes et connectez le réseau d’électrodes au dispositif PSEP.
- Placez le réseau d’électrodes dans l’incubateur à 37 °C pendant au moins 1 h pour permettre à la température de s’équilibrer.
- Cliquez sur le menu déroulant à côté de « Membrane » dans le coin supérieur gauche de l’interface graphique et cliquez sur 400 nm GBO. Répétez cette étape pour « Électrolyte », « Cellules », « Densité d’ensemencement cellulaire » et « Durée de cellule », en sélectionnant DMEM, A431, 200 et 12, respectivement.
REMARQUE : Ces valeurs sont uniquement à des fins d’enregistrement et n’ont pas d’impact sur le fonctionnement de l’appareil. Assurez-vous d’ajuster ces valeurs si nécessaire pour un suivi correct des données. - Tapez 1 dans le champ d’édition Durée post-impulsion (min) sur le côté droit de l’interface graphique pour changer le temps de mesure post-impulsion par défaut à 1 min. Laissez tous les autres paramètres dans l’état par défaut.
REMARQUE : Les paramètres d’impulsion par défaut créent une forme d’onde carrée de 30 volts, 20 Hz, d’une durée de 1 ms et de 200 impulsions. Les paramètres de mesure TEEI par défaut sont 0,5 volts et 100 Hz, 1 000 Hz, 10 000 Hz et 100 000 Hz. - Cliquez sur le bouton Exécuter et entrez les noms appropriés pour les puits 1-3 et 4-6 lorsque vous y êtes invité. Cliquez sur OK pour démarrer l’expérience.
- Retirez le réseau d’électrodes de l’incubateur et transférez les inserts aux emplacements d’origine dans la plaque du puits d’expérience lorsque le programme a fini de s’exécuter.
- Mélangez 2 μL de Hoechst 33342 et 5 μL de calcéine AM avec 123 μL de milieux de culture cellulaire dans un tube de 200 μL.
- Pipeter doucement 10 μL de la solution de coloration dans chaque insert post-impulsion et remettre les inserts dans l’incubateur pendant 5 min.
- Transférez la plaque de puits sur le support de plaque d’un microscope fluorescent avec un objectif à grossissement 5x. Image en fond clair et en fluorescence de chaque colorant. Centrez l’insert sur l’objectif avant de déclencher l’appareil photo.
REMARQUE : Les longueurs d’onde d’excitation pour PI, calcein AM et Hoechst 33342 sont respectivement de 558 nm, 495 nm et 353 nm. Les longueurs d’onde d’émission sont respectivement de 575 nm, 519 nm et 465 nm.
- Expérience de mesure TEEI
- Pipeter 1,5 mL du milieu de culture cellulaire dans chaque puits du réseau d’électrodes. Placez les inserts d’échantillon de cellule dans les puits 1 à 3 et les inserts de contrôle dans les puits 4 à 6, en insérant les pieds de l’insert dans les rainures d’alignement de sorte que l’insert affleure la surface supérieure du puits.
- Vissez le circuit imprimé de l’électrode supérieure sur le dessus des puits du réseau d’électrodes et connectez le réseau d’électrodes au dispositif PSEP.
- Placez le réseau d’électrodes dans l’incubateur à 37 °C pendant au moins 1 h pour permettre à la température de s’équilibrer.
- Cliquez sur le menu déroulant à côté de « Membrane » dans le coin supérieur gauche de l’interface graphique et cliquez sur GBO 400 nm. Répétez cette étape pour « Électrolyte », « Cellules », « Densité d’ensemencement cellulaire » et « Durée de cellule », en sélectionnant DMEM, A431, 200 et 12, respectivement.
REMARQUE : Ces valeurs sont uniquement à des fins d’enregistrement et n’ont pas d’impact sur le fonctionnement de l’appareil. Assurez-vous d’ajuster ces valeurs si nécessaire pour un suivi correct des données. - Laissez tous les paramètres restants dans l’état par défaut.
REMARQUE : Les paramètres d’impulsion par défaut créent une forme d’onde carrée de 30 volts, 20 Hz, d’une durée de 1 ms et de 200 impulsions. Les paramètres de mesure TEEI par défaut sont 0,5 volts et 100 Hz, 1 000 Hz, 10 000 Hz et 100 000 Hz. - Cliquez sur le bouton Exécuter et entrez les noms appropriés pour les puits 1-3 et 4-6 lorsque vous y êtes invité. Cliquez sur OK pour démarrer l’expérience.
- Retirez le réseau d’électrodes de l’incubateur et transférez les inserts aux emplacements d’origine dans la plaque du puits d’expérience lorsque le programme a fini de s’exécuter.
- Mélangez 2 μL de Hoechst 33342, 5 μL de calcéine AM et 10 μL de PI avec 113 μL de milieu de culture cellulaire dans un tube de réaction de 200 μL.
- Pipeter 10 μL de la solution de coloration dans chaque insert post-impulsion et remettre les inserts dans l’incubateur pendant 5 min.
- Transférez la plaque de puits sur le support de plaque d’un microscope à imagerie fluorescente avec un objectif 5x. Image en fond clair et en fluorescence de chaque colorant. Centrez l’insert sur l’objectif avant de déclencher l’appareil photo.
REMARQUE : Les longueurs d’onde d’excitation pour PI, calcein AM et Hoechst 33342 sont respectivement de 558 nm, 495 nm et 353 nm. Les longueurs d’onde d’émission sont respectivement de 575 nm, 519 nm et 465 nm.
4. Analyse des données
- Analyse des données d’image avec le pipeline CellProfiler
- Utilisez le flux de travail CellProfiler personnalisé fourni à l’adresse GitHub :https ://github.com/YangLabUNL/PSEP-TEEI pour traiter les images de livraison et d’expérience de mesure TEEI.
- Analyse TEEI
- Cliquez sur l’onglet Analyse dans l’interface graphique.
- Basculez l’indicateur de type d’impédance sur TEEI en bas de l’interface graphique.
- Cliquez sur la flèche dans la zone en haut à gauche pour afficher tous les noms des expériences dans le fichier de données. Sélectionnez toutes les données de l’échantillon de cellule de l’expérience de mesure TEEI.
- Cliquez sur la flèche dans la case suivante à droite pour afficher tous les noms des expériences dans le fichier de données. Sélectionnez toutes les données d’insert de commande de la mesure TEEI.
- Cliquez sur Exécuter. Une figure de base contenant les données de l’échantillon de cellule sélectionnées à la fréquence de mesure la plus basse apparaîtra.
- Dans la boîte d’options d’exemple sur le côté droit de l’interface graphique, cliquez sur la flèche pour afficher toutes les données d’insertion sélectionnées. Les valeurs aberrantes peuvent être supprimées en sélectionnant les données appropriées et en cliquant sur Supprimer ci-dessous.
REMARQUE : Toutes les données qui ont été supprimées de l’analyse par le dernier clic sur le bouton Supprimer peuvent être récupérées à l’aide du bouton Annuler . - Cliquez sur Terminé pour passer à la figure suivante lorsque les données souhaitées sont affichées dans la figure.
- Répétez les étapes 4.2.6 et 4.2.7 pour les données d’échantillon de cellule et les données de contrôle restantes. Lorsque l’ensemble de données final a été confirmé en cliquant sur « Terminé », la figure d’analyse complète apparaît.
- Enregistrez la figure d’analyse.
Résultats
Le protocole donné établit une méthode d’utilisation des mesures TEEI pour examiner les processus intermédiaires d’électroporation et faire des prédictions de livraison, en particulier pour la lignée cellulaire A431 et le fret PI. Bien que la modification de ce protocole soit abordée plus loin dans l’article, il est important de noter maintenant que même si les valeurs spécifiques peuvent changer, les tendances générales de la réponse restent constantes. Par exemple, l...
Discussion
La figure 2C montre que les augmentations par rapport au minimum et les diminutions par rapport à la ligne de base sont tracées pour chaque tension de forme d’onde PSEP. L’augmentation TEEI crée un arc parabolique, culminant autour de 20 volts avant de diminuer, tandis que la diminution TEEI par rapport à la ligne de base augmente de manière exponentielle à mesure que la tension augmente. L’efficacité de la distribution et les pourcentages de mo...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Nous tenons à souligner le soutien financier de la NSF (Awards 1826135, 1936065, 2143997), des National Institutes of General Medical Sciences P20GM113126 (Nebraska Center for Integrated Biomolecular Communication) et de l’P30GM127200 (Nebraska Center for Nanomedicine), de la Nebraska Collaborative Initiative et du Voelte-Keegan Bioengineering Support. Le dispositif a été fabriqué au NanoEngineering Research Core Facility (NERCF), qui est partiellement financé par la Nebraska Research Initiative.
matériels
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tube | Thermo Scientific | 339651 | |
| 2-Chip Disposable Hemocytometer | Bulldog Bio | DHC-N01 | |
| 75 cm2 Tissue Culture Flask | fisherbrand | FB012937 | |
| A431 Cells | ATCC | CRL-1555 | |
| Calcein AM | Invitrogen | C3099 | |
| Class II Type A2 Biosafety Cabinet | Labgard | NU-543-600 | |
| Custom Components | YangLab | https://github.com/YangLabUNL/PSEP-TEEI | |
| Disposable Centrifuge Tube (50 mL) | fisherbrand | 05-539-6 | |
| DMEM | Gibco | 11965092 | |
| Fetal Bovine Serum | Gibco | A5670401 | |
| Fluid Aspiration System | vacuubrand | 20727403 | |
| HERACELL 240i | Thermo Scientific | 51026331 | |
| Hoechst 33342 | Thermo Scientific | 62249 | |
| Human Plasma Fibronectin | Sigma-Aldrich | FIBRP-RO | |
| Inverted Fluorescent Microscope | Zeiss | 491916-0001-000 | |
| Inverted Microscope | Labomed | TCM 400 | |
| PBS | cytiva | SH30256.02 | |
| PCR Tube 200 µL | Sarstedt | 72.737 | |
| Penicillin / Streptomycin | Gibco | 15140148 | |
| Pipette (0.2-2 µL) | fisherbrand Elite | FBE00002 | |
| Pipette (100-1000 µL) | fisherbrand Elite | FBE01000 | |
| Pipette (20-200 µL) | fisherbrand Elite | FBE00200 | |
| Pipette (2-20 µL) | fisherbrand Elite | FBE00020 | |
| Propidium Iodide | Invitrogen | P1304MP | |
| Reaction Tube 1.5 mL | Sarstedt | 72.690.300 | |
| Sorvall ST 16R Centrifuge | Thermo Scientific | 75004240 | |
| Thincert (24-well) | Greiner Bio-One | 662 641 | 0.4 µm pore diameter, 2x106 cm-2 pore density, transparent PET |
| Tissue Culture Plate (24-well) | fisherbrand | FB012929 | |
| Trypan Blue Solution | Sigma-Aldrich | T8154-20mL | |
| Trypsin | Gibco | 15090046 |
Références
- Brooks, J., et al. High throughput and highly controllable methods for in vitro intracellular delivery. Small. 16 (51), e2004917 (2020).
- Stewart, M. P., Langer, R., Jensen, K. F. Intracellular delivery by membrane disruption: Mechanisms, strategies, and concepts. Chem Rev. 118 (16), 7409-7531 (2018).
- Canatella, P. J., Karr, J. F., Petros, J. A., Prausnitz, M. R. Quantitative study of electroporation-mediated molecular uptake and cell viability. Biophys J. 80 (2), 755-764 (2001).
- Pliquett, U., Gift, E. A., Weaver, J. C. Determination of the electric field and anomalous heating caused by exponential pulses with aluminum electrodes in electroporation experiments. Bioelectrochem Bioenerg. 39 (1), 39-53 (1996).
- Pan, J., et al. Cell membrane damage and cargo delivery in nano-electroporation. Nanoscale. 15 (8), 4080-4089 (2023).
- Boukany, P. E., et al. Nanochannel electroporation delivers precise amounts of biomolecules into living cells. Nat Nanotechnol. 6 (11), 747-754 (2011).
- Chang, L., et al. Micro-/nanoscale electroporation. Lab Chip. 16 (21), 4047-4062 (2016).
- Patino, C. A., et al. Multiplexed high-throughput localized electroporation workflow with deep learning-based analysis for cell engineering. Sci Adv. 8 (29), 7637 (2022).
- Sagvolden, G., Giaever, I., Pettersen, E. O., Feder, J. Cell adhesion force microscopy. Proc Natl Acad Sci U S A. 96 (2), 471-476 (1999).
- Ishibashi, T., Takoh, K., Kaji, H., Abe, T., Nishizawa, M. A porous membrane-based culture substrate for localized in situ electroporation of adherent mammalian cells. Sensors Actuators B: Chem. 128 (1), 5-11 (2007).
- Mukherjee, P., Nathamgari, S. S. P., Kessler, J. A., Espinosa, H. D. Combined numerical and experimental investigation of localized electroporation-based cell transfection and sampling. ACS Nano. 12 (12), 12118-12128 (2018).
- Brooks, J. R., et al. An equivalent circuit model for localized electroporation on porous substrates. Biosens Bioelectron. 199, 113862 (2022).
- Brooks, J. R., et al. Transepithelial electrical impedance increase following porous substrate electroporation enables label-free delivery. Small. 20 (25), 2310221 (2023).
- Vindiš, T., et al. Gene electrotransfer into mammalian cells using commercial cell culture inserts with porous substrate. Pharmaceutics. 14 (9), 1959 (2022).
- Ye, Y., et al. Single-cell electroporation with real-time impedance assessment using a constriction microchannel. Micromachines. 11 (9), 856 (2020).
- Bednarek, R. In vitro methods for measuring the permeability of cell monolayers. Methods Protoc. 5 (1), 17 (2022).
- Harhaj, N. S., Antonetti, D. A. Regulation of tight junctions and loss of barrier function in pathophysiology. Int J Biochem Cell Biol. 36 (7), 1206-1237 (2004).
- Hunter, R. J. . Zeta potential in colloid science: Principles and applications. 2, (2013).
- Wong, P. K., Wang, T. -. H., Deval, J. H., Ho, C. -. M. Electrokinetics in microdevices for biotechnology applications. IEEE/ASME Trans Mechatron. 9 (2), 366-376 (2004).
- Qian, K., Wang, Y., Lei, Y., Yang, Q., Yao, C. An experimental and theoretical study on cell swelling for osmotic imbalance induced by electroporation. Bioelectrochemistry. 157, 108637 (2024).
- Fox, M. B., et al. Electroporation of cells in microfluidic devices: A review. Anal Bioanal Chem. 385 (3), 474-485 (2006).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon