É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Eletroporação Baseada em Substrato Poroso com Monitoramento de Impedância Elétrica Transepitelial
Neste Artigo
Resumo
A eletroporação de substrato poroso (PSEP) combina entrega consistente e de alto rendimento com alta viabilidade celular. A introdução de medições de impedância elétrica transepitelial (TEEI) fornece informações sobre os processos intermediários do PSEP e permite a entrega sem rótulos. Este artigo discute um método para realizar experimentos de entrega de PSEP e análise de medição TEEI simultaneamente.
Resumo
A eletroporação de substrato poroso (PSEP) é um método emergente de eletroporação que fornece alto rendimento e entrega consistente. Como muitos outros tipos de entrega intracelular, o PSEP depende muito de marcadores fluorescentes e microscopia fluorescente para determinar o sucesso da entrega. Para obter informações sobre as etapas intermediárias do processo de eletroporação, foi desenvolvida uma plataforma PSEP com monitoramento integrado de impedância elétrica transepitelial (TEEI). As células são cultivadas em inserções disponíveis comercialmente com membranas porosas. Após um período de incubação de 12 h para permitir a formação de uma monocamada celular totalmente confluente, as inserções são colocadas em meios de transfecção localizados nos poços do dispositivo PSEP. As monocamadas celulares são então submetidas a uma forma de onda definida pelo usuário e a eficiência de entrega é confirmada por microscopia fluorescente. Esse fluxo de trabalho pode ser significativamente aprimorado com medições TEEI entre microscopia pulsante e fluorescente para coletar dados adicionais sobre o processo PSEP, e esses dados TEEI adicionais estão correlacionados com métricas de entrega, como eficiência e viabilidade de entrega. Este artigo descreve um protocolo para a realização de PSEP com medidas TEEI.
Introdução
A eletroporação é uma técnica na qual as células são expostas a um campo elétrico, criando poros temporários na membrana celular através dos quais cargas, incluindo proteínas, RNA e DNA, podem passar 1,2. A versão mais utilizada é a eletroporação em massa (BEP). A BEP é realizada enchendo uma cubeta com um eletrólito contendo milhões de células, expondo o eletrólito a alta voltagem e permitindo que a carga entre nas células por difusão ou endocitose1. Há muitas vantagens no BEP, incluindo alto rendimento e vários sistemas disponíveis comercialmente. No entanto, há limitações para a entrega do BEP. O posicionamento inconsistente da célula em relação aos eletrodos e à blindagem do campo elétrico das células adjacentes causa variabilidade significativa na exposição ao campo elétrico durante o BEP 3,4. A alta voltagem necessária para BEP também tem um impacto negativo significativo na viabilidade celular5. Desde a sua criação em 20116, tem havido um interesse crescente em um método de eletroporação chamado eletroporação por substrato poroso (PSEP), embora às vezes seja referido por outros nomes, incluindo eletroporação localizada e nano ou microeletroporação 1,7,8. Em contraste com a suspensão celular do BEP, o PSEP é conduzido em células aderentes a um substrato poroso. Não apenas um estado aderente é preferido para a maioria das linhagens celulares humanas9, mas os poros do substrato também se concentram na corrente elétrica, localizando o potencial elétrico transmembrana (TMP) em regiões específicas da membrana celular10,11. Essa localização permite uma redução significativa na tensão aplicada, diminuindo os danos e aumentando a viabilidade celular. Essa combinação de efeitos ajuda a controlar o desenvolvimento dos poros da membrana celular, resultando em uma entrega mais consistente e eficiente 1,5,12.
Um estudo recente introduziu um dispositivo PSEP com um feixe de eletrodos banhados a ouro de seis poços para segurar inserções de membrana porosa disponíveis comercialmente13 (Figura 1A, B), uma prática que foi introduzida pela primeira vez por Vindis et al.14. O dispositivo pode aplicar pulsos e medir a impedância elétrica através da monocamada da célula, conhecida como impedância elétrica transepitelial (TEEI), em tempo real13. A interface do usuário do dispositivo permite controle completo sobre a forma de onda e polaridade do eletroporation. É importante ressaltar que as medições de impedância em tempo real podem ser usadas para prever os resultados da entrega sem a necessidade de reagentes caros ou marcadores fluorescentes, um conceito conhecido como entrega sem rótulo15.
A plataforma PSEP consiste em dois componentes elétricos personalizados principais: o corpo principal do dispositivo, que abriga o gerador de pulsos e o equipamento de medição TEEI, e o feixe de eletrodos, onde os substratos porosos são inseridos e ocorre a eletroporação. Diagramas para todos os componentes eletrônicos personalizados e impressos em 3D podem ser encontrados no GitHub: https://github.com/YangLabUNL/PSEP-TEEI. Além da eletrônica personalizada, também é necessário um computador para que a plataforma funcione corretamente. O software personalizado requer o MATLAB (versão 2021a ou posterior) para ser executado e o Microsoft Excel para armazenar e acessar dados para análise. O programa controla a eletrônica personalizada e fornece a interface gráfica do usuário (GUI) para ajustar as configurações. Esses programas também foram disponibilizados no GitHub: https://github.com/YangLabUNL/PSEP-TEEI.
Dados preliminares sugerem que esse processo é possível para diferentes tipos de células aderentes (Figura 1C), mas este artigo discutirá apenas a preparação de células A431 usando parâmetros que foram considerados ideais para essa linhagem celular por Brooks et al.13. Além disso, como a carga de iodeto de propídio (PI) é citotóxica, dois experimentos são realizados, o primeiro com um meio de transfecção de PI de alta concentração para quantificar a eficiência de entrega e o segundo apenas com meios de cultura de células para medir o TEEI em escalas de tempo mais longas. Esses experimentos usam formas de onda de eletroporação idênticas, permitindo que os resultados sejam correlacionados (Figura 1D).
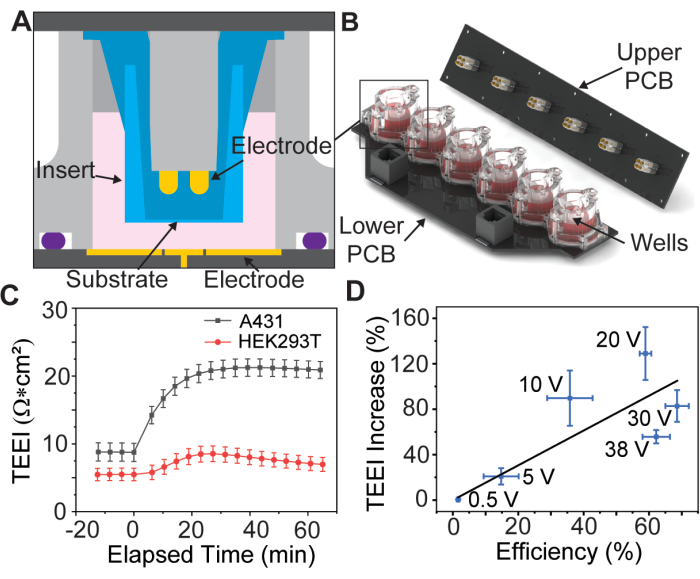
Figura 1: Diagrama de montagem do feixe de eletrodos e dados fundamentais. (A) Modelo CAD de um inserto dentro de um poço do feixe de eletrodos. (B) Modelo CAD do feixe de eletrodos. (C) Aumento da impedância devido ao PSEP para linhagens celulares selecionadas, n = 3 por linhagem celular. Barra de erro: erro padrão da média. (D) Eficiência de entrega vs. TEEI aumenta os dados de correlação. A eficiência de entrega foi calculada dividindo o número de células marcadas nas imagens de PI e calceína dos experimentos de entrega pelo número total de células identificadas com Hoechst. A contagem de células foi determinada usando um pipeline personalizado do CellProfiler, n = 6 por tensão. Barra de erro: (eixos x e y) erro padrão da média. Esta figura é reproduzida de Brooks et al.13 com permissão. Clique aqui para ver uma versão maior desta figura.
Protocolo
Os detalhes dos reagentes e do equipamento utilizado no estudo estão listados na Tabela de Materiais.
1. Preparação de reagentes e cultura celular
- Prepare o meio de cultura celular adicionando 50 mL de soro fetal bovino (FBS) e 5 mL de penicilina-estreptomicina a um recipiente de 500 mL de Dulbecco's Modified Eagle Medium (DMEM). Produzir onze alíquotas de 50 ml para reduzir o risco de contaminação e refrigerar a 4 °C.
- Crie 1 mL de 25 μg/mL de fibronectina plasmática humana em solução salina tamponada com fosfato (PBS) de acordo com as instruções do fabricante.
- Crie 15 mL de iodeto de propídio 0,1 mg / mL em solução estoque de DMEM para permitir experimentos com concentrações de carga variadas.
- Cultivar células A431 num balão T75 contendo 12 ml do meio de cultura celular preparado. As células foram passadas a cada 1-2 dias para manter 50% de confluência.
2. Preparação da amostra
- Revestimento de fibronectina
- Selecione doze pastilhas e duas placas de 24 poços. Coloque as inserções em uma placa de poço, criando duas fileiras de seis. Deixe a segunda placa de poço de lado até mais tarde.
- Crie 1.300 μL de solução de fibronectina de 1 μg/mL misturando 52 μL de solução estoque de fibronectina e 1.248 μL de PBS em um tubo de 1,5 mL.
- Distribua 100 μL da solução de fibronectina em cada inserção. Incubar as pastilhas na placa do poço a 37 °C durante 3 h.
- Ajustando a concentração celular para otimizar a densidade celular
- Cerca de 1 h antes da conclusão da incubação da fibronectina, remova o frasco T75 de células A431 da incubadora para extração celular.
- Remova o meio no frasco com um aspirador e lave com 5 mL de PBS. Remova o PBS da mesma maneira e adicione 3 mL de tripsina. Incube por 3-4 min antes de bater na lateral do frasco para separar completamente as células.
- Adicione 6 mL de meio de cultura de células ao frasco, misturando vigorosamente com uma pipeta para separar as células restantes, e transfira o conteúdo para um tubo de centrífuga de 15 mL. Centrifugue a 100 x g e 20 °C por 5 min.
- Remova o meio de cultura celular e a tripsina do tubo de centrífuga usando um aspirador, tomando cuidado para não perturbar o pellet celular. Adicione 1 mL de meio ao tubo de centrífuga e pipete para frente e para trás (sem produzir bolhas) para quebrar o pellet celular e ressuspender as células.
- Pipete 10 μL da suspensão celular, 40 μL de meio de cultura celular e 50 μL de corante azul de tripano em um tubo de 200 μL, usando uma pipeta para misturar bem.
- Remova 10 μL da mistura de corante e injete-a em um hemocitômetro. Conte as células usando a diluição de 10% da mistura de corantes para estimar a contagem total de células no tubo de centrífuga de 15 mL.
NOTA: Para este protocolo, assuma uma concentração de 5.000.000 células/mL. - Multiplique a densidade de semeadura desejada pela área de superfície da membrana do inserto, divida pelas células contadas/mL na suspensão e multiplique por 1.000 para calcular os microlitros necessários de suspensão celular por inserto.
- Para encontrar a quantidade total de suspensão celular necessária, multiplique esse número por 10 (para garantir células suficientes para 9 amostras, pois 3 das 12 inserções serão controles livres de células) e arredonde para o número inteiro mais próximo. Neste caso, um total de 135 μL da suspensão celular é necessário para este experimento.
- Crie 2.000 μL de solução celular ajustada misturando os 135 μL da suspensão celular previamente calculados com 1.865 μL de meio de cultura celular em um tubo de centrífuga separado de 15 mL.
- Semeando células
- Remova o excesso de fibronectina de cada inserção assim que a incubação da fibronectina estiver completa.
- Lave as inserções duas vezes adicionando 100 μL de água destilada estéril a cada inserção. Remova a água seguindo a mesma ordem em que foi adicionada para garantir um tempo de lavagem consistente entre as inserções.
- Lave novamente a pastilha adicionando 100 μL de meio de cultura celular a cada inserção. Remova a mídia seguindo a mesma ordem em que foi adicionada para garantir um tempo de lavagem consistente entre as inserções.
- Inserções de amostra de célula
- Células de sementes pipetando 200 μL de solução celular ajustada em cada inserção. Para garantir uma confluência consistente entre os insertos, misture a solução celular no tubo de centrífuga antes da distribuição e misture novamente dentro de cada inserto após a distribuição.
- Insertos de controle negativo
- Pipetar 200 μL de meio de cultura celular em cada inserção. Para permanecer consistente com as inserções de amostra de células, use a pipeta para misturar os meios de cultura de células dentro de cada inserção.
- Rotulagem e incubação
- Desenhe uma linha dividindo a segunda placa de poço em duas colunas com três poços de largura (para condições executadas em triplicado) usando um marcador permanente. Separe cada coluna em linhas. Rotule cada região na grade com parâmetros relevantes.
- Adicione 1 mL de meio de cultura celular a cada poço para receber uma inserção para o experimento. Transferir as inserções da placa do alvéolo de preparação para o local adequado na placa do alvéolo rotulado e incubar a 37 °C durante pelo menos 12 h.
3. Procedimento experimental
- Experiência de entrega
- Pipetar 1,5 ml da solução de 0,1 mg/ml de PI em cada alvéolo do feixe de elétrodos. Coloque um inserto em cada poço do feixe de eletrodos, encaixando os pés do inserto nas ranhuras de alinhamento de modo que o inserto fique nivelado com a superfície superior do poço (Figura 1A, B).
- Aparafuse a placa de circuito impresso (PCB) do eletrodo superior na parte superior dos poços do feixe de eletrodos e conecte o feixe de eletrodos ao dispositivo PSEP.
- Coloque o feixe de eletrodos na incubadora de 37 °C por pelo menos 1 h para permitir que a temperatura se equilibre.
- Clique no menu suspenso ao lado de "Membrana" no canto superior esquerdo da GUI e clique em GBO de 400 nm. Repita esta etapa para "Eletrólito", "Células", "Densidade de semeadura celular" e "Duração da célula", selecionando DMEM, A431, 200 e 12, respectivamente.
NOTA: Esses valores são apenas para fins de manutenção de registros e não afetam a função do dispositivo. Certifique-se de ajustar esses valores conforme necessário para o rastreamento correto dos dados. - Digite 1 no campo de edição Duração do tempo pós-pulso (min) no lado direito da GUI para alterar o tempo de medição pós-pulso padrão para 1 min. Deixe todas as outras configurações no estado padrão.
NOTA: Os parâmetros de pulso padrão criam uma forma de onda quadrada com 30 volts, 20 Hz, duração de 1 ms e 200 pulsos. Os parâmetros de medição TEEI padrão são 0,5 volts e 100 Hz, 1.000 Hz, 10.000 Hz e 100.000 Hz. - Clique no botão Executar e insira os nomes apropriados para os poços 1-3 e 4-6 quando solicitado. Clique em OK para iniciar o experimento.
- Remova o feixe de eletrodos da incubadora e transfira as pastilhas de volta para os locais originais na placa do poço do experimento quando o programa terminar de ser executado.
- Misture 2 μL de Hoechst 33342 e 5 μL de calceína AM com 123 μL de meio de cultura celular em um tubo de 200 μL.
- Pipete suavemente 10 μL da solução corante em cada inserto pós-pulso e coloque os insertos de volta na incubadora por 5 min.
- Transfira a placa do poço para o suporte da placa de um microscópio fluorescente com uma objetiva de ampliação de 5x. Imagem usando campo claro e a fluorescência de cada mancha. Centralize a inserção sobre a objetiva antes de acionar a câmera.
NOTA: Os comprimentos de onda de excitação para PI, calceína AM e Hoechst 33342 são 558 nm, 495 nm e 353 nm, respectivamente. Os comprimentos de onda de emissão são 575 nm, 519 nm e 465 nm, respectivamente.
- Experiência de medição TEEI
- Pipete 1,5 mL do meio de cultura celular em cada poço no feixe de eletrodos. Coloque as inserções de amostra de célula nos poços 1-3 e as pastilhas de controle nos poços 4-6, encaixando os pés da pastilha nas ranhuras de alinhamento de modo que a pastilha fique nivelada com a superfície superior do poço.
- Aparafuse a placa de circuito impresso do eletrodo superior na parte superior dos poços do feixe de eletrodos e conecte o conjunto de eletrodos ao dispositivo PSEP.
- Coloque o feixe de eletrodos na incubadora de 37 °C por pelo menos 1 h para permitir que a temperatura se equilibre.
- Clique no menu suspenso ao lado de "Membrana" no canto superior esquerdo da GUI e clique em 400 nm GBO. Repita esta etapa para "Eletrólito", "Células", "Densidade de semeadura celular" e "Duração da célula", selecionando DMEM, A431, 200 e 12, respectivamente.
NOTA: Esses valores são apenas para fins de manutenção de registros e não afetam a função do dispositivo. Certifique-se de ajustar esses valores conforme necessário para o rastreamento correto dos dados. - Deixe todas as configurações restantes no estado padrão.
NOTA: Os parâmetros de pulso padrão criam uma forma de onda quadrada com 30 volts, 20 Hz, duração de 1 ms e 200 pulsos. Os parâmetros de medição TEEI padrão são 0,5 volts e 100 Hz, 1.000 Hz, 10.000 Hz e 100.000 Hz. - Clique no botão Executar e insira os nomes apropriados para os poços 1-3 e 4-6 quando solicitado. Clique em OK para iniciar o experimento.
- Remova o feixe de eletrodos da incubadora e transfira as pastilhas de volta para os locais originais na placa do poço do experimento quando o programa terminar de ser executado.
- Misture 2 μL de Hoechst 33342, 5 μL de calceína AM e 10 μL de PI com 113 μL de meio de cultura celular em um tubo de reação de 200 μL.
- Pipete 10 μL da solução corante em cada inserto pós-pulso e coloque os insertos de volta na incubadora por 5 min.
- Transfira a placa do poço para o suporte da placa de um microscópio de imagem fluorescente com uma lente objetiva de 5x. Imagem usando campo claro e a fluorescência de cada mancha. Centralize a inserção sobre a lente antes de acionar a câmera.
NOTA: Os comprimentos de onda de excitação para PI, calceína AM e Hoechst 33342 são 558 nm, 495 nm e 353 nm, respectivamente. Os comprimentos de onda de emissão são 575 nm, 519 nm e 465 nm, respectivamente.
4. Análise dos dados
- Analisando dados de imagem com o pipeline do CellProfiler
- Use o fluxo de trabalho personalizado do CellProfiler fornecido em GitHub:https://github.com/YangLabUNL/PSEP-TEEI para processar as imagens do experimento de medição de entrega e TEEI.
- Análise – TEEI
- Clique na guia Análise na GUI.
- Alterne o indicador de tipo de impedância para TEEI na parte inferior da GUI.
- Clique na seta na caixa superior esquerda para mostrar todos os nomes dos experimentos no arquivo de dados. Selecione todos os dados de amostra de célula do experimento de medição TEEI.
- Clique na seta na próxima caixa à direita para mostrar todos os nomes de experimentos no arquivo de dados. Selecione todos os dados de inserção de controle da medição TEEI.
- Clique em Executar. Uma figura básica contendo dados de amostra de células selecionadas na frequência de medição mais baixa aparecerá.
- Na caixa de opções de exemplo no lado direito da GUI, clique na seta para mostrar todos os dados de inserção selecionados. Os valores discrepantes podem ser removidos selecionando os dados apropriados e clicando em Remover abaixo.
NOTA: Todos os dados que foram removidos da análise pelo último clique do botão Remover podem ser recuperados pelo botão Desfazer . - Clique em Concluído para passar para a próxima figura quando os dados desejados forem mostrados na figura.
- Repita as etapas 4.2.6 e 4.2.7 para os dados de amostra de célula restantes e dados de controle. Quando o conjunto de dados final for confirmado clicando em "Concluído", a figura completa da análise aparecerá.
- Gravar a figura de análise.
Resultados
O protocolo fornecido estabelece um método para usar medições TEEI para examinar os processos intermediários de eletroporação e fazer previsões de entrega, especificamente para a linha celular A431 e carga PI. Embora a modificação deste protocolo seja discutida mais adiante no artigo, é importante observar agora que, embora os valores específicos possam mudar, as tendências gerais na resposta permanecem consistentes. Por exemplo, os dados TEEI que caem abaixo da linha de base...
Discussão
A Figura 2C demonstra que os aumentos de TEEI do mínimo e as diminuições da linha de base são plotados para cada tensão da forma de onda PSEP. O aumento do TEEI cria um arco parabólico, atingindo um pico de cerca de 20 volts antes de reduzir, enquanto a diminuição do TEEI da linha de base aumenta exponencialmente à medida que a tensão aumenta. A eficiência de entrega e as porcentagens de morte na Figura 2D refletem es...
Divulgações
Os autores declaram não haver conflito de interesses.
Agradecimentos
Reconhecemos o apoio financeiro da NSF (Awards 1826135, 1936065, 2143997), dos Institutos Nacionais de Ciências Médicas Gerais do NIH P20GM113126 (Centro de Comunicação Biomolecular Integrada de Nebraska) e P30GM127200 (Centro de Nanomedicina de Nebraska), da Iniciativa Colaborativa de Nebraska e do Apoio à Bioengenharia Voelte-Keegan. O dispositivo foi fabricado no NanoEngineering Research Core Facility (NERCF), que é parcialmente financiado pela Nebraska Research Initiative.
Materiais
| Name | Company | Catalog Number | Comments |
| 15 mL Conical Centrifuge Tube | Thermo Scientific | 339651 | |
| 2-Chip Disposable Hemocytometer | Bulldog Bio | DHC-N01 | |
| 75 cm2 Tissue Culture Flask | fisherbrand | FB012937 | |
| A431 Cells | ATCC | CRL-1555 | |
| Calcein AM | Invitrogen | C3099 | |
| Class II Type A2 Biosafety Cabinet | Labgard | NU-543-600 | |
| Custom Components | YangLab | https://github.com/YangLabUNL/PSEP-TEEI | |
| Disposable Centrifuge Tube (50 mL) | fisherbrand | 05-539-6 | |
| DMEM | Gibco | 11965092 | |
| Fetal Bovine Serum | Gibco | A5670401 | |
| Fluid Aspiration System | vacuubrand | 20727403 | |
| HERACELL 240i | Thermo Scientific | 51026331 | |
| Hoechst 33342 | Thermo Scientific | 62249 | |
| Human Plasma Fibronectin | Sigma-Aldrich | FIBRP-RO | |
| Inverted Fluorescent Microscope | Zeiss | 491916-0001-000 | |
| Inverted Microscope | Labomed | TCM 400 | |
| PBS | cytiva | SH30256.02 | |
| PCR Tube 200 µL | Sarstedt | 72.737 | |
| Penicillin / Streptomycin | Gibco | 15140148 | |
| Pipette (0.2-2 µL) | fisherbrand Elite | FBE00002 | |
| Pipette (100-1000 µL) | fisherbrand Elite | FBE01000 | |
| Pipette (20-200 µL) | fisherbrand Elite | FBE00200 | |
| Pipette (2-20 µL) | fisherbrand Elite | FBE00020 | |
| Propidium Iodide | Invitrogen | P1304MP | |
| Reaction Tube 1.5 mL | Sarstedt | 72.690.300 | |
| Sorvall ST 16R Centrifuge | Thermo Scientific | 75004240 | |
| Thincert (24-well) | Greiner Bio-One | 662 641 | 0.4 µm pore diameter, 2x106 cm-2 pore density, transparent PET |
| Tissue Culture Plate (24-well) | fisherbrand | FB012929 | |
| Trypan Blue Solution | Sigma-Aldrich | T8154-20mL | |
| Trypsin | Gibco | 15090046 |
Referências
- Brooks, J., et al. High throughput and highly controllable methods for in vitro intracellular delivery. Small. 16 (51), e2004917 (2020).
- Stewart, M. P., Langer, R., Jensen, K. F. Intracellular delivery by membrane disruption: Mechanisms, strategies, and concepts. Chem Rev. 118 (16), 7409-7531 (2018).
- Canatella, P. J., Karr, J. F., Petros, J. A., Prausnitz, M. R. Quantitative study of electroporation-mediated molecular uptake and cell viability. Biophys J. 80 (2), 755-764 (2001).
- Pliquett, U., Gift, E. A., Weaver, J. C. Determination of the electric field and anomalous heating caused by exponential pulses with aluminum electrodes in electroporation experiments. Bioelectrochem Bioenerg. 39 (1), 39-53 (1996).
- Pan, J., et al. Cell membrane damage and cargo delivery in nano-electroporation. Nanoscale. 15 (8), 4080-4089 (2023).
- Boukany, P. E., et al. Nanochannel electroporation delivers precise amounts of biomolecules into living cells. Nat Nanotechnol. 6 (11), 747-754 (2011).
- Chang, L., et al. Micro-/nanoscale electroporation. Lab Chip. 16 (21), 4047-4062 (2016).
- Patino, C. A., et al. Multiplexed high-throughput localized electroporation workflow with deep learning-based analysis for cell engineering. Sci Adv. 8 (29), 7637 (2022).
- Sagvolden, G., Giaever, I., Pettersen, E. O., Feder, J. Cell adhesion force microscopy. Proc Natl Acad Sci U S A. 96 (2), 471-476 (1999).
- Ishibashi, T., Takoh, K., Kaji, H., Abe, T., Nishizawa, M. A porous membrane-based culture substrate for localized in situ electroporation of adherent mammalian cells. Sensors Actuators B: Chem. 128 (1), 5-11 (2007).
- Mukherjee, P., Nathamgari, S. S. P., Kessler, J. A., Espinosa, H. D. Combined numerical and experimental investigation of localized electroporation-based cell transfection and sampling. ACS Nano. 12 (12), 12118-12128 (2018).
- Brooks, J. R., et al. An equivalent circuit model for localized electroporation on porous substrates. Biosens Bioelectron. 199, 113862 (2022).
- Brooks, J. R., et al. Transepithelial electrical impedance increase following porous substrate electroporation enables label-free delivery. Small. 20 (25), 2310221 (2023).
- Vindiš, T., et al. Gene electrotransfer into mammalian cells using commercial cell culture inserts with porous substrate. Pharmaceutics. 14 (9), 1959 (2022).
- Ye, Y., et al. Single-cell electroporation with real-time impedance assessment using a constriction microchannel. Micromachines. 11 (9), 856 (2020).
- Bednarek, R. In vitro methods for measuring the permeability of cell monolayers. Methods Protoc. 5 (1), 17 (2022).
- Harhaj, N. S., Antonetti, D. A. Regulation of tight junctions and loss of barrier function in pathophysiology. Int J Biochem Cell Biol. 36 (7), 1206-1237 (2004).
- Hunter, R. J. . Zeta potential in colloid science: Principles and applications. 2, (2013).
- Wong, P. K., Wang, T. -. H., Deval, J. H., Ho, C. -. M. Electrokinetics in microdevices for biotechnology applications. IEEE/ASME Trans Mechatron. 9 (2), 366-376 (2004).
- Qian, K., Wang, Y., Lei, Y., Yang, Q., Yao, C. An experimental and theoretical study on cell swelling for osmotic imbalance induced by electroporation. Bioelectrochemistry. 157, 108637 (2024).
- Fox, M. B., et al. Electroporation of cells in microfluidic devices: A review. Anal Bioanal Chem. 385 (3), 474-485 (2006).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados