Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse quantitative de la réaction en chaîne par polymérase de la muqueuse gastrique pour détecter Helicobacter pylori et la résistance aux antibiotiques
Dans cet article
Résumé
Une méthode rapide et précise de détection de H. pylori et de test de résistance aux médicaments est très importante pour éradiquer efficacement H. pylori dans la pratique clinique. Ce protocole vise à présenter une méthodologie spécifique impliquant la réaction en chaîne quantitative par polymérase de la muqueuse gastrique (qPCR) pour la détection rapide de H. pylori et de la résistance aux antibiotiques.
Résumé
Helicobacter pylori est un agent pathogène principal qui infecte près de la moitié de la population mondiale et menace la santé publique en raison de sa résistance croissante aux antibiotiques. En outre, Helicobacter pylori est également responsable de gastrite chronique, d’ulcères gastriques et duodénaux, de carcinome gastrique et de lymphome du tissu lymphoïde associé à la muqueuse gastrique (MALT). Par conséquent, il est essentiel d’effectuer un diagnostic rapide et précis de H. pylori et de déterminer sa résistance aux antibiotiques. De nos jours, les méthodes existantes de diagnostic de H. pylori comprennent principalement le test rapide à l’uréase (RUT), le test respiratoire à l’urée (UBT), le test d’anticorps sériques, le test antigénique, la gastroscopie et la culture bactérienne. Cependant, les bactéries n’ont pas pu être cultivées à l’aide des cinq premières méthodes de détection, sans parler de la détection de la résistance aux médicaments. La culture bactérienne prend du temps et les tests de sensibilité aux antibiotiques ne peuvent pas être effectués rapidement et régulièrement. En milieu clinique, l’identification rapide et précise de H. pylori et de sa sensibilité aux antibiotiques est cruciale pour son élimination efficace. L’objectif de ce protocole est de décrire une approche ciblée utilisant l’amplification en chaîne par polymérase quantitative (qPCR) sur des échantillons de muqueuse gastrique afin d’accélérer le diagnostic de H. pylori et d’évaluer sa résistance aux agents antimicrobiens. La qPCR a été exploitée pour détecter le gène ureA pour l’infection à H. pylori et les mutations dans les gènes 23S rRNA et gyrA associées à la résistance à la clarithromycine et aux quinolones, respectivement. À l’heure actuelle, la qPCR de la muqueuse gastrique reste difficile en raison de l’absence de procédures opératoires standard. Par conséquent, il est essentiel de partager les méthodologies avec les détails expérimentaux pour assurer une communication précise des procédures expérimentales, contribuant ainsi à des protocoles de référence qui permettent une plus grande transparence. Dans l’ensemble, ce protocole offre une alternative économique et rapide aux méthodes conventionnelles d’évaluation de l’infection à H. pylori et de sa résistance aux antibiotiques grâce à l’application de la technologie d’amplification en chaîne par polymérase quantitative (qPCR).
Introduction
H. pylori est une bactérie à Gram négatif qui peut survivre en présence d’un faible niveau d’oxygène. L’organisme appartient à plusieurs populations génétiques distinctes et présente une grande diversité génétique. Bien que l’organisme soit généralement en forme de spirale, il pourrait être transformé en bâtonnet1. C’est un agent pathogène principal qui infecte près de 50 % de la population mondiale2. La plupart du temps, l’infection se produit lorsque les patients restent porteurs sains pendant l’enfance et que les symptômes se manifestent plus tard à l’âge adulte. Il a été rapporté qu’il est relatif à la gastrite chronique, aux ulcères gastriques et duodénaux, au carcinome gastrique et au lymphome du tissu lymphoïde associé à la muqueuse gastrique (MALT)3. Les différences dans les manifestations de l’infection à H. pylori pourraient provenir des éléments de pathogénicité de souches bactériennes distinctes, ainsi que des attributs de l’hôte et de ses habitudes alimentaires4. La recherche indique que les hommes qui fument et consomment de l’alcool courent un risque plus élevé de contracter des infections à H. pylori 5. L’efficacité de l’élimination de H. pylori pour prévenir le cancer de l’estomac et les lésions précancéreuses a été vérifiée dans plusieurs études 6,7. Par conséquent, l’éradication de H. pylori a été recommandée à titre préventif par le Centre international de recherche sur le cancer de l’Organisation mondiale de la santé (OMS)8.
Le diagnostic rapide et précis de l’infection à H. pylori est un élément essentiel du traitement pour la plupart des personnes souffrant de dyspepsie asymptomatique. Le diagnostic de H. pylori implique une combinaison d’approches invasives et non invasives. Les techniques invasives englobent généralement l’endoscopie pour la visualisation directe, l’analyse histologique d’échantillons de tissus, le test rapide de l’uréase (RUT) pour détecter l’activité bactérienne et la culture de la bactérie. D’autre part, les stratégies non invasives comprennent le test respiratoire à l’urée (UBT), qui mesure le métabolisme bactérien ; le test d’antigène dans les selles (SAT), qui détecte les protéines bactériennes dans les selles ; des tests sérologiques qui recherchent des anticorps dans le sang ; et les diagnostics moléculaires qui utilisent le matériel génétique pour la détection. Bien que chacune de ces méthodes de diagnostic présente son propre ensemble d’avantages et d’inconvénients, dans le domaine de la pratique clinique, il n’existe pas de méthode universellement reconnue comme la référence ultime9.
À l’heure actuelle, le traitement principal des infections à H. pylori est l’antibiothérapie. Cependant, les taux de réussite des thérapies antimicrobiennes visant à éradiquer H. pylori ont diminué, en raison de divers facteurs. Les principales causes d’inefficacité du traitement sont soupçonnées d’être la résistance de la bactérie aux agents antimicrobiens et l’observance du régime prescrit par les patients10. Plus précisément, les souches de H. pylori résistantes à la clarithromycine ont fait l’objet de recherches approfondies11. L’incidence d’une telle résistance est en hausse dans de nombreux pays. La résistance à H. pylori est principalement due à des mutations dans le gène de la région variable de l’ARNr 23S, qui provoque des changements conformationnels dans le ribosome. Par conséquent, le site de liaison de la clarithromycine change, ce qui affaiblit l’affinité entre H. pylori et la clarithromycine, empêchant l’inhibition de la synthèse des protéines bactériennes12. Les mutations aux positions A2143G, A2142G et A2142C dans le segment 2,9 kb du gène de l’ARNr 23S sont connues pour conférer une résistance à la clarithromycine. La propagation de la résistance aux fluoroquinolones a également fait l’objet de nombreuses recherches. Des études ont documenté des taux de résistance à la lévofloxacine de 34,5 % en Chine et de 22,1 % en Italie13. Les médicaments quinolones agissent principalement sur la topoisomérase II de H. pylori en inhibant l’activité enzymatique, affectant la synthèse et la réplication de l’ADN et la structure secondaire, atteignant ainsi des objectifs antibactériens. Si des mutations se produisent dans les gènes codant pour les sous-unités de la topoisomérase, gyrA et gyrB, cela empêchera la liaison d’antibiotiques tels que la lévofloxacine et l’enzyme, ce qui entraînera l’incapacité d’inhiber la réplication du génome de H. pylori , provoquant ainsi une résistance. Parmi ceux-ci, les points chauds des mutations du gène gyrA sont concentrés aux acides aminés 87 et 91, tandis que les mutations du gène gyrB se produisent moins fréquemment et sont souvent accompagnées de mutations dans gyrA14. Les loci de mutation du gène de résistance à la lévofloxacine comprennent principalement les six sites de mutation (A260T, C261A, T261G, G271A, G271T, A272G) situés dans le gène du gyrA. L’identification de mécanismes de résistance issus d’altérations génétiques a entraîné une transition progressive dans la détection de H. pylori, s’éloignant des méthodes basées sur la culture au profit des techniques de diagnostic moléculaire.
UBT et SAT sont les tests non invasifs les plus couramment choisis, mais ils ne peuvent pas fournir d’informations sur la sensibilité aux médicaments. Par conséquent, la mise au point d’une technique rapide et complète de détection de H. pylori et d’évaluation de sa résistance aux médicaments est cruciale pour son élimination efficace en milieu clinique15. Parmi les techniques de détection moléculaire, la réaction en chaîne par polymérase quantitative (qPCR) a connu des progrès significatifs. Contrairement à la PCR standard, la qPCR élimine le besoin d’électrophorèse sur gel et permet la quantification précise de l’ADN ou de l’ARN en incorporant des amorces et des sondes spécifiques pendant la phase de recuit. Les kits qPCR disponibles dans le commerce offrent désormais la possibilité d’identifier les infections à H. pylori et la résistance aux médicaments16.
Fondamentalement, il existe un besoin clinique immédiat d’une approche diagnostique à la fois puissante et complète, capable de détecter les infections à H. pylori et d’évaluer simultanément la résistance aux médicaments. Nous avons adopté l’analyse qPCR de la muqueuse gastrique pour la détection de H. pylori et la résistance aux antibiotiques à l’aide de différentes sondes d’amorce.
Access restricted. Please log in or start a trial to view this content.
Protocole
L’étude existante a été menée conformément aux considérations éthiques établies par le comité d’éthique de l’Hôpital populaire provincial du Guangdong, Université médicale du Sud, Guangzhou, Chine (numéro d’approbation : KY2024-1115-01). Des patients âgés de 18 à 60 ans ont été inclus dans cette étude. Pour cette étude, les participants ont été exclus s’ils avaient récemment pris des antibiotiques, des remèdes antibactériens à base de plantes, des inhibiteurs de la pompe à protons (IPP) ou des antagonistes des récepteursH2 dans les 2 semaines précédant le test. De plus, les personnes qui avaient suivi un traitement anti-H. pylori au cours des 3 derniers mois, ou celles qui présentaient des problèmes cardiaques, hépatiques ou rénaux importants, une neuropathie sévère ou des troubles psychiatriques n’étaient pas éligibles à la participation. Les personnes présentant des contre-indications à l’examen gastroscopique, telles qu’une perforation gastro-intestinale, un âge avancé, des signes vitaux instables, etc., ont été exclues. L’étude n’a pas non plus inclus les femmes enceintes ou allaitantes. Le formulaire de consentement éclairé des sujets comprend principalement les contenus suivants : (1) contexte et objectif de la recherche ; (2) Qui n’est pas apte à participer à ce projet de recherche ? (3) Quelles sont les conditions requises pour participer à la recherche ? (4) Avantages possibles de la participation à la recherche ; (5) Effets indésirables, risques et inconfort possibles ; (6) Protection de la vie privée ; (7) Droits de la personne concernée ; (8) Déclaration de l’objet ; (9) Déclaration du chercheur, etc.
1. Prélèvement de la muqueuse gastrique
- Préparation des consommables d’échantillonnage
- Préparez le tube d’échantillonnage contenant la solution de stockage.
- Préparez une pince à biopsie gastroscopique.
- Préparation des objets d’échantillonnage
- Prélever des échantillons de patients atteints de gastrite chronique, d’ulcère gastrique, d’ulcère duodénal, de cancer gastrique ou de patients suspects. Prélever des échantillons en maintenant les procédures aseptiques.
REMARQUE : Les échantillons prélevés comprenaient du tissu de la muqueuse gastrique de patients ou de patients suspects. - Confirmez les antécédents médicamenteux.
- S’assurer qu’aucun antibiotique et bismuth pour H. pylori n’est pris par le patient dans les 4 semaines et qu’aucun inhibiteur d’acide n’est pris dans les 2 semaines.
- Demander aux patients prenant des médicaments anticoagulants (warfarine, aspirine, clopidogrel, etc.) d’arrêter de prendre des médicaments pendant 5 à 7 jours.
- Prélever des échantillons de patients atteints de gastrite chronique, d’ulcère gastrique, d’ulcère duodénal, de cancer gastrique ou de patients suspects. Prélever des échantillons en maintenant les procédures aseptiques.
- Prélèvement d’échantillons
- Site d’échantillonnage
- Selon les exigences du nouveau système de Sydney, prélevez des échantillons de biopsie de l’antre gastrique, de la grande et de la petite courbure du corps gastrique et du coin gastrique (un total de 5 points). Les sites spécifiques sont illustrés à la figure 1. Une petite courbure du sinus pylorique à 2-3 cm de l’anneau pylorique (A1) et une grande courbure (A2) ; à 4 cm de l’estomac (B1) et une grande courbure à 8 cm du cardia (B2) ; coin gastrique (AI).
- Selon le consensus d’experts sur la normalisation de la biopsie endoscopique digestive et de l’examen pathologique en Chine (projet), prélevez des échantillons de biopsie du petit côté du sinus gastrique à 5 cm du pylore (adjacent au coin gastrique) ou du grand côté du coin gastrique (biopsie 1-2 morceaux).
- Description du lieu de prélèvement des échantillons spéciaux
- Pour les patients sous n’importe quel traitement, après un sevrage pendant 4 semaines, prélever simultanément des échantillons d’antre gastrique et de corps gastrique.
- Pour les patients atteints d’un cancer gastrique, prélevez du tissu de la grande courbure de l’estomac mais pas du tissu tumoral.
- Pour les patients souffrant d’ulcères gastriques, prélevez l’antre gastrique et le tissu du bord de l’ulcère gastrique. Ne collectez pas le bas de l’ulcère.
- Pour les patients atteints de gastrite atrophique, prélever l’antre gastrique et le corps gastrique. Ne prélevez pas d’échantillons dans les zones d’atrophie de la muqueuse gastrique et de métaplasie intestinale.
- Étapes d’échantillonnage
- Ouvrez la valve de la pince de biopsie et déplacez-la lentement vers la main dominante. Lorsque la tête de la pince est dans le champ de vision, ouvrez le rabat de la pince et manipulez l’endoscope au niveau de la muqueuse gastrique du site de prélèvement.
- Appliquez une légère pression, fermez la pince à biopsie et clampez le tissu de la muqueuse gastrique (diamètre : 0,5-1 mm, environ 5-10 mg/bloc). Extrayez les échantillons de tissus à l’aide de la pince dans le tube d’échantillonnage (y compris le liquide de préservation) pour compléter le site.
- Continuer à compléter l’échantillonnage des autres sites et les placer dans le même tube d’échantillonnage (y compris la solution de préservation).
- Terminez l’échantillonnage de tous les sites, serrez le couvercle du tube d’échantillonnage et scellez-le pour éviter le dessèchement. Étiquetez les tubes avec un numéro d’identification unique à l’extérieur.
- Soumettez les échantillons collectés pour analyse dès que possible. Ne placez pas les échantillons à température ambiante (RT) pendant plus de 24 h. Évitez la congélation et la décongélation répétées des échantillons cliniques pendant le transport sur de longues distances. Si une température de -20 ± 5 °C ne peut être assurée, transportez-les à 0-8 °C. Conservez les échantillons pendant une période de cinq mois à -20 ± 5 °C et, pour un stockage à long terme, maintenez-les en dessous de -70 °C.
- Précautions
- Lors du prélèvement, appuyez le plus profondément possible sur la pince à biopsie pour atteindre toute la couche de muqueuse.
- Lorsque vous retirez la pince à biopsie et ouvrez la biopsie, soyez doux pour éviter les déchirures de la muqueuse.
- Site d’échantillonnage
2. Extraction des acides nucléiques
- Réglez à l’avance la température du bain métallique à 100 °C ; si l’échantillon est congelé, retirez-le et restaurez-le dans RT.
- Mélangez bien le lysat pour suspendre la résine d’acide iminodiacétique. Prenez 100 μL de lysat dans le tube centrifuge avec l’élément filtrant, ou ajoutez directement le tissu de la muqueuse gastrique dans le tube de centrifugation avec 200 μL de lysat et mélangez avec l’oscillation vortex.
- Placez le tube de centrifugation dans un bain métallique à 100 °C pendant 10 min, puis retirez-le et laissez-le refroidir à RT.
- Centrifugeuse à 9500 g pendant 5-10 min. Aspirez soigneusement le surnageant dans un autre tube à centrifuger stérilisé et marquez-le.
- Effectuez immédiatement l’expérience suivante ou stockez-la temporairement à 4 °C pour inspection.
3. Détection par qPCR de H. pylori et des gènes de résistance aux médicaments (clarithromycine et quinolones)
- Retirez la sonde d’amorce, la solution enzymatique mixte et le tampon de détection du kit. Faites fondre tous les composants sur de la glace ou entre 2 et 8 °C, et mélangez-les en secouant légèrement et en effectuant une centrifugation instantanée à basse vitesse.
- Mélanger 12,5 μL de tampon de détection + 7,0 μL de sonde d’amorce + 0,5 μL de solution enzymatique. Calculez d’abord la quantité de chaque réactif, ajoutez-le dans le tube de centrifugation de volume approprié, mélangez bien et centrifugez brièvement.
REMARQUE : La quantité totale de la solution de réaction PCR doit être calculée en incluant le nombre d’échantillons de test, un témoin positif fort, un témoin positif faible, un témoin négatif et un témoin négatif PCR. - Une fois la solution de réaction PCR configurée, ajoutez 20,0 μL de la solution de réaction PCR dans chaque puits de réaction PCR. Ensuite, ajoutez 5,0 μL d’acide nucléique à la réaction PCR avec la solution de réaction PCR. N’ajoutez aucun échantillon ou acide nucléique dans le puits de contrôle négatif de la PCR.
- Placez le tube réactionnel dans le thermocycleur PCR fluorescent et réglez les paramètres du cycle comme suit : tout d’abord, chauffez le mélange réactionnel à 42 °C et 95 °C pendant 2 min ; deuxièmement, dénaturer pendant 10 s à 95 °C, recuire et étendre pendant 45 s à 58 °C, répéter l’opération pendant 40 cycles.
- Collectez les signaux de fluorescence sous forme de FAM (mutation du gène de l’ARNr 23S), HEX/VIC (mutation du gène gyrA ), ROX (H. pylori) et CY5 (étalon interne), ainsi que des données à 58 °C.
- Après la réaction, enregistrez les données et analysez-les à l’aide d’un logiciel qPCR spécifique.
Access restricted. Please log in or start a trial to view this content.
Résultats
Évaluation de l’infection à H. pylori et de la résistance aux antibiotiques dans le tissu gastrique par qPCR
Les tests qPCR pour identifier H. pylori ont été réalisés en ciblant le gène ureA , et la résistance aux antibiotiques a été déterminée en examinant des mutations spécifiques dans les gènes 23S rRNA et gyrA (tableau 1). Les données d’assurance de la quali...
Access restricted. Please log in or start a trial to view this content.
Discussion
Les tests traditionnels tels que RUT, UBT, histologie, culture ainsi que la sérologie sont exploités pour la détection de H. pylori. Chaque approche diagnostique offre des avantages distincts et fait face à des défis spécifiques en fonction du contexte clinique17. La culture de H. pylori à partir de biopsies de la muqueuse gastrique est souvent considérée comme la référence pour le diagnostic. Néanmoins, cette méthode demande beaucou...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Aucun.
Remerciements
Ce travail a été soutenu par des subventions du programme d’incubation NSFC du GDPH (8220080645) et du projet du Fonds provincial de recherche en sciences et technologies médicales du Guangdong (A2024108).
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Bath Incubator | ALLSHENG | MK2000-2 | Provide a constant temperature environment |
| Biosafety cabinet | Haier | HR1500-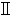 B2 B2 | |
| Centrifuge | Thermo Fisher Scientific | THERMO ST16R | Centrifuge the residual liquid off the wall of the tube. |
| Chelex-100 | sigma | C7901 | Resin |
| Gastroscope biopsy forceps | Boston Scientific Corporation | Sampling of the gastric mucosa | |
| Helicobacter pylori 23S rRNA gene and gyrA gene mutation detection kit | Jiangsu Mole Bioscience | CFDA 20223400137 | qPCR detection kit for H. pylori and drug-resistance genes (clarithromycin and quinolones) |
| Nucleic acid extraction reagent | Jiangsu Mole Bioscience | SEDA 20150076 | For DNA extraction |
| SDS Software | Applied Biosystems | 7300/7500 | Data analysis |
| Thermocylcer | Thermo Fisher Scientific | ABI 7500 | For qPCR detection of H. pylori and drug-resistance genes (clarithromycin and quinolones) |
| Vortex mixer | JOANLAB | VM-5005 | For mixing reagent |
Références
- Sharndama, H. C., Mba, I. E. Helicobacter pylori: an up-to-date overview on the virulence and pathogenesis mechanisms. Braz J Microbiol. 53 (1), 33-50 (2022).
- Ravikumara, M. Helicobacter pylori in children: think before you kill the bug. Therap Adv Gastroenterol. 16 (6), 1-10 (2023).
- Reshetnyak, V. I., Burmistrov, A. I., Maev, I. V. Helicobacter pylori: Commensal, symbiont or pathogen. World J Gastroenterol. 27 (7), 545-560 (2021).
- Al-Ouqaili, M. T. S., et al. Study of vacuolating cytotoxin A (vacA) genotypes of ulcerogenic and non-ulcerogenic strains of Helicobacter pylori and its association with gastric disease. Saudi J Biol Sci. 30 (12), 103867(2023).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Association between alcohol consumption, cigarette smoking, and Helicobacter pylori infection in Iraqi patients submitted to gastrointestinal endoscopy. J Emerg Med Trauma Acute Care. 6, 12(2022).
- Thrift, A. P., Wenker, T. N., El-Serag, H. B. Global burden of gastric cancer: Epidemiological trends, risk factors, screening and prevention. Nat Rev Clin Oncol. 20 (5), 338-349 (2023).
- Liou, J. M., et al. Screening and eradication of Helicobacter pylori for gastric cancer prevention: The Taipei global consensus. Gut. 69 (12), 2093-2112 (2020).
- Gao, T. Y., et al. Cancer burden and risk in the Chinese population aged 55 years and above: A systematic analysis and comparison with the USA and Western Europe. J Glob Health. 14, 04014(2024).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of Helicobacter pylori infection by invasive and non-invasive techniques in patients with gastrointestinal diseases from Iraq: A validation study. PLoS One. 16 (8), e0256393(2021).
- Thung, I., et al. Review article: The global emergence of Helicobacter pylori antibiotic resistance. Aliment Pharmacol Ther. 43 (4), 514-533 (2016).
- Zhang, Y., et al. Mutations in the antibiotic target genes related to clarithromycin, metronidazole and levofloxacin resistance in Helicobacter pylori strains from children in China. Infect Drug Resist. 13 (1), 311-322 (2020).
- Saranathan, R., et al. Helicobacter pylori Infections in the Bronx, New York: Surveying antibiotic susceptibility and strain lineage by whole-genome sequencing. J Clin Microbiol. 58 (3), e01591-e01619 (2020).
- Malfertheiner, P., et al. Management of Helicobacter pylori infection - The Maastricht IV/ Florence consensus report. Gut. 61 (5), 646-664 (2012).
- Palma, G. Z. D., et al. Occurrence of mutations in the antimicrobial target genes related to levofloxacin, clarithromycin, and amoxicillin resistance in Helicobacter pylori isolates from Buenos Aires city. Microb Drug Resist. 23 (3), 351-358 (2017).
- Zhang, L., Zhao, M., Fu, X. S. Gastric microbiota dysbiosis and Helicobacter pylori infection. Front Microbiol. 14, 1153269(2023).
- Peng, X., et al. Gastric juice-based real-time PCR for tailored Helicobacter Pylori treatment: A practical approach. Int J Med Sci. 14 (6), 595-601 (2017).
- Weingart, V., et al. Sensitivity of a novel stool antigen test for detection of Helicobacter pylori in adult outpatients before and after eradication therapy. J Clin Microbiol. 42 (3), 1319-1321 (2004).
- Chi, W. J., et al. A rapid and high-throughput multiplex genetic detection assay for detection, semi-quantification and virulence genotyping of Helicobacter pylori in non-invasive oral samples. Front Cell Infect Microbiol. 13, 1267288(2023).
- Zhang, Y. M., et al. Validation of a high-throughput multiplex genetic detection system for Helicobacter pylori identification, quantification, virulence, and resistance analysis. Front Microbiol. 7, 1401(2016).
- Gisbert, J. P. Empirical or susceptibility-guided treatment for Helicobacter pylori infection? A comprehensive review. Therap Adv Gastroenterol. 13, 1756284820968736(2020).
- Bénéjat, L., et al. Real-time PCR for Helicobacter pylori diagnosis. The best tools available. Helicobacter. 23 (5), e12512(2018).
- Vasapolli, R., et al. Real-time assessment of H. pylori infection to guide molecular antibiotic resistance testing: A combined endoscopy-gastric juice analysis approach. Aliment Pharmacol Ther. 61 (3), 465-471 (2025).
- Hussein, R. A., Al-Ouqaili, M. T. S., Majeed, Y. H. Detection of clarithromycin resistance and 23SrRNA point mutations in clinical isolates of Helicobacter pylori isolates: Phenotypic and molecular methods. Saudi J Biol Sci. 29 (1), 513-520 (2022).
- Wang, Y. H., et al. A systematic review and meta-analysis of genotypic methods for detecting antibiotic resistance in Helicobacter pylori. Helicobacter. 23 (2), e12467(2018).
- Egli, K., et al. Comparison of the diagnostic performance of qPCR, Sanger sequencing, and whole-genome sequencing in determining clarithromycin and levofloxacin resistance in Helicobacter pylori. Front Cell Infect Microbiol. 10, 596371(2020).
- Liu, Y., et al. Relationship between Helicobacter pylori infection and colorectal polyp/colorectal cancer. World J Gastrointest Surg. 16 (4), 1008-1016 (2024).
- Oliver, A. S., Wu, F., Chen, Y. The role of gastric microbiota in gastric cancer. Gut Microbes. 11 (5), 1220-1230 (2020).
- Hulten, K. G., et al. Comparison of culture with antibiogram to next-generation sequencing using bacterial isolates and formalin-fixed, paraffin embedded gastric biopsies. Gastroenterology. 161 (5), 1433-1442 (2021).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon