Method Article
זיהוי של אמנון אגם וירוס באמצעות קונבנציונאלי RT-PCR ו SYBR ירוק RT-qPCR
In This Article
Summary
פרוטוקול זה מאבחן אמנון אגם וירוס (TiLV) ברקמות אמנון באמצעות מתודולוגיות RT-PCR. השיטה כולה מתוארת מן דיסקציה רקמות כדי הכולל החילוץ RNA, ואחריו cDNA סינתזה וזיהוי של TiLV באמצעות PCR קונבנציונאלי או PCR כמותי באמצעות dsDNA מחייב צבע הכריכה פלורסנט.
Abstract
המטרה של שיטה זו היא כדי להקל על זיהוי מהיר, רגיש וספציפי של אמנון אגם וירוס (TiLV) ברקמות אמנון. פרוטוקול זה יכול לשמש כחלק מהתוכניות מעקב, אמצעים biosecurity ובמעבדות המחקר הבסיסי TiLV. תקן הזהב של וירוס אבחון כוללת בדרך כלל וירוס בידוד ואחריו טכניקות משלימים כגון הפוכה-תמלול תגובת שרשרת פולימראזית (RT-PCR) לצורך אימות נוסף. זה יכול להיות מסורבל, זמן רב, בדרך כלל דורש דגימות רקמה בכבדות נגוע בווירוס. השימוש RT-כמותיים (q) PCR זיהוי וירוסים הוא יתרון בשל טבעה כמותית, רגישות גבוהה, ירידה לפרטים, מדרגיות, הגיע הזמן מהיר כתוצאה מכך. כאן, השיטה כולה של PCR גישות מבוססות על TiLV זיהוי מתואר, בין אמנון איברים אופטים, הכולל חומצה ריבונוקלאית (RNA) החילוץ באמצעות a guanidium thiocyanate-פנול-כלורופורם פתרון, כימות RNA, ואחריו ה-PCR שני שלבים פרוטוקול פרוגרמה, סינתזה חומצה deoxyribonucleic משלימים (cDNA), זיהוי של TiLV באמצעות PCR קונבנציונאלי או זיהוי כמותית באמצעות qPCR באמצעות SYBR ירוק אני צובע. PCR קונבנציונלית דורש צעדים פוסט-PCR, פשוט יודיע על הנוכחות של הנגיף. הגישה השנייה תאפשר על כימות מוחלטת של TiLV עד כמה שני עותקים, ולכן הוא שימושי במיוחד עבור TiLV האבחנה במקרים תת קליניים. תיאור מפורט של שתי גישות PCR, נציג תוצאות ממעבדות שני, דיון יסודי פרמטרים קריטיים של שניהם נכללו כדי להבטיח כי חוקרים, מאבחנים למצוא מתאימים שלהם וישימים שיטת זיהוי TiLV.
Introduction
אספקת דגים לנפש העולמי הגיע שיא חדש של 20 ק"ג בשנת 2014, זה היה עקב צמיחה נמרצת של עופות ומידגה. עופות ומידגה נותר אחד של הצמיחה המהירה מזון מן החי לעשיית המגזרים ברחבי העולם, ענף המזון רק בבעלי חיים בייצור כי הוא גדל מהר יותר האוכלוסייה האנושית1. Tilapiine cichilds מהווים את הדגים מים מתוקים השני הכי חשוב מגודלים ברחבי העולם עם הפקה גלובלי הכולל של 6.4 מיליון טון (MT), ערך מוערך 9.8 מיליארד דולר בשנת 20152. המפיקים בעשיריה הפותחת של אמנון הם סין (1.78 MT), אינדונזיה (1.12 MT) למצרים (0.88 MT), ואחריו בנגלדש, וייטנאם, הפיליפינים, ברזיל, תאילנד, קולומביה ו אוגנדה2. הוא צפוי כי אמנון העולמי הייצור יהיה בסביבות 7.3 MT ב- 20303. אמנון הפכו כזה מקור המזון העולמי חשוב לא רק כי הם מקור זול של חלבון4 אלא גם כי הם קלים להתרבות בתפקיד תחת מגוון רחב של המים והאקלים תנאים5,6. רק לפני כמה עשרות שנים, האמינו כי היו כמה מחלות מסחרית משמעותית מאיים אמנון חקלאות אבל זה כבר לא נכון. מחלה נגיפית המתעוררים המכונה אמנון אגם וירוס מחלת (TiLVD) היא המגיפה מחלה קריטית פעם הראשונה נמצאו אמנון כל התעשייה נמצא בסיכון. מחלה זו מהווה איום ישיר על מזון אבטחה עבור מיליוני אנשים ב אפריקה7, אסיה, דרום אמריקה, ויש השלכות סוציו-אקונומי. בתחילת שנת 2018, הארגון העולמי לבריאות בעלי חיים (OIE) דיווחו כי הסוכן etiological של המחלה, TiLV, באופן רשמי זוהו על שלוש יבשות כיסוי מדינות שמונה8 , מאז היה כרטיס מידע זה פתוגן עדכון שם כבר יותר דיווחים על TiLV טנזניה, אוגנדה9, אינדונזיה10, טייוואן11 ופרו12. TiLV הוא נגיף RNA חד גדילי הרומן שמתואר להיות וירוס כמו orthomyxo מכיוון שהוא מכיל מגוון רחב של מאפיינים של אחרים orthomyoxoviruses כגון שפעת או זיהומיות באנמיה סלמון וירוס (ISAV)13. זה זוהה לראשונה בעקבות הפסדים מסיבי של אמנון מעובדים הפראית ב אגם הגליל, ישראל14. לאחר מכן, התפרצות מחלות דומות התייחס בהירושימה הקיץ וגם את חודש דווח על תסמונת התמותה המשויך TiLV זיהום הנילוס אמנון (Oreochromis niloticus) מצרים15 ו הנילוס, היברידית אדום אמנון (Oreochromis spp.) תאילנד16, בהתאמה. זיהוי של וירוסים בעלי חיים ימיים היסטורית מתבצעת על ידי צמיחה ובידוד של וירוסים תרבית תאים. שורות תאים שונים נבדקו עבור הפצת ובידוד של TiLV כולל, E-11 תאים שמקורם ראש-נחש דגים (Ophiocephalus striatus)17,18, לכם, שוייץ שמקורן Oreochromis mossambicus18, ואת OnlB OnlL שמקורם הנילוס אמנון (O. niloticus)19. בעוד וירוס תרבות יש את היתרון שהיא מספקת חומרים לניסויים נוספים, יש לו החיסרון כי זה דורש לפחות 4-7 ימים כדי לבחון את היווצרות של תופעות cytopathic (CPE), ויותר חשוב, וירוסים בית שונים, שהם יותר להשתלב שכפול עשוי להיות מופצות ומפיקים CPE דומה.
במשך העשורים האחרונים, היה מהלך ספורים המסורתית, לעיתים זמן רב שיטות אבחון כמו תרבית תאים, סרולוגיה, זיהוי אנטיגן והחלפת עם חומצת גרעין מהר יותר, רגיש יותר המבוסס על זיהוי בדיקות20, 21. זו ניכרת על ידי העובדה כי מבחני qPCR רבים פותחו כמו שיטות אבחון חשוב עבור מספר רב של מחלות נגיפיות, בעלי חיים ימיים, כגון עבור22,ISAV23, וירוס ויראלי טראומה אלח דם (VHSV)24 ,25, אילנה אסטרייך salmonid27 26,betanodavirus28, דגים iridovirus29, Anguillid ההרפס 1 (AngHV1)30ו- Lymphocystis מחלת וירוס (LCDV)31 . שיטות יציבות למעקב ואבחון הפתוגן דרושים בדחיפות כדי לצמצם את התפשטות TiLV. שיטות כאלה צריך לאפשר גילוי מוקדם של זיהום לפני סימנים קליניים לפתח וזיהוי וירוסים נמוך נטען. עד כה, בפרוטוקולי PCR שונים לרבות RT-PCR14,32, RT-PCR מקוננים18, RT-PCR למחצה מקוננים33ו- RT-qPCR32,34 פותחו עבור זיהוי TiLV ברקמות דגים. השוואה של בידוד RT-qPCR ווירוסים בשורות תאים רגישים לזיהוי TiLV גילה כי RT-qPCR היה רגיש פי אלף יותר מאשר הבידוד של וירוס32. למרות כל שפורסמו PCR פרוטוקול דיווח על רגישויות שונות לאיתור TiLV, רוב מבחני רגישים מאוד עם הגבולות לזיהוי עותקים ויראלי-עותקים 7.533, 7 עותקים18 או עותקים 232 לכל התגובה.
מטרת מאמר זה שיטות היא להסביר בפירוט כיצד לבצע מבחני איתור TiLV, החל מאיסוף רקמות אמנון, כדי החילוץ RNA הכולל, סינתזה cDNA וביססנו PCR ספציפי TiLV מבחני. באופן ספציפי, פרוטוקולים מקיפה הן קונבנציונאלי RT-PCR, והן גם SYBR מבוסס-ירוק RT-qPCR תוארו לערעור בפני מגוון רחב של מדענים במטרה לאתר את TiLV. לשעבר הוא פחות רגיש, אבל הוא בדרך כלל אפשרות זיהוי זולה יותר. האחרון דורש תשתית מורכבים יותר כגון מכונת ה-PCR כמותי, ריאגנטים יקר יותר, אך יש לו את היתרונות של להיות כמותיים, מהיר, רגישה מאוד, כלומר, כי זה יכול לשמש עבור הגילוי של TiLV ב תת קלינית דגים נגועים. RT-PCR ופרוטוקולים RT-qPCR בוצעו במעבדות שונות שני עם מבודד גיאוגרפיים ברורים של TiLV והאר תוצאות כלול הרגישות ואת הפארמצבטית של מבחני המתוארים כאן.
Protocol
פרוטוקול השימוש בבעלי חיים עבור מחקר זה אושרה על ידי ועדת האתיקה חיה של אוניברסיטת Kasetsart תחת אישור מספר ACKU 59-וטרינר-016.
הערה: נא עיין טבלה של חומרים עבור מידע מורחב לגבי ראגנטים וציוד הציע עבור פרוטוקול זה.
1. רקמות דוגמה אוסף
- המתת חסד הדג באמצעות מנת יתר של שמן ציפורן (אמצעי האחסון תלוי בגודל של דגים וריכוז של מוצרים, בדרך כלל יותר מ 3 מ ל/L). לטבול רבע המספריים מלקחיים ומיונז לתוך אתנול 95% (v/v) ואחריו שורף את הציוד באמצעות צורב של אלכוהול כדי לחטא את הציוד.
הערה: Tricaine methanesulfonate (MS-222) יכול לשמש במקום שמן ציפורן. - למצוא את הכבד לחתוך חתיכה קטנה (כ 20-100 מ"ג) או לאסוף µL 200 של ריר באמצעות כיסוי זכוכית או להב כירורגי כדי להסיר, ריר והשתרשה עמוק בלבה את ישבנה של הדג אמנון ומניחים את הדגימות לתוך צינור microcentrifuge 1.5 mL.
- דוגמאות תהליכים באופן מיידי, לאחסן ב- RNA ייצוב פתרון או להעביר אותם אל-80 ° C עד שימוש נוסף.
הערה: המשימה הגדולה ביותר בעבודה עם ה-RNA הוא הכנת מולקולות RNA תקין, ולשמור אותם תקינים לאורך כל handlings הבאים. עמוד השדרה ה-RNA הוא מולד רגיש יותר נזק מאשר ה-DNA. חילוץ ובידוד של RNA הכולל מתאי רקמת מחייבת טכניקה מעבדה זהיר; לקחת את כל הוראות למניעת זיהום RNase על-ידי לבישת כפפות, באמצעות מים חינם RNase, ריאגנטים, ציוד, כלי פלסטיק, זכוכית, מרחב עבודה, באמצעות טיפים מסנן עבור pipetting.
2. Guanidium Thiocyanate - פנול - כלורופורם החילוץ של RNA
- להוסיף 1 מ"ל של פתרון monophasic המכיל isothiocyanate פנול, guanidine לתוך צינור המכיל לדגימות מהסעיף 1.
התראה: פתרון זה הוא רעיל מאוד, חייבים להיות מטופלים עם טיפול תא למינארי עם ציוד מגן, על-ידי לבישת כפפות משקפי שמש, בגדים ובטיחות הגנה נאותה. - לטחון את דגימת רקמה באמצעות רקמות שהעקב מהמגן עד הומוגנית.
הערה: דגימות יכול גם להיות הומוגני באמצעות מהמגן כוח בשילוב עם חרוזים קרמיים. להבטיח כי דגימת הרקמות הוא הומוגני לגמרי לפני שתמשיך לשלב הבא או לעצור את הפרוטוקול פה ולאחסן את הדוגמאות באופן מלא homogenized ב-80 מעלות צלזיוס עד שימוש נוסף. - להוסיף 200 µL של כלורופורם עבור שלב ההפרדה.
התראה הכלורופורם סם פוטנציאליים והוא מאוד מסוכנים. זה צריך להיות מטופל עם טיפול תא למינארי עם ציוד מגן, כמו גם על-ידי לבישת כפפות משקפי שמש, בגדים ובטיחות הגנה נאותה. כמו פחות רעיל, אלטרנטיבי, 1-ברומו-3-chloropropane יכול לשמש גם.
הערה: גודל אמצעי האחסון, למעלה או למטה במקומות המתאימים. לדוגמה, אם רק 500 µL של monophasic, לאודנום isothiocyanate פנול, guanidine שימש, ואז להוסיף רק 100 µL של כלורופורם בשלב זה.- לערבב דגימות היטב על ידי היפוך עבור 15 s.
- דגירה בדגימות למשך 3 דקות בטמפרטורת החדר (RT).
- צנטריפוגה עבור 15 דקות ב 12,000 × g ו- 4 מעלות צלזיוס.
הערה: צריך להיות הפרדה ברורה לתוך שלב אורגני התחתון, לאטמוספרה לבן של שלב מימית העליון המכיל RNA. השלב העליון הזה הוא בדרך כלל חסר צבע אך בהתאם הסוג והכמות של רקמות הומוגני, זה יכול להיות מראה ורוד בהיר. - העברה השכבה העליונה מימית (כ-500 µL) צינור microcentrifuge טריים מבלי להפריע את לאטמוספרה.
הערה: אל תנסו להעביר את שלב מימית כולו, להשאיר כמות קטנה כדי למנוע כל אפשרות של הרנ א המכיל פאזה מימית עם האורגני זיהום או לאטמוספרה. - להוסיף 1 נפח של 100% אלכוהול איזופרופיל, כדי לזרז את הרנ א.
- לחלופין, אם שימשו בכמויות קטנות מאוד של רקמות, לאחר מכן להוסיף 1 µL (5-10 µg) של גליקוגן נטולת RNase כל מדגם לקידום יעיל RNA משקעים. זה יסייע זיהוי בגדר RNA בשלב 2.8.
הערה: הגליקוגן משמש כנשא של RNA, תמנע כמויות קטנות של RNA נדבקות הצד של הצינור. - לערבב צינורות היטב על ידי היפוך מספר פעמים.
- לאחסן את הדגימות עבור 2 h ללון ב-20 ° C.
- לחלופין, אם שימשו בכמויות קטנות מאוד של רקמות, לאחר מכן להוסיף 1 µL (5-10 µg) של גליקוגן נטולת RNase כל מדגם לקידום יעיל RNA משקעים. זה יסייע זיהוי בגדר RNA בשלב 2.8.
- צנטריפוגה דגימות למשך 15 דקות ב- 12,000- g ו- 4 מעלות צלזיוס.
- למחוק את תגובת שיקוע, נזהר לא להוציא בגדר RNA בתחתית של התחתית microcentrifuge.
- להוסיף 1 מ"ל של 75% אתנול (v/v) ולערבב דגימות ה-RNA על-ידי היפוך ברכבת התחתית מספר פעמים.
- צנטריפוגה למשך 15 דקות ב 10,000 x g ו- 4 מעלות צלזיוס.
הערה: הפרוטוקול ניתן לעצור כאן, ניתן לאחסן את הדגימות הכוללת בגדר RNA 75% אתנול ב-20 ° C עד שימוש נוסף. - למחוק את תגובת שיקוע, להיות הפקוחה לא להוציא בגדר RNA בתחתית של התחתית microcentrifuge.
- באופן אופציונלי, חזור על השלבים 2.9-2.11 באמצעות אתנול 70% (v/v). ביסודיות לשטוף בגדר RNA יצמצם לך מלח או מזהם carry-over שעלולות לפגוע יהיה רגיש יישומים במורד הזרם.
- למשוך האתנול הנותרים בעזרת פיפטה של, ואז מילה נהדרת בגדר RNA בטמפרטורת החדר במשך לא יותר מ 5-10 דקות.
הערה: כדורי יתר מיובשים יהיה קשה להשעות מחדש. - להוסיף 30-60 µL של מים נטולי RNase, מראש התחמם עד 55-60 ° C עד solubilize בגדר RNA.
- מניחים את הרנ א על הקרח לשימוש מיידי או חנות ב-80 מעלות צלזיוס לשימוש מאוחר יותר.
3. לכמת ריכוז ה-RNA באמצעות ספקטרופוטומטרים מיקרו בנפח
- לעבור את ההגדרות של ספקטרופוטומטרים ה-RNA.
- השתמש µL 1-2 של מים נטולי RNase ריקים.
- השתמש µL 1-2 של כל מדגם ה-RNA כדי להעריך את כמות ה-RNA.
- להקליט את הקריאות ב- 230 nm, 260 ננומטר, 280 ננומטר עבור כל דגימה.
- לדלל את הרנ א ל-200 ng/µL באמצעות RNase ללא מים.
4. סינתזה של DNA משלימים (cDNA) בעזרת RNA סה
- לערבב 1 µg של RNA הכולל של פרוטוקול 2, 2 מיקרומטר oligo (dT), תערובת dNTPs 0.5 מ מ, להביא את עוצמת הקול הסופי µL 10 במים נטולי נוקלאז. בשביל זה, להכין RT-מאסטר-תערובת על פי מספר דוגמאות ופקדים להיבדק.
הערה: הפקדים הם מדגם חיסור-טרנסקריפטאז (-RT) שבו האנזים RT מוחלף במים נטולי נוקלאז (ראה שלב 4.3) ופקד אין תבנית (NTC) שבו מים נטולי נוקלאז מתווסף המאסטר-מיקס במקום תבנית ה-RNA.- מערבבים את הדגימות היטב על ידי pipetting ואחריו צנטריפוגה קצרה.
- דגירה בדגימות ב 65 מעלות צלזיוס למשך 5 דקות ולאחריו דגירה 2 דקות על קרח.
- Centrifuge בקצרה את הדוגמאות כדי לאסוף את כל הנוזל בתחתית של הצינורות.
- להוסיף 1 x רוורס טרנסקריפטאז מאגר, 100 U רוורס טרנסקריפטאז ולהביא הנפח הסופי של כל מדגם µL 20 בעזרת נוקלאז ללא מים.
- מערבבים את הדגימות היטב על ידי pipetting ואחריו צנטריפוגה קצרה.
- דגירה בדגימות ב 42 מעלות צלזיוס למשך 60 דקות ולאחריו 85 מעלות צלזיוס במשך 5 דקות.
- לדלל את cDNA מסונתז כדי ריכוז הרצוי על-ידי הוספת אמצעי אחסון המתאים של מים נטולי נוקלאז למקם את cDNA בקרח לשימוש מיידי או לאחסן אותו ב-20 ° C לשימוש מאוחר יותר.
5. TiLV PCR קונבנציונלי
- השתמש cDNA, דגימות פקדים, המופקים פרוטוקול סעיף 4 כתבניות לתגובה PCR באמצעות כל אחד הזוגות פריימר הוקמה מפורט בטבלה 1, יחד עם ה-DNA פולימראז של בחירה.
הערה: פקד מתבנית לא נוספים (NTC) יש לכלול כאן על ידי החלפת cDNA למים ללא נוקלאז בעקבות תגובות PCR. פקד חיובי, אם הוא זמין, צריך גם להיות כלול הכוללת לאמת בעבר TiLV דוגמאות חיוביות או השבר cDNA TiLV המתאים משוכפלות ב פלסמיד. - להכין PCR מאסטר-שילוב בהתאם לקווים המנחים של מערכת ה-DNA פולימראז השימוש ומספר דגימות ופקדים להיבדק. תערובת זו צריכה לכלול את פריימר לפנים, פריימר הפוכה, dNTPs, MgCl2 ו- DNA פולימראז שנבחר, יחד עם המאגר שלו.
- על פי ההנחיות של פולימראז שנבחר ה-DNA, לשלב נפח המיועד של מאסטר-מיקס בסכום המוצע של cDNA דוגמאות ודוגמאות שליטה.
הערה: הכנת של 0.5 x עודף התגובה היא כדאית במקרים רבים מאז חלק המאסטר-לערבב אובדים במהלך pipetting. - לבצע PCR תנאים בהתאם לקווים המנחים של מערכת שימוש DNA פולימראז רכיבה על אופניים, שימוש של הטמפרטורה המתאימה מחזק תחל בשימוש (טבלה 1). בדרך כלל, תוכנית כזו יהיה כרוך דנטורציה הראשונית ב 95 מעלות צלזיוס למשך 2-5 דקות, ואחריו 30-40 מחזורי דנטורציה ב 95 ° C ל 30 s, חישול-הטמפרטורה המומלצת עבור s 30 ו התארכות ב-72 מעלות ל 30-s, ואחריו התארכות הסופי ב-72 מעלות במשך 5-10 min.
- לטעון µL 5-15 של כל התגובה PCR ואת סולם הדנ א המתאים ל בארות של ג'ל agarose 1-2%, בהתאם לגודל הצפוי של מוצר ה-PCR. להפריד את ה-DNA מוגבר על-ידי אלקטרופורזה בג'ל, מכתים את הג'ל על ידי אתידיום ברומיד (EthBr) כדי להקל על ויזואלזציה להקות ה-DNA של הגודל הצפוי (טבלה 1) במכונה תיעוד ג'ל באמצעות אור UV.
התראה: EtBr הוא רעיל; זה צריך להיות מטופל בקפידה על-ידי לבישת ביגוד מגן נאותה של כפפות בטיחות.
| המטרה TiLV הגנום פלח | פריימר לפנים 5 - 3' | פריימר הפוכה 5 - 3' | גודל המוצר PCR (bp) | Tm ° C | הפניה מקורית | ||||
| 1 | CCAAACGTTATCTCTTAATTACGCAC | GCAAATATTTCTCTCATTCGCCT | 1641 | 50 | Surachetpong et al., 2017 | ||||
| 1 | CCTCATTCCTCGTTGTGTAAGT | AGGAGTTGCTGTTGGGTTATAG | 1000 | 62 | Mugimba et al., 2018 | ||||
| 2 | ACTCTCTATTACCAAATACATTTACT | TTACCATATATATAGTGAAGGC | 1445 | 45 | Surachetpong et al., 2017 | ||||
| 2 | GTCCAGGGCGGTATGTATTG | CTTACGGCTGACAAGTCTCTAAG | 834 | 62 | Mugimba et al., 2018 | ||||
| 3 | GTTGGGCACAAGGCATCCTA | TATCACGTGCGTACTCGTTCAGT | 250 | 56 | Eyngor et al., 2014 | ||||
| 3 | TATGCAGTACTTTCCCTGCC | TTGCTCTGAGCAAGAGTACC | 491 | 57 | Eyngor et al., 2014 | ||||
| 3 | ACCCCTTAATCCTTAATAGACCGTTA | CCCATAATCCTCTATTAGAACGTCGT | 1352 | 50 | Surachetpong et al., 2017 | ||||
| 3 | GTCGAGGCATTCCAGAAGTAAG | GAGCTAAGGGAACGGCTATTG | 834 | 62 | Mugimba et al., 2018 | ||||
| 4 | AGCAGCAGCAGGAGAAAGAG | ACCGTCCTGTTTCTGAATGG | 358 | 60 | ניקולסון et al., 2017 | ||||
| 4 | CCAAAGTTTACTCCTATTACCCAGA | GCAAATCTTTCTCCAATTACCGTCT | 1250 | 50 | Surachetpong et al., 2017 | ||||
| 4 | GCCCAATGGTTCCCATATCT | GCCCAATGGTTCCCATATCT | 524 | 62 | Mugimba et al., 2018 | ||||
| 5 | CCAAATGTTTCTCTTATCTCAGACTC | CTTTTTCTCAGTTTACCACTTTATG | 1087 | 57 | Surachetpong et al., 2017 | ||||
| 5 | CAACTCTTAGCCTCCGGAATAC | CGTTCTGCACTGGGTTACA | 696 | 62 | Mugimba et al., 2018 | ||||
| 6 | CCAAATTTTACCTCTCGCAT | TCAAGCACTTAAAACTGTACC | 1027 | 45 | Surachetpong et al., 2017 | ||||
| 6 | CCCACACGACAGGACATATAG | GAGTTGGCTTAGGGTGATAAGA | 948 | 62 | Mugimba et al., 2018 | ||||
| 7 | CTCTCTTTGCATTGCATACCGT | GACCAATTATCCCTGCTTTCA | 704 | 57 | Surachetpong et al., 2017 | ||||
| 7 | TCCTTTAGGGATTGGCACTAAC | TTCCATCGACTGCTCCTAGA | 486 | 62 | Mugimba et al., 2018 | ||||
| 8 | ACCTCATCTACACTAACATTTCCA | TCATCATTACACAAATGGAGTAGCT | 637 | 50 | Surachetpong et al., 2017 | ||||
| 8 | CTTAAGGGCCATCCTGTCATC | TGGCTCAAATCCCAACACTAA | 476 | 62 | Mugimba et al., 2018 | ||||
| 9 | TTGGTGATGTCACGATGGATA | AGTTCTATCGCCAGCCATGT | 351 | 60 | ניקולסון et al., 2017 | ||||
| 9 | ACAAGTCCGATTACTTTTTCCGC | TCTTTCTCACGTCCTTAAAGTCA | 530 | 50 | Surachetpong et al., 2017 | ||||
| 9 | GATATCCTCCACATGACCCTTC | GTACGTCACTTTGTGCCATTAC | 261 | 62 | Mugimba et al., 2018 | ||||
| 10 | AACCCTACTAACACCAAATATAGCT | CTTTCCCTCTGACACCCTGT | 450 | 50 | Surachetpong et al., 2017 | ||||
| 10 | TCCTCTCTGTCCCTTCTGTT | CAGGATGAGTGTGGCAGATTAT | 276 | 62 | Mugimba et al., 2018 | ||||
טבלה 1- זוגות פריימר שפורסמו עבור ההגברה של cDNA TiLV משתמש הקצה של ה-PCR. ומהצמיגים להגדיר המוצגות באותיות מודגשות שימשו כדי להפיק את התוצאות נציג שמוצג באיור 3A ו- 3B.
6. TiLV כמותיים תגובת שרשרת פולימראזית (qPCR)
- שימוש של פלסמיד המכיל את TiLV המתאים מקטע גנומי 3 cDNA כסטנדרט, כגון pTiLV32, להכין סדרה דילול טורי 10-fold כפולים או triplicated.
- להתכונן מאסטר-שילוב qPCR כל דגימות, תקנים ופקדים, תוך לקיחה בחשבון התגובות צריכה להתבצע ב שכפל או triplicate ניצול µL 0.4 נוקלאז ללא מים, µL 0.3 של פריימר לפנים, µL 0.3 של פריימר הפוכה 5 µL 2 x SYBR Green Dna פולימראז מאסטר-מיקס לכל תגובה.
- להשתמש תחל את ריכוז של 10 מיקרומטר, ואת המידע תחל את pTiLV רגיל כדלקמן:
קדימה פריימר: TiLV-112F (5'-CTGAGCTAAAGAGGCAATATGGATT-3')
הפוך פריימר: TiLV-112R (5'-CGTGCGTACTCGTTCAGTATAAGTTCT-3')
PTiLV:10 סטנדרט pg/µL
הערה: אם המספר הכולל של דגימות ופקדים הוא 10, יבוצעו ב triplicates זה משווה ל qPCR מאסטר-תערובת הכוללת מים נטולי נוקלאז µL 12, 9 פריימר לפנים µL, 9 µL תחל הפוכה, µL 150 של SYBR Green DNA פולימראז מאסטר-מיקס.... שנרכשו באופן מסחרי 2 x אוניברסלי SYBR Green DNA פולימראז מאסטר-תערובות להכיל את כל הרכיבים הנחוצים עבור התגובה qPCR, כלומר, SYBR Green אני צובע, חם-start Taq DNA פולימראז, dNTPs, MgCl2 ו הפניה פסיבי צבעי. להגן על המיקס מאסטר SYBR Green אור.
- להשתמש תחל את ריכוז של 10 מיקרומטר, ואת המידע תחל את pTiLV רגיל כדלקמן:
- לוותר על 6 µL של qPCR המאסטר-לערבב לתוך צינורות רצועת qPCR או צלחת טוב 96 תואם מכונת qPCR בשימוש.
- להוסיף 4 µL של תבנית cDNA, פקדים או באופן סדרתי מדולל בתקנים TiLV לתוך צינורות או בארות של צלחת טוב 96.
- סגור את הצינורות qPCR או לאטום את הצלחת היטב 96 עם מכסה תואם צלחת למכונת qPCR בשימוש
- בעדינות קפיצי הצינורות qPCR לערבב את הפתרון ואת ספין למטה הצינורות qPCR או 96 צלחת היטב באמצעות צנטריפוגה כדי לאסוף את כל הנוזל בתחתית כלי.
- למקם את צינורות או צלחת הצנטרפוגה תרמי בזמן אמת.
- לתכנת את thermocycler qPCR לביצוע של דנטורציה הראשונית ב 95 מעלות צלזיוס למשך 3 דקות, ואחריו 40 מחזורי 95 ° C עבור 10 s ו- 60 ° C ל 30 s פריימר חישול, התארכות, המסתיימים ההיתוך עקומת צעד של 65 מעלות צלזיוס עד 95 ° C עם תוספת קבועה של 0.5 ° C / 5 s.
- בחר SYBR צבע fluorophore, ולאחר מכן בחר נודע כסוג מדגם והכנס שם לתוך תיבת השם הדגימה.
- פותחים את המכסה של המכונה RT-qPCR למקם את הרצועה qPCR הבארות שהוקצו, ולאחר מכן סגור את המכסה.
- לבצע את הבדיקה RT-qPCR עם התנאים שנבחרו. המכונה יתחיל לפעול לאחר המכסה הגיע לטמפרטורה הרצויה. לאסוף את זריחה של כל מדגם לאחר כל שלב ההרחבה כדי לעקוב אחר ההתקדמות של התגובה.
הערה: המכונה qPCR, תוכנות קשורות באופן אוטומטי לחשב את כל הפרמטרים של וזמינותו ולהציג את עקומות הגברה בזמן אמת, בזמן עיקול רגיל ואת עקומת ההיתוך ייווצר בסוף מחזור qPCR. - לבצע ניתוח נתונים ורכישה על-ידי הבטחת הראשון זה כור ההיתוך ' עקומות ' עבור כל דגימה, תקן יש פסגה אחידה אחת בטמפרטורה הצפוי עבור אמפליקון.
- להעריך את עקומות הגברה של הדגימות, תקנים ולהגדיר הסף באזור היו שקצב הגברה cDNAs אותו הדבר כל הדגימות. זה בדרך כלל מבוצע אוטומטית על ידי התוכנה אך צריך להיבדק בקפידה.
- לחשב את מספר העותקים TiLV באמצעות עקומת סטנדרטי.
תוצאות
בעקבות פרוטוקול המתואר בסעיף 1, אמנון אדעך היברידית אדום מציג סימנים קליניים של זיהום TiLV (איור 1 א') היו מורדמים על ידי רחצה ריכוז גבוה של שמן ציפורן, אשר משמש כחומר הרדמה. סימפטומים קליניים דיווחו משתנה אך התסמינים השכיחים שנראה עייפות, העור סחף, דהוי, exophthalmia, מנותקת קשקשים, פצעים פתוחים/הנגע, התנהגות חריגה15,16,33, 35,36, חלק מהם ניתן לראות בבירור ב איור 1A. לקיר הבטן הוסר כדי לאסוף איברים פנימיים כמו כבד, טחול או כליות ראש (איור 1B). ריר דגימות נאספו גם בשלב זה על ידי גירוד בעדינות את העור מפני הקדמי כדי האחורי של הדג באמצעות כיסוי זכוכית או להב כירורגי37.

איור 1 . ניתוח אמנון ואיסוף מדגם. א. אמנון נגועה TiLV היברידית אדום עם העור leisons, אדמומיות סביב הפה, operculum, העור שחיקה הקרנית אטימות. B. Sectioned אמנון היברידית אדום כדי לאפשר לאיסוף רקמות טחול כבד (בנקודת חץ כחול), או כליות ראש איברים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
לאחר מכן, פרוטוקול מפורט בסעיף 2 לחילוץ Guanidium Thiocyanate-פנול-כלורופורם של RNA הכולל עקבו ובוצע כימות RNA כמתואר בסעיף 3 כדי להעריך מדגם טוהר על ידי חישוב של היחס טוהר ו בחינת ספקטרלי פרופילים (איור 2). איור 2A מראה תוצאה נציג מ מוצלח הכולל RNA מיצוי הליך, בעוד דמות 2B מייצג הכנה RNA המסכן. חומצות גרעין יש ספיגת maxima ב-260 חלבונים יש שלהם ב 280 ננומטר. היחס בין המדידות ב-260 nm ו 280 ננומטר מציינים את הטוהר של כל מדגם וקווים יחסי של 1.9 ל- 2.1 מציינים RNA טהור כמו במקרה של המדגם איור2 א. יחסי A260/280 התחתונה נצפתה איור 2B מצביעים על חלבון או פנול זיהום אפשרי שאריות מההליך מיצוי ה-RNA. ספיגת ב- 230 nm יכול להיות התוצאה של זיהום הדגימה, היחס nm A260/230 מחושב גם מסיבה זו. יחס זה צריך להיות בטווח של 2.0-2.2 על ההכנות RNA טהור כפי שהודגמה בעזרת ערך של 2.03 עבור המדגם ב 2A איור, איור 2B יש יחס A260/230 נמוכה של 1.07 ואילו לפרופיל ספקטרלי מציגה את משמרת השוקת ב- 230 nm לכיוון 240 nm אשר מעיד על guanidine שיורית או פנול במדגם. עבור הדוגמה המוצג באיור 2B, מחדש מאיצים את הרנ א להסיר את הזיהום עשוי לשפר את הטוהר של המדגם.
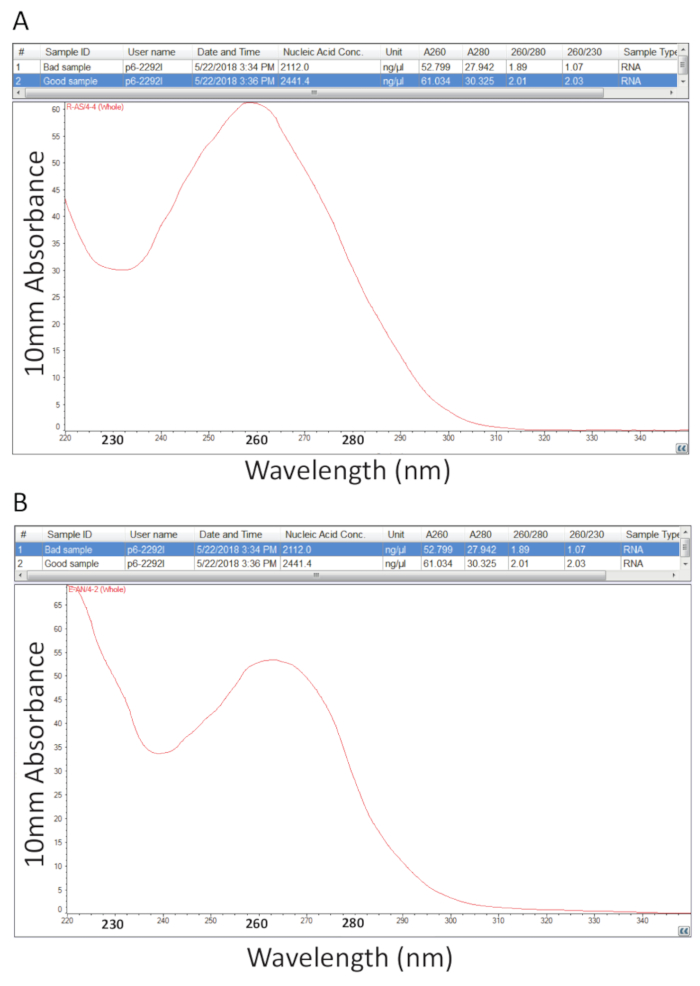
איור 2 . כימות spectrophotometric של RNA הכולל מופק רקמות חולות אמנון. א. יחסי טוהר ופרופילים ספקטרלי של הכנה RNA מוצלחת. B. כמו A, למעט נציג של פרוצדורה החילוץ RNA המסכן. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
כדי לאתר TiLV על ידי ה-RT-PCR, טהור כגון זה מיוצג איור 2A היו הפוכה משועתקים (פרוטוקול 4) לתוך cDNA ודוגמאות שימוש assay תבנית עבור ה-PCR נתונים היסטוריים של סעיף 5, נציג התוצאות מוצגות באיור איור 3 א. תחל שמוצג מודגש ב טבלה 1 שימשו כדי להגביר את קטע bp 491 של מקטע גנומי 3 TiLV14. המוצרים PCR היו מופרדים על ידי אלקטרופורזה בג'ל, מוכתם EtBr להמחשת. איור 3A מציגה את התוצאות של שני שלבים RT-PCR באמצעות 4 דגימות cDNA (S1-S4), נגזר מן הכבד של אמנון ולכוונו מבודד בתאילנד ולאחר בכל מדגם, להקה יחיד נקי של 500 לחץ דם יכול להיות שנצפו, לפיכך, הם דוגמאות 1-4 TiLV חיובי. המוצר PCR הושג מדגם בקרה חיובית, הכוללת cDNA של TiLV קטע 3 משובטים לתוך פלסמיד בגודל32 בעוד אין תבנית הפקד (NTC) לא נכנע מוצרי ה-PCR. הבדיקה ב- 3B איור בוצעה באמצעות תחל את אותו כמו דמות 3A אבל במעבדה שונים, תוך שימוש בגישה RT-PCR צעד אחד, עם 5 דוגמאות RNA נגזר מן הרקמות כליות ראש של אמנון שמקורם במצרים חקלאות ימית 15. נקבע באמצעות הזה assay זיהוי כי דגימות 1, 3 ו-5 הם TiLV חיובי בעוד הם דוגמאות 2 ו- 4 TiLV שלילי מאז אף מוצר ה-PCR נמצאה בגודל הנכון. הפקדים שליליות, כולל שני מינוס רוורס טרנסקריפטאז פקדים, NTCs שני אפשרות לייצר שום מוצרי ה-PCR. Assay RT-PCR צעד אחד בוצעה גם פילוח אמנון ActinB ג'ין. גודל אמפליקון 217 bp נוצר כל מדגם (S1-S5) כמו הצפוי38. Assay הזה שימש פקד לאמינותה של דגימות ה-RNA, כמו גם המאפשרות בדיקה כמותית למחצה של הדגימות החיוביות TiLV. בהתחשב בכך שהמוצר אמנון ActB שנוצר שווה יחסית, אז ההבדלים בכמות המוצר PCR ספציפי TiLV שנוצר אפשר לפרש כמשקף כמות TiLV במדגם רקמות נתון אמיתי.
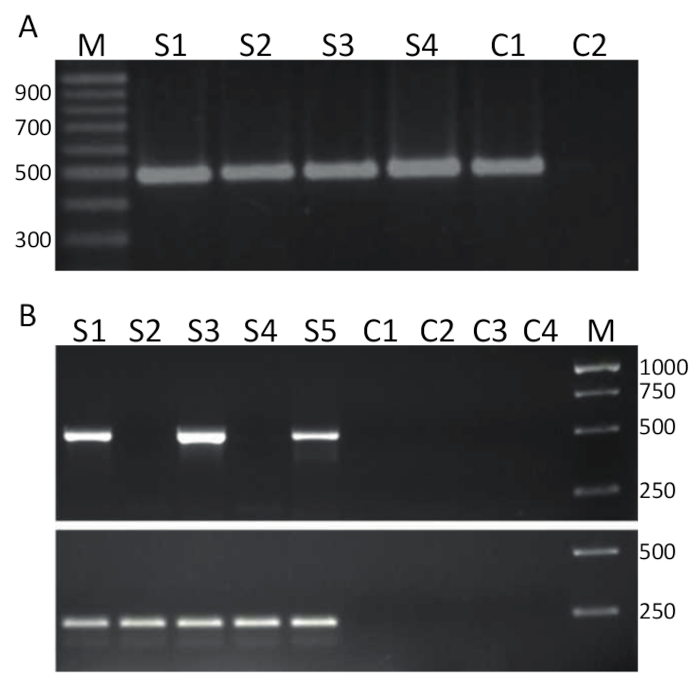
איור 3 . TiLV RT-PCR. א. דגימות cDNA המופק רקמות הכבד של אמנון נגועים, שנאספו מתאילנד היו מסך על זיהום TiLV באמצעות תחל ספציפי עבור מקטע 3 (שמוצג מודגש ב טבלה 1) של TiLV שימוש assay RT-PCR של 2-צעד. M = סמן שמוצג בסיסי-זוגות; S1-S4 = דגימות 1-4; C1 = לשלוט באמצעות pTiLV כתבנית PCR; ו- C2 = שליטה תבנית (NTC). B. צעד אחד RT-PCR באמצעות את תחל אותו כמו A ואסף דגימות רקמות כליה ראש של אמנון חולה בין מצרים15. M = סמן שמוצג בסיסי-זוגות; S1-S5 = דגימות 1-5. פקדים C1-C2 נמצאים מינוס רוורס טרנסקריפטאז פקדים, C3-C4 NTCs. בתחתית החלונית RT-PCR צעד אחד משתמש primers נגד אמנון ActinB38 (ראה טקסט לפרטים) לייצר מוצר ה-PCR של בסיס 217-זוגות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
בניגוד נקודת הקצה מותני מיוצג באיור 3, מבחני qPCR המוסברים בפרוטוקול 6, למדוד את כמות ה-PCR המוצר לאחר כל מחזור ה-PCR. ההגברה של היעד DNA מזוהה באמצעות מולקולות פלורסנט המקיימים אינטראקציה עם ה-DNA שנוצר מתוך כל סיבוב של תגובה. . הנה, SYBR Green אני צובע היה מנוצל, אשר intercalates עם שני גדילי ה-DNA. האות פלורסנט מלווה במהלך התגובה ואת האינטנסיביות שלו מתייחס כמות המוצר נוצרו 39,40,41,42,43. TiLV qPCR מבחני בוצעו כמתואר פרוטוקול 6 במעבדות שונות בעזרת ריאגנטים SYBR Green שונים, מכונות qPCR ודוגמאות ממדינות שונות. הגברה העיקולים וכתוצאה מכך מוצגים באיור 4A , 4B. זה יכול להיות ציין כי עבור כל assay, מהלך הניסוי יש ארבעה שלבים: המהלך הקרקעי ליניארי, מעריכי בשלב מוקדם, בסוף שלב מעריכית, שלב ה-plateau. המהלך הקרקעי ליניארי מתרחשת במהלך המחזורים הראשונים שבו שכפול ה-DNA אין עדיין אפשרות לזהות עקב כמויות ה-DNA לייצר יחס אות/רקע לא מספיקות. קרינה פלואורסצנטית הבסיסית מחושבת במהלך שלב זה. לאחר מכן, מתחיל היעד DNA כפול ריכוז בכל מחזור שייצור את האות הופכות לזיהוי מעל רקע ולהגדיל באופן אקספוננציאלי. יעילות הגברה (E) וזמינותו qPCR היטב אופטימיזציה היא גבוהה מאוד (ליד 100%) בתחילת התגובה ואת נשאר יציב במהלך שלב זה מעריכי מוקדם של ההגברה וזה נמצא בשלב זה כימות מתבצעת, כאשר יעילות התגובה היא עדיין יציב. במחזורים מאוחר יותר מתחיל האות מישור, האינטנסיביות של זריחה הוא כבר לא בקורלציה את המספר ההתחלתי של העתק תבנית כי הרכיבים התגובה הם מותשים44. רוויה עלול להתרחש גם בעקבות התחרות תגובות מחזק מחדש, היחס הריכוז המשתנה של הרכיבים, או את כמות יחידות אנזים כדי במולקולות דנ א המצע. יתכן, פרמטרים כגון בחשבון את ההבדלים בין העקומות הגברה עבור מבחני שמוצג באיור 4A , 4B. הפקדים הכלולים לא ליצור עקומות אלה הגברה אופיינית.
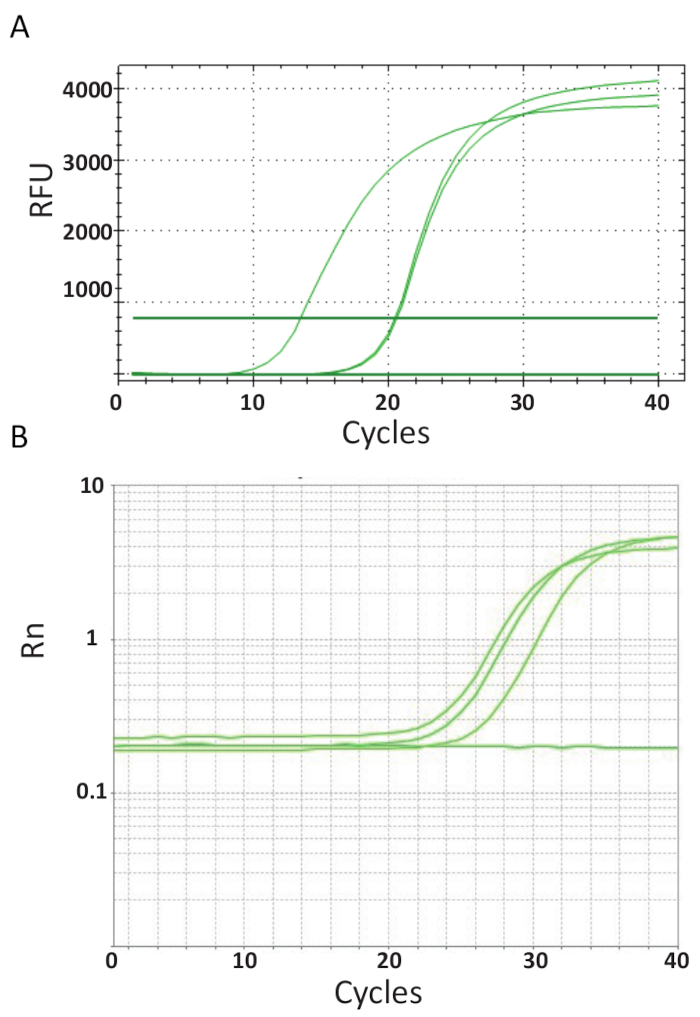
איור 4 . הגברה חלקות להראות ההצטברות של מוצר על-פני משך וזמינותו PCR בזמן אמת. א. עקומות הגברה של דגימות TiLV-חיוביות נגזר תאילנד, NTCs ושליטה פלסמיד חיובי באמצעות SYBR-ירוקה אני דו-שלבי qPCR assay. התרשים נוצר על ידי התוויית היחסי קרינה פלואורסצנטית (RFU) לעומת מחזור מס. B. עקומות הגברה של דגימות חיוביות TiLV נגזר מצרים, כמו באיור 3B ו- NTC. העקומה הגברה הוא זריחה של האות כתב מנורמל ל זריחה של לצבוע רוקס פסיבי הכלולים המספר assay (Rn) לעומת מחזור. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
בסוף thermocycling qPCR על המכונות שונה במעבדה כל הנתונים רכשה, מנותח. איור 5A 5B מראים עקומות ההיתוך נציג מ מבחני שבוצעה במעבדה כל. בכל מחשב qPCR היה מתוכנת לביצוע ניתוח עקומת נמס בסוף. זה הושג על-ידי הגדלת הטמפרטורה בהדרגה ובפיקוח על ידי קרינה פלואורסצנטית כפונקציה של הטמפרטורה. כאשר הטמפרטורה גבוהה מספיק כדי denature dsDNA, ירידה גדולה פלורסצנטיות נרשם כי המולקולה fluorophore הוא שוחרר. התוכנה של כל מכשיר qPCR מחושב הטמפרטורה מחזק (Tm) מהנתונים עקומת נמס על ידי התוויית הטמפרטורה vs נגזרות הראשונה שלילית (איור 5). ניתן לראות כי איור 5A , 5B והמוצרים שנוצר בערכות דגימה שונים יש אחיד מעבר נמס בטמפרטורה הצפוי של 80 ° C עבור וזמינותו. אין פסגות אחרות בטמפרטורות נמוכות נצפו. בשל גודלם הקטן, ה-Tm של פריימר-הדימרים הוא נמוך בדרך כלל מזה של המטרה רצף ה-DNA. לכן, ההבדל בין ה Tmשל מקל לזהות פוטנציאל פריימר-הדימרים או מוצרים אחרים שאינם ספציפיים הגברה. הפקדים אינם מפיקים להמיס עקומות כמו דגימות חיוביות TiLV ותקנים, ניתן לראות קו אופקי כמעט בתחתית של התרשימים איור 5A , 5B.
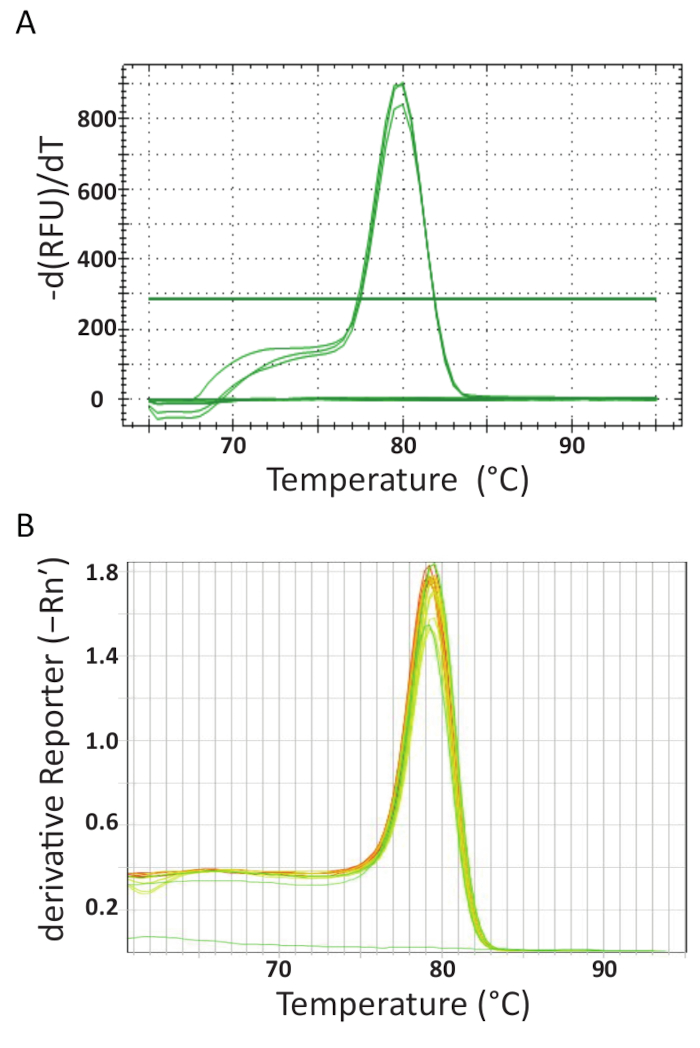
איור 5 . ממיסים עקומת ניתוח כדי להבטיח ירידה לפרטים assay ומוצרים שונים PCR יכול להיות מובחן על-ידי תכונות ההיתוך שלהם. א להמיס עקומת ניתוח דוגמאות TiLV-חיוביות שמקורם בתאילנד, שליטה שלילי ושליטה פלסמיד חיובי. B. להמיס עקומת ניתוח של דגימות חיוביות נגזר מצרים, pTiLV בסטנדרטים NTC TiLV. התרשימים ב A ו- B שניהם הצג את השינוי בזריחה מחולק בשינוי הטמפרטורה להתוות נגד טמפרטורה לייצר תמונה ברורה של הדינמיקות נמס. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
רוב מכשירי qPCR באים עם התוכנה, הקלת הערכה נוספת qPCR ולרוץ לכמת את הדוגמאות על ידי יצירת עיקול רגיל ידי באופן אוטומטי את הסף מחזור (סיטי) נגד הלוגריתם של התבנית של הסטנדרטים pTiLV העתק מספר כפי שמוצג באיור 6A , 6B למעבדות עצמאית שני. בקיצור, ה-Ct היא יחידת המשמש להערכת התוצאות qPCR. ערךt C מציין את מספר מחזורי נדרש להגיע לרמה של האות פלורסצנטיות קביעת הסף. גדול יותר כמות החל תבנית, כמה שפחות מחזורים זה לוקח כדי להשיג רמה לזיהוי קרינה פלואורסצנטית. אכן, דגימות עם עומס גבוה של TiLV יהיו ערכיםt C נמוכים יותר מאשר הדגימות עם עומס נמוך של TiLV כגון דגים עם זיהום תת קליניים. כדי לקבוע ערכיt C, רמות קרינה פלואורסצנטית רקע קודם מופחתות מן הנתונים הגולמיים. בשלב הבא, התוכנה המשויכים המכשיר qPCR יבחר באופן אוטומטי סף פלורסצנטיות על-ידי חיפוש העקומות נתונים עבור כל דגימה, שילובt C מייצג שבו המדגם חצה את הסף. זה נעשה בנפרד עבור כל assay, כל הסף צריך ניתן להעריך בזהירות, המבטיח כי הסף הוגדר בחלק לוגריתמי של עקומות הגברה, במקום שבו כל עקומות מקבילים. לפיכך, C ספציפיt רכשה הוא ערך יחסי וזה יחסית ההתחלה תבנית עותק מספר45, אבל זה גם ספציפיות עבור המכונה qPCR, ריאגנטים בשימוש, את היעילות של הגברה PCR ואת הרגישות של זיהוי. פרמטרים אלה תורמים ההבדלים שנמדדו באמצעות וזמינותו אותו באיור 6.
מן העקומות רגיל באיור 6, ניתוח רגרסיה, כולל חישוב של מדרונות עיקול רגיל (ז), כפי שאמרתי, הגברה יעילות (100 x (101/ז -1))46 , ליניאריות התגובה בוצעו. עיקול רגיל ניתוחים שימשו גם לאשר רגישות (גבול של זיהוי), הדיר, הפארמצבטית של וזמינותו. באופן תיאורטי, כמות ה-DNA מוכפל PCR בכל מחזור, כלומר היעילות (E) הוא שווה ל- 100%. עם זאת, בפועל יעילות כל כך אידיאלי נגיש רק לעתים רחוקות בשל תנאי ה-PCR תת אופטימלית כגון, ה-DNA פולימראז עיכוב, מזהמים, מדי cDNA ו pipetting שגיאות47. בדרך כלל, הגברה E נע בין 90-110% עבור מבחני טוב, ב- 6A איור יעילות של 94.5% מחושב באמצעות 8 דגימות pTiLV באופן סדרתי מדולל, תוך כדי, היעילות assay וזמינותו באיור איור 6B באמצעות pTiLV באופן סדרתי מדולל 7 דוגמאות היה 101.2%. יעילות של למעלה מ- 100% בדרך כלל בשל נוכחותם של מעכבי PCR ב וזמינותו. ניתוח של רגרסיה ליניארית של העלילה רגיל מאפשר גם לחישוב מספר העותקים TiLV בתוך כל מדגם41,42,45, כמו יכול להיות שנצפו על הדגימות TiLV שלושה שמוצג 6B אדום איור אשר עולה בקנה אחד עם התוצאות עבור דגימות S1, S3 S5 שמוצג באיור 3B.
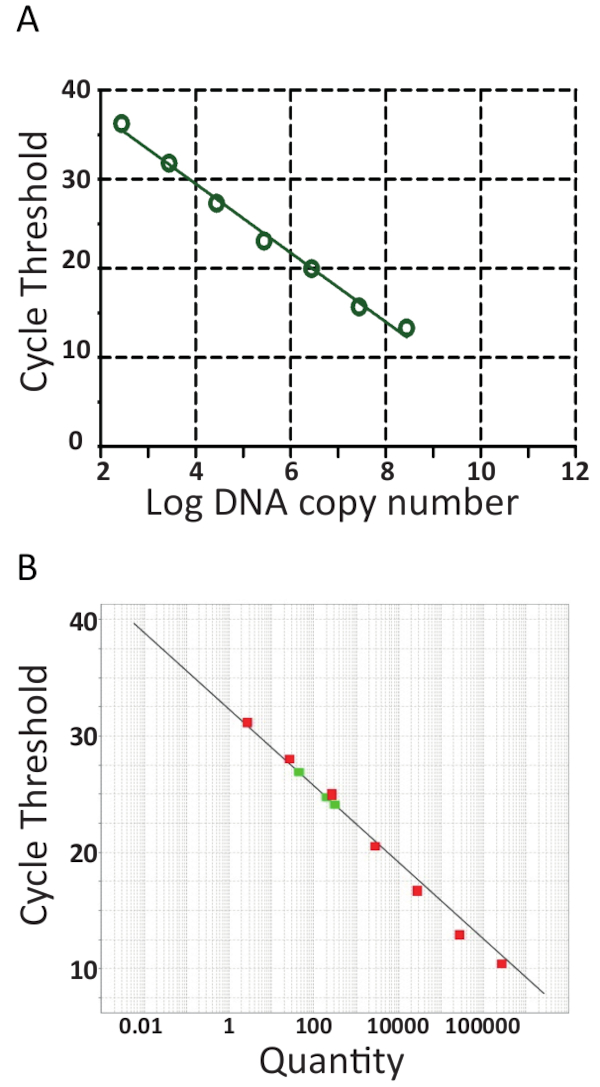
איור 6 . RT-qPCR רגיל עקומות. PCR בזמן אמת של דילולים טורי 10-fold של pTiLV, התקן בשימוש בשתי מעבדות. א 8 pTiLV באופן סדרתי מדולל היו נבדק, כל הריכוז הידוע ודוגמאות בקורלציה מספר העותקים TiLV / תגובה. העקומה רגיל נוצר על ידי התוויית יומן עותק מספר לעומת מחזור הסף (סיטי). השיפוע =-3.462, R2 = 0.9992 היעילות היא 94.47%. B. כמו A, למעט 7 pTiLV באופן סדרתי מדולל דגימות (ירוק) נבדקו, התרשים מציג את מחזור הסף על ציר ה-y ואת המספר עותק של TiLV (כמות) בציר ה-x. החיתוך-y = 32.327, שיפוע =-3.292, R2 = 0.98 היעילות היא 101.2%. עבור שני עיקולים סטנדרטים בתוך A ו- B, מדרון, חיתוך-y וערכי מקדם המתאם (R2) מנוצלים כדי להבין את הביצועים של וזמינותו. חשוב לציין, R2 ערך צריך להיות קרוב ל- 1 שכן הוא מדד של ליניאריות של העקומה סטנדרטי. המדרון משמש כדי למדוד יעילות ה-PCR שבה 100% יעילות מקביל מדרון של-3.32, לראות את הטקסט העיקרי עבור המשוואה ופרטים נוספים. תגובה qPCR טוב בדרך כלל יש יעילות בין 90-110% מתאם אל מדרון של בין-3.58 ל--3.10. העקומה סטנדרטי משמש עבור כימות מוחלטת של דגימות חיוביות TiLV לא ידוע וקובע את המספר המדויק של TiLV עותקים / תגובה, כמו המקרה על הדגימות החיוביות שלושה TiLV בצבע אדום ב'.
Discussion
TiLV דווחה לראשונה בשנת 2014 ישראל14 , מאז, היא זוהתה במדינות רבות כולל מצרים, קולומביה, הודו, מלזיה, אוגנדה, טנזניה, תאילנד15,16,18, 35 , 48. מודעות עולמית, במיוחד, באמנונים בהפקת מדינות יש להציב יותר תשומת לב על הוירוס ולא הגבלות שונות, אמצעי בקרה מרשויות הממשלה יושמו בניסיון למנוע את התפשטות TiLV. כאן, פרוטוקול מפורט לגילוי TiLV ברקמות אמנון, כיסוי איסוף הדגימה, בידוד RNA, מבחני cDNA סינתזה, PCR, qPCR יש כבר הסביר. ישנם היבטים שונים לשיטות אלו המחייב דיון ספציפי. TiLV זוהתה ב דגים המתפרסות על-פני מגוון גדלים9,12,14,15,49 עם מינים של אמנון עד כה, כולל אמנון היברידית מעובדים (O. niloticus x aureus O.)11,14, הנילוס אמנון (O. niloticus)9,10,14,15,16, 33 , 35 , 36 , 49 , אמנון 50 ו אדום (Oreochromis sp.)16,33,48,51, כמו גם בר הנילוס אמנון9,12, שחור אמנון51, zilli ט14,15, ס galilaeus, O. aureus ו ט סימוניס הדממה:14 ו לאחרונה TiLV זוהה פראי קרפיון (Barbonymus schwanenfeldii)52. דגימות רקמות איברים פנימיים (גיל, טחול, כבד, לב, כליות ראש) או ריר37 ניתן שנאסף אמנון בריא, כמו גם אדעך ללא הבדלי גיל, גודל או מינים ולעבד לבידוד RNA. פרוטוקול הכולל של מיצוי RNA המתוארים כאן משתמש פתרון monophasic של פנול, guanidinium thiocyanate, אשר הוא סוכן denaturing chaotropic. הרקמות הן ישירות הומוגני בפתרון זה ואחריו התוספת של כלורופורם וב צנטריפוגה כדי להשיג הפרדה השלב שבו נוצרת של RNA ברור המכיל העליון פאזה מימית, לאטמוספרה וגם שלב אורגני התחתון. ה-RNA היא מבודדת מהשלב מימית על ידי משקעים אלכוהול איזופרופיל, ולאחריה שטיפה של RNA התאושש להיפטר של מזהמים. בידוד של RNA על ידי מתודולוגיה זו היה חלוץ ידי פיוטר Chomczynski ו- Nicoletta סאקי, היה המכונה guanidinium-thiocyanate-פנול-כלורופורם החילוץ-53,-54. יכול להיות שנרכשו או שנעשו במעבדה מסחרית סוג זה של ריאגנט המשמש עבור החילוץ-RNA (ראה את הטבלה של חומרים למידע נוסף). פרוטוקול זה לוקח מעט יותר מאשר מבוססי עמודה שיטות כגון הטיהור מבוססות-סיליקה, אבל באופן כללי, היא עלות יותר אפקטיבי, התשואות RNA יותר.
ב פרוטוקול זה, לכימות RNA באמצעות ערכים A260 היה המתוארים לפיה ספקטרופוטומטר ערכים יכולה להצביע על איכות ה-RNA (A260/A280 = 1.9-2.1). ואילו בשיטה זו ייתן סימן טוב טוהר לדוגמה, לא יכול בהחלט להודיע על איכות RNA שחולצו. כדי לקבוע כראוי אם הרנ א הוא שלם או חלקי מפורק, דגימות יכול להיות הפרדה על ידי agarose בג'ל שבו מריחת של EtBr צבעונית 18S, 28S rRNA להקות לציין RNA השפלה. אימות נוסף של איכות RNA עשוי לכלול שימוש בכלי מעבדה-על-שבב. יתר על כן, זה גם חשוב לעכל את הרנ א מטוהרים עם DNase אני כדי להסיר מזהמות מארח הדנ א, אשר בהתאם היישומים במורד הזרם עלול להוביל תוצאות שגויות. אם המחשב המארח gDNA עדיין מזהם את הדגימה RNA במידה רבה, טיפול DNaseI נוספים ניתן גם לבצע בסופו של ההליך החילוץ RNA (ראה טבלה של חומרים).
סינתזת דנ א משלים ולהשפיע רבות על תוצאות qPCR הכוללת, היא היבט של השיטה שבה באפשרותך להציג את הווריאציה. פרוטוקול ה-cDNA דגלו כאן כוללת של הגדרת רכיב יחיד באמצעות oligo (dT), ובכך רק transcribes mRNAs המכיל תיקים עשויים זנבות. זה מאפשר בקרת משתמש בדיוק אילו רכיבים להשתמש התגובה שעתוק במהופך, במצב זה של cDNA סינתזה הוכיח מוצלח עבור זיהוי TiLV32. חלופה זו הגדרת הוא מאסטר-מיקס מסחרית קנה המכיל כל הרכיבים הדרושים עבור התגובה שעתוק במהופך, והוא מאוד מהירה ופשוטה ללא פרוטוקול רב שלבי, pipetting, טמפרטורה רב מהרגיל. זה יתרון כי זה ממזער טיפול ומקדם אחידות בכל הדוגמאות. מאסטר-תערובות כזו לעיתים קרובות כוללים הן oligo(dT) והן תחל אקראי הפיכתה תבניות RNA החלים שונות, הפקת cDNA נציג עותקים של רצפי מ לכל אורך RNAs באוכלוסיה (ויראלי, אמנון לארח mRNA), באופן תיאורטי, כל מין RNA הרצוי ואז נמדד על ידי קונבנציונאלי PCR או qPCR מתוך כזה מדגם. רבגוניות זו הוא היתרון העיקרי של גישה RT-PCR 2-צעד; הוא מספק בריכה לטווח ארוך יכול לשמש עבור ניסויים שונים רבים. בתוצאות, מיוצגת גישה RT-PCR חד-שלבית שבה תחל רצף ספציפי (טבלה 1) שימשו ו RT ו- PCR בוצעו בשפופרת אחת (ראה רשימת חומרים). באופן כללי, רצף ספציפי תחל לאפשר יעילות גבוהה יותר RT של יעד ספציפי RNA מאשר שימוש אקראי לקרקע, אבל המטרה ספציפי RNA היא היחידה שלא ניתן לכמת במדגם כזה cDNA אשר עשוי להיות המטרה היחידה של מעבדות מסוימים (ראה טבלה של חומרים עבור הצעות למוצרים סינתזה cDNA).
בעוד קונבנציונאלי RT-PCR מופיעה כדי להיות נפוץ עד כה ב TiLV אבחון9,13,14,15,16,17,18, 33 , 35 , 48 , 55. RT-qPCR הוכח להיות כלי יותר חזק זיהוי, כימות של כמויות קטנות של TiLV רקמות דגים או32,ריר37. באופן כללי, qPCR נעשה שימוש נרחב במעבדות לאבחון קליני וירולוגיה בשל רגישות גבוהה שלה, ירידה לפרטים, טוב הפארמצבטית, טווח דינמי רחב מהירות21. בעוד qPCR עשוי להיות בתחילה יקר יותר ליישום מאשר קונבנציונאלי RT-PCR, זה מציע מספר יתרונות חשובים על-קונבנציונאלי PCR; יש זמן יותר מהיר סיבוב מהדגימה לתוצאות, זה לא דורש שום צעדים פוסט-PCR. בנקודה האחרונה כלומר הסיכון הוא מינימלי עבור המעבדה זיהום זה ניתן להתאים בקלות רבה יותר תפוקה גבוהה במצבים כמו למשל במקרה של התפרצויות. יתר על כן, זה מטבעו רגיש יותר קונבנציונאלי RT PCR, שהינו בעל חשיבות לזהות נמוך המון נגיפי דלקות תת קליניים21. זה ידרוש גישה PCR מקוננים הדורשים שעתוק במהופך, שתי תגובות PCR נוסף ולאחר מכן ניתוח לפי agarose ג'ל אלקטרופורזה. השלבים רבים לקחת הרבה זמן ולהגדיל את הסיכויים של שגיאות או זיהום. בכל זאת, עקב רגישות גבוהה שלה, RT-qPCR דורשת הבנה מעמיקה של כימות טכניקות ליצירת תוצאות מדויקות56,57ועיצוב מוקפד ניסיוני.
Fluorophore מחייב ה-DNA, SYBR Green הוכח ב פרוטוקול זה. צבע הכריכה dsDNA DNA לא ספציפי, ובכך ייחודה של וזמינותו טמון לחלוטין את קבוצת צבעי בסיס, אשר עשויה ליצור תוצאות חיוביות שגויות58. לכן, בזמן dsDNA נמס ניתוח עקומת שבוצעה בסופו של כל ה-PCR היא חלק חשוב במיוחד התגובה PCR כי היא מאשרת את אמפליקון PCR אחד בלבד של ה-T הנכוןמ' מופק (זה צריך גם להיות מושגת על ידי ג'ל אלקטרופורזה כאשר מבחני חדש במקדמי). Tm של קטע DNA היא תלויה מגוון רחב של תכונות כגון שלה אורך, קומפוזיציה GC, רצף, משלימים את החסר סטרנד, ריכוז גם על מאגר הרכיבים של משפרי PCR. הניתוחים עקומת ההיתוך בתוצאות נציג המוצגות בין 2 מעבדות שתק ולא גילה את קיומו של פריימר-הדימרים או מוצרים אחרים-PCR לא רצויים, אבל אם זה נצפית עם דגימות אחרות ו/או set-ups ניסיוני, אז הבדיקה צריך להיות אופטימיזציה מחדש. טכנולוגיות מתקדמות יותר qPCR אינם דורשים ההיתוך עקומת צעד כזה, ואכן, מאז זה שיטות העיתון נכתב, TaqMan המבוסס על TiLV RT-qPCR פותחה תחל שני וחומר מכשיר בדיקה מקשה מאוד ספציפי TiLV34.
אין ספק, תחל תוכנן עבור מבחני RT-qPCR היסוד להצלחת assay, תחל כאן עוצבו על פי בציבור TiLV גנומית הנתונים הזמן32. עם זאת, וירוסי RNA ידועים להפגין מוטציה גבוהה המחירים, זנים אפשריים ימלט הבדיקות האבחוניות הנוכחי, כפי נצפתה עבור ISAV59. זה תמיד יהיה קשה עבור סוגים כאלה וירוס להפיק הפשר פן אוניברסלי-TiLV-qPCR assay, כגון מבחני בלבד ללא הרף ישתפר אם TiLV גנומית נתונים נוספים מיקומים מרחיקות לכת ותקופות זמן הופכים לזמינים.
בסופו של דבר, זה חיוני כדי להפעיל כפולים או במידת האפשר, triplicate תגובות ב- "אינטרה" שני, אינטר qPCR מבחני. אם ערכיt C הן גבוהות מאוד, ולאחר מכן שימוש ושכפול זה חשוב במיוחד כדי לוודא כי התגובה PCR הוא אמין לשחזור. באופן כללי, אם לשחזר נתונים מ תגובות משתנה מחזורים יותר מ 0.5, התגובות לחזור אם הערכיםt C משתנים באופן עקבי > 0.5 מחזורים ב ושכפול של, צריך להיות מותאם מחדש וזמינותו. השימוש של רובוט pipetting qPCR משולב עוזר מאוד בעניין זה, אך זהו כלי יוקרה. כמו עם כל הניסוי, הפקדים הכללה ועובדיהם הם בעל חשיבות עליונה להתפתחות של מבחני מולקולריים חזקים, בעיקר במעבדות לאבחון שבו מבחני כזה צריך להיות מוכר. פקדים צריך לכלול חיובי (מדגם TiLV חיובית, פלסמיד TiLV רגיל), פקדים שלילי (NTC ו- RT) דוגמאות, כמו גם זיהוי גנים משק בית אמנון אנדוגני. פקדים אלה אי אפשר לזלזל, צריכים להיכלל בכל assay להבין כראוי את האיכות של כל שלב של וזמינותו, לפרש כראוי את התוצאות.
Disclosures
המחברים אין לחשוף.
Acknowledgements
אנחנו אסירי תודה מכון של בקטריולוגיה וטרינרית, Vetsuisse הפקולטה, אוניברסיטת ברן לתמיכה שלהם עבודה זו מומן על ידי הוועדה לשוויון קידום אקדמי של חוקרים את הקריירה מוקדם ומגדר בפקולטה Vetsuisse, אוניברסיטת ברן באמצעות מימון דגם 120% מוענק PN. WS ויחסי ציבור נתמכים על ידי המרכז ללימודים מתקדמים לחקלאות, מזון, המכון ללימודים מתקדמים, אוניברסיטת Kasetsart, בנגקוק, תאילנד תחת להשכלה גבוהה קידום המחקר הלאומי מחקר באוניברסיטה פרויקט תאילנד, Office של תאילנד עמלה, משרד החינוך, ההשכלה הגבוהה. ברצוננו להודות ד ר Kwanrawee Sirikanchana שלה הקריינות Piyawatchara Sikarin עבור עריכת הוידאו.
Materials
| Name | Company | Catalog Number | Comments |
| Tissue collection | Step 1 | ||
| Tricaine methanesulfonate | Sigma-Aldrich | E10521 | An alternative to clove oil. Step 1.1 |
| RNAlater stabilization solution | Thermo Fisher Scientific | AM7020 | For storing tissues if they cannot be processed immediately Step 1.3 |
| RNA extraction | Step 2 | ||
| TRIreagent | Sigma-Aldrich | Step 2.1 | |
| TRIzol | Thermo Fisher Scientific (Invitrogen) | 15596026 | Step 2.1 |
| GENEzol | Geneaid | GZR100 | Step 2.1 |
| Trisure | Bioline | BIO-38032 | Step 2.1 |
| Homemade solution | - | - | 94.53 g/L (800 mM) guanidine thiocyanate 30.45 g/L (400 mM) ammonium thiocyanate 8.20 g/L (100 mM) sodium acetate 380 mL/L (38 % v/v) phenol 50 mL/L (5 % v/v) glycerol 1.0 g/L (0.1 % w/v) 8-quinolinol, pH 5.0 Store up to 2 years at 4oC Step 2.1 |
| MagNA Lyser Green Beads | Roche | 3358941001 | An alternative tissue homogenization method used in conjunction with tissue lysing machines detailed below Step 2.2 |
| Lysing Matrix D, 2 mL Tube | MP BIOMEDICALS | 116913050 | |
| Chloroform | Sigma-Aldrich | C2432 | Step 2.3 |
| Chloroform | RCI Labscan | AR1027E-G2.5L | Step 2.3 |
| 1-Bromo-3-chloropropane | Sigma-Aldrich | B9673 | A less toxic alternative to chloroform Step 2.3 |
| Isopropanol (GC) ≥ 99.8 % | Sigma-Aldrich | 59300 | Step 2.6 |
| Isopropanol (ACS, ISO Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.09634.2500 | Step 2.6 |
| Glycogen, molecular biology grade (e.g., Sigma, cat. no. G1767) | Thermo Fisher Scientific (Thermo Scientific) | R0551 | Useful step if tissue starting material is small to maximise RNA precipitation optional |
| Ethanol (purity (GC) ≥ 99.9 % | Sigma-Aldrich (EMD Millipore) | 1.00983 | Step 2.9 |
| Ethanol (ACS, ISO Reag. Ph Eur) | Merck (EMSURE) | 1.00983.2500 | Step 2.9 |
| Nuclease-free water | Promega | P1193 | Step 2.13 |
| Nuclease-free water | Multicell | 809-115-CL | Step 2.13 |
| Ambion TURBO DNA-free kit | Thermo Fisher Scientific (Invitrogen) | AM1907 | Can be performed at the end of the RNA extraction protocol optional |
| cDNA synthesis | Step 4 | ||
| Viva cDNA Synthesis Kit | Vivantis | cDSK01 | Step 4.1 & 4.3 |
| ReverTra Ace qPCR RT MasterMix with gDNA remover | Toyobo | A1172K | An alternative option see discussion |
| ReverTra Ace qPCR RT Kit | Toyobo | FSQ-101 | An alternative option see discussion |
| AffinityScript Multiple Temperature Reverse Transcriptase | Agilent Technologies | 600107 | An alternative option |
| PCR | Step 5 | ||
| DNA polymerase systems: | Step 5.2 | ||
| - Platinum II Hot-Start Green PCR Master Mix (2X) | Thermo Fisher Scientific (Invitrogen) | 14001012a | Step 5.2 |
| - GoTaq Mastermix | Promega | M7122 | Step 5.2 |
| Separate PCR mixture components: | Step 5.2 | ||
| 10mM dNTP Mix | Vivantis | NP2409 | Step 5.2 |
| 25mM MgCl2 | Thermo Fisher Scientific | R0971 | Step 5.2 |
| 10X Taq Buffer with KCl | Thermo Fisher Scientific | 00348114 | Step 5.2 |
| Taq DNA polymerase | Vivantis | PL1202 | Step 5.2 |
| - Verso 1-step RT-PCR ReddyMix with ThermoPrime Taq | Thermo Fisher Scientific | AB1454 | One step RT-PCR exemplified in Figure 3B |
| Gel electrophoresis: | For visulation of PCR products from steps 5.1-5.4 | ||
| Ethidium Bromide solution (10 mg/mL) | Thermo Fisher Scientific | 17898 | Step 5.5 |
| Tris/Acetic/EDTA (TAE) buffer: | Step 5.5 | ||
| - Tris | Vivantis | PR0612-1KG | Step 5.5 |
| - Acetic acid (glacial) (ACS, ISO, Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.00063.2500 | Step 5.5 |
| - Ethylenediaminetetraacetic acid (EDTA) | BIO-RAD | 161-0729 | Step 5.5 |
| Agarose | Vivantis | PC0701-100G | Step 5.5 |
| DNA ladders and markers | Vivantis | NL1405 | Step 5.5 |
| DNA gel loading dye (6X) | Thermo Fisher Scientific | R0611 | Step 5.5 |
| qPCR | Step 6 | ||
| PowerUP SYBR Green Master Mix | Thermo Fisher Scientific (Applied Biosystems) | A25779 | Exemplified in Figures 4-6B Step 6.2 |
| iTaq Universal SYBR Green Supermix | BIO-RAD | 1725120 | Exemplified in the video and in Figures 4-6A Step 6.2 |
| Equipment | |||
| Dounce tissue grinder pestle | Sigma-Aldrich | P1110 | Protocol 2 |
| MagNA Lyser Instrument | Roche | 3358976001 | An alternative tissue homogenizing option for protocol 2 which are used in conjunction with the lysing beads detailed above Step 2.2 |
| FastPrep-24 5G Homogenizer | MP BIOMEDICALS | 116005500 | |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5427R | Protocol 2 Step 2.4, 2.7 & 2.10 |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5418R | |
| Heat box | Labnet | AccuBlock Digital Dry Bath | Protocol 2 Step 2.13 |
| Microvolume spectrophotometer | Thermo Fisher Scientific (Applied Biosystems) | Nanodrop 2000 | Protocol 3 Step 3.1 - 3.4 |
| PCR machine | BIO-RAD | T100 Thermal Cycler | Protocol 5 Step 5.4 |
| Power supply | BIO-RAD | PowerPac HC | Protocol 5 Step 5.5 |
| Horizontal gel electrophoresis | BIO-RAD | Mini ReadySub-Cell GT Cell #1704487edu | Protocol 5 Step 5.5 |
| Mini microcentrifuge | Corning | LSE 6766 | Useful to quickly spin down PCR reaction tubes in protocols 4, 5 & 6 Step 6.5.1 |
| Microcentrifuge | LioFuge | LM-60 | Step 6.5.1 |
| qPCR machine and software | Thermo Fisher Scientific | 7500 Fast Real-Time PCR System with 7500 Software v2.0 | Protocol 6 Step 6.6-6.8 |
| qPCR machine and software | BIO-RAD | CFX96 Touch Real-Time PCR Detection System with CFX Manager software | |
| General Materials | |||
| Mayo scissors | Step 1.1-1.2 | ||
| Forceps | Step 1.1-1.2 | ||
| Pipette | Rainin | Pipette-Lite XLS | |
| Aerosol-barrier pipette tips | Sigma-Aldrich | Z333328, Z333336, Z333344 | |
| Nuclease-free 1.5-ml microcentrifuge tubes | Eppendorf |
References
- FAO. . The State of World Fisheries and Aquaculture, 2014. Opportunities and Challenges. , (2014).
- FAO. . The State of World Fisheries and Aquaculture, 2016. Contributing to Food Security and Nutrition for all. , (2016).
- WorldBank. . FISH TO 2030: Prospects for Fisheries and Aquaculture. Agriculture and Environmental Services Discussion Paper 03. , (2013).
- Wing-Keong, N., Nicholas, R. A review of the nutrition and feeding management of farmed tilapia throughout the culture cycle. Reviews in Aquaculture. 5 (4), 220-254 (2013).
- Cleasby, N., et al. The socio-economic context for improving food security through land based aquaculture in Solomon Islands: A peri-urban case study. Marine Policy. 45, 89-97 (2014).
- Ponzoni Raul, W., et al. Genetic improvement of Nile tilapia (Oreochromis niloticus) with special reference to the work conducted by the WorldFish Center with the GIFT strain. Reviews in Aquaculture. 3 (1), 27-41 (2011).
- Hounmanou, Y. M. G., et al. Tilapia lake virus threatens tilapiines farming and food security: Socio-economic challenges and preventive measures in Sub-Saharan Africa. Aquaculture (Amsterdam, Netherlands). 493, 123-129 (2018).
- OIE. . Tilapia Lake Virus (TiLV) - a novel orthomyxo-like virus. OIE technical disease cards. , (2018).
- Mugimba, K. K., et al. Detection of tilapia lake virus (TiLV) infection by PCR in farmed and wild Nile tilapia (Oreochromis niloticus) from Lake Victoria. Journal of Fish Diseases. , (2018).
- Koesharyani, I., Gardenia, L., Widowati, Z., Khumaira, D. D., Rustianti, Studi kasus infeksi tilapia lake virus (tilv) pada ikan nila (Oreochromis niloticus). Jurnal Riset Akuakultur. 13 (1), 85-92 (2018).
- OIE. . Tilapia lake virus disease (TiLV), Chinese Taipei. Immediate Notification. , (2017).
- OIE. . Tilapia Lake Virus Disease (TiLV), Peru. Immediate Notification. , (2018).
- Bacharach, E., et al. Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia. MBio. 7 (2), e00431-e00416 (2016).
- Eyngor, M., et al. Identification of a novel RNA virus lethal to tilapia. Journal of Clinical Microbiology. 52 (12), 4137-4146 (2014).
- Nicholson, P., et al. Detection of Tilapia Lake Virus in Egyptian fish farms experiencing high mortalities in 2015. Journal of Fish Diseases. 40 (12), 1925-1928 (2017).
- Surachetpong, W., et al. Outbreaks of Tilapia Lake Virus Infection, Thailand, 2015-2016. Emerging Infectious Diseases. 23 (6), 1031-1033 (2017).
- Tattiyapong, P., Dachavichitlead, W., Surachetpong, W. Experimental infection of Tilapia Lake Virus (TiLV) in Nile tilapia (Oreochromis niloticus) and red tilapia (Oreochromis spp.). Veterinary Microbiology. 207, 170-177 (2017).
- Kembou Tsofack, J. E., et al. Detection of Tilapia Lake Virus in Clinical Samples by Culturing and Nested Reverse Transcription-PCR. Journal of Clinical Microbiology. 55 (3), 759-767 (2017).
- Thangaraj, R. S., et al. Derivation of two tilapia (Oreochromis niloticus) cell lines for efficient propagation of Tilapia Lake Virus (TiLV). Aquaculture (Amsterdam, Netherlands). 492, 206-214 (2018).
- Hanson, L. A., Rudis, M. R., Vasquez-Lee, M., Montgomery, R. D. A broadly applicable method to characterize large DNA viruses and adenoviruses based on the DNA polymerase gene. Virology Journal. 3, 28-28 (2006).
- Josko, D. Molecular virology in the clinical laboratory. Clinical Laboratory Science. 23 (4), 231-236 (2010).
- Munir, K., Kibenge, F. S. Detection of infectious salmon anaemia virus by real-time RT-PCR. Journal of Virological Methods. 117 (1), 37-47 (2004).
- Snow, M., et al. Developement, application and validation of a Taqman real-time RT-PCR assay for the detection of infectious salmon anaemia virus (ISAV) in Atlantic salmon (Salmo salar). Developments in Biologicals. 126, 133-145 (2006).
- Matejusova, I., McKay, P., McBeath, A. J., Collet, B., Snow, M. Development of a sensitive and controlled real-time RT-PCR assay for viral haemorrhagic septicaemia virus (VHSV) in marine salmonid aquaculture. Diseases of Aquatic Organisms. 80 (2), 137-144 (2008).
- Garver, K. A., et al. Development and validation of a reverse transcription quantitative PCR for universal detection of viral hemorrhagic septicemia virus. Diseases of Aquatic Organisms. 95 (2), 97-112 (2011).
- Dalla Valle, L., et al. Development of a sensitive and quantitative diagnostic assay for fish nervous necrosis virus based on two-target real-time PCR. Veterinary Microbiology. 110 (3-4), 167-179 (2005).
- Hodneland, K., Garcia, R., Balbuena, J. A., Zarza, C., Fouz, B. Real-time RT-PCR detection of betanodavirus in naturally and experimentally infected fish from Spain. Journal of Fish Diseases. 34 (3), 189-202 (2011).
- Hodneland, K., Endresen, C. Sensitive and specific detection of Salmonid alphavirus using real-time PCR (TaqMan). Journal of Virological Methods. 131 (2), 184-192 (2006).
- Wang, X. W., Ao, J. Q., Li, Q. G., Chen, X. H. Quantitative detection of a marine fish iridovirus isolated from large yellow croaker, Pseudosciaena crocea, using a molecular beacon. Journal of Virological Methods. 133 (1), 76-81 (2006).
- van Beurden, S. J., et al. Development and validation of a real-time PCR assay for the detection of anguillid herpesvirus 1. Journal of Fish Diseases. 39 (1), 95-104 (2016).
- Ciulli, S., et al. Development and application of a real-time PCR assay for the detection and quantitation of lymphocystis disease virus. Journal of Virological Methods. 213, 164-173 (2015).
- Tattiyapong, P., Sirikanchana, K., Surachetpong, W. Development and validation of a reverse transcription quantitative polymerase chain reaction for tilapia lake virus detection in clinical samples and experimentally challenged fish. Journal of Fish Diseases. 41 (2), 255-261 (2018).
- Dong, H. T., et al. Emergence of tilapia lake virus in Thailand and an alternative semi-nested RT-PCR for detection. Aquaculture (Amsterdam, Netherlands). 476, 111-118 (2017).
- Waiyamitra, P., et al. A TaqMan RT-qPCR assay for tilapia lake virus (TiLV) detection in tilapia. Aquaculture (Amsterdam, Netherlands). 497, 184-188 (2018).
- Behera, B. K., et al. Emergence of Tilapia Lake Virus associated with mortalities of farmed Nile Tilapia Oreochromis niloticus (Linnaeus 1758) in India. Aquaculture (Amsterdam, Netherlands). 484, 168-174 (2018).
- Ferguson, H. W., et al. Syncytial hepatitis of farmed tilapia, Oreochromis niloticus (L.): a case report. Journal of Fish Diseases. 37 (6), 583-589 (2014).
- Liamnimitr, P., Thammatorn, W., U-thoomporn, S., Tattiyapong, P., Surachetpong, W. Non-lethal sampling for Tilapia Lake Virus detection by RT-qPCR and cell culture. Aquaculture (Amsterdam, Netherlands). 486, 75-80 (2018).
- Yang, C. G., et al. Evaluation of reference genes for quantitative real-time RT-PCR analysis of gene expression in Nile tilapia (Oreochromis niloticus). Gene. 527 (1), 183-192 (2013).
- Bustin, S. A. Real-time, fluorescence-based quantitative PCR: a snapshot of current procedures and preferences. Expert Review of Molecular Diagnostics. 5 (4), 493-498 (2005).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27 (2-3), 126-139 (2006).
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27 (2-3), 95-125 (2006).
- Mackay, I. M., Arden, K. E., Nitsche, A. Real-time PCR in virology. Nucleic Acids Research. 30 (6), 1292-1305 (2002).
- Wong, M. L., Medrano, J. F. Real-time PCR for mRNA quantitation. Biotechniques. 39 (1), 75-85 (2005).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. Journal of Molecular Endocrinology. 25 (2), 169-193 (2000).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Research. 6 (10), 986-994 (1996).
- Rutledge, R. G., Côté, C. Mathematics of quantitative kinetic PCR and the application of standard curves. Nucleic Acids Research. 31 (16), e93-e93 (2003).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Amal, M. N. A., et al. A case of natural co-infection of Tilapia Lake Virus and Aeromonas veronii in a Malaysian red hybrid tilapia (Oreochromis niloticus × O. mossambicus) farm experiencing high mortality. Aquaculture (Amsterdam, Netherlands). 485, 12-16 (2018).
- Fathi, M., et al. Identification of Tilapia Lake Virus in Egypt in Nile tilapia affected by ‘summer mortality’ syndrome. Aquaculture (Amsterdam, Netherlands). 473, 430-432 (2017).
- OIE. . Tilapia Lake Virus disease (TiLV), Philippines. Immediate Notification. , (2017).
- OIE. . Tilapia lake virus disease (TiLV), Malaysia. Immediate Notification. , (2017).
- Abdullah, A., et al. First detection of tilapia lake virus (TiLV) in wild river carp (Barbonymus schwanenfeldii) at Timah Tasoh Lake, Malaysia. Journal of Fish Diseases. 41 (9), 1459-1462 (2018).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Del-Pozo, J., et al. Syncytial Hepatitis of Tilapia ( Oreochromis niloticus L.) is Associated With Orthomyxovirus-Like Virions in Hepatocytes. Veterinary Pathology. 54 (1), 164-170 (2017).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Purcell, M. K., Getchell, R. G., McClure, C. A., Garver, K. A. Quantitative polymerase chain reaction (PCR) for detection of aquatic animal pathogens in a diagnostic laboratory setting. Journal of Aquatic Animal Health. 23 (3), 148-161 (2011).
- Simpson, D. A., Feeney, S., Boyle, C., Stitt, A. W. Retinal VEGF mRNA measured by SYBR green I fluorescence: A versatile approach to quantitative PCR. Molecular Vision. 6, 178-183 (2000).
- Kibenge, M. J., et al. Discovery of variant infectious salmon anaemia virus (ISAV) of European genotype in British Columbia, Canada. Virology Journal. 13, 3 (2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved