Method Article
Tilapia Gölü virüs kullanarak geleneksel RT-PCR ve SYBR yeşil RT-qPCR
Bu Makalede
Özet
Bu iletişim kuralı Tilapia Gölü virüs (TiLV) RT-PCR yöntemlerini kullanarak tilapia dokularda tanılar. Tüm yöntemi doku diseksiyon toplam RNA emme, ardından cDNA sentezi ve TiLV geleneksel PCR veya kantitatif PCR dsDNA bir floresan bağlama boya bağlama kullanarak tarafından algılanması için tanımlanır.
Özet
Bu yöntem, Tilapia Gölü virüs (TiLV) hızlı, hassas ve belirli algılama tilapia dokularda kolaylaştırmak için amaçtır. Bu iletişim kuralının bir parçası olarak gözetleme programlarının, Biyogüvenlik önlemleri TiLV temel araştırma laboratuarlarında kullanılabilir. Altın standart virüs teşhis genelde virüs yalıtımı gibi ters transkripsiyon polimeraz zincir tepkimesi (RT-PCR) daha fazla doğrulama için tamamlayıcı teknikleri takip içerir. Bu hantal, zaman alıcı olabilir ve genellikle ağır virüs bulaşmış doku örneği gerektirir. Kantitatif RT-virüs tespiti (q) PCR kullanımı nicel doğası, yüksek duyarlılık, özgüllük, ölçeklenebilirlik ve hızlı zamanı neden için nedeniyle avantajlıdır. Burada, PCR tüm yöntemi algılama kullanarak bir guanidium thiocyanate-fenol-kloroform çözüm, RNA miktar, bir iki adım PCR tarafından takip tilapia organ parça, toplam ribonükleik asit (RNA) çıkarma üzerinden tarif TiLV için yaklaşımlar tabanlı iletişim kuralı gerektiren, tamamlayıcı Deoksiribonükleik asit (cDNA) sentezi ve TiLV geleneksel PCR ya da qPCR ile SYBR yeşil kullanarak sayısal kimlik tespiti boya. Geleneksel PCR sonrası-PCR adımları gerektirir ve sadece virüs varlığı hakkında bilgi verecek. İkinci yaklaşımı TiLV mutlak miktar az 2 kopya aşağı için izin verir ve böylece alt klinik durumlarda TiLV tanı için son derece yararlı. İki PCR yaklaşımlar, temsilcisi iki laboratuar sonuçlarını ayrıntılı bir açıklamasını ve kapsamlı bir tartışma her iki kritik parametrelerin araştırmacılar ve doktorlarla onların en uygun ve uygulanabilir bulmak sağlamak için dahil edilmiştir TiLV algılama yöntemi.
Giriş
Küresel düşen balık tedarik 2014 yılında 20 kg yeni bir kayıt ulaştı ve bu su ürünleri güçlü büyüme nedeniyle. Su ürünleri en hızlı büyüyen hayvan gıda üreten sektörlerde dünya çapında biri olmaya devam etmektedir ve insan nüfusunun1' den daha hızlı büyüyor sadece hayvan yemi üretim sektörüdür. Tilapiine cichilds dünya çapında toplam üretiminin 6.4 milyon ton (MT) ve tahmini değeri 9,8 milyar dolarlık 20152ile çiftlik en önemli ikinci tatlı su balık oluşturmaktadır. Çin tilapia ilk on ürete ülkelerdir (1,78 MT), Endonezya (1.12 MT) ve Mısır (0,88 MT), Bangladeş, Vietnam, Philippines, Brezilya, Tayland, Kolombiya ve Uganda2ardından. Bu küresel tilapia üretim yaklaşık 7.3 olacağı bekleniyor MT 2030 ile3. Tilapia altında su ve İklim koşulları5,6geniş bir kapasite doğurmak kolay oldukları için sadece onlar protein4 ucuz bir kaynak ama aynı zamanda çünkü böyle bir önemli küresel gıda kaynağı haline gelmiştir. Sadece birkaç on yıl önce bu birkaç ticari açıdan önemli hastalıklar tilapia tarım tehdit edildi ama bu artık doğru değil inanılıyordu. Tilapia Gölü virüs hastalığı (TiLVD) denilen gelişmekte olan bir viral hastalık ilk hiç kritik hastalık salgını tilapia içinde bulunan ve tüm sanayi tehlikede olduğunu. Bu hastalığın ciddi sosyo-ekonomik sonuçları ve gıda güvenliği milyonlarca insan Afrika7, Asya ve Güney Amerika için doğrudan bir tehdit. 2018 başlangıcında, Dünya Örgütü için hayvan sağlık (OIE) bu hastalığı, TiLV, etiyolojik Ajan resmen sekiz ülkede8 kapsayan üç kıtada ve bu patojen bilgi kartı yana ulaştırılmalarını bildirdi Orada güncelleme TiLV Tanzanya, Uganda9, Endonezya10, Tayvan11 ve Peru12daha fazla rapor edilmiştir. TiLV bir roman tek iplikçikli RNA a değişiklik-in diğer orthomyoxoviruses gibi grip veya enfeksiyöz somon anemi (kansızlık) virüs (ISAV)13anımsatan özellikleri içerdiğinden bir orthomyxo gibi virüs olmak açıklanan virüstür. İlk büyük kayıp vahşi ve çiftlik tilapia Celile Gölü, İsrail14sonrasında tespit edilmiştir. Bundan sonra benzer hastalık salgınları yaz Scotlan olarak ve bir ölüm sendromu TiLV enfeksiyonu ile ilişkili olduğunu bildirdi Nil tilapia (Oreochromis niloticus) Mısır15 ve Nil ve kırmızı hibrid tilapia ay sevk (Oreochromis spp.) Tayland16, anılan sıraya göre. Suda yaşayan hayvan virüs algılama analizine tarihsel olarak büyüme ve yalıtım hücre kültüründe virüslerin tarafından yürütülen. Çeşitli hücre satırları yayılma ve TiLV dahil olmak üzere, yılanbaşı balık (Ophiocephalus striatus)17,18, türetilmiş E-11 hücreleri OmB ve Oreochromis kaynaklanan TmB yalıtım için test edilmiştir Mossambicus18ve OnlB ve Nil tilapia (O. niloticus)19kaynaklanan OnlL. Virüs kültür için daha fazla deney materyalleri sağlar avantajı olsa da, en az 4-7 gün cytopathic etkileri (CPE) oluşumu gözlemlemek için gerektirir ve en önemlisi, farklı piscine virüsler, için daha uygun olan dezavantajı vardır çoğaltma yayılır ve benzer CPE üretmek.
Son birkaç on yıl içinde geleneksel, genellikle zaman alıcı tanı yöntemleri hücre kültürü, seroloji ve antijen algılama ve yerine göre daha hızlı ve daha hassas nükleik asit algılama testleri20gibi uzak bir hamle oldu, 21. Bu birçok qPCR deneyleri için hayvanlar, viral hastalıklarda çok sayıda önemli tanı yöntemleri olarak gibi ISAV22,23için viral hemorajik septisemi virüs (VHSV)24 geliştirilmiştir Aslında tarafından belirgindir ,25, betanodavirus26,27 salmonid alphavirus28, balık iridovirus29, Anguillid herpesvirus 1 (AngHV1)30ve Lymphocystis hastalık virüsü (LCDV)31 . Tanı ve patojen gözetimi için güçlü Yöntemler TiLV yayılmasını azaltmak için şiddetle ihtiyaç vardır. Bu tür yöntemleri klinik belirtiler geliştirmeden önce enfeksiyon erken teşhis ve düşük virüs yükler tespiti izin vermelisiniz. RT-PCR14,32, dahil olmak üzere farklı PCR protokolleri bugüne kadar iç içe geçmiş RT-PCR18, yarı iç içe RT-PCR33ve RT-qPCR32,34 TiLV tespiti için geliştirilmiştir Balık dokularda. RT-qPCR ve virüs izolasyonu TiLV algılama için duyarlı hücre hatlarında bir karşılaştırmasını RT-qPCR virüs yalıtım321000 kat daha duyarlı olduğunu ortaya koydu. Yayımlanmış her PCR Protokolü TiLV tespiti için farklı hassasiyetleri bildirdi, çoğu deneyleri 7.5 kopya33, 7 kopya18 veya başına 2 kopya32 viral kopyaları algılama sınırları ile son derece hassas olmakla birlikte reaksiyon.
Bu yöntemleri makalenin amacı, TiLV algılama deneyleri, toplam RNA ayıklama, tilapia doku koleksiyonuna cDNA sentez başlayarak gerçekleştirmek nasıl ayrıntılı olarak açıklamak için ve daha sonra TiLV belirli PCR deneyleri temel. Özellikle, hem geleneksel RT-PCR ve aynı zamanda SYBR yeşil tabanlı RT-qPCR kapsamlı iletişim kuralları hitap TiLV algılamak için hedefleyen Bilim adamları geniş bir yelpazesi için tarif edilmiştir. Eski daha az duyarlıdır ancak genellikle daha ucuz bir algılama seçenektir. İkinci bir kantitatif PCR makinesi ve daha pahalı kimyasalları gibi daha ayrıntılı altyapı gerektirir, ancak nicel, hızlı ve son derece hassas, bu TiLV içinde tespiti için alt klinik olarak kullanılabileceğini anlam olmanın avantajı vardır Balık bulaşmış. RT-PCR ve RT-qPCR iletişim kuralları TiLV ve dahil sonuçları vurgu duyarlılığı ayrı coğrafi yalıtır ve burada açıklanan deneyleri tekrarlanabilirlik ile iki farklı olarak yapıldı.
Protokol
Bu çalışma için hayvan Kullanım Protokolü Kasetsart Üniversitesi hayvan Etik Komitesi izni numarası ACKU 59-VET-016 altında tarafından kabul edildi.
Not: Tablo reçetesi reaktifler ve bu iletişim kuralı için önerilen ekipman ile ilgili geniş bilgi için bakınız.
1. doku örneği koleksiyonu
- Karanfil yağı (birim balık boyutunu ve konsantrasyon ürünleri, genellikle birden fazla 3 mL/L bağlıdır) aşırı dozda kullanarak balık ötenazi. Forseps ve mayo makas dörtte birinden bir alkol brülör ekipman sterilize etmek için kullanarak ekipman yakarak takip %95 (v/v) etanol içine bırakın.
Not: Tricaine methanesulfonate (MS-222) karanfil yağı yerine kullanılabilir. - Karaciğer bulmak ve küçük bir parça (yaklaşık 20-100 mg) kesin ya da 200 µL tilapia balık posterior anterior üzerinden mukus kaldırmak ve örnekleri bir 1.5 mL microcentrifuge tüp içine yerleştirmek için cam ya da cerrahi bıçak kullanarak mukus toplamak.
- İşlem örnekleri hemen, çözüm sabitleme bir RNA depolamak veya-80 ° C'ye kadar daha fazla kullanılmasını taşıyın.
Not: RNA ile çalışmak en büyük görev sağlam RNA molekülleri hazırlama ve onları herhangi bir sonraki handlings boyunca hasarsız tutmak. RNA belkemiği DNA hasar için doğuştan daha duyarlıdır. Çıkarma ve doku hücrelerinin toplam RNA'ın yalıtım gerektirir dikkatli Laboratuvar tekniği; eldivenler, kullanarak RNase free su, reaktifler, donanımları, plastik eşya, cam eşyalar, çalışma alanı ve pipetting için filtre ipuçları kullanarak RNase kontaminasyonu önlemek için tüm hükümler al.
2. Guanidium Thiocyanate - fenol - kloroform ayıklama RNA'ın
- 1 mL 1 bölümünden doku örneği içeren bir tüp içine fenol ve guanidin isothiocyanate içeren monophasic çözeltisi ekleyin.
Uyarı: Bu çözüm çok zehirli ve laminar akış başlıklı koruyucu ekipman ve uygun koruyucu gözlük, giyim ve emanet eldiven giyiyor tarafından itina ile ele alınması gerekir. - Bir doku havaneli homogenizer kadar homojen kullanarak doku örneği eziyet.
Not: Örnekleri de seramik boncuklar ile kombine bir güç homogenizer kullanarak homojen. Doku örneği tamamen sonraki adıma devam etmeden önce homojenize sağlamak ya da iletişim kuralı burada durmak ve-80 ° C'de tam homojenize örnekleri daha fazla kullanmak kadar saklamak. - Kloroform faz ayrılması için 200 µL ekleyin.
DİKKAT kloroform potansiyel bir narkotik ve son derece tehlikeli. Laminar akış başlıklı koruyucu ekipman ile yanı sıra uygun koruyucu gözlük, giyim ve emanet eldiven giyiyor tarafından özenle ele alınmalıdır. Bir daha az toksik olarak alternatif, 1-Bromo-3-sersemletici de kullanılabilir.
Not: uygun olan yerlerde, yukarı veya aşağı birimleri ölçeklendirme. Fenol ve guanidin isothiocyanate içeren monophasic çözüm 500 µL kullanılan yalnızca, örneğin, daha sonra sadece 100 µL kloroform, bu adımda ekleyin.- Mix örnekleri de tarafından inversiyon 15 s.
- Örnekleri, oda sıcaklığında (RT) 3 min için kuluçkaya.
- Santrifüj için 12.000 × g de 15 dk ve 4 ° c
Not: Daha düşük bir organik faz, beyaz bir interfaz ve RNA içeren bir üst sulu faz içine temiz bir ayrılık olmalıdır. Bu üst aşamasıdır normalde renksiz ama türü ve homojenize doku miktarı bağlı olarak hafif pembe bir görünüm olabilir. - Üst sulu katmanı (yaklaşık 500 µL) taze microcentrifuge tüp Interphase bozmadan aktarın.
Not: tüm sulu faz transfer, organik sulu faz içeren RNA'ın herhangi bir potansiyel kontaminasyonu önlemek için küçük bir miktar bırakın veya Interphase çalışmayın. - %1 100 isopropanol RNA çökelti hacmi ekleyin.
- Doku çok az miktarda kullandıysanız, isteğe bağlı olarak, daha sonra 1 µL (5-10 µg) RNase free glikojen verimli RNA yağış tanıtmak için her örnek için ekleyin. Bu adımda 2.8 RNA Pelet tanımlaması yardımcı olacak.
Not: Glikojen bir taşıyıcı RNA olarak davranır ve RNA'ın küçük miktarlarda tüp tarafına yapışmasını engeller. - Mix de INVERSION tarafından birkaç kez tüpleri.
- Örnekleri için 2 h-20 ° C'de bir gecede depolamak
- Doku çok az miktarda kullandıysanız, isteğe bağlı olarak, daha sonra 1 µL (5-10 µg) RNase free glikojen verimli RNA yağış tanıtmak için her örnek için ekleyin. Bu adımda 2.8 RNA Pelet tanımlaması yardımcı olacak.
- 12.000 x g ve 4 ° c de 15 dakika santrifüj örnekleri
- Microcentrifuge tüp alt RNA Pelet çıkarmak değil dikkatli olmak süpernatant, atın.
- 1 mL % 75 etanol (v/v) ve RNA örnekleri tarafından ters çevirme tüp birkaç kez karıştırın.
- 10.000 x g ve 4 ° c ' 15 dakika santrifüj
Not: Protokol burada durdurulabilir ve RNA Pelet % 75 etanol içinde oluşan örnekleri-20 ° C'de daha fazla kullanılmasını kadar saklanabilir. - Microcentrifuge tüp alt RNA Pelet çıkarmak değil dikkatli olmak süpernatant, atın.
- İsteğe bağlı olarak, adımları yineleyin 2,9-%2,11 70 etanol (v/v) kullanarak. İyice RNA Pelet yıkama tuz en aza indirmek olacaktır ya da engel olabilecek kirletici etkilenmişimdir hassas aşağı akım uygulamaları.
- Bir pipet kullanarak kalan etanol çizin ve sonra kuruması için artık 5-10 dk daha oda sıcaklığında RNA Pelet.
Not: aşırı Kuru granül yeniden askıya almak zor olacak. - 30-60 µL 55-60 ° C-RNA Pelet solubilize için önceden sıcak su, RNase free ekleyin.
- RNA buz acele kullanma veya mağaza-80 ° c daha sonra kullanmak için yerleştirin.
3. RNA bir mikro-cilt spektrofotometre kullanarak toplama ölçmek
- Spektrofotometre ayarları için RNA geçin.
- 1-2 µL RNase free su boşluk kullanın.
- 1-2 µL her RNA örneğinin RNA miktarı değerlendirmek için kullanın.
- Kayıt okuma at 230 nm, 260 nm ve 280 nm her örnek için.
- RNA 200 seyreltik ng/µL RNase free su kullanarak.
4. tamamlayıcı DNA (cDNA) kullanarak toplam RNA sentezi
- Toplam RNA protokolden 2, 2 µM oligo (dT), 0,5 mM dNTPs karışımı 1 µg karıştırıp nükleaz ücretsiz su ile 10 µL son hacim getirmek. Bunun için bir RT-master-test edilecek mix örnekleri ve denetimleri sayısına göre hazırlayın.
Not: Bir eksi transkriptaz örnek denetimleridir (-RT) neyin RT enzim nükleaz ücretsiz su ile değiştirilir (bkz. Adım 4.3) ve onda nükleaz ücretsiz su RNA şablon yerine ana karışıma eklenir bir şablon kontrol (NTC).- Örnekleri de kısa aralıklarla tarafından takip pipetting tarafından karıştırın.
- Örnekleri 65 ° c 5 min 2 dk kuluçka tarafından buz üzerinde takip için kuluçkaya.
- Kısaca tüm sıvı tüpler alt toplamak için örnekler santrifüj kapasitesi.
- 1 x transkriptaz arabellek, 100 U ters transkriptaz ekleyin ve her örnek son hacmi 20 µL nükleaz ücretsiz su kullanarak getirmek.
- Örnekleri de kısa aralıklarla tarafından takip pipetting tarafından karıştırın.
- 60 dk 85 ° C 5 min için takip için 42 ° C'de örnekleri kuluçkaya.
- İstenen bir konsantrasyon için sentezlenmiş cDNA nükleaz ücretsiz su uygun bir hacmi ekleyerek sulandırmak ve cDNA buz için acele kullanma yerleştirin veya daha sonra kullanmak için-20 ° C'de saklayabilirsiniz.
5. TiLV geleneksel PCR
- CDNA, örnekleri ve şablonlar için Tablo 1' de, bir DNA polimeraz ile birlikte seçim ayrıntılı kurulan astar çiftleri birini kullanarak bir PCR reaksiyon olarak Bölüm 4 iletişim kuralında oluşturulan denetimleri kullanın.
Not: Ek bir no-şablonu denetim (NTC) burada cDNA nükleaz ücretsiz su PCR reaksiyon için yerine kullanarak dahil edilmelidir. Olumlu bir denetim, varsa, aynı zamanda önceden doğrulanmış TiLV olumlu örnekleri oluşan dahil edilecek veya uygun TiLV cDNA parça içinde bir plazmid klonlanmış. - PCR master mix ve örnekleri ve test edilmesi için denetim sayısı DNA polimeraz sisteminin esaslarına göre hazırlayın. Bu karışımı ileri astar, ters astar, dNTPs, MgCl2 ve kendi arabelleğini birlikte seçilen DNA polimeraz içermelidir.
- Seçili DNA polimeraz kurallarına göre belirlenen birim mix-master cDNA örnekleri ve kontrol örnekleri önerilen miktarı ile birleştirin.
Not: bazı master Mix kayıp pipetting sırasında reaksiyon aşırı x 0.5 hazırlanıyor kez yararlı olduğu için. - Kullanılan DNA polimeraz sisteminin yönergelere uygun koşullar Bisiklete binme ve astar kullanılan (Tablo 1) için uygun bir tavlama sıcaklığı kullanarak PCR gerçekleştirin. Genellikle, böyle bir program bir ilk denatürasyon 95 ° c için 2-5 dk, 30-40 döngüleri denatürasyon 95 ° C'de 30 ardından içerecektir s, 30 s ve uzama 30 72 ° C'de için önerilen sıcaklıkta tavlama 72 ° C 5-10 için son bir uzama ardından s Min.
- 5-15 µL her PCR reaksiyon ve beklenen PCR ürünü boyutuna bağlı olarak % 1-2 özel jel kuyu için bir uygun DNA merdiveni yük. Jel Elektroforez tarafından güçlendirilmiş DNA ayrı ve jel etidyum bromür (EthBr) görüntüleme DNA gruplarından biri beklenen boyutu (Tablo 1) kolaylaştırmak için bir jel dokümantasyon makine UV ışık kullanarak leke.
Dikkat: EtBr toksik; Bu uygun koruyucu giysi ve emanet eldiven giyerek dikkatle ele alınmalıdır.
| Hedef TiLV genom kesimi | İleri astar 5' - 3' | Ters astar 5' - 3' | PCR ürünü boyutu (bp) | Tm ° C | Orijinal başvuru | ||||
| 1 | CCAAACGTTATCTCTTAATTACGCAC | GCAAATATTTCTCTCATTCGCCT | 1641 | 50 | Surachetpong ve ark., 2017 | ||||
| 1 | CCTCATTCCTCGTTGTGTAAGT | AGGAGTTGCTGTTGGGTTATAG | 1000 | 62 | Mugimba ve ark., 2018 | ||||
| 2 | ACTCTCTATTACCAAATACATTTACT | TTACCATATATATAGTGAAGGC | 1445 | 45 | Surachetpong ve ark., 2017 | ||||
| 2 | GTCCAGGGCGGTATGTATTG | CTTACGGCTGACAAGTCTCTAAG | 834 | 62 | Mugimba ve ark., 2018 | ||||
| 3 | GTTGGGCACAAGGCATCCTA | TATCACGTGCGTACTCGTTCAGT | 250 | 56 | Eyngor ve ark., 2014 | ||||
| 3 | TATGCAGTACTTTCCCTGCC | TTGCTCTGAGCAAGAGTACC | 491 | 57 | Eyngor ve ark., 2014 | ||||
| 3 | ACCCCTTAATCCTTAATAGACCGTTA | CCCATAATCCTCTATTAGAACGTCGT | 1352 | 50 | Surachetpong ve ark., 2017 | ||||
| 3 | GTCGAGGCATTCCAGAAGTAAG | GAGCTAAGGGAACGGCTATTG | 834 | 62 | Mugimba ve ark., 2018 | ||||
| 4 | AGCAGCAGCAGGAGAAAGAG | ACCGTCCTGTTTCTGAATGG | 358 | 60 | Nicholson ve ark., 2017 | ||||
| 4 | CCAAAGTTTACTCCTATTACCCAGA | GCAAATCTTTCTCCAATTACCGTCT | 1250 | 50 | Surachetpong ve ark., 2017 | ||||
| 4 | GCCCAATGGTTCCCATATCT | GCCCAATGGTTCCCATATCT | 524 | 62 | Mugimba ve ark., 2018 | ||||
| 5 | CCAAATGTTTCTCTTATCTCAGACTC | CTTTTTCTCAGTTTACCACTTTATG | 1087 | 57 | Surachetpong ve ark., 2017 | ||||
| 5 | CAACTCTTAGCCTCCGGAATAC | CGTTCTGCACTGGGTTACA | 696 | 62 | Mugimba ve ark., 2018 | ||||
| 6 | CCAAATTTTACCTCTCGCAT | TCAAGCACTTAAAACTGTACC | 1027 | 45 | Surachetpong ve ark., 2017 | ||||
| 6 | CCCACACGACAGGACATATAG | GAGTTGGCTTAGGGTGATAAGA | 948 | 62 | Mugimba ve ark., 2018 | ||||
| 7 | CTCTCTTTGCATTGCATACCGT | GACCAATTATCCCTGCTTTCA | 704 | 57 | Surachetpong ve ark., 2017 | ||||
| 7 | TCCTTTAGGGATTGGCACTAAC | TTCCATCGACTGCTCCTAGA | 486 | 62 | Mugimba ve ark., 2018 | ||||
| 8 | ACCTCATCTACACTAACATTTCCA | TCATCATTACACAAATGGAGTAGCT | 637 | 50 | Surachetpong ve ark., 2017 | ||||
| 8 | CTTAAGGGCCATCCTGTCATC | TGGCTCAAATCCCAACACTAA | 476 | 62 | Mugimba ve ark., 2018 | ||||
| 9 | TTGGTGATGTCACGATGGATA | AGTTCTATCGCCAGCCATGT | 351 | 60 | Nicholson ve ark., 2017 | ||||
| 9 | ACAAGTCCGATTACTTTTTCCGC | TCTTTCTCACGTCCTTAAAGTCA | 530 | 50 | Surachetpong ve ark., 2017 | ||||
| 9 | GATATCCTCCACATGACCCTTC | GTACGTCACTTTGTGCCATTAC | 261 | 62 | Mugimba ve ark., 2018 | ||||
| 10 | AACCCTACTAACACCAAATATAGCT | CTTTCCCTCTGACACCCTGT | 450 | 50 | Surachetpong ve ark., 2017 | ||||
| 10 | TCCTCTCTGTCCCTTCTGTT | CAGGATGAGTGTGGCAGATTAT | 276 | 62 | Mugimba ve ark., 2018 | ||||
Tablo 1. Bitiş noktası PCR kullanarak TiLV cDNA amplifikasyon için yayımlanmış astar çiftleri. Kalın olarak gösterilen ayarla astar Şekil 3A ve 3B temsilcisi sonuçlar oluşturmak için kullanılmıştır.
6. TiLV nicel polimeraz zincir reaksiyonu (qPCR)
- Uygun TiLV içeren bir plazmid kullanarak genomik kesimi 3 cDNA pTiLV32gibi bir standart olarak bir yinelenen veya triplicated 10 kat seri seyreltme serisi hazır olun.
- Tüm örnekler, standartları ve denetimleri için bir qPCR usta-karışım hazırlamak, tepkiler-meli var olmak kılınmak içinde dikkate alarak çoğaltmak veya kullanan 0.4 µL nükleaz ücretsiz su, 0.3 µL ileri astar, 0.3 µL ters astar ve 2 SYBR yeşil x 5 µL onaylatılacak DNA polimeraz master-mix tepki başına.
- Astar astar bilgi ve standart pTiLV ve 10 µM, konsantrasyon aşağıdaki gibi kullanın:
İleri astar: TiLV-112F (5'-CTGAGCTAAAGAGGCAATATGGATT-3')
Astar: TiLV ters-112R (5'-CGTGCGTACTCGTTCAGTATAAGTTCT-3')
Standart pTiLV:10 pg/µL
Not: örnekleri ve denetimleri toplam sayısı 10 ise ve triplicates içinde gerçekleştirilecek, bu bir qPCR usta-12 µL nükleaz ücretsiz su, 9 µL ileri astar, 9 µL ters astar ve SYBR yeşil DNA polimeraz master Mix-150 µL oluşan karışıma eşittir. Ticari olarak satın 2 x Evrensel SYBR yeşil DNA polimeraz master-karışımları tüm gerekli bileşenleri qPCR reaksiyon için yani, SYBR ben boya yeşil, sıcak-başlangıç Taq DNA polimeraz, dNTPs, MgCl2 ve pasif başvuru boyalar içerir. SYBR yeşil ana karışım ışıktan korumak.
- Astar astar bilgi ve standart pTiLV ve 10 µM, konsantrasyon aşağıdaki gibi kullanın:
- QPCR şerit tüpler veya 96 iyi plaka kullanımda qPCR makine ile uyumlu içine qPCR master Mix-6 µL dağıtmak.
- CDNA şablon, denetimleri veya seri olarak seyreltilmiş TiLV standartları 4 µL tüpler veya 96 iyi plaka kuyu ekleyin.
- QPCR tüpleri kapatmak veya qPCR makine kullanmak için uyumlu plaka kapak ile 96 iyi plaka mühür
- Yavaşça qPCR tüplere mix çözüm ve spin qPCR tüpler veya 96 iyi plaka tüm sıvı damarları alt toplamak için bir santrifüj kullanarak aşağı hafifçe vur.
- Tüpler veya plaka gerçek zamanlı termal cycler yerleştirin.
- Bir ilk denatürasyon 95 ° C'de 3 min 95 ° C 40 döngüsü tarafından takip için gerçekleştirmek için qPCR thermocycler programı 10 s ve 60 ° C 30 için astar tavlama ve uzama, erime eğrisi ile bir adım 65 ° c ila 95 ° C 0,5 ° C bir artışı ile biten için s / 5 s.
- SYBR bir fluorophore boya seçin sonra bilinmeyen bir örnek türü olarak seçin ve bir örnek adı kutusuna bir ad ekleyin.
- RT-qPCR makine kapağını açın ve qPCR şerit atanan kuyu yerleştirin, sonra kapağını kapatın.
- RT-qPCR tahlil Seçilen koşulları ile gerçekleştirin. Makinenin kapağı istenilen dereceye geldi sonra çalışmaya başlar. Her örnek floresans tepki ilerlemesini izlemek için her uzantısı adım sonra toplamak.
Not: QPCR makine ve ilgili yazılım otomatik olarak tüm parametreleri testin hesaplamak ve amplifikasyon eğrileri gerçek zamanlı olarak görüntülemek, standart eğri ve erime eğrisi qPCR döngüsü sonunda oluşturulan iken. - Veri analizi ve erime eğrileri ilk sağlayarak edinme her örnek için gerçekleştirmek ve standart amplicon için beklenen sıcaklığında bir üniforma tepe var.
- Amplifikasyon eğrileri örnekleri değerlendirmek ve standartları ve küme eşik bölgesinde cDNAs güçlendirme oranı tüm örneklerinde aynıdır. Bu normalde otomatik olarak yazılım tarafından gerçekleştirilir ancak dikkatli bir şekilde denetlenmelidir.
- Standart eğri kullanarak TiLV kopya sayısını hesaplayın.
Sonuçlar
1 bölümünde açıklanan protokolü TiLV enfeksiyonu (Şekil 1A) klinik belirtileri gösteren can çekişen kırmızı hibrid tilapia karanfil yağı, bir anestezik davranan yüksek konsantrasyon yıkanan tarafından euthanized. Bildirilen klinik belirtiler değişkendir ama yaygın belirtileri uyuşukluk, cilt erozyon ve renk değişikliği, exophthalmia, Müstakil ölçekler, açık yaralar/lezyon ve anormal davranış15,16,33gibi görünür, 35,36, bunlardan bazıları açıkça görülebilir Şekil 1A'. Karın duvarının iç organlara karaciğer, dalak veya baş böbrek (Şekil 1B) gibi toplamak için kaldırıldı. Mukus örnekleri de bu aşamada yavaşça ön arka cam veya cerrahi bıçak37kullanarak balık için deriden kazıma toplanmıştır.

Resim 1 . Tilapia diseksiyon ve örnek koleksiyon. A. kırmızı hibrid TiLV enfekte tilapia ile cilt leisons, ağız ve operculum, cilt erozyon ve kornea opaklık çevresinde kızarıklık. B. Sectioned kırmızı hibrid tilapia (noktasında mavi ok), karaciğer dalak veya baş böbrek organ doku koleksiyonu için izin vermek için. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bundan sonra Bölüm 2 toplam RNA Guanidium Thiocyanate-fenol-kloroform çıkarım için ayrıntılı protokolü takip etti ve madde 3 te özetlenen RNA miktar gerçekleştirilen örnek saflık değerlendirmek için saflık oranları hesaplama tarafından ve Spektral profilleri (Şekil 2) incelenmesi. Zavallı bir RNA hazırlık Şekil 2B temsil ederken Şekil 2A başarılı bir toplam RNA çıkarma yordamı, temsili bir sonucu gösterir. Nükleik asitler proteinler onların 280 varken 260 absorbans maxima var nm. Oranı ölçümlerin yüzüne 260 nm ve 280 nm gösteren her örnek saflığı ve oranları 1.9-2,1 gösterir saf RNA Şekil 2Aörneğinde olduğu gibi. Şekil 2B içinde gözlenen alt A260/280 oranları olası protein veya fenol kirlenme RNA ayıklama yordamdan artık gösterir. Absorbans 230 nm örnek kirlenme sonucu olabilir ve A260/230 nm oranı da bu nedenle hesaplanır. Bu oran 2.0-2.2 saf RNA hazırlıkları için aralığında olmalıdır süre 2B rakam vardır 1,07 düşük A260/230 oranında ve spektral profil at 230 yalak bir kayma gösterir 2,03 Şekil 2A, örnek için bir değeri tarafından gösterildiği gibi doğru 240 nm Artık guanidin veya fenol örnek göstergesidir nm. Şekil 2Bgösterilen örnek için yeniden kirlenme kaldırmak için RNA presipite örnek saflığı artırabilir.
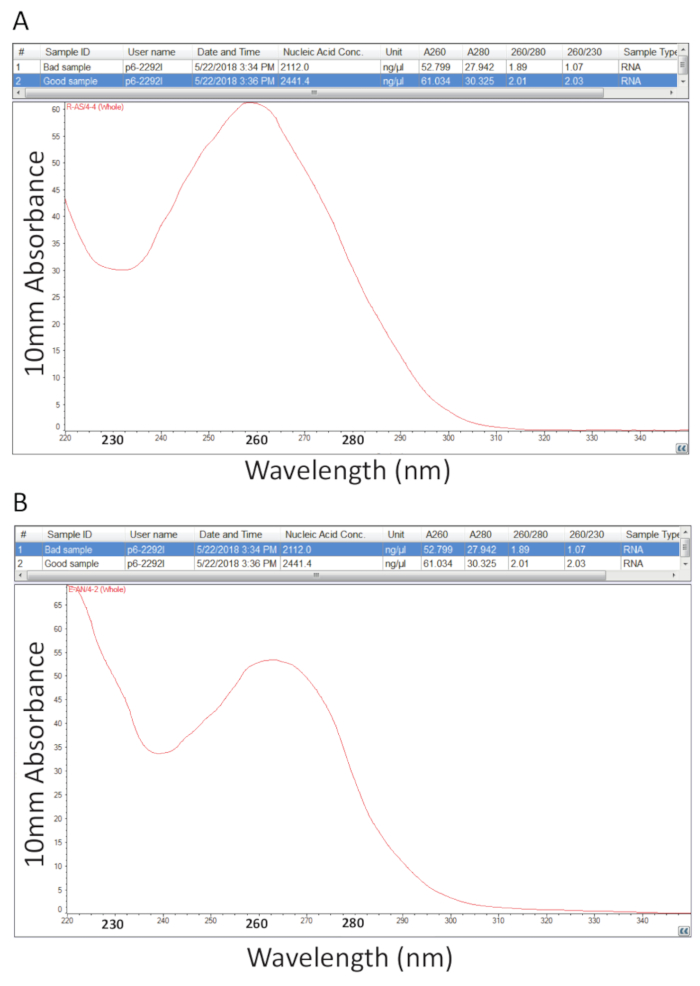
Resim 2 . Spektrofotometrik miktar toplam RNA hastalıklı tilapia dokulardan ayıklanır. A. saflık oranları ve başarılı bir RNA hazırlık spektral profillerden. B. olarak A, zavallı bir RNA çıkarma yordamı temsilcisi hariç. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
TiLV RT-PCR tarafından tespit etmek için 2A yolunu temsil gibi saf örnekleri ters kopya etmek (iletişim kuralı 4) cDNA içine edildi ve kullanılan PCR için bir şablon tahlil ayrıntılı olarak açıklandığı Bölüm 5 ve temsilcisi sonuçları 3A rakamgösterilir. Tablo 1 ' de gösterilen astar kalın TiLV genomik kesimi 314491 bp parçası yükseltmek için kullanıldı. PCR ürünleri jel elektroforez tarafından ayrılmış ve EtBr ile görselleştirme için lekeli. Şekil 3A hastalıklı tilapia Tayland ve her örnek, yaklaşık 500 temiz tek bir bant izole karaciğer türetilen 4 cDNA örnekleri (S1-S4) kullanarak bir iki adım RT-PCR sonuçlarını gösterir bp görülebilir ve bu yüzden, TiLV örnekleri 1-4. olumlu. Aynı PCR ürünü hiçbir şablon kontrolü (NTC) PCR ürünler vermemiştir iken plazmid32 klonlanmış cDNA TiLV segmentin 3 oluşan olumlu denetim örneğinden elde edildi. Şekil 3B tahlil Şekil 3A olduğu gibi ama bir tek adımlı RT-PCR yaklaşım kullanarak farklı bir laboratuvar ve baş böbrek doku içinde Mısır su ürünleri kaynaklanan tilapia türetilen 5 RNA örnekleri ile aynı primerler kullanılarak gerçekleştirildi 15. bu kullanarak bu algılama tahlil örnekleri 1, 3 ve 5 TiLV pozitif iken hiçbir PCR ürünü doğru boyutta bulduğumda 2 ve 4 TiLV negatif olduğu belirlendi. Negatif denetimleri transkriptaz denetimleri ve iki NTCs eksi 2 de dahil olmak üzere herhangi bir PCR ürününün oluşturmadı. Bir tek adımlı RT-PCR tahlil de tilapia ActinB gen hedefleme gerçekleştirildi. 217 amplicon boyutunu bp beklenen38her örnek (S1-S5) oluşturulur. Bu tahlil TiLV olumlu örnekler yarı nicel bir sınav için izin yanı sıra, bir denetim için RNA örnekleri bütünlüğünü olarak görev yaptı. Verilen bu oluşturulan Tilapia ActB ürün nispeten eşit, TiLV belirli PCR ürünü oluşturulan miktarı farklılıkları TiLV miktarına verilen doku örneği olarak doğru bir yansıması olarak yorumlanabilir.
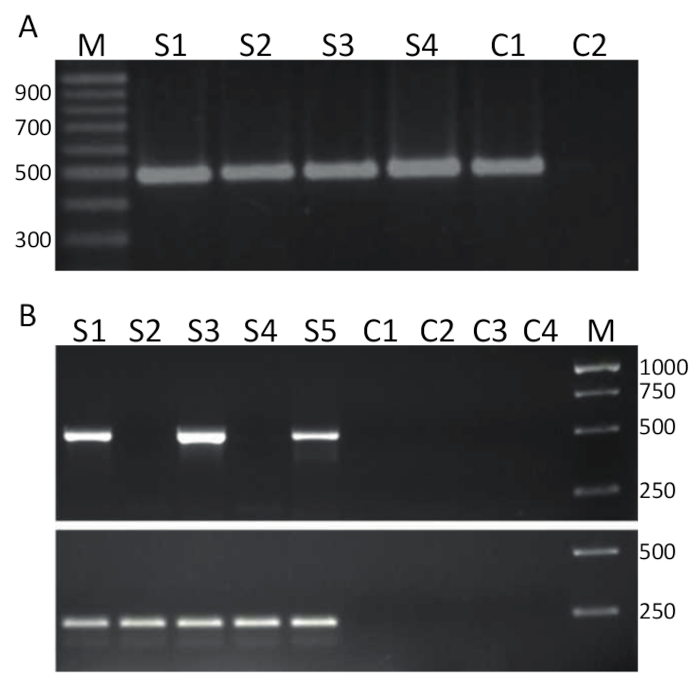
Şekil 3 . TiLV RT-PCR. A. hastalıklı Tilapia, karaciğer dokulardan üretilen cDNA örnekleri toplanmıştır Tayland'dan ekran 3 segment için belirli primerler kullanılarak TiLV enfeksiyon için (gösterilen kalın olarak Tablo 1), 2-adım RT-PCR tahlil kullanarak TiLV. M baz çifti olarak; gösterilen işaretçisi = S1-S4 örnekleri 1-4; = C1 = pozitif kontrol pTiLV kullanarak PCR şablon olarak; ve C2 hiçbir şablon kontrolü (NTC) =. B. tek adımlı RT-PCR A olduğu gibi aynı primerler kullanılarak ve hastalıklı tilapia baş böbrek doku örnekleri toplanan Mısır15. M baz çifti olarak; gösterilen işaretçisi = S1-S5 örnekleri 1-5 =. Denetimleri C1-C2 transkriptaz denetimleri ve C3-C4 NTCs. alt paneli bir tek adımlı RT-PCR tilapia ActinB38 (Ayrıntılar için bkz: metin) üreten karşı yönetmen astar kullanıyor 217 baz çifti, PCR ürünü. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bitiş noktası Şekil 3, 6, iletişim kuralında açıklanan qPCR deneyleri temsil PCR aksine her PCR döngüsü sonra PCR ürün miktarını ölçmek. Hedef DNA amplifikasyon tepki her turda oluşturulan DNA ile etkileşim floresan molekülleri kullanarak algılanır. Burada, hangi çift iplikçikli DNA ile intercalates SYBR yeşil ben boya kullanılmıştır. Floresan sinyal reaksiyonu sırasında takip edilir ve şiddeti ile ilgilidir ürün miktarını 39,40,41,42,43kurdu. TiLV qPCR deneyleri Protokolü 6 farklı SYBR yeşil reaktifler, qPCR makineleri ve örnek farklı ülkeden kullanarak farklı laboratuarlarında açıklandığı gibi yapılmıştır. Elde edilen amplifikasyon eğrileri Şekil 4A ve 4Bgösterilir. Her tahlil için deneme seyri dört aşama vardır görülebilir: doğrusal zemin aşama, erken üssel faz, geç üssel faz ve Plato aşama. Doğrusal zemin faz nerede DNA çoğaltılması henüz yetersiz sinyal/arka plan oranı üreten DNA miktarları nedeniyle tanımlanamıyor erken döngüsü sırasında oluşur. Temel floresans bu evre sırasında hesaplanır. Bundan sonra hedef DNA çift konsantrasyon katlanarak artırmak için arka plan üstüne algılanabilir sinyal inducing her döngüsü ile başlar. İyi optimize edilmiş qPCR tahlil amplifikasyon verimliliği (E) çok yüksek (%100 bu aşamasında erken üstel amplifikasyon ve bu tepki ve kalır kararlı başında bu noktada miktar, ne zaman gerçekleştirildiğini değil reaksiyon verimliliği hala sabit. Daha sonra döngülerle sinyal Plato başlar ve reaksiyon bileşenleri yorgun44olduğundan floresan yoğunluğu artık başlangıç şablonu kopya numarası için ilişkilidir. Doygunluk da DNA substrat moleküllere yeniden tavlama reaksiyonlar, bileşenler veya enzim birimi miktarını değişen konsantrasyon oranları üzerinden rekabet nedeniyle oluşabilir. Belki de, bu tür parametreleri amplifikasyon eğrileri Şekil 4A ve 4Bgösterilen deneyleri için arasındaki farklar için hesap. Dahil denetimleri bu karakteristik amplifikasyon eğrileri oluşturmadı.
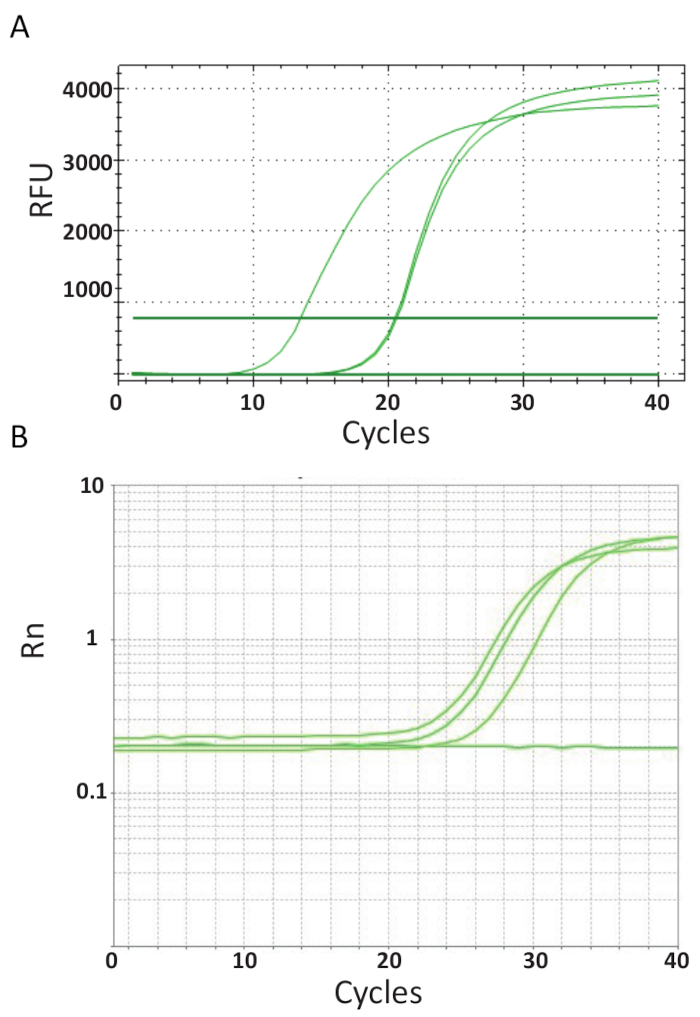
Şekil 4 . Amplifikasyon ürünü birikimi gerçek zamanlı PCR testin süresine göstermek çizer. A. TiLV pozitif örnekleri amplifikasyon eğrileri türetilmiş Tayland, NTCs ve SYBR yeşil ile pozitif plazmid kontrolü ben 2 adım qPCR tahlil. Grafik göreli Floresan (RFU) vs döngüsü numarası komplo tarafından oluşturuldu. B. TiLV olumlu örnekler amplifikasyon eğrileri elde edilen Mısır, Şekil 3B olduğu gibi ve bir NTC. Amplifikasyon eğrisi tahlil (Rn) karşı döngüsü sayısında dahil pasif ROX boya floresan için normalleştirilmiş muhabir sinyal floresan var. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
QPCR thermocycling her laboratuvar farklı makinelerde sonunda, verileri alınan ve analiz. Şekil 5A ve 5B gerçekleştirilen her laboratuvar deneyleri temsilcisi erime eğrileri göster. Her qPCR makine vasıl belgili tanımlık son bir erime eğrisi analizi yapmak için programlanmıştı. Bu artımlı olarak artan sıcaklık ve sıcaklık bir fonksiyonu olarak floresans izleme tarafından sağlanır. Sıcaklık dsDNA denatüre için yüksek olduğunda, fluorophore molekül serbest, çünkü floresans ele kaydedilir. Her qPCR araç yazılım tavlama sıcaklığı (Tm) negatif ilk türev vs sıcaklık (Şekil 5) komplo tarafından erime eğrisi verilerden hesaplanan. O Şekil 5A ve 5B ürünleri farklı örnek kümelerinde oluşan üniforma erime geçiş yaklaşık 80 ° C tahlil için beklenen sıcaklık var görülebilir. Hiçbir diğer zirveleri daha düşük sıcaklıklarda tespit edildi. Kendi küçük boyutu nedeniyle, astar-dimer Tm genellikle hedef DNA dizisi daha düşük. Bu nedenle, Tm'sarasındaki bu fark potansiyel astar-dimer veya diğer non-spesifik amplifikasyon ürünleri tanımlamak kolaylaştırır. Denetimler eritebilir eğrileri TiLV olumlu örnekler ve standartları gibi oluşturmadı ve Şekil 5A ve 5Blistelerinde alt neredeyse yatay bir çizgi olarak görülebilir.
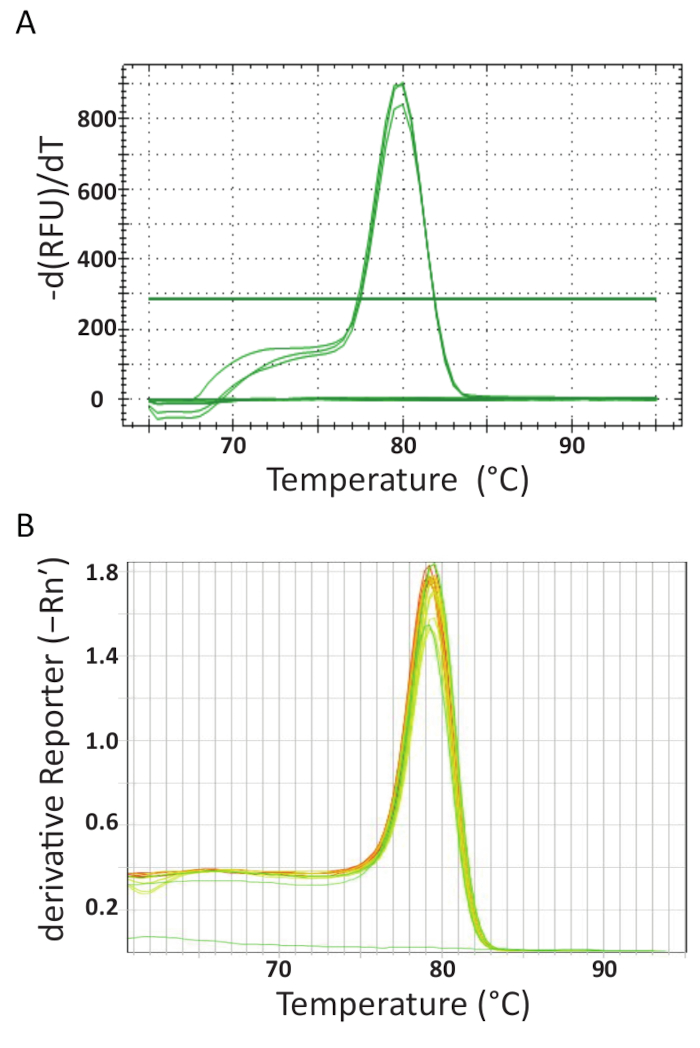
Şekil 5 . Eğri analiz tahlil özgüllük ve farklı PCR ürünleri erime özelliklerine göre farklılaşmış emin olmak için eritebilir. A. eğrisi analiz Tayland, negatif kontrol ve olumlu plazmid denetim'TiLV pozitif örnekleri eritebilir. B. Melt olumlu örnekleri elde edilen Mısır, pTiLV standartları ve bir NTC TiLV eğrisi analizi. İçinde A ve B her ikisi de sıcaklık değişikliği bölü floresans değişiklik gösterir grafikler sıcaklık karşı net bir resim erime dinamiklerinin üretmek için çizilen. Bu rakam daha büyük bir versiyonunu görüntülemek için buraya tıklayınız.
Bir yazılım daha fazla değerlendirme qPCR çalıştırmak ve örnekleri döngüsü eşik (Ct) pTiLV standartları şablon logaritması karşı komplo tarafından otomatik olarak standart bir eğri üreterek ölçmek kolaylaştırılması ile birçok qPCR makinesi gel Şekil 6A ve 6B iki bağımsız laboratuvarlar için gösterildiği gibi numarasını kopyalayın. Kısaca, Ct qPCR sonuçları değerlendirmek için kullanılan birimdir. Ct değeri döngüleri küme eşik floresans sinyal seviyesi ulaşmak için gereken süreyi gösterir. Büyük şablon, az başlangıç miktarını bu tespit floresans seviyeye ulaşmak için alır çevrimleri. Gerçekten, TiLV yüksek bir yük ile örnekleri örnekleri ile TiLV gibi düşük bir yük olarak balık ile bir alt klinik enfeksiyon daha düşük Ct değerleri olacaktır. Ct değerlerini belirlemek için arka plan floresans düzeylerine ilk ham veri düşülür. Daha sonra qPCR aygıtla ilişkili yazılım otomatik floresans eşik her örnek için veri eğrileri arama ve nerede örnek eşik geçti gösteren bir Ct birleşmeyle seçer. Bu ayrı ayrı her tahlil için yapılır ve her eşik dikkatle değerlendirilmelidir, eşik amplifikasyon eğrilerinin ve tüm eğrileri paralel nerede bir yerde Logaritmik bölümünde ayarla sağlanması. Böylece, alınan belirli Ct göreli bir değerdir ve başlangıç şablonu kopyala sayı45göre ancak belirli qPCR makine ve kullanılan reaktifler, PCR güçlendirme verimliliğini ve algılama hassasiyeti için de. Bu parametreler aynı tahlil Şekil 6' kullanma gözlenen farklılıklar katkıda bulunur.
Şekil 6, regresyon analizleri, standart eğriler üzerinden standart eğri yamaçları (m) ve yakaladığını, amplifikasyon verimliliği (100 (101/m -1) x)46 ve doğrusallık tepki de dahil olmak üzere gerçekleştirilen. Standart eğri analizleri de duyarlılık (sınır tespiti), tekrarlanabilirlik ve tekrarlanabilirlik testin onaylamak için kullanılmıştır. Teorik olarak, her PCR ile DNA miktarı iki katına döngüsü, verimlilik (E) % 100'e eşit olduğu anlamına gelir. Ancak, pratikte ideal bir verimlilik nadiren gibi DNA polimeraz inhibisyon, kirleticiler, çok fazla cDNA ve pipetting hatalar47alt-optimal PCR koşulları nedeniyle ulaşılır. 90-%110 %94.5 bir verimlilik süre, tahlil etkinlik 7 seri olarak seyreltilmiş pTiLV kullanarak 6B rakam gösterilen tahlil 8 seri olarak seyreltilmiş pTiLV numuneleri kullanılarak hesaplanmıştır Şekil 6A ' iyi deneyleri için genellikle, amplifikasyon E aralığından örnekleri %101,2 idi. Bir verimlilik % 100'den fazla genellikle PCR inhibitörleri tahlil varlığı nedeniyle var. Doğrusal regresyon analiz standart Arsa da her örnek41,42,45, TiLV kopya sayısını hesaplanması için kırmızı Şekil 6B gösterilen üç TiLV örnek için gözlemlediği gibi sağlar örnekleri S1, S3 ve Şekil 3B' gösterilen S5 için sonuçlar doğrultusunda olduğu.
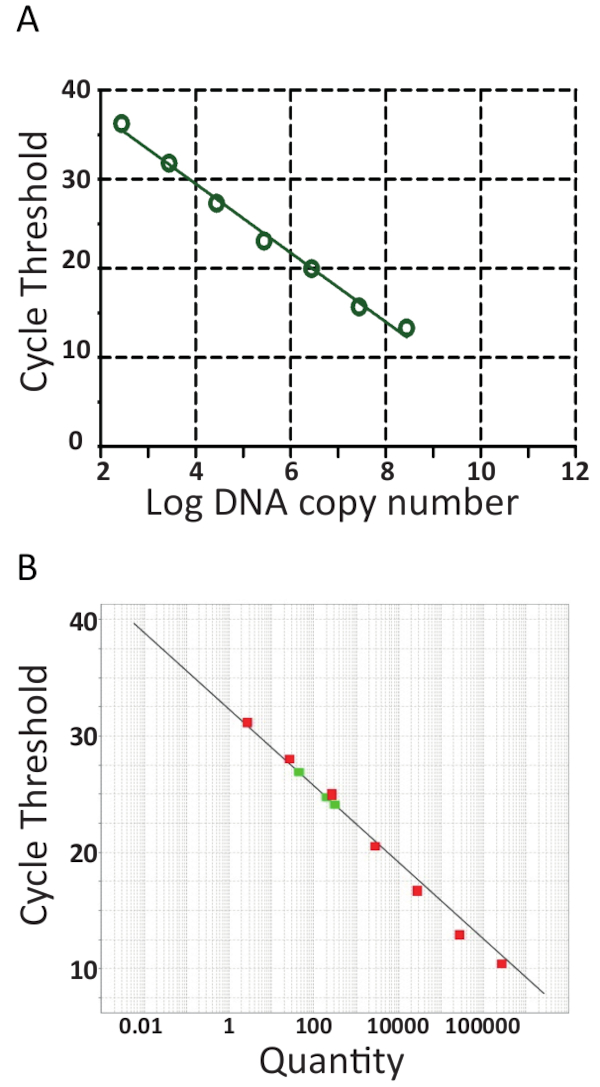
Şekil 6 . RT-qPCR standart eğrileri. Gerçek zamanlı PCR pTiLV, her iki laboratuvarlarda kullanılan standart 10 kat seri dilutions. A. 8 seri olarak seyreltilmiş pTiLV örnekleri test, tüm bilinen konsantrasyon ve TiLV kopya sayısını korelasyon / tepki. Standart eğri günlük kopya numarası vs döngüsü eşik (Ct) komplo tarafından oluşturuldu. Yamaç-3.462, R2 = 0.9992 = ve verimliliği %94.47. B. A olduğu gibi dışında 7 seri olarak seyreltilmiş pTiLV örnekleri (yeşil) test edildi ve y ekseni ve TiLV (miktar) kopya sayısına x ekseni üzerinde eşik döngüsü grafiği görüntüler. Y-kesişim noktası = 32.327, yamaç-3.292, R2 = 0,98 = ve verimliliği %101,2. Her iki standartları eğriler için tahlil performansını anlamak için içinde A ve B, yamaç, y kesişim noktası ve korelasyon katsayısı değerleri (R2) kullanılmaktadır. Önemlisi, standart eğri doğrusallık ölçüsüdür beri R2 değeri 1 yakın olmalıdır. Yamaç neyin-3.32 bir eğimi % 100 verim karşılık gelen PCR verimliliği ölçmek, denklem ve daha fazla bilgi için ana metni görmek için kullanılır. İyi qPCR tepki genellikle-3.58 ve-3.10 arasında bir eğim ile % 90-110 arasında bir verimlilik vardır. Standart eğri bilinmeyen TiLV olumlu örnekleri mutlak miktar için kullanılır ve TiLV kopya tam sayısını belirler / tepki olarak, üç TiLV olumlu örnek için durum renkli kırmızı b
Tartışmalar
TiLV, İsrail-14 2014 yılında ilk rapor ve o zamandan beri Mısır, Kolombiya, Hindistan, Malezya, Uganda, Tanzanya ve Tayland15,16,18, dahil olmak üzere birden çok ülkede saptanmıştır 35 , 48. küresel bilinç, özellikle, tilapia üreten ülkelerde önümüze daha fazla dikkat virüs ve çeşitli sınırlamalar ve kontrol tedbirleri hükümet yetkililerinden TiLV yayılmasını önlemek çalışırken uygulanmıştır. Burada, TiLV algılama tilapia doku, kapsayan örnek koleksiyon, RNA izolasyon, cDNA sentez, PCR ve qPCR deneyleri için detaylı bir protokol ifade edildi. Belirli tartışma garanti bu yöntemler için çeşitli yönleri vardır. TiLV çeşitli boyutları9,12,14,15,49 ve türler çiftlik hibrid tilapia (O. dahil olmak üzere tilapia defa, balıkta olduğu belirlendi niloticus x O. aureus)11,14, Nil tilapia (O. niloticus)9,10,14,15,16, 33 , 35 , 36 , 49 , 50 ve kırmızı tilapia (de vahşi Nil tilapia9olduğu gibi,12, siyah gibiOreochromis sp.)16,33,48,51, tilapia51, T. zilli14,15, S. galilaeus, O. aureus ve T. simonis intermedia14 ve son zamanlarda TiLV vahşi Sazan (Barbonymus tespit yapıldı. schwanenfeldii)52. Doku örnekleri iç organlar (gill, dalak, karaciğer, kalp, baş böbrek) veya mukus37 yaş, boyutu veya türü ne olursa olsun sağlıklı hem de can çekişen tilapia toplanan ve RNA izolasyon için işlenir. Özetlenen toplam RNA ayıklama iletişim kuralı burada bir chaotropic denaturing Ajan fenol ve guanidinium thiocyanate bir monophasic çözüm kullanır. Dokuları doğrudan kloroform ve faz ayrılık neyin üst sulu faz, bir interfaz ve daha düşük bir organik faz içeren açık bir RNA oluşturulur ulaşmak için Santrifüjü eklenmesi tarafından takip bu çözümde homojenize. RNA sulu faz isopropanol yağış, kirleticiler kurtulmak için kurtarılan RNA yıkayarak takip tarafından izole edilmiştir. RNA izolasyon Bu metodoloji tarafından Piotr Chomczynski ve Nicoletta Sacchi tarafından öncülük ve için guanidinium thiocyanate-fenol-kloroform ayıklama53,54anılıyordu. Bu tür bir RNA çıkarma için kullanılan reaktif olabilir ticari olarak satın alınan veya laboratuvarda yapılan ( Tablo malzeme daha fazla bilgi için bkz:). Bu iletişim kuralı gibi silika tabanlı arıtma yöntemleri sütunlara göre biraz uzun sürer, ama genel olarak, daha uygun maliyetli ve daha fazla RNA verir.
Sayede RNA kalite spectrophotometry değerleri belirtebilirsiniz bu protokol için A260 değerleri kullanarak RNA miktarının özetlenen (A260/A280 = 1,9-2.1). Bu yöntem iyi bir gösterge örnek saflık verirken, kesinlikle ayıklanan RNA kalitesi hakkında bilgi veremem. Düzgün RNA bozulmamış veya kısmen bozulmuş olup olmadığını belirlemek için örnekleri ayırma tarafından özel jel elektroforez neyin EtBr bulaşması 18S lekeli olabilir ve 28S rRNA bantları RNA Bozulması gösterir. Daha fazla doğrulama RNA kalite laboratuvar-on-a-chip enstrüman kullanarak içerebilir. Ayrıca, aynı zamanda olduğunu DNaz ile arıtılmış RNA sindirmek önemli ben kirletici kaldırmak için hangi yanlış sonuçlara neden olabilir bağlı olarak aşağı akım uygulamaları genomik DNA, ev sahibi. Ana bilgisayar gDNA hala büyük ölçüde RNA örnek bulaşıcı, ek bir DNaseI tedavi de RNA ayıklama yordamın bitiminde yapılabilir ( Tablo malzemelerigörmek).
Tamamlayıcı DNA sentezi genel qPCR sonuçları büyük ölçüde etkileyebilir ve varyasyon tanıtabilirsiniz yöntemi bir yönüdür. Burada savunduğu cDNA protokolü bir tek bileşen up oluşur ve böylece yalnızca transkripsiyonun polyA kuyrukları içeren mRNA oligo (dT) kullanarak. Tam olarak hangi bileşenlerinin Ters transkripsiyon tepki ve cDNA bu modu kullanmak için sentez TiLV algılama32için başarılı kanıtlamıştır Kullanıcı denetimi sağlar. Bu kurulum için bir alternatif master-mix içeren bir ticari satın Ters transkripsiyon reaksiyon için gerekli tüm bileşenleri ve çok hızlı ve her zamankinden çok adımlı, pipetting ve çok sıcaklık protokolü olmadan basit. Çünkü o işleme en aza indirir ve tekdüzelik tüm örnekler üzerindeki teşvik avantajlıdır. Böyle ana karışımları genellikle oligo(dT) ve uygulanabilir farklı RNA şablonlar yapma ve tüm uzunluğu boyunca bir popülasyondaki RNA'ların üzerinden dizileri kopyalarını temsilcisi cDNA üreten rasgele astar içerir (viral ve mRNA ev sahipliği tilapia) ve teorik olarak, istenen her RNA tür sonra geleneksel PCR ya da qPCR böyle bir örnek üzerinden tarafından ölçülebilir. Bu çok yönlü bir 2-adım RT-PCR yaklaşım büyük avantajı olduğunu; birçok farklı deneyler için kullanılan uzun vadeli bir havuz sağlar. Sonuçlarda, bir tek adımlı RT-PCR yaklaşım temsil neyin sıra belirli astar (Tablo 1) kullanılmıştır ve RT ve PCR bir tüpte yapıldı (malzeme listeye bakın). Genel olarak, rasgele macun kullanarak daha yüksek bir RT verimlilik belirli hedef RNA için sıra belirli astar izin ama belirli hedef RNA (bkz: bazı laboratuvarlar tek amacı olabilir böyle bir cDNA örnek sayılabilir tek kişi Tablo malzemelerin cDNA sentez ürün öneriler için).
Yaygın olarak kullanılan geleneksel RT-PCR görünür iken TiLV tanı9,13,14,15,16,17,18, içinde defa 33 , 35 , 48 , 55. RT-qPCR algılama ve TiLV az miktarda balık dokularda veya mukus32,37miktar için daha güçlü bir araç olarak gösterilir. Genel olarak, qPCR yüksek duyarlılık, özgüllük, iyi tekrarlanabilirlik, geniş dinamik alan ve hız21klinik Viroloji teşhis laboratuvarlarında yaygın olarak kullanılır. QPCR geleneksel RT-PCR uygulamak başlangıçta daha pahalı olsa da, geleneksel PCR birçok önemli avantajlar sunuyor; bir daha hızlı teslimi etrafında zaman örnek sonuçları vardır ve bu herhangi bir post-PCR adımları gerektirmez. Bu ikinci noktada laboratuvar kirlenme için en az risk ve salgınlar halinde gibi yüksek üretilen iş durumlara daha kolay adapte edilebilir anlamına gelir. Ayrıca, doğal olarak daha geleneksel RT-PCR, hangi alt klinik enfeksiyonlar21' düşük viral yük algılamak için hayati önem taşımaktadır daha duyarlı olduğunu. Bu ters transkripsiyon gerektiren bir iç içe geçmiş PCR yaklaşım gerektirir, iki daha fazla PCR reaksiyonları ve sonra özel göre analiz Elektroforez jel. Birçok adımları almak biraz çok-in zaman ve hataları veya bulaşma şansını artırır. Yine de, yüksek hassasiyeti nedeniyle RT-qPCR titiz deneysel tasarım ve kesin sonuçlar56,57üretmek için miktar teknikleri kapsamlı bir anlayış gerektirir.
DNA bağlama fluorophore, SYBR yeşil bu protokol için göstermiştir. Bu bir dsDNA belirsiz DNA bağlama boya ve böylece tamamen yanlış pozitif58oluşturabilir astar kümesinde testin özgüllüğü yatıyor. Bu nedenle, her PCR sonunda gerçekleştirilen eğrisi Analizi erime dsDNA PCR reaksiyon özellikle önemli bir parçası iken o sadece bir PCR amplicon doğru t onaylar çünküm elde edilir (Bu da jel tarafından elde edilmelidir Elektroforez) ne zaman yeni deneyleri uygulanıyor. Bir DNA parçasının Tm a değişiklik-in şekil öyle aynı derecede onun uzunluğu, GC kompozisyon, sıra, strand tamamlayıcılık bağlıdır, konsantrasyon de kadar üzerinde arabellek bileşenleri ve PCR arttırıcılar. İki laboratuar astar-dimer veya diğer istenmeyen PCR ürünleri varlığını ortaya değil ama bu diğer örnekleri ve/veya deneysel set-up ile görülmektedir, sonra tahlil olmalıdır temsilcisi sonuçlar içinde eriyen eğrisi analizleri yeniden en iyi duruma getirilmiş. Daha gelişmiş qPCR teknolojileri erime eğrisi adım gerektirmeyen ve gerçekten de, kağıt yazılmıştır, bu yöntemleri beri dayalı bir TaqMan TiLV RT-qPCR iki astar ve son derece TiLV belirli34yapma bir sonda kullanarak geliştirilmiştir.
Hiç şüphesiz, RT-qPCR deneyleri için tasarlanmış astar tahlil başarısı için temel ve astar burada halk için elde edilebilir TiLV genomik veri saat32bağlı tasarlanmıştır. Ancak, RNA virüsleri yüksek mutasyon oranları sergilemek için iyi bilinen ve olası suşları geçerli tanı sınamaları, ISAV59için gözlendi gibi kaçış olacak. Her zaman böyle virüs türleri için bir evrensel pan-TiLV RT oluşturmak zor olacak-qPCR tahlil ve bu tür deneyleri sadece sürekli geliştirilmiş olabilir geniş kapsamlı mekanlar ve süreler daha fazla TiLV genomik veri yayımlanırsa.
Son olarak, yinelenen çalıştırmak veya mümkünse, her iki içi tepkileri onaylatılacak ve qPCR deneyleri Inter esastır. Ct değerleri çok yüksek ise, o zaman çoğaltır kullanımı PCR reaksiyon güvenilir ve tekrarlanabilir olduğunu tespit etmek özellikle önemlidir. Genel olarak, verileri çoğaltmak tepkiler değişir birden fazla 0,5 döngüleri, tepkiler yinelenmelidir ve Ct değerleri sürekli olarak > 0,5 döngülerle farklılık gösteriyorsa çoğaltır, tahlil yeniden optimize edilmelidir. Bir entegre qPCR pipetting robot kullanımı son derece bu konuda yardımcı olur, ama bu bir lüks araç. Olarak özellikle tanı laboratuvarları akredite olmak için bu tür deneyleri olduğu içinde sağlam moleküler deneyleri gelişimi için son derece önemlidir dahil yeterli ve uygun denetimleri ile herhangi bir deneme, vardır. Olumlu (pozitif TiLV örnek, TiLV plazmid standart) ve negatif denetimleri (NTC ve -RT) örnekleri gibi endojen tilapia temizlik genlerin tespiti denetimleri eklemeniz gerekir. Bu tür denetimler göz ardı edemez ve her tahlil düzgün tahlil her adımında kalitesini anlamak ve düzgün sonuçlar yorumlamak için dahil edilmelidir.
Açıklamalar
Yazarlar ifşa gerek yok.
Teşekkürler
Verdikleri destek için veteriner Bakteriyolojinin Enstitüsü, Vetsuisse Fakültesi, Bern Üniversitesi için sana şükrediyoruz. Bu eser PN için layık, erken kariyer araştırmacı akademik yükselme ve cinsiyet eşitliği modeli %120 fon tarafından Bern Üniversitesi Vetsuisse Fakültesi Komitesi tarafından finanse edildi. WS ve Halkla İlişkiler Merkezi tarafından ileri araştırmalar için desteklenen tarım ve gıda, Institute for Advanced Studies, Kasetsart Üniversitesi, Bangkok, Tayland altında Yükseköğretim araştırma promosyon ve Ulusal Araştırma Üniversitesi proje Tayland, Office için Yüksek öğretim Komisyonu, Milli Eğitim Bakanlığı, Tayland. Onun anlatım için Dr Kwanrawee Sirikanchana ve Piyawatchara Sikarin düzenleme video için teşekkür etmek istiyorum.
Malzemeler
| Name | Company | Catalog Number | Comments |
| Tissue collection | Step 1 | ||
| Tricaine methanesulfonate | Sigma-Aldrich | E10521 | An alternative to clove oil. Step 1.1 |
| RNAlater stabilization solution | Thermo Fisher Scientific | AM7020 | For storing tissues if they cannot be processed immediately Step 1.3 |
| RNA extraction | Step 2 | ||
| TRIreagent | Sigma-Aldrich | Step 2.1 | |
| TRIzol | Thermo Fisher Scientific (Invitrogen) | 15596026 | Step 2.1 |
| GENEzol | Geneaid | GZR100 | Step 2.1 |
| Trisure | Bioline | BIO-38032 | Step 2.1 |
| Homemade solution | - | - | 94.53 g/L (800 mM) guanidine thiocyanate 30.45 g/L (400 mM) ammonium thiocyanate 8.20 g/L (100 mM) sodium acetate 380 mL/L (38 % v/v) phenol 50 mL/L (5 % v/v) glycerol 1.0 g/L (0.1 % w/v) 8-quinolinol, pH 5.0 Store up to 2 years at 4oC Step 2.1 |
| MagNA Lyser Green Beads | Roche | 3358941001 | An alternative tissue homogenization method used in conjunction with tissue lysing machines detailed below Step 2.2 |
| Lysing Matrix D, 2 mL Tube | MP BIOMEDICALS | 116913050 | |
| Chloroform | Sigma-Aldrich | C2432 | Step 2.3 |
| Chloroform | RCI Labscan | AR1027E-G2.5L | Step 2.3 |
| 1-Bromo-3-chloropropane | Sigma-Aldrich | B9673 | A less toxic alternative to chloroform Step 2.3 |
| Isopropanol (GC) ≥ 99.8 % | Sigma-Aldrich | 59300 | Step 2.6 |
| Isopropanol (ACS, ISO Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.09634.2500 | Step 2.6 |
| Glycogen, molecular biology grade (e.g., Sigma, cat. no. G1767) | Thermo Fisher Scientific (Thermo Scientific) | R0551 | Useful step if tissue starting material is small to maximise RNA precipitation optional |
| Ethanol (purity (GC) ≥ 99.9 % | Sigma-Aldrich (EMD Millipore) | 1.00983 | Step 2.9 |
| Ethanol (ACS, ISO Reag. Ph Eur) | Merck (EMSURE) | 1.00983.2500 | Step 2.9 |
| Nuclease-free water | Promega | P1193 | Step 2.13 |
| Nuclease-free water | Multicell | 809-115-CL | Step 2.13 |
| Ambion TURBO DNA-free kit | Thermo Fisher Scientific (Invitrogen) | AM1907 | Can be performed at the end of the RNA extraction protocol optional |
| cDNA synthesis | Step 4 | ||
| Viva cDNA Synthesis Kit | Vivantis | cDSK01 | Step 4.1 & 4.3 |
| ReverTra Ace qPCR RT MasterMix with gDNA remover | Toyobo | A1172K | An alternative option see discussion |
| ReverTra Ace qPCR RT Kit | Toyobo | FSQ-101 | An alternative option see discussion |
| AffinityScript Multiple Temperature Reverse Transcriptase | Agilent Technologies | 600107 | An alternative option |
| PCR | Step 5 | ||
| DNA polymerase systems: | Step 5.2 | ||
| - Platinum II Hot-Start Green PCR Master Mix (2X) | Thermo Fisher Scientific (Invitrogen) | 14001012a | Step 5.2 |
| - GoTaq Mastermix | Promega | M7122 | Step 5.2 |
| Separate PCR mixture components: | Step 5.2 | ||
| 10mM dNTP Mix | Vivantis | NP2409 | Step 5.2 |
| 25mM MgCl2 | Thermo Fisher Scientific | R0971 | Step 5.2 |
| 10X Taq Buffer with KCl | Thermo Fisher Scientific | 00348114 | Step 5.2 |
| Taq DNA polymerase | Vivantis | PL1202 | Step 5.2 |
| - Verso 1-step RT-PCR ReddyMix with ThermoPrime Taq | Thermo Fisher Scientific | AB1454 | One step RT-PCR exemplified in Figure 3B |
| Gel electrophoresis: | For visulation of PCR products from steps 5.1-5.4 | ||
| Ethidium Bromide solution (10 mg/mL) | Thermo Fisher Scientific | 17898 | Step 5.5 |
| Tris/Acetic/EDTA (TAE) buffer: | Step 5.5 | ||
| - Tris | Vivantis | PR0612-1KG | Step 5.5 |
| - Acetic acid (glacial) (ACS, ISO, Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.00063.2500 | Step 5.5 |
| - Ethylenediaminetetraacetic acid (EDTA) | BIO-RAD | 161-0729 | Step 5.5 |
| Agarose | Vivantis | PC0701-100G | Step 5.5 |
| DNA ladders and markers | Vivantis | NL1405 | Step 5.5 |
| DNA gel loading dye (6X) | Thermo Fisher Scientific | R0611 | Step 5.5 |
| qPCR | Step 6 | ||
| PowerUP SYBR Green Master Mix | Thermo Fisher Scientific (Applied Biosystems) | A25779 | Exemplified in Figures 4-6B Step 6.2 |
| iTaq Universal SYBR Green Supermix | BIO-RAD | 1725120 | Exemplified in the video and in Figures 4-6A Step 6.2 |
| Equipment | |||
| Dounce tissue grinder pestle | Sigma-Aldrich | P1110 | Protocol 2 |
| MagNA Lyser Instrument | Roche | 3358976001 | An alternative tissue homogenizing option for protocol 2 which are used in conjunction with the lysing beads detailed above Step 2.2 |
| FastPrep-24 5G Homogenizer | MP BIOMEDICALS | 116005500 | |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5427R | Protocol 2 Step 2.4, 2.7 & 2.10 |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5418R | |
| Heat box | Labnet | AccuBlock Digital Dry Bath | Protocol 2 Step 2.13 |
| Microvolume spectrophotometer | Thermo Fisher Scientific (Applied Biosystems) | Nanodrop 2000 | Protocol 3 Step 3.1 - 3.4 |
| PCR machine | BIO-RAD | T100 Thermal Cycler | Protocol 5 Step 5.4 |
| Power supply | BIO-RAD | PowerPac HC | Protocol 5 Step 5.5 |
| Horizontal gel electrophoresis | BIO-RAD | Mini ReadySub-Cell GT Cell #1704487edu | Protocol 5 Step 5.5 |
| Mini microcentrifuge | Corning | LSE 6766 | Useful to quickly spin down PCR reaction tubes in protocols 4, 5 & 6 Step 6.5.1 |
| Microcentrifuge | LioFuge | LM-60 | Step 6.5.1 |
| qPCR machine and software | Thermo Fisher Scientific | 7500 Fast Real-Time PCR System with 7500 Software v2.0 | Protocol 6 Step 6.6-6.8 |
| qPCR machine and software | BIO-RAD | CFX96 Touch Real-Time PCR Detection System with CFX Manager software | |
| General Materials | |||
| Mayo scissors | Step 1.1-1.2 | ||
| Forceps | Step 1.1-1.2 | ||
| Pipette | Rainin | Pipette-Lite XLS | |
| Aerosol-barrier pipette tips | Sigma-Aldrich | Z333328, Z333336, Z333344 | |
| Nuclease-free 1.5-ml microcentrifuge tubes | Eppendorf |
Referanslar
- FAO. . The State of World Fisheries and Aquaculture, 2014. Opportunities and Challenges. , (2014).
- FAO. . The State of World Fisheries and Aquaculture, 2016. Contributing to Food Security and Nutrition for all. , (2016).
- WorldBank. . FISH TO 2030: Prospects for Fisheries and Aquaculture. Agriculture and Environmental Services Discussion Paper 03. , (2013).
- Wing-Keong, N., Nicholas, R. A review of the nutrition and feeding management of farmed tilapia throughout the culture cycle. Reviews in Aquaculture. 5 (4), 220-254 (2013).
- Cleasby, N., et al. The socio-economic context for improving food security through land based aquaculture in Solomon Islands: A peri-urban case study. Marine Policy. 45, 89-97 (2014).
- Ponzoni Raul, W., et al. Genetic improvement of Nile tilapia (Oreochromis niloticus) with special reference to the work conducted by the WorldFish Center with the GIFT strain. Reviews in Aquaculture. 3 (1), 27-41 (2011).
- Hounmanou, Y. M. G., et al. Tilapia lake virus threatens tilapiines farming and food security: Socio-economic challenges and preventive measures in Sub-Saharan Africa. Aquaculture (Amsterdam, Netherlands). 493, 123-129 (2018).
- OIE. . Tilapia Lake Virus (TiLV) - a novel orthomyxo-like virus. OIE technical disease cards. , (2018).
- Mugimba, K. K., et al. Detection of tilapia lake virus (TiLV) infection by PCR in farmed and wild Nile tilapia (Oreochromis niloticus) from Lake Victoria. Journal of Fish Diseases. , (2018).
- Koesharyani, I., Gardenia, L., Widowati, Z., Khumaira, D. D., Rustianti, Studi kasus infeksi tilapia lake virus (tilv) pada ikan nila (Oreochromis niloticus). Jurnal Riset Akuakultur. 13 (1), 85-92 (2018).
- OIE. . Tilapia lake virus disease (TiLV), Chinese Taipei. Immediate Notification. , (2017).
- OIE. . Tilapia Lake Virus Disease (TiLV), Peru. Immediate Notification. , (2018).
- Bacharach, E., et al. Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia. MBio. 7 (2), e00431-e00416 (2016).
- Eyngor, M., et al. Identification of a novel RNA virus lethal to tilapia. Journal of Clinical Microbiology. 52 (12), 4137-4146 (2014).
- Nicholson, P., et al. Detection of Tilapia Lake Virus in Egyptian fish farms experiencing high mortalities in 2015. Journal of Fish Diseases. 40 (12), 1925-1928 (2017).
- Surachetpong, W., et al. Outbreaks of Tilapia Lake Virus Infection, Thailand, 2015-2016. Emerging Infectious Diseases. 23 (6), 1031-1033 (2017).
- Tattiyapong, P., Dachavichitlead, W., Surachetpong, W. Experimental infection of Tilapia Lake Virus (TiLV) in Nile tilapia (Oreochromis niloticus) and red tilapia (Oreochromis spp.). Veterinary Microbiology. 207, 170-177 (2017).
- Kembou Tsofack, J. E., et al. Detection of Tilapia Lake Virus in Clinical Samples by Culturing and Nested Reverse Transcription-PCR. Journal of Clinical Microbiology. 55 (3), 759-767 (2017).
- Thangaraj, R. S., et al. Derivation of two tilapia (Oreochromis niloticus) cell lines for efficient propagation of Tilapia Lake Virus (TiLV). Aquaculture (Amsterdam, Netherlands). 492, 206-214 (2018).
- Hanson, L. A., Rudis, M. R., Vasquez-Lee, M., Montgomery, R. D. A broadly applicable method to characterize large DNA viruses and adenoviruses based on the DNA polymerase gene. Virology Journal. 3, 28-28 (2006).
- Josko, D. Molecular virology in the clinical laboratory. Clinical Laboratory Science. 23 (4), 231-236 (2010).
- Munir, K., Kibenge, F. S. Detection of infectious salmon anaemia virus by real-time RT-PCR. Journal of Virological Methods. 117 (1), 37-47 (2004).
- Snow, M., et al. Developement, application and validation of a Taqman real-time RT-PCR assay for the detection of infectious salmon anaemia virus (ISAV) in Atlantic salmon (Salmo salar). Developments in Biologicals. 126, 133-145 (2006).
- Matejusova, I., McKay, P., McBeath, A. J., Collet, B., Snow, M. Development of a sensitive and controlled real-time RT-PCR assay for viral haemorrhagic septicaemia virus (VHSV) in marine salmonid aquaculture. Diseases of Aquatic Organisms. 80 (2), 137-144 (2008).
- Garver, K. A., et al. Development and validation of a reverse transcription quantitative PCR for universal detection of viral hemorrhagic septicemia virus. Diseases of Aquatic Organisms. 95 (2), 97-112 (2011).
- Dalla Valle, L., et al. Development of a sensitive and quantitative diagnostic assay for fish nervous necrosis virus based on two-target real-time PCR. Veterinary Microbiology. 110 (3-4), 167-179 (2005).
- Hodneland, K., Garcia, R., Balbuena, J. A., Zarza, C., Fouz, B. Real-time RT-PCR detection of betanodavirus in naturally and experimentally infected fish from Spain. Journal of Fish Diseases. 34 (3), 189-202 (2011).
- Hodneland, K., Endresen, C. Sensitive and specific detection of Salmonid alphavirus using real-time PCR (TaqMan). Journal of Virological Methods. 131 (2), 184-192 (2006).
- Wang, X. W., Ao, J. Q., Li, Q. G., Chen, X. H. Quantitative detection of a marine fish iridovirus isolated from large yellow croaker, Pseudosciaena crocea, using a molecular beacon. Journal of Virological Methods. 133 (1), 76-81 (2006).
- van Beurden, S. J., et al. Development and validation of a real-time PCR assay for the detection of anguillid herpesvirus 1. Journal of Fish Diseases. 39 (1), 95-104 (2016).
- Ciulli, S., et al. Development and application of a real-time PCR assay for the detection and quantitation of lymphocystis disease virus. Journal of Virological Methods. 213, 164-173 (2015).
- Tattiyapong, P., Sirikanchana, K., Surachetpong, W. Development and validation of a reverse transcription quantitative polymerase chain reaction for tilapia lake virus detection in clinical samples and experimentally challenged fish. Journal of Fish Diseases. 41 (2), 255-261 (2018).
- Dong, H. T., et al. Emergence of tilapia lake virus in Thailand and an alternative semi-nested RT-PCR for detection. Aquaculture (Amsterdam, Netherlands). 476, 111-118 (2017).
- Waiyamitra, P., et al. A TaqMan RT-qPCR assay for tilapia lake virus (TiLV) detection in tilapia. Aquaculture (Amsterdam, Netherlands). 497, 184-188 (2018).
- Behera, B. K., et al. Emergence of Tilapia Lake Virus associated with mortalities of farmed Nile Tilapia Oreochromis niloticus (Linnaeus 1758) in India. Aquaculture (Amsterdam, Netherlands). 484, 168-174 (2018).
- Ferguson, H. W., et al. Syncytial hepatitis of farmed tilapia, Oreochromis niloticus (L.): a case report. Journal of Fish Diseases. 37 (6), 583-589 (2014).
- Liamnimitr, P., Thammatorn, W., U-thoomporn, S., Tattiyapong, P., Surachetpong, W. Non-lethal sampling for Tilapia Lake Virus detection by RT-qPCR and cell culture. Aquaculture (Amsterdam, Netherlands). 486, 75-80 (2018).
- Yang, C. G., et al. Evaluation of reference genes for quantitative real-time RT-PCR analysis of gene expression in Nile tilapia (Oreochromis niloticus). Gene. 527 (1), 183-192 (2013).
- Bustin, S. A. Real-time, fluorescence-based quantitative PCR: a snapshot of current procedures and preferences. Expert Review of Molecular Diagnostics. 5 (4), 493-498 (2005).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27 (2-3), 126-139 (2006).
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27 (2-3), 95-125 (2006).
- Mackay, I. M., Arden, K. E., Nitsche, A. Real-time PCR in virology. Nucleic Acids Research. 30 (6), 1292-1305 (2002).
- Wong, M. L., Medrano, J. F. Real-time PCR for mRNA quantitation. Biotechniques. 39 (1), 75-85 (2005).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. Journal of Molecular Endocrinology. 25 (2), 169-193 (2000).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Research. 6 (10), 986-994 (1996).
- Rutledge, R. G., Côté, C. Mathematics of quantitative kinetic PCR and the application of standard curves. Nucleic Acids Research. 31 (16), e93-e93 (2003).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Amal, M. N. A., et al. A case of natural co-infection of Tilapia Lake Virus and Aeromonas veronii in a Malaysian red hybrid tilapia (Oreochromis niloticus × O. mossambicus) farm experiencing high mortality. Aquaculture (Amsterdam, Netherlands). 485, 12-16 (2018).
- Fathi, M., et al. Identification of Tilapia Lake Virus in Egypt in Nile tilapia affected by ‘summer mortality’ syndrome. Aquaculture (Amsterdam, Netherlands). 473, 430-432 (2017).
- OIE. . Tilapia Lake Virus disease (TiLV), Philippines. Immediate Notification. , (2017).
- OIE. . Tilapia lake virus disease (TiLV), Malaysia. Immediate Notification. , (2017).
- Abdullah, A., et al. First detection of tilapia lake virus (TiLV) in wild river carp (Barbonymus schwanenfeldii) at Timah Tasoh Lake, Malaysia. Journal of Fish Diseases. 41 (9), 1459-1462 (2018).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Del-Pozo, J., et al. Syncytial Hepatitis of Tilapia ( Oreochromis niloticus L.) is Associated With Orthomyxovirus-Like Virions in Hepatocytes. Veterinary Pathology. 54 (1), 164-170 (2017).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Purcell, M. K., Getchell, R. G., McClure, C. A., Garver, K. A. Quantitative polymerase chain reaction (PCR) for detection of aquatic animal pathogens in a diagnostic laboratory setting. Journal of Aquatic Animal Health. 23 (3), 148-161 (2011).
- Simpson, D. A., Feeney, S., Boyle, C., Stitt, A. W. Retinal VEGF mRNA measured by SYBR green I fluorescence: A versatile approach to quantitative PCR. Molecular Vision. 6, 178-183 (2000).
- Kibenge, M. J., et al. Discovery of variant infectious salmon anaemia virus (ISAV) of European genotype in British Columbia, Canada. Virology Journal. 13, 3 (2016).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır