Method Article
ティラピア湖ウイルスを使用して従来 RT-PCR および SYBR の検出グリーン Rt-qpcr
要約
このプロトコルは、RT-PCR の方法論を利用したティラピア生体ティラピア湖ウイルス (TiLV) を診断します。全体の方法は、総 RNA の抽出、続いて cDNA を合成して、従来の PCR またはバインディングの蛍光色素を結合 dsDNA を使用して量的な PCR によって TiLV の検出に組織郭清から記載されます。
要約
このメソッドの目的は、ティラピア組織におけるティラピア湖ウイルス (TiLV) の高速、高感度および特定の検出を容易にすることです。このプロトコルは、TiLV 基礎研究所監視プログラム、バイオ セキュリティ対策の一部として使用することができます。ウイルス診断法のゴールド スタンダードは普通逆転写ポリメラーゼ連鎖反応 (RT-PCR) さらに詳しく調べるなどの補完的な手法によって続いてウイルス分離を含みます。これは面倒で時間のかかることができます、通常大きくウイルス感染組織サンプルが必要です。RT 定量的 (q) ウイルスの検出に PCR の使用は、その計量的性質、高い感度、特異性、スケーラビリティおよびその急速な時間が発生するため便利です。ここでは、PCR 法の全体のアプローチ TiLV 検出が記載、guanidium チオシアン酸-フェノール-クロロホルム ソリューション、2 段階 PCR によって続いた RNA 定量を用いたティラピア オルガン断面、合計リボ核酸 (RNA) 抽出私はプロトコルを伴う、相補的デオキシリボ核酸 (cDNA) 合成と従来の PCR または量的識別を介してqPCR SYBR の緑を使用してのいずれかによって TiLV の検出を染めます。従来の PCR はポスト PCR のステップを必要とし、単にウイルスの存在を知らせます。後者のアプローチとして 2 枚まで TiLV の絶対定量が可能となる TiLV サブ臨床診断に非常に役立ちます.2 つの PCR 方法、2 つの研究室からの代表的な結果の詳細な説明と徹底した議論両方の重要なパラメーターの研究者とにとって、最も適していると適用を見つけることを確認する含まれていますTiLV 検出の方法です。
概要
2014 年に 20 キロの新記録に達した世界の一人当たりの魚の供給、これは養殖漁業の活発な成長によるものです。養殖は最も成長が著しい動物食糧生産のセクターを世界の一つで、人口1よりも速く成長している唯一の動物食品製造部門。Tilapiine cichilds 640 万トン (MT) の総世界生産 2015年298 億米ドルの推定値と世界的養殖 2 番目の最も重要な淡水魚で構成されます。ティラピアのトップ 10 の生産者が中国 (1.78 MT)、インドネシア (1.12 MT)、エジプト (0.88 MT)、バングラデシュ、ベトナム、フィリピン、ブラジル、タイ、コロンビア、ウガンダ2の順。グローバル ティラピアの生産が約 7.3 になると予想は 20303MT。彼らは水と気候条件5,6の広い範囲の下で容量で繁殖しやすいのでも蛋白質4の安価なソースを彼らだけでなく、ティラピアのような重要な世界的な食糧源となっています。わずか数十年前に、ティラピア農業を脅すいくつかの商業的に重要な疾患があったが、これはもはや真実を信じられていた。最初の重大な病気の流行は、ティラピアと業界全体がリスクではティラピア湖ウイルス病 (TiLVD) と呼ばれる新興ウイルス性疾患です。この病気は深刻な社会経済的結果を持ち、アフリカ7アジア、南アメリカの人々 の何百万のための食料安全保障に関する直接の脅威です。2018 の開始、健康局 (Oie) 報告したこの病原体情報カードだったので、3 つの大陸をカバーする 8 つの国8 TiLV、この病気の病因が正式に検出されそこ更新と、ペルー12台湾11インドネシア10ウガンダ9タンザニアの TiLV のより多くの報告がされています。TiLV は小説の一本鎖 RNA ウイルスはインフルエンザや伝染性サケ貧血ウイルス (ISAV)13など他の orthomyoxoviruses を連想させる特徴の様々 なが含まれているために、orthomyxo のようなウイルスをあるために記述します。それは、ガリラヤ湖、イスラエル共和国の14の野生と養殖のティラピアの巨額の損失の余波で初めて確認されました。夏死亡とナイル川のテラピア (試行)15エジプトのナイル川と赤いハイブリッド ティラピア TiLV 感染症に関連付けられている死亡症候群症例数 1 ヶ月その後、似たような病気の発生が指す(外部属)タイ16、それぞれ。水生動物ウイルスの検出歴史的に成長と細胞培養におけるウイルスの分離によって実行されます。各種細胞株が伝播し、E-11 細胞に由来する蛇頭魚 (Ophiocephalus 柄)17,18, OmB と TmB外部から発信されたを含む TiLV の分離テストされています。mossambicus18, と OnlB と OnlL ナイル ティラピア (O. niloticus)19発します。ウイルス培養には、さらに実験のための材料を提供することの利点がありますが、それは不利な点、それは細胞変性効果 (CPE) の形成を観察する少なくとも 4-7 日を必要とし、重大に、ヒッパルコスの異なるウイルスにより適合していることレプリケートが伝播し、同様 CPE を生成します。
過去数十年では細胞培養、血清と抗原検出ベースより速くより敏感の核酸検出テスト20、交換など、時間のかかる伝統的な診断法から移動されています。 21。これは、多くの qPCR アッセイは、ISAV22,23, ウイルスの出血性敗血症のウイルス (VHSV)24 のように多数の水生動物のウイルスの病気の重要な診断法として開発されているという事実によって明らか ,25, ベータノダ ウイルス26,27サケ科アルファウイルス28, 魚イリド29、Anguillid ヘルペス ウイルス 1 (AngHV1)30、およびリンホシスチス病病ウイルス (LCDV)31.診断と病原体サーベイランスのための強力なメソッドは緊急 TiLV の広がりを減らす必要があります。このような方法はように臨床徴候を開発前に感染症の早期発見と低ウイルス負荷の検出です。TiLV の検出のためまで、RT-PCR14,32, を含む別の PCR のプロトコル開発されている、入れ子になった RT-PCR18、半入れ子になった RT-PCR33、および RT qPCR32,34魚の体内に。TiLV 検出の感受性細胞における RT qPCR およびウイルス分離の比較は、RT qPCR はウイルス分離32よりも 1,000 倍以上敏感ことをわかった。各パブリッシュされた PCR のプロトコルは、TiLV の検出のための異なった感受性を報告が、ほとんどの試金が 7.5 コピー33、7 のコピー18または 2 コピー32あたりでウイルスのコピーの検出限界と高感度反応。
この方法の記事の目的は、TiLV 検出アッセイ、ティラピアの組織コレクション、総 RNA の抽出に始まる cDNA 合成を実行する方法の詳細に説明して、TiLV 特定の PCR 基づいた試金。具体的には、従来の RT-PCR と SYBR グリーン ベース Rt-qpcr 両方の包括的なプロトコルは、TiLV の検出を目指している科学者の広い範囲にアピールする記載されています。前者は敏感が通常安価な検出オプションです。定量的 PCR 機械など高価な試薬より精巧なインフラストラクチャが必要です、後者ですが量的、高速、高感度、それがサブ臨床で TiLV を検出するため使用ことできますを意味あることの利点感染した魚。RT-PCR と Rt-qpcr プロトコルは、TiLV および含まれて結果の強調表示、感度の異なる地理的な分離およびここで説明アッセイの再現性を持つ 2 つの異なる研究室で行われました。
プロトコル
この研究のための動物の使用プロトコルは [許可番号 ACKU 59-獣医-016 カセサート大学動物倫理委員会によって承認されました。
注: は、試薬及び機器は、このプロトコルの提案に関する拡張情報を材料表をご覧ください。
1. 組織検体の採取
- クローブ オイル (ボリューム魚のサイズと異なります、製品の濃度通常 3 mL/L 以上) の過剰摂取を使用して魚を安楽死させます。95% (v/v) エタノールの燃焼機器を消毒するアルコール バーナーを使用して設備の順に鉗子とマヨのはさみの 4 分の 1 を浸します。
注: トリカインメタネスルフォネイト (MS-222) は、クローブ油の代わりに使用できます。 - 肝臓を見つけると小さな部分 (約 20-100 mg) を遮断または粘液ティラピア魚の後方前方から粘液を除去し、1.5 mL 遠心チューブにサンプルを置きにカバーガラスまたは外科ブレードを使用しての 200 μ L を収集します。
- プロセスのサンプルは、すぐにソリューションを安定させる RNA に格納または-80 ° C にさらに使用するまで移動します。
注: RNA を扱う際の最大の課題はそのまま RNA 分子を準備して、それらをすべての後続の処理中に破損していない維持します。RNA バックボーンは DNA よりもダメージを本質的に敏感です。抽出および組織細胞からの総 RNA の隔離が必要になります注意検査手技;RNase 自由水、試薬、機器、プラスチック製品、ガラス製品、作業スペースを使用して、手袋を着用するピペッティングのフィルターのヒントを使用して、RNase の混入を防ぐためにすべての規定を取る。
2 RNA の Guanidium チオシアン酸 - フェノール - クロロホルム抽出
- 単相性溶液フェノールとグアニジン イソチオシアネート セクション 1 から組織サンプルを含んでいる管に 1 mL を追加します。
注意: このソリューションは非常に有毒であると保護装置と適切な保護眼鏡・服・安全手袋を着用する層流フードに注意して処理する必要があります。 - 均質になるまでティッシュ杵ホモジナイザーを使用して組織サンプルを挽きます。
注: サンプルすることができますも均質にするセラミック ビーズと組み合わせて電源ホモジナイザーを使用します。組織サンプルは、次のステップに進む前に完全に均質化されたことを確認またはここのプロトコルを停止し、さらに使用するまで-80 ° C で完全に均質化試料を保存します。 - 相分離のクロロホルムの 200 μ L を追加します。
注意クロロホルムは潜在的な麻薬を非常に危険です。それは、適切な保護眼鏡・服・安全手袋を着用するだけでなく、保護具の層流フードの注意して処理する必要があります。毒性の低いとして代替、1-ブロモ-3-クロロも使用できます。
注: スケール、ボリューム アップまたは適切な場所をダウンします。たとえば、だけ相性溶液フェノールとグアニジン イソチオシアネート 500 μ L を使用した場合のみ追加します。 100 μ L のクロロホルムのこのステップで。- 15 の反転によってよくサンプルをミックス s。
- 部屋の温度 (RT) で 3 分のサンプルをインキュベートします。
- 4 ° C、12,000 × gで 15 分遠心分離
注: 下の有機相、白い中間期および RNA を含む上層の水相に明確な分離が必要があります。この上のフェーズは通常無色、型、均質化組織量によってそれは光ピンクの外観を持つことができます。 - 新鮮な遠心管に、界面を乱すことがなく上方の水層 (約 500 μ L) を転送します。
注: 転送全体の水相とオーガニック水性相を含む RNA の任意の潜在的な汚染を防ぐために少量を残したり、中間期のするしないでください。 - RNA の沈殿物に 100% イソプロパノールの 1 ボリュームを追加します。
- 必要に応じて、組織の非常に少量を使用した場合、追加 1 μ L (5-10 μ g) グリコーゲンの RNase フリーの効率的な RNA の沈殿物を促進するために各サンプルに。これは 2.8 の手順で RNA の餌の識別を支援します。
注: グリコーゲンは RNA のキャリアとして機能し、チューブの側面に付着微量 RNA を防ぐ。 - ミックスは数回反転によってよくチューブします。
- -20 ° C で一晩に 2 h のサンプルを格納します。
- 必要に応じて、組織の非常に少量を使用した場合、追加 1 μ L (5-10 μ g) グリコーゲンの RNase フリーの効率的な RNA の沈殿物を促進するために各サンプルに。これは 2.8 の手順で RNA の餌の識別を支援します。
- 12,000 × gと 4 ° C で 15 分間遠心分離機サンプル
- 微量遠心チューブの下部に RNA の餌が外れないように注意しながら、上清を捨てます。
- エタノール 75% (v/v) 1 mL を加え、数回チューブを反転 RNA サンプルを混ぜます。
- 10,000 x gと 4 ° C で 15 分間遠心します。
注: プロトコルはここで停止することができます、さらに使用するまで-20 ° C で 75% のエタノールの RNA ペレットで構成されるサンプルを格納できます。 - 微量遠心チューブの下部に RNA の餌が外れないように監視されて、上澄みを廃棄します。
- 必要に応じて、手順 2.9 2.11 70% エタノール (v/v) を使用します。徹底的に塩を最小限に抑える RNA ペレットを洗浄、または妨げる可能性のある汚染物質のキャリー オーバーが敏感なダウン ストリーム アプリケーション。
- ピペットを使用して残りのエタノールを引き出すし、室温は 5 〜 10 分より長い RNA の餌を風乾します。
注: 過剰乾燥ペレットは再停止することは困難になります。 - 30-60 μ L の RNA ペレットの可溶化 55-60 ° C に加温、RNase フリーの水を追加します。
- 即座の使用またはストア後で使用するため-80 ° c の氷の上には、RNA を配置します。
3. マイクロ ボリューム分光光度計を用いた RNA 濃度を定量化します。
- RNA に分光光度計の設定を切り替えます。
- 空白として RNase フリーの水の 1-2 μ L を使用します。
- RNA 量を評価するためにそれぞれの RNA のサンプルの 1-2 μ L を使用します。
- 230 で測定値を記録 nm、260 nm と 280 nm の各サンプルのため。
- 200 に RNA を希釈 ng/μ L の RNase フリーの水を使用しています。
4. 総 RNA を用いた相補的 DNA (cDNA) の合成
- プロトコル 2、2 μ M オリゴ (dT) は、0.5 mM の dNTPs の混合物からの総 RNA の 1 μ g を混合し、ヌクレアーゼ フリー水 10 μ L に最終巻をもたらします。これは、ミックスを調製、RT-マスター - サンプルやコントロールの数によるとテストします。
注: コントロールは、マイナス逆転写酵素サンプル (-RT) 前記 RT 酵素はヌクレアーゼ フリー水に置き換えられます (手順 4.3 参照) と、テンプレートなしのコントロール (NTC) 前記ヌクレアーゼ フリー水が RNA テンプレートの代わりにマスター ミックスに追加されます。- 簡単なの遠心分離によって続いてピペッティングでよくサンプルを混ぜます。
- 続いて 2 分インキュベーション氷の上 5 分の 65 ° C でサンプルをインキュベートします。
- 簡単に遠心管の底で液体のすべてを収集するサンプル。
- 1 x 逆転写酵素バッファー、100 U 逆転写酵素を追加し、20 μ L ヌクレアーゼ フリー水を使用する各サンプルの最終巻。
- 簡単なの遠心分離によって続いてピペッティングでよくサンプルを混ぜます。
- 85 ° C、5 分続く 60 分の 42 ° C でサンプルをインキュベートします。
- ヌクレアーゼ フリー水の適切な量を追加することによって望ましい集中に合成した cDNA を希釈し即座の使用のための氷の cDNA を置くか後で使用のための-20 ° C で保存します。
5. TiLV 従来の PCR
- CDNA、サンプルおよび選択の DNA ポリメラーゼと共にテーブル 1、詳細な確立されたプライマー対のいずれかを使用して PCR の反作用のためのテンプレートとしてプロトコル セクション 4 で生成されたコントロールを使用します。
注: 追加のないテンプレート コントロール (NTC) は、PCR の反作用のヌクレアーゼ フリー水のための cDNA を置き換えることによってここで含める必要があります。肯定的な制御、利用可能な場合は以前に検証 TiLV 肯定的なサンプルを含む含まれている必要があります。 またはプラスミッドに適切な TiLV cDNA 断片クローンで作成されます。 - 使用、サンプルとテストするコントロールの数における DNA ポリメラーゼのガイドラインに従って PCR マスター ミックスを準備します。このミックスは、前方のプライマー、逆プライマーや dNTPs、MgCl2バッファーと共に、選択した DNA ポリメラーゼを含める必要があります。
- 選択した DNA ポリメラーゼのガイドラインに従って cDNA サンプル、コントロールのサンプルの推奨量とマスター ミックスの指定された量を兼ね備えてください。
ピペッティング時にマスター ミックスのいくつかが失われるので注: が有益な多くの場合反応過剰 × 0.5 を準備です。 - DNA ポリメラーゼを利用したシステムのガイドラインに従って条件をサイクリングや適切なアニーリング温度を用いた (表 1) のプライマー PCR を実行します。通常、このようなプログラムは、95 ° C、30 の 95 ° C で変性の 30-40 のサイクルに続いて、2-5 分で初期変性 s、30 s および 30 のための 72 ° C で伸長の推奨温度で焼鈍 s、5-10 のための 72 ° c 最終伸長続く 分。
- 各 PCR の反作用と予想の PCR の製品のサイズに応じて、1-2% の agarose のゲルの井戸への適切な DNA の梯子の 5-15 μ L をロードします。増幅された DNA をゲル電気泳動による分離し、臭化エチジウム予想されるサイズ (表 1) の DNA バンドの視覚化を容易にする (EthBr) によって紫外線を使用してゲルのマニュアル機でゲルを染色します。
注意: EtBr は有毒である;それは、適切な防護服と安全手袋を着用して慎重に取り扱う必要があります。
| ターゲット TiLV ゲノム セグメント | 前方のプライマー 5'-3 ' | 逆プライマー 5'-3 ' | PCR の製品のサイズ (bp) | Tm ° C | 元の参照 | ||||
| 1 | CCAAACGTTATCTCTTAATTACGCAC | GCAAATATTTCTCTCATTCGCCT | 1641 | 50 | Surachetpong et al., 2017 | ||||
| 1 | CCTCATTCCTCGTTGTGTAAGT | AGGAGTTGCTGTTGGGTTATAG | 1000 | 62 | Mugimba et al., 2018 | ||||
| 2 | ACTCTCTATTACCAAATACATTTACT | TTACCATATATATAGTGAAGGC | 1445 | 45 | Surachetpong et al., 2017 | ||||
| 2 | GTCCAGGGCGGTATGTATTG | CTTACGGCTGACAAGTCTCTAAG | 834 | 62 | Mugimba et al., 2018 | ||||
| 3 | GTTGGGCACAAGGCATCCTA | TATCACGTGCGTACTCGTTCAGT | 250 | 56 | Eyngor et al., 2014 | ||||
| 3 | TATGCAGTACTTTCCCTGCC | TTGCTCTGAGCAAGAGTACC | 491 | 57 | Eyngor et al., 2014 | ||||
| 3 | ACCCCTTAATCCTTAATAGACCGTTA | CCCATAATCCTCTATTAGAACGTCGT | 1352 | 50 | Surachetpong et al., 2017 | ||||
| 3 | GTCGAGGCATTCCAGAAGTAAG | GAGCTAAGGGAACGGCTATTG | 834 | 62 | Mugimba et al., 2018 | ||||
| 4 | AGCAGCAGCAGGAGAAAGAG | ACCGTCCTGTTTCTGAATGG | 358 | 60 | ・ ニコルソンら、2017 | ||||
| 4 | CCAAAGTTTACTCCTATTACCCAGA | GCAAATCTTTCTCCAATTACCGTCT | 1250 | 50 | Surachetpong et al., 2017 | ||||
| 4 | GCCCAATGGTTCCCATATCT | GCCCAATGGTTCCCATATCT | 524 | 62 | Mugimba et al., 2018 | ||||
| 5 | CCAAATGTTTCTCTTATCTCAGACTC | CTTTTTCTCAGTTTACCACTTTATG | 1087 | 57 | Surachetpong et al., 2017 | ||||
| 5 | CAACTCTTAGCCTCCGGAATAC | CGTTCTGCACTGGGTTACA | 696 | 62 | Mugimba et al., 2018 | ||||
| 6 | CCAAATTTTACCTCTCGCAT | TCAAGCACTTAAAACTGTACC | 1027 | 45 | Surachetpong et al., 2017 | ||||
| 6 | CCCACACGACAGGACATATAG | GAGTTGGCTTAGGGTGATAAGA | 948 | 62 | Mugimba et al., 2018 | ||||
| 7 | CTCTCTTTGCATTGCATACCGT | GACCAATTATCCCTGCTTTCA | 704 | 57 | Surachetpong et al., 2017 | ||||
| 7 | TCCTTTAGGGATTGGCACTAAC | TTCCATCGACTGCTCCTAGA | 486 | 62 | Mugimba et al., 2018 | ||||
| 8 | ACCTCATCTACACTAACATTTCCA | TCATCATTACACAAATGGAGTAGCT | 637 | 50 | Surachetpong et al., 2017 | ||||
| 8 | CTTAAGGGCCATCCTGTCATC | TGGCTCAAATCCCAACACTAA | 476 | 62 | Mugimba et al., 2018 | ||||
| 9 | TTGGTGATGTCACGATGGATA | AGTTCTATCGCCAGCCATGT | 351 | 60 | ・ ニコルソンら、2017 | ||||
| 9 | ACAAGTCCGATTACTTTTTCCGC | TCTTTCTCACGTCCTTAAAGTCA | 530 | 50 | Surachetpong et al., 2017 | ||||
| 9 | GATATCCTCCACATGACCCTTC | GTACGTCACTTTGTGCCATTAC | 261 | 62 | Mugimba et al., 2018 | ||||
| 10 | AACCCTACTAACACCAAATATAGCT | CTTTCCCTCTGACACCCTGT | 450 | 50 | Surachetpong et al., 2017 | ||||
| 10 | TCCTCTCTGTCCCTTCTGTT | CAGGATGAGTGTGGCAGATTAT | 276 | 62 | Mugimba et al., 2018 | ||||
表 1.公開されたプライマー対 TiLV cDNA エンドポイント PCR を使用して増幅。太字で示されているプライマーは、図 3Aと 3B に示す代表的な結果を生成する使用されました。
6. TiLV 量的なポリメラーゼの連鎖反応 (qPCR)
- 適切な TiLV を含んでいるプラスミッドを使用してゲノム セグメント 3 cDNA pTiLV32, などの標準としては、重複または三重の 10 倍連続希釈系列を準備します。
- サンプル、標準コントロールの qPCR のマスター ミックスを準備、複製またはトリプリケートのヌクレアーゼ フリー水 0.4 μ、前方のプライマーの 0.3 μ L、逆プライマーの 0.3 μ L、SYBR グリーン × 2 の 5 μ L を利用した反応を実行するアカウントに考慮Dna ポリメラーゼ反応あたりマスター ミックス。
- 次のようで標準的な pTiLV とプライマー情報 10 μ M の濃度でプライマーを使用します。
プライマーを転送: TiLV-112F (5'-CTGAGCTAAAGAGGCAATATGGATT-3')
逆プライマー: TiLV-112R (5'-CGTGCGTACTCGTTCAGTATAAGTTCT-3')
標準 pTiLV:10 pg/μ l
注: サンプルやコントロールの合計数が 10、トリプリケートで行われる場合、これは 12 μ L ヌクレアーゼ フリー水、9 μ L 前方プライマー、9 μ L 逆プライマー、SYBR グリーン DNA ポリメラーゼ マスター ミックスの 150 μ L を含む qPCR マスター ミックスに相当します。市販ユニバーサル SYBR グリーン DNA × 2 を購入したポリメラーゼ マスター ミックスはすべて必要なコンポーネント qPCR 反応、すなわちの私は色素 SYBR グリーン、ホット スタート Taq DNA ポリメラーゼ、dNTPs、MgCl2パッシブ参照染料を含まれています。SYBR グリーン マスター ミックスを光から保護します。
- 次のようで標準的な pTiLV とプライマー情報 10 μ M の濃度でプライマーを使用します。
- QPCR ストリップ管または 96 ウェル プレート使用の qPCR のマシンと互換性に qPCR マスター ミックスの 6 μ L を調剤します。
- チューブや 96 ウェル プレートの井戸に cDNA テンプレート、コントロールまたは希釈した TiLV 規格の 4 μ L を追加します。
- QPCR チューブを閉じたり、使用 qPCR マシン用の互換性のあるプレート カバー付き 96 well プレートのシール
- ソリューションと qPCR チューブや船舶の底に液体のすべてを収集する遠心分離機を使用して 96 well プレートのスピンダウンをミックスする qPCR チューブに軽くフリックします。
- リアルタイムのサーマルサイクラーにチューブやプレートを配置します。
- 3 分、95 ° C の 40 のサイクルが続く 95 ° C で初期変性を実行する qPCR たちをプログラム 10 s および 60 ° C、30 のプライマーのアニーリングと伸長、0.5 の ° C の増加が 65 ° C から 95 ° C 融解曲線ステップで終わる s/5s。
- 蛍光染料として SYBR を選択し、サンプル タイプとして未知を選択サンプル名] ボックスに名前を挿入します。
- Rt-qpcr マシンのふたを開けてし、割り当てられた井戸に qPCR ストリップを置き、ふたを閉じる。
- 選択した条件で RT qPCR 測定を実行します。マシンは、蓋が所望の温度に達した後に実行が開始されます。反応の進行を監視する各拡張ステップ後の各サンプルの蛍光を収集します。
注: qPCR マシンおよび関連ソフトウェアが自動的にアッセイのすべてのパラメーターを計算して増幅曲線をリアルタイムで表示、標準曲線と融解曲線は qPCR サイクルの最後に生成されます。 - データ分析を行い、各サンプルの融解曲線最初確保による買収と標準、私アンプリコンの予想される温度で均一な 1 つのピークがあります。
- サンプルの増幅曲線の評価し、基準と領域でしきい値のセットされた Cdna の増幅率はすべてのサンプルで同じ。これは通常、ソフトウェアによって自動的に実行されますが、慎重にチェックする必要があります。
- 標準曲線を用いた TiLV コピーの数を計算します。
結果
セクション 1 で説明されているプロトコルに従い TiLV 感染 (図 1 a) の臨床徴候を表示する瀕死の赤いハイブリッド ティラピアは麻酔薬として機能するクローブ オイルの高い濃度の入浴で安楽死されました。臨床症状は変数ですが、一般的な症状は、無気力、皮膚びらんと変色、exophthalmia、戸のスケール、オープン傷/病変や異常行動15,16,33、ように見える 35,36, これらのいくつか見ることができる明確に図 1 aに。腹壁は、肝臓、脾臓 (図 1 b) 頭腎など内臓を収集するために削除されました。粘液も採取したこの段階でカバーガラスまたは外科ブレード37を使用して魚の後方に前方から皮膚を軽く削って.

図 1.ティラピア郭清およびサンプル コレクション。A. 口と蓋、皮膚びらん、角膜の不透明度周囲の発赤皮膚 leisons と TiLV に感染した赤いハイブリッド ティラピア。B. Sectioned 赤ハイブリッド ティラピア (青い矢印) の時点で肝脾臓または頭腎の臓器から組織の採取を許可します。この図の拡大版を表示するのにはここをクリックしてください。
その後、総 RNA の Guanidium チオシアン酸-フェノール-クロロホルム抽出法は、セクション 2 の詳細なプロトコルが続いたし、純度比率の計算による RNA 定量セクション 3 で説明しているサンプル純度を評価するために行ったとスペクトル プロフィール (図 2) の検査。図 2 aは、図 2 bは、RNA の準備不足から成功の総 RNA 抽出法、代表的な結果を示しています。核酸蛋白質は 280 で彼らを持っている間 260 で吸光度の極大がある nm。260 測定比 nm と 280 nm は、各サンプルの純度を示す、1.9 2.1 への比率は、図 2 aのサンプルの場合、純粋な RNA を示します。図 2 bで低い A260/280 比率 RNA 抽出プロシージャから残り、タンパク質またはフェノールに可能な汚染を示します。吸光度 230 nm サンプル汚染の結果になること、このため A260/230 nm 比を計算しても。この比率は 2.0 から 2.2 純粋な RNA の準備のための範囲べき 1.07 低 A260/230 比は、図 2 bとスペクトルの形状は 230 でトラフのシフトを示していますでは図 2 a、サンプルに対して 2.03 の値で示すように向かって 240 nm 残留グアニジンまたはサンプルのフェノールの示唆されている nm。サンプルは図 2 bに示すように、再汚染物質を除去する RNA を沈殿させるサンプルの純度が向上します。
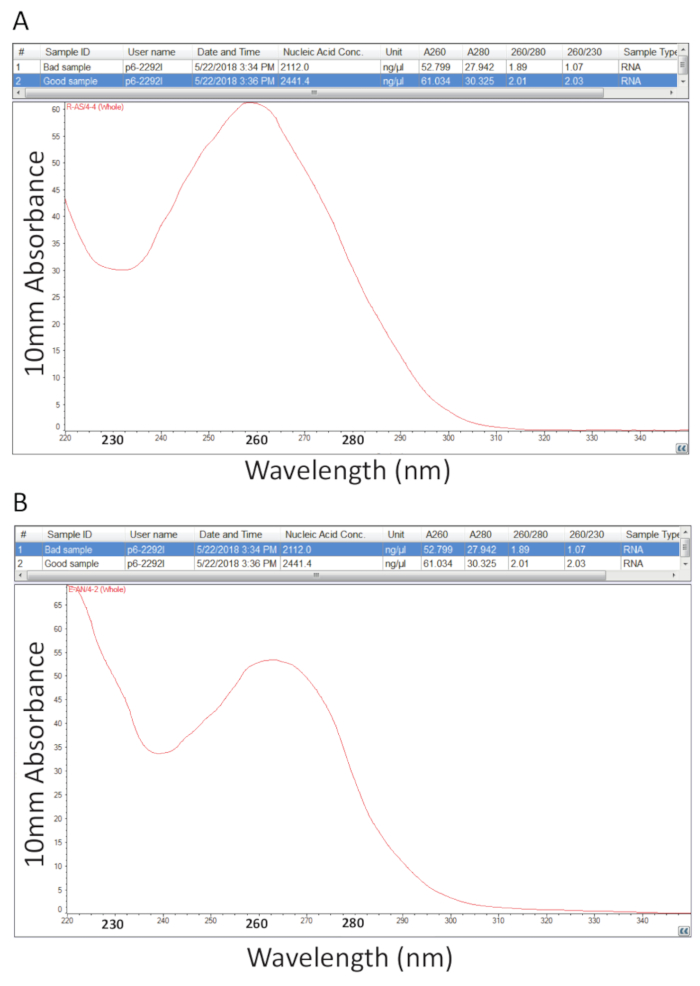
図 2.病気にかかったティラピア組織から抽出した総 RNA の吸光光度定量。A. 純度比率および成功した RNA の準備からスペクトルのプロファイル。B. として、貧しい RNA 抽出法の代表者を除く。この図の拡大版を表示するのにはここをクリックしてください。
RT-PCR による TiLV を検出するには、など図 2Aで表される 1 つの純粋なサンプル逆転写 (プロトコル 4) cDNA にされとして使用 PCR のテンプレート アッセイの詳細セクション 5、代表的な結果を図 3 aに示します。表1 に示すようにプライマーが大胆な TiLV ゲノム セグメント 314491 の bp のフラグメントを増幅するために使用されました。PCR の製品は電気泳動によって分離され、可視化の EtBr 染色します。図 3 aは 2 段階の RT-PCR の結果を示しています 4 cDNA サンプル (S1 S4)、タイ、および各サンプルでは、約 500 のきれいな単一のバンドで分離された病気のティラピアの肝臓から派生を使用して bp を観察することができます、したがって、サンプル 1-4 TiLV肯定的です。同じ PCR の製品はないのテンプレート コントロール (NTC) は PCR の製品を屈服したプラスミド32にクローン TiLV セグメント 3 の cDNA を構成する肯定的な制御サンプルから得られました。図 3 aのようにワンステップ RT-PCR 法のアプローチを使用して、異なる研究室で、元のエジプトの養殖のティラピアの頭腎組織から派生した 5 つの RNA サンプルと同じプライマーを使用して図 3Bのアッセイを行った15. サンプル 1、3、5 は、適切なサイズで PCR の製品が見つかりませんでしたので、サンプル 2 と 4 が TiLV 否定的な肯定的な TiLV をこの検出アッセイを使用して決定されました。ネガティブ コントロール マイナスの逆転写酵素コントロールと 2 つの NTCs 2 名を含む任意の PCR 産物を生成しませんでした。ワンステップ RT-PCR の試金はティラピア ActinB 遺伝子をターゲットにも行った。増幅サイズ 217 の bp が予想される38としてすべてのサンプル (S1 S5) で生成されました。この試金は TiLV 肯定的なサンプルの半定量的な検討を可能にすると同様に、RNA サンプルの完全性のコントロールとして提供しています。生成されたティラピア ActB 製品は比較的等しい、TiLV 特定の PCR 産物を生成量の違いは所定の組織サンプルの TiLV の量の真の反射として解釈できます。
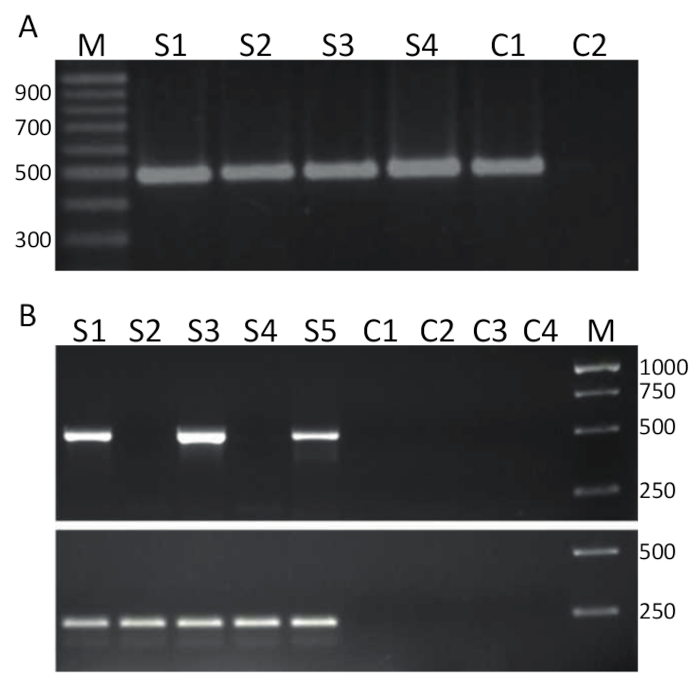
図 3.TiLV RT-PCR。A. 画面は、TiLV 感染特異的プライマーを用いたセグメント 3 タイから収集された cDNA サンプルでは病気にかかった鯛の肝組織から生産 (に示すように表 1に大胆な) 2 ステップ PCR アッセイを用いた TiLV の。M =; 塩基対のマーカーS1 S4 = サンプル 1-4;C1 = PCR テンプレートとして pTiLV を使用して肯定的な制御C2 = テンプレート コントロール (NTC) はありません。B。 ワンステップ RT-PCR 法 A のように同じプライマーを使用してエジプト15から病気にかかったティラピアの頭腎組織から採取し。M =; 塩基対のマーカーS1-S5 サンプル 1-5 を =。逆転写酵素コントロール マイナス コントロール C1 C2 があり、C3-C4 は NTCs。 下のパネルがワンステップ RT-PCR ティラピア ActinB38 (詳細についてはを参照してくださいテキスト) 生産に対する監督のプライマーを使用して 217 塩基対の PCR の製品。この図の拡大版を表示するのにはここをクリックしてください。
PCRs では図 3qPCR の試金プロトコル 6 で説明する表されるエンドポイントとは異なり各 PCR のサイクルの後の PCR の製品の量を測定します。ターゲット DNA の増幅は、各ラウンドの反応から生成される DNA と相互作用する蛍光分子を使用して検出されます。ここでは、私色素 SYBR グリーンは、二本鎖 dna を主体に利用されました。蛍光信号が反応中に続いて、その強度に関連する製品の量は39,40,41,42,43を形成します。TiLV qPCR の試金はプロトコル異なる SYBR グリーン試薬、qPCR 機、サンプルを使用して、さまざまな国から異なる研究室で 6 で説明するよう行われました。結果として得られる増幅曲線は、図 4 aおよび4 bのとおりです。それぞれの試金のための実験コースは 4 つの段階を観察できる: 線形地面相、早期指数、指数後期、プラトー相。線形地面フェーズは、DNA 複製は、DNA 量が不足しているシグナル/バック グラウンド比の生産のため識別することはできませんまだ初期サイクル中に発生します。このフェーズでは、ベースライン蛍光が計算されます。その後、ターゲット DNA は各サイクルで検出可能な背景の上になるし、指数関数的に増加する信号の誘導濃度のダブルを開始します。よく最適化された qPCR アッセイの増幅効率 (E) が非常に高い (100%) に近いこの初期段階指数関数的増幅の反応と安定したままの初めにはこの時点で、定量化が実行されます反応効率がまだ安定しています。後のサイクルの信号が高原、開始され反応コンポーネントが疲れ44の蛍光強度は開始テンプレート コピー数に相関はもはや。彩度が DNA の基質分子へ再アニーリング反応、コンポーネント、または酵素単位量の変化の濃度比から競争のためもあります。おそらく、このようなパラメーターは、図 4 a 4 bに示すように試金のための増幅曲線の違いを考慮します。含まれているコントロールは、これらの特徴的な増幅曲線を生成しませんでした。
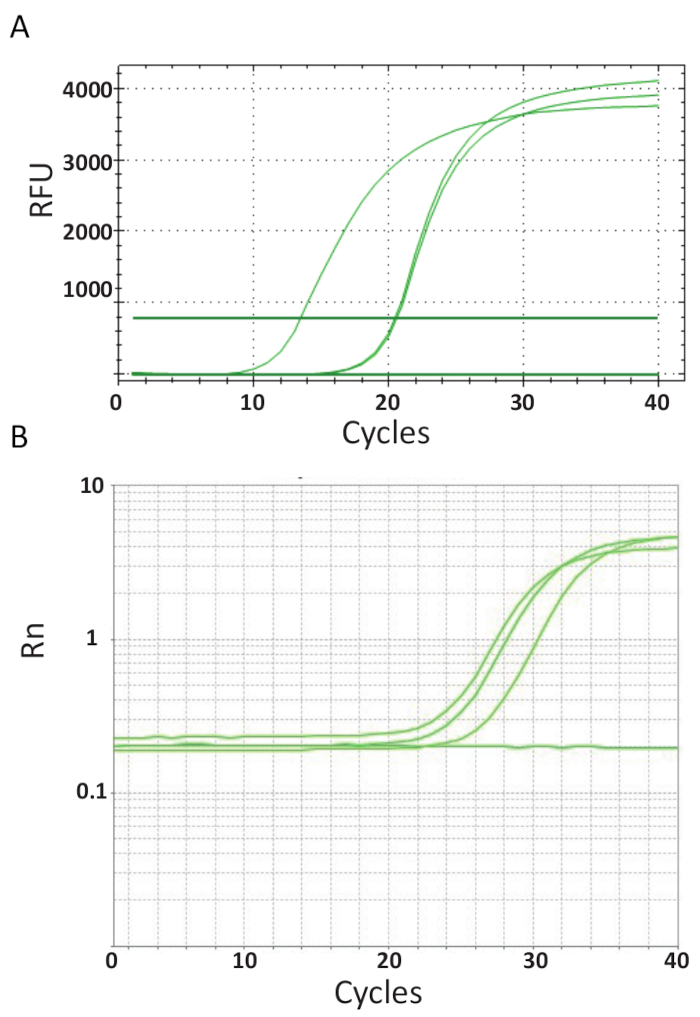
図 4.増幅のリアルタイム PCR の試金の期間にわたって製品の蓄積を示すプロットします。A. TiLV 陽性検体の増幅曲線はマイク 2 ステップ qPCR アッセイ タイや NTCs、SYBR グリーンを使用して肯定的なプラスミッド コントロールから派生します。グラフは、相対的な蛍光 (RFU)とサイクル数の関係をプロットすることによって生成されました。B. TiLV 肯定的なサンプルの増幅曲線が図 3 bのように、エジプトと、NTC から派生しました。増幅曲線は、サイクル数対アッセイ (Rn) に含まれている受動の ROX 色素の蛍光に正規化された記者信号の蛍光です。この図の拡大版を表示するのにはここをクリックしてください。
それぞれの研究室の異なった機械 qPCR サイクルの最後に、データは買収され、分析しました。図 5Aと5Bは、各研究室で実行される試金から代表の融解曲線を表示します。各 qPCR マシンは、最後に融解曲線解析を実行するプログラムされました。これは、増分、温度の上昇や温度の関数として蛍光を監視によって達成されました。温度が十分に高い dsDNA を変性するため、蛍光分子が放出されて蛍光性の大規模なドロップが記録されます。QPCR の各計測器のソフトウェアは、負の最初の誘導体対温度 (図 5) をプロットすることによって融解曲線データからアニーリングの温度 (Tm) を計算しました。図 5Aと5B製品が異なるサンプル セットで形成される試金のための約 80 の ° C の予想される温が均一の融解転移あること見ることができます。低温の他のピークは認められなかった。サイズが小さいため、プライマー二量体の Tmはターゲット DNA シーケンスのそれより普通低い。したがって、この Tmの差の場合、潜在的なプライマー二量体またはその他の非特異的増幅の製品を識別するために簡単になります。コントロールは、TiLV 肯定的なサンプルおよび標準のような溶解曲線を生成しませんでしたし、図 5Aと5Bでグラフの下部にほぼ水平線として見なすことができます。
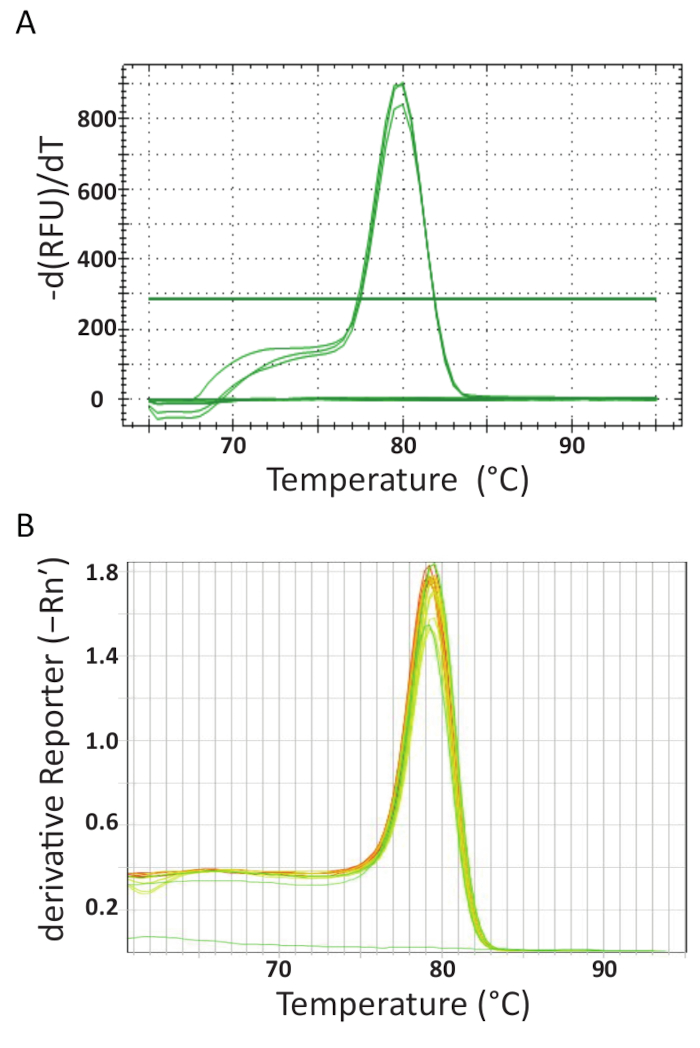
図 5.アッセイの特異性と別の PCR の製品は、溶解の機能によって区別できますように曲線解析を溶かします。A.タイ、負の制御、および肯定的なプラスミッド コントロールから TiLV 陽性検体の曲線解析を溶かします。B. TiLV 肯定的なサンプルに由来するエジプト、pTiLV 基準、NTC の曲線解析を溶かします。温度の変化によって分けられる蛍光の A と B 両方を表示するグラフは、融解ダイナミクスの鮮明な画像を生成する温度に対してプロットしました。この図の拡大版を表示するのにはここをクリックしてください。
ほとんどの qPCR のマシンはさらに評価、qPCR を実行し、pTiLV 標準のテンプレートの対数に対してサイクルしきい値 (Ct) を自動的にプロット標準曲線を生成することで、サンプルを定量化を促進するソフトウェアが付属します。図 6Aおよび6Bの 2 つの独立した実験室のように、番号をコピーします。簡単に言えば、Ctは qPCR 結果の評価に使われる単位です。Ct値は、閾値を設定する蛍光信号レベルに達するに必要なサイクルの数を示します。少ないテンプレートを開始する量サイクルそれ検出可能な蛍光レベルを達成するのにかかる。確かに、TiLV の負荷の高いサンプルはサブ臨床感染症で魚のように TiLV の低負荷のサンプルより下の Ctの値を持ちます。Ctの値を決定するには、バック グラウンド蛍光レベルは raw データから最初に控除されます。次に、qPCR デバイスに関連付けられたソフトウェアを使用して、各サンプルのデータ曲線を検索とサンプルがしきい値を交差を表す Ctを組み込むことによって蛍光しきい値を選択することが自動的に。個別にそれぞれの試金のためこれは、しきい値ごとに、慎重に検討、増幅曲線のすべてのカーブが平行の場所で対数に関するしきい値が設定されていることを確保します。したがって、取得特定 Ct相対値と開始テンプレート コピー数45、基準としていますがも qPCR 機械および使用試薬の PCR 増幅の効率と検出の感度を特定。これらのパラメーターは、図 6に同じアッセイを使用して見られた差異に貢献します。
図 6、回帰分析の標準曲線から直線性反応の増幅効率 ((101/m -1) × 100)46とインターセプト標準曲線斜面 (m) の計算を含む行った。標準曲線の解析も感度 (検出限界)、再現性、測定の再現性を確認するために使用されました。あらゆる PCR で DNA の量を倍増する理論的には、サイクルの効率 (E) が 100% に等しいことを意味します。ただし、実際にこのような理想的な効率はめったに DNA ポリメラーゼの阻害、汚染物質、あまり cDNA とピペッティング エラー47など最適の PCR 条件のため達されます。図 6A 6B を図7 希釈した pTiLV を使用しての試金の分析効率ながら、8 の希釈した pTiLV サンプルを使用して 94.5% の効率を算出したの良いの試金のための 90-110% から通常、増幅 E 範囲サンプルは、101.2% であった。100% 以上の効率は通常、アッセイの PCR 阻害物質の存在のために。赤図 6 b に示すように 3 つの TiLV サンプルの観察することができます。、各サンプル41,42,45, TiLV コピーの数の計算の標準的なプロットの回帰分析もできます。S1、S3 と S5図 3Bに示すサンプルの結果に沿ってであります。
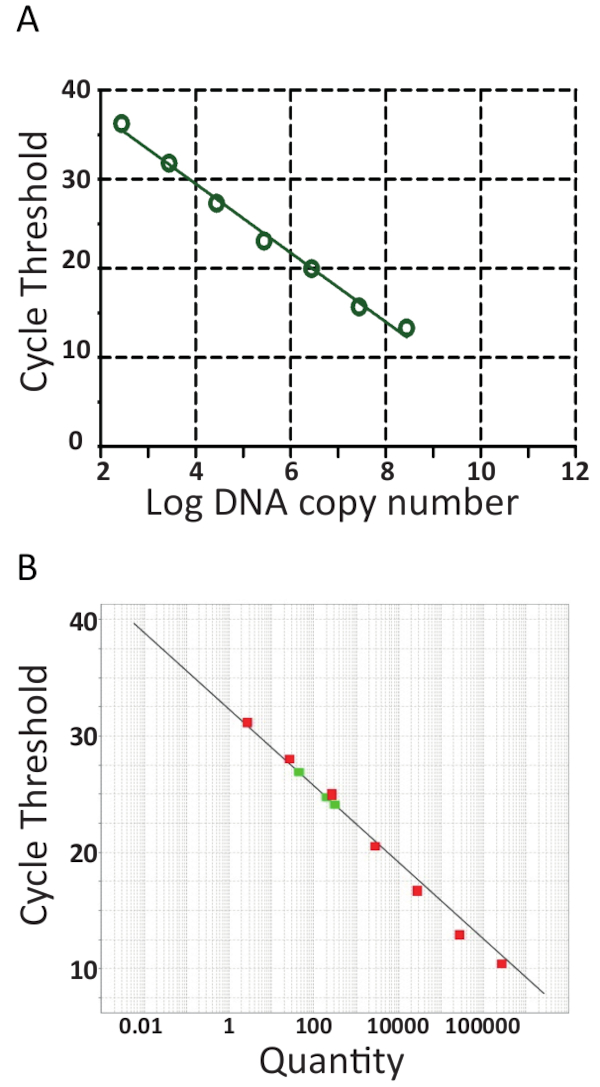
図 6.RT qPCR 標準曲線。PTiLV、両方の研究室で使用される標準のシリアル希釈液 10 倍のリアルタイム PCR は。A. 8 希釈した pTiLV サンプルが、濃度既知のすべてをテストし、TiLV のコピーの数に相関/反応。標準的な曲線は、ログ コピー数とサイクルの閾値 (Ct) をプロットすることによって生成されました。斜面-3.462、R2 = = 0.9992 効率は 94.47% と。B. のように 7 希釈した pTiLV を除いてサンプル (グリーン) テストされ、y 軸と x 軸上の TiLV (数量) のコピー数のしきい値のサイクルが表示されます。Y 切片 = 32.327、斜面-3.292、R2 = = 0.98 効率が 101.2%。両方の標準曲線のアッセイのパフォーマンスを理解するために A と B の傾き、y 切片と相関係数 (R2) が活用されます。重要なことは、R2値に近くなります 1 以来、それは標準的なカーブの直線性の測定です。傾きを使用して 100% の効率が-3.32 の傾きに対応する前記 PCR 効率を測定、メイン テキスト方程式および詳細を参照してください。良い qPCR 反応-3.58 と-3.10 の間の斜面に相関 90 110% 間の効率を通常されます。標準曲線は未知の TiLV 肯定的なサンプルの絶対定量に使用され TiLV コピーの正確な数を決定/反応、そのまま B の 3 つの TiLV 肯定的なサンプルの場合赤色
ディスカッション
TiLV はイスラエル共和国の14の 2014 年に最初に報告された、その後、エジプト、コロンビア、インド、マレーシア、ウガンダ、タンザニア、タイ15,16,18,を含む複数の国で認定されています35,48。 グローバルな意識、特に、ティラピア生産国ではもっと注意に配置ウイルスおよび様々 な制限と対策政府当局からは、TiLV の拡散を防止しようとして実装されています。ここでは、サンプルのコレクション、RNA の隔離、cDNA 合成、PCR と qPCR アッセイを覆うティラピア組織における TiLV 検出のための詳しいプロトコルを説明されています。特定のディスカッションを保証するこれらのメソッドのさまざまな側面があります。TiLV が様々 なサイズ9,12,14,15,49とティラピア養殖ハイブリッド (o. を含むこれまでのところ、ティラピアの種にまたがる魚に発見されましたO. 球菌 x niloticus)11,14, ナイル川テラピア (O. niloticus)9,10,14,15,16,33,35,36,49,50と赤テラピア (外部sp.)16,33,48,51, ティラピア ナイル野生9,12と同様ブラックとティラピア51 t. ジリ14,15 s. galilaeus、 O. 球菌とt. シモニス intermedia14非常に最近 TiLV 野生鯉 (Barbonymus で識別されました。schwanenfeldii)52。内臓 (ギル、脾臓、肝臓、心臓、頭腎) または粘液37から組織サンプルは、年齢、サイズや魚種に関係なく瀕死の状態と同様、健全なティラピアを採集し、RNA の隔離のため処理できます。概説総 RNA の抽出のプロトコルはここカオトロ ピック変性剤であるフェノールとためのチオシアンの単相性のソリューションを使用します。組織が直接クロロホルムおよび上層の水相、中間期およびより低い有機相を含む明確な RNA の生成前記相分離を達成するために遠心分離の添加に続いてこのソリューションで均質化されました。RNA はイソプロパノールの沈殿物、汚染物質を取り除くために回収された RNA の洗浄の順で水相から隔離されます。この方法で RNA の隔離 Piotr Chomczynski、ニコレッタ サッキによって開拓された、ためチオシアン酸-フェノール-クロロホルム抽出53,54と呼ばれました。RNA の抽出に使用される試薬のこのタイプが市販購入または実験室で行われた (詳細については、材料の表を参照してください)。このプロトコルはシリカ系浄化など列ベースの方法よりも少しかかりますが、一般に、それはより費用効果より多くの RNA が得られます。
このプロトコルでは吸光光度法の値は、RNA の品質を示すことができますという説明した A260 値を使用して RNA の定量化 (A260/A280 = 1.9 2.1)。一方、このメソッドは、サンプル純度の良い指標を与えるが、それは絶対に抽出した RNA の品質について通知できません。RNA はそのまままたは部分的に劣化したかどうかを適切に決定、サンプルは 18 歳が前記ステンド EtBr のスミア アガロース電気泳動による分離をすることができ、28 s rRNA バンドを示す RNA の劣化。RNA の品質のさらなる検証ラボ-オン-チップ計測器を使用してがあります。さらに、それはまた DNase で浄化された RNA を消化する重要な汚染を削除するホスト DNA を下位アプリケーションによって偽の結果につながる可能性があります。追加 DNaseI 治療にはホスト gDNA は大部分は RNA のサンプルを汚染まだ場合 RNA 抽出手順の最後で実行できます (材料の表を参照してください)。
相補的な DNA 合成は全体的な qPCR 結果に大きく影響することができます、変異を導入することが法の一側面です。ここで提唱した cDNA プロトコルを単一コンポーネント セットアップの構成オリゴ (dT) を使用し、したがってのみ polyA 尾を含む Mrna を転写。まさに逆転写反応と cDNA のこのモードで使用するコンポーネントの合成が TiLV 検出32成功ユーザー制御できます。このセットアップに代わる逆転写反応に必要なすべてのコンポーネントを含む市販買ったマスター ミックスは、非常に速く、通常のマルチ ステップ、ピペッティングおよび複数温度プロトコルを使用せずに単純です。それは処理を最小限に抑え、すべてのサンプルの均一性を促進するため、これは有利です。このようなマスター ミックスがありますがそれに別に適用される RNA テンプレートを作ると人口における rna 全体の長さから代表的な cDNA シーケンスのコピーを生成するランダムなプライマーとランダムプライマー (ウイルスおよびティラピア mRNA をホスト) と理論的には、すべての目的の RNA の種は、従来の PCR またはそのようなサンプルから qPCR で測定できます。この汎用性は 2 段 RT-PCR 方法の主な利点様々 な実験に使用することができます長期的なプールを提供しています。結果で、ワンステップ RT-PCR 法のアプローチを表現されています前記シーケンス特異的プライマー (表 1) が使用され、1 つの管で RT と PCR を行った (マテリアル リストを参照してください)。一般的に、ランダムなプライミングを使用するよりも特定のターゲット RNA の高い RT の効率を可能にするシーケンス特異的プライマーが、RNA の特定の対象は 1 つだけ (を参照してください特定の研究所の唯一の目的となるような cDNA のサンプルで定量化することができます。材料の表cDNA 合成製品に関するご提案)。
従来の RT-PCR は通常使用に見えますがところ TiLV 診断9,13,14,15,16,17,の18,33,35,48,55。 RT qPCR 検出及び魚肉の粘液32,37TiLV の少量の定量化のより強力なツールであることが示されています。一般に、qPCR 広く高い感度、特異性、良い再現性、広いダイナミック レンジとスピード21による臨床ウイルス診断ラボで使用します。QPCR は当初より高価な従来の RT-PCR より実装するかもしれませんが、それは従来 pcr 法に比べて多くの重要な利点を提供してください。結果をサンプルからより高速なターンアラウンド タイムがあり、任意のポスト PCR の手順は必要ありません。この後者の点では研究室の汚染のための最小限のリスクがある、それはより簡単にアウト ブレークが発生した場合など、高スループットの状況に適応することができます。さらに、サブ臨床感染症21の低ウイルス量を検出する重要性が従来 RT-PCR より本質的に敏感です。逆のトランスクリプションが必要な入れ子になった PCR アプローチが必要だろうこれ、2 つさらに PCR の反作用とアガロースゲルを用いたゲルの電気泳動。これらの多くの手順は多くの時間を取るし、エラーまたは汚染の可能性を高めます。それにもかかわらず、その高感度のため Rt-qpcr 要求綿密な実験的なデザインと正確な結果56,57を生成するための定量化方法を十分に理解します。
DNA の結合 fluorophore SYBR グリーン私はこのプロトコルで実証されています。DsDNA の非特異的 DNA 結合色素であり従ってアッセイの特異性は完全に偽陽性58を生成可能性がありますプライマー セットあります。したがって、正しい T の 1 つだけの PCR 増幅を確認するため、融解曲線解析各 PCR の側で実行される dsDNA は PCR の反作用の特に重要な部分は、mが生成されます (これも達成されるべきゲル電気泳動新しいアッセイが実装されている場合)。DNA のフラグメントの Tmは様々 なその長さ、GC の構成、シーケンス、鎖の相補性などの機能に依存して、同様に濃度バッファー コンポーネントと PCR エンハンサー。研究所には、プライマー二量体またはその他の不要な PCR の製品の存在は明らかにしていないが、これは他のサンプルや実験のセットアップと見られる場合、アッセイはする必要があります 2 つの代表の結果の融解曲線解析再最適化されています。QPCR のより高度な技術は、このような融解曲線のステップを必要としないし、確かに、以来ペーパーは、このメソッドに基づいて TaqMan TiLV RT qPCR 開発されています 2 つのプライマーとプローブの高 TiLV 特定34ことを利用しました。
確かに、RT qPCR の試金のために設計したプライマーはアッセイの成功に不可欠とプライマーをここでは公開時間32TiLV ゲノム データに基づいて設計されました。しかし、RNA ウイルスの高い突然変異率を展示するよく知られているが、ISAV59でみられたように可能な系統が現在の診断テストを脱出します。常に普遍的なパン TiLV RT を生成するには、このようなウイルス型困難になります-qPCR 法及びこのような試金のみ継続的に改善される広範囲の場所と時間帯からのより多くの TiLV ゲノム データが利用可能になる場合。
最後に、重複実行または可能であれば、両方の内で反応をトリプリケートし、qPCR アッセイを間に不可欠です。Ctの値が非常に高い場合複製の使用は PCR の反作用は信頼性が高く、再現性のあることを確認するために特に重要であります。一般からデータをレプリケートする場合反応変化以上 0.5 サイクル反応が繰り返されることし、Ct値は一貫して > 0.5 サイクルを異なる場合にレプリケート、アッセイを再最適化する必要があります。統合 qPCR ピペッティング ロボットの使用は、この問題に非常に役立ちますが、高級ツールです。任意の実験と同様に、含める適切かつ適切なコントロールは、堅牢な分子アッセイを特にこのような試金が認定されることが診断の研究所で開発に最も重要なは。コントロールには、ポジティブ (肯定的な TiLV サンプル、TiLV プラスミド標準) ネガティブ コントロール (NTC と-RT) サンプルと内因性ティラピア ハウスキーピング遺伝子の検出を含める必要があります。このようなコントロールは過小評価することはできません、各試金、試金の各ステップの品質を正しく理解し、正しく結果を解釈するために含まれるべき。
開示事項
著者が明らかに何もありません。
謝辞
獣医細菌学研究所、Vetsuisse 部、ベルン大学を彼らのサポートに感謝しております。この作品は、PN に与えられる Vetsuisse 部、ベルン大学 120% モデルの資金によって初期のキャリアの研究者の学術振興とジェンダー平等のための委員会によって賄われていた。WS と PR のでサポートされるセンター総合研究所総合研究、カセサート大学、バンコク、タイ [高等教育研究の推進と国家研究プロジェクトのタイ大学、オフィス、食品農業高等教育委員会、文部科学省のタイ。彼女のナレーションの博士 Kwanrawee の Sirikanchana と Piyawatchara シカリン ビデオを編集するために感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| Tissue collection | Step 1 | ||
| Tricaine methanesulfonate | Sigma-Aldrich | E10521 | An alternative to clove oil. Step 1.1 |
| RNAlater stabilization solution | Thermo Fisher Scientific | AM7020 | For storing tissues if they cannot be processed immediately Step 1.3 |
| RNA extraction | Step 2 | ||
| TRIreagent | Sigma-Aldrich | Step 2.1 | |
| TRIzol | Thermo Fisher Scientific (Invitrogen) | 15596026 | Step 2.1 |
| GENEzol | Geneaid | GZR100 | Step 2.1 |
| Trisure | Bioline | BIO-38032 | Step 2.1 |
| Homemade solution | - | - | 94.53 g/L (800 mM) guanidine thiocyanate 30.45 g/L (400 mM) ammonium thiocyanate 8.20 g/L (100 mM) sodium acetate 380 mL/L (38 % v/v) phenol 50 mL/L (5 % v/v) glycerol 1.0 g/L (0.1 % w/v) 8-quinolinol, pH 5.0 Store up to 2 years at 4oC Step 2.1 |
| MagNA Lyser Green Beads | Roche | 3358941001 | An alternative tissue homogenization method used in conjunction with tissue lysing machines detailed below Step 2.2 |
| Lysing Matrix D, 2 mL Tube | MP BIOMEDICALS | 116913050 | |
| Chloroform | Sigma-Aldrich | C2432 | Step 2.3 |
| Chloroform | RCI Labscan | AR1027E-G2.5L | Step 2.3 |
| 1-Bromo-3-chloropropane | Sigma-Aldrich | B9673 | A less toxic alternative to chloroform Step 2.3 |
| Isopropanol (GC) ≥ 99.8 % | Sigma-Aldrich | 59300 | Step 2.6 |
| Isopropanol (ACS, ISO Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.09634.2500 | Step 2.6 |
| Glycogen, molecular biology grade (e.g., Sigma, cat. no. G1767) | Thermo Fisher Scientific (Thermo Scientific) | R0551 | Useful step if tissue starting material is small to maximise RNA precipitation optional |
| Ethanol (purity (GC) ≥ 99.9 % | Sigma-Aldrich (EMD Millipore) | 1.00983 | Step 2.9 |
| Ethanol (ACS, ISO Reag. Ph Eur) | Merck (EMSURE) | 1.00983.2500 | Step 2.9 |
| Nuclease-free water | Promega | P1193 | Step 2.13 |
| Nuclease-free water | Multicell | 809-115-CL | Step 2.13 |
| Ambion TURBO DNA-free kit | Thermo Fisher Scientific (Invitrogen) | AM1907 | Can be performed at the end of the RNA extraction protocol optional |
| cDNA synthesis | Step 4 | ||
| Viva cDNA Synthesis Kit | Vivantis | cDSK01 | Step 4.1 & 4.3 |
| ReverTra Ace qPCR RT MasterMix with gDNA remover | Toyobo | A1172K | An alternative option see discussion |
| ReverTra Ace qPCR RT Kit | Toyobo | FSQ-101 | An alternative option see discussion |
| AffinityScript Multiple Temperature Reverse Transcriptase | Agilent Technologies | 600107 | An alternative option |
| PCR | Step 5 | ||
| DNA polymerase systems: | Step 5.2 | ||
| - Platinum II Hot-Start Green PCR Master Mix (2X) | Thermo Fisher Scientific (Invitrogen) | 14001012a | Step 5.2 |
| - GoTaq Mastermix | Promega | M7122 | Step 5.2 |
| Separate PCR mixture components: | Step 5.2 | ||
| 10mM dNTP Mix | Vivantis | NP2409 | Step 5.2 |
| 25mM MgCl2 | Thermo Fisher Scientific | R0971 | Step 5.2 |
| 10X Taq Buffer with KCl | Thermo Fisher Scientific | 00348114 | Step 5.2 |
| Taq DNA polymerase | Vivantis | PL1202 | Step 5.2 |
| - Verso 1-step RT-PCR ReddyMix with ThermoPrime Taq | Thermo Fisher Scientific | AB1454 | One step RT-PCR exemplified in Figure 3B |
| Gel electrophoresis: | For visulation of PCR products from steps 5.1-5.4 | ||
| Ethidium Bromide solution (10 mg/mL) | Thermo Fisher Scientific | 17898 | Step 5.5 |
| Tris/Acetic/EDTA (TAE) buffer: | Step 5.5 | ||
| - Tris | Vivantis | PR0612-1KG | Step 5.5 |
| - Acetic acid (glacial) (ACS, ISO, Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.00063.2500 | Step 5.5 |
| - Ethylenediaminetetraacetic acid (EDTA) | BIO-RAD | 161-0729 | Step 5.5 |
| Agarose | Vivantis | PC0701-100G | Step 5.5 |
| DNA ladders and markers | Vivantis | NL1405 | Step 5.5 |
| DNA gel loading dye (6X) | Thermo Fisher Scientific | R0611 | Step 5.5 |
| qPCR | Step 6 | ||
| PowerUP SYBR Green Master Mix | Thermo Fisher Scientific (Applied Biosystems) | A25779 | Exemplified in Figures 4-6B Step 6.2 |
| iTaq Universal SYBR Green Supermix | BIO-RAD | 1725120 | Exemplified in the video and in Figures 4-6A Step 6.2 |
| Equipment | |||
| Dounce tissue grinder pestle | Sigma-Aldrich | P1110 | Protocol 2 |
| MagNA Lyser Instrument | Roche | 3358976001 | An alternative tissue homogenizing option for protocol 2 which are used in conjunction with the lysing beads detailed above Step 2.2 |
| FastPrep-24 5G Homogenizer | MP BIOMEDICALS | 116005500 | |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5427R | Protocol 2 Step 2.4, 2.7 & 2.10 |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5418R | |
| Heat box | Labnet | AccuBlock Digital Dry Bath | Protocol 2 Step 2.13 |
| Microvolume spectrophotometer | Thermo Fisher Scientific (Applied Biosystems) | Nanodrop 2000 | Protocol 3 Step 3.1 - 3.4 |
| PCR machine | BIO-RAD | T100 Thermal Cycler | Protocol 5 Step 5.4 |
| Power supply | BIO-RAD | PowerPac HC | Protocol 5 Step 5.5 |
| Horizontal gel electrophoresis | BIO-RAD | Mini ReadySub-Cell GT Cell #1704487edu | Protocol 5 Step 5.5 |
| Mini microcentrifuge | Corning | LSE 6766 | Useful to quickly spin down PCR reaction tubes in protocols 4, 5 & 6 Step 6.5.1 |
| Microcentrifuge | LioFuge | LM-60 | Step 6.5.1 |
| qPCR machine and software | Thermo Fisher Scientific | 7500 Fast Real-Time PCR System with 7500 Software v2.0 | Protocol 6 Step 6.6-6.8 |
| qPCR machine and software | BIO-RAD | CFX96 Touch Real-Time PCR Detection System with CFX Manager software | |
| General Materials | |||
| Mayo scissors | Step 1.1-1.2 | ||
| Forceps | Step 1.1-1.2 | ||
| Pipette | Rainin | Pipette-Lite XLS | |
| Aerosol-barrier pipette tips | Sigma-Aldrich | Z333328, Z333336, Z333344 | |
| Nuclease-free 1.5-ml microcentrifuge tubes | Eppendorf |
参考文献
- FAO. . The State of World Fisheries and Aquaculture, 2014. Opportunities and Challenges. , (2014).
- FAO. . The State of World Fisheries and Aquaculture, 2016. Contributing to Food Security and Nutrition for all. , (2016).
- WorldBank. . FISH TO 2030: Prospects for Fisheries and Aquaculture. Agriculture and Environmental Services Discussion Paper 03. , (2013).
- Wing-Keong, N., Nicholas, R. A review of the nutrition and feeding management of farmed tilapia throughout the culture cycle. Reviews in Aquaculture. 5 (4), 220-254 (2013).
- Cleasby, N., et al. The socio-economic context for improving food security through land based aquaculture in Solomon Islands: A peri-urban case study. Marine Policy. 45, 89-97 (2014).
- Ponzoni Raul, W., et al. Genetic improvement of Nile tilapia (Oreochromis niloticus) with special reference to the work conducted by the WorldFish Center with the GIFT strain. Reviews in Aquaculture. 3 (1), 27-41 (2011).
- Hounmanou, Y. M. G., et al. Tilapia lake virus threatens tilapiines farming and food security: Socio-economic challenges and preventive measures in Sub-Saharan Africa. Aquaculture (Amsterdam, Netherlands). 493, 123-129 (2018).
- OIE. . Tilapia Lake Virus (TiLV) - a novel orthomyxo-like virus. OIE technical disease cards. , (2018).
- Mugimba, K. K., et al. Detection of tilapia lake virus (TiLV) infection by PCR in farmed and wild Nile tilapia (Oreochromis niloticus) from Lake Victoria. Journal of Fish Diseases. , (2018).
- Koesharyani, I., Gardenia, L., Widowati, Z., Khumaira, D. D., Rustianti, Studi kasus infeksi tilapia lake virus (tilv) pada ikan nila (Oreochromis niloticus). Jurnal Riset Akuakultur. 13 (1), 85-92 (2018).
- OIE. . Tilapia lake virus disease (TiLV), Chinese Taipei. Immediate Notification. , (2017).
- OIE. . Tilapia Lake Virus Disease (TiLV), Peru. Immediate Notification. , (2018).
- Bacharach, E., et al. Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia. MBio. 7 (2), e00431-e00416 (2016).
- Eyngor, M., et al. Identification of a novel RNA virus lethal to tilapia. Journal of Clinical Microbiology. 52 (12), 4137-4146 (2014).
- Nicholson, P., et al. Detection of Tilapia Lake Virus in Egyptian fish farms experiencing high mortalities in 2015. Journal of Fish Diseases. 40 (12), 1925-1928 (2017).
- Surachetpong, W., et al. Outbreaks of Tilapia Lake Virus Infection, Thailand, 2015-2016. Emerging Infectious Diseases. 23 (6), 1031-1033 (2017).
- Tattiyapong, P., Dachavichitlead, W., Surachetpong, W. Experimental infection of Tilapia Lake Virus (TiLV) in Nile tilapia (Oreochromis niloticus) and red tilapia (Oreochromis spp.). Veterinary Microbiology. 207, 170-177 (2017).
- Kembou Tsofack, J. E., et al. Detection of Tilapia Lake Virus in Clinical Samples by Culturing and Nested Reverse Transcription-PCR. Journal of Clinical Microbiology. 55 (3), 759-767 (2017).
- Thangaraj, R. S., et al. Derivation of two tilapia (Oreochromis niloticus) cell lines for efficient propagation of Tilapia Lake Virus (TiLV). Aquaculture (Amsterdam, Netherlands). 492, 206-214 (2018).
- Hanson, L. A., Rudis, M. R., Vasquez-Lee, M., Montgomery, R. D. A broadly applicable method to characterize large DNA viruses and adenoviruses based on the DNA polymerase gene. Virology Journal. 3, 28-28 (2006).
- Josko, D. Molecular virology in the clinical laboratory. Clinical Laboratory Science. 23 (4), 231-236 (2010).
- Munir, K., Kibenge, F. S. Detection of infectious salmon anaemia virus by real-time RT-PCR. Journal of Virological Methods. 117 (1), 37-47 (2004).
- Snow, M., et al. Developement, application and validation of a Taqman real-time RT-PCR assay for the detection of infectious salmon anaemia virus (ISAV) in Atlantic salmon (Salmo salar). Developments in Biologicals. 126, 133-145 (2006).
- Matejusova, I., McKay, P., McBeath, A. J., Collet, B., Snow, M. Development of a sensitive and controlled real-time RT-PCR assay for viral haemorrhagic septicaemia virus (VHSV) in marine salmonid aquaculture. Diseases of Aquatic Organisms. 80 (2), 137-144 (2008).
- Garver, K. A., et al. Development and validation of a reverse transcription quantitative PCR for universal detection of viral hemorrhagic septicemia virus. Diseases of Aquatic Organisms. 95 (2), 97-112 (2011).
- Dalla Valle, L., et al. Development of a sensitive and quantitative diagnostic assay for fish nervous necrosis virus based on two-target real-time PCR. Veterinary Microbiology. 110 (3-4), 167-179 (2005).
- Hodneland, K., Garcia, R., Balbuena, J. A., Zarza, C., Fouz, B. Real-time RT-PCR detection of betanodavirus in naturally and experimentally infected fish from Spain. Journal of Fish Diseases. 34 (3), 189-202 (2011).
- Hodneland, K., Endresen, C. Sensitive and specific detection of Salmonid alphavirus using real-time PCR (TaqMan). Journal of Virological Methods. 131 (2), 184-192 (2006).
- Wang, X. W., Ao, J. Q., Li, Q. G., Chen, X. H. Quantitative detection of a marine fish iridovirus isolated from large yellow croaker, Pseudosciaena crocea, using a molecular beacon. Journal of Virological Methods. 133 (1), 76-81 (2006).
- van Beurden, S. J., et al. Development and validation of a real-time PCR assay for the detection of anguillid herpesvirus 1. Journal of Fish Diseases. 39 (1), 95-104 (2016).
- Ciulli, S., et al. Development and application of a real-time PCR assay for the detection and quantitation of lymphocystis disease virus. Journal of Virological Methods. 213, 164-173 (2015).
- Tattiyapong, P., Sirikanchana, K., Surachetpong, W. Development and validation of a reverse transcription quantitative polymerase chain reaction for tilapia lake virus detection in clinical samples and experimentally challenged fish. Journal of Fish Diseases. 41 (2), 255-261 (2018).
- Dong, H. T., et al. Emergence of tilapia lake virus in Thailand and an alternative semi-nested RT-PCR for detection. Aquaculture (Amsterdam, Netherlands). 476, 111-118 (2017).
- Waiyamitra, P., et al. A TaqMan RT-qPCR assay for tilapia lake virus (TiLV) detection in tilapia. Aquaculture (Amsterdam, Netherlands). 497, 184-188 (2018).
- Behera, B. K., et al. Emergence of Tilapia Lake Virus associated with mortalities of farmed Nile Tilapia Oreochromis niloticus (Linnaeus 1758) in India. Aquaculture (Amsterdam, Netherlands). 484, 168-174 (2018).
- Ferguson, H. W., et al. Syncytial hepatitis of farmed tilapia, Oreochromis niloticus (L.): a case report. Journal of Fish Diseases. 37 (6), 583-589 (2014).
- Liamnimitr, P., Thammatorn, W., U-thoomporn, S., Tattiyapong, P., Surachetpong, W. Non-lethal sampling for Tilapia Lake Virus detection by RT-qPCR and cell culture. Aquaculture (Amsterdam, Netherlands). 486, 75-80 (2018).
- Yang, C. G., et al. Evaluation of reference genes for quantitative real-time RT-PCR analysis of gene expression in Nile tilapia (Oreochromis niloticus). Gene. 527 (1), 183-192 (2013).
- Bustin, S. A. Real-time, fluorescence-based quantitative PCR: a snapshot of current procedures and preferences. Expert Review of Molecular Diagnostics. 5 (4), 493-498 (2005).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27 (2-3), 126-139 (2006).
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27 (2-3), 95-125 (2006).
- Mackay, I. M., Arden, K. E., Nitsche, A. Real-time PCR in virology. Nucleic Acids Research. 30 (6), 1292-1305 (2002).
- Wong, M. L., Medrano, J. F. Real-time PCR for mRNA quantitation. Biotechniques. 39 (1), 75-85 (2005).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. Journal of Molecular Endocrinology. 25 (2), 169-193 (2000).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Research. 6 (10), 986-994 (1996).
- Rutledge, R. G., Côté, C. Mathematics of quantitative kinetic PCR and the application of standard curves. Nucleic Acids Research. 31 (16), e93-e93 (2003).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Amal, M. N. A., et al. A case of natural co-infection of Tilapia Lake Virus and Aeromonas veronii in a Malaysian red hybrid tilapia (Oreochromis niloticus × O. mossambicus) farm experiencing high mortality. Aquaculture (Amsterdam, Netherlands). 485, 12-16 (2018).
- Fathi, M., et al. Identification of Tilapia Lake Virus in Egypt in Nile tilapia affected by ‘summer mortality’ syndrome. Aquaculture (Amsterdam, Netherlands). 473, 430-432 (2017).
- OIE. . Tilapia Lake Virus disease (TiLV), Philippines. Immediate Notification. , (2017).
- OIE. . Tilapia lake virus disease (TiLV), Malaysia. Immediate Notification. , (2017).
- Abdullah, A., et al. First detection of tilapia lake virus (TiLV) in wild river carp (Barbonymus schwanenfeldii) at Timah Tasoh Lake, Malaysia. Journal of Fish Diseases. 41 (9), 1459-1462 (2018).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Del-Pozo, J., et al. Syncytial Hepatitis of Tilapia ( Oreochromis niloticus L.) is Associated With Orthomyxovirus-Like Virions in Hepatocytes. Veterinary Pathology. 54 (1), 164-170 (2017).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Purcell, M. K., Getchell, R. G., McClure, C. A., Garver, K. A. Quantitative polymerase chain reaction (PCR) for detection of aquatic animal pathogens in a diagnostic laboratory setting. Journal of Aquatic Animal Health. 23 (3), 148-161 (2011).
- Simpson, D. A., Feeney, S., Boyle, C., Stitt, A. W. Retinal VEGF mRNA measured by SYBR green I fluorescence: A versatile approach to quantitative PCR. Molecular Vision. 6, 178-183 (2000).
- Kibenge, M. J., et al. Discovery of variant infectious salmon anaemia virus (ISAV) of European genotype in British Columbia, Canada. Virology Journal. 13, 3 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved