Method Article
Tilapia 호수 바이러스를 사용 하 여 기존의 RT-PCR 및 SYBR의 탐지 RT 정량 그린
요약
이 프로토콜 tilapia 조직 RT PCR 방법론을 사용 하 여에 Tilapia 호수 바이러스 (TiLV)를 진단 한다. 전체 메서드 총 RNA 추출, cDNA 합성 및 기존의 PCR 또는 양이 많은 PCR dsDNA 염료를 형광 바인딩 바인딩을 사용 하 여 TiLV의 탐지를 조직 해 부에서 설명 되어 있습니다.
초록
이 방법의 목표 tilapia 조직에 Tilapia 호수 바이러스 (TiLV)의 빠르고, 과민 하 고 특정 검색을 용이 하 게 하는 것입니다. 이 프로토콜의 일부로 감시 프로그램, 생물 측정 그리고 TiLV 기초 연구 실험실에서 사용할 수 있습니다. 바이러스 진단의 황금 표준 일반적으로 바이러스 격리를 리버스 전사 연쇄 반응 (RT-PCR) 추가 확인 등 보완 기술 다음을 포함 한다. 이 성가신, 시간이 많이 소요 될 수 있습니다 하며 일반적으로 무 겁 게 바이러스에 감염 된 조직 샘플. RT-양적 (q) PCR 바이러스의 검출에의 사용 그것의 양적 성격, 높은 감도, 특이성, 확장성 및 결과에 그것의 급속 한 시간 때문에 유리 하다. PCR의 전체 방법 탐지는 tilapia 기관 단면, 총 ribonucleic 산 (RNA) 추출에서 guanidium 안산 클로 페 놀 프롬-솔루션, RNA 정량화, 뒤에 2 단계 PCR를 사용 하 여 설명 하는 TiLV에 대 한 접근을 기반으로 여기, 프로토콜을 수 반하는, 보완 deoxyribonucleic acid (cDNA) 합성 및 기존의 PCR 또는 양적 식별을 통해 정량 SYBR 녹색을 사용 하 여 TiLV의 감지 나 염료. 기존의 PCR PCR 게시물 단계 필요 하 고 단순히 바이러스의 존재에 대해 알릴 것 이다. 후자의 접근 방식을 조금 2 부 다운 TiLV의 절대 정량화에 대 한 고 따라서 하위 임상의 경우에서 TiLV 진단에 매우 유용. 두 개의 PCR 방법, 2 개의 실험실에서 대표 결과 대 한 자세한 설명 및 둘 다의 중요 한 매개 변수에 대 한 철저 한 토론 연구자와 diagnosticians 그들의 가장 적합 하 고 해당 찾을 수 있도록 포함 되었습니다. TiLV 검출의 방법입니다.
서문
글로벌 1 인당 물고기 공급 2014 년에서 20 kg의 새로운 기록을 도달 하 고 이것은 양식 업에 활발 한 성장 때문. 양식 전세계 급성장 동물 식품 생산 부문 중 하나 이며 인구1보다 더 빨리 성장 하 고 유일한 동물 식품 생산 부문. Tilapiine cichilds 6.4 백만 톤 (MT)의 총 세계 생산 및 201529.8 십억 미국 달러의 추정된 가치 전세계 농장 2 차로 가장 중요 한 민물 생선으로 구성 됩니다. Tilapia의 톱 10 생산자는 중국 (1.78 MT), 인도네시아 (1.12 MT)와 이집트 (0.88 MT), 방글라데시, 베트남, 필리핀, 브라질, 태국, 콜롬비아, 우간다2뒤. 글로벌 tilapia 생산 약 7.3 될 것으로 예상 된다 20303몬태나. 뿐만 아니라 단백질4 의 저렴 한 소스 뿐만 아니라 때문에 그들은 물과 기후 조건5,6의 다양 한 범위에서 용량에 번 식 하기 쉽기 때문에 tilapia 같은 중요 한 글로벌 식품 소스 되고있다. 그냥 몇 년 전, 몇 가지 상업적으로 중요 한 질병 tilapia 농업을 위협 했지만이 사실이 더 이상 믿어 졌다. Tilapia 호수 바이러스 질환 (TiLVD) 이라고 하는 신흥 바이러스 성 질병은 첫번째 이제까지 중요 한 질병 전염병 tilapia에서 발견 하 고 전체 산업은 위험에. 이 질병은 심각한 사회 경제적 결과 이며 아시아와 남미 아프리카7, 사람들의 수백만 위한 식량 안보에 직접적인 위협. 2018의 시작, 동물 건강 (OIE)에 대 한 세계 기구 보고 8 국가8 를 다루는 3 개의 대륙에 그리고이 병원 체 정보 카드는 TiLV,이 질병의 etiological 대리인 공식적으로 발견 되었습니다 했다 거기 업데이트 우간다9, 인도네시아10, 대만11 및 페루12탄자니아에 TiLV의 더 보고 되었습니다. TiLV은 다양 한 연상 독감 이나 전염 성 연어 빈혈증 바이러스 (ISAV)13등 다른 orthomyoxoviruses 특성을 포함 하기 때문에 orthomyxo 같은 바이러스를 되도록 설명 소설 단일 가닥 RNA 바이러스. 그것은 먼저 갈릴리 호수, 이스라엘14야생 및 농장 tilapia의 대규모 손실의 여파에 확인 되었다. 그 후, 비슷한 질병 발생 추천 여름 mortalities 및 TiLV 감염 연관 사망률 증후군 나 일 틸 라 피아 (Oreochromis niloticus) 이집트15 에 나 일에 빨간색 하이브리드 tilapia 보도 했다 한 달 (Oreochromis 종) 태국16, 각각. 수생 동물 바이러스의 검출은 역사적으로 성장 및 세포 배양에서 바이러스의 분리에 의해 수행 됩니다. 다양 한 셀 라인 전파 및 TiLV 포함, E-11 셀 독 사 물고기 (Ophiocephalus striatus)17,18에서 파생 된 OmB Oreochromis 에서 발생 하는 TmB의 격리에 대 한 테스트 mossambicus18, OnlB 및 OnlL 나 일 틸 라 피아 (O. niloticus)19에서 발생. 바이러스 문화는 추가 실험에 대 한 자료를 제공 한다는 이점이 있다, 그러나 그것은 cytopathic 효과 (CPE)의 형성을 관찰 하기 위해 적어도 4-7 일을 요구 하 고 결정적인 다른 시세 바이러스는 더 적합 하는 단점이 있다 복제는 전파 될 수 있습니다 하 고 유사한 CPE 생산.
지난 몇 십년에 세포 배양, 혈 청 학 및 항 원 탐지 및 신속 하 고 더 민감한 핵 산 기반 탐지 테스트20, 교체 등 전통, 수시로 시간이 걸리는 진단 방법 멀리 이동이 되었습니다. 21. 이것은 많은 정량 분석 실험 개발 되었습니다 다양 한 수생 동물의 바이러스 성 질병에 대 한 중요 한 진단 방법으로와 같은 ISAV22,23, 바이러스 haemorrhagic septicaemia 바이러스 (VHSV)24에 대 한 사실에 의해 분명 하다 ,25, betanodavirus26,27 salmonid alphavirus28, 물고기 iridovirus29, Anguillid herpesvirus 1 (AngHV1)30및 Lymphocystis 질병 바이러스 (LCDV)31 . 진단 및 병원 체 감시에 대 한 강력한 방법 긴급 TiLV의 확산을 줄일 필요가 있습니다. 이러한 방법을 임상 증상을 개발 하기 전에 감염의 조기 발견 및 낮은 바이러스 부하의 검출을 허용 해야 합니다. 날짜 하려면, RT-PCR14,32를 포함 하 여 다른 PCR 프로토콜 중첩된 RT-PCR18, 세미 중첩된 RT-PCR33, 그리고 실시간 정량32,34 개발 되었습니다 TiLV의 검출에 대 한 물고기의 조직에서 TiLV 검색 취약 셀 라인에서 실시간 정량 및 바이러스 격리의 비교 계시 실시간 정량 바이러스 격리32보다 1000 배 더 과민 했다. 각 게시 된 PCR 프로토콜은 TiLV의 검출을 위한 다른 감도 보고, 대부분 분석 매우 민감한 7.5 복사본33, 7 복사본18 또는 당 2 부32 바이러스의 검출 한계는 반응입니다.
이 방법 문서의 목표는, 자세히, tilapia 조직 컬렉션, 총 RNA 추출, cDNA 합성 시작 TiLV 탐지 분석 실험을 수행 하는 방법을 설명 하 고 TiLV 특정 PCR 분석 실험을 기반으로. 특히, 기존의 RT-PCR와 또한 SYBR 녹색 기반 실시간 정량의 포괄적인 프로토콜 TiLV를 감지 하는 것을 목표로 하는 과학자의 넓은 범위에 호소를 설명 있다. 전 덜 민감 하지만 일반적으로 더 싼 검색 옵션. 후자 정량 PCR 기계 등 더 비싼 시 약, 더 정교한 인프라를 필요로 하지만 그것은 양적, 신속 하 고 매우 중요 한, 그것은 사용할 수 있는 TiLV에서의 검출에 대 한 하위 임상 의미의 이점이 있다 물고기를 감염. RT-PCR 및 RT-정량 Pcr 프로토콜 TiLV 및 포함된 결과 강조 감도의 명료한 지리적 격리와 여기에서 설명 하는 분석 실험의 재현성 두 다른 실험실에서 수행 했다.
프로토콜
이 연구의 동물 사용 프로토콜 허가 번호 ACKU 59-수 의사-016 Kasetsart 대학 동물 윤리 위원회에 의해 승인 되었다.
참고: 확장 된 사항은 시 약 및 장비가이 프로토콜에 대 한 제안에 대 한 테이블의 자료 를 참조 하십시오.
1. 조직 샘플 컬렉션
- 정 향 오일 (볼륨에 따라 물고기의 크기와 농도의 제품, 보통 이상의 3 mL/L)의 과다를 사용 하 여 물고기 안락사 95% (v/v) 에탄올 알코올 버너를 사용 하 여 장비를 소독 하는 장비를 레코딩하여 뒤에 집게와 메이 요가 위 1-4를 담가.
참고: Tricaine methanesulfonate (MS-222) 정 향 기름 대신 사용할 수 있습니다. - 찾아 간 작은 조각 (약 20-100 mg)을 잘라 또는 후부 tilapia 물고기의 앞쪽에서 점액을 제거 하 고 샘플 1.5 mL microcentrifuge 튜브를 커버 유리 또는 외과 블레이드를 사용 하 여 점액의 200 µ L를 수집.
- 프로세스 샘플 즉시, RNA 안정화 솔루션에에서 저장 하거나 추가 사용까지-80 ° C로 이동.
참고: RNA 작업에 가장 큰 작업 준비 그대로 RNA 분자 이며 그들을 유지 하는 모든 후속 보정을 통해 손상 되지 않은. RNA 백본 innately DNA 보다 손상에 더 과민 하다. 추출 및 조직 세포에서 총 RNA의 격리 필요로 조심 실험실 기술; 오염을 방지 하기 위해 RNase RNase 무료 물, 시 약, 장비, 플라스틱 도자기, 유리, 작업 공간을 사용 하 여 장갑을 끼고 고 pipetting에 대 한 필터 팁을 사용 하 여 모든 규정을 가져가 라.
2. Guanidium 안산-페 놀-클로 프롬 RNA 추출
- 섹션 1에서에서 조직 샘플을 포함 하는 관으로 페 놀 및 guanidine isothiocyanate를 포함 하는 monophasic 솔루션의 1 mL를 추가 합니다.
주의:이 솔루션은 매우 독성이 고 보호 장비와 적절 한 보호 안경, 의류, 안전 장갑 착용 하 여 층 류 두건에 관리와 처리 해야 합니다. - 갈기는 조직 유 봉 균질 화기까지 균질를 사용 하 여 조직 샘플.
참고: 샘플 수 또한 될 무 균 세라믹 구슬 함께 전원 균질 화기를 사용 하 여. 조직 샘플은 다음 단계를 진행 하기 전에 완전히 무 균을 확인 하거나 프로토콜 여기 중지 하 고 추가 사용까지-80 ° C에서 완전 무 균된 샘플을 저장. - 단계 구분에 대 한 클로 프롬의 200 µ L를 추가 합니다.
주의 클로 프롬 잠재적인 마약 이며 매우 위험한. 그것은 층 류 두건 보호 장비 뿐만 아니라 적절 한 보호 안경, 의류, 안전 장갑 입고 관리와 함께 처리 되어야 합니다. 적은 독성으로 대체, 1-브로 모-3-chloropropane 사용할 수 있습니다.
참고: 확장 볼륨을 위 또는 아래로 적절 한. 예를 들어 포함 된 페 놀 및 guanidine isothiocyanate monophasic 솔루션의 500 µ L를 사용 하는 경우에 다음만 추가 100 µ L 클로 프롬의이 단계에서.- 샘플 15 반전 하 여 잘 혼합 s.
- 실 온 (RT)에서 3 분에 대 한 샘플을 품 어.
- 12000 × g 에서 15 분 및 4 ° c.에 대 한 원심 분리기
참고: 낮은 유기 단계, 흰색 interphase 및 RNA를 포함 하는 위 수성 단계에는 명확 하 게 구분을 하 고 있어야 합니다. 이 상위 단계는 일반적으로 무색 하지만 종류와 무 균된 조직의 금액에 따라 밝은 분홍색 외관을 가질 수 있습니다. - Interphase는 방해 하지 않고 신선한 microcentrifuge 튜브 위 수성 층 (약 500 µ L) 전송.
참고: 전체 수성 단계 전송, 유기와 수성 단계를 포함 하는 RNA의 어떤 잠재적인 오염을 방지 하기 위해 작은 금액을 두고 또는 interphase 하려고 하지 마십시오. - RNA 침전을 100% 소 프로 파 놀의 1 볼륨을 추가 합니다.
- 선택적으로, 아주 적은 양의 조직 사용한 경우 다음 추가 1 µ L (5-10 µ g) RNase 무료 glycogen의 효율적 RNA 강 수를 홍보 하는 각 샘플을. 이 단계 2.8에서에서 RNA 펠 릿의 식별을 도움이 됩니다.
참고: 있는 glycogen RNA의 운반대로 작동 하 고 튜브의 측면에 집착에서 RNA의 소량을 방지할 것 이다. - 믹스 전도 의해 잘 여러 번 튜브합니다.
- -20 ° c.에 하룻밤 2 h에 대 한 샘플 저장
- 선택적으로, 아주 적은 양의 조직 사용한 경우 다음 추가 1 µ L (5-10 µ g) RNase 무료 glycogen의 효율적 RNA 강 수를 홍보 하는 각 샘플을. 이 단계 2.8에서에서 RNA 펠 릿의 식별을 도움이 됩니다.
- 12000 x g 와 4 ° C에서 15 분 동안 원심 분리기 샘플
- Microcentrifuge 튜브의 하단에 RNA 펠 릿을 꺼내 려 하지 않도록 주의 되 고 상쾌한을 삭제 합니다.
- 75% 에탄올 (v/v)의 1 mL을 추가 하 고 여러 번 튜브를 거꾸로 하 여 RNA 샘플을 혼합.
- 10000 x g 와 4 ° C에서 15 분 동안 원심 분리기
참고: 프로토콜 여기 중지 될 수 있습니다 그리고 75% 에탄올에 RNA 펠 릿으로 구성 된 샘플 추가 사용까지-20 ° C에서 저장 될 수 있다. - Microcentrifuge 튜브의 하단에 RNA 펠 릿을 꺼내 려 하지 감시 되 고 상쾌한을 삭제 합니다.
- 필요에 따라 반복 단계 2.9-2.11 70% 에탄올 (v/v)를 사용 하 여. 철저 하 게 어떤 소금 최소화 RNA 펠 릿을 세척 또는 방해 오염 이성 민감한 다운스트림 응용 프로그램을 것입니다.
- 피 펫을 사용 하 여 나머지 에탄올 밖으로 그리고 5 ~ 10 분 보다는 더 이상에 대 한 실 온에서 RNA 펠 릿 건조 합니다.
참고:-건조 펠 릿을 다시 중단 어려울 것 이다. - 30-60 µ L RNase 무료 물, RNA 펠 릿을 solubilize를 55-60 ° C에 미리 예 열을 추가 합니다.
- 즉시 사용 하거나 나중에 사용-80 ° C에 게 얼음에 RNA를 놓습니다.
3. 계량 마이크로 볼륨 분 광 광도 계를 사용 하 여 RNA 농도
- RNA는 분 광 광도 계의 설정을 전환 합니다.
- RNase 무료 물 1-2 µ L를 사용 하 여 빈으로.
- 각 RNA 샘플의 1-2 µ L를 사용 하 여 RNA 수량 평가.
- 230에서 판독 기록 nm, 260 nm와 280 nm 각 샘플에 대 한.
- 200 RNA 희석 ng / µ L RNase 무료 물을 사용 하 여.
4. 총 RNA를 사용 하 여 보완 DNA (cDNA)의 합성
- 프로토콜 2, 2 µ M 올리고 (dT), 0.5 m m dNTPs 혼합물에서에서 총 RNA의 1 µ g을 혼합 하 고 nuclease 무료 물으로 10 µ L를 최종 볼륨을가지고. 이 위해, RT-마스터-혼합 샘플 및 컨트롤 수에 따라 테스트를 준비 합니다.
참고: 컨트롤은 마이너스-역전사 샘플 (-RT) 어떤 점에서 RT 효소 nuclease 무료 물으로 대체 됩니다 (단계 4.3 참조)와 아무 템플릿 컨트롤 (NTC) 어떤 점에서 nuclease 무료 물 RNA 서식 파일 대신 마스터 믹스에 추가 됩니다.- 잘 뒤에 간단한 원심 pipetting으로 샘플을 믹스.
- 5 분 2 분 부 화 뒤에 얼음에 65 ° C에서 샘플을 품 어.
- 짧게 원심 튜브의 하단에 있는 액체의 모두 수집 하도록 샘플.
- 1 x 역전사 버퍼, 100 U 역전사 추가 각 샘플의 최종 볼륨 20 µ L nuclease 무료 물을 사용 하 여.
- 잘 뒤에 간단한 원심 pipetting으로 샘플을 믹스.
- 42 ° C 60 분 5 분 85 ° c에서 샘플을 품 어.
- 물 nuclease 무료의 적절 한 볼륨을 추가 하 여 원하는 농도에 합성된 cDNA를 희석 즉각적인 사용을 위해 얼음에는 cDNA를 장소 또는 나중에 사용-20 ° C에서 그것을 저장.
5. TiLV 기존의 PCR
- CDNA, 샘플 및 컨트롤, 템플릿 설립된 뇌관 쌍 선택의 DNA 중 합 효소와 함께 표 1에 자세히 설명 중 하나를 사용 하 여 PCR 반응에 대 한 프로토콜 섹션 4에서에서 생성 된 사용 합니다.
참고: 추가 없음-템플릿 컨트롤 (NTC) nuclease 무료 물이 PCR 반응에 대 한 cDNA를 대체 하 여 여기 포함 되어야 합니다. 긍정적인 통제, 가능 하다 면, 또한 이전 확인된 TiLV 긍정적인 샘플 구성 포함 되어야 합니다 또는 적절 한 TiLV cDNA 조각 한 플라스 미드에 복제 합니다. - 샘플 및 컨트롤을 테스트할 수 및 사용 중인 DNA 중 합 효소 시스템의 지침에 따라 PCR 마스터 믹스를 준비 합니다. 이 믹스는 앞으로 뇌관, 역방향 뇌관, dNTPs, MgCl2 와 선택 된 DNA 중 합 효소, 버퍼 함께 포함 되어야 합니다.
- 선택 된 DNA 중 합 효소의 지침에 따라 마스터-믹스의 볼륨을 지정 된 cDNA 샘플 및 제어 샘플의 제안된 금액 결합 되어 있습니다.
참고: 반응 초과 x 0.5 준비는 자주 도움이 이후 마스터-믹스의 일부 pipetting 동안 손실 됩니다. - 자전거 사용된 DNA 중 합 효소 시스템의 지침에 따라 조건 및 사용 (표 1) 프라이 머에 대 한 적절 한 어 닐 링 온도 사용 하 여 PCR을 수행 합니다. 일반적으로, 이러한 프로그램 30 95 ° C에서 변성의 30-40 주기 다음 2-5 분 동안 95 ° C에서 초기 변성을 포함 한다 s, 30 s 및 30에 대 한 72 ° C에서 신장에 대 한 권장된 온도에서 어 닐 링 s, 72 ° C 5-10에 최종 신장 뒤 분입니다.
- 각 PCR 반응 및 예상된 PCR 제품의 크기에 따라 1-2 %agarose 젤의 우물에는 적절 한 DNA 사다리의 5-15 µ L을 로드 합니다. 젤 전기 이동 법에 의해 증폭 된 DNA를 분리 하 고 자외선을 사용 하 여 젤 설명서 기계에서 젤 ethidium 평범한 사람 (EthBr)의 예상된 크기 (표 1) DNA 밴드의 시각화를 용이 하 게 하 여 얼룩.
주의: EtBr은 독성; 그것은 치료와 함께 적절 한 보호복 및 안전 장갑을 착용 하 여 처리 되어야 합니다.
| 대상 TiLV 게놈 세그먼트 | 앞으로 뇌관 5'-3 ' | 반전 뇌관 5'-3 ' | PCR 제품 크기 (bp) | Tm ° C | 원래 참조 | ||||
| 1 | CCAAACGTTATCTCTTAATTACGCAC | GCAAATATTTCTCTCATTCGCCT | 1641 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 1 | CCTCATTCCTCGTTGTGTAAGT | AGGAGTTGCTGTTGGGTTATAG | 1000 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 2 | ACTCTCTATTACCAAATACATTTACT | TTACCATATATATAGTGAAGGC | 1445 | 45 | Surachetpong 그 외 여러분, 2017 | ||||
| 2 | GTCCAGGGCGGTATGTATTG | CTTACGGCTGACAAGTCTCTAAG | 834 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 3 | GTTGGGCACAAGGCATCCTA | TATCACGTGCGTACTCGTTCAGT | 250 | 56 | Eyngor 그 외 여러분, 2014 | ||||
| 3 | TATGCAGTACTTTCCCTGCC | TTGCTCTGAGCAAGAGTACC | 491 | 57 | Eyngor 그 외 여러분, 2014 | ||||
| 3 | ACCCCTTAATCCTTAATAGACCGTTA | CCCATAATCCTCTATTAGAACGTCGT | 1352 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 3 | GTCGAGGCATTCCAGAAGTAAG | GAGCTAAGGGAACGGCTATTG | 834 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 4 | AGCAGCAGCAGGAGAAAGAG | ACCGTCCTGTTTCTGAATGG | 358 | 60 | 니콜 슨 외., 2017 | ||||
| 4 | CCAAAGTTTACTCCTATTACCCAGA | GCAAATCTTTCTCCAATTACCGTCT | 1250 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 4 | GCCCAATGGTTCCCATATCT | GCCCAATGGTTCCCATATCT | 524 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 5 | CCAAATGTTTCTCTTATCTCAGACTC | CTTTTTCTCAGTTTACCACTTTATG | 1087 | 57 | Surachetpong 그 외 여러분, 2017 | ||||
| 5 | CAACTCTTAGCCTCCGGAATAC | CGTTCTGCACTGGGTTACA | 696 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 6 | CCAAATTTTACCTCTCGCAT | TCAAGCACTTAAAACTGTACC | 1027 | 45 | Surachetpong 그 외 여러분, 2017 | ||||
| 6 | CCCACACGACAGGACATATAG | GAGTTGGCTTAGGGTGATAAGA | 948 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 7 | CTCTCTTTGCATTGCATACCGT | GACCAATTATCCCTGCTTTCA | 704 | 57 | Surachetpong 그 외 여러분, 2017 | ||||
| 7 | TCCTTTAGGGATTGGCACTAAC | TTCCATCGACTGCTCCTAGA | 486 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 8 | ACCTCATCTACACTAACATTTCCA | TCATCATTACACAAATGGAGTAGCT | 637 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 8 | CTTAAGGGCCATCCTGTCATC | TGGCTCAAATCCCAACACTAA | 476 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 9 | TTGGTGATGTCACGATGGATA | AGTTCTATCGCCAGCCATGT | 351 | 60 | 니콜 슨 외., 2017 | ||||
| 9 | ACAAGTCCGATTACTTTTTCCGC | TCTTTCTCACGTCCTTAAAGTCA | 530 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 9 | GATATCCTCCACATGACCCTTC | GTACGTCACTTTGTGCCATTAC | 261 | 62 | Mugimba 그 외 여러분, 2018 | ||||
| 10 | AACCCTACTAACACCAAATATAGCT | CTTTCCCTCTGACACCCTGT | 450 | 50 | Surachetpong 그 외 여러분, 2017 | ||||
| 10 | TCCTCTCTGTCCCTTCTGTT | CAGGATGAGTGTGGCAGATTAT | 276 | 62 | Mugimba 그 외 여러분, 2018 | ||||
표 1. 끝점 PCR를 사용 하 여 TiLV cDNA의 증폭에 대 한 게시 된 뇌관 쌍. 굵게 표시 된 설정 뇌관 그림 3A 및 3B에 표시 하는 대표적인 결과 생성 하 사용 되었다.
6. TiLV 정량적 중 합 효소 연쇄 반응 (정량)
- 적절 한 TiLV를 포함 하는 플라스 미드를 사용 하 여 pTiLV32, 같은 표준으로 게놈 세그먼트 3 cDNA 중복 또는 triplicated 10 직렬 희석 시리즈 준비.
- 모든 샘플, 표준 및 컨트롤에 대 한 정량 마스터-믹스를 준비, 복제 또는 triplicate nuclease 무료 물 0.4 µ L, 앞으로 뇌관의 0.3 µ L, 역방향 뇌관의 0.3 µ L, SYBR 녹색 x 2의 5 µ L을 이용 하는 반응에서 수행 해야 하는 계정에 Dna 중 합 효소 반응 당 마스터 믹스입니다.
- 10 µ M 및 뇌관 정보 및 표준 pTiLV의 농도에 프라이 머를 다음과 같이 사용.
뇌관을 앞으로: TiLV 112F (5'-CTGAGCTAAAGAGGCAATATGGATT-3')
반전 뇌관: TiLV-112R (5'-CGTGCGTACTCGTTCAGTATAAGTTCT-3')
표준 pTiLV:10 pg / µ l
참고: 샘플 및 컨트롤의 총 수는 10 triplicates에서 수행 될 경우이 정량 마스터-믹스 12 µ L nuclease 무료 물, 9 µ L 앞으로 뇌관, 9 µ L 역 뇌관 및 SYBR 녹색 DNA 중 합 효소 마스터-믹스의 150 µ L에과 동일한. 상업적으로 보편적인 SYBR 녹색 DNA x 2 구입 중 합 효소 마스터-믹스 포함 모든 필요한 구성 요소 정량 반응에 대 한 즉, SYBR 녹색 나 염색, 핫 스타트 Taq DNA 중 합 효소, dNTPs, MgCl2 고 수동 참조 염료. 빛에서 SYBR 녹색 마스터 믹스를 보호 합니다.
- 10 µ M 및 뇌관 정보 및 표준 pTiLV의 농도에 프라이 머를 다음과 같이 사용.
- 정량 Pcr 스트립 튜브 또는 96 잘 접시 사용에서 정량 Pcr 기계와 호환으로 정량 마스터-믹스의 6 µ L을 분배.
- 튜브 또는 96 잘 접시의 우물에 cDNA 템플릿, 컨트롤 또는 순차적으로 희석된 TiLV 표준의 4 µ L를 추가 합니다.
- 정량 Pcr 튜브를 닫거나 사용에서 정량 Pcr 기계에 대 한 호환 플레이트 커버 96 잘 접시를 봉인
- 부드럽게 터치 하는 혼합 솔루션 및 정량 튜브 또는 96 잘 접시 모든 혈관의 바닥에 액체를 수집 하는 원심 분리기를 사용 하 여 스핀 정량 튜브.
- 실시간 열 cycler에 튜브 또는 접시를 놓습니다.
- 95 ° C의 40 주기 다음 3 분 동안 95 ° C에서 초기 변성을 수행 하기 위해 정량 thermocycler 프로그램 10 s와 30에 대 한 60 ° C에 대 한 뇌관 어 닐 링 및 신장, 녹는 곡선 단계 65 ° c 95 ° C를 0.5 ° C의 증가 함께 끝나는 s / 5 s입니다.
- SYBR fluorophore 염료로 다음 샘플 형식으로 알 수 없는 선택 선택한 샘플 이름 상자에 이름을 삽입 합니다.
- 실시간 정량 Pcr 기계의 뚜껑을 열고 할당 된 우물에 정량 스트립을 놓습니다 다음 뚜껑을 닫습니다.
- 선택한 조건 실시간 정량 분석을 수행 합니다. 기계는 뚜껑 원하는 온도 도달 하면 실행을 시작 합니다. 반응의 진행 상황을 모니터링할 각 확장 단계 후에 각 샘플의 형광을 수집 합니다.
참고: 정량 Pcr 기계 및 관련된 소프트웨어 자동으로 분석 결과의 모든 매개 변수를 계산 하 고 실시간으로 증폭 곡선 표시, 표준 곡선과 용융 곡선은 정량 사이클의 끝에 생성 됩니다. - 각 샘플에 대 한 데이터 분석 및 곡선 녹는 첫 함으로써 수집을 수행 하 고 표준은 amplicon에 대 한 예상된 온도에서 균일 한 피크를가지고.
- 샘플의 증폭 곡선을 평가 하 고 표준 및 설정 영역에서 임계값은 cDNAs의 증폭 율은 모든 샘플에서 동일 했다. 이것은 일반적으로 소프트웨어에 의해 자동으로 수행 하지만 신중 하 게 검사 되어야 한다.
- 표준 곡선을 사용 하 여 TiLV 사본의 수를 계산 합니다.
결과
섹션 1에에서 설명 된 프로토콜에 따라 죽어가는 빨간 하이브리드 tilapia TiLV 감염 (그림 1A)의 임상 표시를 표시는 입욕 마 취 역할 정 향 오일의 높은 농도에 의해 안락사 했다. 보고 된 임상 증상은 변수 하지만 일반적인 증상 혼 수, 피부 부식 및 변색, exophthalmia, 분리 된 비늘, 오픈 상처/병 변 및 비정상적인 행동15,,1633, 표시 35,36, 이들 중 일부 볼 수 있는 명확 하 게 그림 1A. 복 부 벽은 내부 장기는 간, 비장, 또는 머리 신장 (그림 1B) 등을 수집 하 제거 되었습니다. 점액 샘플 또한 커버 유리 또는 외과 블레이드37을 사용 하 여 물고기의 후부를 앞쪽에서 피부를 부드럽게 된다고 하 여이 단계에서 수집 되었습니다.

그림 1 . Tilapia 해 부 및 샘플 수집. A. 피부 leisons, 입 및 operculum, 피부 부식 및 각 막 불투명도 빨갛게와 빨간 하이브리드 TiLV 감염 tilapia. B. Sectioned 빨간색 하이브리드 tilapia (파란색 화살표), 지점 간 비장 또는 머리 신장 장기에서 조직 컬렉션에 대 한 허용. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
그 후, 제 2 Guanidium 안산-페 놀-클로 프롬 총 RNA의 추출에에서 대 한 자세한 프로토콜 뒤를 이었다 고 순도 비율의 계산에 의해 RNA 정량화 섹션 3에에서 설명 된 대로 샘플 순수성 평가를 수행한 및 스펙트럼 프로필 (그림 2)의 시험. 그림 2A 그림 2B 나타냅니다 가난한 RNA 준비 하는 동안 성공적인 총 RNA 추출 절차에서 대표적인 결과 보여 줍니다. 핵 산 260에서 흡 광도 맥시 마는 동안 단백질 그들의 280에 nm. 260에 측정 비율 nm 및 280 nm 각 샘플의 순도 나타내고 비율 1.9 2.1의 그림 2A에서 샘플에 대 한 경우 순수 RNA를 나타냅니다. 낮은 A260/280 비율 그림 2B 에서 관찰 가능한 단백질 또는 페 놀 오염을 RNA 추출 절차에서 남은 나타냅니다. 230에서 흡 광도 nm 샘플 오염의 결과 수와 A260/230 nm 비율 또한이 계산 됩니다. 이 비율은 1.07의 낮은 A260/230 비율은 그림 2B와 스펙트럼 프로필 230에서 여 물통에 변화를 표시 하는 동안 그림 2A, 샘플 2.03의 값에 의해 그림과 같이 2.0-2.2 순수 RNA 준비의 범위에 있어야 240으로 nm 잔여 guanidine 또는 샘플에서 페 놀의 지표는 nm 그림 2B의 샘플에 대 한 다시 계기 오염을 제거 하는 RNA 샘플의 순도 높일 수 있습니다.
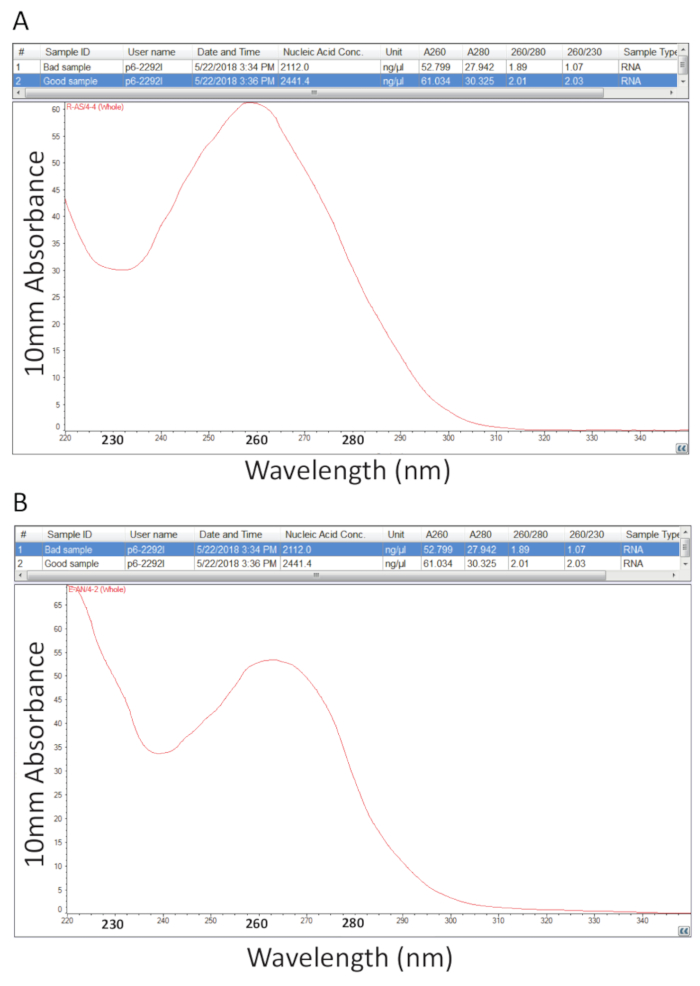
그림 2 . Spectrophotometric 병 tilapia 조직에서 추출한 총 RNA의 정량화. A. 순도 비율 및 성공적인 RNA 준비에서 스펙트럼 프로필. B. A로, 가난한 RNA 추출 절차의 대표를 제외 하 고. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
감지 RT-PCR에 의해 TiLV, 그림 2A에 표시 하는 나와 같은 순수한 샘플 역방향 베낀된 (프로토콜 4) cDNA로 되었고 사용 하는 PCR에 대 한 서식 파일에서 섹션 5 시험 하 고 대표적인 결과 3A를 그림에 표시 됩니다. 표 1 에서 뇌관에 표시 된 굵은 TiLV 게놈 세그먼트 314491 bp 파편을 증폭 하는 데 사용 했다. PCR 제품 젤 전기 이동 법으로 분리 되었고 EtBr 시각화를 위한 물. 그림 3A 에서는 2 단계 RT-PCR의 결과 4 cDNA 샘플 (S1 ~ S4), 태국, 그리고 각 샘플, 약 500의 깨끗 한 단일 밴드 격리 병 tilapia의 간에서 파생 된을 사용 하 여 혈압을 관찰 될 수 있으며 따라서, 샘플 1-4 TiLV 긍정적인. 같은 PCR 제품 TiLV 세그먼트 3의 cDNA 없습니다 템플릿 컨트롤 (NTC) PCR 제품을 생성 하지 않았다 하는 동안32 플라스 미드 복제를 구성 하는 긍정적인 컨트롤 샘플에서 얻은 했다. 그림 3B 에서 분석 결과 그림 3A 와 같이 하지만 원스텝 RT-PCR 방법을 사용 하 여 다른 실험실에서 및 tilapia 이집트 양식에서의 머리 신장 조직에서 파생 된 5 RNA 샘플 같은 뇌관을 사용 하 여 수행한 15. 예제 2 및 4는 부정적인 TiLV 때문에 PCR 제품이 올바른 크기로 발견 샘플 1, 3, 5는 긍정적인 TiLV이 검출 분석 결과 사용 하 여 결정 했다. 부정적인 컨트롤 역전사 컨트롤 및 두 개의 NTCs 마이너스 2 등 했 어 생성 하지 어떤 PCR 제품. 원스텝 RT-PCR 분석 결과 또한 tilapia ActinB 유전자를 대상으로 수행 되었다. 217의 amplicon 크기 bp 예상된38모든 샘플 (S1-s 5)에서 생성 했다. 이 분석 결과 RNA 샘플의 무결성에 대 한 제어 뿐만 아니라 TiLV 긍정적인 샘플의 반 정량 검사에 대 한 허용으로 재직 했습니다. 생성 된 Tilapia ActB 제품은 상대적으로 동등한, TiLV 특정 PCR 제품 생성의 차이 주어진된 조직 샘플에서 TiLV의 금액의 진정한 반성으로 해석할 수 있다.
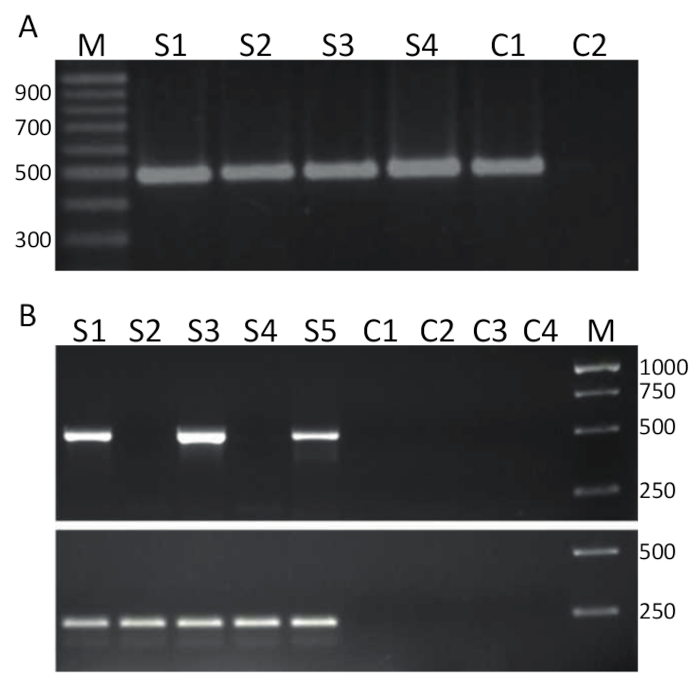
그림 3 . TiLV RT PCR A. 병 tilapia의 간 조직에서 생산 하는 cDNA 샘플 수집 태국에서 세그먼트 3에 대 한 특정 뇌관을 사용 하 여 TiLV 감염에 대 한 화면을 했다 (에 표시 된 표 1에 굵게) 2 단계 RT-PCR 분석 결과 사용 하 여 TiLV의. M = 기본적인 쌍;에 표시 된 마커 S1-S4 = 샘플 1-4; C1 = PCR 템플릿으로; pTiLV을 사용 하 여 긍정적인 컨트롤 및 C2 = 템플릿 제어 (NTC). B. 원스텝 RT-PCR A에서 동일한 뇌관을 사용 하 여 이집트15병 tilapia의 머리 신장 조직에서 샘플을 수집 하 고. M = 기본적인 쌍;에 표시 된 마커 S1-s 5 샘플 1-5 =. 컨트롤 C1-C2 역전사 컨트롤 마이너스 이며 C3-C4 NTCs 하단 패널 원스텝 RT-PCR 뇌관 tilapia ActinB38 (자세한 내용은 참조 텍스트) 생산에 대 한 지시를 사용 하는 217의 기본적인 쌍의 PCR 제품. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
끝점 PCRs 그림 3, 정량 분석 실험 프로토콜, 6에서에서 설명에서 달리 각 PCR 주기 후 PCR 제품의 크기를 측정 합니다. 표적 DNA의 증폭 반응의 각 라운드에서 생성 된 DNA와 상호 작용 하는 형광 분자를 사용 하 여 검색 됩니다. 여기, SYBR 녹색 나 염색 이용 되었다는 이중 가닥 DNA와 intercalates. 형광 신호 반응 동안 뒤 고 강도에 관한 제품의 양을 형성 39,40,41,,4243. TiLV 정량 분석 실험 프로토콜 다른 나라에서 다른 SYBR 녹색 시 약, 정량 Pcr 기계 및 샘플을 사용 하 여 다른 실험실에서 6에에서 설명 된 대로 실행 되었다. 결과 증폭 곡선 그림 4A 와 4B에 표시 됩니다. 각 분석 결과 대 한 실험의 과정 4 단계는 관찰 될 수 있다: 선형 지상 단계 초기 지 수 단계, 후반 지 수 단계, 고원 단계. 선형 지상 단계는 DNA 복제 DNA 수량 부족 신호/배경 비율 생산 때문 식별할 수 아직 없는 초기 주기 동안 발생 합니다. 기준선 형광이 단계 계산 됩니다. 그 후, 표적 DNA 농도에서 두 번 배경 위에서 감지 되 고 기 하 급수적으로 증가 신호 유도 각 주기 시작 합니다. 증폭 효율 (E) 잘 최적화 된 정량 분석 결과의 이다 (100%)에 가까운 매우 높은 증폭 및 그것의이 초기 지 수 단계 남아 안정 하 고 반응의 시작은이 시점에서 그 정량화를 수행 때 반응 효율은 여전히 꾸준한. 이후 주기 신호 고원, 시작 하 고 형광의 강도 시작 템플릿 복사 수 상관 이상 반응 구성 요소는 지쳐44. 채도는 DNA 기질 분자를 다시 어 닐 링 반응, 구성 요소, 또는 효소 단위 금액의 변화 농도 비율에서에서 경쟁으로 인해 발생할 수 있습니다. 아마도, 이러한 매개 변수는 그림 4A 와 4B에 표시 된 분석 실험에 대 한 증폭 곡선의 차이점에 대 한 계정. 포함 된 컨트롤은 이러한 특성 증폭 곡선 생성 하지 않았다.
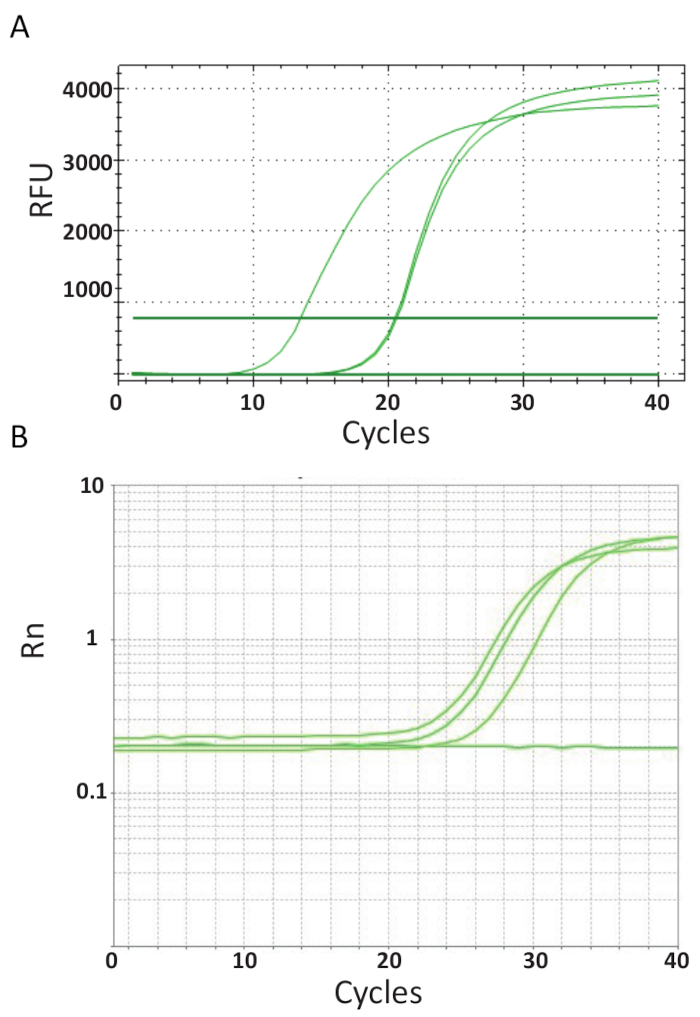
그림 4 . 증폭 표시 실시간 PCR 분석 결과의 기간 동안 제품의 축적을 플롯 합니다. A. TiLV-긍정적인 샘플의 증폭 곡선에서 파생 된 태국, NTCs, 및 긍정적인 플라스 미드 제어 SYBR 녹색을 사용 하 여 내가 2 단계 정량 분석 결과. 차트 주기 수 대 상대 형광 (RFU)를 그려서 생성 되었습니다. B. TiLV 긍정적인 샘플의 증폭 곡선 그림 3B 에서 이집트와는 NTC에서 파생 된. 증폭 곡선 기자 신호 주기 수 대 (Rn)의 분석 결과에 포함 된 수동를 이용한 염료의 형광으로 정규화의 형광 이다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
각 실험실에 다른 기계에 정량 thermocycling의 끝에, 데이터 취득 되었고 분석. 그림 5A 와 5B 각 연구실에서 수행 하는 분석 실험에서 대표적인 용융 곡선을 표시 합니다. 정량 Pcr 기계 각 끝에 녹는 곡선 분석을 수행 하도록 프로그램 되었습니다. 이것은 점차적으로 증가 하는 온도 및 온도의 기능으로 형광 모니터링에 의해 달성 되었다. 온도가 충분히 dsDNA 변성 높은 때 fluorophore 분자 출시 때문에 형광의 큰 드롭 기록 됩니다. 각 정량 악기의 소프트웨어 네거티브 첫번째 파생 vs 온도 (그림 5)를 그려서 녹는 곡선 데이터에서 어 닐 링 온도 (Tm)을 계산 합니다. 그것은 그림 5A 와 5B 는 제품 다른 샘플 세트에 형성에 균일 한 용융 전환 분석 결과 약 80 ° C의 예상된 온도에서 볼 수 있습니다. 낮은 온도에서 다른 봉우리 관찰 되었다. 그들의 작은 크기로 인해 뇌관 이합체의 Tm 은 대상 DNA 시퀀스의 보다 일반적으로 낮은. 따라서, Tm의이 차이 쉽게 잠재적인 뇌관 이합체 또는 다른 일반적인 증폭 제품 식별할 수 있습니다. 컨트롤은 TiLV 긍정적인 샘플 및 표준 처럼 용융 곡선을 생성 하지 않았다 고 그림 5A 와 5B차트의 하단에 거의 수평 라인으로 볼 수 있습니다.
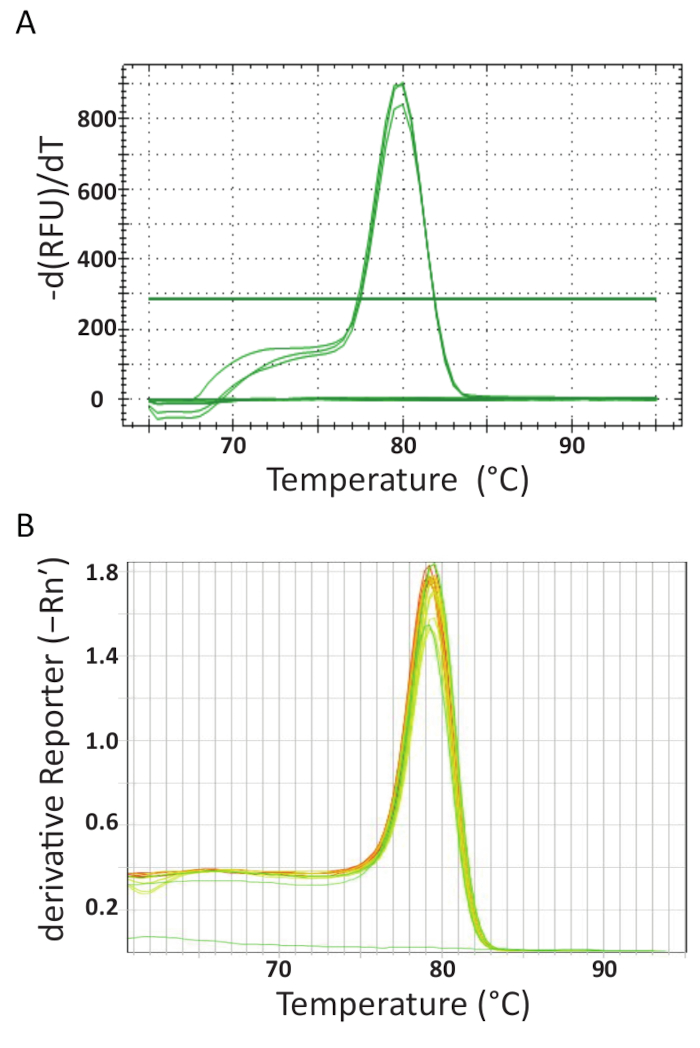
그림 5 . 곡선 분석 분석 결과 특이성 및 다른 PCR 제품의 용 해 기능에 의해 분화 될 수 있다 되도록 녹으 십시오. A. 태국, 부정적인 컨트롤 및 긍정적인 플라스 미드 컨트롤에서 발생 하는 TiLV 양성 샘플의 곡선 분석을 녹여. B. TiLV 이집트, pTiLV 표준과 NTC에서 파생 된 긍정적인 샘플의 곡선 분석을 녹여. A와 B 둘 다 형광 변화 온도에 의해 분할에 변화를 표시 하는 차트는 녹는 역학의 분명 한 그림을 생성 하는 온도 대 한 플롯. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
대부분의 정량 기계와 함께 추가 평가 정량 실행 하 고 자동으로 주기 임계값 (Ct)를 그려서 pTiLV 표준 서식 파일의 로그에 대 한 표준 곡선을 생성 하 여 샘플을 계량 소프트웨어 촉진 그림 6A 6B 두 개의 독립적인 실험실에 표시 된 번호를 복사 합니다. 짧게, Ct 는 정량 결과 평가 하기 위해 사용 하는 단위입니다. Ct 값 설정된 임계값 형광 신호 레벨에 도달 하는 데 필요한 사이클의 수를 나타냅니다. 큰 시작 하는 서식 파일에 적은 양의 주기 그것 감지 형광 레벨을 달성 하는 데 걸리는입니다. 실제로, TiLV의 높은 부하와 낮은 Ct 값 하위 임상 감염을 가진 물고기에와 같은 TiLV의 낮은 부하와 있을 것 이다. Ct 값을 확인 하려면 배경 형광 수준은 먼저 원시 데이터에서 공제 된다. 다음, 정량 장치과 관련 된 소프트웨어 자동으로 검색 하는 각 샘플에 대 한 데이터 곡선 및 샘플 임계값을 넘어 대표 Ct 통합 하 여 형광 임계값을 선택 합니다. 각 분석 결과 경우에 별도로 이렇게 하 고 각 임계값 신중 하 게 평가 해야, 임계값 로그 부분 증폭 곡선의와 모든 커브 병렬 한 장소에 설정 되어 있는지 확인 합니다. 따라서, 특정 Ct 인수는 상대 가치는 시작 템플릿을 복사 번호45, 상대적입니다 하지만 그것도 정량 Pcr 기계 및 시 약 사용, PCR 확대의 효율성과 검색의 감도 대 한 특정. 이러한 매개 변수는 그림 6에 동일한 분석 결과 사용 하 여 관찰 하는 차이에 기여 한다.
그림 6, 회귀 분석, 표준 곡선에서 표준 곡선 슬로프 (m)와 교차, 증폭 효율 (101/m -1) x (100)46 및 반응의 선형의 계산을 포함 하 여 수행 했다. 표준 곡선 분석 감도 (탐지 한계), 반복성 및 재현성 분석 결과의 확인에 사용 되었다. 모든 PCR와 DNA의 양을 두 배로 이론적으로, 사이클, 효율 (E)은 100%를 의미. 연습에서의 이러한 이상적인 효율 드물게 DNA 중 합 효소 저해, 오염 물질, 너무 많은 cDNA 및 pipetting 오류47등 최적의 PCR 사정 도달 된다. 일반적으로, 증폭 전자에 이르기까지 좋은 분석 실험, 그림 6A 94.5%의 효율성 동안, 7 직렬 희석된 pTiLV를 사용 하 여 그림 6B 에 나와 분석 결과에서 분석 결과 효율성 8 직렬 희석된 pTiLV 샘플을 사용 하 여 계산에 대 한 90-110% 샘플은 101.2% 이었다. 100%의 효율이 대부분 분석 결과에서 PCR 억제제의 존재 때문입니다. 선형 회귀 분석의 표준 줄거리 또한 수 있습니다 각 샘플41,,4245, TiLV 사본의 수의 계산에 대 한 빨간색 그림 6B에 나와 3 TiLV 샘플에 대 한 관찰 될 수 있다 는 S1, S3, S5 그림 3B에 표시 된 샘플에 대 한 결과 맞춰.
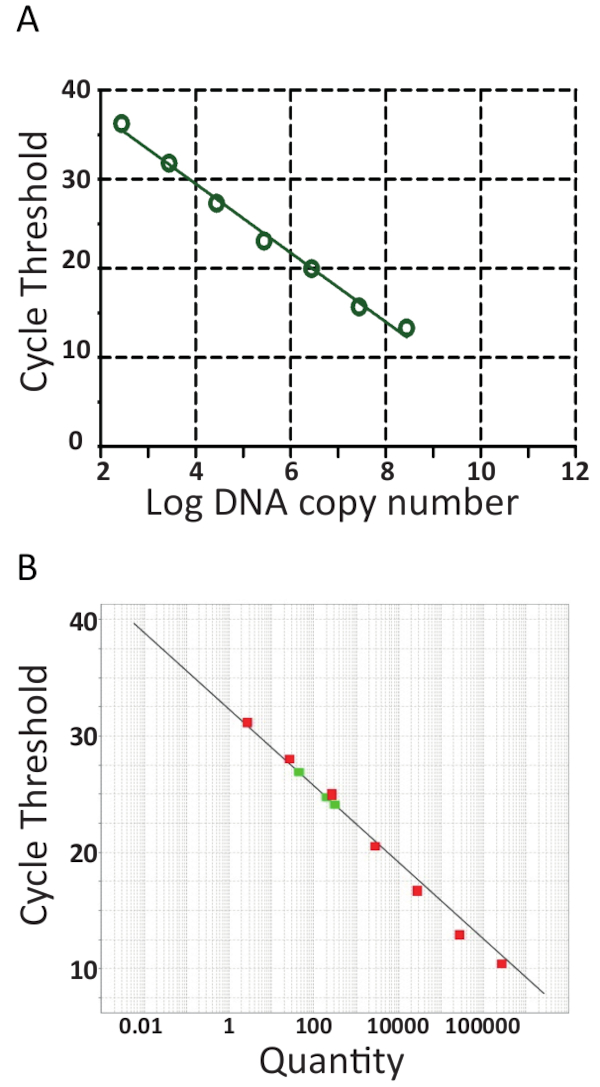
그림 6 . 실시간 정량 Pcr 표준 곡선입니다. PTiLV, 두 실험실에서 사용 되는 표준의 10 연속 희석의 실시간 PCR A. 8 직렬 희석된 pTiLV 샘플, 모든 알려진된 농도의 테스트 되었고 TiLV 복사본의 수에 상관 / 반응. 표준 곡선 로그 복사 번호 대 주기 임계값 (Ct)를 그려서 생성 되었습니다. 기울기 =-3.462, R2 = 0.9992 효율은 94.47%. B. A에서 7 직렬 희석된 pTiLV 제외 하 고 샘플 (그린) 했다 테스트 하 고 차트에 y 축과 x 축에 TiLV (양)의 복사본 수에 임계값 주기 표시 됩니다. Y-절편 = 32.327, 슬로프-3.292, R2 = 0.98 = 효율성은 101.2%. 두 표준 곡선에서 A와 B, 기울기, y 절편와 상관 계수 값 (R2) 분석 결과의 성능을 이해 하 활용 됩니다. 중요 한 것은, R2 값은 표준 곡선의 선형성 측정 이므로 1 가까이 있어야 한다. 기울기 측정 PCR 효율 100%-3.32의 경사에 해당 하는 점에서, 방정식 및 자세한 내용은 본문을 참조 하는 데 사용 됩니다. 좋은 정량 반응에는 일반적으로-3.58와-3.10 사이 경사를 상호 연결 하는 90-110% 사이 효율성이 있다. 표준 곡선의 알 수 없는 TiLV 긍정적인 샘플 절대 정량화에 사용 되 고 TiLV 사본의 정확한 수를 결정 합니다 / 반응은 3 TiLV 긍정적인 샘플 케이스 b에서 빨간색
토론
TiLV 이스라엘14 2014에 처음 알려졌다 고 그 이후로, 그것은 이집트, 콜롬비아, 인도, 말레이시아, 우간다, 탄자니아, 태국15,,1618, 를 포함 한 여러 나라에서 발견 되었습니다. 35 , 48. 세계 인식, 특히, tilapia 생산 국가에 더 많은 관심에 배치 바이러스 및 다양 한 제한 및 정부 기관에서 대책 시도 TiLV의 확산을 방지 하기 위해 구현 되었습니다. 여기, tilapia 조직, 샘플 수집, RNA 격리, cDNA 합성, PCR과 정량 분석 실험에서에서 TiLV 검색에 대 한 상세한 프로토콜 설명 되었습니다. 특정 토론을 보증 하는 경우 이러한 방법에 다양 한 측면을 확인 하 고 있습니다. TiLV는 물고기는 다양 한 크기9,12,,1415,49 와 지금까지 농장된 하이브리드 tilapia (o.를 포함 한 tilapia의 종에서 발견 되었습니다. 오 균 x niloticus)11,14, 나 일 틸 라 피아 (O. niloticus)9,10,,1415,16, 33 , 35 , 36 , 49 , 50 와 붉은 틸 라 피아 (Oreochromis sp.)16,33,,4851, 뿐만 아니라 야생 나 일 틸 라 피아9,12, 블랙으로 tilapia51, T. zilli14,15, S. galilaeus, 오 균 과 T. simonis intermedia14 그리고 아주 최근에 TiLV 야생 잉어 (Barbonymus에서에서 확인 된 schwanenfeldii)52. 내부 장기 (길, 비장, 간, 심장, 머리 신장) 또는 점액37 에서 조직 샘플 나이, 크기 또는 종에 건강으로 죽어가는 tilapia에서 수집 하 고 RNA 격리에 대 한 처리 수 있습니다. 설명 된 총 RNA 추출 프로토콜 여기 chaotropic 변성 에이전트를은 페 놀 및 guanidinium 안산의 monophasic 솔루션을 사용 합니다. 조직에는 클로 프롬 및 위 수성 단계는 interphase, 낮은 유기 단계를 포함 하는 분명 RNA 생성 되는 어떤 점에서 단계 분리를 달성 하기 위해 원심 분리의 추가 의해 다음이 솔루션에 직접 무 균. RNA는 소 프로 파 놀 강 수, 오염 물질의 제거에 복구 된 RNA의 세척에 의해 다음에 의해 수성 단계에서 격리 됩니다. 이 방법론에 의해 RNA의 격리 Piotr Chomczynski와 니콜 Sacchi에 의해 개척 되었다 고 guanidinium 안산 클로 페 놀 프롬-추출53,54로 불렸다. 사용할 RNA 추출 시 약의이 종류를 상업적으로 구입한 하 또는 실험실에서 수 있습니다 (자세한 내용은 재료의 표 참조). 이 프로토콜은 실리 카 기반 정화 등 열 기반 메서드는 보다 약간 이상 하지만 일반적으로, 더 비용 효과적 이며 더 많은 RNA를 생성.
이 프로토콜에서 RNA A260 값을 사용 하 여 측정 설명 했다 분 광 광도 학 값 RNA 품질을 나타낼 수 있다 그것에 의하여 (A260/A280 = 1.9-2.1). 이 메서드는 샘플 순도의 좋은 표시를 줄 것 이다, 하는 동안 그것은 절대적으로 추출 된 RNA의 품질에 대 한 알릴 수 없습니다. 제대로 되는지 확인 하려면 RNA 그대로 또는 부분적으로 저하, 샘플 수 점에서 18S 스테인드는 EtBr의 번짐 agarose 젤 전기 이동 법으로 분리 되며 28S rRNA 밴드 RNA 저하를 나타냅니다. RNA 품질의 더 검증 랩 온 칩 악기를 사용 하 여 포함할 수 있습니다. 또한, 그것은 또한 DNase와 순화 된 RNA를 소화 하는 것이 중요 오염 제거 하려면 호스트 게놈 DNA를 다운스트림 응용 프로그램에 따라 잘못 된 결과가 발생할 수 있습니다. RNA 추출 절차의 끝에 추가 DNaseI 처리를 수행할 수도 있습니다 경우 호스트 gDNA 여전히 대부분 RNA 샘플을 오염 이다, ( 재료의 표참조).
보완 DNA 합성 전체 정량 결과 크게 영향을 미칠 수 있습니다 하 고 변화를 발생 시킬 수 있는 방법의 한 측면 이다. 단일 구성 요소 설정의 구성 하 고 여기에 주 창 하는 cDNA 프로토콜 올리고 (dT)를 사용 하 고 따라서만 polyA 꼬리를 포함 하는 mRNAs를 transcribes. 그것은 정확 하 게 반전 녹음 방송 반응 및 cDNA의이 모드에서 사용 하는 구성 요소의 합성은 성공적으로 입증 TiLV 탐지32에 대 한 사용자 정의 컨트롤을 수 있습니다. 이 설정 대신 포함 하는 상업적으로 구입 마스터 믹스 반전 녹음 방송 반응에 필요한 모든 구성 요소 이며 매우 신속 하 고 일반적인 다단계, pipetting 및 멀티 온도 프로토콜 없이 간단. 그것은 처리를 최소화 하 고 모든 샘플에서 균일성을 촉진 하기 때문에 이것은 유리입니다. 같은 마스터-믹스 자주 등 모두 oligo(dT) 인구에 RNAs의 전체 길이에서 시퀀스의 대표적인 cDNA 복사본을 생성 하는 다른 적용 가능한 RNA 서식 파일을 만드는 무작위 뇌관 (바이러스 및 tilapia mRNA 호스트)와 이론, 모든 원하는 RNA 종 다음 기존의 PCR 또는 이러한 샘플에서 정량으로 측정할 수 있습니다. 이 다양성은 주요 활용 2 단계 RT-PCR 접근; 그것은 많은 다른 실험에 사용할 수 있는 장기 수영장을 제공 합니다. 결과에 원스텝 RT-PCR 접근 표현 하고있다 어떤 점에서 시퀀스 특정 뇌관 (표 1) 사용 되 고 고 RT PCR 한 튜브에서 수행 했다 (소재 목록 참조). 일반적으로, 순서 특정 한 뇌관 임의의 뇌관을 사용 하 여 보다 구체적인 대상 RNA의 높은 RT 효율성에 대 한 허용 하지만 특정 대상 RNA는 유일 하 게 같은 cDNA 샘플 ( 를 참조 하는 특정 실험실의 유일한 목적은 될 수 있는 양이 정해질 수 있는 자료의 테이블 cDNA 합성 제품 제안을 위해).
그러나 기존의 RT-PCR 나타납니다 일반적으로 사용 되는 TiLV 진단9,13,14,15,,1617,18, 에서 지금까지 33 , 35 , 48 , 55. 실시간 정량 검출 및 정량화 물고기 조직 또는 점액32,37TiLV의 작은 금액에 대 한 더 강력한 도구에 표시 되었습니다. 일반적으로, 정량은 높은 감도, 특이성, 좋은 재현성, 넓은 동적 범위 및 속도21바이러스학 임상 진단 실험실에서 널리 이용 된다. 정량은 처음 기존의 RT-PCR 보다 구현 비용이 있을 수 있습니다, 그것은 많은 중요 한 이점을 기존의 PCR; 제공지 않습니다. 그것은 보다 빠른 설정-주위 시간 샘플에서 결과를 하 고 어떤 게시물-PCR는 단계를 필요 하지 않습니다. 이 후자의 포인트 실험실 오염에 대 한 최소한의 위험이 발생 시와 같은 높은 처리량 상황에 더 쉽게 적용할 수 있습니다 의미 합니다. 또한, 그것은 본질적으로 기존의 RT-PCR의 중요 한 하위 임상 감염21낮은 바이러스 부하를 감지 하는 보다 더 민감한. 이것은 중첩 된 PCR 방법은 반전 녹음 방송 필요로 해야, 두 개의 추가 PCR 반응 그리고 분석 agarose 젤 전기 이동 법. 이러한 많은 단계 시간을 많이 하 고 오류 또는 오염의 기회를 증가. 그럼에도 불구 하 고, 그것의 높은 감도 때문에 실시간 정량 세심 한 실험 설계 및 정확한 결과56,57를 생성 하기 위한 정량화 기법에 대 한 철저 한 이해를 요구 한다.
DNA 바인딩 fluorophore, SYBR 녹색 내가이 프로토콜에서 입증 되었습니다. 난다 DNA 바인딩 dsDNA 염료 이며 따라서 분석 결과의 특이성 뇌관, 가양성58를 생성할 수 있습니다 집합에 완전히 거짓말. 따라서, 녹는 곡선 분석 끝에 각 PCR 수행 dsDNA PCR 반응의 특히 중요 한 부분 이므로 그 하나만 PCR amplicon 올바른 T의 확인 하는 동안m 생산 (이 또한 의해 달성 되어야 젤 전기 이동 법 새로운 분석 구현 되는 경우). DNA 파편의 Tm 다양 한 길이, GC 구성, 시퀀스, 스트랜드 complementarity 기능에 따라 달라 집니다, 그리고에 뿐만 아니라 집중 버퍼 구성 요소 및 PCR 강화. 실험실 뇌관 이합체 또는 다른 원치 않는 PCR 제품의 존재를 공개 하지 않았다 그러나 경우이 다른 샘플 및 실험적인 체제과 관찰, 분석 결과 이어야 한다 2에서 표시 된 대표 결과에 녹는 곡선 분석 다시 최적화. 고급 정량 기술 같은 녹는 곡선 단계를 필요로 하지 않는 하 고 실제로, 종이 작성 되었습니다이 방법을 기반으로 TaqMan부터 TiLV RT-정량 개발 되었습니다 2 개의 뇌관 및 높은 TiLV 특정34만들기 검색을 활용 하 여.
의심의 여지가, 실시간 정량 Pcr 분석 실험을 위한 뇌관 분석 결과의 성공에 필수적 이며 여기에 뇌관32시간 공개적으로 사용 가능한 TiLV 게놈 데이터에 따라 설계 되었습니다. 그러나, RNA 바이러스는 잘 알려진 높은 돌연변이 비율을 전시 하 고 가능한 긴장 ISAV59에 대 한 관찰로 현재 진단 테스트를 탈출 한다. 그것은 어려울 것 이다 항상 보편적인 팬-TiLV RT 생성 하와 같은 바이러스 종류-정량 분석 결과 같은 분석 실험만 지속적으로 향상 됩니다 광범위 한 위치 및 시간 기간에서 더 많은 TiLV 게놈 데이터를 사용할 수 있게 하는 경우.
마지막으로, 중복 실행 또는 가능 하다 면, 둘 다 내부에서 반응 triplicate 인터 정량 분석 실험을 필수적 이다. Ct 값이 매우 높은 경우, 복제의 사용은 특히 PCR 반응 안정적이 고 재현 가능한가 확인 하는 것이 중요. 일반적으로 데이터를 복제 하는 경우 반응 다릅니다 이상 0.5 사이클, 반응과 반복 해야 경우 Ct 값 > 0.5 사이클에서 일관 되 게 복제, 분석 결과 다시 최적화 해야 합니다. 통합된 정량 pipetting 로봇의 사용은 대단히이 문제와 도움이 됩니다 하지만 그것은 고급 도구입니다. 어떤 실험을 포함 한 적절 하 고 적절 한 컨트롤으로 이러한 분석 실험 인가 해야 진단 실험실에서 특히 강력한 분자 분석 실험의 발달에 매우 중요입니다. 컨트롤 내 생 tilapia 하우스키핑 유전자의 검출 뿐만 아니라 포지티브 (긍정적인 TiLV 샘플, 표준 TiLV 플라스 미드)와 부정적인 컨트롤 (NTC와-RT) 샘플을 포함 해야 합니다. 이러한 통제 과소평가 될 수 없다 고 제대로 각 단계 분석 결과의 품질을 이해 하 고 제대로 결과 해석 하는 각 분석 결과에 포함 되어야 합니다.
공개
저자는 공개 없다.
감사의 말
우리는 그들의 지원에 대 한 수의 세균 학 연구소, Vetsuisse 교수, 베른 대학에 감사입니다. 이 작품은 pn 수 여 Vetsuisse 교수, 120% 모델 자금에 의해 베른 대학에서 초기 경력 연구원의 학술 진흥 및 성 평등을 위한 위원회에 의해 투자 되었다. WS 및 홍보는에서 지원 센터 고급 연구를 위한 농업 및 식품, 고급 연구, Kasetsart 대학, 방콕, 태국 등 교육 연구 진흥 및 국가 연구 대학 프로젝트의 태국, 오피스 연구소 고 등 교육 위원회, 교육 과학기술부, 태국. 우리는 그녀의 내레이션에 대 한 박사 Kwanrawee Sirikanchana Piyawatchara Sikarin 비디오 편집을 위한 감사 하 고 싶습니다.
자료
| Name | Company | Catalog Number | Comments |
| Tissue collection | Step 1 | ||
| Tricaine methanesulfonate | Sigma-Aldrich | E10521 | An alternative to clove oil. Step 1.1 |
| RNAlater stabilization solution | Thermo Fisher Scientific | AM7020 | For storing tissues if they cannot be processed immediately Step 1.3 |
| RNA extraction | Step 2 | ||
| TRIreagent | Sigma-Aldrich | Step 2.1 | |
| TRIzol | Thermo Fisher Scientific (Invitrogen) | 15596026 | Step 2.1 |
| GENEzol | Geneaid | GZR100 | Step 2.1 |
| Trisure | Bioline | BIO-38032 | Step 2.1 |
| Homemade solution | - | - | 94.53 g/L (800 mM) guanidine thiocyanate 30.45 g/L (400 mM) ammonium thiocyanate 8.20 g/L (100 mM) sodium acetate 380 mL/L (38 % v/v) phenol 50 mL/L (5 % v/v) glycerol 1.0 g/L (0.1 % w/v) 8-quinolinol, pH 5.0 Store up to 2 years at 4oC Step 2.1 |
| MagNA Lyser Green Beads | Roche | 3358941001 | An alternative tissue homogenization method used in conjunction with tissue lysing machines detailed below Step 2.2 |
| Lysing Matrix D, 2 mL Tube | MP BIOMEDICALS | 116913050 | |
| Chloroform | Sigma-Aldrich | C2432 | Step 2.3 |
| Chloroform | RCI Labscan | AR1027E-G2.5L | Step 2.3 |
| 1-Bromo-3-chloropropane | Sigma-Aldrich | B9673 | A less toxic alternative to chloroform Step 2.3 |
| Isopropanol (GC) ≥ 99.8 % | Sigma-Aldrich | 59300 | Step 2.6 |
| Isopropanol (ACS, ISO Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.09634.2500 | Step 2.6 |
| Glycogen, molecular biology grade (e.g., Sigma, cat. no. G1767) | Thermo Fisher Scientific (Thermo Scientific) | R0551 | Useful step if tissue starting material is small to maximise RNA precipitation optional |
| Ethanol (purity (GC) ≥ 99.9 % | Sigma-Aldrich (EMD Millipore) | 1.00983 | Step 2.9 |
| Ethanol (ACS, ISO Reag. Ph Eur) | Merck (EMSURE) | 1.00983.2500 | Step 2.9 |
| Nuclease-free water | Promega | P1193 | Step 2.13 |
| Nuclease-free water | Multicell | 809-115-CL | Step 2.13 |
| Ambion TURBO DNA-free kit | Thermo Fisher Scientific (Invitrogen) | AM1907 | Can be performed at the end of the RNA extraction protocol optional |
| cDNA synthesis | Step 4 | ||
| Viva cDNA Synthesis Kit | Vivantis | cDSK01 | Step 4.1 & 4.3 |
| ReverTra Ace qPCR RT MasterMix with gDNA remover | Toyobo | A1172K | An alternative option see discussion |
| ReverTra Ace qPCR RT Kit | Toyobo | FSQ-101 | An alternative option see discussion |
| AffinityScript Multiple Temperature Reverse Transcriptase | Agilent Technologies | 600107 | An alternative option |
| PCR | Step 5 | ||
| DNA polymerase systems: | Step 5.2 | ||
| - Platinum II Hot-Start Green PCR Master Mix (2X) | Thermo Fisher Scientific (Invitrogen) | 14001012a | Step 5.2 |
| - GoTaq Mastermix | Promega | M7122 | Step 5.2 |
| Separate PCR mixture components: | Step 5.2 | ||
| 10mM dNTP Mix | Vivantis | NP2409 | Step 5.2 |
| 25mM MgCl2 | Thermo Fisher Scientific | R0971 | Step 5.2 |
| 10X Taq Buffer with KCl | Thermo Fisher Scientific | 00348114 | Step 5.2 |
| Taq DNA polymerase | Vivantis | PL1202 | Step 5.2 |
| - Verso 1-step RT-PCR ReddyMix with ThermoPrime Taq | Thermo Fisher Scientific | AB1454 | One step RT-PCR exemplified in Figure 3B |
| Gel electrophoresis: | For visulation of PCR products from steps 5.1-5.4 | ||
| Ethidium Bromide solution (10 mg/mL) | Thermo Fisher Scientific | 17898 | Step 5.5 |
| Tris/Acetic/EDTA (TAE) buffer: | Step 5.5 | ||
| - Tris | Vivantis | PR0612-1KG | Step 5.5 |
| - Acetic acid (glacial) (ACS, ISO, Reag. Ph Eur) | Merck KGaA (EMSURE) | 1.00063.2500 | Step 5.5 |
| - Ethylenediaminetetraacetic acid (EDTA) | BIO-RAD | 161-0729 | Step 5.5 |
| Agarose | Vivantis | PC0701-100G | Step 5.5 |
| DNA ladders and markers | Vivantis | NL1405 | Step 5.5 |
| DNA gel loading dye (6X) | Thermo Fisher Scientific | R0611 | Step 5.5 |
| qPCR | Step 6 | ||
| PowerUP SYBR Green Master Mix | Thermo Fisher Scientific (Applied Biosystems) | A25779 | Exemplified in Figures 4-6B Step 6.2 |
| iTaq Universal SYBR Green Supermix | BIO-RAD | 1725120 | Exemplified in the video and in Figures 4-6A Step 6.2 |
| Equipment | |||
| Dounce tissue grinder pestle | Sigma-Aldrich | P1110 | Protocol 2 |
| MagNA Lyser Instrument | Roche | 3358976001 | An alternative tissue homogenizing option for protocol 2 which are used in conjunction with the lysing beads detailed above Step 2.2 |
| FastPrep-24 5G Homogenizer | MP BIOMEDICALS | 116005500 | |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5427R | Protocol 2 Step 2.4, 2.7 & 2.10 |
| Refrigerated microcentrifuge | Eppendorf | Eppendorf 5418R | |
| Heat box | Labnet | AccuBlock Digital Dry Bath | Protocol 2 Step 2.13 |
| Microvolume spectrophotometer | Thermo Fisher Scientific (Applied Biosystems) | Nanodrop 2000 | Protocol 3 Step 3.1 - 3.4 |
| PCR machine | BIO-RAD | T100 Thermal Cycler | Protocol 5 Step 5.4 |
| Power supply | BIO-RAD | PowerPac HC | Protocol 5 Step 5.5 |
| Horizontal gel electrophoresis | BIO-RAD | Mini ReadySub-Cell GT Cell #1704487edu | Protocol 5 Step 5.5 |
| Mini microcentrifuge | Corning | LSE 6766 | Useful to quickly spin down PCR reaction tubes in protocols 4, 5 & 6 Step 6.5.1 |
| Microcentrifuge | LioFuge | LM-60 | Step 6.5.1 |
| qPCR machine and software | Thermo Fisher Scientific | 7500 Fast Real-Time PCR System with 7500 Software v2.0 | Protocol 6 Step 6.6-6.8 |
| qPCR machine and software | BIO-RAD | CFX96 Touch Real-Time PCR Detection System with CFX Manager software | |
| General Materials | |||
| Mayo scissors | Step 1.1-1.2 | ||
| Forceps | Step 1.1-1.2 | ||
| Pipette | Rainin | Pipette-Lite XLS | |
| Aerosol-barrier pipette tips | Sigma-Aldrich | Z333328, Z333336, Z333344 | |
| Nuclease-free 1.5-ml microcentrifuge tubes | Eppendorf |
참고문헌
- FAO. . The State of World Fisheries and Aquaculture, 2014. Opportunities and Challenges. , (2014).
- FAO. . The State of World Fisheries and Aquaculture, 2016. Contributing to Food Security and Nutrition for all. , (2016).
- WorldBank. . FISH TO 2030: Prospects for Fisheries and Aquaculture. Agriculture and Environmental Services Discussion Paper 03. , (2013).
- Wing-Keong, N., Nicholas, R. A review of the nutrition and feeding management of farmed tilapia throughout the culture cycle. Reviews in Aquaculture. 5 (4), 220-254 (2013).
- Cleasby, N., et al. The socio-economic context for improving food security through land based aquaculture in Solomon Islands: A peri-urban case study. Marine Policy. 45, 89-97 (2014).
- Ponzoni Raul, W., et al. Genetic improvement of Nile tilapia (Oreochromis niloticus) with special reference to the work conducted by the WorldFish Center with the GIFT strain. Reviews in Aquaculture. 3 (1), 27-41 (2011).
- Hounmanou, Y. M. G., et al. Tilapia lake virus threatens tilapiines farming and food security: Socio-economic challenges and preventive measures in Sub-Saharan Africa. Aquaculture (Amsterdam, Netherlands). 493, 123-129 (2018).
- OIE. . Tilapia Lake Virus (TiLV) - a novel orthomyxo-like virus. OIE technical disease cards. , (2018).
- Mugimba, K. K., et al. Detection of tilapia lake virus (TiLV) infection by PCR in farmed and wild Nile tilapia (Oreochromis niloticus) from Lake Victoria. Journal of Fish Diseases. , (2018).
- Koesharyani, I., Gardenia, L., Widowati, Z., Khumaira, D. D., Rustianti, Studi kasus infeksi tilapia lake virus (tilv) pada ikan nila (Oreochromis niloticus). Jurnal Riset Akuakultur. 13 (1), 85-92 (2018).
- OIE. . Tilapia lake virus disease (TiLV), Chinese Taipei. Immediate Notification. , (2017).
- OIE. . Tilapia Lake Virus Disease (TiLV), Peru. Immediate Notification. , (2018).
- Bacharach, E., et al. Characterization of a Novel Orthomyxo-like Virus Causing Mass Die-Offs of Tilapia. MBio. 7 (2), e00431-e00416 (2016).
- Eyngor, M., et al. Identification of a novel RNA virus lethal to tilapia. Journal of Clinical Microbiology. 52 (12), 4137-4146 (2014).
- Nicholson, P., et al. Detection of Tilapia Lake Virus in Egyptian fish farms experiencing high mortalities in 2015. Journal of Fish Diseases. 40 (12), 1925-1928 (2017).
- Surachetpong, W., et al. Outbreaks of Tilapia Lake Virus Infection, Thailand, 2015-2016. Emerging Infectious Diseases. 23 (6), 1031-1033 (2017).
- Tattiyapong, P., Dachavichitlead, W., Surachetpong, W. Experimental infection of Tilapia Lake Virus (TiLV) in Nile tilapia (Oreochromis niloticus) and red tilapia (Oreochromis spp.). Veterinary Microbiology. 207, 170-177 (2017).
- Kembou Tsofack, J. E., et al. Detection of Tilapia Lake Virus in Clinical Samples by Culturing and Nested Reverse Transcription-PCR. Journal of Clinical Microbiology. 55 (3), 759-767 (2017).
- Thangaraj, R. S., et al. Derivation of two tilapia (Oreochromis niloticus) cell lines for efficient propagation of Tilapia Lake Virus (TiLV). Aquaculture (Amsterdam, Netherlands). 492, 206-214 (2018).
- Hanson, L. A., Rudis, M. R., Vasquez-Lee, M., Montgomery, R. D. A broadly applicable method to characterize large DNA viruses and adenoviruses based on the DNA polymerase gene. Virology Journal. 3, 28-28 (2006).
- Josko, D. Molecular virology in the clinical laboratory. Clinical Laboratory Science. 23 (4), 231-236 (2010).
- Munir, K., Kibenge, F. S. Detection of infectious salmon anaemia virus by real-time RT-PCR. Journal of Virological Methods. 117 (1), 37-47 (2004).
- Snow, M., et al. Developement, application and validation of a Taqman real-time RT-PCR assay for the detection of infectious salmon anaemia virus (ISAV) in Atlantic salmon (Salmo salar). Developments in Biologicals. 126, 133-145 (2006).
- Matejusova, I., McKay, P., McBeath, A. J., Collet, B., Snow, M. Development of a sensitive and controlled real-time RT-PCR assay for viral haemorrhagic septicaemia virus (VHSV) in marine salmonid aquaculture. Diseases of Aquatic Organisms. 80 (2), 137-144 (2008).
- Garver, K. A., et al. Development and validation of a reverse transcription quantitative PCR for universal detection of viral hemorrhagic septicemia virus. Diseases of Aquatic Organisms. 95 (2), 97-112 (2011).
- Dalla Valle, L., et al. Development of a sensitive and quantitative diagnostic assay for fish nervous necrosis virus based on two-target real-time PCR. Veterinary Microbiology. 110 (3-4), 167-179 (2005).
- Hodneland, K., Garcia, R., Balbuena, J. A., Zarza, C., Fouz, B. Real-time RT-PCR detection of betanodavirus in naturally and experimentally infected fish from Spain. Journal of Fish Diseases. 34 (3), 189-202 (2011).
- Hodneland, K., Endresen, C. Sensitive and specific detection of Salmonid alphavirus using real-time PCR (TaqMan). Journal of Virological Methods. 131 (2), 184-192 (2006).
- Wang, X. W., Ao, J. Q., Li, Q. G., Chen, X. H. Quantitative detection of a marine fish iridovirus isolated from large yellow croaker, Pseudosciaena crocea, using a molecular beacon. Journal of Virological Methods. 133 (1), 76-81 (2006).
- van Beurden, S. J., et al. Development and validation of a real-time PCR assay for the detection of anguillid herpesvirus 1. Journal of Fish Diseases. 39 (1), 95-104 (2016).
- Ciulli, S., et al. Development and application of a real-time PCR assay for the detection and quantitation of lymphocystis disease virus. Journal of Virological Methods. 213, 164-173 (2015).
- Tattiyapong, P., Sirikanchana, K., Surachetpong, W. Development and validation of a reverse transcription quantitative polymerase chain reaction for tilapia lake virus detection in clinical samples and experimentally challenged fish. Journal of Fish Diseases. 41 (2), 255-261 (2018).
- Dong, H. T., et al. Emergence of tilapia lake virus in Thailand and an alternative semi-nested RT-PCR for detection. Aquaculture (Amsterdam, Netherlands). 476, 111-118 (2017).
- Waiyamitra, P., et al. A TaqMan RT-qPCR assay for tilapia lake virus (TiLV) detection in tilapia. Aquaculture (Amsterdam, Netherlands). 497, 184-188 (2018).
- Behera, B. K., et al. Emergence of Tilapia Lake Virus associated with mortalities of farmed Nile Tilapia Oreochromis niloticus (Linnaeus 1758) in India. Aquaculture (Amsterdam, Netherlands). 484, 168-174 (2018).
- Ferguson, H. W., et al. Syncytial hepatitis of farmed tilapia, Oreochromis niloticus (L.): a case report. Journal of Fish Diseases. 37 (6), 583-589 (2014).
- Liamnimitr, P., Thammatorn, W., U-thoomporn, S., Tattiyapong, P., Surachetpong, W. Non-lethal sampling for Tilapia Lake Virus detection by RT-qPCR and cell culture. Aquaculture (Amsterdam, Netherlands). 486, 75-80 (2018).
- Yang, C. G., et al. Evaluation of reference genes for quantitative real-time RT-PCR analysis of gene expression in Nile tilapia (Oreochromis niloticus). Gene. 527 (1), 183-192 (2013).
- Bustin, S. A. Real-time, fluorescence-based quantitative PCR: a snapshot of current procedures and preferences. Expert Review of Molecular Diagnostics. 5 (4), 493-498 (2005).
- Fleige, S., Pfaffl, M. W. RNA integrity and the effect on the real-time qRT-PCR performance. Molecular Aspects of Medicine. 27 (2-3), 126-139 (2006).
- Kubista, M., et al. The real-time polymerase chain reaction. Molecular Aspects of Medicine. 27 (2-3), 95-125 (2006).
- Mackay, I. M., Arden, K. E., Nitsche, A. Real-time PCR in virology. Nucleic Acids Research. 30 (6), 1292-1305 (2002).
- Wong, M. L., Medrano, J. F. Real-time PCR for mRNA quantitation. Biotechniques. 39 (1), 75-85 (2005).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. Journal of Molecular Endocrinology. 25 (2), 169-193 (2000).
- Heid, C. A., Stevens, J., Livak, K. J., Williams, P. M. Real time quantitative PCR. Genome Research. 6 (10), 986-994 (1996).
- Rutledge, R. G., Côté, C. Mathematics of quantitative kinetic PCR and the application of standard curves. Nucleic Acids Research. 31 (16), e93-e93 (2003).
- Svec, D., Tichopad, A., Novosadova, V., Pfaffl, M. W., Kubista, M. How good is a PCR efficiency estimate: Recommendations for precise and robust qPCR efficiency assessments. Biomolecular Detection and Quantification. 3, 9-16 (2015).
- Amal, M. N. A., et al. A case of natural co-infection of Tilapia Lake Virus and Aeromonas veronii in a Malaysian red hybrid tilapia (Oreochromis niloticus × O. mossambicus) farm experiencing high mortality. Aquaculture (Amsterdam, Netherlands). 485, 12-16 (2018).
- Fathi, M., et al. Identification of Tilapia Lake Virus in Egypt in Nile tilapia affected by ‘summer mortality’ syndrome. Aquaculture (Amsterdam, Netherlands). 473, 430-432 (2017).
- OIE. . Tilapia Lake Virus disease (TiLV), Philippines. Immediate Notification. , (2017).
- OIE. . Tilapia lake virus disease (TiLV), Malaysia. Immediate Notification. , (2017).
- Abdullah, A., et al. First detection of tilapia lake virus (TiLV) in wild river carp (Barbonymus schwanenfeldii) at Timah Tasoh Lake, Malaysia. Journal of Fish Diseases. 41 (9), 1459-1462 (2018).
- Chomczynski, P., Sacchi, N. Single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction. Analytical Biochemistry. 162 (1), 156-159 (1987).
- Chomczynski, P., Sacchi, N. The single-step method of RNA isolation by acid guanidinium thiocyanate-phenol-chloroform extraction: twenty-something years on. Nature Protocols. 1 (2), 581-585 (2006).
- Del-Pozo, J., et al. Syncytial Hepatitis of Tilapia ( Oreochromis niloticus L.) is Associated With Orthomyxovirus-Like Virions in Hepatocytes. Veterinary Pathology. 54 (1), 164-170 (2017).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry. 55 (4), 611-622 (2009).
- Purcell, M. K., Getchell, R. G., McClure, C. A., Garver, K. A. Quantitative polymerase chain reaction (PCR) for detection of aquatic animal pathogens in a diagnostic laboratory setting. Journal of Aquatic Animal Health. 23 (3), 148-161 (2011).
- Simpson, D. A., Feeney, S., Boyle, C., Stitt, A. W. Retinal VEGF mRNA measured by SYBR green I fluorescence: A versatile approach to quantitative PCR. Molecular Vision. 6, 178-183 (2000).
- Kibenge, M. J., et al. Discovery of variant infectious salmon anaemia virus (ISAV) of European genotype in British Columbia, Canada. Virology Journal. 13, 3 (2016).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유