Method Article
פילוח RNA בכל מקום וריאקמה בדרוסופילים מלנוגאסטר באמצעות CRISPR/CasRx
In This Article
Summary
מאמר זה מתאר פרוטוקול מפורט לשימוש באנזים Cas13D הממוקד ברנ"א (RfxCas13D) בזבובים.
Abstract
CasRx, חברה במשפחת Cas13 הממוקדת ברנ"א, היא תוספת חדשה ומבטיחה של טכנולוגיות CRISPR/Cas בהפחתת תמליל גנים יעילה עם פרופיל אטרקטיבי מחוץ למטרה הן ברמה התאית והן ברמה האורגנית. לאחרונה דווח כי מערכת CRISPR/CasRx יכולה לשמש להשגת הפחתת תעתיק גנים בכל מקום ורקמות ספציפית לדרוסופיל מלנוגאסטר. מאמר זה מפרט את השיטות מהעבודה האחרונה, המורכבת משלושה חלקים: 1) בכל מקום ב- vivo אנדוגני RNA פילוח באמצעות מערכת CasRx שני רכיבים; 2) בכל מקום ב- vivo אקסוגני RNA מיקוד באמצעות מערכת CasRx שלושה רכיבים; ו-3) פילוח RNA ספציפי לרקמות באמצעות מערכת CasRx בעלת שלושה רכיבים. ההשפעות של פילוח RNA שנצפו כוללות שינויים פנוטיפיים ספציפיים לגנים ממוקדים, הפחתת תעתיק RNA ממוקדת, ופנוטיפים קטלניים מדי פעם הקשורים לביטוי גבוה של חלבון CasRx ופעילות נלווית. בסך הכל, תוצאות אלה הראו כי מערכת CasRx מסוגלת למקד הפחתת תעתיק RNA ברמה האורגנית באופן ניתן לתכנות ויעיל, המוכיח כי ב- vivo transcriptome פילוח, והנדסה היא אפשרית ומניחה את הבסיס לטכנולוגיות פילוח RNA מבוססות VIVO CRISPR.
Introduction
מאז הופעתן של טכנולוגיות פלינדרומיות קצרות (CRISPR), רוב המיקוד בתחום זה היה בעריכת דנ"א, המציע יישומים טרנספורמטיביים ברפואה וביוטכנולוגיה1. שינוי קבוע של רצפי DNA, עם זאת, לא תמיד רצוי משיקולים אתיים. לאור זאת, מחקרים שנערכו לאחרונה החלו לפתח כלים מבוססי CRISPR למיקוד RNA והוכיחו כי אכן ניתן להשתמש בטכנולוגיות CRISPR למיקוד RNA במגוון מערכות ביולוגיות2,3,4,5,6,7. ברבות מהמערכות הללו שנבדקו, הגישה הנפוצה כיום למיקוד RNA והפחתת תמלילים היא הפרעות RNA (RNAi), אשר רחוק מלהיות מושלם, לעתים קרובות מציג יעילות מגוונת ופעילות גבוהה מחוץ למטרה כאשר נעשה שימוש vivo8,9,10,11,12,13,14,15,16,17 . לכן, בהתחשב במצב של טכנולוגיות אלה, כדאי לבחון עוד יותר את הפוטנציאלים של כלים מבוססי CRISPR עבור פילוח RNA.
מחקר אחד בולט שנערך לאחרונה דיווח כי ריבונוקלאז CasRx, חבר בכיתת Cas13d, יכול להפחית ביעילות את רמות תעתיק הגנים בתרבות התאים האנושיים ובעל פרופיל אטרקטיבי מחוץ למטרה4. ממצא זה הוביל לשאלה אם ריבונוקלאז חדש זה יכול לשמור על יעילותו ושיעור נמוך מחוץ למטרה עבור RNA מיקוד ברמה האורגנית. מחקר שנערך לאחרונה התייחס לשאלה זו על ידי מראה כי מערכת CasRx ניתן להשתמש כדי להשיג בכל מקום ורקמות ספציפי תעתיק תעתיק Drosophila melanogaster5.
כדי לייעל את השימושיות של גישה זו שפורסמה לאחרונה, פרוטוקול זה מפרט את השיטות מעבודה זו לאחרונה, המורכבת משלושה חלקים עיקריים: 1) הנמצאים בכל מקום במיקוד RNA של vivo באמצעות מערכת CasRx בעלת שני רכיבים; 2) בכל מקום ב- vivo אקסוגני RNA מיקוד באמצעות מערכת CasRx שלושה רכיבים; ו-3) פילוח RNA ספציפי לרקמות באמצעות מערכת CasRx בעלת שלושה רכיבים.
מדריך RNAs (gRNA) מיקוד גנים יעד שונים תחת שליטתו של מקדם בכל מקום תוכננו וקווי זבוב המבטאים מבנים המכילים gRNA אלה נוצרו. מבנים CasRx תחת שליטתו של מקדם בכל מקום, או מקדם רצף הפעלה במעלה הזרם מותנה (UASt) הניתן להפעלה על ידי גורם התמלול GAL4, תוכננו גם הם וקווים מעופפים המטפחים מבנים המכילים CasRx אלה שנוצרו. מבנים CasRx לא פעילים קטליטית, dCasRx, תוכננו ושימשו כפקדים שליליים. RNA בכל מקום מיקוד זבובים מושגת על ידי חציית קווי זבובים ביטוי gRNA עם קווי זבובים בכל מקום CasRx ביטוי. האבן המבטאת הן את מבנה ה- gRNA המתמקד בתעתיק גנים מסוים והן בחלבון CasRx יש הפחתה בכל מקום של תמלילי גנים ממוקדים. RNA ספציפי לרקמות הממוקד בזבובים מושג על ידי חציית זבובים הראשונים המביעים GRNA עם UASt-CasRx המבטאים זבובים, המשיגים זבובים טרנסהטרוזיגוס הנושאים מבנים GRNA ו- UASt-CasRx. זבובים כאלה בתורם חוצים עם זבובים ספציפיים לרקמות GAL4 ביטוי, וכתוצאה מכך ייצור של ביטוי CasRx ספציפי לרקמות ומיקוד RNA בזבובים.
האופי הניתן לתכנות של מערכת CasRx מציע אפשרות להתאמה אישית ואופטימיזציה כדי לסייע בהשגת יעילות גבוהה ופעילות נמוכה מחוץ ליעד עבור פילוח RNA של vivo. יישומים פוטנציאליים של פילוח RNA מבוסס CRISPR הם רבים, כולל החלפת RNAi במעבדה ותרומת בקרת וקטור חרקים בטבע. של האחרון, אחד הצרכים העולמיים ללא מענה הוא פיתוח כלים יעילים כדי להילחם בזיהומים של וירוסי RNA המועברים באמצעות יתושים. נגיפי RNA רבים, כגון דנגי, זיקה ווירוס צ'יקונגוניה, מועברים באמצעות יתושים, המשפיעים על בריאות האדם ותורמת לתמותה. הצעות רבות להנדסת אוכלוסיות יתושים עם עמידות לנגיף למניעת מחלות הובאו; עם זאת, אף טכנולוגיה נוכחית אינה מסוגלת להפוך יתושים לעמידים בו זמנית בפני כל נגיפי ה- RNA המשמעותיים18,19,20,21,22,23. מערכות Cas המיועדות ל-RNA עשויות לספק נקודת התחלה לטכנולוגיה כזו על-ידי הפעלת פלטפורמה ניתנת לתכנות למיקוד כל נגיפי הרנ"א המועברים על ידי יתושים.
Protocol
1. בכל מקום ב- vivo RNA פילוח באמצעות מערכת CasRx שני רכיבים
- יצירת וקטור ביטוי Ubiq-CasRx ו- Ubiq-dCasRx
- להגביר את רצף CasRx באמצעות תגובת שרשרת פולימראז (PCR) עם פריימר 1050E. סי-3 ו-1050-אי. C4 ואת ה-CasRx המקורי לבנות pNLS-RfxCas13d-NLS-HA (pCasRx); ולהגביר את רצף dCasRx באמצעות PCR עם פריימר 1050E. סי-3 ו-1050-אי. C4 ואת המקורי dCasRx לבנות pNLS-dRfxCas13d-NLS-HA (pdCasRx)4 (טבלה 1). טהרו את שברי ה-CasRx וה-dCasRx המוגברים לאחר מכן באמצעות ערכת טיהור ג'ל.
- תקציר וקטור בסיס (Addgene plasmid #112686) עם אנזימי הגבלה SwaI ו PacI24. במוצרים המתקבלים, השתמש בערכה כדי לטהר את החלק הגדול יותר, הנקרא עמוד השדרה הווקטורי הבסיסי.
- להרכיב את וקטור Ubiq-CasRx עם עמוד השדרה וקטור הבסיס ואת שבר CasRx באמצעות שיטת ההרכבה גיבסון; להרכיב את וקטור Ubiq-dCasRx עם עמוד השדרה וקטור הבסיס ואת קטע dCasRx באמצעות שיטת ההרכבה גיבסון25.
הערה: מזהה התוספת של וקטור Ubiq-CasRx (OA-1050E) הוא #132416, ומזהה ה-Addgene של וקטור Ubiq-dCasRx (OA-1050R) הוא #132417.
- יצירת וקטור ביטוי gRNA
- עצב כל שבר gRNA בהתבסס על הקריטריונים הבאים: רצף היעד הוא 30 נוקלאוטידים באורך; האורך המרבי של מתיחות פולי-U ברצף היעד הוא 4 זוגות בסיס; תוכן GC רצף היעד להיות בטווח של 30% - 70%; רצף היעד צפוי לא ליצור מבני סיכת ראש RNA חזקים; ורצף היעד המכיל מבנה RNA משני או שלשלעי מינימלי5.
הערה: מחקר זה תכנן כל gRNA כ-4 רצפי טנדם באורך של כל 30 נוקלאוטידים, במרווחים של 36 חזרות ישירות ארוכות של נוקלאוטיד, ועם שליחות קטלנית של 7 תימין בשני הקצוות5. עבור גן היעד האקסוגני, GFP, אותם קריטריונים כמו לעיל היה אחריו עם תוספת של קטע OpIE2-GFP5. - להגביר את רצף מקדם U6:3 באמצעות PCR עם פריימרים 1043.C1 ו- 1043.C23 ו- Addgene plasmid #112688 (טבלה 1)26. טהר את שברי U6:3 המוגברים באמצעות ערכת טיהור ג'ל.
- תקציר תוסיפן פלסמיד #112688 עם אנזים הגבלה AscI ו- XbaI24. במוצרים המתקבלים, השתמש בערכה כדי לטהר את השברים הגדולים יותר, הנקרא עמוד השדרה הווקטורי שלפני הבסיס.
- להרכיב את וקטור הבסיס עם עמוד השדרה הווקטורי לפני הבסיס ואת קטע U6:3 באמצעות שיטת ההרכבה גיבסון25. וקטור הבסיס להלן נקרא OA-1043.
הערה: מזהה ה-Addgene של פלסמיד OA-1043 הוא #164586. - לסנתז שבר gRNA של גן היעד באמצעות שירות סינתזת גנים חיצוני.
- לעכל את וקטור הבסיס OA-1043 עם אנזים הגבלה PstI ו NotI24. שמור את כל מוצר העיכול, אשר נקרא OA-1043 מעוכל.
- להרכיב וקטור ביטוי gRNA עם OA-1043 מעוכל ושבר gRNA גן היעד באמצעות שיטת ההרכבה גיבסון25.
הערה: ארבעה גנים היעד נחקרו: שלושה היו אנדוגני (לבן, חריץ, צהוב), אחד היה אקסוגני (GFP). תעודות הזהות של Addgene הן: #132420 (gRNAw), #132421(gRNAN), #132425 (gRNAy) ו-#133304 (gRNAGFP).
- עצב כל שבר gRNA בהתבסס על הקריטריונים הבאים: רצף היעד הוא 30 נוקלאוטידים באורך; האורך המרבי של מתיחות פולי-U ברצף היעד הוא 4 זוגות בסיס; תוכן GC רצף היעד להיות בטווח של 30% - 70%; רצף היעד צפוי לא ליצור מבני סיכת ראש RNA חזקים; ורצף היעד המכיל מבנה RNA משני או שלשלעי מינימלי5.
- יצירת זבובים מהונדסים
- הזרקו וקטורים של ביטוי לעוברים מעופפים באמצעות שירות הזרקת עוברי זבובים חיצוניים ועוברים מזבובים המכילים אתרי אינטגרציה ØC31. תחלקו את העוברים המוזרקים במהירות של 26 מעלות צלזיוס.
הערה: attp40w (עם אתרי אינטגרציה על קו כרומוזום 2) שימש ליצירת קווי CasRx ו 8622 (עם אתרי שילוב על כרומוזום 3) שימש ליצירת קווי gRNA שונים. - שמור את הזבובים או כמו קווי הומוזיגוס או כמו קווים הטרוזיגוס מאוזנים.
הערה: זבובי Ubiq-CasRx ו- Ubiq-dCasRx נשמרו כקווים מאוזנים עם CyO כמאזן. בנוסף, הן Ubiq-CasRx והן וקטורים Ubiq-dCasRx מכילים סמן dsRed. כתוצאה מכך, זבובי Ubiq-CasRx ו- Ubiq-dCasRx כוללים את שלושת הפנוטיפים הבאים: dsRed-חיובי, כנפיים מתולתלות ועיניים לבנות. הזבובים המביעים GRNA נשמרו כקווי הומוזיגוס. מספרי המניות שלהם ב-Bloomington Drosophila Stock Center (BDSC) הם: #84118 (Ubiq-CasRx), #84119 (Ubiq-dCasRx), #84124 (gRNAw), #84122 (gRNAN), #84123 (gRNAy), #84986 (gRNAGFP).
- הזרקו וקטורים של ביטוי לעוברים מעופפים באמצעות שירות הזרקת עוברי זבובים חיצוניים ועוברים מזבובים המכילים אתרי אינטגרציה ØC31. תחלקו את העוברים המוזרקים במהירות של 26 מעלות צלזיוס.
- גנטיקה של זבובים (איור 1A)
- לאסוף 10 זבובים נקבה בוגרת בתולה מקו gRNA הומוזיגוס ולאסוף 5 זבובים זכר בוגרים מן הטרוזיגוס מאוזן Ubiq-CasRx / CyO קו. שים את הזבובים הנשיים והזכריים שנאספו, הנקראים זבובי ההורים, במין בתוספת אבקת שמרים יבשים (איור 1A).
- חזור על השלב הקודם 3 פעמים כדי ליצור 3 שכפולים. עבור קבוצת הביקורת, השתמש בקו Ubiq-dCasRx/CyO תוך שמירה על כל השאר זהה.
הערה: עבור בקבוקון זכוכית רגיל של מזון, 0.1 גרם של אבקת שמרים יבשים מספיק. מתכון מזון הזבובים של ה-BDSC משמש. - תחלקו את הבקבוקונים המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות. ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (50 °F) לפחות 20 ימים.
- שימו לב לבקבוקונים כל יום כדי לראות אם צאצאים בוגרים חדשים יצאו מהגולם של דור F1. אם כן, יש למרדים אותם עם פחמן דו-חמצני על ידי החדרת צינור המחובר למיכל דו תחמוצת הפחמן בתוך בקבוקוני הזבוב, ואז להפעיל את מתג הזרימה למשך 10 שניות.
- ברגע שהזבובים הופכים לבלתי מובילים, רוקנו אותם מהבילול אל משטח זבובים, המחובר גם למיכל הפחמן הדו-חמצני ופחמן דו-חמצני זורם ללא הרף דרך משטח הזבוב.
- ציון הפנוטיפ של זבובים מרדים ולצלם אותם באמצעות מצלמת צבע מחובר סטרימיקורוסקופ פלואורסצנטי. לספור את המספרים של צאצאים עם פנוטיפים שונים. השתמש בתוכנת עיבוד תמונה עבור תמונה לאחר עיבוד והידור (איור 2A - 2D).
הערה: בהתבסס על הגנטיקה של מנדליה, שני סוגים של זבובים צפויים בין הצאצאים לכל צלב (איור 1A).
- RNA-Seq (איור 2E – 2G)
- אוסף לדוגמה
הערה: בחר שיטת איסוף דוגמאות מתאימה מתוך 3 הדוגמאות שלהלן; נדרשים 3 שכפולים עבור כל סוג דגימה נפרד.- אוסף דוגמאות לראש זבוב למבוגרים
- לאסוף 10 זבובים נקבה בוגרת בתולה מקו gRNA הומוזיגוס. לאסוף 5 זבובים זכר בוגרים מן הטרוזיגוס מאוזן Ubiq-CasRx / CyO קו. שים את זבובי הנקבה והזכר שנאספו, שהם זבובי ההורים, במין בתוספת אבקת שמרים יבשים.
- חזור על השלב הקודם 3 פעמים עבור 3 שכפולים. עבור קבוצת הביקורת, השתמש בקו Ubiq-dCasRx/CyO תוך שמירה על כל השאר זהה.
- תחלקו את הבקבוקונים המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות. ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (55 °F) עד צאצאים לצאת גלמים.
- לאסוף 10 זבובים מבוגר יום אחד עם פנוטיפ הנכון. להרדים את הזבובים עם פחמן דו חמצני, ואז לחתוך את ראש הזבוב ולשים את הראשים בצינור צנטריפוגה 1.5 מ"ל על קרח יבש. לאחסן את צינור הצנטריפוגה ב -80 °C (80 °F). חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- אוסף מדגמי עובר בן 17 – 20 שעות
- לאסוף 8-10 זבובים נקבה בוגרת בתולה מקו gRNA הומוזיגוס. לאסוף 4-5 זבובים זכר בוגרים מן הטרוזיגוס מאוזן Ubiq-CasRx / CyO קו. שים את זבובי הנקבה והזכר שנאספו, שהם זבובי ההורים, במין בתוספת אבקת שמרים יבשים.
- חזור על השלב הקודם 3 פעמים עבור 3 שכפולים. עבור קבוצת הביקורת, השתמש בקו Ubiq-dCasRx/CyO תוך שמירה על כל השאר זהה.
- תחלקו את הבקבוקונים המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות.
- הכן תא אוסף אחד של מיץ ענבים לכל שכפול בעקבות המתכון הזה: 376 מ"ל מים, 126 מ"ל של מיץ ענבים, 15 גרם אגר ו -6 גרם סוכרוז. שים את התקשורת בתוך 1 L כוסות ומיקרוגל אותו על גבוה במשך 5-6 דקות תוך שמירה מקרוב על התקשורת בכוס כדי לבדוק אם בועות / קצף מופיע. אם כן, לעצור את המיקרוגל ולתת הבועה / קצף להתיישב. המשך למיקרוגל בדרך זו עד הבועה מתבהרת. אין להסתובב עד שכל הבועות יתבהרו. לבסוף, להוסיף 10 מ"ל של 100% אלכוהול ו 5 מ"ל של חומצה אצטית. מערבבים היטב, ואז מציפים את המדיה לתוך 35 מ"מ פטרי מנות עם צינור סרולוגי 25 מ"ל. כאשר התקשורת מתמצק בצלחת פטרי, היא מוכנה לשימוש.
- בסוף הדגירה בת 48 השעות, העבירו זבובי הורים לתאי איסוף העוברים של מיץ ענבים ודגרו אותם ב-26 מעלות צלזיוס למשך 3 שעות. לאחר מכן להסיר את הזבובים הבוגרים תוך שמירה על העוברים טריים הניח על צלחות מיץ ענבים עוד 17 שעות ב 26 °C (70 °F).
- לאחר הדגירה, לאסוף את 50 - 100 עוברים מן הצלחות מיץ ענבים, לנקות את פני העובר על ידי שקוע אותם במים deionized, ולאחר מכן להעביר אותם צינור צנטריפוגה 1.5 מ"ל על קרח. לאחסן אותם ב -80 °C (50 °F). חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- אוסף דגימות זחלי Instar הראשון
- לאסוף 8-10 זבובים נקבה בוגרת בתולה מקו gRNA הומוזיגוס. לאסוף 4-5 זבובים זכר בוגרים מן הטרוזיגוס מאוזן Ubiq-CasRx / CyO קו. שים את זבובי הנקבה והזכר שנאספו, שהם זבובי ההורים, במין בתוספת אבקת שמרים יבשים. חזור על שלב זה 3 פעמים עבור 3 שכפולים. עבור קבוצת הביקורת, השתמש בקו Ubiq-dCasRx/CyO תוך שמירה על כל השאר זהה.
- תחלקו את הבקבוקונים המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות. לאחר מכן להעביר את הזבובים למבוגרים לקבאל מזון רגיל חדש אחר לדגירה לילית (16 שעות) בשעה 26 מעלות צלזיוס. לאחר מכן, הסר את הזבובים הבוגרים.
- שמור את הנקניקייה המכילה עובר ב 26 °C (24 °F) עבור 24 שעות, ולאחר מכן להבקיע את זחלי instar הראשון transheterozygous מתחת למיקרוסקופ באמצעות סמנים נפרדים מבוססים. לאסוף 15-30 זחלים עם פנוטיפים נכונים ולשים אותם לתוך צינור צנטריפוגה 1.5 מ"ל ולאחסן אותם ב -80 °C (80 °F). חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- אוסף דוגמאות לראש זבוב למבוגרים
- רצף
- מיצוי RNA: השתמש בערכת חילוץ RNA זמינה מסחרית ובצע את הוראת הערכה לעיבוד כל הדגימות. לאחר מכן, לדגור על דגימות RNA שחולצו עם deoxyribonuclease זמין מסחרית ופעל לפי ההוראות שלה כדי להסיר כל DNA מזהם מן הדגימות.
- מדוד ריכוז RNA באמצעות ספקטרופוטומטר UV-vis זמין מסחרית. מדוד את תקינות הרנ"א בדגימות באמצעות התקנה של בדיקת תקינות RNA הזמינה מסחרית.
- בנה את ספריות ה- RNA-seq באמצעות ערכת הכנת ספריית RNA הזמינה מסחרית.
- השתמש בשירות רצף חיצוני עבור ריצוף ספריה עם ההגדרות הבאות: מצב קריאה בודד; אורך קריאה: 50nt, עומק: 20 מיליון קריאות לכל ספריה. בצע שיחות בסיס עם RTA 1.18.64 ולאחר מכן המיר את הנתונים ל- FASTQ באמצעות bcl2fastq 1.8.4.
הערה: נתוני רצף גולמיים ניתן למצוא במרכז הלאומי עבור ביוטכנולוגיה מידע ריצוף ארכיון לקרוא (מזהה השליחה: SUB6818910 [BioProject: PRJNA600654]).
- ביואינפורמטיקה
- המפה קוראת מנתוני הרצף כדי לשחרר 6 גנום drosophila melanogaster מפרויקט הגנום של ברקלי Drosophila (מספר גישה GenBank: GCA_000001215.4) ואת רצפי CasRx ו - GFP אקסוגני באמצעות הגדרת הפרמטר ברירת המחדל של STAR aligner28 עם תוספת של אפשרות המסנן "-outFilterType BySJout" ו- "-sjdbGTFfile Drosophila_melanogaster. BDGP6.22.97.gtf" קובץ פורמט העברת גנים מ- ENSEMBL.
- קבע את ספירת התמלילים הגולמית עבור כל תעתיק מובא עם התכונה Counts35 באמצעות האפשרויות "-t exon -g gene_id -M-O -- fraction". לאחר מכן, נרמל ספירות תמלילים גולמיות באמצעות ספירת תמלילים כוללת באמצעות סקריפט פרל "addTpmFpkmToFeatureCounts.pl".
- השתמש בשיטה האחורית המרבית עם אומדן הכיווץ המקורי בצנרת DESeq2 כדי להעריך את שינוי הקיפול הלוגריתמי (LFC) של כל גן.
- אוסף לדוגמה
2. בכל מקום ב- vivo אקסוגני RNA פילוח באמצעות מערכת CasRx שלושה רכיבים
- יצירת וקטור ביטוי אקסוגני בכל מקום
- PCR להגביר את שבר מקדם Ubiq באמצעות פריימרים 1052B. סי-1 ו-1052-בי. C2 ו הוסיף plasmid #112686 26. לאחר מכן, PCR להגביר את שבר T2A-eGFP מוגבר מ Addgene plasmid #112686 עם פריימרים 908A.1 ו 908A.2 (טבלה 1)26. לאחר מכן, PCR להגביר את קטע מקדם Ubiq כמו רצף הפוך באמצעות Addgene plasmid #112686 עם פריימרים 908A.3 ו 908A.4 (טבלה 1)26. טהר את שבר מקדם Ubiq, את שבר T2A-eGFP ואת שבר מקדם Ubiq ההפוך באמצעות ערכת טיהור ג'ל.
- הזמינו רצף קידוד לוציפראז גחלילית מותאם אישית ושבר מותאם אישית המכיל שבר p10 3'UTR, רנה לוציפראז הפוכה ואחריו שבר SV40 3'UTR.
- תקציר תוסיפן פלסמיד #112688 עם אנזים הגבלה AscI ו- XbaI24. במוצרים המתקבלים, ג'ל לטהר את השברים הגדולים יותר באמצעות ערכת טיהור ג'ל, אשר נקרא עמוד השדרה וקטור הבסיס.
- השתמש בשיטת ההרכבה של גיבסון כדי להרכיב וקטור בסיס עם עמוד השדרה וקטור הבסיס ואת השברים הבאים: שבר מקדם Ubiq, שבר T2A-eGFP, שבר מקדם Ubiq הפוך, רצף קידוד לוציפראז גחלילית, ואת לוציפראז רנה הפוכה ואחריו שבר SV40 3'UTR25.
הערה: מזהה Addgene של וקטור הביטוי הלוציפראז הכפול המתקבל (OA-1052B) הוא #132426.
- יצירת וקטור ביטוי gRNA
- PCR להגביר את רצף מקדם U6:3 באמצעות פריימרים 1043.C1 ו 1043.C23 ו הוסיף plasmid #112688 (טבלה 1)26. טהר את שברי U6:3 המוגברים באמצעות ערכת טיהור ג'ל.
- תקציר Addgene plasmid #112688 עם אנזים הגבלה AscI ו XbaI24. במוצרים המתקבלים, ג'ל לטהר את השברים הגדולים יותר, אשר נקרא עמוד השדרה וקטור טרום הבסיס, באמצעות ערכת טיהור ג'ל.
- להרכיב וקטור בסיס עם עמוד השדרה הווקטורי לפני הבסיס ואת קטע U6:3 באמצעות שיטת ההרכבה גיבסון25. וקטור הבסיס להלן נקרא OA-1043.
- לסנתז שבר gRNA של גן היעד באמצעות שירות סינתזת גנים חיצוני.
- לעכל את וקטור הבסיס OA-1043 עם אנזים הגבלה PstI ו NotI24. שמור את כל מוצר העיכול, אשר נקרא מעוכל OA-1043.
- להרכיב וקטור ביטוי gRNA עם OA-1043 מעוכל ושבר gRNA גן היעד באמצעות שיטת ההרכבה גיבסון25.
הערה: מזהה התוספת של הפלסמיד המתקבל (OA-1052K) הוא #132422.
- יצירת זבובים מהונדסים
- הזרקו וקטור OA-1052B לעוברים מעופפים באמצעות עוברים מזבובים המכילים אתר אינטגרציה ØC31 בכרומוזום השלישי, מלאי זבוב BDSC מספר 9744, באמצעות שירות הזרקת עוברי זבובים חיצוניים. באופן דומה, הזריקו וקטור OA-1052K לעוברים מעופפים באמצעות עוברים מזבובים המכילים אתר אינטגרציה ØC31 בכרומוזום השלישי, מלאי זבוב BDSC מספר 8622. תחלקו את העוברים המוזרקים במהירות של 26 מעלות צלזיוס.
- שמור על זבובים כפולים-לוציפראז ואת זבובי ה- gRNA כקווי הומוזיגוס; שמור על קווי Ubiq-CasRx כקווי הטרוזיגווס מאוזנים כפולים על ידי עקירת קו הטרוזיגוס Ubiq-CasRx המאוזן ביותר שנוצר בסעיף 1 לקווים מאזניים הנושאים כרומוזום מאזן TM6 עם סמן זיפים (Stb) ולשמור רק על הצאצאים המאוזנים הכפולים עם עיניים לבנות, כנפיים מתולתלות ופנוטיפים dsRed-פלואורסצנטיים בו זמנית.
הערה: מספרי המניות לטוס BDSC הם: #84127 (Ubiq-Fluc-Rluc), #84125 (gRNAFluc).
- גנטיקה של זבובים (איור 1B ואיור 3A)
- לאסוף 8-10 זבובים בוגרים בתולה מהקו הדו-לוציפראז מבטא. לאסוף 4-5 זבובים זכר בוגר מן הטרוזיגוס מאוזן Ubiq-CasRx / CyO; +/TM6, קו Stb המציג כנפיים לבנות עיניים, כנפיים מתולתלות, ופלואורסצנטיות dsRed בו זמנית. שים את זבובי הנקבה והזכר שנאספו, שהם זבובי ההורים, במין בתוספת אבקת שמרים יבשים (להלן נקרא שלב 1 קרוס).
- חזור על השלב הקודם 3 פעמים עבור 3 שכפולים. עבור קבוצת הביקורת, השתמש ב- Ubiq-dCasRx/CyO; +/TM6, קו Stb תוך שמירה על כל השאר אותו הדבר.
- דרג את בקבוקוני הצלב שלב 1 המכילים את זבובי ההורים ב 26 °C (48 שעות). ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (55 °F) לפחות 14 ימים. במהלך תקופה זו, לאסוף 8-10 בתולות נקבה מקו ה-gRNA של הגרנ"א הומוזיגוס. חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- שימו לב לבקבוקוני הצלב שלב 1 כל יום כדי לראות אם זבוב מבוגר חדש מגיח מגולם. אם כן, להרדים אותם עם פחמן דו חמצני, לאסוף 5 זבובים זכרים המבטאים הן Ubiq-CasRx (או Ubiq-dCasRx) ואת כתב לוציפרז כפול מן הצאצאים ולשים אותם לתוך בריון חדש יחד עם 10 נקבות בתולה מקו gRNA לוציפראז גליפראז גחלילית (להלן נקרא שלב 2 קרוס). חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- לאסוף עוד 5 גברים יום אחד מבטאים הן Ubiq-CasRx (או Ubiq-dCasRx) ואת כתב לוציפראז כפול מבקבוקוני הצלב שלב 1 ודגרה אותם במשך 2 - 4 ימים ב 26 °C (70 °F). לאחר מכן, להעביר אותם לתוך צינור צנטריפוגה 1.5 מ"ל ולאחסן אותם ב -80 °C (70 °F). חזור על שלב זה 3 פעמים עבור שלושה שכפולים.
- תחלקו את בקבוקוני הצלב שלב 2 המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות. ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (50 °F) לפחות 20 ימים.
- שימו לב לבקבוקונים של שלב 2 קרוס כל יום כדי לראות אם צאצאים בוגרים חדשים יצאו מהגולם. אם כן, למרדים אותם עם פחמן דו חמצני, להבקיע את הפנוטיפים של זבובים מרדים ולדמות אותם באמצעות מצלמת צבע מצויד סטריאוטירוסקופ פלואורסצנטי. לספור את המספרים של צאצאים עם פנוטיפים שונים. השתמש בתוכנת עיבוד תמונה עבור תמונה לאחר עיבוד והידור (איור 3B – 3C).
הערה: הגנטיקה של מנדליה מצביעה על כך שאם זבובים הם כולם בני קיימא, צפויים בין הצאצאים 4 סוגי זבובים מ-Step 2 Cross, שכל אחד מהם מהווה 25% מהאוכלוסייה (איור 1B ואיור 3A).
- אסיפת לוציפרזה (איור 3D)
- צור את זבובי הטרנסהטרוז'יגו המשולשים, כמו גם את זבובי הבקרה על ידי חזרה על שלבים 2.4.1-2.4.5. לאסוף זבובים זכרים בלידה ולהזדקן אותם עד גיל 3 ימים.
- העבר את זבובים 3 ימים לתוך צינורות צנטריפוגות 1.5 מ"ל ולזל אותם באמצעות עלה ואת מאגר תמוגה לוציפראז של ערכת בדיקת לוציפראז זמין מסחרית.
- השתמש 5 μL של רקמה lysed מכל מדגם כדי למדוד הן גחלילית ופעילות לוציפראז רנילה באמצעות ערכת בדיקת לוציפראז זמין מסחרית לומינומטר.
3. פילוח RNA ספציפי לרקמות באמצעות מערכת CasRx בעלת שלושה רכיבים
- יצירת וקטור ביטוי UAST-CasRx ו- UAST-dCasRx
- PCR להגביר את רצף מקדם UASt באמצעות pJFRC81 plasmid ו פריימרים 1041.C9 ו 1041.C11; לאחר מכן, PCR להגביר את קטע CasRx באמצעות plasmid OA-1050E (מזהה Addgene #132416) ו פריימרים 1050L. סי-1 ו-1050 אי. ג4; ולאחר מכן PCR להגביר את שברי dCasRx באמצעות plasmid OA-1050R (מזהה Addgene #132417) ו פריימרים 1050L. סי-1 ו-1050 אי. ג4 (טבלה 1)26. טהר את רצף מקדם UASt המוגבר, CasRx ו- dCasRx באמצעות ערכת טיהור ג'ל.
- תקציר וקטור בסיס (Addgene plasmid #112686) עם אנזימי הגבלה NotI ו PacI24. במוצרים המתקבלים, ג'ל לטהר את שבר גדול יותר, אשר נקרא עמוד השדרה וקטור הבסיס, באמצעות ערכת טיהור ג'ל.
- להרכיב את וקטור UASt-CasRx עם עמוד השדרה וקטור הבסיס, רצף מקדם UASt, וקטע CasRx באמצעות הרכבת גיבסון; לאחר מכן להרכיב את וקטור UASt-dCasRx עם עמוד השדרה וקטור הבסיס, רצף מקדם UAST, ואת קטע dCasRx באמצעות שיטת ההרכבה גיבסון25.
הערה: וקטור UASt-CasRx הוא Addgene plasmid #132418, והווקטור UASt-dCasRx הוא Addgene plasmid #132419
- יצירת זבובים מהונדסים
- הזרקת וקטור UASt-CasRx לעוברים מעופפים באמצעות שירות הזרקת עובר זבובים ועוברים מאתר האינטגרציה ØC31 8621 בכרומוזומים השניים שלהם; ואז להזריק וקטור UASt-dCasRx לתוך עוברים זבוב באמצעות שירות הזרקת עובר זבוב ועוברים מן זבובים ØC31 שילוב אתר 8621 על הכרומוזומים השני שלהם. תחלקו את העוברים המוזרקים במהירות של 26 מעלות צלזיוס.
- שמור על הזבובים כקווי הטרוזיגוס מאוזנים כפולים עם סמני CyO ו- Sb. הערה: מזהי קווי הטיסה ב- BDSC הם 84121 (UASt-CasRx) ו- 84120 (UASt-dCasRx).
- גנטיקה של זבובים (איור 1C)
- סדר שורות GAL4 הרצויות מ- BDSC; קבל קווי gRNA רלוונטיים מהשלב 3.2.2 (או מ- BDSC).
הערה: נעשה שימוש ב-2 זבובי GAL4 הבאים מ- BDSC: GAL4-GMR (מזהה BDSC: #29967), GAL4-y (מזהה BDSC: #44373). נעשה שימוש באותם 3 קווי gRNA שנוצרו בסעיף הראשון: gRNAw (מזהה BDSC: #84124), gRNAN (מזהה BDSC #84122), gRNAy (מזהה BDSC: #84123). - לאסוף 5-10 בתולה מבוגרות זבובים מקו gRNA. לאסוף 2-4 זבובים זכר בוגר מן הטרוזיגוס מאוזן כפול UAST-CasRx / CyO; +/TM6, קו Sb המציג עיניים לבנות, כנפיים מתולתלות ופלואורסצנטיות dsRed בו זמנית. שים את זבובי הנקבה והזכר שנאספו, שהם זבובי ההורים, במין מזון רגיל (להלן נקרא שלב 1 קרוס). חזור על שלב זה 3 פעמים עבור 3 שכפולים. עבור קבוצת הביקורת, השתמש ב- UASt-dCasRx/CyO; +/TM6, קו Sb תוך שמירה על כל השאר אותו הדבר.
- דרג את בקבוקוני הצלב שלב 1 המכילים את זבובי ההורים ב 26 °C (48 שעות). ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (55 °F) לפחות 14 ימים. במהלך תקופה זו, לאסוף 5-10 בתולות נקבה מקו GAL4. חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- שימו לב לבקבוקוני הצלב שלב 1 כל יום כדי לראות אם זבוב מבוגר חדש מגיח מגולם. אם כן, למרדים אותם עם פחמן דו חמצני, לאסוף 2-4 זבובים זכרים המבטאים הן את UASt-CasRx (או UASt-dCasRx) ואת וקטור gRNA מן האברכים אשר בו זמנית יש פנוטיפים dsRed-פלואורסצנטי וזיפים. שים את הזכרים שנאספו מצעד 1 קרוס לתוך בוחן חדש יחד עם 5-10 נקבה בתולה שנאספו מקו GAL4 (להלן נקרא שלב 2 קרוס). חזור על שלב זה 3 פעמים עבור 3 שכפולים.
- תחלקו את בקבוקוני הצלב שלב 2 המכילים את זבובי ההורים ב-26 מעלות צלזיוס למשך 48 שעות. ואז להסיר את כל זבובי ההורים מכל מקטורון. לאחר מכן לשמור על הבקבוקונים ב 26 °C (50 °F) לפחות 20 ימים.
- שימו לב לבקבוקונים של שלב 2 קרוס כל יום כדי לראות אם זבוב מבוגר חדש מגיח מהגולם. אם כן, למרדים אותם עם פחמן דו חמצני, להבקיע את הפנוטיפים של זבובים מרדים ולדמות אותם באמצעות מצלמת צבע מצויד סטריאוטירוסקופ פלואורסצנטי. לספור את המספרים של צאצאים עם פנוטיפים שונים. השתמש בתוכנת עיבוד תמונה עבור תמונה לאחר עיבוד והידור (איור 4).
הערה: הגנטיקה של מנדליה מצביעה על כך שאם זבובים הם כולם בני קיימא, צפויים בין הצאצאים 4 סוגים של זבובים מ-Step 2 Cross, שכל אחד מהם מהווה 25% מהאוכלוסייה (איור 1C).
- סדר שורות GAL4 הרצויות מ- BDSC; קבל קווי gRNA רלוונטיים מהשלב 3.2.2 (או מ- BDSC).
תוצאות
בכל מקום ב-vivo RNA פילוח באמצעות מערכת CasRx בעלת שני רכיבים
זבובי הטרנסהטרוזיגוus F1 המבטאים הן את ה-Ubiq-CasRx והן את ה-GRNA (המתמקדים הן בגנים אנדוגניים והן גנים אקסוגניים) הראו פנוטיפים מסומנים בהשוואה לזבובי הבקרה המבטאים את מבני Ubiq-dCasRx ו-gRNA (איור 2 ואיור 4). באופן ספציפי, לזבובי ה-CasRx transheterozygous יש רמות נמוכות משמעותית של שיעור הישרדות בהשוואה לזבובי dCasRx הטרנס-טרנזיים, מה שמעיד על רעילות של מערכת Ubiq-CasRx (איור 2A ואיור 4A). ראוי לציין כי הן transheterozygous CasRx והן זבובי dCasRx יש פחות מ 50% שיעור ירושה, המהווה את היחס הצפוי המבוסס על גנטיקה מנדליאנית. מתוך שלושת הגנים היעד, Ubiq-CasRx /+; U6-gRNAN/+ טסים ו-Ubiq-CasRx/+; זבובי U6-gRNAy/+ אינם ברי קיימא (0% ירושה) ולא גדלו מעבר לשלב הזחלים השני (איור 2A-2B). Ubiq-CasRx ששרד / +; זבובי U6-gRNAw/+ נחלתם הייתה 12.9%, הראו פנוטיפ ברור בעל עיניים לבנות (איור 2B). בנוסף לתכונות נצפות הקשורות ל-CasRx, הצלחנו לאשר הפחתה משמעותית של תמלילי גנים ממוקדים עבור 3 גנים ממוקדים: חריץ, צהוב ו-GFP (איור 2E-2G). הפחתת תמלילי גנים לבנים נצפתה בזבובי Ubiq-CasRx/+, U6-gRNAw/+ בהשוואה לזבובי Ubiq-dCasRx/+, U6-gRNAw/+ עף, אם כי ההפחתה לא הייתה משמעותית סטטיסטית (איור 2E - 2F). נמצאו ראיות לפעילות מחוץ למטרה שנגרמה על-ידי CasRx כאשר משווים בין התמלילים המובעים באופן דיפרנציאלי בין דגימות מזבובים המביעים CasRx לבין דגימות מזבובים המביעים dCasRx (איור 2E, 2G). מספר התמלילים שאינם מטרות המתבטאים באופן דיפרנציאלי באופן משמעותי הם כדלקמן: לבן, 253 (1.4% מסך התמלילים); חריץ,300 (1.7%); צהוב, 41 (0.23%); GFP, 5,880 (33%) (איור 2G). מתוך 17,779 תמלילים שונים, 6 תמלילים שאינם מטרות באו לידי ביטוי באופן דיפרנציאלי משמעותי בכל 4 קבוצות הדגימות. אחד מ-6 התמלילים שזוהו היה Gadd45, גן המעורב באפופטוזיס ובמעצר תאי בזבובים, מה שמעלה את האפשרות שהפעולה האנזימטית של CasRx עלולה לעורר ישירות אפופטוזיס תאי או לגרום בעקיפין לביטוי שגוי של גנים אחרים, מה שמוביל לאפופטוזיס. לבסוף, ראוי לציין כי זבובי Ubiq-CasRx ו- Ubiq-dCasRx לא הוקמו כמניות הומוזיגוס, ככל הנראה בשל רעילות שהוענקה על ידי ביטוי גבוה בכל מקום. כתוצאה מכך, הזבובים הטרוזיגוס Ubiq-CasRx/CyO ו- Ubiq-dCasRx/CyO שימשו למעבר עם קווי זבוב gRNA הומוזיגוס. לסיכום, מערכת Ubiq-CasRx בעלת שני הרכיבים מסוגלת להשיג מיקוד RNA בכל מקום עבור מטרות אנדוגניות ואקסוגניות וכתוצאה מכך פנוטיפים נצפים והפחתת תעתיק. תוצאות אלה הראו גם כי פילוח RNA בתיווך CasRx עלול להציג רעילות ב vivo.
בכל מקום ב-vivo אקסוגני RNA פילוח באמצעות מערכת CasRx שלושה רכיבים
התוצאות של הצלב הדו-שלבי הראו שלמרות האופי האקסוגני של גן המטרה (כלומר, Fluc), המבטא את כל שלושת הטרנסג'נים בטרנסהטרוזיגואטים משולשים F2 (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) הביא לקטלניות של 100% בהשוואה לצלבי שליטה הקשורים ל- Ubiq-dCasRx, שם לא נצפתה קטלניות בתמרות המשולשות F2 (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) (איור 3B-C ). ליתר דיוק, רק השילוב של כל שלושת הטרנסג'נים (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) הביא ל-100% קטלניות (איור 3B ו-D), בעוד (Ubiq-CasRx/+; gRNAFluc/TM6) ו-Ubiq-CasRx/+; גנוטיפים של Ubiq-Fluc-Ubiq-Rluc/TM6) היו בני קיימא וחסרים פנוטיפים עם שיעורי הירושה שלהם התואמים את שיעורי ההדבקה הצפויים של מנדליה, מה שמצביע על כך שזמינות רצף היעד (כלומר, לוציפראז גחלילית) בשילוב עם Ubiq-CasRx/+ וה- gRNAFluc הוא מה שגרם לפנוטיפים הקטלניים הנצפים, ככל הנראה הנובעים מפעילות המשנית של אנזימי Cas132,8 . בנוסף, אין פנוטיפים בולטים או השפעה דרמטית על ירושה בטרנסהטרוזיגוטס F1 (Ubiq-CasRx/+; gRNAFluc/+ או Ubiq-CasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) נצפו בהשוואה לבקרות Ubiq-dCasRx (Ubiq-dCasRx/+; gRNAFluc/+ או Ubiq-dCasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) (איור 3B), המציין כי אנזים פעיל קטליטית חיוני כדי להשיג את הפנוטיפים הקטלניים שנצפו. יתר על כן, רמות הביטוי Fluc ו Rluc בזבובים של כל הגנוטיפים קיימא לא הראו ירידה משמעותית ביטוי Fluc ב- Ubiq-dCasRx משולש transheterozygotes (Ubiq-dCasRx/+; gRNAFluc / Ubiq-Fluc-Ubiq-Rluc) לעומת פקדים כפולים של כתב לוציפראז. הדבר מצביע על כך שרמות ביטוי החלבון של Fluc לא הופחתו על ידי פילוח dCasRx (איור 3D). יחד, פנוטיפ הקטלניות הנפוץ בשני ניסויי מיקוד RNA שונים בתיווך CasRx בכל מקום מצביעים על כך שכאשר משתמשים בהם על רקמות בכל מקום, מיקוד RNA בתיווך CasRx יכול להיות רעיל לאורגניזם.
פילוח RNA ייחודי לרקמות באמצעות מערכת CasRx בעלת שלושה רכיבים
הרמה הגבוהה של רעילות שנצפתה בניסויי מיקוד RNA בכל מקום גרמה לנו לחקור את מיקוד ה- RNA הספציפי לרקמות באמצעות תכנון מערכת CasRx בעל שלושה רכיבים המפורט בסעיף השיטות. ואכן, רמת הרעילות שנצפתה הופחתה כאשר הביטוי הכללי CasRx הופחת באמצעות מקדם UASt בהשוואה לזה של מקדם Ubiq, זה בא לידי ביטוי בשלושה היבטים: 1) קווי UASt-CasRx ו- UASt-dCasRx נשמרו כקווי הומוזיגוס, אם כי בהתבסס על תוכנית הצלב הדו-שלבית כפול מאוזן UASt-CasRx ו- UASt-dCasRx שימשו לביצוע הצלבים, 2) כל שיעורי הירושה המשולשים של דור F2 dCasRx תואמים את שיעור הירושה הצפוי של 25% מנדליאנית, ו-3) פנוטיפ הקטלניות הטרנסה-טרנסהטרוזיגוס משולשת של דור ה-F2 הופחת במתינות. בניסוי פילוח לבן, מתוך 25% שיעורי הירושה המנדליים הצפויים בטרנסהטרוזיגוטים המשולשים F2, נצפו רק 0.57% זבובים בוגרים בני קיימא (UASt-CasRx/+; gRNAw/GMR-Gal4), שכולם הציגו פיגמנטציה חמורה ספציפית לעיניים ופנוטיפים מורפולוגיים (איור 4A ו-4B). עבור הצלב בעל המיקוד הלבן, שיעור הירושה המשולש של CasRx-express transheterozygous F2 היה נמוך משמעותית מזה של קבוצת הבקרה הטרנסה-טרנסהטרוזיגוס (27.6%) (איור 4A). בניסוי מיקוד חריץ, שיעור הירושה של בקרת DCasRx היה 29.3% (איור 4A). בניסוי פילוח צהוב, F2 משולש transheterozygous CasRx-מבטא, gRNAy, ו y-GAL4 הראה הפחתת פיגמנט כיטין שולית כתמים קטנים של עור צהוב על בית החזה והבטן עם שיעור ירושה של 2.67%, נמוך בהרבה מזה של קבוצת הביקורת dCasRx (25.2%) (איור 4A ). כל זבובי ה-dCasRx שולטים בטרנסהטרוזיגוס משולשת לא הציגו פנוטיפים ברורים כזבובים המביעים CasRx, מה שמצביע על כך שפעילות קטליטית של CasRx תרמה לפנוטיפים שנצפו. שיעור הירושה הנמוך בקבוצת הטרנסהטרוזיגוס המשולשת CasRx הציע כי קיימים שני מקורות לרעילות בפילוח הרנ"א של CasRx: האחד קשור לביטוי גבוה של CasRx, שהרעילות שלו הופחתה על ידי ביטוי CasRx מגביל, השני קשור לפעילות המשנית. יחד, תוצאות אלה הראו כי מערכת CasRx יכולה להשיג פילוח RNA ספציפי לרקמות על ידי מינוף מערכת Gal4 / UASt הקלאסית ובינתיים להפחית את הרעילות. עם זאת, רעילות ופנוטיפים קטלניים מדי פעם עדיין נצפו ברמה נמוכה יותר של חומרה לעומת זה של הגישות בכל מקום, המציין כי פעילות מחשוף בטחונות קשורה רעילות.
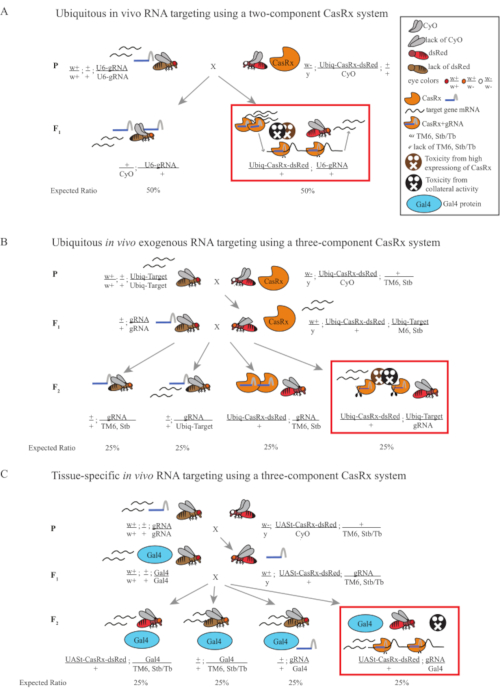
איור 1: סקירה כללית של פילוח RNA באמצעות מערכת Cas13D. (A) שרטוטים של הצלב הגנטי בעל השלב האחד במיקוד הרנ"א של vivo בכל מקום באמצעות מערכת CasRx בעלת שני רכיבים. (B) שרטוטים של צלב גנטי דו-שלבי בכל מקום ב- vivo אקסוגני RNA מיקוד באמצעות מערכת CasRx שלושה רכיבים. (C) שרטוטים של צלב גנטי דו-שלבי ב- RNA ספציפי לרקמה באמצעות מערכת CasRx בעלת שלושה רכיבים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
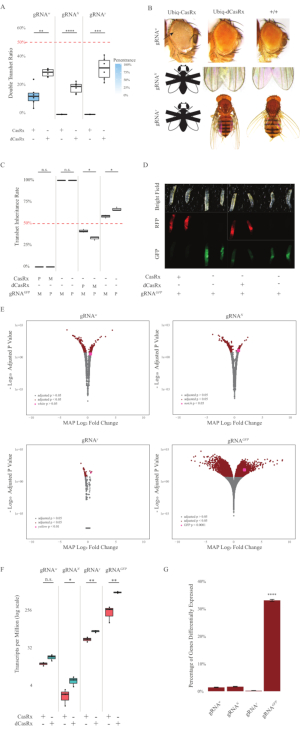
איור 2: בכל מקום ב-vivo RNA פילוח באמצעות מערכת CasRx דו-רכיבים (reprinted5). (A) אחוזי ירושה כוללים של זבובי טרנסהטרוזיגו היורשים את Ubiq-CasRx (או Ubiq-dCasRx) ו- gRNAs. הצללה כחולה בעלילת התיבה מצביעה על חנונים פנוטיפ. (B) פנוטיפים של זבובי טרנסהטרוזיגוס. החצים מצביעים על נמק רקמה בעין. זבוב שחור-לבן המסומן ב-'X' מייצג קטלניות. (ג) אחוזי ירושה כוללים של זבובי טרנסהטרוזיגוס של צלבים דו-כיווניים בין Ubiq-CasRx (או Ubiq-dCasRx) לבין זבובי gRNAGFP-OpIE2-GFP. M, ירושה אימהית של CasRx; P, ירושה אבהית של CasRx. (ד) צאצאי זחלי F1 בצלב האבהי. (ה) מקסימום הערכות אחוריות לשינוי הקיפול הלוגריתמי. נעשה שימוש בצינור DESeq2. (ו) תמלילים למיליון (TPM) המיועדים ל- CasRx או dCasRx. (ז) CasRx-depentent ביטא באופן דיפרנציאלי אחוז תמלילים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
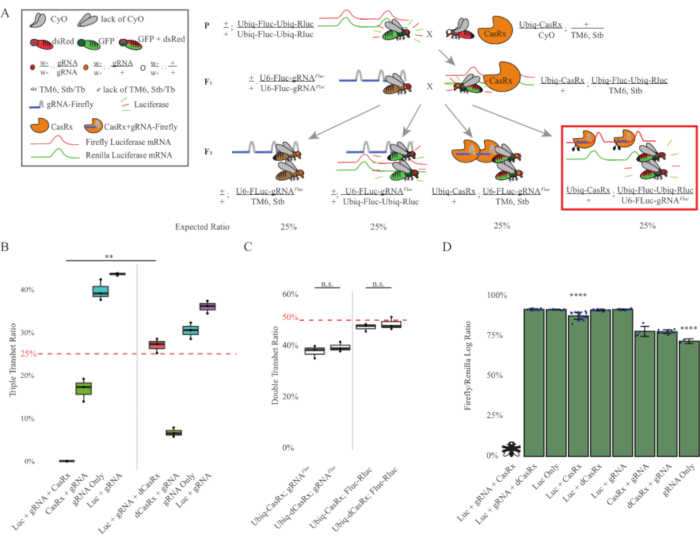
איור 3: בכל מקום ב-vivo exogenous RNA מיקוד באמצעות מערכת CasRx שלושה רכיבים. (A) שרטוטים של הצלב הגנטי דו-שלבי. (ב) סה"כ אחוזי ירושה עבור כל הגנוטיפים המתעוררים בדור F2 . בירושת כל שלושת הטרנסגנים (Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc ו- gRNAFLuc) בצאצאי F2 הביאה ל-100% קטלניות והייתה נמוכה משמעותית בהשוואה לקבוצת הביקורת הטרנסהטרוציגוטס המשולשת Ubiq-dCasRx (p = 0.001, t-test). (ג) נשיאת Ubiq-CasRx/gRNAFluc לבדה או Ubiq-CasRx ו- Ubiq-Fluc-Ubiq-Rluc לבדה לא הובילה לקטלניות חמורה, ויחסי הירושה בין Ubiq-CasRx ל- Ubiq-dCasRx transheterozygotes לא היו שונים באופן משמעותי (p = 0.41 ו- p = 0.51, בהתאמה, t-test). (ד) לוציפראז מנרמל קריאות Fluc לקריאות Rluc. זבובי טרנסהטרוזיגוס משולשים המבטאות את Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc, gRNAFLuc היו קטלניים עובריים, אשר היו מיוצגים על ידי זבוב עם "X", וכתוצאה מכך ביטוי לוציפראז לא נמדד. יחס Fluc/Rluc של Ubiq-CasRx/+, Ubiq-Fluc-Ubiq-Rluc/TM6, טרנסהטרוזיגוטס Stb היה נמוך משמעותית מזה של קבוצות ההבעה האחרות של Ubiq-Fluc-Ubiq-Rluc (p = 1.2e-06 ומטה, t-test). התוצאות של קבוצת gRNAFLuc בלבד היו נמוכות משמעותית מאלה של כל הקבוצות האחרות (p = 1.2e-06 ומטה, t-test). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
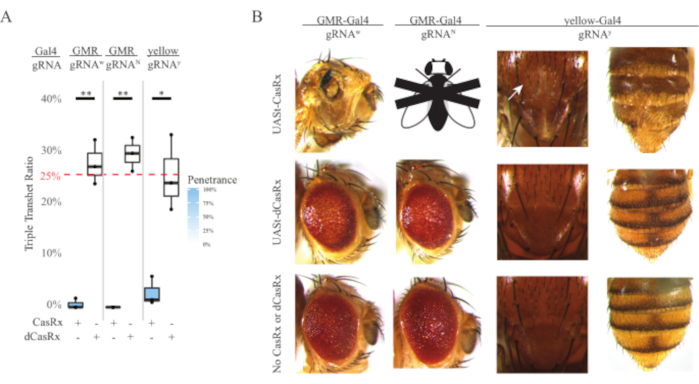
איור 4: פילוח RNA ספציפי לרקמות באמצעות מערכת CasRx בעלת שלושה רכיבים (מודפסת מחדש5). (A) אחוז הירושה הכולל של זבובי טרנס-טרנסג'ן (UASt-CasRx או UASt-dCasRx, gRNAs ו- Gal4-driver. (ב) פנוטיפים של זבובי הטרנסהטרוזיגו המשולשים. החץ הלבן מצביע על הפחתת פיגמנט כיטין בבית החזה. זבוב שחור-לבן המסומן ב-'X' מייצג קטלניות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
| מבנה | תיאור | תחל | רצף פריימר (5' עד 3') | תבנית PCR |
| OA-1050E | CasRx | 1050E. C3 | TACTAATTTTCCAC ATCTATTTTGAC CCGCAGATTAATTA אטגאגאקאקאגה אגהא | pNLS-RfxCas13d-NLS-HA (pCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTGG AACA | |||
| OA-1050R | dCasRx | 1050E. C3 | TACTAATTTTCCAC ATCTATTTTGAC CCGCAGATTAATTA אטגאגאקאקאגה אגהא | pNLS-dRfxCas13d-NLS-HA (pdCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTG GAACA | |||
| OA-1050L | מקדם UASt | 1041.C9 | GCGGGTTCTCGA CGGTCACGGCGG GCATGTCGACGC GGCCGCAACCAA CAACACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCACC TTTCTCTCTCTCT TGGCTCATGT TTAAACCCAATT CCCTATTCAGA | |||
| CasRx | 1050 ל'. C1 | אטאקאגאגה גקטקטאטה GGGAATTGGGT TTAAACATGAGC CCCAAGAAGAA | pCasRx | |
| 1050E. C4 | CAATTGATTTGT טאטאטאאאאק GATTCATTCTA GCTAGCTTAAG CGTAATCTGGA ACA | |||
| OA-1050S | מקדם UASt | 1041.C9 | GCGGGTTCTC GACGGTCACG GCGGGCATGT CGACGCGGCC GCAACCAACAA קקטאג | pJFRC81 |
| 1041.C11 | CTGGCCTCCA CCTCTCTC TTCTTGGGGCT CATGTTTAAAC CCAATTCCCTA TTCAGA | |||
| dCasRx | 1050 ל'. C1 | אטאקאג אגקטקטגאאט אגגאאטאג TTTAAACATGAG CCCCAAGAAGAA | pdCasRx | |
| 1050E. C4 | CAATTGATTTGT טאטאטאאאאק GATTCATTCTAG CTAGCTTAAGCG טאאטקטגאקה | |||
| OA-1043 | U6:3 מקדם | 1043.C1 | GGGAATTGGGA ATTGGGCAAT TTAAATGGCGGC GCGCCGAATTCT TTTGCTCACCT | Addgene plasmid #164586 |
| 1043.C23 | אקקטאגטאגאט CTAGAGGTAC CGTTGCGGCCG CAAAAAAGTTGT AATAGCCCCTCA AAACTGGACCTT CCACAACTGCAG CCGACGTTAAAT TGAAA | |||
| OA-1052B | מקדם Ubiq | 1052-בי. C1 | GGGAATTGGGCA אטטאאטאגק GGCTGCAGCGC GCAGATCGCCGAT | Addgene plasmid #112686 |
| 1052-בי. C2 | TTTCTTTATGTTT TTGGCGTCTTCC ATCCTAGGTCTG CGGGTCAAAATA גאגאטג | |||
| T2A-eGFP | 908A1 | אטאאאגקאאג AAGGGCGGAAA GATCGCCGTGG אגקאגאגה AGTCTTCTAACAT GC | Addgene plasmid #112686 | |
| 908A2 | TTGTTATTTTAAAA ACGATTCATTCTA GGCGATCGCTTA CTTGTACAGCTC GTCCATGCC | |||
| מקדם Ubiq הפוך | 908A3 | ACCGTGACCTAC ATCGTCGACACTA GTGGATCTCTAGA CGCGCAGATCGC CGATG | Addgene plasmid #112686 | |
| 908A4 | GGATCATAAACTT TCGAAGTCATGC GGCCGCTCTGCG GGTCAAAATAGAG ATGT |
טבלה 1: רשימת מבנים מולקולריים פריימרים המשמשים במחקר זה. רשימה זו כוללת את כל המבנים (הן המזהה והן התיאור) ואת הפריימרים המשויכים לכל מבנה (הן המזהה והן הרצפים (5' עד 3')) ותבניות המשמשות.
Discussion
עם שלושה עיצובי יישומים שונים של מערכת CasRx, עבודה זו הדגימה RNA לתכנות vivoprogramming בזבובים. האסטרטגיות השונות מספקות צרכי פרויקט שונים, כגון פילוח גנים אנדוגני לעומת אקסוגני ומיקוד RNA בכל מקום לעומת פילוח RNA ספציפי לרקמות. ההשפעות של פילוח RNA כללו שינויים פנוטיפיים ספציפיים לגן היעד, הפחתת תעתיק RNA היעד, ופנוטיפים קטלניות מדי פעם הקשורים לביטוי גבוה של חלבון CasRx ופעילות נלווית. בסך הכל, תוצאות אלה הראו כי מערכת CasRx מסוגלת למקד הפחתת תעתיק RNA ברמה האורגנית באופן ניתן לתכנות ויעיל.
אחד הגורמים העיקריים להתאמה אישית מוצלחת של מערכת CasRx הוא העיצוב של gRNAs. באופן ספציפי, העצה הבאה תהיה לשים לב: רצף היעד הוא סביב 30 נוקלאוטידים באורך, אורך מתיחות poly-U ברצף היעד הוא 4 זוגות בסיס או פחות, תוכן GC רצף היעד הוא בטווח של 30% - 70%, רצף היעד אינו צפוי ליצור מבני סיכת ראש RNA חזקים, ואת רצף היעד מכיל מבנה RNA משני או שלשלני חזוי מינימלי5.
בנוסף לתכנוני ה-gRNA, שלב הגנטיקה של הזבוב בכל פרוטוקול הוא קריטי גם ביישום מוצלח. נוכחות או היעדר פנוטיפים מוגדרים שהועברו מההורים בצאצאים חשובים לזיהוי וכימות פנוטיפים הנגרמים על ידי מערכת CasRx בצאצאי transheterozygous. כמו כן, הגדרת צלבי בקרה באמצעות זבובי dCasRx במקביל מועילים גם לפנוטיפים לא ספציפיים בצאצאי transheterozygous.
ראוי לציין כי תוצאות אלה חשפו בעיית רעילות שהוצגה על ידי הבעת CasRx וחלבון dCasRx בכל מקום בזבוב, מגבלה של מערכת CasRx. ביטוי בכל מקום של CasRx או dCasRx תחת מקדם Ubiq לבד, ללא gRNAs, הגיע עם עלויות כושר לא טריוויאלי, כמו לא זבובי Ubiq-CasRx ולא זבובי Ubiq-dCasRx יכול להיות מבוסס כמו קווי הומוזיגוס. להיפך, UASt-CasRx ו UASt-dCasRx זבובים ניתן לבסס כמו מניות הומוזיגוס בריא, אם כי בשל העיצוב של ערכת הצלב הם נשמרו כמו מלאי מאוזן כפול, עובדה התומכת בקיומה של רעילות הנגרמת על ידי ביטוי חלבון CasRx בכל מקום. ראיה תומכת נוספת היא כי בניסויי בקרה מעורבים dCasRx, אשר אינו פעיל מבחינה קטליטית, אחוזי הזבובים הנושאים הן dCasRx והן gRNA מבנים מתוך המספר הכולל של זבובים בדור F1 היו נמוכים בעקביות מ -50%, היחס הצפוי בהתבסס על גנטיקה מנדליאנית אם לא היה קיים רעילות הקשורה ל- dCasRx. זה הצביע על כך בכל מקום להביע dCasRx, יחד עם gRNAs, גורמים רעילות בזבוב, וכתוצאה מכך יחס ירושה פחות מהצפוי. יחסי הירושה של transheterozygous UASt-dCasRx, gRNA, זבובי GAL4 עקבו אחר הגנטיקה המנדליאנית, אשר שוב מרמז על הרעילות הנגרמת במיוחד על ידי ביטוי בכל מקום של חלבוני CasRx ו- dCasRx. רעילות במערכת CRISPR/Cas אינה חדשה. כמויות גבוהות של חלבון Cas9 הוכח להיות רעיל במספר אורגניזמים, כולל זבובים 29,30,31,32. מחקר שנערך לאחרונה פיתח מערכת GAL4/UAS מותאמת אישית שיכולה לכוונן את כמות חלבון Cas9 המתבטאת בזבובים על ידי הוספת מסגרת קריאה פתוחה באורך משתנה בין רצף UAS לבין רצף Cas9 במבנה UAS-Cas933. לכן, כדאי לבחון דרכים להפחית רעילות הנגרמת על ידי CasRx על ידי כוונון רמת ביטוי חלבון CasRx.
מלבד הרעילות הנגרמת על ידי ביטוי בכל מקום של חלבוני CasRx ו- dCasRx, התוצאות הראו גם קטלניות הקשורה לאפקטים החוץ ספציפיים של מערכת CasRx, תכונה של מערכות CRISPR רבות1,2,7,34. בחלק מהזבובים CasRx ו- gRNA הגן הלא חיוני מבטאים זבובים כפולים או משולשים transheterozygous, למשל בעת מיקוד Notch, זבובי Transheterozygous CasRx יש רמות נמוכות משמעותית של שיעור הישרדות לעומת זבובי dCasRx transheterozgyous. בניתוח RNA-seq של זבובי הטרנסה-טרנס-ארק אלה ומבטאי gRNA, נצפו הן הפחתת רמות תעתיק גן היעד והן הפחתת תמלילי גנים שאינם מטרה. השפעות נלוות אלה היו תלויות CasRx ותלויות מטרה, שכן הן נצפו רק בזבובי טרנסהטרוזיגוס המבטאות הן את חלבון ה- CasRx והן את ה- gRNA. ראוי לציין כי אחד הגנים היעד, לבן, הראה רק ירידה מוגבלת, לא סטטיסטית משמעותית בתעתיקים כאשר הגן הלבן היה ממוקד על ידי CasRx, אשר היה בניגוד פנוטיפ הפחתת פיגמנט ברור. ההשערה היא כי ייתכן שהסיבה לכך היא ש- 1) התזמון של איסוף מדגם RNA-seq לא היה מיושר היטב עם התזמון כאשר הגן הלבן מגיע לביטוי השיא שלו במהלך ההתפתחות המוקדמת, ו -2) הביטוי המקומי של הגן הלבן בעיניים הופך אותו למאתגר לאסוף את הרקמות הרלוונטיות בשלב הפיתוח המוקדם כאשר רק איסוף מדגם של כל הגוף הוא אפשרי. כדי להפחית את הפעילות המשנית במערכת CasRx, מחקרים עתידיים נקראים להבין באופן מלא את המנגנונים שבבסיס מערכת התופעה מחוץ למטרה ברמה האורגנית.
מעניין, מחקר שנערך לאחרונה35 המתאר כלי Cas13 ממוקדי RNA בזבובים נראה כדי לשפר את הרעילות הכללית הקשורה ביטוי CasRx, מכמה סיבות אפשריות. ראשית, המחברים מקודדים מחדש את הטרנסג'נס Cas13 כדי לייעל את הביטוי בדרוסופילה והשתמשו במקדם חלש יותר (actin 5C) בהשוואה למקדם היוביקוויטין המשמש במחקר הנוכחי, מה שהוביל ככל הנראה לרמות נמוכות יותר של ביטוי Cas13 ובכך פחות רעילות. ואכן, זה נתמך על ידי התצפיות כי UAST מונע CasRx וביטוי dCasRx לא היה, כשלעצמו, רעיל, כמו מחקר זה (ואת המחברים ב 35) לא הבחין כל קטלניות ברורה זבובים UASt-CasRx. יתר על כן, מחברים אלה מקודדים gRNAs שלהם באופן שונה בהשוואה למחקר זה, אשר עשוי להשפיע על הביטוי שלהם והפחית את הרעילות של המערכת זבובי transheterozygous Cas13/gRNA. לדוגמה, במחקר שלהם שני gRNAs באו לידי ביטוי באמצעות מקדם U6:3 ומאוגף על ידי tRNAs כדי לאפשר עיבוד gRNA על התבגרות tRNA ללא צורך CasRx35. לעומת זאת, במחקר זה, gRNAs קודדו כערכים המתמקדים עד 4 מיקומים לכל גן ומחקים את מבנה מערך Cas13 אנדוגני שנמצא בחיידקים, אשר דורש את האנזים Cas13 לעבד כל gRNA. גישות שונות אלה עשויות להוביל להבדלים ברמות ביטוי gRNA וגורמים אחרים שעשויים להיות להם השפעות מובנות על הרעילות של המערכת כולה. לבסוף, Huynh ואח ' ממוקד גנים שונים מאלה ממוקדים במחקר הנוכחי, אשר לגרום הבדלים באינטראקציה היעד-Cas / gRNA ופעילות בטחונות ועשויים להיות השפעות על רמות הנצפות של קטלניות. הבדלים אלה ברעילות שנצפתה מצדיקים חקירה נוספת כדי לזהות דרכים שבהן ניתן לשפר את המערכות הכוללות.
בסך הכל, מחקר זה הוא ההדגמה הראשונה של מערכת Cas ממוקדת גנטית פונקציונלית לתכנות ב- D. melanogaster, אם כי אופטימיזציה נוספת של מערכת CasRx (בהתאם למה שדווח35) תידרש להפחית עוד יותר את הקטלניות הקשורה למטרה ולהגדיל את היעילות של מחשוף CasRx על היעד. פילוח RNA עם אנזימי Cas הוא תחום המתפתח במהירות עם יישומים פוטנציאליים רבים החל בקרה וקטור חרקים לשימושים טיפוליים1,2,3,4,5,6,7, ופרוטוקול זה מציע חבילת התחלה לכל מי שמעוניין לעצב את מערכת CasRx הראשונה שלהם בזבובים, תוך התאמה להתאמה אישית ואופטימיזציה נוספת של המערכת. הדוגמאות המוצגות כאן מדגימים מגוון תוצאות שניתן להיתקל בהן במהלך יישום מערכת זו ב- vivo ועשויות לשמש אמות מידה עבור משתמשים אחרים בהערכת הביצועים של מערכת CasRx ביישומים שלהם.
Disclosures
O.S.A הוא מייסד Agragene, Inc. , יש אינטרס הון עצמי, ומכהן בוועדה המדעית המייעצת של החברה. תנאי הסדר זה נבדקו ואושרו על ידי אוניברסיטת קליפורניה בסן דייגו בהתאם למדיניות ניגוד העניינים שלה. כל הסופרים האחרים מצהירים שאין אינטרסים מתחרים.
Acknowledgements
עבודה זו נתמכה בחלקה על ידי מימון מענק תוכנית הגנים הבטוחים של DARPA (HR0011-17-2-0047), ופרסים של NIH (R21RAI149161A, DP2AI152071) שהוענק ל- O.S.A.
Materials
| Name | Company | Catalog Number | Comments |
| 100% Grape Juice | Welch Foods Inc. | N/A | |
| Active Dry Yeast (908g) | Red Star Yeast Company, LLC | N/A | |
| DMC4500 color camera | Leica Microsystems | DMC4500 | |
| Dual-Luciferase Reporter Assay System | Promega | E1910 | |
| GAL4-GMR flies | Bloomington Drosophila Stock Center | 29967 | |
| GAL4-y flies | Bloomington Drosophila Stock Center | 44373 | |
| Glomax 20/20 Luminometer | Promega | E5331 | |
| Illumina HiSeq2500 Sequencer | Illumina, Inc. | HiSeq2500 | |
| M165FC fluorescent stereomicroscope | Leica Microsystems | M165FC | |
| Nanodrop OneC UV-vis spectrophotometer | ThermoFisher | NDONEC-W | |
| NEBNext Ultra II RNA Library Prep Kit | New England Biolabs, Inc. | E7770 | |
| plasmid # 112686 | Addgene | 112686 | |
| plasmid # 112688 | Addgene | 112688 | |
| plasmid # 132416 | Addgene | 132416 | |
| plasmid # 132417 | Addgene | 132417 | |
| plasmid # 132419 | Addgene | 132419 | |
| plasmid # 132420 | Addgene | 132420 | |
| plasmid # 132421 | Addgene | 132421 | |
| plasmid # 132422 | Addgene | 132422 | |
| plasmid # 132425 | Addgene | 132425 | |
| plasmid # 132426 | Addgene | 132426 | |
| plasmid # 133304 | Addgene | 133304 | |
| plasmid # 164586 | Addgene | 164586 | |
| plasmid #132418 | Addgene | 132418 | |
| plasmid pJFRC81 | Addgene | 36432 | |
| Qiagen RNeasy Mini Kit | Qiagen | 74104 | |
| Restriction endonucleases AscI | New England Biolabs Inc. | R0558L | |
| Restriction endonucleases NotI | New England Biolabs Inc. | R0189L | |
| Restriction endonucleases PacI | New England Biolabs Inc. | R0547L | |
| Restriction endonucleases PstI | New England Biolabs Inc. | R0140L | |
| Restriction endonucleases SwaI | New England Biolabs Inc. | R0604L | |
| Restriction endonucleases XbaI | New England Biolabs Inc. | R0145L | |
| RNA 6000 Pico Kit for Bioanalyzer | Agilent Technologies | 5067-1513 | |
| Turbo DNase | Invitrogen | AM2238 | |
| U6-3:4-gRNA-Fluc flies | Bloomington Drosophila Stock Center | 84125 | |
| U6-3:4-gRNA-GFP; OpIE2-GFP flies | Bloomington Drosophila Stock Center | 84986 | |
| U6-3:4-gRNA-N flies | Bloomington Drosophila Stock Center | 84122 | |
| U6-3:4-gRNA-w flies | Bloomington Drosophila Stock Center | 84124 | |
| U6-3:4-gRNA-y flies | Bloomington Drosophila Stock Center | 84123 | |
| UASt-CasRx flies | Bloomington Drosophila Stock Center | 84121 | |
| UASt-dCasRx flies | Bloomington Drosophila Stock Center | 84120 | |
| Ubiq-CasRx flies | Bloomington Drosophila Stock Center | 84118 | |
| Ubiq-dCasRx flies | Bloomington Drosophila Stock Center | 84119 | |
| Ubiq-Firefly-T2A-eGFP-Ubiq-Renilla flies | Bloomington Drosophila Stock Center | 84127 | |
| Zymoclean Gel DNA Recovery Kits | Zymo Research Corporation | D4007 |
References
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nat Communications. 9, 1911 (2018).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), 5573 (2016).
- East-Seletsky, A., O'Connell, M., Burstein, D., Knott, G., Doudna, J. RNA Targeting by Functionally Orthogonal Type VI-A CRISPR-Cas Enzymes. Molecular Cell. 66 (3), 373-383 (2017).
- Konermann, S., Lotfy, P., Brideau, N., Oki, J., Shokhirev, M., Hsu, P. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell. 173 (3), 665-676 (2018).
- Buchman, A., Brogan, D., Sun, R., Yang, T., Hsu, P., Akbari, O. Programmable RNA Targeting Using CasRx in Flies. The CRISPR Journal. 3 (3), 164-176 (2020).
- Kushawah, G., et al. CRISPR-Cas13d Induces Efficient mRNA Knockdown in Animal Embryos. Developmental Cell. 54 (6), 805-817 (2020).
- Abudayyeh, O., et al. RNA targeting with CRISPR-Cas13. Nature. 550, 280-284 (2017).
- Perrimon, N., Ni, J., Perkins, L. In vivo RNAi: today and tomorrow. Cold Spring Harbor Perspectives in Biology. 2, 003640 (2010).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
- Ni, J., et al. A Drosophila resource of transgenic RNAi lines for neurogenetics. Genetics. 182 (4), 1089-1100 (2009).
- Ni, J., et al. A genome-scale shRNA resource for transgenic RNAi in Drosophila. Nature Methods. 8, 405-407 (2011).
- Ni, J., et al. Vector and parameters for targeted transgenic RNA interference in Drosophila melanogaster. Nature Methods. 5, 49-51 (2008).
- Heigwer, F., Port, F., Boutros, M. RNA Interference (RNAi) Screening in Drosophila. Genetics. 208 (3), 853-874 (2018).
- Kulkarni, M., et al. Evidence of off-target effects associated with long dsRNAs in Drosophila melanogaster cell-based assays. Nature Methods. 3, 833-838 (2006).
- Ma, Y., Creanga, A., Lum, L., Beachy, P. Prevalence of off-target effects in Drosophila RNA interference screens. Nature. 443, 359-363 (2006).
- Perrimon, N., Mathey-Prevot, B. Matter arising: off-targets and genome-scale RNAi screens in Drosophila. Fly. 1 (1), 1-5 (2007).
- Markstein, M., Pitsouli, C., Villalta, C., Celniker, S., Perrimon, N. Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nature Genetics. 40, 476-483 (2008).
- Champer, J., Buchman, A., Akbari, O. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nature Review Genetics. 17 (3), 146-159 (2016).
- Buchman, A., et al. Broad dengue neutralization in mosquitoes expressing an engineered antibody. PLoS Pathogens. 16 (4), 1008103 (2020).
- Mathur, G., Sanchez-Vargas, I., Alvarez, D., Olson, K., Marinotti, O., James, A. Transgene-mediated suppression of dengue viruses in the salivary glands of the yellow fever mosquito, Aedes aegypti. Insect Molecular Biology. 19 (6), 753-763 (2011).
- Franz, A., et al. Engineering RNA interference-based resistance to dengue virus type 2 in genetically modified Aedes aegypti. Proceedings of the National Academy of Sciences of the United States of America. 103 (11), 4198-4203 (2006).
- Yen, P., James, A., Li, J., Chen, C., Failloux, A. Synthetic miRNAs induce dual arboviral-resistance phenotypes in the vector mosquito Aedes aegypti. Communications Biology. 1, 11 (2018).
- Buchman, A., et al. Engineered resistance to Zika virus in transgenic Aedes aegypti expressing a polycistronic cluster of synthetic small RNAs. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3656-3661 (2019).
- . Addgene Available from: https://www.addgene.org/protocols/restriction-digest/ (2020)
- Gibson, D., Young, L., Chuang, R., Venter, J., Hutchison, C., Smith, H. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Addgene Available from: https://www.addgene.org/protocols/pcr/ (2020)
- . Indiana University Bloomington Available from: https://bdsc.indiana.edu/information/recipes/bloomfood.htmL (2020)
- Dobin, A., et al. STAR:ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Jiang, W., Brueggeman, A., Horken, K., Plucinak, T., Weeks, D. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii. Eukaryotic Cell. 13 (11), 1465-1469 (2014).
- Poe, A., et al. Robust CRISPR/Cas9-Mediated Tissue-Specific mutagenesis reveals gene redundancy and perdurance in Drosophila. Genetics. 211 (2), 459-472 (2019).
- Port, F., Chen, H., Lee, T., Bullock, S. Optimized CRISPR/Cas tools for efficient ger mLine and somatic genome engineering in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 111 (29), 2967-2976 (2014).
- Yang, S., Li, S., Li, X. Shortening the Half-Life of Cas9 maintains its gene editing ability and reduces neuronal toxicity. Cell Reports. 25 (10), 2653-2659 (2018).
- Port, F., et al. A large-scale resource for tissue-specific CRISPR mutagenesis in Drosophila. eLife. 9, 53865 (2020).
- Zhang, X., Tee, L., Wang, X., Huang, Q., Yang, S. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4 (17), 264 (2015).
- Huynh, N., Depner, N., Larson, R., King-Jones, K. A versatile toolkit for CRISPR-Cas13-based RNA manipulation in Drosophila. Genome Biology. 21, 279 (2020).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved