Method Article
Ubiquitäres und gewebespezifisches RNA-Targeting in Drosophila Melanogaster mittels CRISPR/CasRx
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt ein detailliertes Protokoll für die Verwendung des RNA-Targeting-Enzyms Cas13D (RfxCas13D) in Fliegen.
Zusammenfassung
CasRx, ein Mitglied der RNA-Targeting-Cas13-Familie, ist eine vielversprechende neue Ergänzung der CRISPR/Cas-Technologien in der effizienten Reduktion von Gentranskripten mit einem attraktiven Off-Target-Profil sowohl auf zellulärer als auch auf organismischer Ebene. Kürzlich wurde berichtet, dass das CRISPR/CasRx-System verwendet werden kann, um eine ubiquitäre und gewebespezifische Reduktion von Gentranskripten in Drosophila melanogaster zu erreichen. Dieses Papier beschreibt die Methoden aus der jüngsten Arbeit, bestehend aus drei Teilen: 1) ubiquitäres in vivo endogenes RNA-Targeting mit einem Zwei-Komponenten-CasRx-System; 2) ubiquitäre in vivo exogene RNA-Targeting mit einem Drei-Komponenten-CasRx-System; und 3) gewebespezifisches in vivo RNA-Targeting mit einem Drei-Komponenten-CasRx-System. Zu den beobachteten Wirkungen des RNA-Targetings gehören gezielte genspezifische phänotypische Veränderungen, eine gezielte RNA-Transkriptreduktion und gelegentliche Letalitätsphänotypen, die mit einer hohen Expression des CasRx-Proteins und der Kollateralaktivität assoziiert sind. Insgesamt zeigten diese Ergebnisse, dass das CasRx-System in der Lage ist, die RNA-Transkriptreduktion auf organismischer Ebene programmierbar und effizient zu reduzieren, was zeigt, dass in vivo Transkriptom-Targeting und Engineering machbar ist und die Grundlage für zukünftige in vivo CRISPR-basierte RNA-Targeting-Technologien legt.
Einleitung
Seit dem Aufkommen der CRISPR-Technologien (Clustered Regularly Interspaced Short Palindromic Repeats) liegt ein Großteil des Fokus in diesem Bereich auf der DNA-Editierung, die transformative Anwendungen in der Medizin und Biotechnologie bietet1. Eine dauerhafte Veränderung von DNA-Sequenzen ist jedoch aus ethischen Gründen nicht immer erwünscht. Vor diesem Hintergrund begannen jüngste Studien mit der Entwicklung von CRISPR-basierten Werkzeugen für das Targeting von RNA und zeigten, dass CRISPR-Technologien tatsächlich für das RNA-Targeting in einer Vielzahl von biologischen Systemen verwendet werden können2,3,4,5,6,7. In vielen dieser getesteten Systeme ist der derzeit weit verbreitete Ansatz für die Targeting-RNA und Transkriptreduktion die RNA-Interferenz (RNAi), die bei weitem nicht perfekt ist und oft eine unterschiedliche Wirksamkeit und eine hohe Off-Target-Aktivität aufweist, wenn sie in vivo verwendet wird8,9,10,11,12,13,14,15,16,17 . Angesichts des Status dieser Technologien lohnt es sich daher, die Potenziale von CRISPR-basierten Tools für das RNA-Targeting weiter zu erforschen.
Eine bemerkenswerte aktuelle Studie berichtete, dass die Ribonuklease CasRx, ein Mitglied der Cas13d-Klasse, die Gentranskriptspiegel in der menschlichen Zellkultur effizient reduzieren kann und ein attraktives Off-Target-Profil besitzt4. Dieser Befund führte zu der Frage, ob diese neue Ribonuklease ihre Wirksamkeit und niedrige Off-Target-Rate für das RNA-Targeting auf organismischer Ebene aufrechterhalten kann. Eine kürzlich durchgeführte Studie befasste sich mit dieser Frage, indem sie zeigte, dass das CasRx-System verwendet werden kann, um eine ubiquitäre und gewebespezifische Reduktion von Gentranskripten in Drosophila melanogaster5 zu erreichen.
Um die Benutzerfreundlichkeit dieses kürzlich veröffentlichten Ansatzes zu rationalisieren, beschreibt dieses Protokoll die Methoden aus dieser jüngsten Arbeit, die aus drei Hauptteilen besteht: 1) allgegenwärtiges In-vivo-RNA-Targeting mit einem Zwei-Komponenten-CasRx-System; 2) ubiquitäre in vivo exogene RNA-Targeting mit einem Drei-Komponenten-CasRx-System; und 3) gewebespezifisches In-vivo-RNA-Targeting mit einem Drei-Komponenten-CasRx-System.
Guide RNAs (gRNA), die auf verschiedene Zielgene unter der Kontrolle eines ubiquitären Promotors abzielen, wurden entworfen und Fliegenlinien erzeugt, die diese gRNA-haltigen Konstrukte exprimieren. CasRx-Konstrukte, die entweder unter der Kontrolle eines ubiquitären Promotors oder eines UFSt-Promotors (Conditional Upstream Activation Sequence) stehen, der durch den GAL4-Transkriptionsfaktor aktivierbar ist, wurden ebenfalls entworfen und Flylines erzeugt, die diese CasRx-haltigen Konstrukte beherbergen. Katalytisch inaktive CasRx-Konstrukte, dCasRx, wurden als Negativkontrollen konzipiert und verwendet. Das ubiquitäre RNA-Targeting in Fliegen wird durch Kreuzung von gRNA-exprimierenden Fliegenlinien mit ubiquitär CasRx-exprimierenden Fliegenlinien erreicht. Die Nachkommen, die sowohl das gRNA-Konstrukt exprimieren, das auf ein bestimmtes Gentranskript abzielt, als auch das CasRx-Protein haben eine allgegenwärtige Reduktion der zielgerichteten Gentranskripte. Ein gewebespezifisches RNA-Targeting in Fliegen wird erreicht, indem zunächst gRNA-exprimierende Fliegen mit UASt-CasRx-exprimierenden Fliegen kreuzen und transheterozygote Fliegen erhalten werden, die sowohl gRNA- als auch UASt-CasRx-Konstrukte tragen. Solche Fliegen wiederum werden mit gewebespezifischen GAL4-exprimierenden Fliegen gekreuzt, was zur Erzeugung einer gewebespezifischen CasRx-Expression und eines RNA-Targetings bei Fliegen führt.
Die programmierbare Natur des CasRx-Systems bietet die Möglichkeit der Anpassung und Optimierung, um eine hohe Wirksamkeit und eine geringe Off-Target-Aktivität für das In-vivo-RNA-Targeting zu erreichen. Mögliche Anwendungen von CRISPR-basiertem RNA-Targeting sind zahlreich, einschließlich des Ersatzes von RNAi im Labor und des Beitrags zur Insektenvektorkontrolle in freier Wildbahn. Von letzterem ist einer der globalen unerfüllten Bedürfnisse die Entwicklung effizienter Werkzeuge zur Bekämpfung von Infektionen von RNA-Viren, die über Moskitos übertragen werden. Viele RNA-Viren wie Dengue-, Zika- und Chikungunya-Virus werden über Moskitos übertragen, was die menschliche Gesundheit beeinträchtigt und zur Mortalität beiträgt. Es wurden viele Vorschläge für die Entwicklung von Mückenpopulationen mit Virusresistenz zur Krankheitsprävention gemacht; Keine aktuelle Technologie ist jedoch in der Lage, Moskitos gleichzeitig gegen alle signifikanten RNA-Viren resistent zu machen18,19,20,21,22,23. RNA-targeting-Cas-Systeme können einen Ausgangspunkt für eine solche Technologie bieten, indem sie eine programmierbare Plattform für das Targeting aller durch Mücken übertragenen RNA-Viren ermöglichen.
Protokoll
1. Ubiquitäres In-vivo-RNA-Targeting mit einem Zwei-Komponenten-CasRx-System
- Generieren von Ubiq-CasRx- und Ubiq-dCasRx-Ausdrucksvektoren
- Amplifizieren Sie die CasRx-Sequenz mit einer Polymerase-Kettenreaktion (PCR) mit Primer 1050E. C3 und 1050E. C4 und das ursprüngliche CasRx-Konstrukt pNLS-RfxCas13d-NLS-HA (pCasRx); und amplifizieren Sie die dCasRx-Sequenz mittels PCR mit Primer 1050E. C3 und 1050E. C4 und das ursprüngliche dCasRx-Konstrukt pNLS-dRfxCas13d-NLS-HA (pdCasRx)4 (Tabelle 1). Anschließende Gelreinigung der amplifizierten CasRx- und dCasRx-Fragmente mit einem Gelreinigungskit.
- Digest-Basenvektor (Addgen-Plasmid #112686) mit Restriktionsenzymen SwaI und PacI24. Verwenden Sie in den resultierenden Produkten ein Kit, um das größere Fragment, das als Basisvektor-Rückgrat bezeichnet wird, zu gelreinigen.
- Assemblieren Sie den Ubiq-CasRx-Vektor mit dem Basisvektor-Backbone und dem CasRx-Fragment mit der Gibson-Assembly-Methode. Assemblieren Sie den Ubiq-dCasRx-Vektor mit dem Basisvektor-Backbone und das dCasRx-Fragment mit der Gibson-Assembly-Methode25.
HINWEIS: Die Addgene-ID des Ubiq-CasRx-Vektors (OA-1050E) ist #132416, und die Addgene-ID des Ubiq-dCasRx-Vektors (OA-1050R) ist #132417.
- Generieren eines gRNA-Expressionsvektors
- Entwerfen Sie jedes gRNA-Fragment basierend auf den folgenden Kriterien: Zielsequenz mit einer Länge von 30 Nukleotiden; die maximale Länge von Poly-U-Dehnungen in der Zielsequenz beträgt 4 Basenpaare; der GC-Gehalt der Zielsequenz liegt im Bereich von 30% - 70%; die Zielsequenz prognostizierte, keine starken RNA-Haarnadelstrukturen zu bilden; und die Zielsequenz mit minimal vorhergesagter sekundärer oder tertiärer RNA-Struktur5.
HINWEIS: Diese Studie entwarf jede gRNA als 4 Tandemsequenzen mit jeweils 30 Nukleotiden längen, im Abstand von 36 nukleotidlangen direkten Wiederholungen und mit einem 7-Thymin-Terminator an beiden Enden5. Für das exogene Zielgen GFP wurden die gleichen Kriterien wie oben mit einer Zugabe eines OpIE2-GFP-Fragments5 befolgt. - Amplifizieren Sie die U6:3-Promotorsequenz mittels PCR mit den Primern 1043.C1 und 1043.C23 und dem Addgen-Plasmid #112688 (Tabelle 1)26. Gel-Reinigung der amplifizierten U6: 3-Fragmente mit Gelreinigungskit.
- Digest Addgene Plasmid #112688 mit Restriktionsenzym AscI und XbaI24. Verwenden Sie in den resultierenden Produkten ein Kit, um die größeren Fragmente zu gelreinigen, was als Präbasis-Vektor-Backbone bezeichnet wird.
- Setzen Sie den Basisvektor mit dem Pre-Base-Vektor-Backbone und dem U6:3-Fragment mit der Gibson-Assembly-Methode zusammen25. Der nachfolgende Basisvektor heißt OA-1043.
HINWEIS: Die Addgene-ID des Plasmids OA-1043 ist #164586. - Synthetisieren Sie das gRNA-Fragment des Zielgens mit einem externen Gensynthesedienst.
- Verdauen Sie den Basenvektor OA-1043 mit Restriktionsenzym PstI und NotI24. Bewahren Sie das gesamte Verdauungsprodukt auf, das als verdautes OA-1043 bezeichnet wird.
- Assemblieren Sie den gRNA-Expressionsvektor mit dem verdauten OA-1043 und dem Zielgen-gRNA-Fragment unter Verwendung der Gibson-Assemblierungsmethode25.
ANMERKUNG: Vier Zielgene wurden untersucht: drei waren endogen (weiß, Notch, gelb), eines war exogen (GFP). Ihre Addgene-IDs sind: #132420 (gRNAw), #132421(gRNAN), #132425 (gRNAy) und #133304 (gRNAGFP).
- Entwerfen Sie jedes gRNA-Fragment basierend auf den folgenden Kriterien: Zielsequenz mit einer Länge von 30 Nukleotiden; die maximale Länge von Poly-U-Dehnungen in der Zielsequenz beträgt 4 Basenpaare; der GC-Gehalt der Zielsequenz liegt im Bereich von 30% - 70%; die Zielsequenz prognostizierte, keine starken RNA-Haarnadelstrukturen zu bilden; und die Zielsequenz mit minimal vorhergesagter sekundärer oder tertiärer RNA-Struktur5.
- Erzeugung transgener Fliegen
- Injektion von Expressionsvektoren in Fliegenembryonen unter Verwendung eines externen Fliegenembryo-Injektionsdienstes und Embryonen von Fliegen, die ØC31-Integrationsstellen enthalten. Aufziehen der injizierten Embryonen bei 26 °C.
HINWEIS: Die attp40w-Linie (mit Integrationsstellen auf dem 2. Chromosom) wurde verwendet, um CasRx-Linien zu erzeugen, und die 8622-Linie (mit Integrationsstellen auf dem 3. Chromosom) wurde verwendet, um verschiedene gRNA-Linien zu erzeugen. - Halten Sie die Fliegen entweder als homozygote Linien oder als ausgeglichene heterozygote Linien.
HINWEIS: Ubiq-CasRx- und Ubiq-dCasRx-Fliegen wurden als heterozygote balancierte Linien mit dem CyO als Balancer gehalten. Darüber hinaus enthalten sowohl Ubiq-CasRx- als auch Ubiq-dCasRx-Vektoren einen dsRed-Marker. Infolgedessen haben die Ubiq-CasRx- und Ubiq-dCasRx-Fliegen die folgenden drei Phänotypen: dsRed-positive, lockige Flügel und weiße Augen. Die gRNA-exprimierenden Fliegen wurden als homozygote Linien gehalten. Ihre Bloomington Drosophila Stock Center (BDSC) Fliegenbestandszahlen sind: #84118 (Ubiq-CasRx), #84119 (Ubiq-dCasRx), #84124 (gRNAw), #84122 (gRNAN), #84123 (gRNAy), #84986 (gRNAGFP).
- Injektion von Expressionsvektoren in Fliegenembryonen unter Verwendung eines externen Fliegenembryo-Injektionsdienstes und Embryonen von Fliegen, die ØC31-Integrationsstellen enthalten. Aufziehen der injizierten Embryonen bei 26 °C.
- Fliegengenetik (Abbildung 1A)
- Sammeln Sie 10 jungfräuliche erwachsene weibliche Fliegen aus der homozygoten gRNA-Linie und sammeln Sie 5 erwachsene männliche Fliegen aus der ausgewogenen heterozygoten Ubiq-CasRx / CyO-Linie. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die als Elternfliegen bezeichnet werden, in eine Durchstechflasche, die mit Trockenhefepulver ergänzt wird (Abbildung 1A).
- Wiederholen Sie den vorherigen Schritt 3 Mal, um 3 Replikate zu generieren. Verwenden Sie für die Kontrollgruppe die Ubiq-dCasRx/CyO-Linie, während alles andere gleich bleibt.
HINWEIS: Für eine normale Glasfläschchen mit Lebensmitteln sind 0,1 g Trockenhefepulver ausreichend. Das Fliegenfutterrezept des BDSC wird verwendet. - Legen Sie die Fläschchen mit den Elternfliegen 48 Stunden lang bei 26 °C auf. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Bewahren Sie die Durchstechflaschen dann mindestens 20 Tage lang bei 26 °C auf.
- Beobachten Sie die Fläschchen jeden Tag, um zu sehen, ob neue erwachsene Nachkommen aus Puppen der F1-Generation hervorgegangen sind. Wenn ja, betäuben Sie sie mit Kohlendioxid, indem Sie einen Schlauch einführen, der mit einem Kohlendioxidtank in den Fliegenfläschchen verbunden ist, und dann den Durchflussschalter für 10 Sekunden einschalten.
- Sobald die Fliegen unbeweglich werden, entleeren Sie sie aus der Durchstechflasche auf ein Fliegenpolster, das auch mit dem Kohlendioxidtank verbunden ist und Kohlendioxid kontinuierlich durch das Fliegenpolster fließt.
- Bewerten Sie den Phänotyp der anästhesierten Fliegen und bilden Sie sie mit einer Farbkamera ab, die mit einem fluoreszierenden Stereomikroskop verbunden ist. Zählen Sie die Anzahl der Nachkommen mit verschiedenen Phänotypen. Verwenden Sie Bildverarbeitungssoftware für die Nachbearbeitung und Kompilierung von Bildern (Abbildung 2A - 2D).
HINWEIS: Basierend auf der Mendelschen Genetik werden zwei Arten von Fliegen unter den Nachkommen für jede Kreuzung erwartet (Abbildung 1A).
- RNA-Seq (Abbildung 2E – 2G)
- Musterkollektion
HINWEIS: Wählen Sie eine geeignete Probenentnahmemethode aus den folgenden 3 Beispielen aus. Für jeden probentyp sind 3 Replikate erforderlich.- Adulte Fliegenkopfprobenentnahme
- Sammeln Sie 10 jungfräuliche erwachsene weibliche Fliegen aus der homozygoten gRNA-Linie. Sammle 5 erwachsene männliche Fliegen aus der ausgewogenen heterozygoten Ubiq-CasRx/CyO-Linie. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die die elterlichen Fliegen sind, in eine Durchstechflasche, die mit Trockenhefepulver ergänzt wird.
- Wiederholen Sie den vorherigen Schritt 3 Mal für 3 Replikate. Verwenden Sie für die Kontrollgruppe die Ubiq-dCasRx/CyO-Linie, während alles andere gleich bleibt.
- Legen Sie die Fläschchen mit den Elternfliegen 48 Stunden lang bei 26 °C auf. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Halten Sie dann die Fläschchen bei 26 ° C, bis Nachkommen aus Puppen austreten.
- Sammle 10 1 Tag alte erwachsene Fliegen mit dem richtigen Phänotyp. Betäuben Sie die Fliegen mit Kohlendioxid, schneiden Sie dann den Fliegenkopf ab und legen Sie die Köpfe in ein 1,5 ml Zentrifugenrohr auf Trockeneis. Lagern Sie das Zentrifugenröhrchen bei -80 °C. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- 17 – 20 Stunden alte Embryonenprobenentnahme
- Sammeln Sie 8-10 jungfräuliche erwachsene weibliche Fliegen aus der homozygoten gRNA-Linie. Sammle 4-5 erwachsene männliche Fliegen aus der ausgewogenen heterozygoten Ubiq-CasRx/CyO-Linie. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die die elterlichen Fliegen sind, in eine Durchstechflasche, die mit Trockenhefepulver ergänzt wird.
- Wiederholen Sie den vorherigen Schritt 3 Mal für 3 Replikate. Verwenden Sie für die Kontrollgruppe die Ubiq-dCasRx/CyO-Linie, während alles andere gleich bleibt.
- Legen Sie die Fläschchen mit den Elternfliegen 48 Stunden lang bei 26 °C auf.
- Bereiten Sie eine Traubensaft-Embryo-Sammelkammer für jedes Replikat nach diesem Rezept vor: 376 ml Wasser, 126 ml Traubensaft, 15 g Agar und 6 g Saccharose. Legen Sie das Medium in ein 1-Liter-Becherglas und erhitzen Sie es für 5-6 Minuten in der Mikrowelle, während Sie die Medien im Becherglas genau im Auge behalten, um zu überprüfen, ob Blasen / Schaum auftreten. Wenn ja, stoppen Sie die Mikrowelle und lassen Sie die Blase / den Schaum absetzen. Fahren Sie auf diese Weise mit der Mikrowelle fort, bis die Blase klar wird. Nicht schwenken, bis alle Blasen klar sind. Schließlich fügen Sie 10 ml 100% Alkohol und 5 ml Essigsäure hinzu. Gut mischen, dann das Medium in 35 mm Petrischalen mit einem 25 ml serologischen Pipett pipettieren. Wenn sich das Medium in der Petrischale erstarrt, ist es gebrauchsfertig.
- Am Ende der 48-stündigen Inkubation werden die elterlichen Fliegen in die Traubensaft-Embryo-Sammelkammern übertragen und bei 26 °C für 3 h inkubiert. Dann entfernen Sie die erwachsenen Fliegen, während Sie die frisch gelegten Embryonen für weitere 17 h bei 26 °C auf den Traubensaftplatten aufbewahren.
- Sammeln Sie nach der Inkubation die 50 – 100 Embryonen von den Traubensaftplatten, reinigen Sie die Embryooberfläche, indem Sie sie in entionisiertes Wasser tauchen, und übertragen Sie sie dann in ein 1,5 ml Zentrifugenröhrchen auf Eis. Lagern Sie sie bei -80 °C. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Erste Instar-Larvenprobenentnahme
- Sammeln Sie 8-10 jungfräuliche erwachsene weibliche Fliegen aus der homozygoten gRNA-Linie. Sammle 4-5 erwachsene männliche Fliegen aus der ausgewogenen heterozygoten Ubiq-CasRx/CyO-Linie. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die die elterlichen Fliegen sind, in eine Durchstechflasche, die mit Trockenhefepulver ergänzt wird. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate. Verwenden Sie für die Kontrollgruppe die Ubiq-dCasRx/CyO-Linie, während alles andere gleich bleibt.
- Legen Sie die Fläschchen mit den Elternfliegen 48 Stunden lang bei 26 °C auf. Anschließend werden die erwachsenen Fliegen zur Inkubation über Nacht (16 h) bei 26 °C in ein anderes neues normales Lebensmittelfläschchen überführt. Entfernen Sie dann die erwachsenen Fliegen.
- Bewahren Sie die embryonenhaltige Durchstechflasche 24 h lang bei 26 °C auf und bewerten Sie dann die transheterozygoten Larven des ersten Stadiums unter dem Mikroskop mit unterschiedlichen Markern. 15-30 Larven mit korrekten Phänotypen sammeln und in ein 1,5 ml Zentrifugenröhrchen geben und bei -80 °C lagern. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Adulte Fliegenkopfprobenentnahme
- Sequenzierung
- RNA-Extraktion: Verwenden Sie ein handelsübliches RNA-Extraktionskit und befolgen Sie die Anweisungen des Kits, um alle Proben zu verarbeiten. Inkubieren Sie dann die extrahierten RNA-Proben mit handelsüblicher Desoxyribonuklease und befolgen Sie deren Anweisung, kontaminierte DNA aus den Proben zu entfernen.
- Messen Sie die RNA-Konzentration mit einem handelsüblichen UV-Vis-Spektralphotometer. Messen Sie die RNA-Integrität in den Proben mit handelsüblichen RNA-Integritätsassays.
- Konstruieren Sie die RNA-seq-Bibliotheken unter Verwendung eines kommerziell erhältlichen RNA-Bibliotheksvorbereitungskits.
- Verwenden Sie den externen Sequenzierungsdienst für die Bibliothekssequenzierung mit den folgenden Einstellungen: Einzellesemodus; Lesedauer: 50nt, Tiefe: 20 Millionen Lesevorgänge pro Bibliothek. Führen Sie Basisaufrufe mit RTA 1.18.64 durch und konvertieren Sie die Daten dann mit bcl2fastq 1.8.4 in FASTQ.
HINWEIS: Rohsequenzierungsdaten finden Sie im National Center for Biotechnology Information Sequencing Read Archive (Einreichungs-ID: SUB6818910 [BioProject: PRJNA600654]).
- Bioinformatik
- Karte liest aus den Sequenzierungsdaten zu Release 6 Drosophila melanogaster Genom aus dem Berkeley Drosophila Genome Project (GenBank Accession Number: GCA_000001215.4) und den exogenen CasRx - und GFP-Sequenzen unter Verwendung der Standardparametereinstellung von STAR aligner28 mit der Filteroption "-outFilterType BySJout" und "-sjdbGTFfile Drosophila_melanogaster. BDGP6.22.97.gtf" Gentransfer-Formatdatei von ENSEMBL.
- Bestimmen Sie die Rohtranskriptanzahl für jedes kommentierte Transkript mit der Funktion Counts35 mit den Optionen "-t exon -g gene_id -M -O --fraction". Normalisieren Sie dann die rohen Transkriptzählungen mit der Gesamtzahl der Transkriptzählungen mit dem Perl-Skript "addTpmFpkmToFeatureCounts.pl".
- Verwenden Sie die Maximum-Posteriori-Methode mit dem ursprünglichen Schrumpfungsschätzer in der DESeq2-Pipeline, um die logarithmische Faltenänderung (LFC) der Transkripte jedes Gens zu schätzen.
- Musterkollektion
2. Allgegenwärtiges in vivo exogenes RNA-Targeting mit einem Drei-Komponenten-CasRx-System
- Generieren eines exogenen ubiquitären Zielexpressionsvektors
- PCR amplifizieren das Ubiq-Promotorfragment mit Primern 1052B. C1 und 1052B. C2 und das Addgen-Plasmid #112686 26. Anschließend verstärkt die PCR das T2A-eGFP-Fragment, das aus dem Addgen-Plasmid #112686 mit den Primern 908A.1 und 908A.2 amplifiziert wurde (Tabelle 1)26. Anschließend verstärkt die PCR das Ubiq-Promotorfragment als umgekehrte Sequenz unter Verwendung des Addgen-Plasmids #112686 mit den Primern 908A.3 und 908A.4 (Tabelle 1)26. Gel-Reinigung des Ubiq-Promotor-Fragments, des T2A-eGFP-Fragments und des umgekehrten Ubiq-Promotor-Fragments mit Gel-Reinigungs-Kit.
- Bestellen Sie eine benutzerdefinierte Glühwürmchen-Luciferase-codierende Sequenz und ein benutzerdefiniertes Fragment, das ein p10 3'UTR-Fragment enthält, umgekehrte Renilla-Luciferase, gefolgt von einem SV40 3'UTR-Fragment.
- Digest Addgene Plasmid #112688 mit Restriktionsenzym AscI und XbaI24. In den resultierenden Produkten reinigen Sie die größeren Fragmente mit einem Gelreinigungskit, das als Basisvektor-Rückgrat bezeichnet wird.
- Verwenden Sie die Gibson-Assemblierungsmethode, um den Basisvektor mit dem Basisvektor-Backbone und den folgenden Fragmenten zusammenzusetzen: Ubiq-Promotorfragment, das T2A-eGFP-Fragment, das umgekehrte Ubiq-Promotorfragment, die Glühwürmchen-Luciferase-kodierende Sequenz und die umgekehrte Renilla-Luciferase, gefolgt von einem SV40 3'UTR-Fragment25.
HINWEIS: Die Addgene-ID des resultierenden Dual-Luciferase-Expressionsvektors (OA-1052B) ist #132426.
- Generieren eines gRNA-Expressionsvektors
- Die PCR verstärkt die U6:3-Promotorsequenz mit den Primern 1043.C1 und 1043.C23 sowie dem Addgenplasmid #112688 (Tabelle 1)26. Gel-Reinigung der amplifizierten U6: 3-Fragmente mit Gelreinigungskit.
- Digest Addgene Plasmid #112688 mit Restriktionsenzym AscI und XbaI24. In den resultierenden Produkten reinigen Sie die größeren Fragmente, die als Präbasisvektor-Rückgrat bezeichnet werden, mit einem Gelreinigungskit.
- Montieren Sie den Basisvektor mit dem Pre-Base-Vektor-Backbone und dem U6:3-Fragment mit der Gibson-Assembly-Methode25. Der nachfolgende Basisvektor heißt OA-1043.
- Synthetisieren Sie das gRNA-Fragment des Zielgens mit einem externen Gensynthesedienst.
- Verdauen Sie den Basenvektor OA-1043 mit Restriktionsenzym PstI und NotI24. Bewahren Sie das gesamte Verdauungsprodukt auf, das als verdautes OA-1043 bezeichnet wird.
- Assemblieren Sie den gRNA-Expressionsvektor mit dem verdauten OA-1043 und dem Zielgen-gRNA-Fragment unter Verwendung der Gibson-Assemblierungsmethode25.
HINWEIS: Die Addgen-ID des resultierenden Plasmids (OA-1052K) ist #132422.
- Erzeugung transgener Fliegen
- Injektion des OA-1052B-Vektors in Fliegenembryonen unter Verwendung von Embryonen von Fliegen, die ØC31-Integrationsstelle auf dem 3. Chromosom, BDSC-Fliegenbestand Nummer 9744, über einen externen Fliegenembryo-Injektionsservice enthalten. In ähnlicher Weise injiziert OA-1052K-Vektor in Fliegenembryonen unter Verwendung von Embryonen von Fliegen, die ØC31-Integrationsstelle auf dem 3. Chromosom, BDSC-Fliegenbestand Nummer 8622, enthalten. Aufziehen der injizierten Embryonen bei 26 °C.
- Halten Sie die Dual-Luciferase-exprimierenden Fliegen und die gRNA-Fliegen als homozygote Linien; Halten Sie die Ubiq-CasRx-Linien als doppelt ausbalancierte heterozygote Linien, indem Sie die in Abschnitt 1 erzeugte einfach-ausgeglichene heterozygote Ubiq-CasRx-Linie zu Balancer-Linien auskreuzen, die TM6-Balancer-Chromosom mit Stoppelmarker (Stb) tragen, und behalten Sie nur die doppelt ausbalancierten Nachkommen mit weißäugigen, lockigen Flügeln und dsRed-fluoreszierenden Phänotypen gleichzeitig bei.
HINWEIS: Die BDSC-Fliegenbestandsnummern lauten: #84127 (Ubiq-Fluc-Rluc), #84125 (gRNAFluc).
- Fliegengenetik (Abbildung 1B und Abbildung 3A)
- Sammle 8-10 jungfräuliche erwachsene weibliche Fliegen aus der Dual-Luciferase-exprimierenden Linie. Sammeln Sie 4-5 erwachsene männliche Fliegen aus dem ausgewogenen heterozygoten Ubiq-CasRx / CyO; +/TM6, Stb-Linie, die gleichzeitig weißäugige, lockige Flügel und dsRed-Fluoreszenz zeigen. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die die Elterlichen Fliegen sind, in eine Durchstechflasche, die mit Trockenhefepulver (im Folgenden Schritt 1 Kreuz genannt) ergänzt wird.
- Wiederholen Sie den vorherigen Schritt 3 Mal für 3 Replikate. Verwenden Sie für die Kontrollgruppe den Ubiq-dCasRx/CyO; +/TM6, Stb-Linie, während alles andere gleich bleibt.
- Ziehen Sie die Step 1 Cross-Fläschchen mit den Elterlichen Fliegen bei 26 °C für 48 Stunden zurück. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Bewahren Sie die Durchstechflaschen dann mindestens 14 Tage lang bei 26 °C auf. Sammeln Sie während dieser Zeit 8-10 weibliche Jungfrauen aus der homozygoten Glühwürmchen-Luziferase-Targeting-gRNA-Linie. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Beobachten Sie jeden Tag die Step 1 Cross Fläschchen, um zu sehen, ob eine neue erwachsene Fliege aus Puppen auftaucht. Wenn ja, betäuben Sie sie mit Kohlendioxid, sammeln Sie 5 männliche Fliegen, die sowohl die Ubiq-CasRx (oder Ubiq-dCasRx) als auch den Dual-Luciferase-Reporter aus den Nachkommen exprimieren, und legen Sie sie zusammen mit 10 jungfräulichen Weibchen aus der Glühwürmchen-Luciferase-targeting gRNA-Linie in ein neues Fläschchen (im Folgenden Step 2 Cross genannt). Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Sammeln Sie weitere 5 ein Tage alte Männchen, die sowohl den Ubiq-CasRx (oder Ubiq-dCasRx) als auch den Dual-Luciferase-Reporter aus den Step 1 Cross-Fläschchen ausdrücken, und inkubieren Sie sie für 2 – 4 Tage bei 26 ° C. Anschließend in ein 1,5-ml-Zentrifugenröhrchen geben und bei -80 °C lagern. Wiederholen Sie diesen Schritt 3 Mal für drei Wiederholungen.
- Hinter den Step 2 Cross Fläschchen mit den Elternfliegen bei 26 °C für 48 Stunden. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Bewahren Sie die Durchstechflaschen dann mindestens 20 Tage lang bei 26 °C auf.
- Beobachten Sie jeden Tag die Step 2 Cross Fläschchen, um zu sehen, ob neue erwachsene Nachkommen aus Puppen hervorgegangen sind. Wenn ja, betäuben Sie sie mit Kohlendioxid, bewerten Sie die Phänotypen der betäubten Fliegen und bilden Sie sie mit einer Farbkamera ab, die mit einem fluoreszierenden Stereomikroskop ausgestattet ist. Zählen Sie die Anzahl der Nachkommen mit verschiedenen Phänotypen. Verwenden Sie Bildverarbeitungssoftware für die Nachbearbeitung und Kompilierung von Bildern (Abbildung 3B – 3C).
HINWEIS: Die Mendelsche Genetik legt nahe, dass, wenn Fliegen alle lebensfähig sind, 4 Arten von Fliegen unter den Nachkommen von Schritt 2 Cross erwartet werden, die jeweils 25% der Bevölkerung ausmachen (Abbildung 1B und Abbildung 3A).
- Luciferase-Assay (Abbildung 3D)
- Erzeugen Sie die dreifachen transheterozygoten Fliegen sowie die Kontrollfliegen, indem Sie die Schritte 2.4.1-2.4.5 wiederholen. Sammle männliche Fliegen bei der Geburt und altere sie bis zum Alter von 3 Tagen.
- Übertragen Sie die 3 Tage alten Fliegen in 1,5 ml Zentrifugenröhrchen und lysieren Sie sie mit einem Stößel und dem Luciferase-Lysepuffer des kommerziell erhältlichen Luciferase-Assay-Kits.
- Verwenden Sie 5 μL lysiertes Gewebe aus jeder Probe, um sowohl die Glühwürmchen- als auch die Renilla-Luciferase-Aktivität mit handelsüblichen Luciferase-Assay-Kit und Luminometer zu messen.
3. Gewebespezifisches in vivo RNA-Targeting mit einem Drei-Komponenten-CasRx-System
- Generieren von UASt-CasRx- und UASt-dCasRx-Ausdrucksvektoren
- PCR amplifizieren die UASt-Promotorsequenz unter Verwendung des Plasmids pJFRC81 und der Primer 1041.C9 und 1041.C11; Anschließend amplifizieren die PCR das CasRx-Fragment mit dem Plasmid OA-1050E (Addgene ID #132416) und den Primern 1050L. C1 und 1050E. C4; und dann PCR amplifizieren dCasRx Fragmente mit Plasmid OA-1050R (Addgene ID #132417) und Primern 1050L. C1 und 1050E. C4 (Tabelle 1)26. Gel-Reinigung der amplifizierten UASt-Promotorsequenz, CasRx und dCasRx-Fragmente mit einem Gelreinigungskit.
- Digest-Basenvektor (Addgen-Plasmid #112686) mit Restriktionsenzymen NotI und PacI24. In den resultierenden Produkten reinigen Sie das größere Fragment, das als Basisvektor-Rückgrat bezeichnet wird, mit einem Gelreinigungskit.
- Setzen Sie den UASt-CasRx-Vektor mit dem Basisvektor-Backbone, der UASt-Promotorsequenz und dem CasRx-Fragment unter Verwendung der Gibson-Assembly zusammen. Setzen Sie dann den UASt-dCasRx-Vektor mit dem Basisvektor-Backbone, der UASt-Promotorsequenz und dem dCasRx-Fragment mit der Gibson-Assembly-Methode zusammen25.
HINWEIS: Der UASt-CasRx-Vektor ist Addgen-Plasmid #132418, und der UASt-dCasRx-Vektor ist Addgene-Plasmid #132419
- Erzeugung transgener Fliegen
- Injektion von UASt-CasRx-Vektor in Fliegenembryonen mittels Fliegenembryo-Injektionsdienst und Embryonen von Fliegen ØC31 Integrationsstelle 8621 auf ihren 2. Chromosomen ; Dann injizieren Sie UASt-dCasRx-Vektor in Fliegenembryonen mit Hilfe des Fliegenembryo-Injektionsdienstes und Embryonen von Fliegen ØC31 Integrationsstelle 8621 auf ihren 2. Chromosomen . Aufziehen der injizierten Embryonen bei 26 °C.
- Halten Sie die Fliegen als doppelt ausbalancierte heterozygote Linien mit CyO- und Sb-Markern. HINWEIS: Die IDs der Flylines in BDSC sind 84121 (UASt-CasRx) und 84120 (UASt-dCasRx).
- Fliegengenetik (Abbildung 1C)
- Bestellen Sie die gewünschten GAL4-Zeilen von BDSC; Erhalten Sie relevante gRNA-Linien aus Schritt 3.2.2 (oder aus BDSC).
HINWEIS: Die folgenden 2 GAL4-Fliegen von BDSC wurden verwendet: GAL4-GMR (BDSC ID: #29967), GAL4-y (BDSC ID: #44373). Die gleichen 3 gRNA-Linien, die im ersten Abschnitt generiert wurden, wurden verwendet: gRNAw (BDSC ID: #84124), gRNAN (BDSC ID #84122), gRNAy(BDSC ID: #84123). - Sammeln Sie 5-10 jungfräuliche erwachsene weibliche Fliegen aus der gRNA-Linie. Sammeln Sie 2-4 erwachsene männliche Fliegen aus dem doppelt ausbalancierten heterozygoten UASt-CasRx / CyO; +/TM6, Sb-Linie, die gleichzeitig weißäugige, lockige Flügel und dsRed-Fluoreszenz zeigen. Legen Sie die gesammelten weiblichen und männlichen Fliegen, die die elterlichen Fliegen sind, in ein normales Lebensmittelfläschchen (im Folgenden Schritt 1 Kreuz genannt). Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate. Verwenden Sie für die Kontrollgruppe UASt-dCasRx/CyO. +/TM6, Sb-Linie, während alles andere gleich bleibt.
- Ziehen Sie die Step 1 Cross-Fläschchen mit den Elterlichen Fliegen bei 26 °C für 48 Stunden zurück. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Bewahren Sie die Durchstechflaschen dann mindestens 14 Tage lang bei 26 °C auf. Sammeln Sie während dieser Zeit 5-10 weibliche Jungfrauen aus der GAL4-Linie. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Beobachten Sie jeden Tag die Step 1 Cross Fläschchen, um zu sehen, ob eine neue erwachsene Fliege aus Puppen auftaucht. Wenn ja, betäuben Sie sie mit Kohlendioxid, sammeln Sie 2-4 männliche Fliegen, die sowohl den UASt-CasRx (oder UASt-dCasRx) als auch den gRNA-Vektor aus den Nachkommen exprimieren, die gleichzeitig dsRed-fluoreszierende und stoppelige Phänotypen haben. Legen Sie die gesammelten Männchen von Step 1 Cross zusammen mit 5-10 gesammelten jungfräulichen Weibchen aus der GAL4-Linie (im Folgenden Step 2 Cross genannt) in ein neues Fläschchen. Wiederholen Sie diesen Schritt 3 Mal für 3 Replikate.
- Hinter den Step 2 Cross Fläschchen mit den Elternfliegen bei 26 °C für 48 Stunden. Entfernen Sie dann alle elterlichen Fliegen aus jeder Durchstechflasche. Bewahren Sie die Durchstechflaschen dann mindestens 20 Tage lang bei 26 °C auf.
- Beobachten Sie jeden Tag die Step 2 Cross Fläschchen, um zu sehen, ob ein neuer erwachsener Fliege aus Puppen hervorgeht. Wenn ja, betäuben Sie sie mit Kohlendioxid, bewerten Sie die Phänotypen der betäubten Fliegen und bilden Sie sie mit einer Farbkamera ab, die mit einem fluoreszierenden Stereomikroskop ausgestattet ist. Zählen Sie die Anzahl der Nachkommen mit verschiedenen Phänotypen. Verwenden Sie Bildverarbeitungssoftware für die Nachbearbeitung und Kompilierung von Bildern (Abbildung 4).
HINWEIS: Die Mendelsche Genetik legt nahe, dass, wenn Fliegen alle lebensfähig sind, 4 Arten von Fliegen unter den Nachkommen von Schritt 2 Cross erwartet werden, die jeweils 25% der Bevölkerung ausmachen (Abbildung 1C).
- Bestellen Sie die gewünschten GAL4-Zeilen von BDSC; Erhalten Sie relevante gRNA-Linien aus Schritt 3.2.2 (oder aus BDSC).
Ergebnisse
Ubiquitäres In-vivo-RNA-Targeting mit einem Zwei-Komponenten-CasRx-System
Die F1-Transheterozygot-Fliegen, die sowohl die Ubiq-CasRx- als auch die gRNA-Konstrukte (die sowohl auf endogene als auch auf exogene Gene abzielen) exprimieren, zeigten ausgeprägte Phänotypen im Vergleich zu den Kontrollfliegen, die die Ubiq-dCasRx- und gRNA-Konstrukte exprimieren (Abbildung 2 und Abbildung 4). Insbesondere haben die transheterozygoten CasRx-Fliegen im Vergleich zu den transheterozgyen dCasRx-Fliegen eine signifikant niedrigere Überlebensrate, was auf eine Toxizität des Ubiq-CasRx-Systems hinweist (Abbildung 2A und Abbildung 4A). Es ist erwähnenswert, dass sowohl transheterozygote CasRx- als auch dCasRx-Fliegen eine Vererbungsrate von weniger als 50% aufweisen, was das erwartete Verhältnis basierend auf der Mendelschen Genetik ist. Von den drei Zielgenen ist das Ubiq-CasRx/+; U6-gRNAN/+ fliegt und Ubiq-CasRx/+; U6-gRNAy/+ Fliegen sind nicht lebensfähig (0% Vererbung) und wuchsen nicht über das zweite Larvenstadium hinaus (Abbildung 2A-2B). Die überlebende Ubiq-CasRx/+; U6-gRNAw/+ Fliegen, deren Vererbung 12,9% betrug, zeigten einen ausgeprägten vollständig durchdringenden Weißäugigen Phänotyp (Abbildung 2B). Zusätzlich zu den beobachtbaren Merkmalen, die mit CasRx assoziiert sind, konnten wir eine signifikante Reduktion der Zielgentranskripte für 3 Zielgene bestätigen: Notch, Gelb und GFP (Abbildung 2E-2G). Eine Reduktion der weißen Gentranskripte wurde bei Ubiq-CasRx/+-, U6-gRNAw/+-Fliegen im Vergleich zu den Kontrollfliegen Ubiq-dCasRx/+, U6-gRNAw/+ beobachtet, obwohl die Reduktion statistisch nicht signifikant war (Abbildung 2E - 2F). Hinweise auf eine durch CasRx induzierte Off-Target-Aktivität wurden beim Vergleich der differentiell exprimierten Transkripte zwischen Proben von CasRx-exprimierenden Fliegen und Proben von dCasRx-exprimierenden Fliegen gefunden (Abbildung 2E, 2G). Die Anzahl der Nicht-Ziel-Transkripte, die signifikant unterschiedlich ausgedrückt werden, ist wie folgt: weiß, 253 (1,4% der gesamten Transkripte); Kerbe, 300 (1,7%); gelb, 41 (0,23%); GFP, 5.880 (33%) (Abbildung2G). Von den insgesamt 17.779 verschiedenen Transkripten wurden 6 Nichtzieltranskripte in allen 4 Gruppen von Stichproben signifikant unterschiedlich exprimiert. Eines der 6 identifizierten Transkripte war Gadd45, ein Gen, das an apoptose und zellulärem Stillstand bei Fliegen beteiligt ist, was die Möglichkeit erhöht, dass die enzymatische Wirkung von CasRx entweder direkt zelluläre Apoptose auslösen oder indirekt eine Fehlexpression anderer Gene auslösen kann, was wiederum zu Apoptose führt. Schließlich ist es erwähnenswert, dass die Fliegen Ubiq-CasRx und Ubiq-dCasRx nicht als homozygote Bestände etabliert wurden, vermutlich aufgrund der Toxizität, die durch eine hohe ubiquitäre Expression verliehen wird. Infolgedessen wurden heterozygote Ubiq-CasRx/CyO- und Ubiq-dCasRx/CyO-Fliegen für die Kreuzung mit homozygoten gRNA-Fliegenlinien verwendet. Zusammenfassend lässt sich sagen, dass das Zwei-Komponenten-Ubiq-CasRx-System in der Lage ist, ein ubiquitäres RNA-Targeting sowohl für endogene als auch für exogene Ziele zu erreichen, was zu beobachtbaren Phänotypen und Transkriptreduktion führt. Diese Ergebnisse zeigten auch, dass CasRx-vermitteltes RNA-Targeting Toxizität in vivo einführen kann.
Allgegenwärtiges in vivo exogenes RNA-Targeting mit einem Drei-Komponenten-CasRx-System
Die Ergebnisse des zweistufigen Kreuzes zeigten, dass trotz der exogenen Natur des Zielgens (d. h. Fluc) die Exprimierung aller drei Transgene in F2-Dreifachtransheterozygoten (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) zu einer Letalität von 100% im Vergleich zu Kontrollkreuzungen mit Ubiq-dCasRx führte, bei denen bei den F2-Dreifach-Transheterozygoten (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) keine Letalität beobachtet wurde (Abbildung 3B-C ). Genauer gesagt führte nur die Kombination aller drei Transgene (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) zu einer 100%igen Letalität (Abbildung 3B und D), während (Ubiq-CasRx/+; gRNAFluc/TM6) und (Ubiq-CasRx/+; Ubiq-Fluc-Ubiq-Rluc/TM6) Genotypen waren lebensfähig und es fehlten Phänotypen, deren Vererbungsraten den erwarteten Mendelschen Übertragungsraten entsprachen, was darauf hindeutet, dass die Verfügbarkeit der Zielsequenz (d.h. Glühwürmchen-Luciferase) in Kombination mit Ubiq-CasRx/+ und dem gRNAFluc zu den beobachteten Letalitätsphänotypen führte, die vermutlich auf die Kollateralaktivität von Cas13-Enzymen zurückzuführen sind2,8 . Darüber hinaus gibt es keine unterscheidbaren Phänotypen oder dramatischen Einfluss auf die Vererbung in F1-Transheterozygoten (Ubiq-CasRx/+; gRNAFluc/+ oder Ubiq-CasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) wurden im Vergleich zu Ubiq-dCasRx-Kontrollen (Ubiq-dCasRx/+; gRNAFluc/+ oder Ubiq-dCasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) (Abbildung 3B), was darauf hindeutet, dass ein katalytisch aktives Enzym essentiell ist, um die beobachteten Letalitätsphänotypen zu erhalten. Darüber hinaus zeigten die Fluc- und Rluc-Expressionsniveaus bei Fliegen aller lebensfähigen Genotypen keine signifikante Reduktion der Fluc-Expression in den Ubiq-dCasRx-Dreifachtransheterozygoten (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) im Vergleich zu dualen Luciferase-Reporterkontrollen. Dies deutet darauf hin, dass die Fluc-Proteinexpressionsniveaus durch dCasRx-Targeting nicht reduziert wurden (Abbildung 3D). Zusammengenommen zeigt der gemeinsame Letalitätsphänotyp in den beiden verschiedenen CasRx-vermittelten ubiquitären RNA-Targeting-Experimenten, dass CasRx-vermitteltes RNA-Targeting bei ubiquitärer Verwendung auf Geweben für den Organismus toxisch sein kann.
Gewebespezifisches in vivo RNA-Targeting mit einem Drei-Komponenten-CasRx-System
Die hohe Toxizität, die in ubiquitären RNA-Targeting-Experimenten beobachtet wurde, veranlasste uns, das gewebespezifische RNA-Targeting mit einem dreikomponentigen CasRx-Systemdesign zu untersuchen, das im Methodenabschnitt beschrieben wird. In der Tat wurde der Grad der beobachteten Toxizität reduziert, wenn die Gesamtex-CasRx-Expression unter Verwendung des UASt-Promotors im Vergleich zu der des Ubiq-Promotors gesenkt wurde, dies wird durch drei Aspekte veranschaulicht: 1) die UASt-CasRx- und UASt-dCasRx-Linien wurden als homozygote Linien beibehalten, obwohl auf der Grundlage des zweistufigen Kreuzschemas doppelt ausbalancierte UASt-CasRx- und UASt-dCasRx-Linien zur Durchführung der Kreuze verwendet wurden, 2) alle dreifach transheterozygoten Vererbungsraten der F2-Generation dCasRx entsprachen der erwarteten Mendelschen Vererbungsrate von 25%, und 3) der CasRx-Dreifachtransheterozygot-Letalitätsphänotyp der F2-Generation war moderat reduziert. Im White-Targeting-Experiment wurden von den 25% Mendelschen Vererbungsraten, die in den F2-Dreifachtransheterozygoten erwartet wurden, nur 0,57% lebensfähige erwachsene Fliegen (UASt-CasRx/+; gRNAw/GMR-Gal4) beobachtet, die alle schwere augenspezifische Pigmentierungs- und Morphologiephänotypen aufwiesen (Abbildung 4A und 4B). Für das White-Targeting-Kreuz war die CasRx-exprimierende dreifache transheterozygote F2-Vererbungsrate signifikant niedriger als die der dCasRx-exprimierenden dreifachen transheterozygoten Kontrollgruppe (27,6%) (Abbildung 4A). Im Notch-Targeting-Experiment waren CasRx-exprimierende dreifache Tranheterozygous, die alle drei Transgene trugen, zu 100% tödlich, während die Vererbungsrate der dCasRx-Kontrolle 29,3% betrug (Abbildung 4A). Im Gelb-Targeting-Experiment zeigten F2 Triple Transheterozygous CasRx-exprimierend, gRNAy und y-GAL4 eine marginale Chitinpigmentreduktion als kleine Flecken gelber Kutikula am Brustkorb und Abdomen mit einer Vererbungsrate von 2,67%, viel niedriger als die der dCasRx-Kontrollgruppe (25,2%) (Abbildung 4A ). Alle dCasRx-Kontroll-Dreifach-Transheterozygot-Fliegen wiesen keine offensichtlichen Phänotypen wie die CasRx-exprimierenden Fliegen auf, was darauf hindeutet, dass die katalytische Aktivität von CasRx zu den beobachteten Phänotypen beitrug. Die niedrige Vererbungsrate in der CasRx-Dreifachtransheterozygot-Gruppe deutete darauf hin, dass im CasRx-RNA-Targeting zwei Toxizitätsquellen existieren: Eine ist mit einer hohen Expression von CasRx verbunden, deren Toxizität durch restriktive CasRx-Expression reduziert wurde, die andere ist mit der Kollateralaktivität verbunden. Zusammengenommen zeigten diese Ergebnisse, dass das CasRx-System durch die Nutzung des klassischen Gal4/UASt-Systems ein gewebespezifisches in vivo RNA-Targeting erreichen und in der Zwischenzeit die Toxizität reduzieren kann. Toxizitäts- und gelegentliche Letalitätsphänotypen wurden jedoch im Vergleich zu den allgegenwärtigen Ansätzen immer noch bei einem niedrigeren Schweregrad beobachtet, was darauf hindeutet, dass die Kollateralspaltungsaktivität mit der Toxizität verbunden ist.
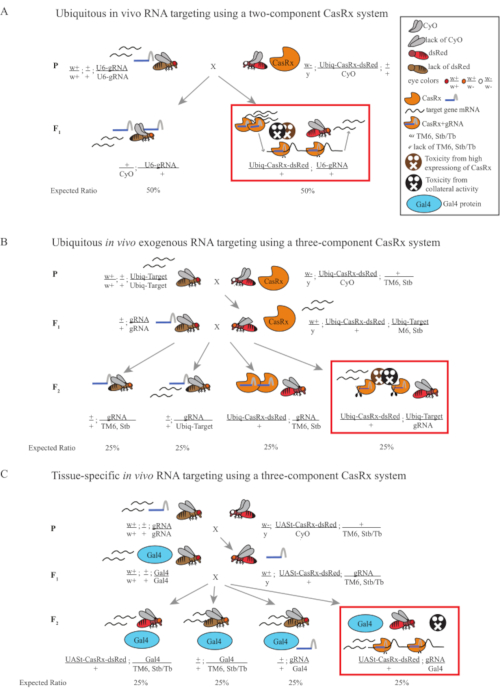
Abbildung 1: Allgemeiner Überblick über das RNA-Targeting mit einem Cas13D-System. (A) Schematische Darstellung des einstufigen genetischen Kreuzes im ubiquitären in vivo RNA-Targeting mit einem Zwei-Komponenten-CasRx-System. (B) Schematische Darstellung einer zweistufigen genetischen Kreuzung in der ubiquitären in vivo exogenen RNA, die unter Verwendung des Drei-Komponenten-CasRx-Systems abzielt. (C) Schematische Darstellung einer zweistufigen genetischen Kreuzung in der gewebespezifischen in vivo RNA, die mit einem Drei-Komponenten-CasRx-System zielt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
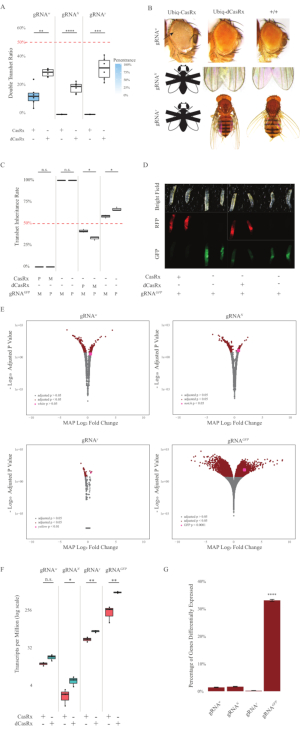
Abbildung 2: Ubiquitous in vivo RNA targeting using a two-component CasRx system (reprinted5). (A) Total inheritance percentages of transheterozygous flies inheriting Ubiq-CasRx (or Ubiq-dCasRx) and gRNAs. Die blaue Schattierung im Box-Plot zeigt die Penetranz des Phänotyps an. (B) Phänotypen von transheterozygoten Fliegen. Pfeile zeigen Gewebenekrose im Auge an. Schwarz-weiße Fliege, die mit ''X'' markiert ist, steht für Letalität. (C) Gesamtvererbungsprozentsätze von transheterozygoten Fliegen bidirektionaler Kreuzungen zwischen Ubiq-CasRx (oder Ubiq-dCasRx) und gRNAGFP-OpIE2-GFP-Fliegen. M, mütterliches Erbe von CasRx; P, väterliches Erbe von CasRx. (D) F1-Larven-Nachkommen im väterlichen Kreuz. (E) Maximale a posteriori-Schätzungen der Transkripte für die logarithmische Faltenänderung. DESeq2-Pipeline wurde verwendet. (F) Transkripte pro Million (TPM), die mit CasRx oder dCasRx angestrebt werden. (G) CasRx-abhängiger differenziell ausgedrückter Transkriptprozentsatz von Transkripten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
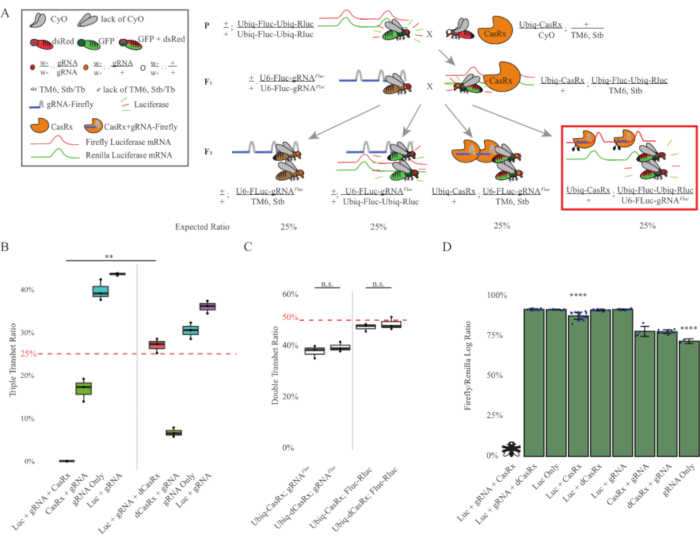
Abbildung 3: Ubiquitäres in vivo exogenes RNA-Targeting unter Verwendung eines Drei-Komponenten-CasRx-Systems. (A) Schemata der zweistufigen genetischen Kreuzung. (B) Gesamtvererbungsprozentsätze für alle Genotypen, die in der F2-Generation entstehen. Die Vererbung aller drei Transgene (Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc und gRNAFLuc) in F2-Nachkommen ergab eine 100%ige Letalität und war im Vergleich zur Ubiq-dCasRx-Dreifach-Transheterozygotes-Kontrollgruppe signifikant niedriger (p = 0,001, t-Test). (C) Das Tragen von Ubiq-CasRx/gRNAFluc allein oder Ubiq-CasRx und Ubiq-Fluc-Ubiq-Rluc allein führte nicht zu schwerer Letalität, und die Vererbungsverhältnisse zwischen Ubiq-CasRx und Ubiq-dCasRx-Transheterozygoten unterschieden sich nicht signifikant (p = 0,41 bzw. p = 0,51, t-Test). (D) Luziferase-Verhältnis von normalisierenden Fluc-Messwerten zu Rluc-Messwerten. Dreifache transheterozygote Fliegen, die Ubiq-CasRx exprimieren, Ubiq-Fluc-Ubiq-Rluc, gRNAFLuc waren embryonal letal, was durch eine Fliege mit einem "X" dargestellt wurde, und infolgedessen wurde die Luciferase-Expression nicht gemessen. Fluc/Rluc-Verhältnis von Ubiq-CasRx/+, Ubiq-Fluc-Ubiq-Rluc/TM6, Stb-Transheterozygoten war signifikant niedriger als das der anderen Ubiq-Fluc-Ubiq-Rluc-exprimierenden Gruppen (p = 1,2e-06 oder niedriger, t-Test). Die Ergebnisse aus der gRNAFLuc-only-Gruppe waren signifikant niedriger als die aller anderen Gruppen (p = 1,2e-06 oder niedriger, t-Test). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
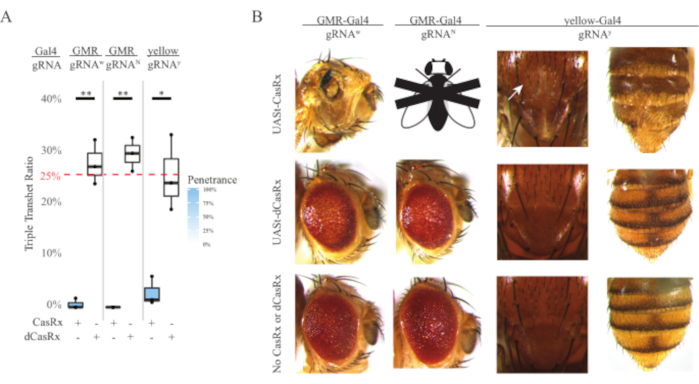
Abbildung 4: Gewebespezifisches in vivo RNA-Targeting mit einem Drei-Komponenten-CasRx-System (nachgedruckt5). (A) Gesamtvererbungsprozentsatz von dreifach transheterozygoten Fliegen, die drei Transgene (UASt-CasRx oder UASt-dCasRx, gRNAs und Gal4-Treiber) tragen. (B) Phänotypen der dreifach transheterozygoten Fliegen. Der weiße Pfeil zeigt eine Chitinpigmentreduktion im Thorax an. Schwarz-weiße Fliege, die mit ''X'' markiert ist, steht für Letalität. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Bauen | Beschreibung | Fibel | Primer-Sequenz (5' bis 3') | PCR-Vorlage |
| OA-1050E | CasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA AGAA | pNLS-RfxCas13d-NLS-HA (pCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTGG AACA | |||
| OA-1050R | dCasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA AGAA | pNLS-dRfxCas13d-NLS-HA (pdCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTG GAACA | |||
| OA-1050L | UASt-Projektträger | 1041.C9 | GCGGGTTCTCGA CGGTCACGGCGG GCATGTCGACGC GGCCGCAACCAA CAACACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCACC TTTCTCTTCTTCT TGGGGCTCATGT TTAAACCCAATT CCCTATTCAGA | |||
| CasRx | 1050L. C1 | AATACAAGAAGA GAACTCTGAATA GGGAATTGGGT TTAAACATGAGC CCCAAGAAGAA | pCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTA GCTAGCTTAAG CGTAATCTGGA ACA | |||
| OA-1050S | UASt-Projektträger | 1041.C9 | GCGGGTTCTC GACGGTCACG GCGGGCATGT CGACGCGGCC GCAACCAACAA CACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCA CCTTTCTCTTC TTCTTGGGGCT CATGTTTAAAC CCAATTCCCTA TTCAGA | |||
| dCasRx | 1050L. C1 | AATACAAGAAG AGAACTCTGAAT AGGGAATTGGG TTTAAACATGAG CCCCAAGAAGAA | pdCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTAG CTAGCTTAAGCG TAATCTGGAACA | |||
| OA-1043 | U6:3 Veranstalter | 1043.C1 | GGGAATTGGGA ATTGGGCAATAT TTAAATGGCGGC GCGCCGAATTCT TTTTTGCTCACCT | Addgen-Plasmid #164586 |
| 1043.C23 | ACACTAGTGGAT CTCTAGAGGTAC CGTTGCGGCCG CAAAAAAGTTGT AATAGCCCCTCA AAACTGGACCTT CCACAACTGCAG CCGACGTTAAAT TGAAA | |||
| OA-1052B | Ubiq-Projektträger | Nr. 1052B. C1 | GGGAATTGGGCA ATATTTAAATGGC GGCTGCAGCGC GCAGATCGCCGAT | Addgenplasmid #112686 |
| Nr. 1052B. C2 | TTTCTTTATGTTT TTGGCGTCTTCC ATCCTAGGTCTG CGGGTCAAAATA GAGATG | |||
| T2A-eGFP | 908A1 | ATAAAGGCCAAG AAGGGCGGAAA GATCGCCGTGG AGGGCAGAGGA AGTCTTCTAACAT GC | Addgenplasmid #112686 | |
| 908A2 | TTGTTATTTTAAAA ACGATTCATTCTA GGCGATCGCTTA CTTGTACAGCTC GTCCATGCC | |||
| Umgekehrter Ubiq-Promoter | 908A3 | ACCGTGACCTAC ATCGTCGACACTA AGBGGATCTCTAGA CGCGCAGATCGC CGATG | Addgenplasmid #112686 | |
| 908A4 | GGATCATAAACTT TCGAAGTCATGC GGCCGCTCTGCG GGTCAAAATAGAG ATGT |
Tabelle 1: Liste der molekularen Konstrukte und Primer, die in dieser Studie verwendet wurden. Diese Liste enthält alle Konstrukte (sowohl die ID als auch die Beschreibung) und die jedem Konstrukt zugeordneten Primer (sowohl die ID als auch die Sequenzen (5' bis 3')) und die verwendeten Vorlagen.
Diskussion
Mit drei verschiedenen Anwendungsdesigns des CasRx-Systems demonstrierte diese Arbeit in vivoprogrammierbares RNA-Targeting in Fliegen. Die verschiedenen Strategien sind auf unterschiedliche Projektanforderungen ausgerichtet, z. B. endogenes versus exogenes Gen-Targeting und allgegenwärtiges versus gewebespezifisches RNA-Targeting. Die Wirkungen des RNA-Targetings umfassten zielgenspezifische phänotypische Veränderungen, die Reduzierung des Ziel-RNA-Transkripts und gelegentliche Letalitätsphänotypen, die mit einer hohen Expression des CasRx-Proteins und der Kollateralaktivität assoziiert sind. Insgesamt zeigten diese Ergebnisse, dass das CasRx-System in der Lage ist, die RNA-Transkriptreduktion auf organismischer Ebene programmierbar und effizient zu reduzieren.
Einer der Schlüsselfaktoren für ein erfolgreiches Customizing des CasRx-Systems ist das Design von gRNAs. Insbesondere ist der folgende Rat zu beachten: Die Zielsequenz ist etwa 30 Nukleotide lang, die Länge der Poly-U-Dehnungen in der Zielsequenz beträgt 4 Basenpaare oder weniger, der GC-Gehalt der Zielsequenz liegt im Bereich von 30% - 70%, die Zielsequenz wird nicht vorhergesagt, um starke RNA-Haarnadelstrukturen zu bilden, und die Zielsequenz enthält eine minimale vorhergesagte sekundäre oder tertiäre RNA-Struktur5.
Neben den gRNA-Designs ist auch der Schritt der Fliegengenetik in jedem Protokoll entscheidend für eine erfolgreiche Implementierung. Das Vorhandensein oder Fehlen der definierten Phänotypen, die von den Eltern in den Nachkommen weitergegeben werden, ist wichtig für die Identifizierung und Quantifizierung von Phänotypen, die durch das CasRx-System in den transheterozygoten Nachkommen induziert werden. Auch das parallele Einrichten von Kontrollkreuzen mit den dCasRx-Fliegen ist hilfreich, um unspezifische Phänotypen in den transheterozygoten Nachkommen auszuschließen.
Es ist erwähnenswert, dass diese Ergebnisse ein Toxizitätsproblem aufdeckten, das durch die allgegenwärtige Expression von CasRx- und dCasRx-Protein im Flug eingeführt wurde, eine Einschränkung des CasRx-Systems. Die allgegenwärtige Expression von CasRx oder dCasRx unter dem Ubiq-Promotor allein, ohne gRNAs, war mit nichttrivialen Fitnesskosten verbunden, da weder Ubiq-CasRx- noch Ubiq-dCasRx-Fliegen als homozygote Linien etabliert werden konnten. Im Gegenteil, UASt-CasRx- und UASt-dCasRx-Fliegen können als gesunde homozygote Bestände etabliert werden, obwohl sie aufgrund des Designs des Kreuzschemas als doppelt ausgewogene Bestände gehalten wurden, eine Tatsache, die die Existenz einer Toxizität unterstützt, die durch die allgegenwärtige CasRx-Proteinexpression induziert wird. Ein weiterer unterstützender Beweis ist, dass in Kontrollexperimenten mit dCasRx, das katalytisch inaktiv ist, die Prozentsätze der Fliegen, die sowohl dCasRx- als auch gRNA-Konstrukte aus der Gesamtzahl der Fliegen in der F1-Generation tragen, durchweg niedriger als 50% waren, das Verhältnis, das basierend auf der Mendelschen Genetik erwartet wurde, wenn keine dCasRx-assoziierte Toxizität vorhanden war. Dies deutete darauf hin, dass die ubiquitäre Expression von dCasRx zusammen mit gRNAs eine Toxizität in der Fliege induziert, was zu einem geringeren Vererbungsverhältnis als erwartet führt. Die Vererbungsverhältnisse von transheterozygoten UASt-dCasRx-, gRNA-, GAL4-Fliegen folgten der Mendelschen Genetik, was wiederum auf die Toxizität hindeutet, die spezifisch durch die ubiquitäre Expression von CasRx- und dCasRx-Proteinen induziert wird. Die Toxizität im CRISPR/Cas-System ist nicht neu. Es hat sich gezeigt, dass hohe Mengen an Cas9-Protein in mehreren Organismen toxisch sind, einschließlich Fliegen29,30,31,32. Eine kürzlich durchgeführte Studie hat ein maßgeschneidertes GAL4 / UAS-System entwickelt, das die Menge an Cas9-Protein, die in Fliegen exprimiert wird, abstimmen kann, indem ein offener Leserahmen unterschiedlicher Länge zwischen der UAS-Sequenz und der Cas9-Sequenz im UAS-Cas9-Konstrukt hinzugefügt wird33. Daher lohnt es sich, Wege zu erkunden, um die CasRx-induzierte Toxizität durch Abstimmung des CasRx-Proteinexpressionsniveaus zu reduzieren.
Abgesehen von der Toxizität, die durch die ubiquitäre Expression von CasRx- und dCasRx-Proteinen induziert wird, zeigten die Ergebnisse auch eine Letalität im Zusammenhang mit den unspezifischen kollateralen Off-Target-Effekten des CasRx-Systems, ein Merkmal vieler CRISPR-Systeme1,2,7,34. In einigen der CasRx- und nicht-essentiellen Gen-gRNA-exprimierenden Doppel- oder Dreifach-Transheterozygot-Fliegen, zum Beispiel beim Targeting von Notch, haben die transheterozygoten CasRx-Fliegen im Vergleich zu den transheterozgyen dCasRx-Fliegen eine signifikant niedrigere Überlebensrate. In der RNA-seq-Analyse dieser CasRx- und gRNA-exprimierenden transheterozygoten Fliegen wurde sowohl die Reduktion der Ziel-Gentranskript-Spiegel als auch die Reduktion von Nicht-Ziel-Gentranskripten beobachtet. Diese Kollateraleffekte waren CasRx-abhängig und zielabhängig, da sie nur bei transheterozygoten Fliegen beobachtet wurden, die sowohl das CasRx-Protein als auch die gRNA exprimierten. Es ist erwähnenswert, dass eines der Zielgene, weiß, nur eine begrenzte, nicht statistisch signifikante Reduktion der Transkripte zeigte, wenn das weiße Gen von CasRx angegriffen wurde, was im Gegensatz zum klaren Pigmentreduktionsphänotyp stand. Es wird angenommen, dass dies auf die Tatsache zurückzuführen sein könnte, dass 1) der Zeitpunkt der RNA-seq-Probenentnahme nicht gut auf den Zeitpunkt abgestimmt war, zu dem das weiße Gen während der frühen Entwicklung seine maximale Expression erreicht, und 2) die lokalisierte Expression des weißen Gens in den Augen es schwierig macht, die relevanten Gewebe während der frühen Entwicklungsphase zu sammeln, wenn nur die Ganzkörperprobenentnahme möglich ist. Um die Kollateralaktivität im CasRx-System zu reduzieren, sind zukünftige Studien erforderlich, um die Mechanismen, die dem Off-Target-Phänomensystem auf organismischer Ebene zugrunde liegen, vollständig zu verstehen.
Interessanterweise schien eine kürzlich durchgeführte Studie35 , in der RNA-Targeting-Cas13-Werkzeuge in Fliegen beschrieben wurden, die allgemeine Toxizität im Zusammenhang mit der CasRx-Expression aus mehreren möglichen Gründen zu verbessern. Erstens kodierten die Autoren die Cas13-Transgene neu, um die Expression in Drosophila zu optimieren, und verwendeten einen schwächer exprimierenden Promotor (Actin 5C) im Vergleich zu dem in der vorliegenden Studie verwendeten Ubiquitin-Promotor, was wahrscheinlich zu niedrigeren Cas13-Expressionsraten und damit zu einer geringeren Toxizität führte. Tatsächlich wird dies durch die Beobachtungen gestützt, dass die UASt-gesteuerte CasRx- und dCasRx-Expression an sich nicht toxisch war, da diese Studie (und die Autoren in 35) keine offensichtliche Letalität bei UASt-CasRx-Fliegen beobachteten. Darüber hinaus kodierten diese Autoren ihre gRNAs anders als in dieser Studie, was ihre Expression beeinflusst und die Toxizität des Systems in transheterozygoten Cas13/gRNA-Fliegen reduziert haben könnte. Zum Beispiel wurden in ihrer Studie zwei gRNAs mit dem U6: 3-Promotor exprimiert und von tRNAs flankiert, um die gRNA-Verarbeitung bei der tRNA-Reifung zu ermöglichen, ohne CasRx35 zu benötigen. Umgekehrt wurden die gRNAs in dieser Studie als Arrays kodiert, die auf bis zu 4 Stellen pro Gen abzielen und die endogene Cas13-Array-Struktur in Bakterien nachahmen, die das Cas13-Enzym benötigt, um jede gRNA zu verarbeiten. Diese unterschiedlichen Ansätze könnten zu Unterschieden in der gRNA-Expression und anderen Faktoren geführt haben, die inhärente Auswirkungen auf die Toxizität des gesamten Systems haben können. Schließlich zielten Huynh et al. auf andere Gene ab als die in der vorliegenden Studie angesprochenen, was zu Unterschieden in der Target-Cas/gRNA-Interaktion und der Kollateralaktivität führt und Auswirkungen auf die beobachtete Letalität haben kann. Diese Unterschiede in der beobachteten Toxizität rechtfertigen weitere Untersuchungen, um Wege zu finden, wie die Gesamtsysteme verbessert werden können.
Insgesamt ist diese Studie die erste Demonstration eines funktionellen, genetisch kodierten programmierbaren RNA-Targeting-Cas-Systems in D. melanogaster, obwohl eine weitere Optimierung des CasRx-Systems (im Einklang mit dem, was berichtet wird35) erforderlich sein wird, um die Off-Target-assoziierte Letalität weiter zu reduzieren und die Wirksamkeit der CasRx-On-Target-Spaltung zu erhöhen. RNA-Targeting mit Cas-Enzymen ist ein sich schnell entwickelndes Feld mit vielen potenziellen Anwendungen, die von der Insektenvektorkontrolle bis hin zu therapeutischen Anwendungen reichen1,2,3,4,5,6,7, und dieses Protokoll bietet ein Starterpaket für alle, die daran interessiert sind, ihr erstes CasRx-System in Fliegen zu entwerfen, während es mit der Anpassung und weiteren Optimierung des Systems kompatibel ist. Die hier vorgestellten Beispiele zeigen eine Reihe von Ergebnissen, die bei der Implementierung dieses Systems in vivo auftreten können, und können als Benchmarks für andere Benutzer bei der Bewertung der Leistung des CasRx-Systems in ihren Anwendungen dienen.
Offenlegungen
O.S.A ist einer der Gründer von Agragene, Inc., hält eine Kapitalbeteiligung und ist Mitglied des wissenschaftlichen Beirats des Unternehmens. Die Bedingungen dieser Vereinbarung wurden von der University of California, San Diego, in Übereinstimmung mit ihren Richtlinien zu Interessenkonflikten geprüft und genehmigt. Alle anderen Autoren erklären keine Gegeninteressen.
Danksagungen
Diese Arbeit wurde zum Teil durch die Finanzierung durch einen DARPA Safe Genes Program Grant (HR0011-17-2-0047) und NIH-Auszeichnungen (R21RAI149161A, DP2AI152071) an O.S.A. unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 100% Grape Juice | Welch Foods Inc. | N/A | |
| Active Dry Yeast (908g) | Red Star Yeast Company, LLC | N/A | |
| DMC4500 color camera | Leica Microsystems | DMC4500 | |
| Dual-Luciferase Reporter Assay System | Promega | E1910 | |
| GAL4-GMR flies | Bloomington Drosophila Stock Center | 29967 | |
| GAL4-y flies | Bloomington Drosophila Stock Center | 44373 | |
| Glomax 20/20 Luminometer | Promega | E5331 | |
| Illumina HiSeq2500 Sequencer | Illumina, Inc. | HiSeq2500 | |
| M165FC fluorescent stereomicroscope | Leica Microsystems | M165FC | |
| Nanodrop OneC UV-vis spectrophotometer | ThermoFisher | NDONEC-W | |
| NEBNext Ultra II RNA Library Prep Kit | New England Biolabs, Inc. | E7770 | |
| plasmid # 112686 | Addgene | 112686 | |
| plasmid # 112688 | Addgene | 112688 | |
| plasmid # 132416 | Addgene | 132416 | |
| plasmid # 132417 | Addgene | 132417 | |
| plasmid # 132419 | Addgene | 132419 | |
| plasmid # 132420 | Addgene | 132420 | |
| plasmid # 132421 | Addgene | 132421 | |
| plasmid # 132422 | Addgene | 132422 | |
| plasmid # 132425 | Addgene | 132425 | |
| plasmid # 132426 | Addgene | 132426 | |
| plasmid # 133304 | Addgene | 133304 | |
| plasmid # 164586 | Addgene | 164586 | |
| plasmid #132418 | Addgene | 132418 | |
| plasmid pJFRC81 | Addgene | 36432 | |
| Qiagen RNeasy Mini Kit | Qiagen | 74104 | |
| Restriction endonucleases AscI | New England Biolabs Inc. | R0558L | |
| Restriction endonucleases NotI | New England Biolabs Inc. | R0189L | |
| Restriction endonucleases PacI | New England Biolabs Inc. | R0547L | |
| Restriction endonucleases PstI | New England Biolabs Inc. | R0140L | |
| Restriction endonucleases SwaI | New England Biolabs Inc. | R0604L | |
| Restriction endonucleases XbaI | New England Biolabs Inc. | R0145L | |
| RNA 6000 Pico Kit for Bioanalyzer | Agilent Technologies | 5067-1513 | |
| Turbo DNase | Invitrogen | AM2238 | |
| U6-3:4-gRNA-Fluc flies | Bloomington Drosophila Stock Center | 84125 | |
| U6-3:4-gRNA-GFP; OpIE2-GFP flies | Bloomington Drosophila Stock Center | 84986 | |
| U6-3:4-gRNA-N flies | Bloomington Drosophila Stock Center | 84122 | |
| U6-3:4-gRNA-w flies | Bloomington Drosophila Stock Center | 84124 | |
| U6-3:4-gRNA-y flies | Bloomington Drosophila Stock Center | 84123 | |
| UASt-CasRx flies | Bloomington Drosophila Stock Center | 84121 | |
| UASt-dCasRx flies | Bloomington Drosophila Stock Center | 84120 | |
| Ubiq-CasRx flies | Bloomington Drosophila Stock Center | 84118 | |
| Ubiq-dCasRx flies | Bloomington Drosophila Stock Center | 84119 | |
| Ubiq-Firefly-T2A-eGFP-Ubiq-Renilla flies | Bloomington Drosophila Stock Center | 84127 | |
| Zymoclean Gel DNA Recovery Kits | Zymo Research Corporation | D4007 |
Referenzen
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nat Communications. 9, 1911 (2018).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), 5573 (2016).
- East-Seletsky, A., O'Connell, M., Burstein, D., Knott, G., Doudna, J. RNA Targeting by Functionally Orthogonal Type VI-A CRISPR-Cas Enzymes. Molecular Cell. 66 (3), 373-383 (2017).
- Konermann, S., Lotfy, P., Brideau, N., Oki, J., Shokhirev, M., Hsu, P. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell. 173 (3), 665-676 (2018).
- Buchman, A., Brogan, D., Sun, R., Yang, T., Hsu, P., Akbari, O. Programmable RNA Targeting Using CasRx in Flies. The CRISPR Journal. 3 (3), 164-176 (2020).
- Kushawah, G., et al. CRISPR-Cas13d Induces Efficient mRNA Knockdown in Animal Embryos. Developmental Cell. 54 (6), 805-817 (2020).
- Abudayyeh, O., et al. RNA targeting with CRISPR-Cas13. Nature. 550, 280-284 (2017).
- Perrimon, N., Ni, J., Perkins, L. In vivo RNAi: today and tomorrow. Cold Spring Harbor Perspectives in Biology. 2, 003640 (2010).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
- Ni, J., et al. A Drosophila resource of transgenic RNAi lines for neurogenetics. Genetics. 182 (4), 1089-1100 (2009).
- Ni, J., et al. A genome-scale shRNA resource for transgenic RNAi in Drosophila. Nature Methods. 8, 405-407 (2011).
- Ni, J., et al. Vector and parameters for targeted transgenic RNA interference in Drosophila melanogaster. Nature Methods. 5, 49-51 (2008).
- Heigwer, F., Port, F., Boutros, M. RNA Interference (RNAi) Screening in Drosophila. Genetics. 208 (3), 853-874 (2018).
- Kulkarni, M., et al. Evidence of off-target effects associated with long dsRNAs in Drosophila melanogaster cell-based assays. Nature Methods. 3, 833-838 (2006).
- Ma, Y., Creanga, A., Lum, L., Beachy, P. Prevalence of off-target effects in Drosophila RNA interference screens. Nature. 443, 359-363 (2006).
- Perrimon, N., Mathey-Prevot, B. Matter arising: off-targets and genome-scale RNAi screens in Drosophila. Fly. 1 (1), 1-5 (2007).
- Markstein, M., Pitsouli, C., Villalta, C., Celniker, S., Perrimon, N. Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nature Genetics. 40, 476-483 (2008).
- Champer, J., Buchman, A., Akbari, O. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nature Review Genetics. 17 (3), 146-159 (2016).
- Buchman, A., et al. Broad dengue neutralization in mosquitoes expressing an engineered antibody. PLoS Pathogens. 16 (4), 1008103 (2020).
- Mathur, G., Sanchez-Vargas, I., Alvarez, D., Olson, K., Marinotti, O., James, A. Transgene-mediated suppression of dengue viruses in the salivary glands of the yellow fever mosquito, Aedes aegypti. Insect Molecular Biology. 19 (6), 753-763 (2011).
- Franz, A., et al. Engineering RNA interference-based resistance to dengue virus type 2 in genetically modified Aedes aegypti. Proceedings of the National Academy of Sciences of the United States of America. 103 (11), 4198-4203 (2006).
- Yen, P., James, A., Li, J., Chen, C., Failloux, A. Synthetic miRNAs induce dual arboviral-resistance phenotypes in the vector mosquito Aedes aegypti. Communications Biology. 1, 11 (2018).
- Buchman, A., et al. Engineered resistance to Zika virus in transgenic Aedes aegypti expressing a polycistronic cluster of synthetic small RNAs. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3656-3661 (2019).
- . Addgene Available from: https://www.addgene.org/protocols/restriction-digest/ (2020)
- Gibson, D., Young, L., Chuang, R., Venter, J., Hutchison, C., Smith, H. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Addgene Available from: https://www.addgene.org/protocols/pcr/ (2020)
- . Indiana University Bloomington Available from: https://bdsc.indiana.edu/information/recipes/bloomfood.htmL (2020)
- Dobin, A., et al. STAR:ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Jiang, W., Brueggeman, A., Horken, K., Plucinak, T., Weeks, D. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii. Eukaryotic Cell. 13 (11), 1465-1469 (2014).
- Poe, A., et al. Robust CRISPR/Cas9-Mediated Tissue-Specific mutagenesis reveals gene redundancy and perdurance in Drosophila. Genetics. 211 (2), 459-472 (2019).
- Port, F., Chen, H., Lee, T., Bullock, S. Optimized CRISPR/Cas tools for efficient ger mLine and somatic genome engineering in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 111 (29), 2967-2976 (2014).
- Yang, S., Li, S., Li, X. Shortening the Half-Life of Cas9 maintains its gene editing ability and reduces neuronal toxicity. Cell Reports. 25 (10), 2653-2659 (2018).
- Port, F., et al. A large-scale resource for tissue-specific CRISPR mutagenesis in Drosophila. eLife. 9, 53865 (2020).
- Zhang, X., Tee, L., Wang, X., Huang, Q., Yang, S. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4 (17), 264 (2015).
- Huynh, N., Depner, N., Larson, R., King-Jones, K. A versatile toolkit for CRISPR-Cas13-based RNA manipulation in Drosophila. Genome Biology. 21, 279 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten