Method Article
Ciblage de l’ARN omniprésent et spécifique aux tissus chez Drosophila Melanogaster à l’aide de CRISPR/CasRx
Dans cet article
Résumé
Cet article décrit un protocole détaillé pour l’utilisation de l’enzyme Cas13D ciblant l’ARN (RfxCas13D) chez les mouches.
Résumé
CasRx, un membre de la famille Cas13 ciblant l’ARN, est un nouvel ajout prometteur des technologies CRISPR / Cas dans la réduction efficace des transcriptions géniques avec un profil hors cible attrayant aux niveaux cellulaire et organisationnel. Il a récemment été rapporté que le système CRISPR/CasRx peut être utilisé pour obtenir une réduction ubiquitaire et spécifique du transcript génique tissulaire chez Drosophila melanogaster. Cet article détaille les méthodes issues des travaux récents, composés de trois parties: 1) ciblage in vivo omniprésent de l’ARN endogène à l’aide d’un système CasRx à deux composants; 2) ciblage in vivo de l’ARN exogène omniprésent à l’aide d’un système CasRx à trois composants; et 3) le ciblage in vivo de l’ARN in vivo spécifique aux tissus à l’aide d’un système CasRx à trois composants. Les effets du ciblage de l’ARN observés comprennent des changements phénotypiques spécifiques aux gènes ciblés, une réduction ciblée des transcriptions de l’ARN et des phénotypes de létalité occasionnels associés à une expression élevée de la protéine CasRx et à une activité collatérale. Dans l’ensemble, ces résultats ont montré que le système CasRx est capable de cibler la réduction des transcriptions d’ARN au niveau de l’organisme de manière programmable et efficace, démontrant que le ciblage in vivo du transcriptome et l’ingénierie sont réalisables et jettent les bases de futures technologies de ciblage de l’ARN in vivo basées sur CRISPR.
Introduction
Depuis l’avènement des technologies CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats), l’accent a été mis en grande partie dans ce domaine sur l’édition de l’ADN, qui offre des applications transformatrices en médecine et en biotechnologie1. Cependant, l’altération permanente des séquences d’ADN n’est pas toujours souhaitée pour des raisons éthiques. À la lumière de cela, des études récentes ont commencé à développer des outils basés sur CRISPR pour cibler l’ARN et ont démontré que les technologies CRISPR peuvent effectivement être utilisées pour le ciblage de l’ARN dans une variété de systèmes biologiques2,3,4,5,6,7. Dans bon nombre de ces systèmes testés, l’approche actuelle largement utilisée pour cibler la réduction de l’ARN et des transcriptions est l’interférence ARN (ARNi), qui est loin d’être parfaite, présentant souvent une efficacité variée et une activité hors cible élevée lorsqu’elle est utilisée in vivo8,9,10,11,12,13,14,15,16,17 . Par conséquent, compte tenu de l’état de ces technologies, il convient d’explorer davantage les potentiels des outils basés sur CRISPR pour le ciblage de l’ARN.
Une étude récente notable a rapporté que la ribonucléase CasRx, un membre de la classe Cas13d, peut réduire efficacement les niveaux de transcription des gènes dans la culture cellulaire humaine et possède un profil hors cible attrayant4. Cette découverte a conduit à la question de savoir si cette nouvelle ribonucléase peut maintenir son efficacité et son faible taux hors cible pour le ciblage de l’ARN au niveau de l’organisme. Une étude récente a abordé cette question en montrant que le système CasRx peut être utilisé pour obtenir une réduction du transcript génique omniprésente et spécifique aux tissus chez Drosophila melanogaster5.
Pour rationaliser la convivialité de cette approche récemment publiée, ce protocole détaille les méthodes de ce travail récent, qui se compose de trois parties principales: 1) ciblage in vivo omniprésent de l’ARN à l’aide d’un système CasRx à deux composants; 2) ciblage in vivo de l’ARN exogène omniprésent à l’aide d’un système CasRx à trois composants; et 3) le ciblage de l’ARN in vivo spécifique aux tissus à l’aide d’un système CasRx à trois composants.
Des ARN guides (ARNg) ciblant différents gènes cibles sous le contrôle d’un promoteur omniprésent ont été conçus et des lignées de mouches exprimant ces constructions contenant de l’ARNg ont été générées. Des constructions CasRx sous le contrôle d’un promoteur omniprésent ou d’un promoteur de séquence d’activation conditionnelle en amont (UASt) activable par le facteur de transcription GAL4 ont également été conçues et des lignes de vol hébergeant ces constructions contenant CasRx générées. Les constructions CasRx catalytiquement inactives, dCasRx, ont été conçues et utilisées comme témoins négatifs. Le ciblage de l’ARN omniprésent chez les mouches est réalisé en croisant des lignées de mouches exprimant l’ARNg avec des lignes de mouches exprimant CasRx de manière omniprésente. La progéniture exprimant à la fois la construction de l’ARNg ciblant un transcrit de gène spécifique et la protéine CasRx a une réduction omniprésente des transcriptions de gènes ciblés. Le ciblage de l’ARN spécifique aux tissus chez les mouches est réalisé en croisant d’abord des mouches exprimant l’ARNg avec des mouches exprimant UASt-CasRx, obtenant des mouches transhétérozygotes portant à la fois des constructions d’ARNg et d’UASt-CasRx. Ces mouches sont à leur tour croisées avec des mouches exprimant GAL4 spécifiques aux tissus, ce qui entraîne la génération d’expression de CasRx spécifique aux tissus et le ciblage de l’ARN chez les mouches.
La nature programmable du système CasRx offre la possibilité de personnalisation et d’optimisation pour aider à atteindre une efficacité élevée et une faible activité hors cible pour le ciblage in vivo de l’ARN. Les applications potentielles du ciblage de l’ARN à base de CRISPR sont nombreuses, notamment le remplacement de l’ARNi en laboratoire et la contribution à la lutte contre les insectes vecteurs dans la nature. Parmi ces derniers, l’un des besoins mondiaux non satisfaits est le développement d’outils efficaces pour lutter contre les infections par les virus à ARN transmis par les moustiques. De nombreux virus à ARN, tels que la dengue, le Zika et le chikungunya, sont transmis par les moustiques, ce qui affecte la santé humaine et contribue à la mortalité. De nombreuses propositions visant à concevoir des populations de moustiques résistantes aux virus pour la prévention des maladies ont été faites; cependant, aucune technologie actuelle n’est capable de rendre les moustiques simultanément résistants à tous les virus à ARN importants18,19,20,21,22,23. Les systèmes Cas ciblant l’ARN peuvent fournir un point de départ pour une telle technologie en permettant une plate-forme programmable pour cibler tous les virus à ARN transmis par les moustiques.
Protocole
1. Ciblage in vivo de l’ARN in vivo omniprésent à l’aide d’un système CasRx à deux composants
- Génération du vecteur d’expression Ubiq-CasRx et Ubiq-dCasRx
- Amplifier la séquence CasRx à l’aide d’une réaction en chaîne par polymérase (PCR) avec l’amorce 1050E. C3 et 1050E. C4 et la construction CasRx originale pNLS-RfxCas13d-NLS-HA (pCasRx); et amplifier la séquence dCasRx en utilisant la PCR avec l’amorce 1050E. C3 et 1050E. C4 et la construction dCasRx d’origine pNLS-dRfxCas13d-NLS-HA (pdCasRx)4 (Tableau 1). Purifiez ensuite au gel les fragments casRx et dCasRx amplifiés à l’aide d’un kit de purification de gel.
- Digeste vecteur de base (plasmide Addgene #112686) avec les enzymes de restriction SwaI et PacI24. Dans les produits résultants, utilisez un kit pour purifier le plus grand fragment, appelé colonne vertébrale du vecteur de base.
- Assemblez le vecteur Ubiq-CasRx avec l’épine dorsale du vecteur de base et le fragment CasRx à l’aide de la méthode d’assemblage Gibson; assembler le vecteur Ubiq-dCasRx avec l’épine dorsale du vecteur de base et le fragment dCasRx à l’aide de la méthode d’assemblage Gibson25.
REMARQUE : L’ID Addgene du vecteur Ubiq-CasRx (OA-1050E) est #132416, et l’ID Addgene du vecteur Ubiq-dCasRx (OA-1050R) est #132417.
- Génération d’un vecteur d’expression d’ARNg
- Concevoir chaque fragment d’ARNg en fonction des critères suivants : séquence cible de 30 nucléotides de longueur; la longueur maximale des étirements en poly-U dans la séquence cible étant de 4 paires de bases; la teneur en GC de la séquence cible étant comprise entre 30 % et 70 %; la séquence cible prévue pour ne pas former de fortes structures en épingle à cheveux de l’ARN; et la séquence cible contenant une structure secondaire ou tertiaire minimale prédite de l’ARN5.
REMARQUE: Cette étude a conçu chaque ARNg comme 4 séquences en tandem de 30 nucléotides de long, espacées de 36 répétitions directes de nucléotides et avec un terminateur de 7 thymines aux deux extrémités5. Pour le gène cible exogène, GFP, les mêmes critères que ci-dessus ont été suivis avec l’ajout d’un fragment OpIE2-GFP5. - Amplifier la séquence promotrice U6:3 à l’aide de la PCR avec les amorces 1043.C1 et 1043.C23 et le plasmide Addgene #112688 (Tableau 1)26. Purifiez en gel les fragments amplifiés U6:3 à l’aide d’un kit de purification en gel.
- Digest Addgene plasmid #112688 avec l’enzyme de restriction AscI et XbaI24. Dans les produits résultants, utilisez un kit pour purifier au gel les plus gros fragments, appelé l’épine dorsale du vecteur pré-base.
- Assemblez le vecteur de base avec l’épine dorsale du vecteur pré-base et le fragment U6:3 à l’aide de la méthode d’assemblage Gibson25. Le vecteur de base ci-après est nommé OA-1043.
REMARQUE: L’ID Addgene du plasmide OA-1043 est #164586. - Synthétisez le fragment d’ARNm du gène cible à l’aide d’un service externe de synthèse de gènes.
- Digérer le vecteur de base OA-1043 avec l’enzyme de restriction PstI et NotI24. Conservez tout le produit de digestion, appelé OA-1043 digéré.
- Assemblez le vecteur d’expression de l’ARNg avec l’OA-1043 digéré et le fragment d’ARNg du gène cible à l’aide de la méthode d’assemblage de Gibson25.
NOTE: Quatre gènes cibles ont été étudiés: trois étaient endogènes (blanc, Notch, jaune), un était exogène (GFP). Leurs ID Addgene sont : #132420 (gRNAw), #132421 (gRNAN), #132425 (gRNAy) et #133304 (gRNAGFP).
- Concevoir chaque fragment d’ARNg en fonction des critères suivants : séquence cible de 30 nucléotides de longueur; la longueur maximale des étirements en poly-U dans la séquence cible étant de 4 paires de bases; la teneur en GC de la séquence cible étant comprise entre 30 % et 70 %; la séquence cible prévue pour ne pas former de fortes structures en épingle à cheveux de l’ARN; et la séquence cible contenant une structure secondaire ou tertiaire minimale prédite de l’ARN5.
- Génération de mouches transgéniques
- Injecter des vecteurs d’expression dans des embryons de mouches à l’aide d’un service externe d’injection d’embryons de mouches et d’embryons de mouches contenant des sites d’intégration ØC31. Élever les embryons injectés à 26 °C.
REMARQUE: La ligne attp40w (avec des sites d’intégration sur le 2ème chromosome) a été utilisée pour générer des lignes CasRx et la ligne 8622 (avec des sites d’intégration sur le 3ème chromosome) a été utilisée pour générer diverses lignées d’ARNg. - Gardez les mouches soit comme des lignes homozygotes, soit comme des lignes hétérozygotes équilibrées.
REMARQUE: Les mouches Ubiq-CasRx et Ubiq-dCasRx ont été conservées en tant que lignes équilibrées hétérozygotes avec le CyO comme équilibreur. De plus, les vecteurs Ubiq-CasRx et Ubiq-dCasRx contiennent un marqueur dsRed. En conséquence, les mouches Ubiq-CasRx et Ubiq-dCasRx ont les trois phénotypes suivants: dsRed-positif, ailes bouclées et yeux blancs. Les mouches exprimant l’ARNg ont été conservées comme des lignées homozygotes. Leurs numéros de stock de mouches du Bloomington Drosophila Stock Center (BDSC) sont les suivants : #84118 (Ubiq-CasRx), #84119 (Ubiq-dCasRx), #84124 (gRNAw), #84122 (gRNAN), #84123 (gRNAy), #84986 (gRNAGFP).
- Injecter des vecteurs d’expression dans des embryons de mouches à l’aide d’un service externe d’injection d’embryons de mouches et d’embryons de mouches contenant des sites d’intégration ØC31. Élever les embryons injectés à 26 °C.
- Génétique des mouches (Figure 1A)
- Recueillez 10 mouches femelles adultes vierges de la lignée homozygote de l’ARNg et recueillez 5 mouches mâles adultes de la lignée hétérozygote équilibrée Ubiq-CasRx/CyO. Mettez les mouches femelles et mâles collectées, appelées mouches parentales, dans un flacon complété par de la levure sèche en poudre (Figure 1A).
- Répétez l’étape précédente 3 fois pour générer 3 réplications. Pour le groupe témoin, utilisez la ligne Ubiq-dCasRx/CyO tout en gardant tout le reste identique.
REMARQUE: Pour un flacon en verre ordinaire de nourriture, 0,1 g de levure sèche en poudre est suffisant. La recette de la nourriture à la mouche de la BDSC est utilisée. - Élevez les flacons contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Conservez ensuite les flacons à 26 °C pendant au moins 20 jours.
- Observez les flacons tous les jours pour voir si de nouvelles progénitures adultes ont émergé de pupes de génération F1. Si c’est le cas, anesthésiez-les avec du dioxyde de carbone en insérant un tube relié à un réservoir de dioxyde de carbone à l’intérieur des flacons de mouche, puis allumez l’interrupteur de débit pendant 10 secondes.
- Une fois que les mouches deviennent immobiles, videz-les du flacon sur un fly-pad, qui est également connecté au réservoir de dioxyde de carbone et le dioxyde de carbone s’écoule continuellement à travers le fly pad.
- Notez le phénotype des mouches anesthésiées et imagez-les à l’aide d’une caméra couleur connectée à un stéréomicroscope fluorescent. Comptez le nombre de progénitures avec différents phénotypes. Utilisez un logiciel de traitement d’image pour le post-traitement et la compilation d’images (Figure 2A - 2D).
NOTE: D’après la génétique mendélienne, deux types de mouches sont attendus parmi les progénitures pour chaque croisement (Figure 1A).
- Séquençage de l’ARN (Graphique 2E – 2G)
- Prélèvement d’échantillons
REMARQUE: Choisissez une méthode de prélèvement d’échantillons appropriée parmi les 3 exemples ci-dessous; 3 répétitions pour chaque type d’échantillon distinct sont requises.- Prélèvement d’échantillons de tête de mouche adulte
- Recueillez 10 mouches femelles adultes vierges de la lignée d’ARNg homozygote. Recueillez 5 mouches mâles adultes de la lignée hétérozygote équilibrée Ubiq-CasRx/CyO. Mettez les mouches femelles et mâles collectées, qui sont les mouches parentales, dans un flacon complété par de la poudre de levure sèche.
- Répétez l’étape précédente 3 fois pour 3 réplications. Pour le groupe témoin, utilisez la ligne Ubiq-dCasRx/CyO tout en gardant tout le reste identique.
- Élevez les flacons contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Ensuite, maintenez les flacons à 26 °C jusqu’à ce que des progénitures émergent des nymphes.
- Recueillez 10 mouches adultes âgées de 1 jour avec le phénotype correct. Anesthésiez les mouches avec du dioxyde de carbone, puis coupez la tête de la mouche et placez les têtes dans un tube de centrifugeuse de 1,5 mL sur de la glace carbonique. Conserver le tube de centrifugeuse à -80 °C. Répétez cette étape 3 fois pour 3 réplications.
- Prélèvement d’échantillons d’embryons âgés de 17 à 20 heures
- Recueillir 8 à 10 mouches femelles adultes vierges de la lignée homozygote de l’ARNg. Recueillez 4 à 5 mouches mâles adultes de la lignée hétérozygote équilibrée Ubiq-CasRx/CyO. Mettez les mouches femelles et mâles collectées, qui sont les mouches parentales, dans un flacon complété par de la poudre de levure sèche.
- Répétez l’étape précédente 3 fois pour 3 réplications. Pour le groupe témoin, utilisez la ligne Ubiq-dCasRx/CyO tout en gardant tout le reste identique.
- Élevez les flacons contenant les mouches parentales à 26 °C pendant 48 heures.
- Préparez une chambre de collecte d’embryons de jus de raisin pour chaque répétition suivant cette recette: 376 mL d’eau, 126 mL de jus de raisin, 15 g de gélose et 6 g de saccharose. Mettez le support dans un bécher de 1 L et mettez-le au micro-ondes pendant 5 à 6 minutes tout en gardant un œil attentif sur le support dans le bécher pour vérifier si des bulles / mousse apparaissent. Si c’est le cas, arrêtez le micro-ondes et laissez la bulle/mousse se déposer. Continuez à micro-ondes de cette façon jusqu’à ce que la bulle devienne claire. Ne tourbillonnez pas tant que toutes les bulles ne sont pas claires. Enfin, ajoutez 10 mL d’alcool à 100 % et 5 mL d’acide acétique. Bien mélanger, puis pipeter le milieu dans des boîtes de Petri de 35 mm avec une pipette sérologique de 25 mL. Lorsque le support se solidifie dans la boîte de Pétri, il est prêt à l’emploi.
- À la fin de l’incubation de 48 heures, transférer les mouches parentales dans les chambres de collecte d’embryons de jus de raisin et les incuber à 26 °C pendant 3 h. Retirez ensuite les mouches adultes tout en conservant les embryons fraîchement pondus sur les assiettes de jus de raisin pendant encore 17 h à 26 °C.
- Après l’incubation, prélever les 50 à 100 embryons dans les assiettes de jus de raisin, nettoyer la surface de l’embryon en les immergeant dans de l’eau désionisée, puis les transférer dans un tube de centrifugeuse de 1,5 mL sur de la glace. Conservez-les à -80 °C. Répétez cette étape 3 fois pour 3 réplications.
- Premier prélèvement d’échantillons de larves d’instar
- Recueillir 8 à 10 mouches femelles adultes vierges de la lignée homozygote de l’ARNg. Recueillez 4 à 5 mouches mâles adultes de la lignée hétérozygote équilibrée Ubiq-CasRx/CyO. Mettez les mouches femelles et mâles collectées, qui sont les mouches parentales, dans un flacon complété par de la poudre de levure sèche. Répétez cette étape 3 fois pour 3 réplications. Pour le groupe témoin, utilisez la ligne Ubiq-dCasRx/CyO tout en gardant tout le reste identique.
- Élevez les flacons contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, transférez les mouches adultes dans un autre nouveau flacon de nourriture ordinaire pour une incubation de nuit (16 h) à 26 ° C. Ensuite, retirez les mouches adultes.
- Conserver le flacon contenant l’embryon à 26 °C pendant 24 h, puis noter les larves transhétérozygotes du premier stade au microscope à l’aide de marqueurs distincts basés. Recueillir 15 à 30 larves avec des phénotypes corrects et les mettre dans un tube de centrifugeuse de 1,5 mL et les stocker à -80 ° C. Répétez cette étape 3 fois pour 3 réplications.
- Prélèvement d’échantillons de tête de mouche adulte
- Séquençage
- Extraction d’ARN : utilisez un kit d’extraction d’ARN disponible dans le commerce et suivez les instructions du kit pour traiter tous les échantillons. Ensuite, incubez les échantillons d’ARN extraits avec de la désoxyribonucléase disponible dans le commerce et suivez ses instructions pour éliminer tout ADN contaminant des échantillons.
- Mesurer la concentration d’ARN à l’aide d’un spectrophotomètre UV-vis disponible dans le commerce. Mesurez l’intégrité de l’ARN dans les échantillons à l’aide d’un test d’intégrité de l’ARN disponible dans le commerce.
- Construisez les bibliothèques RNA-seq à l’aide du kit de préparation de bibliothèque d’ARN disponible dans le commerce.
- Utilisez le service de séquençage externe pour le séquençage de bibliothèque avec les paramètres suivants : mode de lecture unique ; longueur de lecture: 50nt, profondeur: 20 millions de lectures par bibliothèque. Effectuez des appels de base avec RTA 1.18.64, puis convertissez les données en FASTQ à l’aide de bcl2fastq 1.8.4.
REMARQUE: Les données brutes de séquençage peuvent être trouvées dans les archives de lecture du National Center for Biotechnology Information Sequencing (ID de soumission: SUB6818910 [BioProject: PRJNA600654]).
- Bioinformatique
- La carte lit à partir des données de séquençage le génome de Drosophila melanogaster de la version 6 du Berkeley Drosophila Genome Project (numéro d’acquisition GenBank: GCA_000001215.4) et les séquences exogènes CasRx et GFP en utilisant le paramètre par défaut de STAR aligner28 avec l’ajout de l’option de filtre « -outFilterType BySJout » et « -sjdbGTFfile Drosophila_melanogaster. BDGP6.22.97.gtf » fichier de format de transfert de gènes de l’ENSEMBL.
- Déterminez le nombre brut de transcriptions pour chaque transcription annotée avec la fonction Counts35 à l’aide des options « -t exon -g gene_id -M -O --fraction ». Ensuite, normalisez le nombre de transcriptions brutes à l’aide du nombre total de transcriptions à l’aide du script Perl « addTpmFpkmToFeatureCounts.pl ».
- Utilisez la méthode du maximum a posteriori avec l’estimateur de retrait d’origine dans le pipeline DESeq2 pour estimer le changement de pli logarithmique (LFC) des transcriptions de chaque gène.
- Prélèvement d’échantillons
2. Ciblage in vivo de l’ARN exogène omniprésent à l’aide d’un système CasRx à trois composants
- Génération d’un vecteur d’expression ubiquitaire cible exogène
- La PCR amplifie le fragment promoteur Ubiq à l’aide des amorces 1052B. C1 et 1052B. C2 et le plasmide Addgene #112686 26. Ensuite, la PCR amplifie le fragment T2A-eGFP amplifié à partir du plasmide Addgene #112686 avec les amorces 908A.1 et 908A.2 (Tableau 1)26. Ensuite, la PCR amplifie le fragment promoteur Ubiq sous forme de séquence inversée à l’aide du plasmide Addgene #112686 avec les amorces 908A.3 et 908A.4 (Tableau 1)26. Purifiez en gel le fragment de promoteur Ubiq, le fragment T2A-eGFP et le fragment de promoteur Ubiq inversé à l’aide d’un kit de purification en gel.
- Commandez une séquence de codage de luciole luciférase personnalisée et un fragment personnalisé contenant un fragment p10 3'UTR, une renilla luciferase inversée suivie d’un fragment SV40 3'UTR.
- Digest Addgene plasmid #112688 avec l’enzyme de restriction AscI et XbaI24. Dans les produits résultants, purifiez les plus gros fragments à l’aide d’un kit de purification de gel, appelé colonne vertébrale du vecteur de base.
- Utilisez la méthode d’assemblage gibson pour assembler le vecteur de base avec l’épine dorsale du vecteur de base et les fragments suivants : le fragment promoteur Ubiq, le fragment T2A-eGFP, le fragment promoteur Ubiq inversé, la séquence codante luciférase luciole et la luciférase renilla inversée suivie d’un fragment SV40 3'UTR25.
REMARQUE: L’ID Addgene du vecteur d’expression de la double luciférase résultant (OA-1052B) est #132426.
- Génération d’un vecteur d’expression d’ARNg
- La PCR amplifie la séquence du promoteur U6:3 à l’aide des amorces 1043.C1 et 1043.C23 et du plasmide Addgene #112688 (Tableau 1)26. Purifiez en gel les fragments amplifiés U6:3 à l’aide d’un kit de purification en gel.
- Digest Addgene plasmid #112688 avec l’enzyme de restriction AscI et XbaI24. Dans les produits résultants, purifiez le gel des plus gros fragments, appelé l’épine dorsale du vecteur pré-base, à l’aide d’un kit de purification en gel.
- Assemblez le vecteur de base avec l’épine dorsale du vecteur pré-base et le fragment U6:3 à l’aide de la méthode d’assemblage Gibson25. Le vecteur de base ci-après est nommé OA-1043.
- Synthétisez le fragment d’ARNm du gène cible à l’aide d’un service externe de synthèse de gènes.
- Digérer le vecteur de base OA-1043 avec l’enzyme de restriction PstI et NotI24. Conservez l’ensemble du produit de digestion, appelé OA-1043 digéré.
- Assemblez le vecteur d’expression de l’ARNg avec l’OA-1043 digéré et le fragment d’ARNg du gène cible à l’aide de la méthode d’assemblage de Gibson25.
REMARQUE: L’ID Addgene du plasmide résultant (OA-1052K) est #132422.
- Génération de mouches transgéniques
- Injecter le vecteur OA-1052B dans des embryons de mouches à l’aide d’embryons de mouches contenant le site d’intégration ØC31 sur le 3e chromosome, numéro de stock de mouches BDSC 9744, via un service externe d’injection d’embryons de mouches. De même, injecter le vecteur OA-1052K dans des embryons de mouches en utilisant des embryons de mouches contenant le site d’intégration ØC31 sur le 3ème chromosome, numéro de stock de mouche BDSC 8622. Élever les embryons injectés à 26 °C.
- Gardez les mouches exprimant la double luciférase et les mouches de l’ARNg comme des lignes homozygotes; conserver les lignes Ubiq-CasRx en tant que lignes hétérozygotes à double équilibre en croisant la ligne hétérozygote Ubiq-CasRx à simple équilibre générée dans la section 1 aux lignes d’équilibre portant le chromosome d’équilibre TM6 avec marqueur de chaume (Stb) et ne conserver simultanément que les progénitures à double équilibre avec des ailes bouclées aux yeux blancs et des phénotypes fluorescents dsRed.
REMARQUE: Les numéros de stock de mouches BDSC sont: # 84127 (Ubiq-Fluc-Rluc), # 84125 (gRNAFluc).
- Génétique des mouches (Figure 1B et Figure 3A)
- Collectez 8 à 10 mouches femelles adultes vierges de la ligne exprimant la double luciférase. Recueillir 4 à 5 mouches mâles adultes de l’hétérozygote équilibré Ubiq-CasRx/CyO; +/TM6, ligne Stb qui montre simultanément des ailes bouclées aux yeux blancs et une fluorescence dsRed. Mettez les mouches femelles et mâles collectées, qui sont les mouches parentales, dans un flacon complété par de la poudre de levure sèche (ci-après appelée Étape 1 Cross).
- Répétez l’étape précédente 3 fois pour 3 réplications. Pour le groupe témoin, utilisez l’Ubiq-dCasRx/CyO; +/TM6, ligne Stb tout en gardant tout le reste identique.
- Remontez les flacons Step 1 Cross contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Conservez ensuite les flacons à 26 °C pendant au moins 14 jours. Pendant ce temps, collectez 8 à 10 vierges femelles de la lignée d’ARNg ciblant la luciférase homozygote. Répétez cette étape 3 fois pour 3 réplications.
- Observez les flacons de l’étape 1 Cross tous les jours pour voir si une nouvelle mouche adulte émerge des nymphes. Si c’est le cas, anesthésiez-les avec du dioxyde de carbone, collectez 5 mouches mâles exprimant à la fois l’Ubiq-CasRx (ou Ubiq-dCasRx) et le rapporteur de la double luciférase des progénitures et mettez-les dans un nouveau flacon avec 10 femelles vierges de la ligne d’ARNg ciblant la luciférase (ci-après appelée Étape 2 Cross). Répétez cette étape 3 fois pour 3 réplications.
- Récupérez 5 autres mâles d’un jour exprimant à la fois l’Ubiq-CasRx (ou Ubiq-dCasRx) et le rapporteur à double luciférase des flacons Step 1 Cross et incurez-les pendant 2 à 4 jours à 26 °C. Ensuite, transférez-les dans un tube de centrifugeuse de 1,5 mL et stockez-les à -80 °C. Répétez cette étape 3 fois pour trois réplications.
- Remontez les flacons Step 2 Cross contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Conservez ensuite les flacons à 26 °C pendant au moins 20 jours.
- Observez les flacons de l’étape 2 Cross tous les jours pour voir si de nouvelles progénitures adultes ont émergé des nymphes. Si c’est le cas, anesthésiez-les avec du dioxyde de carbone, notez les phénotypes des mouches anesthésiées et imagez-les à l’aide d’une caméra couleur équipée d’un stéréomicroscope fluorescent. Comptez le nombre de progénitures avec différents phénotypes. Utilisez un logiciel de traitement d’image pour le post-traitement et la compilation d’images (Figure 3B – 3C).
NOTE: La génétique mendélienne suggère que, si les mouches sont toutes viables, 4 types de mouches sont attendus parmi les descendants de l’étape 2 Cross, chacun représentant 25% de la population (Figure 1B et Figure 3A).
- Dosage de la luciférase (Figure 3D)
- Générez les triples mouches transhétérozygotes ainsi que les mouches de contrôle en répétant les étapes 2.4.1 à 2.4.5. Recueillez les mouches mâles à la naissance et vieillissez-les jusqu’à l’âge de 3 jours.
- Transférez les mouches âgées de 3 jours dans des tubes centrifuges de 1,5 mL et lysez-les à l’aide d’un pilon et du tampon de lyse de la luciférase du kit de dosage de la luciférase disponible dans le commerce.
- Utilisez 5 μL de tissu lysé de chaque échantillon pour mesurer l’activité de la luciole et de la renilla luciferase à l’aide d’un kit de dosage de la luciférase et d’un luminomètre disponibles dans le commerce.
3. Ciblage de l’ARN in vivo spécifique aux tissus à l’aide d’un système CasRx à trois composants
- Génération du vecteur d’expression UASt-CasRx et UASt-dCasRx
- La PCR amplifie la séquence du promoteur UASt à l’aide du plasmide pJFRC81 et des amorces 1041.C9 et 1041.C11; ensuite, la PCR amplifie le fragment CasRx en utilisant le plasmide OA-1050E (Addgene ID #132416) et les amorces 1050L. C1 et 1050E. C4; puis la PCR amplifie les fragments dCasRx en utilisant le plasmide OA-1050R (Addgene ID #132417) et les amorces 1050L. C1 et 1050E. C4 (tableau 1)26. Purifiez en gel la séquence de promoteur UASt amplifiée, les fragments CasRx et dCasRx à l’aide du kit de purification en gel.
- Digeste vecteur de base (plasmide Addgene #112686) avec les enzymes de restriction NotI et PacI24. Dans les produits résultants, purifiez le plus gros fragment, appelé colonne vertébrale du vecteur de base, à l’aide d’un kit de purification en gel.
- Assemblez le vecteur UASt-CasRx avec l’épine dorsale du vecteur de base, la séquence de promoteurs UASt et le fragment CasRx à l’aide de l’assemblage Gibson ; puis assemblez le vecteur UASt-dCasRx avec l’épine dorsale du vecteur de base, la séquence de promoteur UASt et le fragment dCasRx à l’aide de la méthode d’assemblage Gibson25.
REMARQUE: Le vecteur UASt-CasRx est le plasmide Addgene #132418, et le vecteur UASt-dCasRx est le plasmide Addgene #132419
- Génération de mouches transgéniques
- Injecter le vecteur UASt-CasRx dans des embryons de mouches à l’aide d’un service d’injection d’embryons de mouches et d’embryons de mouches du site d’intégration ØC31 8621 sur leurs 2èmes chromosomes; puis injecter le vecteur UASt-dCasRx dans des embryons de mouches en utilisant le service d’injection d’embryons de mouches et des embryons de mouches du site d’intégration ØC31 8621 sur leurs 2èmes chromosomes. Élever les embryons injectés à 26 °C.
- Gardez les mouches comme des lignes hétérozygotes à double équilibre avec des marqueurs CyO et Sb. REMARQUE: Les ID des lignes de mouche dans BDSC sont 84121 (UASt-CasRx) et 84120 (UASt-dCasRx).
- Génétique des mouches (Figure 1C)
- Commandez les lignes GAL4 souhaitées auprès de BDSC; Obtenir les lignées d’ARNg pertinentes à partir de l’étape 3.2.2 (ou de BDSC).
REMARQUE : Les 2 mouches GAL4 suivantes de BDSC ont été utilisées : GAL4-GMR (ID BDSC : #29967), GAL4-y (ID BDSC : #44373). Les mêmes 3 lignées d’ARNg générées dans la première section ont été utilisées : gRNAw (ID BDSC : #84124), gRNAN (ID BDSC #84122), gRNAy (ID BDSC : #84123). - Recueillez 5 à 10 mouches femelles adultes vierges de la lignée d’ARNg. Recueillir 2 à 4 mouches mâles adultes de l’UASt-CasRx/CyO hétérozygote double équilibré; +/TM6, ligne Sb qui montrent simultanément des ailes bouclées aux yeux blancs et une fluorescence dsRed. Mettez les mouches femelles et mâles collectées, qui sont les mouches parentales, dans un flacon de nourriture ordinaire (ci-après appelé Étape 1 Croix). Répétez cette étape 3 fois pour 3 réplications. Pour le groupe témoin, utilisez l’UASt-dCasRx/CyO; +/TM6, ligne Sb tout en gardant tout le reste identique.
- Remontez les flacons Step 1 Cross contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Conservez ensuite les flacons à 26 °C pendant au moins 14 jours. Pendant ce temps, collectez 5 à 10 vierges femelles de la ligne GAL4. Répétez cette étape 3 fois pour 3 réplications.
- Observez les flacons de l’étape 1 Cross tous les jours pour voir si une nouvelle mouche adulte émerge des nymphes. Si c’est le cas, anesthésiez-les avec du dioxyde de carbone, collectez 2 à 4 mouches mâles exprimant à la fois l’UASt-CasRx (ou UASt-dCasRx) et le vecteur aRNg des progénitures qui ont simultanément des phénotypes fluorescents et chaumes. Mettez les mâles collectés de l’étape 1 Cross dans un nouveau flacon avec 5-10 femelles vierges collectées de la lignée GAL4 (ci-après appelée Step 2 Cross). Répétez cette étape 3 fois pour 3 réplications.
- Remontez les flacons Step 2 Cross contenant les mouches parentales à 26 °C pendant 48 heures. Ensuite, retirez toutes les mouches parentales de chaque flacon. Conservez ensuite les flacons à 26 °C pendant au moins 20 jours.
- Observez les flacons de l’étape 2 Cross tous les jours pour voir si de nouvelles mouches adultes émettent des nymphes. Si c’est le cas, anesthésiez-les avec du dioxyde de carbone, notez les phénotypes des mouches anesthésiées et imagez-les à l’aide d’une caméra couleur équipée d’un stéréomicroscope fluorescent. Comptez le nombre de progénitures avec différents phénotypes. Utilisez un logiciel de traitement d’image pour le post-traitement et la compilation d’images (Figure 4).
NOTE: La génétique mendélienne suggère que, si les mouches sont toutes viables, 4 types de mouches sont attendus parmi les descendants de l’étape 2 Cross, chacun représentant 25% de la population (Figure 1C).
- Commandez les lignes GAL4 souhaitées auprès de BDSC; Obtenir les lignées d’ARNg pertinentes à partir de l’étape 3.2.2 (ou de BDSC).
Résultats
Ciblage in vivo de l’ARN in vivo omniprésent à l’aide d’un système CasRx à deux composants
Les mouches transhétérozygotes F1 exprimant à la fois l’Ubiq-CasRx et l’ARNg (ciblant à la fois les gènes endogènes et exogènes) ont montré des phénotypes marqués par rapport aux mouches témoins exprimant les constructions Ubiq-dCasRx et gRNA (Figure 2 et Figure 4). Plus précisément, les mouches CasRx transhétérozygotes ont des niveaux de survie significativement plus faibles que les mouches transhétérozgyes dCasRx, ce qui indique la toxicité du système Ubiq-CasRx (Figure 2A et Figure 4A). Il convient de noter que les mouches CasRx transhétérozygotes et dCasRx ont un taux d’hérédité inférieur à 50%, ce qui est le rapport attendu basé sur la génétique mendélienne. Parmi les trois gènes cibles, l’Ubiq-CasRx/+; Mouches U6-gRNAN/+ et Ubiq-CasRx/+; Les mouches U6-gRNAy/+ ne sont pas viables (hérédité de 0 %) et n’ont pas grandi au-delà du stade larvaire du deuxième stade (figure 2A-2B). L’Ubiq-CasRx/+ survivant; Les mouches U6-gRNAw/+, dont l’hérédité était de 12,9 %, présentaient un phénotype distinct aux yeux blancs entièrement pénétrant (figure 2B). En plus des traits observables associés à CasRx, nous avons pu confirmer une réduction significative des transcriptions des gènes cibles pour 3 gènes cibles : Notch, jaune et GFP (Figure 2E-2G). Une réduction des transcriptions des gènes blancs a été observée chez les mouches Ubiq-CasRx/+, U6-gRNAw/+, par rapport aux mouches témoins Ubiq-dCasRx/+, U6-gRNAw/+, bien que la réduction n’ait pas été statistiquement significative (Figure 2E - 2F). Des preuves d’activité hors cible induites par CasRx ont été trouvées en comparant les transcriptions exprimées différemment entre des échantillons de mouches exprimant CasRx et des échantillons de mouches exprimant dCasRx (Figure 2E, 2G). Le nombre de transcriptions non ciblées exprimé de manière significativement différentielle est le suivant : blanc, 253 (1,4 % du total des transcriptions); Notch, 300 (1,7 %); jaune, 41 (0,23 %); GFP, 5 880 (33 %) (Figure2G) Sur un total de 17 779 transcriptions différentes, 6 transcriptions non ciblées ont été exprimées de manière significativement différentielle dans les 4 groupes d’échantillons. L’un des 6 transcrits identifiés était Gadd45, un gène impliqué dans l’apoptose et l’arrêt cellulaire chez les mouches, ce qui soulève la possibilité que l’action enzymatique de CasRx puisse soit déclencher directement l’apoptose cellulaire, soit déclencher indirectement une mauvaise expression d’autres gènes, ce qui à son tour conduit à l’apoptose. Enfin, il convient de noter que les mouches Ubiq-CasRx et Ubiq-dCasRx n’ont pas été établies comme des stocks homozygotes, probablement en raison de la toxicité conférée par une expression ubiquitaire élevée. En conséquence, les mouches hétérozygotes Ubiq-CasRx/CyO et Ubiq-dCasRx/CyO ont été utilisées pour le croisement avec les lignées de mouches homozygotes de l’ARNg. En somme, le système Ubiq-CasRx à deux composants est capable d’atteindre un ciblage d’ARN omniprésent pour des cibles endogènes et exogènes, ce qui entraîne des phénotypes observables et une réduction des transcriptions. Ces résultats ont également montré que le ciblage de l’ARN médié par CasRx peut introduire une toxicité in vivo.
Ciblage in vivo de l’ARN exogène omniprésent à l’aide d’un système CasRx à trois composants
Les résultats du croisement en deux étapes ont montré que, malgré la nature exogène du gène cible (c.-à-d. Fluc), l’expression des trois transgènes dans les triples transhétérosygotes F2 (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) a entraîné une létalité de 100 % par rapport aux croisements de contrôle impliquant Ubiq-dCasRx, où aucune létalité n’a été observée chez les transhétérozygotes triples F2 (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) (Figure 3B-C ). Plus précisément, seule la combinaison des trois transgènes (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) a entraîné une létalité de 100 % (Figure 3B et D), tandis que (Ubiq-CasRx/+; gRNAFluc/TM6) et (Ubiq-CasRx/+; Les génotypes Ubiq-Fluc-Ubiq-Rluc/TM6) étaient viables et manquaient de phénotypes avec des taux d’hérédité correspondant aux taux de transmission mendéliens attendus, ce qui suggère que la disponibilité de la séquence cible (c’est-à-dire la luciole luciférase) en combinaison avec Ubiq-CasRx/+ et le gRNAFluc est ce qui a entraîné les phénotypes de létalité observés, probablement issus de l’activité collatérale des enzymes Cas132,8 . De plus, aucun phénotype distinguable ou influence dramatique sur l’hérédité chez les transhétérosygotes F1 (Ubiq-CasRx/+; gRNAFluc/+ ou Ubiq-CasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) ont été observés par rapport aux témoins Ubiq-dCasRx (Ubiq-dCasRx/+; gRNAFluc/+ ou Ubiq-dCasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) (Figure 3B), indiquant qu’une enzyme catalytiquement active est essentielle pour obtenir les phénotypes de létalité observés. De plus, les niveaux d’expression de Fluc et de Rluc chez les mouches de tous les génotypes viables n’ont pas montré de réduction significative de l’expression de Fluc chez les transhétérosygotes triples Ubiq-dCasRx (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) par rapport aux témoins rapporteurs de luciférase double. Cela suggère que les niveaux d’expression de la protéine Fluc n’ont pas été réduits par le ciblage dCasRx (Figure 3D). Pris ensemble, le phénotype de létalité commun dans les deux différentes expériences de ciblage de l’ARN omniprésent médié par CasRx indique que lorsqu’il est utilisé sur des tissus de manière omniprésente, le ciblage de l’ARN médié par CasRx peut être toxique pour l’organisme.
Ciblage de l’ARN in vivo spécifique aux tissus à l’aide d’un système CasRx à trois composants
Le niveau élevé de toxicité observé dans les expériences de ciblage de l’ARN omniprésent nous a incités à explorer le ciblage de l’ARN spécifique aux tissus à l’aide d’une conception de système CasRx à trois composants détaillée dans la section des méthodes. En effet, le niveau de toxicité observé a été réduit lorsque l’expression globale de CasRx a été abaissée à l’aide du promoteur UASt par rapport à celle du promoteur Ubiq, ceci est illustré par trois aspects: 1) les lignes UASt-CasRx et UASt-dCasRx ont été maintenues en tant que lignes homozygotes, bien que sur la base du schéma croisé en deux étapes, les lignes UASt-CasRx et UASt-dCasRx ont été utilisées pour effectuer les croisements, 2) tous les taux d’hérédité triple transhétérozygote de génération F2 dCasRx correspondaient au taux d’hérédité mendélien attendu de 25 %, et 3) le phénotype de létalité triple transhétérozygote CasRx de génération F2 a été modérément réduit. Dans l’expérience de ciblage blanc, sur les taux d’hérédité mendélienne de 25 % attendus chez les triples transhétérozygotes F2, seulement 0,57 % des mouches adultes viables (UASt-CasRx/+; gRNAw/GMR-Gal4) ont été observées, qui présentaient toutes de graves phénotypes de pigmentation et de morphologie spécifiques à l’œil (figure 4A et 4B). Pour le croisement de ciblage blanc, le taux d’hérédité F2 triple transhétérozygote exprimant CasRx était significativement inférieur à celui du groupe témoin triple transhétérozygote exprimant dCasRx (27,6 %) (figure 4A). Dans l’expérience de ciblage Notch, le triple transhétérozygote exprimant CasRx portant les trois transgènes était mortel à 100%, tandis que le taux d’héritage du contrôle dCasRx était de 29,3% (Figure 4A). Dans l’expérience de ciblage jaune, F2 triple transhétérozygote exprimant CasRx, arnay et y-GAL4 ont montré une réduction marginale du pigment de chitine sous forme de petites plaques de cuticule jaune sur le thorax et l’abdomen avec un taux d’hérédité de 2,67%, beaucoup plus faible que celui du groupe témoin dCasRx (25,2%) (Figure4A ). Toutes les mouches transhétérozygotes triples témoins dCasRx ne présentaient pas de phénotypes évidents comme les mouches exprimant CasRx, ce qui indique que l’activité catalytique de CasRx a contribué aux phénotypes observés. Le faible taux d’hérédité dans le groupe triple transhétérozygote CasRx suggère que deux sources de toxicité existent dans le ciblage de l’ARN CasRx : l’une est associée à une expression élevée de CasRx, dont la toxicité a été réduite par une expression restrictive de CasRx, l’autre est associée à l’activité collatérale. Pris ensemble, ces résultats ont montré que le système CasRx peut atteindre un ciblage in vivo de l’ARN spécifique aux tissus en tirant parti du système classique Gal4 / UASt et, dans l’intervalle, réduire la toxicité. Cependant, la toxicité et les phénotypes de létalité occasionnelle ont encore été observés à un niveau de gravité inférieur à celui des approches omniprésentes, ce qui indique que l’activité de clivage collatéral est associée à la toxicité.
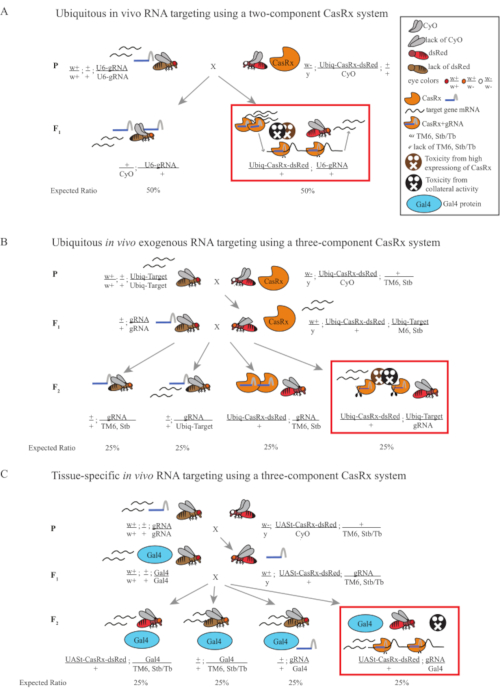
Figure 1 : Aperçu général du ciblage de l’ARN à l’aide d’un système Cas13D. (A) Schémas du croisement génétique en une étape dans le ciblage in vivo omniprésent de l’ARN à l’aide d’un système CasRx à deux composants. (B) Schémas d’un croisement génétique en deux étapes dans l’ARN exogène in vivo omniprésent ciblant à l’aide du système CasRx à trois composants. (C) Schémas d’un croisement génétique en deux étapes dans le ciblage in vivo de l’ARN in vivo spécifique au tissu à l’aide d’un système CasRx à trois composants. Veuillez cliquer ici pour voir une version agrandie de cette figure.
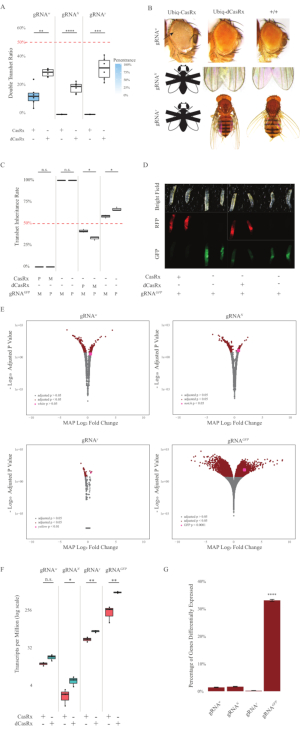
Figure 2 : Ciblage in vivo de l’ARN in vivo omniprésent à l’aide d’un système CasRx à deux composants (réimprimé5). (A) Pourcentages d’hérédité totale des mouches transhétérozygotes héritant d’Ubiq-CasRx (ou Ubiq-dCasRx) et d’ARNg. L’ombrage bleu dans le diagramme de la boîte indique la pénétrance du phénotype. (B) Phénotypes des mouches transhétérozygotes. Les flèches indiquent une nécrose tissulaire dans l’œil. La mouche noire et blanche marquée de « X » représente la létalité. (C) Pourcentages totaux d’hérédité des mouches transhétérozygotes de croisements bidirectionnels entre les mouches Ubiq-CasRx (ou Ubiq-dCasRx) et gRNAGFP-OpIE2-GFP. M, héritage maternel de CasRx; P, héritage paternel de CasRx. (D) Les larves F1 se reproduisent dans le croisement paternel. (E) Estimations a posteriori maximales des transcriptions pour le changement de pli logarithmique. Le pipeline DESeq2 a été utilisé. (F) Transcriptions par million (TPM) ciblées avec CasRx ou dCasRx. (G) Pourcentage de transcription exprimé différemment par CasRx dans les transcriptions. Veuillez cliquer ici pour voir une version agrandie de cette figure.
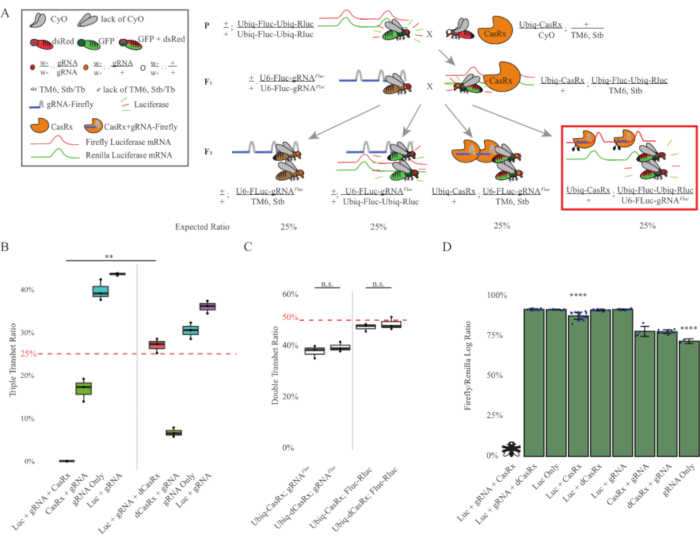
Figure 3 : Ciblage in vivo de l’ARN exogène omniprésent à l’aide d’un système CasRx à trois composants. (A) Schémas du croisement génétique en deux étapes. (B) Pourcentages totaux d’hérédité pour tous les génotypes émergeant dans la génération F2. L’héritage des trois transgènes (Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc et gRNAFLuc) dans la descendance F2 a entraîné une létalité de 100% et était significativement plus faible par rapport au groupe témoin Ubiq-dCasRx triple transhétérosygotes (p = 0,001, test t). (C) Le port d’Ubiq-CasRx/gRNAFluc seul ou d’Ubiq-CasRx et d’Ubiq-Fluc-Ubiq-Rluc seul n’a pas entraîné de létalité sévère, et les rapports héréditaires entre les transhétérosygotes Ubiq-CasRx et Ubiq-dCasRx n’étaient pas significativement différents (p = 0,41 et p = 0,51, respectivement, test t). (D) Rapports de luciférase normalisant les lectures de Fluc à lectures de Rluc. Les mouches transhétérozygotes triples exprimant Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc, gRNAFLuc étaient létales embryonnaires, ce qui était représenté par une mouche avec un « X », et par conséquent l’expression de la luciférase n’a pas été mesurée. Le rapport Fluc/Rluc des transhétérosygotes Ubiq-CasRx/+, Ubiq-Fluc-Ubiq-Rluc/TM6, Stb était significativement inférieur à celui des autres groupes exprimant Ubiq-Fluc-Ubiq-Rluc (p = 1,2e-06 ou moins, test t). Les résultats du groupe gRNAFLuc uniquement étaient significativement inférieurs à ceux de tous les autres groupes (p = 1,2e-06 ou moins, test t). Veuillez cliquer ici pour voir une version agrandie de cette figure.
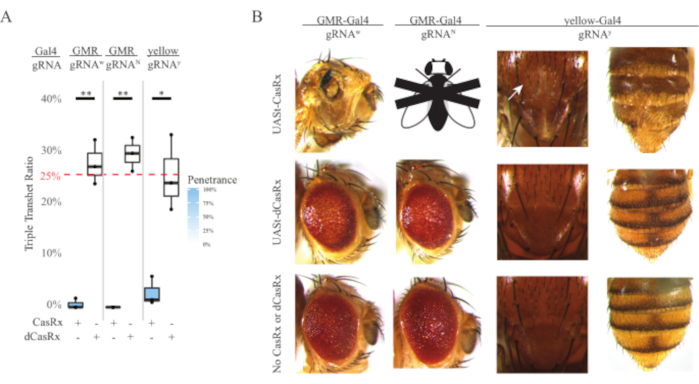
Figure 4 : Ciblage in vivo de l’ARN spécifique aux tissus à l’aide d’un système CasRx à trois composants (réimprimé5). (A) Pourcentage d’hérédité totale des mouches transhétérozygotes triples porteuses de trois transgènes (UASt-CasRx ou UASt-dCasRx, ARNg et Gal4-driver. (B) Phénotypes des mouches transhétérozygotes triples. La flèche blanche indique une réduction du pigment de chitine dans le thorax. La mouche noire et blanche marquée de « X » représente la létalité. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Construire | Description | Amorce | Séquence d’amorce (5' à 3') | Modèle pcr |
| OA-1050E | CasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA L’AGAA | pNLS-RfxCas13d-NLS-HA (pCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTGG L' | |||
| OA-1050R | dCasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA L’AGAA | pNLS-dRfxCas13d-NLS-HA (pdCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTG Le GAACA | |||
| OA-1050L | Promoteur UASt | 1041.C9 | GCGGGTTCTCGA CGGTCACGGCGG GCATGTCGACGC GGCCGCAACCAA CAACACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCACC TTTCTCTTCTTCT TGGGGCTCATGT TTAAACCCAATT CCCTATTCAGA | |||
| CasRx | 1050L. C1 | AATACAAGAAGA GAACTCTGAATA GGGAATTGGGT TTAAACATGAGC CCCAAGAAGAA | pCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTA GCTAGCTTAAG CGTAATCTGGA L' | |||
| OA-1050S | Promoteur UASt | 1041.C9 | GCGGGTTCTC GACGGTCACG GCGGGCATGT CGACGCGGCC GCAACCAACAA CACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCA CCTTTCTCTTC TTCTTGGGGCT CATGTTTAAAC CCAATTCCCTA TTCAGA | |||
| dCasRx | 1050L. C1 | AATACAAGAAG AGAACTCTGAAT AGGGAATTGGG TTTAAACATGAG CCCCAAGAAGAA | pdCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTAG CTAGCTTAAGCG TAATCTGGAACA | |||
| OA-1043 | Promoteur U6:3 | 1043.C1 | GGGAATTGGGA ATTGGGCAATAT TTAAATGGCGGC GCGCCGAATTCT TTTTTGCTCACCT | Plasmide addgène #164586 |
| 1043.C23 | ACACTAGTGGAT CTCTAGAGGTAC CGTTGCGGCCG CAAAAAAGTTGT AATAGCCCCTCA AAACTGGACCTT CCACAACTGCAG CCGACGTTAAAT La TGAAA | |||
| OA-1052B | Promoteur Ubiq | 1052B. C1 | GGGAATTGGGCA ATATTTAAATGGC GGCTGCAGCGC GCAGATCGCCGAT | Plasmide addgène #112686 |
| 1052B. C2 | TTTCTTTATGTTT TTGGCGTCTTCC ATCCTAGGTCTG CGGGTCAAAATA GAGATG | |||
| T2A-eGFP | 908A1 | ATAAAGGCCAAG AAGGGCGGAAA GATCGCCGTGG AGGGCAGAGGA AGTCTTCTAACAT GC | Plasmide addgène #112686 | |
| 908A2 | TTGTTATTTTAAAA ACGATTCATTCTA GGCGATCGCTTA CTTGTACAGCTC GTCCATGCC | |||
| Promoteur Ubiq inversé | 908A3 | ACCGTGACCTAC ATCGTCGACACTA GTGGATCTCTAGA CGCGCAGATCGC Le | Plasmide addgène #112686 | |
| 908A4 | GGATCATAAACTT TCGAAGTCATGC GGCCGCTCTGCG GGTCAAAATAGAG ATGT |
Tableau 1 : Liste des constructions moléculaires et des amorces utilisées dans cette étude. Cette liste comprend toutes les constructions (à la fois l’ID et la description) et les amorces associées à chaque construction (à la fois l’ID et les séquences (5' à 3')) et les modèles utilisés.
Discussion
Avec trois conceptions d’application différentes du système CasRx, ce travail a démontré le ciblage in vivo de l’ARN programmable chez les mouches. Les différentes stratégies répondent à différents besoins du projet, tels que le ciblage des gènes endogènes par rapport aux gènes exogènes et le ciblage de l’ARN omniprésent par rapport à l’ARN spécifique aux tissus. Les effets du ciblage de l’ARN comprenaient des changements phénotypiques spécifiques au gène cible, une réduction du transcript de l’ARN cible et des phénotypes de létalité occasionnels associés à une expression élevée de la protéine CasRx et à une activité collatérale. Dans l’ensemble, ces résultats ont montré que le système CasRx est capable de cibler la réduction des transcriptions d’ARN au niveau de l’organisme de manière programmable et efficace.
L’un des facteurs clés de la personnalisation réussie du système CasRx est la conception des ARNg. Plus précisément, les conseils suivants doivent être pris en compte: la séquence cible est d’environ 30 nucléotides de longueur, la longueur des étirements poly-U dans la séquence cible est de 4 paires de bases ou moins, la teneur en GC de la séquence cible est comprise entre 30% et 70%, la séquence cible ne devrait pas former de fortes structures en épingle à cheveux d’ARN et la séquence cible contient une structure secondaire ou tertiaire minimale prédite5.
En plus des conceptions d’ARNg, l’étape de la génétique des mouches dans chaque protocole est également essentielle à une mise en œuvre réussie. La présence ou l’absence des phénotypes définis transmis par les parents dans les progénitures est importante pour identifier et quantifier les phénotypes induits par le système CasRx dans les progénitures transhétérozygotes. En outre, la mise en place de croisements de contrôle à l’aide des mouches dCasRx en parallèle est également utile pour exclure les phénotypes non spécifiques dans les progénitures transhétérozygotes.
Il convient de noter que ces résultats ont révélé un problème de toxicité introduit par l’expression omniprésente de casrx et de protéines dCasRx chez la mouche, une limitation du système CasRx. L’expression omniprésente de CasRx ou dCasRx sous le promoteur Ubiq seul, sans aRNg, entraînait des coûts de remise en forme non triviaux, car ni les mouches Ubiq-CasRx ni Ubiq-dCasRx ne pouvaient être établies comme des lignées homozygotes. Au contraire, les mouches UASt-CasRx et UASt-dCasRx peuvent être établies comme des stocks homozygotes sains, bien qu’en raison de la conception du schéma croisé, elles aient été conservées en tant que stocks à double équilibre, un fait qui soutient l’existence d’une toxicité induite par l’expression omniprésente de la protéine CasRx. Une autre preuve à l’appui est que dans les expériences de contrôle impliquant dCasRx, qui est catalytiquement inactif, les pourcentages de mouches porteuses à la fois de dCasRx et de constructions d’ARNg sur le nombre total de mouches de la génération F1 étaient systématiquement inférieurs à 50%, le rapport attendu sur la base de la génétique mendélienne si aucune toxicité associée à dCasRx n’était présente. Cela indique que l’expression omniprésente de dCasRx, ainsi que des ARNg, induit une toxicité chez la mouche, ce qui entraîne un rapport d’hérédité inférieur à celui prévu. Les rapports d’hérédité des mouches transhétérozygotes UASt-dCasRx, arno gRNA, GAL4 ont suivi la génétique mendélienne, ce qui suggère à nouveau la toxicité induite spécifiquement par l’expression omniprésente des protéines CasRx et dCasRx. La toxicité dans le système CRISPR/Cas n’est pas nouvelle. Il a été démontré que de grandes quantités de protéine Cas9 sont toxiques chez plusieurs organismes, y compris les mouches29,30,31,32. Une étude récente a développé un système GAL4/UAS personnalisé qui peut régler la quantité de protéine Cas9 exprimée chez les mouches en ajoutant un cadre de lecture ouvert de longueur variable entre la séquence UAS et la séquence Cas9 dans la construction UAS-Cas933. Par conséquent, il vaut la peine d’explorer des moyens de réduire la toxicité induite par CasRx en ajustant le niveau d’expression de la protéine CasRx.
Outre la toxicité induite par l’expression omniprésente des protéines CasRx et dCasRx, les résultats ont également montré une létalité liée aux effets collatéraux non spécifiques hors cible du système CasRx, une caractéristique de nombreux systèmes CRISPR1,2,7,34. Chez certaines des mouches transhétérozygotes double ou triple exprimant l’ARN gRx et le gène non essentiel, par exemple lors du ciblage de Notch, les mouches CasRx transhétérozygotes ont un taux de survie significativement inférieur à celui des mouches dCasRx transhétérozgyes. Dans l’analyse de séquençage de l’ARN de ces mouches transhétéérozygotes exprimant CasRx et gRNA, la réduction des niveaux de transcription des gènes cibles et la réduction des transcriptions des gènes non cibles ont été observées. Ces effets collatéraux étaient dépendants de CasRx et dépendants de la cible, car ils n’ont été observés que chez les mouches transhétérozygotes exprimant à la fois la protéine CasRx et l’ARNg. Il convient de souligner que l’un des gènes cibles, le blanc, n’a montré qu’une réduction limitée et non statistiquement significative des transcriptions lorsque le gène blanc était ciblé par CasRx, ce qui contrastait avec le phénotype de réduction pigmentaire claire. On suppose que cela peut être dû au fait que 1) le moment de la collecte de l’échantillon d’ARN-seq n’était pas bien aligné avec le moment où le gène blanc atteint son apogée d’expression au début du développement, et 2) l’expression localisée du gène blanc dans les yeux rend difficile la collecte des tissus pertinents au début de la phase de développement lorsque seule la collecte d’échantillons du corps entier est possible. Pour réduire l’activité collatérale dans le système CasRx, des études futures sont nécessaires pour bien comprendre les mécanismes sous-jacents au système de phénomène hors cible au niveau de l’organisme.
Fait intéressant, une étude récente35 décrivant des outils Cas13 ciblant l’ARN chez les mouches semblait améliorer la toxicité générale associée à l’expression de CasRx, pour plusieurs raisons possibles. Tout d’abord, les auteurs ont recodé les transgènes Cas13 pour optimiser l’expression chez la drosophile et ont utilisé un promoteur plus faiblement exprimant (actine 5C) par rapport au promoteur de l’ubiquitine utilisé dans la présente étude, conduisant probablement à des niveaux plus faibles d’expression de Cas13 et donc moins de toxicité. En effet, cela est corroboré par les observations selon lesquelles l’expression de CasRx et de dCasRx pilotée par UASt n’était pas, en soi, toxique, car cette étude (et les auteurs dans 35) n’a observé aucune létalité évidente chez les mouches UASt-CasRx. De plus, ces auteurs ont codé leurs ARNg différemment par rapport à cette étude, ce qui pourrait avoir affecté leur expression et réduit la toxicité du système chez les mouches transhétérozygotes Cas13/ ARNg. Par exemple, dans leur étude, deux ARNg ont été exprimés à l’aide du promoteur U6:3 et flanqués d’ARNt pour permettre le traitement de l’ARNg lors de la maturation de l’ARNt sans nécessiter CasRx35. Inversement, dans cette étude, les ARNg ont été codés comme des réseaux ciblant jusqu’à 4 emplacements par gène et imitant la structure endogène du réseau Cas13 trouvée dans les bactéries, ce qui nécessite l’enzyme Cas13 pour traiter chaque ARNg. Ces différentes approches peuvent avoir conduit à des différences dans les niveaux d’expression de l’ARNg et d’autres facteurs qui peuvent avoir des effets inhérents sur la toxicité de l’ensemble du système. Enfin, Huynh et al. ont ciblé des gènes différents de ceux ciblés dans la présente étude, ce qui entraîne des différences dans l’interaction cible-Cas/ARNg et l’activité collatérale et peut avoir des effets sur les niveaux de létalité observés. Ces différences dans la toxicité observée justifient des recherches plus approfondies afin de déterminer les moyens d’améliorer l’ensemble des systèmes.
Dans l’ensemble, cette étude est la première démonstration d’un système Cas fonctionnel ciblant l’ARN programmable génétiquement codé chez D. melanogaster, bien qu’une optimisation supplémentaire du système CasRx (conformément à ce qui est rapporté35) sera nécessaire pour réduire davantage la létalité associée hors cible et augmenter l’efficacité du clivage CasRx sur cible. Le ciblage de l’ARN avec les enzymes Cas est un domaine en évolution rapide avec de nombreuses applications potentielles allant de la lutte contre les insectes vecteurs aux utilisations thérapeutiques1,2,3,4,5,6,7, et ce protocole offre un package de démarrage pour toute personne intéressée par la conception de son premier système CasRx chez les mouches, tout en étant compatible avec la personnalisation et l’optimisation ultérieure du système. Les exemples présentés ici démontrent une gamme de résultats que l’on peut rencontrer lors de la mise en œuvre de ce système in vivo et peuvent servir de références pour d’autres utilisateurs dans l’évaluation des performances du système CasRx dans leurs applications.
Déclarations de divulgation
O.S.A est l’un des fondateurs d’Agragene, Inc., détient une participation au capital et siège au conseil consultatif scientifique de la société. Les termes de cet arrangement ont été examinés et approuvés par l’Université de Californie à San Diego conformément à ses politiques en matière de conflits d’intérêts. Tous les autres auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Ce travail a été soutenu en partie par un financement provenant d’une subvention du programme DARPA Safe Genes (HR0011-17-2-0047) et de prix NIH (R21RAI149161A, DP2AI152071) décernés à O.S.A.
matériels
| Name | Company | Catalog Number | Comments |
| 100% Grape Juice | Welch Foods Inc. | N/A | |
| Active Dry Yeast (908g) | Red Star Yeast Company, LLC | N/A | |
| DMC4500 color camera | Leica Microsystems | DMC4500 | |
| Dual-Luciferase Reporter Assay System | Promega | E1910 | |
| GAL4-GMR flies | Bloomington Drosophila Stock Center | 29967 | |
| GAL4-y flies | Bloomington Drosophila Stock Center | 44373 | |
| Glomax 20/20 Luminometer | Promega | E5331 | |
| Illumina HiSeq2500 Sequencer | Illumina, Inc. | HiSeq2500 | |
| M165FC fluorescent stereomicroscope | Leica Microsystems | M165FC | |
| Nanodrop OneC UV-vis spectrophotometer | ThermoFisher | NDONEC-W | |
| NEBNext Ultra II RNA Library Prep Kit | New England Biolabs, Inc. | E7770 | |
| plasmid # 112686 | Addgene | 112686 | |
| plasmid # 112688 | Addgene | 112688 | |
| plasmid # 132416 | Addgene | 132416 | |
| plasmid # 132417 | Addgene | 132417 | |
| plasmid # 132419 | Addgene | 132419 | |
| plasmid # 132420 | Addgene | 132420 | |
| plasmid # 132421 | Addgene | 132421 | |
| plasmid # 132422 | Addgene | 132422 | |
| plasmid # 132425 | Addgene | 132425 | |
| plasmid # 132426 | Addgene | 132426 | |
| plasmid # 133304 | Addgene | 133304 | |
| plasmid # 164586 | Addgene | 164586 | |
| plasmid #132418 | Addgene | 132418 | |
| plasmid pJFRC81 | Addgene | 36432 | |
| Qiagen RNeasy Mini Kit | Qiagen | 74104 | |
| Restriction endonucleases AscI | New England Biolabs Inc. | R0558L | |
| Restriction endonucleases NotI | New England Biolabs Inc. | R0189L | |
| Restriction endonucleases PacI | New England Biolabs Inc. | R0547L | |
| Restriction endonucleases PstI | New England Biolabs Inc. | R0140L | |
| Restriction endonucleases SwaI | New England Biolabs Inc. | R0604L | |
| Restriction endonucleases XbaI | New England Biolabs Inc. | R0145L | |
| RNA 6000 Pico Kit for Bioanalyzer | Agilent Technologies | 5067-1513 | |
| Turbo DNase | Invitrogen | AM2238 | |
| U6-3:4-gRNA-Fluc flies | Bloomington Drosophila Stock Center | 84125 | |
| U6-3:4-gRNA-GFP; OpIE2-GFP flies | Bloomington Drosophila Stock Center | 84986 | |
| U6-3:4-gRNA-N flies | Bloomington Drosophila Stock Center | 84122 | |
| U6-3:4-gRNA-w flies | Bloomington Drosophila Stock Center | 84124 | |
| U6-3:4-gRNA-y flies | Bloomington Drosophila Stock Center | 84123 | |
| UASt-CasRx flies | Bloomington Drosophila Stock Center | 84121 | |
| UASt-dCasRx flies | Bloomington Drosophila Stock Center | 84120 | |
| Ubiq-CasRx flies | Bloomington Drosophila Stock Center | 84118 | |
| Ubiq-dCasRx flies | Bloomington Drosophila Stock Center | 84119 | |
| Ubiq-Firefly-T2A-eGFP-Ubiq-Renilla flies | Bloomington Drosophila Stock Center | 84127 | |
| Zymoclean Gel DNA Recovery Kits | Zymo Research Corporation | D4007 |
Références
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nat Communications. 9, 1911 (2018).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), 5573 (2016).
- East-Seletsky, A., O'Connell, M., Burstein, D., Knott, G., Doudna, J. RNA Targeting by Functionally Orthogonal Type VI-A CRISPR-Cas Enzymes. Molecular Cell. 66 (3), 373-383 (2017).
- Konermann, S., Lotfy, P., Brideau, N., Oki, J., Shokhirev, M., Hsu, P. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell. 173 (3), 665-676 (2018).
- Buchman, A., Brogan, D., Sun, R., Yang, T., Hsu, P., Akbari, O. Programmable RNA Targeting Using CasRx in Flies. The CRISPR Journal. 3 (3), 164-176 (2020).
- Kushawah, G., et al. CRISPR-Cas13d Induces Efficient mRNA Knockdown in Animal Embryos. Developmental Cell. 54 (6), 805-817 (2020).
- Abudayyeh, O., et al. RNA targeting with CRISPR-Cas13. Nature. 550, 280-284 (2017).
- Perrimon, N., Ni, J., Perkins, L. In vivo RNAi: today and tomorrow. Cold Spring Harbor Perspectives in Biology. 2, 003640 (2010).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
- Ni, J., et al. A Drosophila resource of transgenic RNAi lines for neurogenetics. Genetics. 182 (4), 1089-1100 (2009).
- Ni, J., et al. A genome-scale shRNA resource for transgenic RNAi in Drosophila. Nature Methods. 8, 405-407 (2011).
- Ni, J., et al. Vector and parameters for targeted transgenic RNA interference in Drosophila melanogaster. Nature Methods. 5, 49-51 (2008).
- Heigwer, F., Port, F., Boutros, M. RNA Interference (RNAi) Screening in Drosophila. Genetics. 208 (3), 853-874 (2018).
- Kulkarni, M., et al. Evidence of off-target effects associated with long dsRNAs in Drosophila melanogaster cell-based assays. Nature Methods. 3, 833-838 (2006).
- Ma, Y., Creanga, A., Lum, L., Beachy, P. Prevalence of off-target effects in Drosophila RNA interference screens. Nature. 443, 359-363 (2006).
- Perrimon, N., Mathey-Prevot, B. Matter arising: off-targets and genome-scale RNAi screens in Drosophila. Fly. 1 (1), 1-5 (2007).
- Markstein, M., Pitsouli, C., Villalta, C., Celniker, S., Perrimon, N. Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nature Genetics. 40, 476-483 (2008).
- Champer, J., Buchman, A., Akbari, O. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nature Review Genetics. 17 (3), 146-159 (2016).
- Buchman, A., et al. Broad dengue neutralization in mosquitoes expressing an engineered antibody. PLoS Pathogens. 16 (4), 1008103 (2020).
- Mathur, G., Sanchez-Vargas, I., Alvarez, D., Olson, K., Marinotti, O., James, A. Transgene-mediated suppression of dengue viruses in the salivary glands of the yellow fever mosquito, Aedes aegypti. Insect Molecular Biology. 19 (6), 753-763 (2011).
- Franz, A., et al. Engineering RNA interference-based resistance to dengue virus type 2 in genetically modified Aedes aegypti. Proceedings of the National Academy of Sciences of the United States of America. 103 (11), 4198-4203 (2006).
- Yen, P., James, A., Li, J., Chen, C., Failloux, A. Synthetic miRNAs induce dual arboviral-resistance phenotypes in the vector mosquito Aedes aegypti. Communications Biology. 1, 11 (2018).
- Buchman, A., et al. Engineered resistance to Zika virus in transgenic Aedes aegypti expressing a polycistronic cluster of synthetic small RNAs. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3656-3661 (2019).
- . Addgene Available from: https://www.addgene.org/protocols/restriction-digest/ (2020)
- Gibson, D., Young, L., Chuang, R., Venter, J., Hutchison, C., Smith, H. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Addgene Available from: https://www.addgene.org/protocols/pcr/ (2020)
- . Indiana University Bloomington Available from: https://bdsc.indiana.edu/information/recipes/bloomfood.htmL (2020)
- Dobin, A., et al. STAR:ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Jiang, W., Brueggeman, A., Horken, K., Plucinak, T., Weeks, D. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii. Eukaryotic Cell. 13 (11), 1465-1469 (2014).
- Poe, A., et al. Robust CRISPR/Cas9-Mediated Tissue-Specific mutagenesis reveals gene redundancy and perdurance in Drosophila. Genetics. 211 (2), 459-472 (2019).
- Port, F., Chen, H., Lee, T., Bullock, S. Optimized CRISPR/Cas tools for efficient ger mLine and somatic genome engineering in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 111 (29), 2967-2976 (2014).
- Yang, S., Li, S., Li, X. Shortening the Half-Life of Cas9 maintains its gene editing ability and reduces neuronal toxicity. Cell Reports. 25 (10), 2653-2659 (2018).
- Port, F., et al. A large-scale resource for tissue-specific CRISPR mutagenesis in Drosophila. eLife. 9, 53865 (2020).
- Zhang, X., Tee, L., Wang, X., Huang, Q., Yang, S. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4 (17), 264 (2015).
- Huynh, N., Depner, N., Larson, R., King-Jones, K. A versatile toolkit for CRISPR-Cas13-based RNA manipulation in Drosophila. Genome Biology. 21, 279 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon