Method Article
RNA onipresente e específico para tecidos em Drosophila Melanogaster usando CRISPR/CasRx
Neste Artigo
Resumo
Este artigo descreve um protocolo detalhado para o uso da enzima Cas13D com destino a RNA (RfxCas13D) em moscas.
Resumo
CasRx, um membro da família Cas13 que visa o RNA, é uma nova adição promissora das tecnologias CRISPR/Cas na redução eficiente da transcrição genética com um perfil atraente fora do alvo em níveis celulares e organismos. Recentemente é relatado que o sistema CRISPR/CasRx pode ser usado para alcançar a redução onipresente e específica da transcrição genética em Drosophila melanogaster. Este artigo detalha os métodos do trabalho recente, composto por três partes: 1) onipresente direcionamento de RNA endógeno in vivo usando um sistema CasRx de dois componentes; 2) alvo onipresente de RNA exógeno in vivo usando um sistema CasRx de três componentes; e 3) segmentação in vivo RNA específica do tecido usando um sistema CasRx de três componentes. Os efeitos do direcionamento do RNA observado incluem alterações fenotípicas específicas de genes direcionados, redução de transcrição de RNA direcionada e fenótipos de letalidade ocasionais associados à alta expressão da proteína CasRx e atividade colateral. No geral, esses resultados mostraram que o sistema CasRx é capaz de direcionar a redução da transcrição do RNA no nível do organismo de forma programável e eficiente, demonstrando que o direcionamento do transcrito in vivo, e a engenharia é viável e estabelece as bases para futuras tecnologias de segmentação de RNA baseadas em CRISPR.
Introdução
Desde o advento das tecnologias Clustered Regularmente Interspaced Short Palindromic Repeats (CRISPR), grande parte do foco neste campo tem sido na edição de DNA, que oferece aplicações transformadoras em medicina e biotecnologia1. A alteração permanente das sequências de DNA, no entanto, nem sempre é desejada devido a considerações éticas. À luz disso, estudos recentes começaram a desenvolver ferramentas baseadas em CRISPR para direcionar o RNA e demonstraram que as tecnologias CRISPR podem de fato ser usadas para direcionamento de RNA em uma variedade de sistemas biológicos2, 3,4,5,6,7. Em muitos desses sistemas testados, a abordagem atual amplamente utilizada para direcionar a redução de RNA e transcrição é a interferência de RNA (RNAi), que está longe de ser perfeita, muitas vezes exibindo eficácia variada e atividade fora do alvo quando usada no vivo8,9,10,11,12,13,14,15,16,17 . Portanto, dado o status dessas tecnologias, vale a pena explorar ainda mais os potenciais de ferramentas baseadas em CRISPR para segmentação de RNA.
Um estudo recente notável relatou que a ribonuclease CasRx, um membro da classe Cas13d, pode reduzir eficientemente os níveis de transcrição genética na cultura celular humana e possui um perfil atraente fora do alvo4. Esse achado levou à questão de saber se essa nova ribonuclease pode manter sua eficácia e baixa taxa de RNA para o alvo do RNA no nível do organismo. Um estudo recente abordou essa questão mostrando que o sistema CasRx pode ser usado para alcançar a redução onipresente e específica da transcrição genética em Drosophila melanogaster5.
Para agilizar a usabilidade desta abordagem recentemente publicada, este protocolo detalha os métodos deste trabalho recente, que consiste em três partes principais: 1) onipresente direcionamento in vivo RNA utilizando um sistema CasRx de dois componentes; 2) alvo onipresente de RNA exógeno in vivo usando um sistema CasRx de três componentes; e 3) segmentação de RNA vivo específica de tecido usando um sistema CasRx de três componentes.
Os RNAs guias (gRNA) que visam diferentes genes-alvo sob o controle de um promotor onipresente foram projetados e linhas de mosca expressando essas construções contendo gRNA foram geradas. As construções casRx sob o controle de um promotor onipresente, ou uma sequência de ativação a montante condicional (UASt) ativada pelo fator de transcrição GAL4, também foram projetadas e linhas de mosca que abrigam esses construtos contendo CasRx gerados. Construções casrx catalyticamente inativas, dCasRx, foram projetadas e usadas como controles negativos. O alvo onipresente do RNA em moscas é alcançado cruzando linhas aéreas que expressam gRNA com linhas aéreas onipresentes de expressão CasRx. A prole que expressa tanto o projeto gRNA visando uma transcrição genética específica quanto a proteína CasRx tem uma redução onipresente das transcrições genéticas direcionadas. O direcionamento de RNA específico para o tecido em moscas é alcançado pela primeira vez cruzando moscas que expressam gRNA com moscas expressas UASt-CasRx, obtendo moscas transheterozigous carregando tanto gRNA quanto construções UASt-CasRx. Tais moscas, por sua vez, são cruzadas com moscas de forma específica de tecido, resultando na geração de expressão CasRx específica do tecido e direcionamento de RNA em moscas.
A natureza programável do sistema CasRx oferece a possibilidade de personalização e otimização para ajudar a alcançar alta eficácia e baixa atividade fora do alvo para o RNA in vivo. As aplicações potenciais de RNA-targeting baseada em CRISPR são numerosas, incluindo a substituição do RNAi no laboratório e a contribuição para o controle de vetores de insetos na natureza. Desta última, uma das necessidades globais não atendidas é o desenvolvimento de ferramentas eficientes para combater infecções de vírus RNA transmitidas por mosquitos. Muitos vírus do RNA, como o vírus da dengue, zika e chikungunya, são transmitidos através de mosquitos, afetando a saúde humana e contribuindo com a mortalidade. Muitas propostas para a engenharia de populações de mosquitos com resistência ao vírus para prevenção de doenças foram feitas; no entanto, nenhuma tecnologia atual é capaz de tornar os mosquitos simultaneamente resistentes a todos os vírus RNA significativos18,19,20,21,22,23. Os sistemas Cas direcionados ao RNA podem fornecer um ponto de partida para tal tecnologia, permitindo uma plataforma programável para atingir todos os vírus RNA transmitidos por mosquitos.
Protocolo
1. Alvo onipresente in vivo RNA usando um sistema CasRx de dois componentes
- Gerando vetor de expressão Ubiq-CasRx e Ubiq-dCasRx
- Amplie a sequência CasRx usando uma reação de corrente de polimerase (PCR) com primer 1050E. C3 e 1050E. C4 e o CasRx original constroem pNLS-RfxCas13d-NLS-HA (pCasRx); e amplificar a sequência dCasRx usando PCR com primer 1050E. C3 e 1050E. C4 e o dCasRx original constroem pNLS-dRfxCas13d-NLS-HA (pdCasRx)4 (Tabela 1). Purificar os fragmentos de CasRx e dCasRx amplificados depois usando um kit de purificação de gel.
- Vetor de base digestora (Addgene plasmid #112686) com enzimas de restrição SwaI e PacI24. Nos produtos resultantes, use um kit para purificar o fragmento maior, que é chamado de espinha dorsal vetorial base.
- Monte o vetor Ubiq-CasRx com a espinha dorsal do vetor base e o fragmento CasRx usando o método de montagem Gibson; montar o vetor Ubiq-dCasRx com a espinha dorsal do vetor base e o fragmento dCasRx usando o método de montagem Gibson25.
NOTA: O Addgene ID do vetor Ubiq-CasRx (OA-1050E) é #132416, e o Addgene ID do vetor Ubiq-dCasRx (OA-1050R) é #132417.
- Gerando vetor de expressão gRNA
- Projetar cada fragmento de gRNA com base nos seguintes critérios: sequência de alvos sendo 30 nucleotídeos de comprimento; o comprimento máximo dos trechos poli-U na sequência de alvos sendo 4 pares de base; o teor de sequência de alvos GC está na faixa de 30% a 70%; a sequência de alvos previu não formar estruturas fortes de grampos de RNA; e a sequência de destino contendo estrutura secundária ou terciária mínima prevista de RNA5.
NOTA: Este estudo projetou cada gRNA como 4 sequências tandem cada 30 nucleotídeos de comprimento, espaçados por 36 repetições diretas de nucleotídeos longos, e com um exterminador de 7 timmina em ambas as extremidades5. Para o gene alvo exógeno, GFP, os mesmos critérios acima foram seguidos com a adição de um fragmento OpIE2-GFP5. - Amplie a sequência de promotores U6:3 usando PCR com primers 1043.C1 e 1043.C23 e o plasmídeo Addgene #112688 (Tabela 1)26. Purificar os fragmentos U6:3 amplificados usando o kit de purificação de gel.
- Digeste Addgene plasmid #112688 com enzima de restrição AscI e XbaI24. Nos produtos resultantes, use um kit para purificar os fragmentos maiores, que é chamado de espinha dorsal vetorial pré-base.
- Monte o vetor base com a espinha dorsal do vetor pré-base e o fragmento U6:3 usando o método de montagem Gibson25. O vetor base daqui em diante se chama OA-1043.
NOTA: O Plasmid OA-1043's Addgene ID é #164586. - Sintetizar o fragmento de gRNA do gene alvo usando o serviço de síntese genética externa.
- Digerir o vetor base OA-1043 com a enzima de restrição PstI e NotI24. Mantenha todo o produto de digestão, que é chamado de OA-1043 digerido.
- Monte o vetor de expressão gRNA com o OA-1043 digerido e o fragmento gRNA genético alvo usando o método de montagem Gibson25.
NOTA: Foram estudados quatro genes-alvo: três eram endógenos (branco, Notch, amarelo), um era exógeno (GFP). Seus IDs Addgene são: #132420 (gRNAw), #132421(gRNAN), #132425 (gRNAy) e #133304 (gRNAGFP).
- Projetar cada fragmento de gRNA com base nos seguintes critérios: sequência de alvos sendo 30 nucleotídeos de comprimento; o comprimento máximo dos trechos poli-U na sequência de alvos sendo 4 pares de base; o teor de sequência de alvos GC está na faixa de 30% a 70%; a sequência de alvos previu não formar estruturas fortes de grampos de RNA; e a sequência de destino contendo estrutura secundária ou terciária mínima prevista de RNA5.
- Gerando moscas transgênicas
- Injete vetores de expressão em embriões de mosca usando serviço externo de injeção de embriões de moscas e embriões de moscas contendo locais de integração ØC31. Guarde os embriões injetados a 26 °C.
NOTA: A linha attp40w (com locais de integração no 2º cromossomo) foi usada para gerar linhas CasRx e a linha 8622 (com locais de integração no 3º cromossomo) foi usada para gerar várias linhas de gRNA. - Mantenha as moscas como linhas homozigas ou como linhas heterozigous equilibradas.
NOTA: As moscas Ubiq-CasRx e Ubiq-dCasRx foram mantidas como linhas heterozigous equilibradas com o CyO como o balanceador. Além disso, tanto os vetores Ubiq-CasRx quanto os vetores Ubiq-dCasRx contêm um marcador dsRed. Como resultado, as moscas Ubiq-CasRx e Ubiq-dCasRx têm os seguintes três fenótipos: dsRed-positivo, asas encaracoladas e olhos brancos. As moscas que expressavam gRNA foram mantidas como linhas homozigas. Seus números de ações da Bloomington Drosophila Stock Center (BDSC) são: #84118 (Ubiq-CasRx), #84119 (Ubiq-dCasRx), #84124 (gRNAw), #84122 (gRNAN), #84123 (gRNAy), #84986 (gRNAGFP).
- Injete vetores de expressão em embriões de mosca usando serviço externo de injeção de embriões de moscas e embriões de moscas contendo locais de integração ØC31. Guarde os embriões injetados a 26 °C.
- Genética de moscas (Figura 1A)
- Colete 10 moscas fêmeas adultas virgens da linha homozigosa gRNA e colete 5 moscas machos adultas da linha heterozigous equilibrada Ubiq-CasRx/CyO. Coloque as moscas femininas e masculinas coletadas, que são chamadas de moscas parentais, em um frasco complementado com fermento seco em pó (Figura 1A).
- Repita a etapa anterior 3 vezes para gerar 3 réplicas. Para o grupo de controle, use a linha Ubiq-dCasRx/CyO, mantendo tudo igual.
NOTA: Para um frasco de vidro regular de alimentos, 0,1 g de fermento seco em pó é suficiente. A receita de comida voadora do BDSC é usada. - Guarde os frascos que contêm as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C por pelo menos 20 dias.
- Observe os frascos todos os dias para ver se algum novo gênio adulto emergiu de pupae da geração F1. Se assim for, anestesia-os com dióxido de carbono inserindo um tubo conectado a um tanque de dióxido de carbono dentro dos frascos de mosca, em seguida, ligando o interruptor de fluxo por 10 segundos.
- Uma vez que as moscas se tornam imóveis, esvazie-as do frasco para um fly-pad, que também está conectado ao tanque de dióxido de carbono e o dióxido de carbono flui continuamente através da plataforma de voo.
- Marque o fenótipo das moscas anestesiadas e imagem-as usando câmera colorida conectada a um estereóscópio fluorescente. Conte o número de gênios com fenótipos diferentes. Use software de processamento de imagem para pós-processamento e compilação de imagens (Figura 2A - 2D).
NOTA: Com base na genética mendeliana, dois tipos de moscas são esperadas entre os progens para cada cruz (Figura 1A).
- RNA-Seq (Figura 2E – 2G)
- Coleta de amostras
NOTA: Escolha um método adequado de coleta de amostras a partir dos 3 exemplos abaixo; São necessárias 3 réplicas para cada tipo de amostra distinta.- Coleção de amostras de cabeça de mosca adulta
- Colete 10 moscas fêmeas adultas virgens da linha homozygous gRNA. Colete 5 moscas masculinas adultas da linha Ubiq-CasRx/CyO equilibrada. Coloque as moscas femininas e machos coletadas, que são as moscas parentais, em um frasco complementado com fermento seco em pó.
- Repita a etapa anterior 3 vezes para 3 réplicas. Para o grupo de controle, use a linha Ubiq-dCasRx/CyO, mantendo tudo igual.
- Guarde os frascos que contêm as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C até que os progênies emergam de pupae.
- Colete moscas adultas de 10 dias com o fenótipo correto. Anestesiar as moscas com dióxido de carbono, depois cortar a cabeça de mosca e colocar as cabeças em um tubo de centrífuga de 1,5 mL em gelo seco. Armazene o tubo de centrífuga a -80 °C. Repita esta etapa 3 vezes para 3 réplicas.
- 17 – coleta de amostras de embriões de 20 horas
- Colete moscas fêmeas adultas virgens de 8-10 da linha homozigoso gRNA. Colete moscas machos adultos de 4-5 da linha Ubiq-CasRx/CyO equilibrada. Coloque as moscas femininas e machos coletadas, que são as moscas parentais, em um frasco complementado com fermento seco em pó.
- Repita a etapa anterior 3 vezes para 3 réplicas. Para o grupo de controle, use a linha Ubiq-dCasRx/CyO, mantendo tudo igual.
- Guarde os frascos que contêm as moscas dos pais a 26 °C durante 48 horas.
- Prepare uma câmara de coleta de embrião de suco de uva para cada réplica seguindo esta receita: 376 mL de água, 126 mL de suco de uva, 15 g de ágar e 6 g de sacarose. Coloque a mídia em um béquer 1 L e micro-ondas em alta por 5-6 minutos enquanto mantém um olho atento na mídia no béquer para verificar se bolhas/espuma aparecem. Se assim for, pare o micro-ondas e deixe a bolha/espuma se instalar. Continue microwaving desta maneira até que a bolha se torne clara. Não gire até que todas as bolhas estejam claras. Por fim, adicione 10 mL de 100% de álcool e 5 mL de ácido acético. Misture bem, em seguida, pipet a mídia em placas de Petri de 35 mm com um pipet sorológico de 25 mL. Quando a mídia se solidifica na placa de Petri, ela está pronta para uso.
- Ao final da incubação de 48 horas, transfira moscas parentais para as câmaras de coleta de embriões de suco de uva e incuba-las a 26 °C por 3 h. Em seguida, remova as moscas adultas enquanto mantém os embriões recém-colocados nas placas de suco de uva por mais 17 h a 26 °C.
- Após a incubação, colete os 50 – 100 embriões das placas de suco de uva, limpe a superfície do embrião submergindo-os em água deionizada e transfira-os para um tubo de centrífugas de 1,5 mL no gelo. Armazene-os a -80 °C. Repita esta etapa 3 vezes para 3 réplicas.
- Primeira coleta de amostras de larvas instar
- Colete moscas fêmeas adultas virgens de 8-10 da linha homozigoso gRNA. Colete moscas machos adultos de 4-5 da linha Ubiq-CasRx/CyO equilibrada. Coloque as moscas femininas e machos coletadas, que são as moscas parentais, em um frasco complementado com fermento seco em pó. Repita esta etapa 3 vezes para 3 réplicas. Para o grupo de controle, use a linha Ubiq-dCasRx/CyO, mantendo tudo igual.
- Guarde os frascos que contêm as moscas dos pais a 26 °C durante 48 horas. Em seguida, transfira as moscas adultas para outro novo frasco de alimentos regulares para incubação noturna (16 h) a 26 °C. Então, remova as moscas adultas.
- Mantenha o frasco contendo embriões a 26 °C por 24 horas e, em seguida, marque a primeira larva instar transheterozúlga sob o microscópio usando marcadores distintos baseados. Colete 15-30 larvas com fenótipos corretos e coloque-as em um tubo de centrífuga de 1,5 mL e armazene-as a -80 °C. Repita esta etapa 3 vezes para 3 réplicas.
- Coleção de amostras de cabeça de mosca adulta
- Seqüenciamento
- Extração de RNA: use um kit de extração de RNA comercialmente disponível e siga as instruções do kit para processar todas as amostras. Em seguida, incubar as amostras de RNA extraídas com desoxyribonuclease comercialmente disponível e seguir sua instrução para remover qualquer DNA contaminante das amostras.
- Meça a concentração de RNA usando espectrofotômetro UV-vis comercialmente disponível. Meça a integridade do RNA nas amostras usando a configuração de ensaio de integridade RNA disponível comercialmente.
- Construa as bibliotecas RNA-seq usando o kit de preparação da biblioteca RNA disponível comercialmente.
- Use o serviço de sequenciamento externo para sequenciamento de biblioteca com as seguintes configurações: modo de leitura única; comprimento de leitura: 50nt, profundidade: 20 milhões de leituras por biblioteca. Realize chamadas base com RTA 1.18.64 e, em seguida, converteu os dados para FASTQ usando bcl2fastq 1.8.4.
NOTA: Os dados de sequenciamento bruto podem ser encontrados no National Center for Biotechnology Information Sequencing Read Archive (ID de submissão: SUB6818910 [BioProject: PRJNA600654]).
- Bioinformática
- O mapa lê os dados de sequenciamento para liberar 6 genomas de melanogaster Drosophila do Projeto Genoma de Berkeley Drosophila (número de adesão do GenBank: GCA_000001215.4) e as exogenous sequências CasRx e GFP usando a configuração padrão do parâmetro STAR aligner28 com a adição da opção de filtro "-outFilterType BySJout" e "-sjdbGTFfile Drosophila_melanogaster. BDGP6.22.97.gtf" arquivo de formato de transferência genética do ENSEMBL.
- Determine a contagem bruta de transcrição para cada transcrição anotada com o recurso Counts35 usando as opções "-t exon -g gene_id -M -O --fraction". Em seguida, normalize a contagem de transcrições brutas usando contagens totais de transcrição usando o script Perl "addTpmFpkmToFeatureCounts.pl".
- Use o método máximo posteriori com o estimador de encolhimento original no pipeline DESeq2 para estimar a mudança de dobra logarítmica (LFC) de cada gene.
- Coleta de amostras
2. Alvo onipresente in vivo exógeno do RNA usando um sistema CasRx de três componentes
- Gerando vetor de expressão onipresente de alvo exógeno
- PCR amplifica o fragmento do promotor Ubiq usando primers 1052B. C1 e 1052B. C2 e o plasmídeo Addgene #112686 26. Em seguida, o PCR amplifica o fragmento T2A-eGFP amplificado a partir de Addgene plasmid #112686 com primers 908A.1 e 908A.2 (Tabela 1)26. Em seguida, o PCR amplifica o fragmento do promotor ubiq como sequência invertida usando addgene plasmid #112686 com primers 908A.3 e 908A.4 (Tabela 1)26. Purificar o fragmento do promotor ubiq, o fragmento T2A-eGFP e o fragmento de promotor ubiq invertido usando o kit de purificação de gel.
- Solicite uma sequência personalizada de codificação de luciferase de vagalume e um fragmento personalizado contendo um fragmento p10 3'UTR, luciferase renilla invertida seguido de um fragmento SV40 3'UTR.
- Digeste Addgene plasmid #112688 com enzima de restrição AscI e XbaI24. Nos produtos resultantes, purificar o gel-purificar os fragmentos maiores usando o kit de purificação de gel, que é chamado de backbone vetorial base.
- Use o método de montagem gibson para montar o vetor base com a espinha dorsal do vetor base e os seguintes fragmentos: fragmento promotor ubiq, o fragmento T2A-eGFP, o fragmento de promotor ubiq invertido, a sequência de codificação da luciferase de vagalume, e a luciferase renilla invertida seguida por um fragmento SV40 3'UTR25.
NOTA: O ID addgene do vetor de expressão de dupla luciferase resultante (OA-1052B) é #132426.
- Gerando vetor de expressão gRNA
- PCR amplifica a sequência de promotores U6:3 usando primers 1043.C1 e 1043.C23 e o plasmídeo Addgene #112688 (Tabela 1)26. Purificar os fragmentos U6:3 amplificados usando o kit de purificação de gel.
- Digeste Addgene plasmid #112688 com enzima de restrição AscI e XbaI24. Nos produtos resultantes, purificar o gel-purificar os fragmentos maiores, que é chamado de backbone vetorial pré-base, usando kit de purificação de gel.
- Monte o vetor base com a espinha dorsal do vetor pré-base e o fragmento U6:3 usando o método de montagem Gibson25. O vetor base daqui em diante se chama OA-1043.
- Sintetizar o fragmento de gRNA do gene alvo usando o serviço de síntese genética externa.
- Digerir o vetor base OA-1043 com a enzima de restrição PstI e NotI24. Mantenha todo o produto de digestão, que é chamado de OA-1043 digerido.
- Monte o vetor de expressão gRNA com o OA-1043 digerido e o fragmento gRNA genético alvo usando o método de montagem Gibson25.
NOTA: O ID addgene do plasmídeo resultante (OA-1052K) é #132422.
- Gerando moscas transgênicas
- Injete vetor OA-1052B em embriões de moscas usando embriões de moscas contendo local de integração ØC31 no 3º cromossomo, BDSC fly stock número 9744, via serviço externo de injeção de embriões de mosca. Da mesma forma, injete vetor OA-1052K em embriões de moscas usando embriões de moscas contendo o local de integração ØC31 no 3º cromossomo, bdsc fly stock número 8622. Guarde os embriões injetados a 26 °C.
- Mantenha as moscas expressas de dupla luciferase e o gRNA voa como linhas homozigas; manter as linhas Ubiq-CasRx como linhas heterozigous de equilíbrio duplo, cruzando a linha Ubiq-CasRx heterozygous de equilíbrio único gerada na seção 1 para linhas balanceirantes que carregam cromossomo balanceador TM6 com marcador stubble (Stb) e reter apenas os progágios de dois balanceados com olhos brancos, asas encaracoladas e fenótipos dsRed-fluorescente simultaneamente.
NOTA: Os números de ações da BDSC são: #84127 (Ubiq-Fluc-Rluc), #84125 (gRNAFluc).
- Genética de Mosca (Figura 1B e Figura 3A)
- Colete 8-10 moscas virgens adultas da linha de expressão de dupla luciferase. Coletar 4-5 moscas masculinas adultas do equilibrado heterozigoso Ubiq-CasRx/CyO; +/TM6, linha Stb que mostra asas de olhos brancos, cacheadas e fluorescência dsRed simultaneamente. Coloque as moscas femininas e masculinas coletadas, que são as moscas parentais, em um frasco complementado com pó de levedura seca (doravante chamado Passo 1 Cruz).
- Repita a etapa anterior 3 vezes para 3 réplicas. Para o grupo de controle, use o Ubiq-dCasRx/CyO; +/TM6, linha Stb mantendo tudo igual.
- Guarde os frascos passo 1 cross contendo as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C por pelo menos 14 dias. Durante este tempo, colete 8-10 virgens femininas da linha homozigórica de luciferase-alvo gRNA. Repita esta etapa 3 vezes para 3 réplicas.
- Observe os frascos da Cruz passo 1 todos os dias para ver se alguma nova mosca adulta emerge da pupae. Se assim for, anestesia-os com dióxido de carbono, colete 5 moscas machos expressando tanto o Ubiq-CasRx (ou Ubiq-dCasRx) quanto o repórter de dupla luciferase dos progenias e coloque-os em um novo frasco junto com 10 fêmeas virgens da linha gRNA de mira de luciferase vagalumes (doravante chamada Passo 2 Cross). Repita esta etapa 3 vezes para 3 réplicas.
- Colete outro homem de 5 dias de idade expressando tanto o Ubiq-CasRx (ou Ubiq-dCasRx) quanto o repórter de dupla luciferase dos frascos Passo 1 Cross e incuba-os por 2 – 4 dias a 26 °C. Em seguida, transfira-os para tubo centrífugo de 1,5 mL e armazene-os a -80 °C. Repita esta etapa 3 vezes para três réplicas.
- Guarde os frascos da Etapa 2 Cross contendo as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C por pelo menos 20 dias.
- Observe os frascos da Cruz passo 2 todos os dias para ver se algum novo gênio adulto emergiu de pupae. Se assim for, anestesia-os com dióxido de carbono, marque os fenótipos das moscas anestesiadas e imagem-as usando câmera colorida equipada com um estereóscópio fluorescente. Conte o número de gênios com fenótipos diferentes. Use software de processamento de imagem para pós-processamento e compilação de imagens (Figura 3B – 3C).
NOTA: A genética mendeliana sugere que, se as moscas forem todas viáveis, são esperados 4 tipos de moscas entre os progens da Etapa 2 Cross, cada uma representando 25% da população (Figura 1B e Figura 3A).
- Ensaio de luciferase (Figura 3D)
- Gere as moscas transheterozigous triplas, bem como as moscas de controle repetindo as etapas 2.4.1-2.4.5. Recolher moscas machos ao nascer e envelheca-as até os 3 dias de idade.
- Transfira as moscas de 3 dias em tubos de centrífugas de 1,5 mL e lise-os usando um pilão e o tampão de luciferase de ensaio de luciferase disponíveis comercialmente.
- Use 5 μL de tecido líscido de cada amostra para medir tanto a atividade de luciferase de vagalume quanto renilla usando kit de ensaio de luciferase e luminômetro disponíveis comercialmente.
3. Segmentação in vivo RNA específica do tecido usando um sistema CasRx de três componentes
- Gerando vetor de expressão UASt-CasRx e UASt-dCasRx
- PCR amplifica a sequência de promotores do UASt usando plasmid pJFRC81 e primers 1041.C9 e 1041.C11; em seguida, pcR amplificar fragmento CasRx usando plasmid OA-1050E (Addgene ID #132416) e primers 1050L. C1 e 1050E. C4; e, em seguida, pcR amplificar fragmentos dCasRx usando plasmid OA-1050R (Addgene ID #132417) e primers 1050L. C1 e 1050E. C4 (Tabela 1)26. Purificar o amplificado UASt, sequência de promotores UASt, CasRx e dCasRx usando kit de purificação de gel.
- Vetor de base digestora (Addgene plasmid #112686) com enzimas de restrição NotI e PacI24. Nos produtos resultantes, purificar o fragmento maior, que é chamado de backbone vetorial base, usando kit de purificação de gel.
- Monte o vetor UASt-CasRx com a espinha dorsal do vetor base, a sequência de promotores UASt e o fragmento CasRx usando o conjunto Gibson; em seguida, monte o vetor UASt-dCasRx com a espinha dorsal do vetor base, a sequência de promotores UASt e o fragmento dCasRx usando o método de montagem Gibson25.
NOTA: O vetor UASt-CasRx é Addgene plasmid #132418, e o vetor UASt-dCasRx é Addgene plasmid #132419
- Gerando moscas transgênicas
- Injete vetor UASt-CasRx em embriões de mosca usando serviço de injeção de embriões de moscas e embriões de moscas ØC31 local de integração 8621 em seus 2º cromossomos; em seguida, injete vetor UASt-dCasRx em embriões de mosca usando serviço de injeção de embriões de moscas e embriões de moscas ØC31 local de integração 8621 em seus 2º cromossomos. Guarde os embriões injetados a 26 °C.
- Mantenha as moscas como linhas heterozigous duplamente equilibradas com marcadores CyO e Sb. NOTA: Os IDs das linhas de mosca em BDSC são 84121 (UASt-CasRx) e 84120 (UASt-dCasRx).
- Genética de moscas (Figura 1C)
- Encomendar linhas GAL4 desejadas do BDSC; Obtenha linhas gRNA relevantes a partir da etapa 3.2.2 (ou do BDSC).
NOTA: Foram utilizadas as seguintes 2 moscas GAL4 da BDSC: GAL4-GMR (BDSC ID: #29967), GAL4-y (BDSC ID: #44373). As mesmas 3 linhas de gRNA geradas na primeira seção foram utilizadas: gRNAw (BDSC ID: #84124), gRNAN (BDSC ID #84122), gRNAy(BDSC ID: #84123). - Colete 5-10 moscas fêmeas adultas virgens da linha gRNA. Coletar 2-4 moscas masculinas adultas do UASt-CasRx/CyO heterozigoso duplo equilibrado; +/TM6, linha Sb que mostra asas de olhos brancos, cacheadas e fluorescência dsRed simultaneamente. Coloque as moscas coletadas do sexo feminino e masculino, que são as moscas parentais, em um frasco de comida regular (doravante chamado Passo 1 Cross). Repita esta etapa 3 vezes para 3 réplicas. Para o grupo de controle, use o UASt-dCasRx/CyO; +/TM6, linha Sb mantendo tudo igual.
- Guarde os frascos passo 1 cross contendo as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C por pelo menos 14 dias. Durante este tempo, colete 5-10 virgens femininas da linha GAL4. Repita esta etapa 3 vezes para 3 réplicas.
- Observe os frascos da Cruz passo 1 todos os dias para ver se alguma nova mosca adulta emerge da pupae. Se assim for, anestesia-os com dióxido de carbono, colete 2-4 moscas machos expressando tanto o UASt-CasRx (ou UASt-dCasRx) quanto o vetor de gRNA dos progenias que simultaneamente têm fenótipos dsRed-fluorescente e stubble. Coloque os machos coletados do Passo 1 Cross em um novo frasco junto com 5-10 fêmeas virgens coletadas da linha GAL4 (doravante chamada Passo 2 Cross). Repita esta etapa 3 vezes para 3 réplicas.
- Guarde os frascos da Etapa 2 Cross contendo as moscas dos pais a 26 °C durante 48 horas. Então remova todas as moscas dos pais de cada frasco. Em seguida, mantenha os frascos a 26 °C por pelo menos 20 dias.
- Observe os frascos da Cruz passo 2 todos os dias para ver se algum novo adulto voa emergs de pupae. Se assim for, anestesia-os com dióxido de carbono, marque os fenótipos das moscas anestesiadas e imagem-as usando câmera colorida equipada com um estereóscópio fluorescente. Conte o número de gênios com fenótipos diferentes. Use software de processamento de imagem para pós-processamento e compilação de imagens (Figura 4).
NOTA: A genética mendeliana sugere que, se as moscas forem todas viáveis, são esperados 4 tipos de moscas entre os progens da Etapa 2 Cross, cada uma representando 25% da população (Figura 1C).
- Encomendar linhas GAL4 desejadas do BDSC; Obtenha linhas gRNA relevantes a partir da etapa 3.2.2 (ou do BDSC).
Resultados
Alvo in vivo in vivo RNA usando um sistema CasRx de dois componentes
As moscas transheterozigous F1 expressando tanto o Ubiq-CasRx quanto o gRNA (visando genes endógenos e exógenos) mostraram fenótipos marcados em comparação com as moscas de controle expressando as construções Ubiq-dCasRx e gRNA (Figura 2 e Figura 4). Especificamente, as moscas CasRx transheterozigous têm níveis significativamente mais baixos de taxa de sobrevivência em comparação com as moscas transheterozgyous dCasRx, indicando toxicidade do sistema Ubiq-CasRx (Figura 2A e Figura 4A). Vale ressaltar que tanto as moscas transheterozigous CasRx quanto dCasRx têm menos de 50% de taxa de herança, que é a razão esperada com base na genética mendeliana. Dos três genes alvo, o Ubiq-CasRx/+; U6-gRNAN/+ voa e Ubiq-CasRx/+; As moscas U6-gRNAy/+ são inviáveis (herança de 0%) e não cresceram além do segundo estágio de larvas instar (Figura 2A-2B). O sobrevivente Ubiq-CasRx/+; As moscas U6-gRNAw/+ , a herança da qual era de 12,9%, mostraram um fenótipo de olhos brancos totalmente penetrante (Figura 2B). Além dos traços observáveis associados ao CasRx, pudemos confirmar uma redução significativa das transcrições genéticas-alvo para 3 genes-alvo: Entalhe, amarelo e GFP (Figura 2E-2G). A redução das transcrições genéticas brancas foi observada em moscas Ubiq-CasRx/+, U6-gRNAw/+ em comparação com o controle Ubiq-dCasRx/+, U6-gRNAw/+ voa, embora a redução não tenha sido estatisticamente significante (Figura 2E - 2F). Evidências de atividade fora do alvo induzida pelo CasRx foram encontradas ao comparar as transcrições expressas diferencialmente entre amostras de moscas expressas por CasRx e amostras de moscas expressas por dCasRx (Figura 2E, 2G). O número de transcrições não-alvos expressas significativamente diferencialmente são os seguintes: branco, 253 (1,4% do total de transcrições); Notch, 300 (1,7%); amarelo, 41 (0,23%); GFP, 5.880 (33%) (Figura2G). Do total de 17.779 transcrições diferentes, 6 transcrições não-alvo foram expressas significativamente diferencialmente em todos os 4 grupos de amostras. Uma das 6 transcrições identificadas foi Gadd45, um gene envolvido em apoptose e prisão celular em moscas, levantando a possibilidade de que a ação enzimática do CasRx possa desencadear diretamente apoptose celular ou desencadear indiretamente a má expressão de outros genes, o que, por sua vez, leva à apoptose. Finalmente, vale a pena notar que as moscas Ubiq-CasRx e Ubiq-dCasRx não foram estabelecidas como estoques homozigosos, presumivelmente devido à toxicidade conferida pela alta expressão onipresente. Como resultado, as moscas heterozigous Ubiq-CasRx/CyO e Ubiq-dCasRx/CyO foram usadas para atravessar com linhas de moscas gRNA homozigous. Em suma, o sistema Ubiq-CasRx de dois componentes é capaz de alcançar alvos onipresentes de RNA para alvos endógenos e exógenos, resultando em fenótipos observáveis e redução de transcrições. Esses resultados também mostraram que o direcionamento de RNA mediado pelo CasRx pode introduzir toxicidade in vivo.
Alvo de RNA exógeno in vivo onipresente usando um sistema CasRx de três componentes
Os resultados da cruz de duas etapas mostraram que, apesar da natureza exógena do gene alvo (ou seja, Fluc), expressando todos os três transgenes em transheterozigotes f2 triplos (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) resultou em 100% de letalidade em comparação com cruzamentos de controle envolvendo Ubiq-dCasRx, onde nenhuma letalidade foi observada nas transheterozygotes F2 triplas (Ubiq-dCasRx/+; gRNAc/Ubiq-Fluc-Ubiq-Rluc) (Figura 3B-C ). Mais especificamente, apenas a combinação de todos os três transgenes (Ubiq-CasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) resultou em 100% de letalidade (Figura 3B e D), enquanto (Ubiq-CasRx/+; gFluRNAc/TM6) e (Ubiq-CasRx/+; Os genótipos Ubiq-Fluc-Ubiq-Rluc/TM6 eram viáveis e não tinham fenótipos com suas taxas de herança que correspondiam às taxas de transmissão mendelianas esperadas, sugerindo que a disponibilidade da sequência de destino (ou seja, luciferase de vagalume) em combinação com Ubiq-CasRx/+ e o gRNAFluc foi o que resultou nos fenótipos de letalidade observados, presumivelmente decorrentes da atividade colateral das enzimas Cas132,8 . Além disso, nenhum fenótipo distinto ou influência dramática sobre a herança em transheterozygotes F1 (Ubiq-CasRx/+; gRNAFluc/+ ou Ubiq-CasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) foram observados em comparação com os controles Ubiq-dCasRx (Ubiq-dCasRx/+; gRNAFluc/+ ou Ubiq-dCasRx/+; Ubiq-Fluc-Ubiq-Rluc/+) (Figura 3B), indicando que uma enzima catalíticamente ativa é essencial para obter os fenótipos de letalidade observados. Além disso, os níveis de expressão fluc e rluc em moscas de todos os genótipos viáveis não mostraram redução significativa na expressão Fluc no Ubiq-dCasRx triple transheterozygotes (Ubiq-dCasRx/+; gRNAFluc/Ubiq-Fluc-Ubiq-Rluc) em comparação com controles duplos de repórteres luciferase. Isso sugere que os níveis de expressão da proteína Fluc não foram reduzidos pela segmentação dCasRx (Figura 3D). Juntos, o fenótipo de letalidade comum nos dois diferentes experimentos de alvo onipresentes do RNA mediados por CasRx indicam que, quando usado em tecidos onipresentemente, o alvo de RNA mediado por CasRx pode ser tóxico para o organismo.
Segmentação in vivo RNA específica do tecido usando um sistema CasRx de três componentes
O alto nível de toxicidade observado em experimentos onipresentes de segmentação de RNA nos levou a explorar o direcionamento de RNA específico do tecido usando um projeto de sistema CasRx de três componentes detalhado na seção de métodos. De fato, o nível de toxicidade observado foi reduzido quando a expressão geral casRx foi reduzida usando o promotor UASt em comparação com o do promotor ubiq, isso é exemplificado em três aspectos: 1) as linhas UASt-CasRx e UASt-dCasRx foram mantidas como linhas homozigas, embora com base no esquema cruzado de duas etapas duplamente equilibradas as linhas UASt-CasRx e UASt-dCasRx foram usadas para executar as cruzes, 2) todas as taxas de herança transheterozigous triplas da geração F2 coincidiram com a taxa de herança mendeliana esperada de 25%, e 3) o fenótipo de letalidade transheterozigous da geração F2 CasRx foi moderadamente reduzido. No experimento de segmentação branca, das taxas de herança mendeliana de 25% esperadas nas transheterozigotes triplas F2, apenas 0,57% foram observadas moscas adultas viáveis (UASt-CasRx/+; gRNAw/GMR-Gal4), todas com pigmentação específica do olho grave e fenótipos de morfologia (Figura 4A e 4B). Para a cruz de alvo branco, a taxa de herança F2 tríplice transheterozigous que expressa CasRx foi significativamente menor do que a do grupo de controle triplo transheterozigos (27,6%) (Figura 4A). No experimento de alvo notch, casRx-expressing triplo tranheterozygous carregando todos os três transgenes foram 100% letais, enquanto a taxa de herança de controle dCasRx foi de 29,3% (Figura 4A). No experimento de mira amarela, f2 triplo transheterozigous CasRx-expressing, gRNAy, e y-GAL4 mostrou redução marginal de pigmento de quitina como pequenas manchas de cutícula amarela no tórax e abdômen com uma taxa de herança de 2,67%, muito menor do que a do grupo de controle dCasRx (25,2%) (Figura4A) (Figura4A ). Todos os dCasRx controlam moscas transheterozigous triplas não apresentaram fenótipos óbvios como as moscas que expressam CasRx, indicando que a atividade catalítica do CasRx contribuiu para os fenótipos observados. A baixa taxa de herança no grupo transheterozigos triplo CasRx sugeriu que existem duas fontes de toxicidade na segmentação casrx RNA: uma está associada à alta expressão do CasRx, a toxicidade da qual foi reduzida pela expressão restritiva CasRx, a outra está associada à atividade colateral. Juntos, esses resultados mostraram que o sistema CasRx pode alcançar o direcionamento in vivo RNA específico do tecido, aproveitando o sistema clássico Gal4/UASt e, entretanto, reduzir a toxicidade. No entanto, a toxicidade e os fenótipos de letalidade ocasionais ainda foram observados em um nível mais baixo de gravidade em comparação com o das abordagens onipresentes, indicando que a atividade de decote colateral está associada à toxicidade.
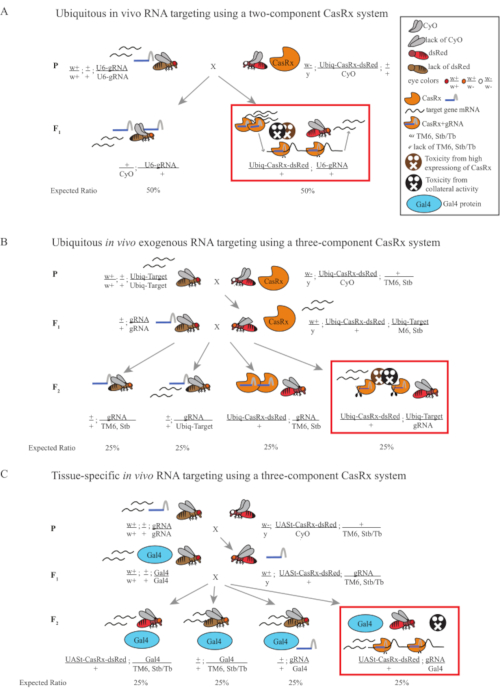
Figura 1: Visão geral geral do alvo do RNA usando um sistema Cas13D. (A) Esquemas da cruz genética de um passo na onipresente segmentação in vivo RNA usando um sistema CasRx de dois componentes. (B) Esquemas de uma cruz genética de duas etapas no onipresente direcionamento de RNA exógeno in vivo usando o sistema CasRx de três componentes. (C) Esquemas de uma cruz genética de duas etapas no alvo in vivo RNA específico do tecido usando um sistema CasRx de três componentes. Clique aqui para ver uma versão maior desta figura.
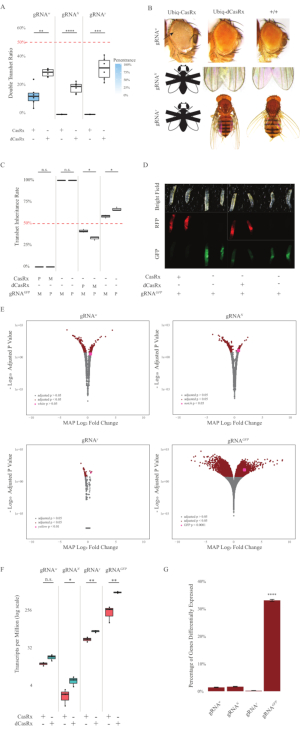
Figura 2: Onipresente in vivo RNA targeting usando um sistema CasRx de dois componentes (reimpressa5). (A) Percentuais totais de herança de moscas transheterozigous herdando Ubiq-CasRx (ou Ubiq-dCasRx) e gRNAs. Sombreamento azul no enredo da caixa indica penetração de fenótipo. (B) Fenótipos de moscas transheterozigous. As flechas indicam necrose tecidual no olho. Mosca preta e branca marcada com ''X'' representa letalidade. (C) Percentua totais de herança de moscas transheterozigous de cruzes bidirecionais entre as moscas Ubiq-CasRx (ou Ubiq-dCasRx) e gRNAGFP-OpIE2-GFP. M, herança materna de CasRx; P, herança paterna de CasRx. (D) Larvas de F1 progenies na cruz paterna. (E) As estimativas máximas de a posteriori das transcrições para a alteração da dobra logarítmica. O gasoduto DESeq2 foi usado. (F) Transcrições por milhão (TPM) direcionadas com CasRx ou dCasRx. (G) CasRx-depentent percentual de transcrição despententetada diferencialmente expressa das transcrições. Clique aqui para ver uma versão maior desta figura.
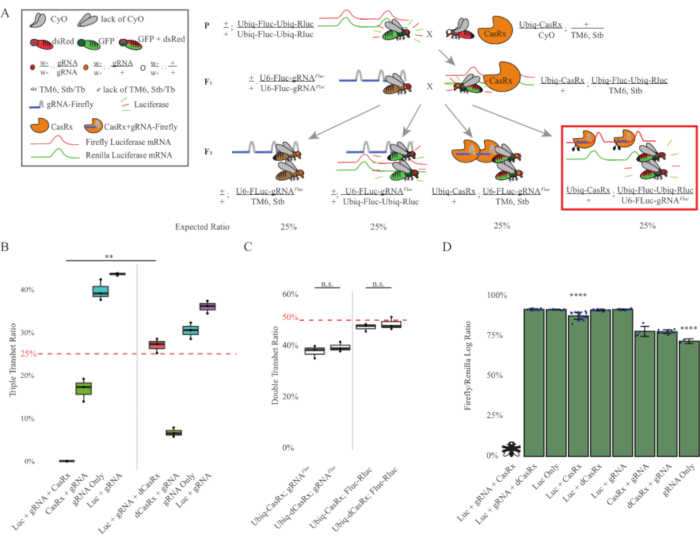
Figura 3: Onipresente in vivo exógeno RNA targeting usando um sistema CasRx de três componentes. (A) Esquemas da cruz genética de duas etapas. (B) Percentuais totais de herança para todos os genótipos emergentes na geração F2 . Herdar todos os três transgenes (Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc e gRNAFLuc) na progênero F2 resultou em 100% de letalidade e foi significativamente menor em comparação com o grupo de controle de transheterozygotes triplo Ubiq-dCasRx (p = 0,001, t-test). (C) Carregar apenas Ubiq-CasRx/gRNAFluc ou Ubiq-CasRx e Ubiq-Fluc-Ubiq-Rluc por si só não levou a letalidade grave, e as razões de herança entre Ubiq-CasRx e Ubiq-dCasRx transheterozygotes não foram significativamente diferentes (p = 0,41 e p = 0,51, respectivamente, t-test). (D) Proporções de luciferase normalizando as leituras fluc para leituras de Rluc. Moscas transheterozigous triplas expressando Ubiq-CasRx, Ubiq-Fluc-Ubiq-Rluc, gRNAFLuc eram letais embrionárias, que era representada por uma mosca com um "X", e como resultado a expressão luciferase não foi medida. A razão Fluc/Rluc de Ubiq-CasRx/+, Ubiq-Fluc-Ubiq-Rluc/TM6, Stb transheterozygotes foi significativamente menor que a dos outros grupos de expressões Ubiq-Fluc-Ubiq-Rluc (p = 1,2e-06 ou inferior, t-test). Os resultados do grupo somente gRNAFLuc foram significativamente inferiores aos de todos os outros grupos (p = 1,2e-06 ou inferior, t-test). Clique aqui para ver uma versão maior desta figura.
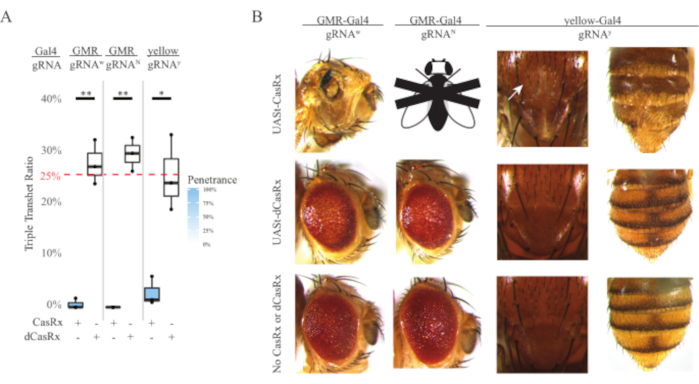
Figura 4: Segmentação in vivo RNA específica do tecido usando um sistema CasRx de três componentes (reimpressa5) (A) Percentual total de herança de moscas transheterozigous triplas transportando três transgenes (UASt-CasRx ou UASt-dCasRx, gRNAs e Gal4-driver. (B) Fenótipos das moscas transheterozigous triplas. A seta branca indica redução do pigmento de quitina no tórax. Mosca preta e branca marcada com ''X'' representa letalidade. Clique aqui para ver uma versão maior desta figura.
| Construir | Descrição | Cartilha | Sequência de primer (5' a 3') | Modelo PCR |
| OA-1050E | CasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA AGAA | pNLS-RfxCas13d-NLS-HA (pCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTGG AACA | |||
| OA-1050R | dCasRx | 1050E. C3 | TACTAATTTTCCAC ATCTCTATTTTGAC CCGCAGATTAATTA ATGAGCCCCAAGA AGAA | pNLS-dRfxCas13d-NLS-HA (pdCasRx) |
| 1050E. C4 | CAATTGATTTGTTA TTTTAAAAACGATT CATTCTAGCTAGCT TAAGCGTAATCTG GAACA | |||
| OA-1050L | Promotor do UASt | 1041.C9 | GCGGGTTCTCGA CGGTCACGGCGG GCATGTCGACGC GGCCGCAACCAA CAACACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCACC TTTCTCTCTTTCTCT TGGGGCTCATGT TTAAACCCAATT CCCTATTCAGA | |||
| CasRx | 1050L. C1 | AATACAAGAAGA GAACTCTGAATA GGGAATTGGGT TTAAACATGAGC CCCAAGAAGAA | pCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTA GCTAGCTTAAG CGTAATCTGGA ACA | |||
| OA-1050S | Promotor do UASt | 1041.C9 | GCGGGTTCTC GACGGTCACG GCGGGCATGT CGACGCGGCC GCAACCAACAA CACTAGTAG | pJFRC81 |
| 1041.C11 | CTGGCCTCCA CCTTTCTCTTC TTCTTGGGGCT CATGTTTAAAC CCAATTCCCTA TTCAGA | |||
| dCasRx | 1050L. C1 | AATACAAGAAG AGAACTCTGAAT AGGGAATTGGG TTTAAACATGAG CCCCAAGAAGAA | pdCasRx | |
| 1050E. C4 | CAATTGATTTGT TATTTTAAAAAC GATTCATTCTAG CTAGCTTAAGCG TAATCTGGAACA | |||
| OA-1043 | U6:3 promotor | 1043.C1 | GGGAATTGGGA ATTGGGCAATAT TTAAATGGCGGC GCGCCGAATTCT TTTTTGCTCACCT | Addgene plasmid #164586 |
| 1043.C23 | ACACTAGTGGAT CTCTAGAGGTAC CGTTGCGGCCG CAAAAAAAGTTGT AATAGCCCCTCA AAACTGGACCTT CCACAACTGCAG CCGACGTTAAAT TGAAA | |||
| OA-1052B | Promotor da Ubiq | 1052B. C1 | GGGAATTGGGCA ATATTTAAATGGC GGCTGCAGC GCAGATCGCCGAT | Addgene plasmid #112686 |
| 1052B. C2 | TTTCTTTATGTTT TTGGCGTCTTCC ATCCTAGGTCTG CGGGTCAAAATA GAGATG | |||
| T2A-eGFP | 908A1 | ATAAAGGCCAAG AAGGGCGGAAA GATCGCCGTGG AGGGCAGAGGA AGTCTTCTAACAT GC | Addgene plasmid #112686 | |
| 908A2 | TTGTTATTTTAAAA ACGATTCATTCTA GGCGATCGCTTA CTTGTACAGCTC GTCCATGCC | |||
| Promotor reverso da Ubiq | 908A3 | ACCGTGACCTAC ATCGTCGACACTA GTGGATCTCTAGA CGCGCAGATCGC CGATG | Addgene plasmid #112686 | |
| 908A4 | GGATCATAAACTT TCGAAGTCATGC GGCCGCTCTGCG GGTCAAAAATAGAG ATGT |
Tabela 1: Lista de construtos moleculares e primers utilizados neste estudo. Esta lista inclui todos os construtos (tanto o ID quanto a descrição) e os primers associados de cada construção (tanto o ID quanto as sequências (5' a 3')) e os modelos utilizados.
Discussão
Com três diferentes projetos de aplicação do sistema CasRx, este trabalho demonstrou direcionamento in vivoprogramável de RNA em moscas. As diferentes estratégias atendem às diferentes necessidades de projeto, como direcionamento genético endógeno versus exógeno e segmentação onipresente versus RNA específica do tecido. Os efeitos do direcionamento do RNA incluíram alterações fenotípicas específicas do gene alvo, redução da transcrição do RNA e fenótipos de letalidade ocasionais associados à alta expressão da proteína CasRx e atividade colateral. No geral, esses resultados mostraram que o sistema CasRx é capaz de direcionar a redução da transcrição do RNA no nível do organismo de forma programável e eficiente.
Um dos principais fatores na personalização bem-sucedida do sistema CasRx é o design de gRNAs. Especificamente, o seguinte conselho deve ser cuidado: a sequência de destino é de cerca de 30 nucleotídeos de comprimento, o comprimento dos trechos poli-U na sequência de destino é de 4 pares de base ou menos, a sequência de destino GC o conteúdo está na faixa de 30% a 70%, a sequência de alvo não é prevista para formar estruturas fortes de grampo de RNA, e a sequência de alvo contém estrutura mínima prevista de RNA secundária ou terciária5.
Além dos projetos de gRNA, a etapa genética da mosca em cada protocolo também é fundamental em uma implementação bem sucedida. A presença ou falta dos fenótipos definidos passados pelos pais nos progens são importantes para identificar e quantificar fenótipos induzidos pelo sistema CasRx nos progens transheterozigos. Além disso, configurar cruzes de controle usando as moscas dCasRx em paralelo também são úteis na excluindo fenótipos não específicos nos progens transheterozigos.
Vale ressaltar que esses resultados revelaram problema de toxicidade introduzido pela onipresente expressão da proteína CasRx e dCasRx na mosca, uma limitação do sistema CasRx. A expressão onipresente de CasRx ou dCasRx sob o promotor Ubiq sozinho, sem gRNAs, veio com custos de fitness não trivial, já que nem as moscas Ubiq-CasRx nem Ubiq-dCasRx poderiam ser estabelecidas como linhas homozigas. Pelo contrário, as moscas UASt-CasRx e UASt-dCasRx podem ser estabelecidas como estoques homozigos saudáveis, embora devido ao desenho do esquema cruzado eles foram mantidos como estoques de equilíbrio duplo, fato que suporta a existência de toxicidade induzida pela onipresente expressão proteica CasRx. Outra evidência de apoio é que em experimentos de controle envolvendo dCasRx, que é cataticamente inativo, as porcentagens de moscas carregando tanto dCasRx quanto construções de gRNA do número total de moscas na geração F1 foram consistentemente inferiores a 50%, a razão esperada com base na genética mendeliana se não houvesse toxicidade associada ao dCasRx. Isso indicou que a expressão onipresente do dCasRx, juntamente com os gRNAs, induz a toxicidade na mosca, resultando em uma razão de herança menor do que a esperada. As razões de herança do UASt-dCasRx transheterozigous, gRNA, GAL4 seguiram a genética mendeliana, o que sugere novamente a toxicidade induzida especificamente pela expressão onipresente das proteínas CasRx e dCasRx. A toxicidade no sistema CRISPR/Cas não é nova. Altas quantidades de proteína Cas9 tem se mostrado tóxicas em vários organismos, incluindo moscas29,30,31,32. Um estudo recente desenvolveu um sistema GAL4/UAS personalizado que pode ajustar a quantidade de proteína Cas9 expressa em moscas adicionando um quadro de leitura aberta de comprimento variado entre a sequência UAS e a sequência Cas9 na construção UAS-Cas933. Portanto, vale a pena explorar maneiras de reduzir a toxicidade induzida pelo CasRx, afinando o nível de expressão da proteína CasRx.
Além da toxicidade induzida pela expressão onipresente das proteínas CasRx e dCasRx, os resultados também mostraram letalidade ligada aos efeitos colaterais não específicos fora do alvo do sistema CasRx, uma característica de muitos sistemas CRISPR1,2,7,34. Em algumas das moscas casrx e não essenciais que expressam gRNA dupla ou triplamente transheterozgous, por exemplo, ao atingir notch, as moscas CasRx transheterozigous têm níveis significativamente mais baixos de taxa de sobrevivência em comparação com as moscas transheterozgyous dCasRx. Na análise rna-seq dessas moscas transheterozigos que expressam CasRx e gRNA, tanto a redução dos níveis de transcrição genética-alvo quanto a redução de transcrições genéticas não-alvo foram observadas. Esses efeitos colaterais foram dependentes de CasRx e dependentes de alvos, pois só foram observados em moscas transheterozigous expressando tanto a proteína CasRx quanto o gRNA. Vale ressaltar que um dos genes-alvo, branco, apresentou apenas uma redução limitada e não estatisticamente significativa nas transcrições quando o gene branco foi alvo do CasRx, que contrastava com o fenótipo de redução de pigmento claro. Acredita-se que isso possa ser devido ao fato de que 1) o tempo da coleta de amostras de RNA-seq não estava bem alinhado com o momento em que o gene branco atinge seu pico de expressão durante o desenvolvimento precoce, e 2) a expressão localizada do gene branco nos olhos torna desafiador coletar os tecidos relevantes durante a fase de desenvolvimento precoce quando apenas a coleta de amostras de corpo inteiro é viável. Para reduzir a atividade colateral no sistema CasRx, estudos futuros são solicitados a compreender completamente os mecanismos subjacentes ao sistema de fenômenos fora do alvo no nível do organismo.
Curiosamente, um estudo recente35 descrevendo ferramentas Cas13 direcionadas ao RNA em moscas parecia amenizar a toxicidade geral associada à expressão CasRx, por várias razões possíveis. Em primeiro lugar, os autores recodificaram os transgênicos Cas13 para otimizar a expressão em Drosophila e utilizaram um promotor mais fraco (actin 5C) em comparação com o promotor de ubiquitina utilizado no presente estudo, provavelmente levando a níveis mais baixos de expressão Cas13 e, portanto, menos toxicidade. De fato, isso é apoiado pelas observações de que a expressão CasRx e dCasRx, impulsionadas pelo UASt, não era, por si só, tóxica, pois este estudo (e os autores em 35) não observaram nenhuma letalidade óbvia nas moscas UASt-CasRx. Além disso, esses autores codificaram suas gRNAs de forma diferente em relação a este estudo, o que pode ter afetado sua expressão e reduzido a toxicidade do sistema em moscas Cas13/gRNA transheterozigous. Por exemplo, em seu estudo dois gRNAs foram expressos usando o promotor U6:3 e ladeados por tRNAs para permitir o processamento de gRNA após o amadurecimento do tRNA sem exigir CasRx35. Por outro lado, neste estudo, os gRNAs foram codificados como matrizes que visam até 4 locais por gene e imitam a estrutura endógena da matriz Cas13 encontrada em bactérias, o que requer que a enzima Cas13 processe cada gRNA. Essas diferentes abordagens podem ter levado a diferenças nos níveis de expressão do gRNA e outros fatores que podem ter efeitos inerentes à toxicidade de todo o sistema. Finalmente, Huynh et al. atingiram genes diferentes dos alvos no presente estudo, que resultam em diferenças na interação alvo-Cas/gRNA e atividade colateral e podem ter efeitos sobre os níveis observados de letalidade. Essas diferenças na toxicidade observada justificam uma investigação mais aprofundada para identificar maneiras de que os sistemas globais possam ser melhorados.
No geral, este estudo é a primeira demonstração de um sistema de Cas programável programável geneticamente codificado funcional em D. melanogaster, embora uma otimização adicional do sistema CasRx (em linha com o que é relatado35) será necessária para reduzir ainda mais a letalidade fora do alvo e aumentar a eficácia do decote casrx no alvo. O direcionamento de RNA com enzimas Cas é um campo em rápida evolução com muitas aplicações potenciais que vão desde o controle de vetores de insetos até usos terapêuticos1,2,3,4,5,6,7, e este protocolo oferece um pacote inicial para qualquer pessoa interessada em projetar seu primeiro sistema CasRx em moscas, ao mesmo tempo em que é compatível com a personalização e otimização do sistema. Os exemplos aqui apresentados demonstram uma série de resultados que se pode encontrar durante a implementação deste sistema in vivo e pode servir de benchmark para outros usuários na avaliação do desempenho do sistema CasRx em seus aplicativos.
Divulgações
A O.S.A é uma das fundadoras da Agragene, Inc., tem participação acionária e atua no Conselho Consultivo Científico da empresa. Os termos deste acordo foram revisados e aprovados pela Universidade da Califórnia, San Diego, de acordo com suas políticas de conflito de interesses. Todos os outros autores não declaram interesses concorrentes.
Agradecimentos
Este trabalho foi apoiado em parte por financiamento de uma Bolsa do Programa de Genes Seguros darpa (HR0011-17-2-0047), e prêmios NIH (R21RAI149161A, DP2AI152071) concedidos à O.S.A.
Materiais
| Name | Company | Catalog Number | Comments |
| 100% Grape Juice | Welch Foods Inc. | N/A | |
| Active Dry Yeast (908g) | Red Star Yeast Company, LLC | N/A | |
| DMC4500 color camera | Leica Microsystems | DMC4500 | |
| Dual-Luciferase Reporter Assay System | Promega | E1910 | |
| GAL4-GMR flies | Bloomington Drosophila Stock Center | 29967 | |
| GAL4-y flies | Bloomington Drosophila Stock Center | 44373 | |
| Glomax 20/20 Luminometer | Promega | E5331 | |
| Illumina HiSeq2500 Sequencer | Illumina, Inc. | HiSeq2500 | |
| M165FC fluorescent stereomicroscope | Leica Microsystems | M165FC | |
| Nanodrop OneC UV-vis spectrophotometer | ThermoFisher | NDONEC-W | |
| NEBNext Ultra II RNA Library Prep Kit | New England Biolabs, Inc. | E7770 | |
| plasmid # 112686 | Addgene | 112686 | |
| plasmid # 112688 | Addgene | 112688 | |
| plasmid # 132416 | Addgene | 132416 | |
| plasmid # 132417 | Addgene | 132417 | |
| plasmid # 132419 | Addgene | 132419 | |
| plasmid # 132420 | Addgene | 132420 | |
| plasmid # 132421 | Addgene | 132421 | |
| plasmid # 132422 | Addgene | 132422 | |
| plasmid # 132425 | Addgene | 132425 | |
| plasmid # 132426 | Addgene | 132426 | |
| plasmid # 133304 | Addgene | 133304 | |
| plasmid # 164586 | Addgene | 164586 | |
| plasmid #132418 | Addgene | 132418 | |
| plasmid pJFRC81 | Addgene | 36432 | |
| Qiagen RNeasy Mini Kit | Qiagen | 74104 | |
| Restriction endonucleases AscI | New England Biolabs Inc. | R0558L | |
| Restriction endonucleases NotI | New England Biolabs Inc. | R0189L | |
| Restriction endonucleases PacI | New England Biolabs Inc. | R0547L | |
| Restriction endonucleases PstI | New England Biolabs Inc. | R0140L | |
| Restriction endonucleases SwaI | New England Biolabs Inc. | R0604L | |
| Restriction endonucleases XbaI | New England Biolabs Inc. | R0145L | |
| RNA 6000 Pico Kit for Bioanalyzer | Agilent Technologies | 5067-1513 | |
| Turbo DNase | Invitrogen | AM2238 | |
| U6-3:4-gRNA-Fluc flies | Bloomington Drosophila Stock Center | 84125 | |
| U6-3:4-gRNA-GFP; OpIE2-GFP flies | Bloomington Drosophila Stock Center | 84986 | |
| U6-3:4-gRNA-N flies | Bloomington Drosophila Stock Center | 84122 | |
| U6-3:4-gRNA-w flies | Bloomington Drosophila Stock Center | 84124 | |
| U6-3:4-gRNA-y flies | Bloomington Drosophila Stock Center | 84123 | |
| UASt-CasRx flies | Bloomington Drosophila Stock Center | 84121 | |
| UASt-dCasRx flies | Bloomington Drosophila Stock Center | 84120 | |
| Ubiq-CasRx flies | Bloomington Drosophila Stock Center | 84118 | |
| Ubiq-dCasRx flies | Bloomington Drosophila Stock Center | 84119 | |
| Ubiq-Firefly-T2A-eGFP-Ubiq-Renilla flies | Bloomington Drosophila Stock Center | 84127 | |
| Zymoclean Gel DNA Recovery Kits | Zymo Research Corporation | D4007 |
Referências
- Adli, M. The CRISPR tool kit for genome editing and beyond. Nat Communications. 9, 1911 (2018).
- Abudayyeh, O., et al. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 353 (6299), 5573 (2016).
- East-Seletsky, A., O'Connell, M., Burstein, D., Knott, G., Doudna, J. RNA Targeting by Functionally Orthogonal Type VI-A CRISPR-Cas Enzymes. Molecular Cell. 66 (3), 373-383 (2017).
- Konermann, S., Lotfy, P., Brideau, N., Oki, J., Shokhirev, M., Hsu, P. Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell. 173 (3), 665-676 (2018).
- Buchman, A., Brogan, D., Sun, R., Yang, T., Hsu, P., Akbari, O. Programmable RNA Targeting Using CasRx in Flies. The CRISPR Journal. 3 (3), 164-176 (2020).
- Kushawah, G., et al. CRISPR-Cas13d Induces Efficient mRNA Knockdown in Animal Embryos. Developmental Cell. 54 (6), 805-817 (2020).
- Abudayyeh, O., et al. RNA targeting with CRISPR-Cas13. Nature. 550, 280-284 (2017).
- Perrimon, N., Ni, J., Perkins, L. In vivo RNAi: today and tomorrow. Cold Spring Harbor Perspectives in Biology. 2, 003640 (2010).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
- Ni, J., et al. A Drosophila resource of transgenic RNAi lines for neurogenetics. Genetics. 182 (4), 1089-1100 (2009).
- Ni, J., et al. A genome-scale shRNA resource for transgenic RNAi in Drosophila. Nature Methods. 8, 405-407 (2011).
- Ni, J., et al. Vector and parameters for targeted transgenic RNA interference in Drosophila melanogaster. Nature Methods. 5, 49-51 (2008).
- Heigwer, F., Port, F., Boutros, M. RNA Interference (RNAi) Screening in Drosophila. Genetics. 208 (3), 853-874 (2018).
- Kulkarni, M., et al. Evidence of off-target effects associated with long dsRNAs in Drosophila melanogaster cell-based assays. Nature Methods. 3, 833-838 (2006).
- Ma, Y., Creanga, A., Lum, L., Beachy, P. Prevalence of off-target effects in Drosophila RNA interference screens. Nature. 443, 359-363 (2006).
- Perrimon, N., Mathey-Prevot, B. Matter arising: off-targets and genome-scale RNAi screens in Drosophila. Fly. 1 (1), 1-5 (2007).
- Markstein, M., Pitsouli, C., Villalta, C., Celniker, S., Perrimon, N. Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nature Genetics. 40, 476-483 (2008).
- Champer, J., Buchman, A., Akbari, O. Cheating evolution: engineering gene drives to manipulate the fate of wild populations. Nature Review Genetics. 17 (3), 146-159 (2016).
- Buchman, A., et al. Broad dengue neutralization in mosquitoes expressing an engineered antibody. PLoS Pathogens. 16 (4), 1008103 (2020).
- Mathur, G., Sanchez-Vargas, I., Alvarez, D., Olson, K., Marinotti, O., James, A. Transgene-mediated suppression of dengue viruses in the salivary glands of the yellow fever mosquito, Aedes aegypti. Insect Molecular Biology. 19 (6), 753-763 (2011).
- Franz, A., et al. Engineering RNA interference-based resistance to dengue virus type 2 in genetically modified Aedes aegypti. Proceedings of the National Academy of Sciences of the United States of America. 103 (11), 4198-4203 (2006).
- Yen, P., James, A., Li, J., Chen, C., Failloux, A. Synthetic miRNAs induce dual arboviral-resistance phenotypes in the vector mosquito Aedes aegypti. Communications Biology. 1, 11 (2018).
- Buchman, A., et al. Engineered resistance to Zika virus in transgenic Aedes aegypti expressing a polycistronic cluster of synthetic small RNAs. Proceedings of the National Academy of Sciences of the United States of America. 116 (9), 3656-3661 (2019).
- . Addgene Available from: https://www.addgene.org/protocols/restriction-digest/ (2020)
- Gibson, D., Young, L., Chuang, R., Venter, J., Hutchison, C., Smith, H. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nature Methods. 6 (5), 343-345 (2009).
- . Addgene Available from: https://www.addgene.org/protocols/pcr/ (2020)
- . Indiana University Bloomington Available from: https://bdsc.indiana.edu/information/recipes/bloomfood.htmL (2020)
- Dobin, A., et al. STAR:ultrafast universal RNA-seq aligner. Bioinformatics. 29 (1), 15-21 (2013).
- Jiang, W., Brueggeman, A., Horken, K., Plucinak, T., Weeks, D. Successful transient expression of Cas9 and single guide RNA genes in Chlamydomonas reinhardtii. Eukaryotic Cell. 13 (11), 1465-1469 (2014).
- Poe, A., et al. Robust CRISPR/Cas9-Mediated Tissue-Specific mutagenesis reveals gene redundancy and perdurance in Drosophila. Genetics. 211 (2), 459-472 (2019).
- Port, F., Chen, H., Lee, T., Bullock, S. Optimized CRISPR/Cas tools for efficient ger mLine and somatic genome engineering in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 111 (29), 2967-2976 (2014).
- Yang, S., Li, S., Li, X. Shortening the Half-Life of Cas9 maintains its gene editing ability and reduces neuronal toxicity. Cell Reports. 25 (10), 2653-2659 (2018).
- Port, F., et al. A large-scale resource for tissue-specific CRISPR mutagenesis in Drosophila. eLife. 9, 53865 (2020).
- Zhang, X., Tee, L., Wang, X., Huang, Q., Yang, S. Off-target Effects in CRISPR/Cas9-mediated Genome Engineering. Molecular Therapy - Nucleic Acids. 4 (17), 264 (2015).
- Huynh, N., Depner, N., Larson, R., King-Jones, K. A versatile toolkit for CRISPR-Cas13-based RNA manipulation in Drosophila. Genome Biology. 21, 279 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados