A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
RGBradford: כימות חלבונים עם מצלמת סמארטפון
In This Article
Summary
מאמר זה מספק פרוטוקול לכימות חלבונים באמצעות בדיקת ברדפורד וטלפון חכם כמכשיר אנליטי. ניתן לכמת את רמות החלבון בדגימות באמצעות נתוני צבע שחולצו מתמונה של צלחת מיקרו שצולמה באמצעות טלפון חכם.
Abstract
כימות חלבונים הוא הליך חיוני במחקר מדעי החיים. בין מספר שיטות אחרות, מבחן ברדפורד הוא אחד הנפוצים ביותר. בגלל נפוצותו, המגבלות והיתרונות של מבחן ברדפורד דווחו באופן ממצה, כולל מספר שינויים של השיטה המקורית כדי לשפר את הביצועים שלה. אחד השינויים בשיטה המקורית הוא השימוש במצלמת סמארטפון כמכשיר אנליטי. תוך ניצול שלוש הצורות של צבע Coomassie Brilliant Blue הקיימות בתנאים של מבחן ברדפורד, מאמר זה מתאר כיצד לכמת במדויק חלבון בדגימות באמצעות נתוני צבע שחולצו מתמונה אחת של מיקרו-לוחית. לאחר ביצוע הבדיקה במיקרו-לוחית, התמונה מצולמת באמצעות מצלמת סמארטפון, ונתוני צבע RGB מופקים מהתמונה באמצעות יישום תוכנה חינמי וקוד פתוח לניתוח תמונות. לאחר מכן, היחס בין עוצמה כחולה לירוקה (בסולם RGB) של דגימות עם ריכוזים לא ידועים של חלבון משמש לחישוב תכולת החלבון בהתבסס על עקומה סטנדרטית. לא נצפה הבדל משמעותי בין ערכים המחושבים באמצעות נתוני צבע RGB לבין ערכים המחושבים באמצעות נתוני ספיגה רגילים.
Introduction
ללא קשר לשימוש במורד הזרם (למשל, ELISA, קינטיקה של אנזימים, כתמים מערביים, טיהור חלבונים וספקטרומטריית מסות), כימות חלבונים חיוני לניתוח מדויק במעבדות מדעי החיים. בנוסף לשימוש בהם כקריאות משניות (כלומר, כדי לחשב רמות יחסיות של אנליטים לכל מסת חלבון), רמות חלבון במדגם יכולות להיות גם הפלט הרצוי עצמו. לדוגמה, אפשר להתעניין ברמות החלבון במשאבי מזון1 או בשתן2. ישנן שיטות רבות זמינות למדידת ריכוז החלבון בדגימות3, כולל קריאות ספיגת UV ישירות4, כלציה חלבון-נחושת 5,6, בדיקות קולורימטריות קושרות צבע חלבון7, ומבחני פלורסנט קושרי צבע חלבון8. הרלוונטיות של כימות החלבונים באה לידי ביטוי בנוכחותם של שני מאמרים המתארים שיטות מדידת חלבונים 5,7 בטופ-3 של הספרות המצוטטת ביותר 9,10. למרות העובדה שמחברים רבים מזניחים את הציטוט האמיתי שלהם על ידי ציטוט הפניות לא ראשוניות או לא מצטטים שום דבר בכלל, המאמרים המקוריים המתארים את בדיקת החלבון של לורי ואת בדיקת חלבון ברדפורד מסתכמים >200,000 ציטוטים כלאחד 10.
הפופולריות של מבחן ברדפורד נובעת מהמחיר, הפשטות, המהירות והרגישות שלו. הבדיקה מבוססת על האינטראקציה בין חלבונים לבין הצבע Coomassie Brilliant Blue G בתנאים חומציים. בתנאי הבדיקה (כלומר, pH נמוך), הצבע קיים בשלוש צורות: צורה קטיונית אדומה עם λmax ב 470 ננומטר; צורה נייטרלית ירוקה עם λmax ב 650 ננומטר; וצורה אניונית כחולה עם λmax ב-590 ננומטר11,12 (איור 1). הצורה הקטיונית שולטת בהיעדר חלבונים. כאשר חלבונים מתקשרים עם הצבע, הם מייצבים את הצורה האניונית הכחולה, וגורמים לשינוי ניכר בצבע התמיסה, מחום לכחול. בדרך כלל, השינוי בריכוז הצורה הכחולה של הצבע מכומת באופן ספקטרופוטומטרי, שספיגתו ב 590-595 ננומטר פרופורציונלית לכמות החלבון בבדיקה.
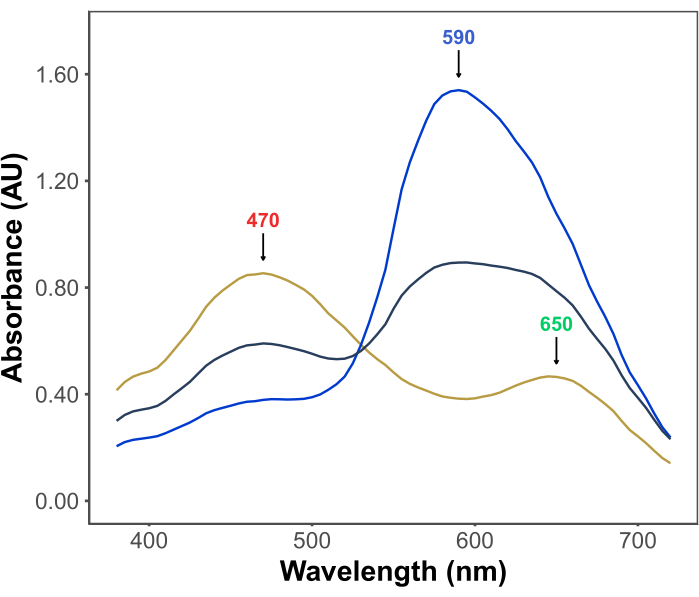
איור 1: ספקטרום בליעת G כחול מבריק של קומאסי בתנאים של ניסוי ברדפורד. שלוש הפסגות העיקריות מסומנות בחצים המציינים את λmax של הצורות האדום (470 ננומטר), הירוק (650 ננומטר) והכחול (590 ננומטר) של הצבע. ספקטרום תועד בהיעדר חלבון (קו צהוב) ובנוכחות 3 מיקרוגרם (קו אפור) ו -10 מיקרוגרם (קו כחול) של אלבומין בסרום בקר. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
השימוש הנרחב בבדיקת ברדפורד הוביל לזיהוי מספר מגבלות (למשל, תגובות משתנות לחלבונים שונים11, והפרעות על ידי ליפידים13 ודטרגנטים7) ופיתוח שינויים לשיפור ביצועיו (למשל, הוספת דטרגנטים14,15, אלקליניזציה14,16 ושימוש ביחס הספיגות17). בנוסף לשינויים בבדיקה עצמה, השימוש במכשירים חלופיים, כגון טלפונים חכמים או מצלמות, כדי ללכוד אותות אנליטיים תוארו גם 18,19,20. ואכן, פיתוח שיטות המשתמשות בסמארטפונים כאנלייזרים כימיים ניידים היה תחום מחקר פעיל. המוטיבציה לשימוש בסמארטפונים נובעת מהמחיר הנוח, הניידות, קלות השימוש והזמינות הרחבה של מכשירים אלה.
מאמר זה מספק פרוטוקול לכימות חלבונים באמצעות בדיקת RGBradford20, המשתמשת בטלפון חכם כמכשיר אנליטי. בניגוד לפרסום המקורי של RGBradford20, כאן הוצג הליך המייעל את תהליך מיצוי הצבע. זה כרוך בשימוש ביישום תוכנה זמין באופן חופשי כדי לחלץ מידע צבע מכל באר של תמונת microplate באופן אוטומטי, חוסך זמן ומאמץ משמעותיים. זוהי חלופה לשיטה הקודמת של רכישה ידנית של נתוני צבע מכל באר אחד אחד באמצעות יישום תוכנת עורך גרפי20. בסופו של דבר, ניתן לכמת את רמות החלבון בדגימות באמצעות נתוני צבע שחולצו מתמונה של מיקרו-צלחת שצולמה באמצעות טלפון חכם.
Protocol
1. הכנת מגיב בדיקת חלבון ברדפורד
- להמיס 100 מ"ג של Coomassie Brilliant Blue G באתנול של 50 מ"ל 95% (עם V). מערבבים עד ש-Coomassie Brilliant Blue G מומס לחלוטין.
אזהרה: אתנול דליק וגורם לגירוי בעיניים. הימנעו מלהבות והשתמשו במשקפי מגן. - לתמיסה הקודמת, הוסף 100 מ"ל של 85% (w/v) חומצה זרחתית בזהירות.
אזהרה: חומצה זרחתית היא קורוזיבית למתכות וגורמת לקורוזיה בעור, נזק חמור לעיניים ורעילות חריפה דרך הפה. יש ללבוש כפפות ומשקפי מגן. - הוסיפו את התמיסה המכילה Coomassie Brilliant Blue G, אתנול וחומצה זרחתית באיטיות ל-600 מ"ל מים שעברו דה-יוניזציה.
- לדלל את התמיסה לנפח סופי של 1,000 מ"ל. קנה מידה למעלה או למטה בהתאם למספר הדגימות שיש לנתח. כפי שתואר בשיטה7 המקורית, הריכוזים הסופיים בריאגנט ברדפורד העובד צריכים להיות 0.01% (w/v) Coomassie Brilliant Blue G, 4.7% (w/v) אתנול ו-8.5% (w/v) חומצה זרחתית.
- הסר כל חומר בלתי מסיס המסנן דרך נייר סינון (נייר Whatman #1 או שווה ערך).
- המגיב יציב למשך מספר שבועות כאשר הוא מאוחסן בטמפרטורת החדר (RT) ומוגן מפני אור. סנן לפי הצורך מכיוון שמשקעים עשויים להיווצר עם הזמן.
הערה: לחלופין, ריאגנטים מוכנים לדלל ולהשתמש בברדפורד זמינים מסחרית. בצע את הוראות היצרן להכנת מגיב והמשך לשלב הבא.
2. הכנת תמיסות סטנדרטיות חלבוניות
- הכינו תמיסת מלאי (עיין בשלב 2.4) של חלבון מבודד לשימוש כסטנדרט. חלבון זול ונפוץ הוא אלבומין בסרום בקר (BSA). אפשרויות אחרות הן ovalbumin ו globulin גמא בקר.
- אם הספיגה המולרית של החלבון המשמש כסטנדרט ידועה, בדוק את ריכוז תמיסת המניות בספקטרופוטומטר.
- עבור BSA, נוסחה נפוצה היא BSA (mg/mL) = (A280/6.6) × 10, כאשר A280 הוא הספיגה ב 280 ננומטר עם אורך נתיב של 1 ס"מ קריאה כנגד ריק מתאים (כלומר,ε 2801% = 6.6)7. לדוגמה, 0.8 מ"ג / מ"ל BSA יש ספיגה של 0.528 ב 280 ננומטר.
- כדי ליצור את העקומה הסטנדרטית, הכינו מספר דילולים של BSA בטווח של 0.025 מ"ג/מ"ל ו-1.0 מ"ג/מ"ל. אלה יביאו 0.25-10 מיקרוגרם של BSA לכל באר לאחר הוספת נפח מדגם של 10 μL לכל באר.
הערה: יש להכין תמיסות סטנדרטיות של חלבונים בתווך בעל אותו הרכב (ריכוז סופי) של המדיום המשמש להכנת דגימות.
3. בדיקה
- יש לדלל את הדגימות כדי להגיע לריכוז חלבון בטווח העקומה הסטנדרטית של 0.025-1.0 מ"ג/מ"ל. יש כמה (לפחות 3) דילולים מדגם בתוך הטווח.
הערה: ניתן לדלל דגימות במי מלח חוצצי פוספט (PBS) או בכל הרכב בינוני/חיץ אחר התואם למגיב ברדפורד. הריכוז הסופי של רכיבים בינוניים לא צריך להיות שונה בין תקן דגימות. - הוסף 10 μL מכל תמיסה סטנדרטית של חלבון לשלוש בארות (כלומר, במשולש) של מיקרו-צלחת בת 96 בארות. עבור נקודת החלבון 0 (אפס), הוסף 10 μL של החיץ/המדיום המשמש להכנת התמיסות הסטנדרטיות ודילול הדגימה.
- לקבוצה אחרת של בארות, הוסף 10 μL מכל דילול מדגם לשלוש בארות (כלומר, במשולש) של אותה מיקרו-צלחת בת 96 בארות. גישה אחת היא להוסיף נפחים שונים של דגימה ולהשלים עם תווך של עד 10 μL לכל באר (למשל, 0.5 μL, 1 μL, 2 μL, 4 μL, ו 8 μL של הדגימה בתוספת 9.5 μL, 9 μL, 8 μL, 6 μL, ו 2 μL של בינוני).
- הוסף 250 μL של מגיב בדיקת חלבון ברדפורד לכל הבארות. הגדרת מיקרו-צלחות טיפוסית מוצגת באיור 2. בדוגמה זו, קבוצה של בארות ריקות המכילות 260 μL (הנפח הסופי לכל באר) מים נכללה כדי לשמש ריק בקורא microplate (שלב 4.5). זה נדרש רק אם ייאספו גם נתוני קליטה.
הערה: הכן עקומה סטנדרטית באותה מיקרו-צלחת של דגימות בכל פעם שהבדיקה מבוצעת. במילים אחרות, אם מספר הדגימות דורש צלחת נוספת, הכינו עקומה סטנדרטית נוספת בלוח השני, וכן הלאה. - רשום תוצאות (סעיף 4) תוך 5-15 דקות.
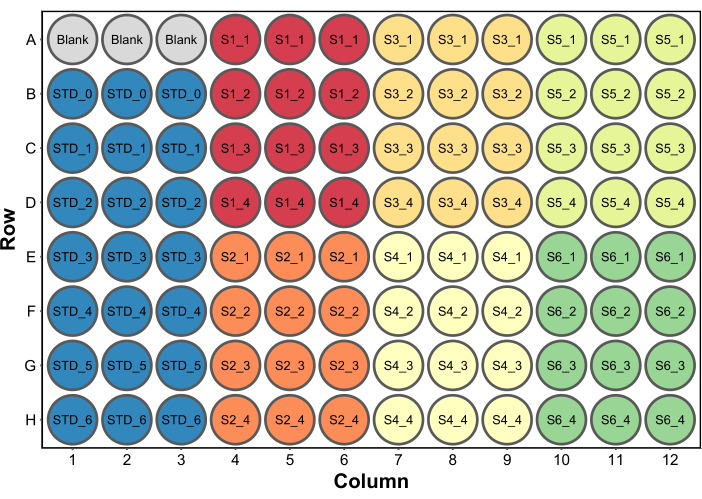
איור 2: פריסת לוחות טיפוסית עבור בדיקת חלבון ברדפורד. Blank מתייחס לשלוש בארות המכילות 260 μL של מים שישמשו ריקים בקורא microplate. STD מתייחס לתקני חלבון. S1-S6 הן שש דוגמאות שונות. SX_1-SX_4 הם ארבעה דילולי מדגם שונים עבור כל דגימה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
4. הקלטת תוצאות
- בחדר מואר היטב, החזיקו ביד אחת את צלחת המיקרו במקביל לספסל על רקע לבן אחיד (למשל, דף נייר). הבטיחו יישור מדויק על ידי הנחת מפלס הבועות על הצלחת.
- עם היד השנייה, החזיקו את הטלפון החכם במקביל (חלק מיישומי המצלמה מציינים באופן שימושי את נטיית המכשיר) לספסל ולצלחת המיקרו וצלמו תמונה אחת או יותר של כל המיקרו-צלחת (איור 3). במכשירי iOS, הפעילו את מחוון רמת המצלמה בהגדרות המצלמה על ידי הפעלת האפשרות רשת . במכשירי Android, הפעל את מחוון רמת המצלמה בהגדרות המצלמה על-ידי הפעלת רמזי מסגור.
- אין צורך במנגנון תאורה מיוחד, אך יש להקפיד להימנע מצללים והשתקפויות. לדוגמה, הימנעו מהצללת הצלחת או הרקע באמצעות הטלפון החכם והימנעו מהצללת הרקע באמצעות המיקרו-לוחית. השתקפויות קטנות בשולי אזור הבאר אינן בעיה; ניתן לחלץ נתוני צבע מאזור קטן מאוד במרכז כל באר.
- בדוק בקצרה את התמונה עבור אחידות רקע, צללים והשתקפויות. כמו כן, תסתכל על הזווית של הבארות; המרכז של כל באר צריך להיות גלוי ישירות (כלומר, לא מאחורי קירות באר).
- אם רוצים להשוות בין קריאות ספיגת מיקרו-לוחות לבין נתוני צבע תמונה, קרא את המיקרו-לוח ב-590 ננומטר וב-450 ננומטר בקורא מיקרו-לוחות21.

איור 3: לכידת התוצאות של בדיקת חלבון ברדפורד. בחדר מואר היטב, צלחת המיקרו ממוקמת במקביל לספסל על רקע אחיד ביד אחת. עם זאת, הטלפון החכם מוחזק במקביל לספסל ולצלחת המיקרו. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
5. חילוץ נתוני צבע באופן אוטומטי
- הורד את ImageJ ואת ReadPlate22, תוסף ImageJ זמין ב https://imagej.nih.gov/ij/plugins/readplate/index.html (זהו קובץ .txt).
- פתח את ImageJ, לחץ על תוספים > התקן ובחר את הקובץ שהורד בשלב 6.1.
- הגדר את פרמטרי המדידה על ידי לחיצה על נתח > קבע מדידות ובדוק את האפשרויות הבאות: שטח; סטיית תקן; ערך אפור מינימלי ומרבי; ערך אפור ממוצע; ערך אפור מודאלי. בתחתית החלון, הגדר ניתוב מחדש אל: ללא ומקומות עשרוניים (0-9): 3.
- עבור אל קובץ > פתח ובחר את התמונה של צלחת המיקרו שצולמה בשלב 4.
- עבור אל תוספים > ReadPlate. קרא את ההוראות ולאחר מכן לחץ על אישור.
- בחר את מספר הבארות: 96.
- באמצעות כלי הבחירה המלבני שנטען אוטומטית על ידי התוסף, ליצור מלבן המתחיל במרכז באר A1 ומסתיים במרכז באר H12. לאחר מכן, לחץ על אישור.
- בחר את הערוץ הכחול ולחץ על אישור.
- אשר את פרמטרי ברירת המחדל על ידי לחיצה על אישור.
- בדוק אם התוכנה תחמה אזור בתוך כל באר ואם האזורים שנבחרו אינם מכסים אזורים עם צללים חריגים או השתקפות. לחץ על אישור.
- שמור את התוצאות וחזור על שלבים 5.8-5.10 עבור הערוץ הירוק.
- חשב את היחס בין כחול לירוק באמצעות המצב עבור כל צבע.
הערה: ניתן לבצע את החישוב באופן ידני או באמצעות התוכנה של העדפות הקורא, כגון R, Microsoft Excel או GraphPad Prism.
6. חילוץ נתוני צבע באופן ידני
- הורד את Inkscape, עורך גרפי חינמי בקוד פתוח.
הערה: ניתן להשתמש בכל תוכנה עם כלי דוגם הצבע (המתואר בדרך כלל כטפטפת) כדי לזהות את הצבע ולחלץ נתוני RGB. - פתח את Inkscape, עבור אל File > Open. בחר את התמונה של לוח המיקרו שצולמה בשלב 4.
- בחרו בכלי בחירה ושינוי צורה של עצמים (S), המתואר כחץ בצד שמאל למעלה, ולחצו על התמונה. גבול קו מקווקו יציין את הבחירה.
- בחרו בכלי בחירת צבעים מתמונה (D), המתואר כטפטפת בצד שמאל.
- לחץ במרכז באר. הצבע בחלונית התחתונה ("מילוי:") ישתנה בהתאם. לחץ על הצבע וכרטיסיית מילוי וקו תופיע בצד ימין.
- שנה את התפריט הנפתח צבע שטוח ל - RGB. רשום את הערכים המוצגים עבור הערוצים הכחולים והירוקים עבור כל באר.
- חשב את היחס בין כחול לירוק באמצעות הערכים הרשומים.
הערה: ניתן לבצע את החישוב באופן ידני או באמצעות התוכנה של העדפות הקורא, כגון R, Microsoft Excel או GraphPad Prism.
7. בניית עקומות סטנדרטיות ואקסטרפולציה של נעלמים
- התווה את יחס העוצמה הממוצע בין ירוק לכחול כפונקציה של ריכוז תקן החלבון.
הערה: התווה את הנתונים באופן ידני או באמצעות התוכנה של העדפות הקורא, כגון R, Microsoft Excel או GraphPad Prism. - חשב את יחס העוצמה הממוצע בין ירוק לכחול עבור כל דגימה ודילול.
- בדוק אם האות המתקבל עבור הדגימה נמצא בטווח הליניארי של תקן החלבון.
- התעלם מערך כלשהו מתחת או מעל ערכי המינימום או המקסימום של העקומה הסטנדרטית.
- השתמש במשוואה הליניארית המתארת את העקומה הסטנדרטית כדי להסיק את כמות החלבון בדגימה. הכפל את הערכים המחושבים בגורם הדילול בהתאם.
תוצאות
איור 4 הוא תמונה של מיקרו-לוחית שממנה חולצו נתוני צבע, ותועדה ספיגה ב-450 ננומטר וב-590 ננומטר. נתוני הצבע RGB שדווחו כאן כמייצגים התקבלו באופן אוטומטי כמתואר בסעיף 5. דפוס אופייני של נתוני צבע הוא עלייה בערכים הכחולים וירידה בערכים האדומים והירוקים (איור 5). שימו ?...
Discussion
מאמר זה מתאר את RGBradford, שיטה המשתמשת במצלמת סמארטפון כדי להקליט נתונים מבדיקת חלבון ברדפורד, לחלץ נתוני צבע ולכמת במדויק את רמות החלבונים בדגימות ביולוגיות כפי שתואר במקורלאחרונה 20. הבדל אחד משיטת RGBradford המקורית הוא שכאן נעשה שימוש בהליך לקבלת נתוני צבע באופן אוטומטי עם תוסף Ima...
Disclosures
למחבר אין ניגודי עניינים להצהיר.
Acknowledgements
עבודה זו מומנה על ידי המועצה הלאומית לפיתוח מדעי וטכנולוגי (CNPq, ברזיל) [מספרי מענקים 428048/2018-8 ו-402556/2022-4] ואוניברסיטת ברזיליה (ברזיל). המחבר מודה לד"ר דוארטה נונו קרבליו וד"ר אוולין סנטוס (i3s, פורטו, פורטוגל) על מתן גישה לסמארטפונים שלהם המשמשים במחקר זה.
Materials
| Name | Company | Catalog Number | Comments |
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
References
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved