Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
RGBradford: Proteinquantifizierung mit einer Smartphone-Kamera
In diesem Artikel
Zusammenfassung
Dieser Artikel enthält ein Protokoll für die Proteinquantifizierung mit dem Bradford-Assay und einem Smartphone als Analysegerät. Der Proteingehalt in Proben kann anhand von Farbdaten quantifiziert werden, die aus einem mit einem Smartphone aufgenommenen Bild einer Mikrotiterplatte extrahiert werden.
Zusammenfassung
Die Proteinquantifizierung ist ein wesentliches Verfahren in der lebenswissenschaftlichen Forschung. Neben mehreren anderen Methoden ist der Bradford-Assay eine der am häufigsten verwendeten. Aufgrund seiner weiten Verbreitung wurden die Grenzen und Vorteile des Bradford-Assays ausführlich beschrieben, einschließlich mehrerer Modifikationen der ursprünglichen Methode zur Verbesserung seiner Leistung. Eine der Abwandlungen der ursprünglichen Methode ist die Verwendung einer Smartphone-Kamera als Analyseinstrument. Unter Ausnutzung der drei Formen des Coomassie Brilliant Blue-Farbstoffs, die unter den Bedingungen des Bradford-Assays vorliegen, wird in diesem Artikel beschrieben, wie Protein in Proben mithilfe von Farbdaten, die aus einem einzigen Bild einer Mikrotiterplatte extrahiert wurden, genau quantifiziert werden können. Nach der Durchführung des Assays in einer Mikroplatte wird ein Bild mit einer Smartphone-Kamera aufgenommen, und RGB-Farbdaten werden mit einer kostenlosen Open-Source-Bildanalysesoftware aus dem Bild extrahiert. Anschließend wird das Verhältnis von Blau- zu Grünintensität (in der RGB-Skala) von Proben mit unbekannten Proteinkonzentrationen verwendet, um den Proteingehalt auf der Grundlage einer Standardkurve zu berechnen. Es wird kein signifikanter Unterschied zwischen Werten beobachtet, die mit RGB-Farbdaten berechnet wurden, und solchen, die mit herkömmlichen Absorptionsdaten berechnet wurden.
Einleitung
Unabhängig von der nachgelagerten Anwendung (z. B. ELISA, Enzymkinetik, Western Blotting, Proteinreinigung und Massenspektrometrie) ist die Proteinquantifizierung entscheidend für eine genaue Analyse in Life-Sciences-Laboren. Zusätzlich zu ihrer Verwendung als sekundäre Messwerte (d. h. zur Berechnung der relativen Konzentrationen von Analyten pro Proteinmasse) können Proteingehalte in einer Probe auch selbst die gewünschte Ausgabe sein. Zum Beispiel kann man sich für den Proteingehalt in Nahrungsressourcen1 oder im Urin2 interessieren. Es stehen viele Methoden zur Verfügung, um die Proteinkonzentration in Proben3 zu messen, einschließlich direkter UV-Absorptionsmesswerte4, Protein-Kupfer-Chelatbildung 5,6, proteinbindende kolorimetrische Assays7 und proteinfarbstoffbindende Fluoreszenzassays8. Die Relevanz der Proteinquantifizierung wird durch das Vorhandensein von zwei Arbeiten, die Proteinmessmethoden beschreiben, 5,7 in den Top-3 der meistzitierten Literaturbelegt 9,10. Trotz der Tatsache, dass viele Autoren ihr tatsächliches Zitieren vernachlässigen, indem sie nicht-primäre Referenzen zitieren oder überhaupt nichts zitieren, belaufen sich die Originalarbeiten, die den Lowry-Protein-Assay und den Bradford-Protein-Assay beschreiben, auf jeweils >200.000 Zitate.
Die Popularität des Bradford-Assays beruht auf seiner Erschwinglichkeit, Einfachheit, Geschwindigkeit und Empfindlichkeit. Der Assay basiert auf der Wechselwirkung zwischen Proteinen und dem Farbstoff Coomassie Brilliant Blue G unter sauren Bedingungen. Unter den Bedingungen des Assays (d. h. niedriger pH-Wert) liegt der Farbstoff in drei Formen vor: einer roten kationischen Form mit λmax bei 470 nm; eine grüne neutrale Form mit λmax bei 650 nm; und eine blaue anionische Form mit λmax bei 590 nm 11,12 (Abbildung 1). Die kationische Form überwiegt in Abwesenheit von Proteinen. Wenn Proteine mit dem Farbstoff interagieren, stabilisieren sie die blaue anionische Form, was zu einer deutlichen Veränderung der Farbe der Lösung von bräunlich zu blau führt. In der Regel wird die Konzentrationsänderung der blauen Form des Farbstoffs spektrophotometrisch quantifiziert, dessen Absorption bei 590-595 nm proportional zur Proteinmenge im Assay ist.
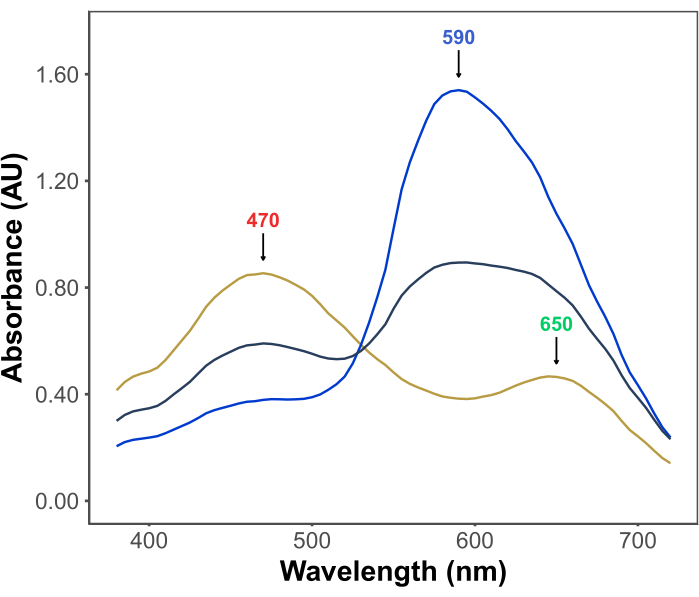
Abbildung 1: Coomassie-Absorptionsspektren für leuchtend blaues G unter den Bedingungen des Bradford-Assays. Die drei Hauptpeaks sind mit Pfeilen markiert, die das λmax der roten (470 nm), grünen (650 nm) und blauen (590 nm) Formen des Farbstoffs anzeigen. Die Spektren wurden in Abwesenheit von Protein (gelbe Linie) und in Gegenwart von 3 μg (graue Linie) und 10 μg (blaue Linie) Rinderserumalbumin aufgenommen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Die weit verbreitete Verwendung des Bradford-Assays hat zur Identifizierung mehrerer Einschränkungen geführt (z. B. unterschiedliche Reaktionen auf verschiedene Proteine11 und Interferenz durch Lipide13 und Detergenzien7) und zur Entwicklung von Modifikationen zur Verbesserung seiner Leistung (z. B. Zugabe von Detergenzien14,15, Alkalisierung14,16 und Verwendung des Verhältnisses von Absorptionen17). Neben Modifikationen des Assays selbst wurde auch die Verwendung alternativer Geräte wie Smartphones oder Kameras zur Erfassung analytischer Signale beschrieben 18,19,20. In der Tat ist die Entwicklung von Methoden, die Smartphones als tragbare chemische Analysatoren nutzen, ein aktives Forschungsgebiet. Die Motivation für die Verwendung von Smartphones ergibt sich aus der Erschwinglichkeit, Portabilität, Benutzerfreundlichkeit und weit verbreiteten Verfügbarkeit dieser Geräte.
Dieser Artikel stellt ein Protokoll für die Proteinquantifizierung mit dem RGBradford-Assay20 vor, der ein Smartphone als Analysegerät verwendet. Im Gegensatz zur ursprünglichen RGBradford-Veröffentlichung20 wurde hier ein Verfahren eingeführt, das den Farbextraktionsprozess rationalisiert. Es beinhaltet die Verwendung einer frei verfügbaren Softwareanwendung, um Farbinformationen aus jeder Vertiefung eines Mikroplattenbildes automatisch zu extrahieren, was viel Zeit und Mühe spart. Dies ist eine Alternative zu dem vorherigen Verfahren des manuellen Erfassens von Farbdaten aus jeder Vertiefung einzeln unter Verwendung einer Grafikeditor-Softwareanwendung20. Letztendlich kann der Proteingehalt in Proben anhand von Farbdaten quantifiziert werden, die aus einem mit einem Smartphone aufgenommenen Bild einer Mikroplatte extrahiert werden.
Protokoll
1. Herstellung des Bradford-Protein-Assay-Reagenzes
- 100 mg Coomassie Brilliant Blue G in 50 ml 95%igem (w/v) Ethanol auflösen. Mixen, bis sich Coomassie Brilliant Blue G vollständig aufgelöst hat.
VORSICHT: Ethanol ist brennbar und verursacht Augenreizungen. Vermeiden Sie Flammen und verwenden Sie eine Schutzbrille. - Zur vorherigen Lösung werden vorsichtig 100 ml 85%ige (w/v) Phosphorsäure zugegeben.
VORSICHT: Phosphorsäure ist korrosiv gegenüber Metallen und verursacht Hautkorrosion, schwere Augenschäden und akute orale Toxizität. Tragen Sie Handschuhe und Schutzbrille. - Die Lösung, die Coomassie Brilliant Blue G, Ethanol und Phosphorsäure enthält, wird langsam zu 600 ml deionisiertem Wasser gegeben.
- Verdünnen Sie die Lösung auf ein Endvolumen von 1.000 ml. Skalieren Sie je nach Anzahl der zu analysierenden Proben nach oben oder unten. Wie in der ursprünglichen Methode7 beschrieben, sollten die Endkonzentrationen im arbeitenden Bradford-Reagenz 0,01 % (w/v) Coomassie Brilliant Blue G, 4,7 % (w/v) Ethanol und 8,5 % (w/v) Phosphorsäure betragen.
- Entfernen Sie alle unlöslichen Materialien, die durch Filterpapier (Whatman #1-Papier oder gleichwertig) gefiltert werden.
- Das Reagenz ist mehrere Wochen stabil, wenn es bei Raumtemperatur (RT) gelagert und vor Licht geschützt wird. Filtern Sie nach Bedarf, da sich im Laufe der Zeit Niederschläge bilden können.
HINWEIS: Alternativ sind verdünnungs- und gebrauchsfertige Bradford-Reagenzien im Handel erhältlich. Befolgen Sie die Anweisungen des Herstellers zur Herstellung des Reagenzes und fahren Sie mit dem nächsten Schritt fort.
2. Herstellung von Protein-Standardlösungen
- Bereiten Sie eine Stammlösung (siehe Schritt 2.4) eines isolierten Proteins vor, das standardmäßig verwendet werden soll. Ein erschwingliches und häufig verwendetes Protein ist Rinderserumalbumin (BSA). Andere Optionen sind Ovalbumin und bovines Gammaglobulin.
- Wenn die molare Absorptionsfähigkeit des standardmäßig verwendeten Proteins bekannt ist, ist die Konzentration der Stammlösung in einem Spektralphotometer zu überprüfen.
- Für BSA ist eine häufig verwendete Formel BSA (mg/ml) = (A280/6,6) × 10, wobei A280 die Absorption bei 280 nm mit einer Schichtdicke von 1 cm ist, die gegen einen geeigneten Blindwert abgelesen wird (d. h. ε2801 % = 6,6)7. Zum Beispiel haben 0,8 mg/ml BSA eine Absorption von 0,528 bei 280 nm.
- Um die Standardkurve zu erstellen, bereiten Sie mehrere Verdünnungen von BSA innerhalb von 0,025 mg/ml und 1,0 mg/ml vor. Diese führen zu 0,25-10 μg BSA pro Well, nachdem ein Probenvolumen von 10 μl pro Well hinzugefügt wurde.
ANMERKUNG: Proteinstandardlösungen sollten in einem Medium mit der gleichen Zusammensetzung (Endkonzentration) wie das zur Probenvorbereitung verwendete Medium hergestellt werden.
3. Gehalt
- Verdünnen Sie die Proben, um eine Proteinkonzentration innerhalb des Standardkurvenbereichs von 0,025-1,0 mg/ml zu erreichen. Halten Sie mehrere (mindestens 3) Probenverdünnungen innerhalb des Bereichs.
HINWEIS: Die Proben können mit phosphatgepufferter Kochsalzlösung (PBS) oder einer anderen Medium/Pufferzusammensetzung verdünnt werden, die mit dem Bradford-Reagenz kompatibel ist. Die Endkonzentration der Medienkomponenten sollte sich zwischen Standard und Proben nicht unterscheiden. - Geben Sie 10 μl jeder Proteinstandardlösung in drei Vertiefungen (d. h. in dreifacher Ausfertigung) einer 96-Well-Mikroplatte. Für den Proteinpunkt 0 (Null) werden 10 μl des Puffers/Mediums zugegeben, das zur Herstellung der Standardlösungen und Probenverdünnungen verwendet wurde.
- Zu einem weiteren Satz von Wells geben Sie 10 μl jeder Probenverdünnung in drei Wells (d. h. in dreifacher Ausfertigung) derselben 96-Well-Mikroplatte. Ein Ansatz besteht darin, verschiedene Volumina einer Probe hinzuzufügen und mit einem Medium von bis zu 10 μl pro Vertiefung zu vervollständigen (z. B. 0,5 μl, 1 μl, 2 μl, 4 μl und 8 μl der Probe plus 9,5 μl, 9 μl, 8 μl, 6 μl und 2 μl Medium).
- Geben Sie 250 μl Bradford-Protein-Assay-Reagenz in alle Vertiefungen. Ein typischer Mikrotiterplattenaufbau ist in Abbildung 2 dargestellt. In diesem Beispiel wurde ein Satz von Blindvertiefungen, die 260 μl (das endgültige Volumen pro Bohrung) Wasser enthielten, als Leerlöcher in einem Mikroplatten-Reader verwendet (Schritt 4.5). Dies ist nur erforderlich, wenn auch Absorptionsdaten erhoben werden.
HINWEIS: Bereiten Sie bei jeder Durchführung des Assays eine Standardkurve in derselben Mikrotiterplatte der Proben vor. Mit anderen Worten, wenn die Anzahl der Proben eine weitere Platte erfordert, bereiten Sie eine weitere Standardkurve in der zweiten Platte vor und so weiter. - Erfassen Sie die Ergebnisse (Abschnitt 4) innerhalb von 5-15 Minuten.
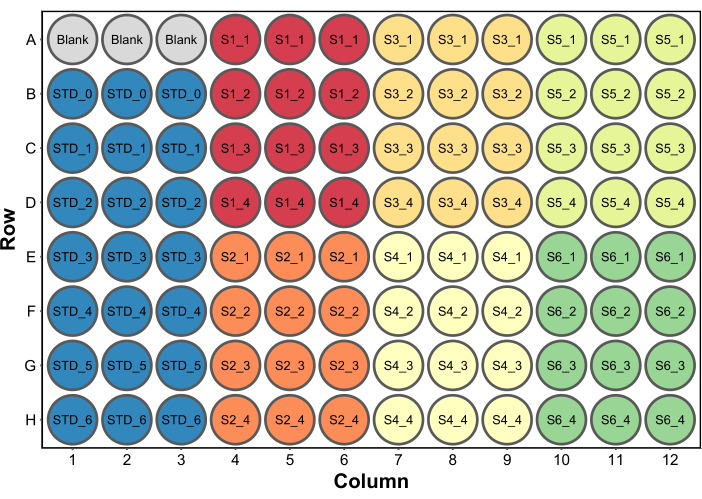
Abbildung 2: Ein typisches Plattenlayout für den Bradford-Protein-Assay. Blank bezieht sich auf drei Wells mit 260 μl Wasser, die als Blindwert in einem Mikroplatten-Reader verwendet werden sollen. STD bezieht sich auf Proteinstandards. S1-S6 sind sechs verschiedene Samples. SX_1-SX_4 gibt vier verschiedene Probenverdünnungen für jede Probe. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
4. Aufzeichnen der Ergebnisse
- Halten Sie die Mikrotiterplatte in einem gut beleuchteten Raum mit einer Hand parallel zur Bank vor einen einheitlichen weißen Hintergrund (z. B. ein Blatt Papier). Stellen Sie eine genaue Ausrichtung sicher, indem Sie eine Wasserwaage auf die Platte legen.
- Halten Sie das Smartphone mit der anderen Hand parallel (einige Kameraanwendungen zeigen die Neigung des Geräts sinnvollerweise an) an die Werkbank und die Mikroplatte und machen Sie ein oder mehrere Bilder der gesamten Mikroplatte (Abbildung 3). Aktivieren Sie auf iOS-Geräten die Anzeige für die Kameraebene in den Kameraeinstellungen, indem Sie die Option Raster aktivieren. Aktivieren Sie auf Android-Geräten die Anzeige für die Kameraebene in den Kameraeinstellungen, indem Sie die Framing-Hinweise aktivieren.
- Es ist keine spezielle Beleuchtungsvorrichtung erforderlich, aber achten Sie darauf, Schatten und Reflexionen zu vermeiden. Vermeiden Sie es beispielsweise, die Platte oder den Hintergrund mit dem Smartphone zu schattieren und vermeiden Sie es, den Hintergrund mit der Mikroplatte zu schattieren. Kleine Reflexionen an den Rändern des Brunnenbereichs sind kein Problem; Farbdaten können aus einem sehr kleinen Bereich in der Mitte jeder Vertiefung extrahiert werden.
- Überprüfen Sie das Bild kurz auf Gleichmäßigkeit des Hintergrunds, Schatten und Reflexionen. Werfen Sie auch einen Blick auf den Winkel der Brunnen; Die Mitte jedes Brunnens sollte direkt sichtbar sein (d. h. nicht hinter Brunnenwänden).
- Wenn der Vergleich zwischen Mikroplatten-Absorptionsmesswerten und Bildfarbdaten gewünscht wird, wird die Mikroplatte bei 590 nm und 450 nm in einem Mikroplatten-Reader21 abgelesen.

Abbildung 3: Erfassung der Ergebnisse des Bradford-Protein-Assays. In einem gut beleuchteten Raum wird die Mikroplatte mit einer Hand parallel zur Bank vor einem einheitlichen Hintergrund positioniert. Mit der anderen Hand wird das Smartphone parallel zur Werkbank und der Mikroplatte gehalten. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Automatisches Extrahieren von Farbdaten
- Laden Sie ImageJ und ReadPlate22 herunter, ein ImageJ-Plugin ist bei https://imagej.nih.gov/ij/plugins/readplate/index.html verfügbar (dies ist eine .txt Datei).
- Öffnen Sie ImageJ, klicken Sie auf Plugins > Installieren und wählen Sie die in Schritt 6.1 heruntergeladene Datei aus.
- Stellen Sie die Messparameter ein, indem Sie auf Analysieren > Messungen festlegen klicken und die folgenden Optionen aktivieren: Fläche; Standardabweichung; Minimaler und maximaler Grauwert; Mittlerer Grauwert; Modaler Grauwert. Legen Sie am unteren Rand des Fensters Umleitung auf Keine und Dezimalstellen (0-9): 3 fest.
- Gehen Sie zu Datei > Öffnen und wählen Sie das Bild der Mikrotiterplatte aus, das in Schritt 4 aufgenommen wurde.
- Gehen Sie zu Plugins > ReadPlate. Lesen Sie die Anweisungen, und klicken Sie dann auf OK.
- Wählen Sie die Anzahl der Wells: 96.
- Erstellen Sie mit dem rechteckigen Auswahlwerkzeug, das automatisch vom Plugin geladen wird, ein Rechteck, das in der Mitte des A1-Wells beginnt und in der Mitte des H12-Bereichs endet. Klicken Sie dann auf OK.
- Wählen Sie den blauen Kanal aus und klicken Sie auf OK.
- Bestätigen Sie die Standardparameter mit einem Klick auf OK.
- Überprüfen Sie, ob die Software einen Bereich in jedem Well abgegrenzt hat und ob die ausgewählten Bereiche keine Bereiche mit abnormalen Schatten oder Reflexionen abdecken. Klicken Sie auf OK.
- Speichern Sie die Ergebnisse und wiederholen Sie die Schritte 5.8-5.10 für den grünen Kanal.
- Berechnen Sie das Blau-Grün-Verhältnis mit dem Modus für jede Farbe.
HINWEIS: Die Berechnung kann manuell oder mit der Software der Leseeinstellungen wie R, Microsoft Excel oder GraphPad Prism durchgeführt werden.
6. Extrahieren von Farbdaten - Manuell
- Laden Sie Inkscape herunter, einen kostenlosen Open-Source-Grafikeditor.
HINWEIS: Jede Software mit dem Farbwähler-Werkzeug (normalerweise als Pipette dargestellt) kann verwendet werden, um die Farbe zu identifizieren und RGB-Daten zu extrahieren. - Öffnen Sie Inkscape, gehen Sie zu Datei > Öffnen. Wählen Sie das Bild der Mikroplatte aus, das in Schritt 4 aufgenommen wurde.
- Wählen Sie das Werkzeug Objekte auswählen und transformieren (S), das oben links als Pfeil dargestellt wird, und klicken Sie auf das Bild. Ein gestrichelter Linienrand zeigt die Auswahl an.
- Wählen Sie das Werkzeug Farben aus Bild auswählen (D) aus, das auf der linken Seite als Pipette dargestellt wird.
- Klicken Sie in die Mitte eines Brunnens. Die Farbe im unteren Bereich ("Füllen:") ändert sich entsprechend. Klicken Sie auf die Farbe und auf der rechten Seite wird eine Registerkarte Fläche und Kontur angezeigt.
- Ändern Sie das Dropdown-Menü "Flache Farbe" in RGB. Notieren Sie die Werte, die für den blauen und den grünen Kanal für jede Vertiefung angezeigt werden.
- Berechnen Sie das Blau-Grün-Verhältnis anhand der aufgezeichneten Werte.
HINWEIS: Die Berechnung kann manuell oder mit der Software der Leseeinstellungen wie R, Microsoft Excel oder GraphPad Prism durchgeführt werden.
7. Erstellen von Standardkurven und Extrapolieren von Unbekannten
- Zeichnen Sie das durchschnittliche Verhältnis von Grün zu Blau als Funktion der Proteinstandardkonzentration auf.
HINWEIS: Zeichnen Sie die Daten manuell oder mit der Software der Leseeinstellungen, z. B. R, Microsoft Excel oder GraphPad Prism. - Berechnen Sie das durchschnittliche Verhältnis von Grün zu Blau für jede Probe und Verdünnung.
- Prüfen Sie, ob das für die Probe erhaltene Signal innerhalb des linearen Bereichs des Proteinstandards liegt.
- Ignorieren Sie alle Werte, die unter oder über den Minimal- oder Maximalwerten der Standardkurve liegen.
- Verwenden Sie die lineare Gleichung, die die Standardkurve beschreibt, um die Proteinmenge in der Probe zu extrapolieren. Multiplizieren Sie die berechneten Werte entsprechend mit dem Verdünnungsfaktor.
Ergebnisse
Abbildung 4 zeigt ein Bild einer Mikroplatte, aus der Farbdaten extrahiert und die Absorption bei 450 nm und 590 nm aufgezeichnet wurde. Die RGB-Farbdaten, die hier als repräsentativ angegeben werden, wurden automatisch erhalten, wie in Abschnitt 5 beschrieben. Ein typisches Muster von Farbdaten ist eine Zunahme der Blauwerte und eine Abnahme der Rot- und Grünwerte (Abbildung 5). Beachten Sie, dass trotz der offensichtlichen Reflexion in allen Vertiefungen und...
Diskussion
In diesem Artikel wird RGBradford beschrieben, eine Methode, bei der eine Smartphone-Kamera verwendet wird, um Daten aus einem Bradford-Protein-Assay aufzuzeichnen, Farbdaten zu extrahieren und den Proteingehalt in biologischen Proben genau zu quantifizieren, wie ursprünglich kürzlich beschrieben20. Ein Unterschied zum ursprünglichen RGBradford-Verfahren besteht darin, dass hier ein Verfahren zum automatischen Abrufen von Farbdaten mit einem ImageJ-Plugin22 verwendet wur...
Offenlegungen
Der Autor hat keine Interessenkonflikte zu deklarieren.
Danksagungen
Diese Arbeit wurde vom Nationalen Rat für wissenschaftliche und technologische Entwicklung (CNPq, Brasilien) [Fördernummern 428048/2018-8 und 402556/2022-4] und der Universität Brasília (Brasilien) finanziert. Der Autor bedankt sich bei Dr. Duarte Nuno Carvalho und Dr. Evelyn Santos (i3s, Porto, Portugal) für den Zugang zu ihren Smartphones, die in dieser Studie verwendet wurden.
Materialien
| Name | Company | Catalog Number | Comments |
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
Referenzen
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten