JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
RGBradford: 스마트폰 카메라를 이용한 단백질 정량 분석
요약
이 논문은 Bradford 분석과 스마트폰을 분석 장치로 사용하여 단백질 정량화를 위한 프로토콜을 제공합니다. 샘플의 단백질 수준은 스마트폰으로 촬영한 마이크로플레이트 사진에서 추출한 색상 데이터를 사용하여 정량화할 수 있습니다.
초록
단백질 정량 분석은 생명 과학 연구에서 필수적인 절차입니다. 다른 여러 방법 중에서 Bradford 분석은 가장 많이 사용되는 방법 중 하나입니다. 널리 퍼져 있기 때문에 Bradford 분석의 한계와 장점은 성능을 개선하기 위해 원래 방법의 몇 가지 수정을 포함하여 철저하게 보고되었습니다. 원래 방법의 변경 사항 중 하나는 스마트폰 카메라를 분석 도구로 사용하는 것입니다. 이 논문은 Bradford 분석의 조건에 존재하는 세 가지 형태의 Coomassie Brilliant Blue 염료를 활용하여 마이크로플레이트의 단일 사진에서 추출한 색상 데이터를 사용하여 샘플의 단백질을 정확하게 정량화하는 방법을 설명합니다. 마이크로플레이트에서 분석을 수행한 후 스마트폰 카메라를 사용하여 사진을 촬영하고 무료 오픈 소스 이미지 분석 소프트웨어 응용 프로그램을 사용하여 사진에서 RGB 색상 데이터를 추출합니다. 그런 다음 단백질 농도를 알 수 없는 샘플의 청색 대 녹색 강도 비율(RGB 척도)을 사용하여 표준 곡선을 기반으로 단백질 함량을 계산합니다. RGB 색상 데이터를 사용하여 계산된 값과 기존 흡광도 데이터를 사용하여 계산된 값 사이에는 유의미한 차이가 관찰되지 않습니다.
서문
다운스트림 사용(예: ELISA, 효소 역학, 웨스턴 블로팅, 단백질 정제 및 질량 분석법)에 관계없이 단백질 정량화는 생명 과학 실험실의 정확한 분석에 매우 중요합니다. 2차 판독(즉, 단백질 질량당 분석물의 상대적 수준을 계산하기 위해)으로 사용하는 것 외에도 샘플의 단백질 수준은 원하는 출력 자체가 될 수도 있습니다. 예를 들어, 음식물 자원1 또는 소변2의 단백질 수치에 관심을 가질수 있다. 직접 UV 흡광도 판독 값4, 단백질-구리 킬레이트화 5,6, 단백질 염료 결합 비색 분석7 및 단백질 염료 결합 형광 분석8을 포함하여 샘플3에서 단백질 농도를 측정하는 데 사용할 수 있는 많은 방법이 있습니다. 단백질 정량의 관련성은 가장 많이 인용된 문헌 9,10의 상위 3개 문헌에 단백질 측정 방법 5,7을 설명하는 두 개의 논문이 있음으로써 입증됩니다. 많은 저자들이 1차 문헌이 아닌 문헌을 인용하거나 전혀 인용하지 않음으로써 실제 인용을 소홀히 하고 있음에도 불구하고, Lowry 단백질 분석과 Bradford 단백질 분석을 설명하는 원본 논문은 각각10>200,000회 인용)에 달합니다.
Bradford 분석의 인기는 경제성, 단순성, 속도 및 감도에서 비롯됩니다. 이 분석은 산성 조건에서 단백질과 염료 Coomassie Brilliant Blue G 간의 상호 작용을 기반으로 합니다. 분석 조건(즉, 낮은 pH)에서 염료는 세 가지 형태로 존재합니다: 470nm에서 λmax를 갖는 적색 양이온 형태; 650nm에서 λmax를 갖는 녹색 중성 형태; 및 590nm에서 λmax를 갖는 청색 음이온 형태11,12 (그림 1). 양이온 형태는 단백질이 없을 때 우세합니다. 단백질이 염료와 상호 작용할 때 청색 음이온 형태를 안정화시켜 용액의 색상이 갈색에서 파란색으로 눈에 띄게 변합니다. 일반적으로 염료의 청색 형태 농도 변화는 분광 광도계로 정량화되며, 590-595nm에서의 흡광도는 분석의 단백질 양에 비례합니다.
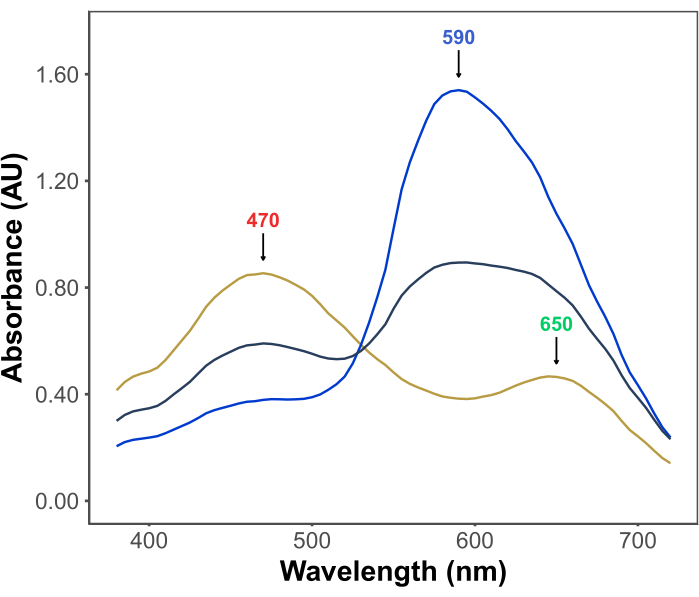
그림 1: Bradford 분석 조건에서 Coomassie 브릴리언트 블루 G 흡수 스펙트럼. 3개의 주요 피크는 염료의 빨간색(470nm), 녹색(650nm) 및 파란색(590nm) 형태의 λmax를 나타내는 화살표로 표시됩니다. 스펙트럼은 단백질(황색 선)이 없는 경우 및 3μg(회색 선) 및 10μg(청색 선)의 소 혈청 알부민이 있는 경우 기록되었습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
Bradford 분석법의 광범위한 사용은 몇 가지 한계(예: 상이한 단백질11에 대한 다양한 반응, 지질13 및 세제7에 의한 간섭)의 확인과 그 성능을 향상시키기 위한 변형의 개발(예: 세제(14,15)의 첨가, 알칼리화(14,16) 및 흡광도(17)의 비율의 사용)을 개발하게 하였다 ). 분석 자체의 수정 외에도 분석 신호를 캡처하기 위해 스마트폰 또는 카메라와 같은 대체 장치를 사용하는 것도 설명되었습니다 18,19,20. 실제로 스마트폰을 휴대용 화학 분석기로 사용하는 방법의 개발은 활발한 연구 분야였습니다. 스마트폰 사용의 동기는 이러한 장치의 경제성, 휴대성, 사용 편의성 및 광범위한 가용성에서 비롯됩니다.
이 논문은 스마트폰을 분석 장치로 사용하는 RGBradford assay20을 사용하여 단백질 정량을 위한 프로토콜을 제공합니다. 원래 RGBradford 간행물20과 달리 여기서는 색상 추출 프로세스를 간소화하는 절차가 도입되었습니다. 여기에는 무료로 사용할 수 있는 소프트웨어 응용 프로그램을 활용하여 마이크로플레이트 그림의 각 웰에서 색상 정보를 자동으로 추출하여 상당한 시간과 노력을 절약하는 것이 포함됩니다. 이것은 그래픽 편집기 소프트웨어 애플리케이션(20)을 사용하여 각 웰로부터 하나씩 수동으로 색상 데이터를 획득하는 이전 방법의 대안이다. 궁극적으로 샘플의 단백질 수준은 스마트폰으로 촬영한 마이크로플레이트 사진에서 추출한 색상 데이터를 사용하여 정량화할 수 있습니다.
프로토콜
1. Bradford 단백질 분석 시약의 준비
- Coomassie Brilliant Blue G 100mg을 50mL 95%(w/v) 에탄올에 녹입니다. 쿠마시 브릴리언트 블루 G가 완전히 녹을 때까지 섞는다.
주의 : 에탄올은 가연성이며 눈에 자극을 일으킵니다. 화염을 피하고 고글을 사용하십시오. - 이전 용액에 100mL의 85%(w/v) 인산을 조심스럽게 첨가합니다.
주의 : 인산은 금속을 부식시키며 피부 부식, 심각한 눈 손상 및 급성 경구 독성을 유발합니다. 장갑과 고글을 착용하십시오. - Coomassie Brilliant Blue G, 에탄올 및 인산이 포함된 용액을 탈이온수 600mL에 천천히 추가합니다.
- 용액을 최종 부피 1,000mL로 희석합니다. 분석할 샘플 수에 따라 확장 또는 축소합니다. 본래의 방법7에 기술된 바와 같이, 작용 브래드포드 시약의 최종 농도는 0.01%(w/v)의 쿠마시 브릴리언트 블루 G, 4.7%(w/v)의 에탄올, 및 8.5%(w/v)의 인산이어야 한다.
- 여과지(Whatman #1 용지 또는 이와 동등한 용지)를 통해 여과되는 불용성 물질을 제거합니다.
- 시약은 실온(RT)에서 보관하고 빛으로부터 보호할 때 몇 주 동안 안정적입니다. 시간이 지남에 따라 침전물이 형성될 수 있으므로 필요에 따라 여과하십시오.
참고: 또는 희석하여 사용할 수 있는 Bradford 시약을 상업적으로 이용할 수 있습니다. 시약 준비에 대한 제조업체의 지침을 따르고 다음 단계로 진행하십시오.
2. 단백질 표준물질 용액의 제조
- 표준으로 사용할 분리된 단백질의 원액(2.4단계 참조)을 준비합니다. 저렴하고 일반적으로 사용되는 단백질은 소 혈청 알부민(BSA)입니다. 다른 옵션으로는 오브알부민(ovalbumin)과 소 감마 글로불린(bovine gamma globulin)이 있습니다.
- 표준으로 사용되는 단백질의 몰 흡수율을 알고 있는 경우 분광 광도계에서 원액의 농도를 확인하십시오.
- BSA의 경우 일반적으로 사용되는 공식은 BSA(mg/mL) = (A280/6.6) × 10이며, 여기서 A280은 280nm에서 흡광도이며 경로 길이는 1cm이고 적절한 블랭크에 대해 판독됩니다(즉,ε 2801% = 6.6)7. 예를 들어, 0.8mg/mL BSA는 280nm에서 0.528의 흡광도를 갖습니다.
- 표준 곡선을 생성하려면 0.025mg/mL 및 1.0mg/mL 내에서 BSA를 여러 번 희석하여 준비합니다. 이는 웰당 10μL의 샘플 부피를 추가한 후 웰당 0.25-10μg의 BSA를 생성합니다.
참고: 단백질 표준 용액은 샘플 준비에 사용된 배지와 동일한 조성(최종 농도)을 가진 배지에서 준비해야 합니다.
3. 분석
- 샘플을 희석하여 0.025-1.0mg/mL의 표준 곡선 범위 내에서 단백질 농도를 얻습니다. 범위 내에 여러 개(최소 3개)의 시료 희석액이 있어야 합니다.
참고: 샘플은 인산염 완충 식염수(PBS) 또는 Bradford 시약과 호환되는 기타 배지/완충액 조성물로 희석할 수 있습니다. 중간 성분의 최종 농도는 표준물질과 샘플 간에 차이가 없어야 합니다. - 각 단백질 표준 용액 10μL를 96웰 마이크로플레이트의 3웰(즉, 3웰)에 추가합니다. 0(영) 단백질 포인트의 경우 표준 용액 및 샘플 희석액을 준비하는 데 사용되는 완충액/배지 10μL를 추가합니다.
- 다른 웰 세트에 각 샘플 희석액 10μL를 동일한 96웰 마이크로플레이트의 웰 3개(즉, 삼중)에 추가합니다. 한 가지 방법은 다양한 부피의 시료를 추가하고 웰당 최대 10μL의 배지(예: 0.5μL, 1μL, 2μL, 4μL 및 8μL의 시료와 9.5μL, 9μL, 8μL, 6μL 및 2μL의 배지)로 완성하는 것입니다.
- 모든 웰에 250μL의 Bradford 단백질 분석 시약을 추가합니다. 일반적인 마이크로플레이트 설정은 그림 2에 나와 있습니다. 이 예에서, 260 μL(웰당 최종 부피)의 물을 포함하는 블랭크 웰 세트를 마이크로플레이트 리더에서 블랭크로 사용하기 위해 포함시켰다(단계 4.5). 이는 흡광도 데이터도 수집되는 경우에만 필요합니다.
참고: 분석이 수행될 때마다 샘플의 동일한 마이크로플레이트에서 표준 곡선을 준비합니다. 즉, 샘플 수에 다른 플레이트가 필요한 경우 두 번째 플레이트에서 다른 표준 곡선을 준비하는 식입니다. - 4-5분 이내에 결과(섹션 15)를 기록합니다.
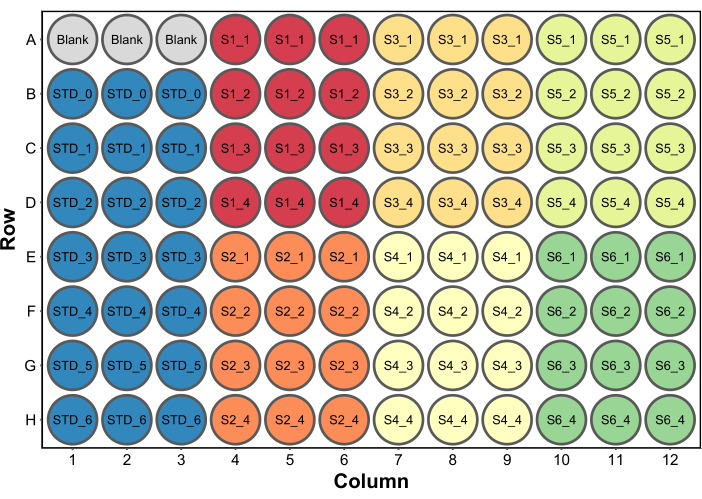
그림 2: Bradford 단백질 분석을 위한 일반적인 플레이트 레이아웃. 블랭크는 마이크로플레이트 리더에서 블랭크로 사용할 260μL의 물이 들어 있는 3개의 웰을 말합니다. 성병은 단백질 표준을 말합니다. S1-S6은 6개의 서로 다른 샘플입니다. SX_1-SX_4는 각 샘플에 대해 4가지 다른 샘플 희석액입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
4. 결과 기록
- 조명이 밝은 방에서 한 손으로 균일한 흰색 배경(예: 종이 시트)에 대해 벤치와 평행한 마이크로플레이트를 잡습니다. 플레이트에 기포 수평을 배치하여 정확한 정렬을 보장합니다.
- 다른 손으로 스마트폰을 벤치와 마이크로플레이트에 평행하게 잡고(일부 카메라 애플리케이션은 장치의 기울기를 유용하게 나타냄) 전체 마이크로플레이트의 사진을 하나 또는 여러 장 찍습니다(그림 3). iOS 장치에서는 그리드 옵션을 활성화하여 카메라 설정에서 카메라 레벨 표시기를 켭니다. Android 장치의 경우 프레임 힌트를 활성화하여 카메라 설정에서 카메라 레벨 표시기를 켭니다.
- 특별한 조명 장치가 필요하지 않지만 그림자와 반사를 피하도록 주의하십시오. 예를 들어, 스마트폰으로 판이나 배경을 음영 처리하지 말고 마이크로플레이트로 배경을 음영 처리하지 마십시오. 우물 영역의 가장자리에 있는 작은 반사는 문제가 되지 않습니다. 색상 데이터는 각 웰 중앙의 매우 작은 영역에서 추출할 수 있습니다.
- 사진에서 배경 균일성, 그림자 및 반사를 간략하게 확인합니다. 또한 우물의 각도를 살펴보십시오. 각 우물의 중심은 직접 볼 수 있어야 합니다(즉, 우물 벽 뒤가 아님).
- 마이크로플레이트 흡광도 판독값과 그림 컬러 데이터 사이의 비교가 필요한 경우, 마이크로플레이트 리더(21)에서 590nm 및 450nm에서 마이크로플레이트를 판독한다.

그림 3: Bradford 단백질 분석 결과 캡처. 조명이 밝은 방에서 마이크로플레이트는 한 손으로 균일한 배경에 대해 벤치와 평행하게 배치됩니다. 다른 손으로는 스마트폰을 벤치와 마이크로플레이트에 평행하게 고정합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
5. 색상 데이터 자동 추출
- ImageJ 및 ReadPlate22를 다운로드하면 ImageJ 플러그인은 https://imagej.nih.gov/ij/plugins/readplate/index.html 에서 사용할 수 있습니다(.txt 파일).
- ImageJ를 열고 플러그인 > 설치를 클릭한 다음 6.1단계에서 다운로드한 파일을 선택합니다.
- Analyze(분석) > Set Measurements(측정 설정)를 클릭하여 측정 매개변수를 설정하고 다음 옵션을 확인합니다. 표준 편차; Min & max 회색 값; 평균 회색 값; 모달 회색 값입니다. 창 아래쪽에서 리디렉션을 다음으로 설정합니다. 없음 및 소수점 이하 자릿수(0-9): 3.
- 파일 > 열기로 이동하여 4단계에서 촬영한 마이크로플레이트 사진을 선택합니다.
- 플러그인 > ReadPlate로 이동합니다. 지침을 읽은 후 확인을 클릭합니다.
- 웰 수를 선택: 96.
- 플러그인에 의해 자동으로 로드된 직사각형 선택 도구를 사용하여 A1 웰의 중앙에서 시작하여 H12 웰의 중앙에서 끝나는 직사각형을 만듭니다. 그런 다음 확인을 클릭합니다.
- 파란색 채널을 선택하고 [확인]을 클릭합니다.
- OK를 클릭하여 기본 매개변수를 확인합니다.
- 소프트웨어가 각 웰 내부의 영역을 묘사했는지, 선택한 영역이 비정상적인 그림자 또는 반사가 있는 영역을 덮고 있지 않은지 확인합니다. 확인을 클릭합니다.
- 결과를 저장하고 녹색 채널에 대해 5.8-5.10단계를 반복합니다.
- 각 색상에 대한 모드를 사용하여 파란색과 녹색의 비율을 계산합니다.
참고: 계산은 수동으로 수행하거나 R, Microsoft Excel 또는 GraphPad Prism과 같은 리더 기본 설정의 소프트웨어를 사용하여 수행할 수 있습니다.
6. 수동으로 색상 데이터 추출
- 무료 오픈 소스 그래픽 편집기인 Inkscape를 다운로드하십시오.
알림: 색상 선택 도구(일반적으로 스포이드로 표시됨)가 있는 모든 소프트웨어를 사용하여 색상을 식별하고 RGB 데이터를 추출할 수 있습니다. - Inkscape를 열고 파일 > 열기로 이동합니다. 4단계에서 촬영한 마이크로플레이트의 사진을 선택합니다.
- 왼쪽 상단에 화살표로 표시된 Select and Transform Objects (S) 도구를 선택하고 그림을 클릭합니다. 점선 테두리는 선택을 나타냅니다.
- 왼쪽에 스포이드로 표시된 이미지에서 색상 선택(D) 도구를 선택합니다.
- 우물 가운데를 클릭합니다. 아래쪽 패널의 색상("채우기:")이 그에 따라 변경됩니다. 색상을 클릭하면 오른쪽에 채우기 및 획 탭이 나타납니다.
- 플랫 컬러(Flat Color) 드롭다운 메뉴를 RGB로 변경합니다. 각 웰의 파란색 및 녹색 채널에 표시된 값을 기록합니다.
- 기록된 값을 사용하여 파란색과 녹색 비율을 계산합니다.
참고: 계산은 수동으로 수행하거나 R, Microsoft Excel 또는 GraphPad Prism과 같은 리더 기본 설정의 소프트웨어를 사용하여 수행할 수 있습니다.
7. 표준 곡선 구축 및 미지수를 외삽합니다
- 평균 녹색-파란색 강도 비율을 단백질 표준 농도의 함수로 플로팅합니다.
참고: 수동으로 또는 R, Microsoft Excel 또는 GraphPad Prism과 같은 리더 기본 설정 소프트웨어를 사용하여 데이터를 플롯합니다. - 각 샘플에 대한 평균 녹색-파란색 강도 비율을 계산하고 희석합니다.
- 샘플에 대해 얻은 신호가 단백질 표준물질의 선형 범위 내에 있는지 확인합니다.
- 표준 곡선의 최소값 또는 최대값보다 작거나 큰 값은 무시합니다.
- 표준 곡선을 설명하는 선형 방정식을 사용하여 샘플의 단백질 양을 추정합니다. 그에 따라 계산된 값에 희석 계수를 곱합니다.
결과
도 4는 컬러 데이터를 추출하고 450nm 및 590nm에서 흡광도를 기록한 마이크로플레이트의 그림입니다. 여기에 대표로 보고된 RGB 색상 데이터는 섹션 5에 설명된 대로 자동으로 얻은 것입니다. 색 데이터의 일반적인 패턴은 파란색 값이 증가하고 빨간색과 녹색 값이 감소하는 것입니다(그림 5). 모든 웰에서 반사가 뚜렷하고 마이크로플레이트가 완벽하게 ?...
토론
이 논문은 스마트폰 카메라를 사용하여 Bradford 단백질 분석의 데이터를 기록하고, 색상 데이터를 추출하고, 최근20년에 설명된 대로 생물학적 샘플의 단백질 수준을 정확하게 정량화하는 방법인 RGBradford에 대해 설명합니다. 본래의 RGBradford 방법과의 한 가지 차이점은 여기서는 ImageJ 플러그인(22 )을 사용하여 색상 데이터를 자동으로 획득하는 절차가 사용되었다...
공개
저자는 선언할 이해 상충이 없습니다.
감사의 말
이 연구는 브라질 국립과학기술개발위원회(CNPq)[보조금 번호 428048/2018-8 및 402556/2022-4]와 브라질리아 대학교(브라질)의 자금 지원을 받았습니다. 저자는 이 연구에 사용된 스마트폰에 대한 액세스를 제공한 Duarte Nuno Carvalho 박사와 Evelyn Santos 박사(i3s, Porto, Portugal)에게 감사를 표합니다.
자료
| Name | Company | Catalog Number | Comments |
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
참고문헌
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유