Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
RGBradford : Quantification des protéines avec l’appareil photo d’un smartphone
Dans cet article
Résumé
Cet article fournit un protocole pour la quantification des protéines à l’aide du test de Bradford et d’un smartphone comme dispositif d’analyse. Les niveaux de protéines dans les échantillons peuvent être quantifiés à l’aide de données de couleur extraites d’une photo d’une microplaque prise avec un smartphone.
Résumé
La quantification des protéines est une procédure essentielle dans la recherche en sciences de la vie. Parmi plusieurs autres méthodes, le test Bradford est l’une des plus utilisées. En raison de sa grande popularité, les limites et les avantages du test Bradford ont été rapportés de manière exhaustive, y compris plusieurs modifications de la méthode originale pour améliorer ses performances. L’une des modifications de la méthode originale est l’utilisation de l’appareil photo d’un smartphone comme instrument d’analyse. En tirant parti des trois formes du colorant bleu brillant de Coomassie qui existent dans les conditions du test de Bradford, cet article décrit comment quantifier avec précision les protéines dans les échantillons à l’aide de données de couleur extraites d’une seule image d’une microplaque. Après avoir effectué le test dans une microplaque, une image est prise à l’aide de l’appareil photo d’un smartphone et les données de couleur RVB sont extraites de l’image à l’aide d’une application logicielle d’analyse d’image gratuite et open-source. Ensuite, le rapport entre l’intensité bleue et l’intensité verte (dans l’échelle RVB) d’échantillons avec des concentrations inconnues de protéines est utilisé pour calculer la teneur en protéines sur la base d’une courbe standard. Aucune différence significative n’est observée entre les valeurs calculées à l’aide des données de couleur RVB et celles calculées à l’aide des données d’absorbance conventionnelles.
Introduction
Quelle que soit l’utilisation en aval (par exemple, ELISA, cinétique enzymatique, transfert Western, purification des protéines et spectrométrie de masse), la quantification des protéines est cruciale pour une analyse précise dans les laboratoires des sciences de la vie. En plus de leur utilisation comme lectures secondaires (c’est-à-dire pour calculer les niveaux relatifs d’analytes par masse de protéines), les niveaux de protéines dans un échantillon peuvent également être le résultat souhaité lui-même. Par exemple, on peut s’intéresser aux teneurs en protéines des ressources alimentaires1 ou dans l’urine2. Il existe de nombreuses méthodes disponibles pour mesurer la concentration de protéines dans les échantillons3, y compris les lectures directes d’absorbance UV4, la chélation protéine-cuivre 5,6, les tests colorimétriques de liaison protéine-colorant7 et les tests fluorescents de liaison protéine-colorant8. La pertinence de la quantification des protéines est mise en évidence par la présence de deux articles décrivant les méthodes de mesure des protéines 5,7 dans le top-3 de la littérature la plus citée 9,10. Malgré le fait que de nombreux auteurs négligent leur citation réelle en citant des références non primaires ou en ne citant rien du tout, les articles originaux décrivant le test protéique de Lowry et le test protéique de Bradford s’élèvent > 200 000 citations chacundes 10.
La popularité du test Bradford découle de son prix abordable, de sa simplicité, de sa rapidité et de sa sensibilité. Le test est basé sur l’interaction entre les protéines et le colorant Coomassie Brilliant Blue G dans des conditions acides. Dans les conditions de l’essai (c’est-à-dire un pH bas), le colorant existe sous trois formes : une forme cationique rouge avec λmax à 470 nm ; une forme neutre verte avec λmax à 650 nm ; et une forme anionique bleue avec λmax à 590 nm11,12 (Figure 1). La forme cationique prédomine en l’absence de protéines. Au fur et à mesure que les protéines interagissent avec le colorant, elles stabilisent la forme anionique bleue, provoquant un changement notable de la couleur de la solution, de brunâtre à bleu. Habituellement, le changement de concentration de la forme bleue du colorant est quantifié par spectrophotométrie, dont l’absorbance à 590-595 nm est proportionnelle à la quantité de protéines dans le test.
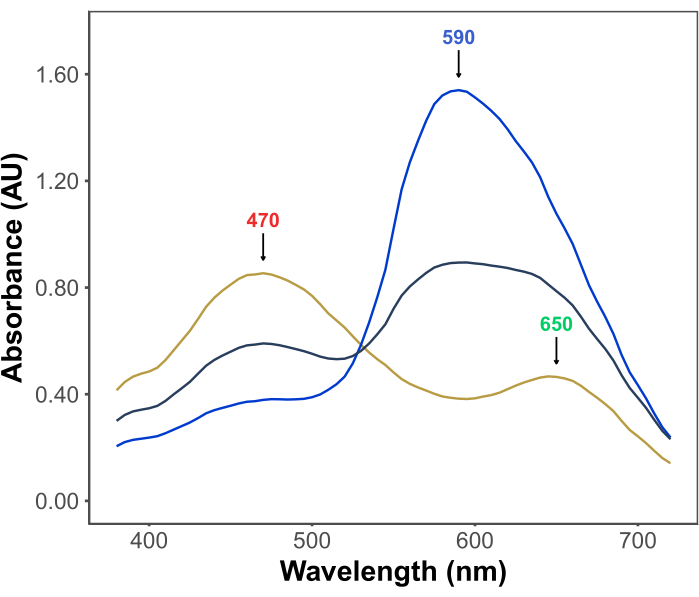
Figure 1 : Spectres d’absorption G bleu brillant de Coomassie dans les conditions de l’essai de Bradford. Les trois pics principaux sont marqués par des flèches indiquant le λmax des formes rouge (470 nm), verte (650 nm) et bleue (590 nm) du colorant. Les spectres ont été enregistrés en l’absence de protéines (raie jaune) et en présence de 3 μg (raie grise) et 10 μg (raie bleue) d’albumine sérique bovine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’utilisation généralisée du test de Bradford a conduit à l’identification de plusieurs limites (par exemple, des réponses variables à différentes protéines11 et à l’interférence des lipides13 et des détergents7) et à la mise au point de modifications pour améliorer ses performances (par exemple, l’ajout de détergents14,15, l’alcalinisation14,16 et l’utilisation du rapport d’absorbances17). En plus des modifications apportées au test lui-même, l’utilisation d’appareils alternatifs, tels que des smartphones ou des appareils photo, pour capturer des signaux analytiques a également été décrite 18,19,20. En effet, le développement de méthodes qui utilisent les smartphones comme analyseurs chimiques portables a été un domaine de recherche actif. La motivation pour l’utilisation des smartphones découle de l’abordabilité, de la portabilité, de la facilité d’utilisation et de la disponibilité généralisée de ces appareils.
Cet article fournit un protocole pour la quantification des protéines à l’aide du test RGBradford20, qui utilise un smartphone comme dispositif d’analyse. Contrairement à la publication originale RGBradford20, ici, une procédure qui rationalise le processus d’extraction des couleurs a été introduite. Il s’agit de l’utilisation d’une application logicielle disponible gratuitement pour extraire automatiquement les informations de couleur de chaque puits d’une image de microplaque, ce qui permet d’économiser beaucoup de temps et d’efforts. Il s’agit d’une alternative à la méthode précédente consistant à acquérir manuellement les données de couleur de chaque puits un par un à l’aide d’un logiciel d’édition graphique20. En fin de compte, les niveaux de protéines dans les échantillons peuvent être quantifiés à l’aide de données de couleur extraites d’une photo d’une microplaque prise avec un smartphone.
Protocole
1. Préparation du réactif de dosage des protéines de Bradford
- Dissoudre 100 mg de Coomassie Brilliant Blue G dans 50 ml d’éthanol à 95 % (p/v). Mélanger jusqu’à ce que Coomassie Brilliant Blue G soit complètement dissous.
ATTENTION : L’éthanol est inflammable et provoque une irritation des yeux. Évitez les flammes et utilisez des lunettes de protection. - Ajouter 100 mL d’acide phosphorique à 85 % (p/v) à la solution précédente.
ATTENTION : L’acide phosphorique est corrosif pour les métaux et provoque une corrosion de la peau, de graves lésions oculaires et une toxicité orale aiguë. Portez des gants et des lunettes de protection. - Ajouter lentement la solution contenant Coomassie Brilliant Blue G, l’éthanol et l’acide phosphorique à 600 mL d’eau déminéralisée.
- Diluer la solution jusqu’à obtenir un volume final de 1 000 ml. Augmentez ou diminuez la taille en fonction du nombre d’échantillons à analyser. Comme décrit dans la méthode7 originale, les concentrations finales dans le réactif Bradford de travail doivent être de 0,01 % (p/v) de bleu brillant de Coomassie G, de 4,7 % (p/v) d’éthanol et de 8,5 % (p/v) d’acide phosphorique.
- Éliminez tout matériau insoluble filtrant à travers du papier filtre (papier Whatman #1 ou équivalent).
- Le réactif est stable pendant plusieurs semaines lorsqu’il est conservé à température ambiante (RT) et à l’abri de la lumière. Filtrez au besoin, car des précipités peuvent se former avec le temps.
REMARQUE : Alternativement, des réactifs Bradford prêts à diluer et à utiliser sont disponibles dans le commerce. Suivez les instructions du fabricant pour préparer le réactif et passez à l’étape suivante.
2. Préparation de solutions étalons protéiques
- Préparer une solution mère (voir l’étape 2.4) d’une protéine isolée à utiliser en standard. L’albumine sérique bovine (BSA) est une protéine abordable et couramment utilisée. D’autres options sont l’ovalbumine et la gammaglobuline bovine.
- Si l’absorptivité molaire de la protéine utilisée comme étalon est connue, vérifier la concentration de la solution mère dans un spectrophotomètre.
- Pour la BSA, une formule couramment utilisée est BSA (mg/mL) = (A280/6,6) × 10, où A280 est l’absorbance à 280 nm avec une longueur de trajet de 1 cm lue par rapport à un blanc approprié (c.-à-d. ε2801 % = 6,6)7. Par exemple, 0,8 mg/mL de BSA a une absorbance de 0,528 à 280 nm.
- Pour générer la courbe standard, préparez plusieurs dilutions de BSA à 0,025 mg/mL et 1,0 mg/mL. Ceux-ci se traduiront par 0,25 à 10 μg de BSA par puits après l’ajout d’un volume d’échantillon de 10 μL par puits.
REMARQUE : Les solutions étalons de protéines doivent être préparées dans un milieu ayant la même composition (concentration finale) que le milieu utilisé pour préparer les échantillons.
3. Dosage
- Diluer les échantillons pour obtenir une concentration en protéines dans la plage de courbe standard de 0,025 à 1,0 mg/mL. Avoir plusieurs (au moins 3) dilutions d’échantillons dans la plage.
REMARQUE : Les échantillons peuvent être dilués avec une solution saline tamponnée au phosphate (PBS) ou toute autre composition de milieu/tampon compatible avec le réactif Bradford. La concentration finale des composants du milieu ne doit pas différer entre l’échantillon standard et l’échantillon. - Ajouter 10 μL de chaque solution étalon protéique dans trois puits (c.-à-d. en trois exemplaires) d’une microplaque à 96 puits. Pour le point protéique 0 (zéro), ajouter 10 μL du tampon/milieu utilisé pour préparer les solutions étalons et les dilutions de l’échantillon.
- À un autre ensemble de puits, ajouter 10 μL de chaque dilution d’échantillon à trois puits (c.-à-d. en trois exemplaires) de la même microplaque à 96 puits. Une approche consiste à ajouter différents volumes d’un échantillon et à compléter avec un milieu allant jusqu’à 10 μL par puits (p. ex., 0,5 μL, 1 μL, 2 μL, 4 μL et 8 μL de l’échantillon plus 9,5 μL, 9 μL, 8 μL, 6 μL et 2 μL de milieu).
- Ajouter 250 μL de réactif d’analyse des protéines de Bradford dans tous les puits. Une configuration typique de microplaque est illustrée à la figure 2. Dans cet exemple, un ensemble de puits vierges contenant 260 μL (le volume final par puits) d’eau a été inclus pour servir de blanc dans un lecteur de microplaques (étape 4.5). Cela n’est nécessaire que si des données d’absorbance doivent également être collectées.
REMARQUE : Préparez une courbe standard dans la même microplaque d’échantillons chaque fois que l’essai est effectué. En d’autres termes, si le nombre d’échantillons nécessite une autre plaque, préparez une autre courbe standard dans la deuxième plaque, et ainsi de suite. - Enregistrez les résultats (section 4) dans un délai de 5 à 15 minutes.
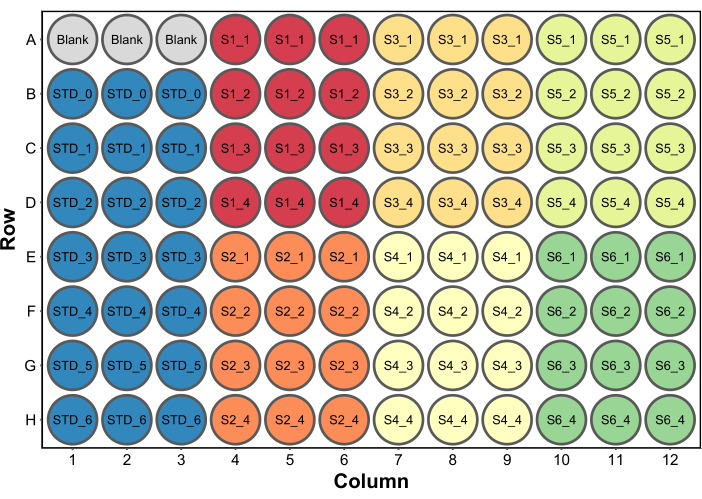
Figure 2 : Disposition typique de la plaque pour le test de la protéine de Bradford. Le terme « blanc » désigne trois puits contenant 260 μL d’eau à utiliser comme blanc dans un lecteur de microplaques. Les MST font référence aux étalons de protéines. S1-S6 sont six échantillons différents. SX_1-SX_4 sont quatre dilutions d’échantillon différentes pour chaque échantillon. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Enregistrement des résultats
- Dans une pièce bien éclairée, tenez la microplaque parallèle à la paillasse sur un fond blanc uniforme (p. ex., une feuille de papier) d’une main. Assurez-vous d’un alignement précis en plaçant un niveau à bulle sur la plaque.
- De l’autre main, tenez le smartphone parallèlement (certaines applications d’appareil photo indiquent utilement l’inclinaison de l’appareil) à la paillasse et à la microplaque et prenez une ou plusieurs photos de l’ensemble de la microplaque (Figure 3). Sur les appareils iOS, activez l’indicateur de niveau de l’appareil photo dans les paramètres de l’appareil photo en activant l’option Grille . Sur les appareils Android, activez l’indicateur de niveau de l’appareil photo dans les paramètres de l’appareil photo en activant les conseils de cadrage.
- Aucun appareil d’éclairage spécial n’est nécessaire, mais veillez à éviter les ombres et les reflets. Par exemple, évitez d’ombrer la plaque ou l’arrière-plan avec le smartphone et évitez d’ombrer l’arrière-plan avec la microplaque. Les petits reflets sur les bords de la zone du puits ne sont pas un problème ; Les données de couleur peuvent être extraites d’une très petite zone au centre de chaque puits.
- Vérifiez brièvement l’uniformité de l’arrière-plan, les ombres et les reflets de l’image. Jetez également un coup d’œil à l’angle des puits ; Le centre de chaque puits doit être directement visible (c.-à-d. qu’il ne doit pas être situé derrière les parois du puits).
- Si l’on souhaite comparer les valeurs d’absorbance des microplaques et les données de couleur de l’image, lire la microplaque à 590 nm et 450 nm dans un lecteur de microplaques21.

Figure 3 : Capture des résultats du test de la protéine de Bradford. Dans une pièce bien éclairée, la microplaque est positionnée parallèlement à la paillasse sur un fond uniforme d’une seule main. De l’autre main, le smartphone est maintenu parallèlement à la paillasse et à la microplaque. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Extraction automatique des données de couleur
- Téléchargez ImageJ et ReadPlate22, un plugin ImageJ est disponible à https://imagej.nih.gov/ij/plugins/readplate/index.html (il s’agit d’un fichier .txt).
- Ouvrez ImageJ, cliquez sur Plugins > Installer et sélectionnez le fichier téléchargé à l’étape 6.1.
- Définissez les paramètres de mesure en cliquant sur Analyser > Définir les mesures et cochez les options suivantes : Surface ; Écart type; Valeur de gris minimale et maximale ; Valeur moyenne des gris ; Valeur modale de gris. En bas de la fenêtre, définissez Redirection vers : Aucun et Décimales (0-9) : 3.
- Allez dans Fichier > Ouvrir et sélectionnez la photo de la microplaque prise à l’étape 4.
- Allez dans Plugins > ReadPlate. Lisez les instructions, puis cliquez sur OK.
- Sélectionnez le nombre de puits : 96.
- À l’aide de l’outil de sélection rectangulaire chargé automatiquement par le plugin, faites un rectangle commençant au centre du puits A1 et se terminant au centre du puits H12. Cliquez ensuite sur OK.
- Sélectionnez le canal bleu et cliquez sur OK.
- Confirmez les paramètres par défaut en cliquant sur OK.
- Vérifiez si le logiciel a délimité une zone à l’intérieur de chaque puits et si les zones sélectionnées ne couvrent pas les zones présentant des ombres ou des reflets anormaux. Cliquez sur OK.
- Enregistrez les résultats et répétez les étapes 5.8 à 5.10 pour le canal vert.
- Calculez le rapport bleu/vert à l’aide du mode de chaque couleur.
REMARQUE : Le calcul peut être effectué manuellement ou à l’aide du logiciel des préférences du lecteur, tel que R, Microsoft Excel ou GraphPad Prism.
6. Extraction manuelle des données de couleur
- Téléchargez Inkscape, un éditeur graphique gratuit et open-source.
REMARQUE : N’importe quel logiciel doté de l’outil de sélection de couleurs (généralement représenté sous la forme d’une pipette) peut être utilisé pour identifier la couleur et extraire les données RVB. - Ouvrez Inkscape, allez dans Fichier > Ouvrir. Sélectionnez la photo de la microplaque prise à l’étape 4.
- Choisissez l’outil Sélectionner et transformer des objets (S ), représenté par une flèche en haut à gauche, puis cliquez sur l’image. Une bordure en pointillés indiquera la sélection.
- Sélectionnez l’outil Choisir les couleurs de l’image (D), qui est représenté par une pipette sur le côté gauche.
- Cliquez au centre d’un puits. La couleur du panneau inférieur (« Remplir : ») changera en conséquence. Cliquez sur la couleur et un onglet Remplissage et contour apparaîtra sur le côté droit.
- Définissez le menu déroulant Flat Color (Couleur plate ) sur RVB (RVB). Notez les valeurs indiquées pour les canaux bleu et vert de chaque puits.
- Calculez le rapport bleu/vert à l’aide des valeurs enregistrées.
REMARQUE : Le calcul peut être effectué manuellement ou à l’aide du logiciel des préférences du lecteur, tel que R, Microsoft Excel ou GraphPad Prism.
7. Construire des courbes standard et extrapoler des inconnues
- Tracez le rapport d’intensité moyen entre le vert et le bleu en fonction de la concentration standard en protéines.
REMARQUE : Tracez les données manuellement ou à l’aide du logiciel des préférences du lecteur, tel que R, Microsoft Excel ou GraphPad Prism. - Calculer le rapport d’intensité moyen entre le vert et le bleu pour chaque échantillon et chaque dilution.
- Vérifiez si le signal obtenu pour l’échantillon se situe dans la plage linéaire de l’étalon protéique.
- Ignorez toute valeur inférieure ou supérieure aux valeurs minimales ou maximales de la courbe standard.
- Utilisez l’équation linéaire décrivant la courbe standard pour extrapoler la quantité de protéines dans l’échantillon. Multipliez les valeurs calculées par le facteur de dilution en conséquence.
Résultats
La figure 4 est une image d’une microplaque à partir de laquelle les données de couleur ont été extraites et l’absorbance à 450 nm et 590 nm a été enregistrée. Les données de couleur RVB rapportées ici comme représentatives ont été obtenues automatiquement comme décrit dans la section 5. Un modèle typique des données de couleur est une augmentation des valeurs bleues et une diminution des valeurs rouges et vertes (Figure 5). Il est à noter q...
Discussion
Cet article décrit RGBradford, une méthode qui utilise l’appareil photo d’un smartphone pour enregistrer les données d’un test de protéine Bradford, extraire les données de couleur et quantifier avec précision les niveaux de protéines dans les échantillons biologiques, comme décrit récemment20. Une différence par rapport à la méthode RGBradford originale est qu’ici une procédure permettant d’obtenir automatiquement des données de couleur avec un plugin ImageJ
Déclarations de divulgation
L’auteur n’a aucun conflit d’intérêts à déclarer.
Remerciements
Ces travaux ont été financés par le Conseil national pour le développement scientifique et technologique (CNPq, Brésil) [numéros de subvention 428048/2018-8 et 402556/2022-4] et l’Université de Brasilia (Brésil). L’auteur remercie le Dr Duarte Nuno Carvalho et le Dr Evelyn Santos (i3s, Porto, Portugal) pour avoir fourni l’accès à leurs smartphones utilisés dans cette recherche.
matériels
| Name | Company | Catalog Number | Comments |
| 96-well flat-bottom polystyrene microtiter plates | Jet Biofil, Guangzhou, China | TCP011096 | Any flat-bottom microplate compativle with optical reading will suffice. |
| Bovine serum albumin | Sigma-Aldrich, St. Louis, MO | A2153 | |
| Coomassie Brilliant Blue G | Sigma-Aldrich, St. Louis, MO | B0770 | |
| Ethyl alcohol | |||
| iPhone 11 | Apple | MWM02BR/A | Can be substituted with other smartphone equiped with a camera |
| iPhone 14 Pro | Apple | N/A | |
| Phosphoric acid | Sigma-Aldrich, St. Louis, MO | 695017 | |
| Redmi Note 9 Pro | XIAOMI | N/A | |
| S22 Ultra | Samsung | N/A | |
| SpectraMax 384 Plus. Microplate reader. | Molecular Devices, San Jose, CA | PLUS 384 | Any microplate reader capable of reading at 450 nm and 590 nm will work. This is optional. The method was actually created to dismiss the need of a microplate reader. |
Références
- Zaguri, M., Kandel, S., Rinehart, S. A., Torsekar, V. R., Hawlena, D. Protein quantification in ecological studies: A literature review and empirical comparisons of standard methodologies. Methods in Ecology and Evolution. 12 (7), 1240-1251 (2021).
- Koga, T., et al. Mild electrical stimulation and heat shock ameliorates progressive proteinuria and renal inflammation in mouse model of Alport syndrome. PLoS One. 7 (8), e43852 (2012).
- Peterson, G. L. Determination of total protein. Methods in Enzymology. 91, 95-119 (1983).
- Goldfarb, A. R., Saidel, L. J., Mosovich, E. The ultraviolet absorption spectra of proteins. The Journal of Biological Chemistry. 193 (1), 397-404 (1951).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. The Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Smith, P. K., et al. Measurement of protein using bicinchoninic acid. Analytical Biochemistry. 150 (1), 76-85 (1985).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Datki, Z., et al. Application of BisANS fluorescent dye for developing a novel protein assay. PLoS One. 14 (4), e0215863 (2019).
- Van Noorden, R., Maher, B., Nuzzo, R. The top 100 papers. Nature. 514 (7524), 550-553 (2014).
- . Scopus Available from: https://www.scopus.com/ (2022)
- Compton, S. J., Jones, C. G. Mechanism of dye response and interference in the Bradford protein assay. Analytical Biochemistry. 151 (2), 369-374 (1985).
- Chial, H. J., Thompson, H. B., Splittgerber, A. G. A spectral study of the charge forms of Coomassie Blue G. Analytical Biochemistry. 209 (2), 258-266 (1993).
- Pande, S. V., Murthy, M. S. R. A modified micro-Bradford procedure for elimination of interference from sodium dodecyl sulfate, other detergents, and lipids. Analytical Biochemistry. 220 (2), 424-426 (1994).
- Gogstad, G. O., Krutnes, M. -. B. Measurement of protein in cell suspensions using the Commassie brilliant blue dye-binding assay. Analytical Biochemistry. 126 (2), 355-359 (1982).
- Friedenauer, S., Berlet, H. H. Sensitivity and variability of the Bradford protein assay in the presence of detergents. Analytical Biochemistry. 178 (2), 263-268 (1989).
- Stoscheck, C. M. Increased uniformity in the response of the Coomassie blue G protein assay to different proteins. Analytical Biochemistry. 184 (1), 111-116 (1990).
- Zor, T., Selinger, Z. Linearization of the Bradford protein assay increases its sensitivity: Theoretical and experimental studies. Analytical Biochemistry. 236 (2), 302-308 (1996).
- Gee, C. T., Kehoe, E., Pomerantz, W. C. K., Penn, R. L. Quantifying protein concentrations using smartphone colorimetry: A new method for an established test. Journal of Chemical Education. 94 (7), 941-945 (2017).
- de Camargo, C., Vicentini, M., Gobbi, A., Martinez, D., Lima, R. Smartphone for point-of-care quantification of protein by Bradford assay. Journal of the Brazilian Chemical Society. 28 (4), 689-693 (2016).
- Moreira, D. C. RGBradford: Accurate measurement of protein concentration using a smartphone camera and the blue to green intensity ratio. Analytical Biochemistry. 655, 114839 (2022).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. Journal of Visualized Experiments. (38), 1918 (2010).
- Angelani, C. R., et al. A metabolic control analysis approach to introduce the study of systems in biochemistry: the glycolytic pathway in the red blood cell: Metabolic control analysis and the glycolytic pathway. Biochemistry and Molecular Biology Education. 46 (5), 502-515 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon