A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניתוח זיהום תאי אפיתל עם שיגלה
In This Article
Summary
הפרוטוקול הנוכחי מתאר בדיקות זיהום כדי לחקור היצמדות לשיגלה , פלישה ושכפול תוך תאי באמצעות קווי תאי אפיתל במבחנה .
Abstract
הפתוגן החיידקי האנטרי המותאם לבני אדם שיגלה גורם למיליוני זיהומים מדי שנה, יוצר השפעות גדילה ארוכות טווח בקרב ילדים ומהווה גורם מוביל למוות משלשולים ברחבי העולם. זיהום גורם לשלשול מימי או דמי כתוצאה מכך שהפתוגן עובר במערכת העיכול ומדביק את תאי האפיתל המצפים את המעי הגס. עם עלייה מדהימה בעמידות לאנטיביוטיקה והמחסור הנוכחי בחיסונים מאושרים, פרוטוקולי מחקר סטנדרטיים הם קריטיים לחקר הפתוגן האימתני הזה. כאן מוצגות מתודולוגיות לבחינת הפתוגנזה המולקולרית של שיגלה באמצעות אנליזות חוץ גופיות של היצמדות חיידקים, פלישה ושכפול תוך-תאי בתאי אפיתל המעי הגס. לפני ניתוח הזיהום, פנוטיפ האלימות של מושבות שיגלה אומת על ידי ספיגת הצבע האדום של קונגו על לוחות אגר. ניתן לשקול גם שימוש בחומרי מעבדה מוספים במהלך תרבית חיידקים כדי לחקות בתנאי in vivo . תאי חיידקים משמשים לאחר מכן בפרוטוקול סטנדרטי כדי להדביק תאי אפיתל המעי הגס בלוחיות תרבית רקמה בריבוי מבוסס של זיהום עם התאמות לנתח כל שלב של זיהום. לצורך בדיקות היצמדות, תאי שיגלה מודגרים עם רמות מדיה מופחתות כדי לקדם מגע חיידקי עם תאי אפיתל. הן עבור פלישה והן עבור מבחני שכפול תוך תאי, גנטמיצין מיושם בפרקי זמן שונים כדי לחסל חיידקים חוץ-תאיים ולאפשר הערכה של פלישה ו / או כימות של שיעורי שכפול תוך תאי. כל פרוטוקולי ההדבקה מונים חיידקים דבקים, פלשו ו / או תוך תאיים על ידי דילול סדרתי של תאי אפיתל נגועים וציפוי יחידות יוצרות מושבת חיידקים ביחס לטיטרים מדביקים על לוחות אגר אדומים בקונגו. יחד, פרוטוקולים אלה מאפשרים אפיון עצמאי והשוואות עבור כל שלב של זיהום Shigella של תאי אפיתל כדי לחקור פתוגן זה בהצלחה.
Introduction
מחלות שלשול הנגרמות על ידי פתוגנים חיידקיים אנטריים מהוות נטל בריאותי עולמי משמעותי. בשנת 2016, מחלות שלשול היו אחראיות ל -1.3 מיליון מקרי מוות ברחבי העולם והיו סיבת המוות הרביעית בשכיחותה בקרב ילדים מתחת לגיל חמש שנים של 1,2. הפתוגן החיידקי הגראם-שלילי, האנטרי, שיגלה, הוא הגורם הסיבתי לשיגלוזיס, גורם עיקרי למוות משלשולים ברחבי העולם3. שיגלוזיס גורם לתחלואה ותמותה משמעותית מדי שנה בילדים ממדינות בעלות הכנסה נמוכה ובינונית 4,5, בעוד זיהומים במדינות בעלות הכנסה גבוהה קשורים להתפרצויות מעונות יום, מזון ומים 6,7,8,9. פיתוח חיסונים לא יעיל10 ועלייה בשיעורי עמידות מיקרוביאלית (AMR)11,12 סיבכו את הניהול של התפרצויות שיגלה בקנה מידה גדול. נתונים עדכניים של המרכז לבקרת מחלות ומניעתן מראים כי כמעט 46% מזיהומי שיגלה בארצות הברית הפגינו עמידות לתרופות בשנת 202013,14, בעוד ארגון הבריאות העולמי הכריז על שיגלה כפתוגן בעדיפות AMR שעבורו יש צורך דחוף בטיפולים חדשים15.
זיהומי שיגלה מועברים בקלות דרך מסלול הצואה-דרך הפה עם בליעת מזון או מים מזוהמים, או באמצעות מגע ישיר עם בני אדם. שיגלה התפתחה להיות פתוגן יעיל המותאם לבני אדם, עם מינון מדבק של 10-100 חיידקים המספיקים כדי לגרום למחלה16. במהלך מעבר במעי הדק, שיגלה חשופה לאותות סביבתיים, כגון טמפרטורה גבוהה ומרה17. זיהוי אותות אלה גורם לשינויי שעתוק לבטא גורמי אלימות המשפרים את יכולתם של החיידקים להדביק את המעי הגס האנושי 17,18,19. שיגלה אינה פולשת לאפיתל המעי הגס מפני השטח האפיקאליים, אלא עוברת דרך שכבת האפיתל בעקבות ספיגה לתאי מיקרופולד מיוחדים מציגי אנטיגן (תאי M) בתוך אפיתל 20,21,22 הקשור לזקיק. לאחר טרנסציטוזה, תאי שיגלה עוברים פאגוציטוזה על ידי מקרופאגים תושבים. שיגלה בורח במהירות מהפאגוזום וגורם למוות של תאי מקרופאגים, וכתוצאה מכך משחררים ציטוקינים מעודדי דלקת 5,23,24. שיגלה פולש אז לתאי אפיתל המעי הגס מהצד הבזולטרלי, ממקם את הוואקול המקרופינוציטי, ומבסס נישה משוכפלת בציטופלסמה 5,25. ציטוקינים מעודדי דלקת, במיוחד אינטרלוקין-8 (IL-8), מגייסים לויקוציטים נויטרופילים פולימורפו-גרעיניים (PMNs) לאתר הזיהום, מה שמחליש צמתים הדוקים אפיתל, ומאפשר חדירה חיידקית של רירית האפיתל כדי להחמיר זיהום בזולטרלי5. PMNs להרוס את רירית אפיתל נגוע כדי להכיל את הזיהום, וכתוצאה מכך הסימפטומים האופייניים של דיזנטריה חיידקית (דמית)5. למרות שמנגנוני הפלישה והשכפול התוך-תאי אופיינו ביסודיות, מחקר חדש מדגים מושגים חדשים חשובים בזיהום שיגלה, כולל ויסות אלימות במהלך מעבר במערכת העיכול (GI)17, היצמדות19, שיפור הגישה הבזולטרלית דרך חדירות מחסום26, והובלה א-סימפטומטית בילדים הסובלים מתת-תזונה27.
היכולת של Shigella spp. לגרום למחלות שלשול מוגבלת לבני אדם ולפרימטים לא אנושיים (NHP)28. מודלים של זיהום מעיים Shigella פותחו עבור דגי זברה29, עכברים30, שרקנים31, ארנבים 21,32,33 וחזירים 34,35. עם זאת, אף אחת ממערכות מודל אלה אינה יכולה לשכפל במדויק את מאפייני המחלה שנצפו במהלך זיהום אנושי36. למרות שמודלים NHP של שיגלוזיס הוקמו כדי לחקור את הפתוגנזה של שיגלה, מערכות מודל אלה יקרות ליישום ודורשות מינונים זיהומיים גבוהים באופן מלאכותי, עד תשעה סדרי גודל גבוהים יותר מהמינון המדבק של בני אדם 37,38,39,40,41,42. לפיכך, ההתאמה יוצאת הדופן של שיגלה להדבקה של פונדקאים אנושיים מחייבת שימוש בתרביות תאים שמקורן בבני אדם כדי ליצור מחדש מודלים רלוונטיים מבחינה פיזיולוגית לחקירה מדויקת של פתוגנזה של שיגלה.
כאן, מתוארים נהלים מפורטים כדי למדוד את שיעורי ההיצמדות של Shigella , פלישה ושכפול בתוך תאי אפיתל המעי הגס HT-29. באמצעות פרוטוקולים סטנדרטיים אלה, ניתן לחקור את המנגנונים המולקולריים שבאמצעותם גנים של אלימות חיידקית ואותות סביבתיים משפיעים על כל שלב של זיהום שיגלה כדי להבין טוב יותר את יחסי הגומלין הדינמיים בין המארח לפתוגן.
Protocol
1. הכנת ריאגנטים וחומרים
הערה: כל אמצעי האחסון תואמים לבדיקה באמצעות שני לוחות בעלי 6 בארות.
- TSB בינוני: הוסף 0.5 ליטר מים נטולי יונים(DI) ל-15 גרם של מרק סויה טריפטי (TSB, ראה טבלת חומרים) בינוני ואוטוקלאב. יש לאחסן בטמפרטורת החדר.
- מלחי מרה בינוניים (TSB + BS): להכנת TSB המכיל 0.4% (w/v) מלחי מרה, יש להשהות מחדש 0.06 גרם מלחי מרה (BS, ראה טבלת חומרים) ב-15 מ"ל של TSB אוטוקלאב. יש לעקר את המסנן באמצעות מסנן PES בגודל 0.22 מיקרומטר.
הערה: מלחי המרה מורכבים מתערובת של 1:1 של נתרן כולאט ונתרן דאוקסיכולאט. יש להכין מדיה טרייה מיד לפני השימוש. - DMEM + 10% (v/v) FBS: הוסף 5 מ"ל של סרום בקר עוברי (FBS) ל-45 מ"ל של מדיום הנשר המותאם של דולבקו (DMEM). יש לאחסן בטמפרטורה של 4°C.
- DMEM + gentamicin: לצינור של 50 מ"ל, הוסף 50 מ"ל DMEM ו- 50 μL של 50 מ"ג / מ"ל גנטמיצין (ראה טבלת חומרים).
הערה: יש להכין אליקוט טרי וחם באמבט מים בטמפרטורה של 37 מעלות צלזיוס לפני כל ניסוי. - PBS + 1% (v/v) Triton X-100: הוסף 150 μL של Triton X-100 עד 15 מ"ל של מלח חוצץ פוספט (PBS).
הערה: יש להכין אליקוט טרי וחם באמבט מים בטמפרטורה של 37 מעלות צלזיוס לפני כל ניסוי. - צלחות חיווי אדומות TSB + קונגו: הוסף 15 גרם TSB, 7.5 גרם אגר נבחר ו- 0.125 גרם צבע אדום קונגו (ראה טבלת חומרים) לבקבוק 1 ליטר. מוסיפים 0.5 ליטר מים DI ואוטוקלאב. יוצקים 10-20 מ"ל של מדיה לתוך צלחות פטרי סטריליות בודדות (100 מ"מ x 15 מ"מ) ולתת להתמצק.
אזהרה: אדום קונגו הוא מסרטן ורעל רבייה. יש לוודא כי הטיפול באדום קונגו מתבצע באמצעות ציוד המגן האישי המתאים. עיין בגיליון נתוני בטיחות המוצר לקבלת מידע נוסף.
הערה: כ-20 לוחות עשויים ממדיה אדומה של קונגו 0.5 ליטר. ניתן להכין צלחות 2-3 ימים מראש ולהשאיר הפוכות בטמפרטורת החדר עד לשימוש. לאחסון לטווח ארוך, הניחו צלחות הפוכות בשרוולים מפלסטיק בטמפרטורה של 4°C למשך עד 3 חודשים. - DMEM + 10% (v/v) FBS ו-5% (v/v) dimethyl sulfoxide (DMSO): הוסף 42.5 מ"ל DMEM, 5 מ"ל FBS ו-2.5 מ"ל DMSO לצינור של 50 מ"ל. יש לאחסן בטמפרטורה של 4°C.
2. הכנת חיידקים
הערה: כל פרוטוקולי הטיפוח והאחסון של מעבדת שיגלה מותאמים מפיין, S. M.43.
זהירות: Shigella spp. הם קבוצת סיכון 2 פתוגנים44. בצע את כל עבודות המעבדה בסביבת BSL-2, עם אמצעי בטיחות נוספים שננקטו כדי להגביל חשיפות מקריות עקב המינון הזיהומי הנמוך של Shigella spp.
- צמיחה של שיגלה ממלאי קפוא
- מעבירים כמות קטנה של תרבית קפואה מהבקבוקון הקריוגני לצלחת אגר אדומה TSB + קונגו באמצעות מוליך סטרילי.
- להבה מעקרת לולאת חיסון ומאפשרת לה להתקרר. פזרו את החיסון קדימה ואחורה על פני רביע אחד של הצלחת. להבעיר את הלולאה, לאפשר לה להתקרר, ואז פסים מהרביע הראשון אל הרביע השני של הצלחת. חזור על הרצף לתוך הרביע השלישי והרביעי של הצלחת.
הערה: לחלופין, יש לפזר את החיסון באמצעות מוליך סטרילי טרי בין כל רביע. - להפוך את הצלחת ולדגור על 37 מעלות צלזיוס במשך הלילה.
הערה: דגירה בטמפרטורות ≥37 מעלות צלזיוס נדרשת לביטוי גורמי אלימות שיגלה הדרושים לתצפית בפנוטיפ45 של קונגו אדום-חיובי (CR+). מושבות ארסיות יהיו בעלות מראה לבן ולא יהיו פולשניות. - אוטמים את הצלחת בסרט פרפין ומאחסנים בקירור בטמפרטורה של 4°C.
הערה: מושבות חיידקים יישארו בנות קיימא על צלחות אגר למשך 1-2 שבועות.
- גידול בן לילה של שיגלה בתרבית נוזלית
- Aliquot 3 מ"ל של מדיה TSB לתוך סטרילי 14 מ"ל צינורות תרבית.
- בחר מושבה אדומה אחת ומבודדת היטב (CR+) באמצעות אפליקטור סטרילי והשהה מחדש במדיה נוזלית.
- לדגור על תרביות לילה (16-18 שעות) ב 37 ° C עם רעד ב 250 סיבובים לדקה (סל"ד).
3. הכנת תאים איקריוטים HT-29
הערה: כל אמצעי האחסון תואמים לבדיקה באמצעות שני לוחות בעלי 6 בארות. קווי תאים HT-29 נרכשו מאוסף תרביות הסוג האמריקאי (ATCC). פרוטוקולי תחזוקה HT-29 מותאמים להמלצות ATCC46. יש לחמם מראש את כל חומרי ההדפסה באמבט מים בטמפרטורה של 37°C לפני השימוש. כל פרוטוקולי התחזוקה של HT-29 צריכים להתבצע בארון בטיחות ביולוגית. הימנע מייצור בועות בעת ערבוב/עבודה עם תאי HT-29 במדיה כדי למנוע שינויים דרמטיים ב- pH.
- הפשרת תאי HT-29 ממלאי קפוא
- הפשיר את בקבוקון תאי HT-29 באמבט מים של 37 מעלות צלזיוס.
הערה: ודא שהמכסה נשאר מלא מעל המים כדי למנוע זיהום. ההפשרה אמורה להימשך פחות מ-2 דקות. - מוציאים את הבקבוקון מהמים מיד לאחר הפשרת התרבית במלואה ומטהרים עם 70% אתנול. ודא שכל השלבים מנקודה זו מבוצעים באמצעות טכניקות אספטיות.
- הוסף את כל תכולת הבקבוקון לצינור צנטריפוגה של 15 מ"ל המכיל 9 מ"ל DMEM + 10% FBS. צנטריפוגה ב 125 x גרם במשך 5 דקות בטמפרטורת החדר.
- דקרו את הסופרנאטנט לתוך מיכל פסולת והשהו מחדש את הגלולה ב 10 מ"ל של DMEM חם + 10% FBS. העבר תאים מרחפים לבקבוק תרבית רקמהבגודל 75 ס"מ (T75) המכיל 10 מ"ל DMEM חם + 10% FBS (נפח כולל של 20 מ"ל).
- לדגור על תאים ב 37 ° C עם 5% CO2 עד התאים מגיעים 90% מפגש (כ 6-7 ימים).
הערה: המפגש נאמד באמצעות קירוב חזותי.
- הפשיר את בקבוקון תאי HT-29 באמבט מים של 37 מעלות צלזיוס.
- זריעת תאי HT-29
- טרום חם 20 מ"ל של PBS ו 50 מ"ל של DMEM + 10% FBS באמבט מים 37 ° C ו 3 מ"ל חם מראש של 0.25% (w / v) Trypsin-EDTA לטמפרטורת החדר.
- ברגע שתאי HT-29 (משלב 3.1) מגיעים למפגש של 90%, תרבית תאי HT-29 מבקבוק T75 הופכת למיכל פסולת. יוצקים ~ 10 מ"ל של PBS חם לתוך הבקבוק ומערבלים בעדינות לשטוף. לרוקן את PBS לתוך מיכל פסולת. לשטוף עם PBS חם שוב decant.
- הוסף 2-3 מ"ל של 0.25% (w/v) טריפסין-EDTA ומערבל בעדינות על פני כל שטח הפנים. יש לדגור בטמפרטורה של 37°C עם 5% CO2 למשך 4 דקות.
- הסר את הבקבוק מהאינקובטור וסובב בעדינות את הטריפסין-EDTA, תוך הבטחה חזותית שכל התאים יתנתקו מפני השטח.
- מיד להוסיף 6 מ"ל של DMEM חם + 10% FBS כדי להשבית את טריפסין. פיפטה למעלה ולמטה כדי לערבב היטב.
- מעבירים את כל התכולה לצינור צנטריפוגה של 15 מ"ל ומסובבים אותו במהירות של 500 x גרם למשך 5 דקות בטמפרטורת החדר.
- דקנט בעדינות supernatant לתוך מיכל פסולת להשעות מחדש את הגלולה ב 6 מ"ל של DMEM חם + 10% FBS.
- מיד לאחר ההשעיה, להעביר 10 μL של תאי HT-29 מרחפים מאמצע התרבית לצינור PCR 0.2 מ"ל. הוסיפו 10 μL של צבע כחול Trypan לצינור ה-PCR וערבבו.
- הוסף 10 μL של תערובת HT-29 תא/טריפאן כחול לשקופית תא מונה חד פעמית של הרוזנת (ראה טבלת חומרים). מנה את מספר התאים החיים וחשב את כדאיות התא.
הערה: בעת תיעוד מספר התאים בדגימה, קרא את המספר תחת ספירת התאים "חיה", ולא את ספירת התאים הכוללת. לחלופין, ספירת תאים יכולה להתבצע באופן ידני באמצעות המוציטומטר. - זרע תאי HT-29 מרחפים מחדש לתוך בקבוק T75 טרי או צלחת 6 בארות.
- עבור צלוחיות T75:
- פיפט בעדינות כדי לערבב, ולאחר מכן להעביר 2.5 x 106 תאים לבקבוק T75 טרי על פי המשוואה הבאה:
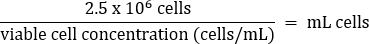
- הוסף מדיה חמה DMEM + 10% FBS לנפח סופי של 20 מ"ל (ריכוז סופי של 1.25 x 105 תאים / מ"ל).
- פזרו תאים באופן שווה על פני הבקבוק על ידי נדנוד עדין קדימה ואחורה.
- יש לדגור בטמפרטורה של 37°C עם 5%CO2 עד שהתאים מגיעים למפגש של 80%.
הערה: לצמיחה מיטבית, החלף את מדיית DMEM + 10% FBS בבקבוק T75 כל ~3 ימים. דקנט מדיה לתוך מיכל פסולת ולהוסיף 10 מ"ל של PBS חם לצלוחית. סובבו את PBS בעדינות והכניסו אותו למיכל הפסולת. לאחר מכן להוסיף 20 מ"ל של DMEM טרי וחם + 10% FBS לבקבוק ולחזור 37 ° C, 5% CO2 אינקובטור.
- פיפט בעדינות כדי לערבב, ולאחר מכן להעביר 2.5 x 106 תאים לבקבוק T75 טרי על פי המשוואה הבאה:
- לצלחת 6 בארות:
- פיפט בעדינות כדי לערבב, ולאחר מכן להעביר 5.85 x 106 תאים לצינור חרוטי טרי 50 מ"ל על פי המשוואה הבאה:
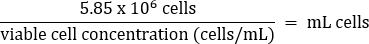
- הוסף מדיה חמה DMEM + 10% FBS לנפח סופי של 26 מ"ל (ריכוז סופי של 2.25 x 105 תאים / מ"ל).
- מפטמים בעדינות כדי לערבב, ואז מוציאים 2 מ"ל (4.5 x 105 תאים) לתוך בארות בודדות של צלחות 6 בארות.
- פזרו תאים באופן שווה על פני הבאר על ידי נדנוד עדין למעלה/למטה ושמאלה / ימינה 2-3x.
- יש לדגור בטמפרטורה של 37°C עם 5%CO2 עד שהתאים מגיעים למפגש של 80%-95% (כ-3-4 ימים).
הערה: 85% מפגש מומלץ למבחני פלישה ושכפול תוך תאי, בעוד 90%-95% מפגש מומלץ למבחני היצמדות. התאים צריכים להגיע למפגש ~85% לאחר דגירה של 48 שעות עם ריכוז סופי של כ 1 x 106 תאים / באר. ייתכן שיידרשו התאמות למספר התאים שנזרעו ולמשך הדגירה.
- פיפט בעדינות כדי לערבב, ולאחר מכן להעביר 5.85 x 106 תאים לצינור חרוטי טרי 50 מ"ל על פי המשוואה הבאה:
- עבור צלוחיות T75:
- ביצוע מניות HT-29 קפואות
- Aliquot 1 מ"ל של DMEM + 10% FBS + 5% מדיה DMSO לתוך בקבוקונים קריוגניים בודדים.
- הוסף 1 x 106 תאי HT-29 משלב 3.2.7 לכל בקבוקון. חשב את נפח התאים לפי הנוסחה הבאה:
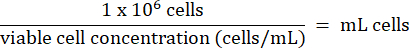
- אחסנו תאי HT-29 לטווח ארוך מתחת ל-130°C במקפיא אחסון אדי חנקן נוזלי.
4. בדיקת דבקות
הערה: כל אמצעי האחסון תואמים לבדיקה באמצעות שני לוחות בעלי 6 בארות.
- תת-תרבות בן לילה תרבויות שיגלה באמצעות דילול 1:50 למדיה רעננה.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
הערה: הגבל את נפח התרבית ל-<20% מנפח בקבוק התרבית או הצינורית כדי להבטיח אוורור נאות. - יש לדגור ב-37°C עם רעידות ב-250 סל"ד עד שהתאים מגיעים לצפיפות אופטית (OD600) של 0.7 (שלב אמצע לוג של צמיחת שיגלה ); בערך 2-2.5 שעות.
הערה: במהלך תת-התרבית, aliquot 50 מ"ל של DMEM ונפח מספיק של PBS עבור כל שלבי הכביסה ומניחים באמבט מים 37 °C. אפשר לחומרי הדפסה להגיע ל-37°C לפני השימוש.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
- העבר 2 x 108 יחידות יוצרות מושבה (CFUs) תת-תרבית Shigella לצינורות מיקרוצנטריפוגות בודדים של 2 מ"ל.
הערה: 2 x 108 יחידות יוצרות מושבה (CFU) מתאימות לכ-1 מ"ל של תאי חיידקים ב-OD600 של 0.7. השתמש בקריאות OD600 כדי להעריך CFU/מ"ל בקירוב בהתאם לכיול של כל ספקטרופוטומטר בנפרד. - שטפו כל דגימת שיגלה 2x עם PBS.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר. שואפים את הסופרנטנט, ואז מוסיפים 1 מ"ל של PBS חם ומרחפים מחדש את הגלולה היטב, בעדינות מעלים ומורידים את הדגימה עד שהתערובת הומוגנית לחלוטין (8-10x).
- חזור על שלב 4.3.1 פעם נוספת אחת.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר, שואפים את supernatant, ו resuspend את הכדורים ב 2 מ"ל של DMEM חם.
הערה: הריכוז הסופי של חיידקים מרחפים יהיה 1 x 108 CFU/mL.
- מערבולת, ולאחר מכן להוסיף 1 מ"ל (1 x 108 CFUs) של Shigella מושעה מחדש לכל באר של HT-29 monolayers אפיתל המעי הגס מוכן בלוחות 6 באר (משלב 3.2.10.2).
הערה: זיהומים מבוצעים בדרך כלל בריבוי זיהומים (MOI; יחס בין תאי חיידקים לתאי אפיתל) של 100. כדי לבדוק MOI שונים, לדלל Shigella resuspended ב DMEM חם לריכוז הרצוי, ולאחר מכן להוסיף 1 מ"ל של חיידקים מדוללים HT-29 monolayers. לדוגמה, כדי לבדוק MOI של 10, לדלל חיידקים ביחס של 1:10 על-ידי הוספת 150 μL של 1 x 108 CFU/mL חיידקים ל-1.35 מ"ל של DMEM חם, ולאחר מכן להחיל 1 מ"ל (1 x 107 CFU) על תאי HT-29. - לדגור על צלחות 6 בארות ב 37 ° C עם 5% CO2 במשך 3 שעות.
- במהלך הדגירה, לקבוע את titer זיהום חיידקי.
- הכינו דילולים סדרתיים פי 10 של תאי Shigella מרחפים מחדש (משלב 4.3.3) לתוך PBS.
- צלחת 100 μL של 1 x 10-5 ו 1 x 10-6 דילול על TSB + קונגו לוחות אדומים לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-5 ו- 1 x 10-6 מתאים למקדם דילול סופי של 1 x 10-6 ו- 1 x 10-7, בהתאמה.
- לאחר הדגירה, לשטוף את monolayers 4-5x עם PBS.
- שאפו מדיה מכל באר.
הערה: בעת שאיפת מדיה מלוחות 6 בארות, יש להנחות את קצה השואף לאורך הצד התחתון של הבארות, בניסיון להימנע ממגע עם תאי HT-29. - מוסיפים 1 מ"ל PBS חמים לכל באר ושוטפים בעדינות.
הערה: כדי לשטוף בעדינות 6 שכבות עם PBS, הזיזו את הצלחת למעלה ולמטה ומצד לצד על הספסל. שטיפת צלחות בתנועה מעגלית ו/או הסרת הצלחת משטח הספסל עלולה לגרום להסרה מכנית של תאים מפלסטיק. - חזור על שלבים 4.7.1 ו- 4.7.2 4x פעמים נוספות.
- שאפו מדיה מכל באר.
- הסר PBS על ידי שאיפה וליז תאי HT-29 על ידי הוספת 1 מ"ל של PBS + 1% Triton X-100 לכל באר.
- לדגור על צלחות 6 בארות ב 37 ° C במשך 5 דקות.
- השתמש מגרד תאים או קצה פיפטה כפוף כדי לגרד את התאים lysed מתחתית הבאר ולהעביר את 1 מ"ל מלא לתוך צינור microcentrifuge טרי 1.7 מ"ל.
- לקבוע את מספר החיידקים הקשורים לתאים.
- מערבול כל צינור (משלב 4.10) במשך 30 שניות לפחות כדי לעקור עוד יותר את שיגלה מהתאים האיקריוטים הליזדים.
- הכינו דילולים סדרתיים פי 10 של ליזטים לתוך PBS.
- צלחת 100 μL של 1 x 10-2, 1 x 10-3, ו 1 x 10-4 דילול על TSB + קונגו לוחות אדומים לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-2, 1 x 10-3 ו- 1 x 10-4 מתאים למקדם דילול סופי של 1 x 10-3, 1 x 10-4 ו- 1 x 10-5, בהתאמה.
5. מבחן הפלישה
הערה: כל אמצעי האחסון תואמים לבדיקה באמצעות שני לוחות בעלי 6 בארות.
- תת-תרבות בן לילה תרבויות שיגלה באמצעות דילול 1:50 למדיה רעננה.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
הערה: הגבל את נפח התרבית ל-<20% מנפח בקבוק התרבית או הצינורית כדי להבטיח אוורור נאות. - דוגרים ב-37°C עם טלטול ב-250 סל"ד עד שהתאים מגיעים ל-OD600 של 0.7 (שלב אמצע לוג של צמיחת Shigella ); בערך 2-2.5 שעות.
הערה: במהלך תת-התרבית, aliquot 50 מ"ל של DMEM + 50 מ"ג / מ"ל gentamicin ונפח מספיק של PBS עבור כל שלבי הכביסה ומניחים באמבט מים 37 °C. אפשר לחומרי הדפסה להגיע ל-37°C לפני השימוש.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
- העבר 2 x 108 יחידות יוצרות מושבה ( CFU) לצינורות מיקרוצנטריפוגות בודדות בתרבית של 2 מ"ל.
הערה: 2 x 108 יחידות יוצרות מושבה (CFU) מתאימות לכ-1 מ"ל של תאי חיידקים ב-OD600 של 0.7. השתמש בקריאות OD600 כדי להעריך CFU/מ"ל בקירוב בהתאם לכיול של כל ספקטרופוטומטר בנפרד. - לשטוף דגימות Shigella 1x עם PBS.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר. שואפים את הסופרנטנט, ואז מוסיפים 1 מ"ל של PBS חם ומרחפים מחדש את הגלולה היטב, בעדינות מעלים ומורידים את הדגימה עד שהתערובת הומוגנית לחלוטין (8-10x).
- חזור על שלב 5.3.1. 1x זמן נוסף.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר, שואפים את supernatant, ו resuspend את הכדורים ב 2 מ"ל של DMEM חם.
הערה: הריכוז הסופי של חיידקים מרחפים יהיה 1 x 108 CFU/mL.
- מערבולת, ולאחר מכן להוסיף 1 מ"ל (1 x 108 CFUs) של Shigella מושעה בתוספת 1 מ"ל של DMEM לכל באר של monolayers אפיתל המעי הגס HT-29 מוכן בלוחות 6 באר (משלב 3.2.10.2).
הערה: זיהומים מבוצעים בדרך כלל בריבוי זיהומים (MOI; יחס בין תאי חיידקים לתאי אפיתל) של 100. כדי לבדוק MOI שונים, לדלל Shigella resuspended ב DMEM לריכוז הרצוי, ולאחר מכן להוסיף 1 מ"ל של חיידקים מדוללים HT-29 monolayers. לדוגמה, כדי לבדוק MOI של 10, לדלל חיידקים ביחס של 1:10 על-ידי הוספת 150 μL של 1 x 108 CFU/mL חיידקים ל-1.35 מ"ל DMEM, ולאחר מכן הוסף 1 מ"ל (1 x 107 CFU) לתאי HT-29. - כדי לקדם מגע חיידקי עם תאי HT-29, צנטריפוגו את לוחות 6 הקידוחים במהירות של 2,000 x גרם למשך 10 דקות בטמפרטורת החדר או 37°C אם ניתן לכוונן את הגדרת הטמפרטורה.
הערה: צנטריפוגה מקדמת מגע חיידקי עם תאי HT-29, אשר עוקף את הצורך בגורמי היצמדות ומאפשר לחיידקים לפלוש במהירות לתאים. - יש לדגור על צלחות 6 בארות בטמפרטורה של 37°C עם 5% CO2 למשך 45 דקות.
- במהלך הדגירה, לקבוע את titer זיהום חיידקי.
- הכינו דילולים סדרתיים פי 10 של תאי Shigella מרחפים מחדש (משלב 5.3.3) לתוך PBS.
- צלחת 100 μL של 1 x 10-5 ו 1 x 10-6 דילול על TSB + קונגו לוחות אדומים לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-5 ו- 1 x 10-6 מתאים למקדם דילול סופי של 1 x 10-6 ו- 1 x 10-7, בהתאמה.
- לשטוף ביסודיות תאי HT-29 נגועים 3x עם 1 מ"ל של PBS.
- שאפו מדיה מכל באר.
הערה: בעת שאיפת מדיה מלוחות 6 בארות, יש להנחות את קצה השואף לאורך הצד התחתון של הבארות, בניסיון להימנע ממגע עם תאי HT-29. - מוסיפים 1 מ"ל PBS חמים לכל באר ושוטפים בעדינות.
הערה: כדי לשטוף בעדינות 6 שכבות עם PBS, הזיזו את הצלחת למעלה ולמטה ומצד לצד על הספסל. שטיפת צלחות בתנועה מעגלית ו/או הסרת הצלחת משטח הספסל עלולה לגרום להסרה מכנית של תאים מפלסטיק. - חזור על שלבים 5.8.1 ו- 5.8.2 פעמיים נוספות.
- שאפו מדיה מכל באר.
- הסר PBS על ידי שאיפה, ולאחר מכן להוסיף 2 מ"ל של DMEM חם בתוספת 50 מיקרוגרם / מ"ל gentamicin לכל באר לדגור במשך 30 דקות ב 37 ° C עם 5% CO2.
- לשטוף ביסודיות תאי HT-29 נגועים 3x עם 1 מ"ל של PBS.
- חזור על שלב 5.8 של הכביסה.
- הסר PBS על ידי שאיפה, ולאחר מכן להוסיף 2 מ"ל של DMEM חם בתוספת 50 מיקרוגרם / מ"ל gentamicin לכל באר לדגור במשך 60 דקות ב 37 ° C עם 5% CO2.
- לשטוף ביסודיות תאי HT-29 נגועים 3x עם 1 מ"ל של PBS.
- חזור על שלב 5.8 של הכביסה.
- הסר PBS על ידי שאיפה וליז תאי HT-29 על ידי הוספת 1 מ"ל של PBS + 1% Triton X-100 לכל באר.
- לדגור על צלחות 6 בארות ב 37 ° C במשך 5 דקות.
- השתמש מגרד תאים או קצה פיפטה כפוף כדי לגרד את התאים lysed מתחתית הבאר ולהעביר את 1 מ"ל מלא לתוך צינור microcentrifuge טרי 1.7 מ"ל.
- לקבוע את מספר חיידקים תאיים.
- מערבול כל צינור (משלב 5.15) במשך 30 שניות לפחות כדי לעקור עוד יותר את שיגלה מהתאים האיקריוטים הליזדים.
- הכינו דילולים סדרתיים פי 10 של ליזטים לתוך PBS.
- צלחת 100 μL של 1 x 10-2 ו 1 x 10-3 דילול על TSB + קונגו לוחות אדומים לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-2 ו- 1 x 10-3 מתאים למקדם דילול סופי של 1 x 10-3 ו- 1 x 10-4, בהתאמה.
6. בדיקת שכפול תוך תאי
הערה: כל אמצעי האחסון תואמים לבדיקה באמצעות שני לוחות בעלי 6 בארות.
- תת-תרבות בן לילה תרבויות שיגלה באמצעות דילול 1:50 למדיה רעננה.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
הערה: הגבל את נפח התרבית ל-<20% מנפח בקבוק התרבית או הצינורית כדי להבטיח אוורור נאות. - דוגרים ב-37°C עם טלטול ב-250 סל"ד עד שהתאים מגיעים ל-OD600 של 0.7 (שלב אמצע לוג של צמיחת Shigella ); בערך 2-2.5 שעות.
הערה: במהלך תת-התרבית, aliquot 50 מ"ל של DMEM + 50 מ"ג / מ"ל gentamicin ונפח מספיק של PBS עבור כל שלבי הכביסה ומניחים באמבט מים 37 °C. אפשר לחומרי הדפסה להגיע ל-37°C לפני השימוש.
- מערבולת, ולאחר מכן להוסיף 100 μL של כל תרבית לילה ל 5 מ"ל של TSB טרי או TSB + BS בצינור תרבית בגודל מתאים.
- העבר 2 x 108 יחידות יוצרות מושבה ( CFU) לצינורות מיקרוצנטריפוגות בודדות בתרבית של 2 מ"ל.
הערה: 2 x 108 יחידות יוצרות מושבה (CFU) מתאימות לכ-1 מ"ל של תאי חיידקים ב-OD600 של 0.7. השתמש בקריאות OD600 כדי להעריך CFU/מ"ל בקירוב בהתאם לכיול של כל ספקטרופוטומטר בנפרד. - לשטוף דגימות Shigella 1x עם PBS.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר. שואפים את הסופרנטנט, ואז מוסיפים 1 מ"ל של PBS חם ומרחפים מחדש את הגלולה היטב, בעדינות מעלים ומורידים את הדגימה עד שהתערובת הומוגנית לחלוטין (8-10x).
- חזור על שלב 6.3.1. 1x זמן נוסף.
- תאי גלולה על ידי צנטריפוגה ב 17,000 x גרם במשך 2 דקות בטמפרטורת החדר, שואפים את supernatant, ו resuspend את הכדורים ב 2 מ"ל של DMEM חם.
הערה: הריכוז הסופי של חיידקים מרחפים יהיה 1 x 108 CFU/mL.
- מערבולת, ולאחר מכן להוסיף 1 מ"ל (1 x 108 CFUs) של Shigella מושעה בתוספת 1 מ"ל של DMEM לכל באר של HT-29 monolayers אפיתל המעי הגס מוכן בלוחות 6 באר (משלב 3.2.10.2).
הערה: זיהומים מבוצעים בדרך כלל בריבוי זיהומים (MOI; יחס בין תאי חיידקים לתאי אפיתל) של 100. כדי לבדוק MOI שונים, לדלל Shigella resuspended ב DMEM לריכוז הרצוי, ולאחר מכן להוסיף 1 מ"ל של חיידקים מדוללים HT-29 monolayers. לדוגמה, כדי לבדוק MOI של 10, לדלל חיידקים 1:10 על ידי הוספת 150 μL של 1 x 108 CFU/mL חיידקים ל 1.35 מ"ל של DMEM, ולאחר מכן להחיל 1 מ"ל (1 x 107 CFUs) על תאי HT-29. - כדי לקדם מגע חיידקי עם תאי HT-29, צנטריפוגו את לוחות 6 הקידוחים במהירות של 2,000 x גרם למשך 10 דקות בטמפרטורת החדר או 37°C אם ניתן לכוונן את הגדרת הטמפרטורה.
הערה: צנטריפוגה מקדמת מגע חיידקי עם תאי HT-29, אשר עוקף את הצורך בגורמי היצמדות ומאפשר לחיידקים לפלוש במהירות לתאים. - יש לדגור על צלחות 6 בארות בטמפרטורה של 37°C עם 5% CO2 למשך 45 דקות.
- במהלך הדגירה, לקבוע את titer זיהום חיידקי.
- הכינו דילולים סדרתיים פי 10 של תאי Shigella מרחפים מחדש (משלב 6.3.3) לתוך PBS.
- צלחת 100 μL של 1 x 10-5 ו 1 x 10-6 דילול על TSB + קונגו לוחות אדומים לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-5 ו- 1 x 10-6 מתאים למקדם דילול סופי של 1 x 10-6 ו- 1 x 10-7, בהתאמה.
- לשטוף ביסודיות תאי HT-29 נגועים 3x עם 1 מ"ל של PBS.
- שאפו מדיה מכל באר.
הערה: בעת שאיפת מדיה מלוחות 6 בארות, יש להנחות את קצה השואף לאורך הצד התחתון של הבארות, בניסיון להימנע ממגע עם תאי HT-29. - מוסיפים 1 מ"ל PBS חמים לכל באר ושוטפים בעדינות.
הערה: כדי לשטוף בעדינות 6 שכבות עם PBS, הזיזו את הצלחת למעלה ולמטה ומצד לצד על הספסל. שטיפת צלחות בתנועה מעגלית ו/או הסרת הצלחת משטח הספסל עלולה לגרום להסרה מכנית של תאים מפלסטיק. - חזור על שלבים 6.8.1 ו- 6.8.2 פעמיים נוספות.
- שאפו מדיה מכל באר.
- הסר PBS על ידי שאיפה, ולאחר מכן להוסיף 2 מ"ל של DMEM חם בתוספת 50 מיקרוגרם / מ"ל gentamicin לכל באר לדגור במשך 30 דקות ב 37 ° C עם 5% CO2.
- לשטוף ביסודיות תאי HT-29 נגועים 3x עם 1 מ"ל של PBS.
- חזור על שלב הכביסה 6.8.
- הסר PBS על ידי שאיפה, ולאחר מכן להוסיף 2 מ"ל של DMEM חם עם 50 מיקרוגרם / מ"ל gentamicin לכל באר של לוחות 6 באר לדגור ב 37 ° C עם 5% CO2 למשך הזמן הרצוי כדי לאפשר שכפול תוך תאי (עד 24 שעות).
- לשטוף ביסודיות תאים 2x עם 1 מ"ל של PBS.
- חזור על שלב הכביסה 6.8.
- הסר PBS על ידי שאיפה וליז תאי HT-29 על ידי הוספת 1 מ"ל של PBS + 1% Triton X-100 לכל באר.
- לדגור על צלחות 6 בארות ב 37 ° C במשך 5 דקות.
- השתמש מגרד תאים או קצה פיפטה כפוף כדי לגרד את התאים lysed מתחתית הבאר ולהעביר את 1 מ"ל מלא לתוך צינור microcentrifuge טרי 1.7 מ"ל.
- לקבוע את מספר חיידקים תאיים.
- מערבול כל צינור (משלב 6.15) במשך 30 שניות לפחות כדי לעקור עוד יותר את שיגלה מהתאים האיקריוטים הליזדים.
- הכינו דילולים סדרתיים פי 10 של ליזטים לתוך PBS.
- צלחת 100 μL של 1 x 10-2, 1 x 10-3, ו 1 x 10-4 דילול על TSB + קונגו צלחות אדומות לדגור לילה ב 37 ° C.
הערה: ציפוי 100 μL מהדילולים 1 x 10-2, 1 x 10-3 ו- 1 x 10-4 מתאים למקדם דילול סופי של 1 x 10-3, 1 x 10-4 ו- 1 x 10-5, בהתאמה.
תוצאות
בדיקות היצמדות, פלישה ושכפול תוך-תאי בוצעו בהשוואה בין S. flexneri 2457T wild type (WT) ל-S. flexneri ΔVF (ΔVF), מוטנט ששיערו כי הוא מווסת באופן שלילי את אלימות Shigella. מכיוון ששיגלה משתמשת במלחי מרה כאות לוויסות אלימות 17,18,47, ניסויים בוצעו לאחר תת-תרבית חיידקית במדיה TSB וכן TSB בתוספת 0.4% (w/v) מ?...
Discussion
פרוטוקול זה מתאר סדרה של שלוש בדיקות סטנדרטיות לחקר היצמדות לשיגלה, פלישה ושכפול תוך-תאי של תאי אפיתל במעי. למרות ששיטות אלה הן בסך הכל גרסאות מותאמות של מבחני גנטמיצין קלאסיים המשמשים לחקר הפלישה והשכפול התוך-תאי של פתוגנים חיידקיים שונים בתוך תאי המאכסן49,50,51
Disclosures
המחברים מצהירים כי אין ניגודי עניינים.
Acknowledgements
התמיכה במחברים כוללת את המחלקה לרפואת ילדים של בית החולים הכללי של מסצ'וסטס, פרס הוועד המנהל למימון תמיכה זמנית במחקר (ISF) לשנת 2022A009041, מענק המכון הלאומי לאלרגיה ומחלות זיהומיות R21AI146405, והמכון הלאומי לסוכרת ומחלות עיכול וכליות מענק תזונה במרכז המחקר להשמנת יתר בהרווארד (NORCH) 2P30DK040561-26. למממנים לא היה כל תפקיד בעיצוב המחקר, באיסוף הנתונים ובניתוחם, בהחלטה על פרסומם או בהכנת כתב היד.
Materials
| Name | Company | Catalog Number | Comments |
| 0.22 μm PES filter | Millipore-Sigma | SCGP00525 | Sterile, polyethersulfone filter for sterilizing up to 50 mL media |
| 14 mL culture tubes | Corning | 352059 | 17 mm x 100 mm polypropylene test tubes with cap |
| 50 mL conical tubes | Corning | 430829 | 50 mL clear polypropylene conical bottom centrifuge tubes with leak-proof cap |
| 6-well tissue culture plates | Corning | 3516 | Plates are treated for optimal cell attachment |
| Bile salts | Sigma-Aldrich | B8756 | 1:1 ratio of cholate to deoxycholate |
| Congo red dye | Sigma-Aldrich | C6277 | A benzidine-based anionic diazo dye, >85% purity |
| Countess cell counting chamber slide | Invitrogen | C10283 | To be used with the Countess Automated Cell Counter |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | A a highly polar organic reagent |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 10569-010 | DMEM is supplemented with high glucose, sodium pyruvate, GlutaMAX, and Phenol Red |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F4135 | Heat-inactivated, sterile |
| Gentamicin | Sigma-Aldrich | G3632 | Stock concentration is 50 mg/mL |
| HT-29 cell line | ATCC | HTB-38 | Adenocarcinoma cell line; colorectal in origin |
| Paraffin film | Bemis | PM999 | Laboratory sealing film |
| Petri dishes | Thermo Fisher Scientific | FB0875713 | 100 mm x 15 mm Petri dishes for solid media |
| Phosphate-buffered saline (PBS) | Thermo Fisher Scientific | 10010049 | 1x concentration; pH 7.4 |
| Select agar | Invitrogen | 30391023 | A mixture of polysaccharides extracted from red seaweed cell walls to make bacterial plating media |
| T75 flasks | Corning | 430641U | Tissue culture flasks |
| Triton X-100 | Sigma-Aldrich | T8787 | A common non-ionic surfactant and emulsifier |
| Trypan blue stain | Invitrogen | T10282 | A dye to detect dead tissue culture cells; only live cells can exclude the dye |
| Trypsin-EDTA | Gibco | 25200-056 | Reagent for cell dissociation for cell line maintenance and passaging |
| Tryptic Soy Broth (TSB) | Sigma-Aldrich | T8907 | Bacterial growth media |
References
- Karambizi, N. U., McMahan, C. S., Blue, C. N., Temesvari, L. A. Global estimated Disability-Adjusted Life-Years (DALYs) of diarrheal diseases: A systematic analysis of data from 28 years of the global burden of disease study. PloS one. 16 (10), e0259077 (2021).
- WHO. WHO methods and data sources for country-level causes of death 2000-2016. World Health Organization. , (2018).
- Kotloff, K. L. Shigella infection in children and adults: a formidable foe. Lancet Glob Health. 5 (12), e1166-e1167 (2017).
- Kotloff, K. L., et al. Burden and aetiology of diarrhoeal disease in infants and young children in developing countries (the Global Enteric Multicenter Study, GEMS): A prospective, case-control study. Lancet. 382 (9888), 209-222 (2013).
- Schroeder, G. N., Hilbi, H. Molecular pathogenesis of Shigella spp.: Controlling host cell signaling, invasion, and death by type III secretion. Clin Microbiol Rev. 21 (1), 134-156 (2008).
- Arvelo, W., et al. Transmission risk factors and treatment of pediatric shigellosis during a large daycare center-associated outbreak of multidrug resistant shigella sonnei: Implications for the management of shigellosis outbreaks among children. Pediatr Infect Dis J. 28 (11), 976-980 (2009).
- Kozyreva, V. K., et al. Recent outbreaks of Shigellosis in California caused by two distinct populations of Shigella sonnei with either increased virulence or fluoroquinolone resistance. mSphere. 1 (6), 1-18 (2016).
- Bowen, A., et al. Importation and domestic transmission of Shigella sonnei resistant to ciprofloxacin - United States, May 2014-February 2015. MMWR Morb Mortal Wkly Rep. 64 (12), 318-320 (2015).
- Tansarli, G. S., et al. Genomic reconstruction and directed interventions in a multidrug-resistant Shigellosis outbreak in Seattle, WA, USA: a genomic surveillance study. Lancet. 3099 (22), 1-11 (2023).
- Barry, E. M., et al. Progress and pitfalls in Shigella vaccine research. Nat Rev Gastroenterol Hepatol. 10 (4), 245-255 (2013).
- Increase in Extensively Drug-Resistant Shigellosis in the United States. CDC Health Alert Network. Centers for Disease Control and Prevention Available from: https://emergency.cdc.gov/han/2023/han00486.asp?ACSTrackingID=USCDC_511-DM100260&ACSTrackingLabel=HAN%20486%20-%20General%20Public&deliveryName=USCDC_511-DM100260 (2023)
- Shiferaw, B., et al. Antimicrobial susceptibility patterns of Shigella isolates in Foodborne Diseases Active Surveillance Network (FoodNet) sites, 2000-2010. Clin Infect Dis. 54, S458-S463 (2012).
- Centers for Disease Control and Prevention. COVID-19: U.S. Impact on Antimicrobial Resistance, Special Report 2022. Atlanta, GA: U.S. Department of Health and Human Services. CDC. , (2022).
- Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019. CDC. 10 (1), (2019).
- WHO. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. WHO. , (2017).
- DuPont, H. L., Levine, M. M., Hornick, R. B., Formal, S. B. Inoculum size in shigellosis and implications for expected mode of transmission. J Infect Dis. 159 (6), 1126-1128 (1989).
- Nickerson, K. P., et al. Analysis of Shigella flexneri resistance, biofilm formation, and transcriptional profile in response to bile salts. Infect Immun. 85 (6), 1-18 (2017).
- Faherty, C. S., Redman, J. C., Rasko, D. A. Shigella flexneri effectors OspE1 and OspE2 mediate induced adherence to the colonic epithelium following bile salts exposure. Mol Microbiol. 85 (1), 107-121 (2012).
- Chanin, R. B., et al. Shigella flexneri adherence factor expression in in vivo-like conditions. mSphere. 4 (6), e00751 (2019).
- Baranov, V., Hammarström, S. Carcinoembryonic antigen (CEA) and CEA-related cell adhesion molecule 1 (CEACAM1), apically expressed on human colonic M cells, are potential receptors for microbial adhesion. Histochem Cell Biol. 121 (2), 83-89 (2004).
- Wassef, J. S., Keren, D. F., Mailloux, J. L. Role of M cells in initial antigen uptake and in ulcer formation in the rabbit intestinal loop model of shigellosis. Infect Immun. 57 (3), 858-863 (1989).
- Sansonetti, P. J., Arondel, J., Cantey, J. R., Prévost, M. C., Huerre, M. Infection of rabbit Peyer's patches by Shigella flexneri: Effect of adhesive or invasive bacterial phenotypes on follicle-associated epithelium. Infect Immun. 64 (7), 2752-2764 (1996).
- Sansonetti, P. J., et al. Caspase-1 activation of IL-1beta and IL-18 are essential for Shigella flexneri-induced inflammation. Immunity. 12 (5), 581-590 (2000).
- Zychlinsky, A., Fitting, C., Cavaillon, J. M., Sansonetti, P. J. Interleukin 1 is released by murine macrophages during apoptosis induced by Shigella flexneri. J Clin Invest. 94 (3), 1328-1332 (1994).
- Sansonetti, P. J., Ryter, A., Clerc, P., Maurelli, A. T., Mounier, J. Multiplication of Shigella flexneri within HeLa cells: lysis of the phagocytic vacuole and plasmid-mediated contact hemolysis. Infect Immun. 51 (2), 461-469 (1986).
- Maldonado-Contreras, A., et al. Shigella depends on SepA to destabilize the intestinal epithelial integrity via cofilin activation. Gut Microbes. 8 (6), 544-560 (2017).
- Collard, J. -. M., et al. High prevalence of small intestine bacteria overgrowth and asymptomatic carriage of enteric pathogens in stunted children in Antananarivo, Madagascar. PLoS Negl Trop Dis. 16 (5), e0009849 (2022).
- Mattock, E., Blocker, A. J. How do the virulence factors of shigella work together to cause disease. Front Cell Infect Microbiol. 7, 1-24 (2017).
- Mostowy, S., et al. The zebrafish as a new model for the in vivo study of Shigella flexneri interaction with phagocytes and bacterial autophagy. PLoS Pathog. 9 (9), e1003588 (2013).
- Martinez-Becerra, F. J., et al. Parenteral immunization with IpaB/IpaD protects mice against lethal pulmonary infection by Shigella. Vaccine. 31 (24), 2667-2672 (2013).
- Shim, D. -. H., et al. New animal model of shigellosis in the Guinea pig: its usefulness for protective efficacy studies. J Immunol. 178 (4), 2476-2482 (2007).
- Marteyn, B., et al. Modulation of Shigella virulence in response to available oxygen in vivo. Nature. 465 (7296), 355-358 (2010).
- West, N. P., et al. Optimization of virulence functions through glucosylation of Shigella LPS. Science. 307 (5713), 1313-1317 (2005).
- Maurelli, A. T., et al. Shigella infection as observed in the experimentally inoculated domestic pig, Sus scrofa domestica. Microbial Pathog. 25 (4), 189-196 (1998).
- Jeong, K. -. I., Zhang, Q., Nunnari, J., Tzipori, S. A piglet model of acute gastroenteritis induced by Shigella dysenteriae Type 1. J Infect Dis. 201 (6), 903-911 (2010).
- Kim, Y. -. J., Yeo, S. -. G., Park, J. -. H., Ko, H. -. J. Shigella vaccine development: prospective animal models and current status. Curr Pharm Biotechnol. 14 (10), 903-912 (2013).
- Kent, T. H., Formal, S. B., LaBrec, E. H., Sprinz, H., Maenza, R. M. Gastric shigellosis in rhesus monkeys. Am J Pathol. 51 (2), 259-267 (1967).
- Shipley, S. T., et al. A challenge model for Shigella dysenteriae 1 in cynomolgus monkeys (Macaca fascicularis). Comp Med. 60 (1), 54-61 (2010).
- Higgins, R., Sauvageau, R., Bonin, P. Shigella flexneri Type 2 Infection in captive nonhuman primates. Can Vet J. 26 (12), 402-403 (1985).
- Oaks, E. V., Hale, T. L., Formal, S. B. Serum immune response to Shigella protein antigens in rhesus monkeys and humans infected with Shigella spp. Infect Immun. 53 (1), 57-63 (1986).
- Formal, S. B., et al. Protection of monkeys against experimental shigellosis with a living attenuated oral polyvalent dysentery vaccine. J Bacteriol. 92 (1), 17-22 (1966).
- Levine, M. M., Kotloff, K. L., Barry, E. M., Pasetti, M. F., Sztein, M. B. Clinical trials of Shigella vaccines: two steps forward and one step back on a long, hard road. Nat Rev Microbiol. 5 (7), 540-553 (2007).
- Payne, S. M. Laboratory cultivation and storage of Shigella. Curr Protoc Microbiol. 55 (1), 93 (2019).
- NIH Guidelines. NIH guidelines for research involving recombinant or synthetic nucleic acid molecules. NIH Guidelines. 2, 142 (2019).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Loss of pigmentation in Shigella flexneri 2a is correlated with loss of virulence and virulence-associated plasmid. Infect Immun. 43 (1), 397-401 (1984).
- HT-29 cell line product sheet. ATCC Available from: https://www.atcc.org/products/htb-38 (2023)
- Sistrunk, J. R., Nickerson, K. P., Chanin, R. B., Rasko, D. A., Faherty, C. S. Survival of the fittest: How bacterial pathogens utilize bile to enhance infection. Clin Microbiol Rev. 29 (4), 819-836 (2016).
- Stensrud, K. F., et al. Deoxycholate interacts with IpaD of Shigella flexneri in inducing the recruitment of IpaB to the type III secretion apparatus needle tip. J Biol Chem. 283 (27), 18646-18654 (2008).
- Mandell, G. L. Interaction of intraleukocytic bacteria and antibiotics. J Clin Invest. 52 (7), 1673-1679 (1973).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymo. 236 (1979), 405-420 (1994).
- Elsinghorst, E. A., Weitz, J. A. Epithelial cell invasion and adherence directed by the enterotoxigenic Escherichia coli tib locus is associated with a 104-kilodalton outer membrane protein. Infect Immun. 62 (8), 3463-3471 (1994).
- Dorman, C. J., McKenna, S., Beloin, C. Regulation of virulence gene expression in Shigella flexneri, a facultative intracellular pathogen. Int J Med Microbiol. 291 (2), 89-96 (2001).
- Porter, M. E., Dorman, C. J. Positive regulation of Shigella flexneri virulence genes by integration host factor. J Bacteriol. 179 (21), 6537-6550 (1997).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Temperature-dependent expression of virulence genes in Shigella species. Infect Immun. 43 (1), 195-201 (1984).
- Schuch, R., Maurelli, A. T. Virulence plasmid instability in Shigella flexneri 2a is induced by virulence gene expression. Infect Immun. 65 (9), 3686-3692 (1997).
- Formal, S. B., Hale, T. L., Sansonetti, P. J. Invasive enteric pathogens. Rev Infect Dis. 5, S702-S707 (1983).
- Pál, T., Hale, T. L. Plasmid-associated adherence of Shigella flexneri in a HeLa cell model. Infect Immun. 57 (8), 2580-2582 (1989).
- Noben, M., et al. Human intestinal epithelium in a dish: Current models for research into gastrointestinal pathophysiology. United European Gastroenterol J. 5 (8), 1073-1081 (2017).
- Liévin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: lessons from cultured, fully differentiated human colon cancer cell lines. Microbiol Mol Biol Rev R. 77 (3), 380-439 (2013).
- Mitchell, D. M., Ball, J. M. Characterization of a spontaneously polarizing HT-29 cell line, HT-29/cl.f8. In Vitro Cell Dev Biol - Anim. 40 (10), 297-302 (2004).
- Gagnon, M., Zihler Berner, A., Chervet, N., Chassard, C., Lacroix, C. Comparison of the Caco-2, HT-29 and the mucus-secreting HT29-MTX intestinal cell models to investigate Salmonella adhesion and invasion. J Microbiol Methods. 94 (3), 274-279 (2013).
- Koestler, B. J., et al. Human intestinal enteroids as a model system of Shigella pathogenesis. Infect Immun. 87 (4), 00733 (2019).
- Ranganathan, S., et al. Evaluating Shigella flexneri pathogenesis in the human enteroid model. Infect Immun. 87 (4), (2019).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiol Spectr. 9 (1), 1-17 (2021).
- Perlman, M., Senger, S., Verma, S., Carey, J., Faherty, C. S. A foundational approach to culture and analyze malnourished organoids. Gut Microbes. 15 (2), 2248713 (2023).
- Pope, L. M., Reed, K. E., Payne, S. M. Increased protein secretion and adherence to HeLa cells by Shigella spp. following growth in the presence of bile salts. Infect Immun. 63 (9), 3642-3648 (1995).
- Faherty, C. S., et al. The synthesis of OspD3 (ShET2) in Shigella flexneri is independent of OspC1. Gut Microbes. 7 (6), 486-502 (2016).
- Ridlon, J. M., Kang, D. -. J., Hylemon, P. B. Bile salt biotransformations by human intestinal bacteria. J Lipid Res. 47 (2), 241-259 (2006).
- Köseoğlu, V. K., Hall, C. P., Rodríguez-López, E. M., Agaisse, H. The Autotransporter IcsA promotes Shigella flexneri biofilm formation in the presence of bile salts. Infect Immun. 87 (7), 1-14 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved