Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyses des infections des cellules épithéliales avec Shigella
Dans cet article
Résumé
Le présent protocole décrit des tests d’infection pour interroger l’adhérence, l’invasion et la réplication intracellulaire de Shigella à l’aide de lignées cellulaires épithéliales in vitro .
Résumé
L’agent pathogène bactérien entérique adapté à l’homme, Shigella , provoque des millions d’infections chaque année, crée des effets de croissance à long terme chez les patients pédiatriques et est l’une des principales causes de décès dus à la diarrhée dans le monde. L’infection induit une diarrhée aqueuse ou sanglante en raison du passage de l’agent pathogène dans le tractus gastro-intestinal et de l’infection des cellules épithéliales tapissant le côlon. Avec l’augmentation stupéfiante de la résistance aux antibiotiques et le manque actuel de vaccins approuvés, des protocoles de recherche standardisés sont essentiels pour étudier ce formidable agent pathogène. Ici, des méthodologies sont présentées pour examiner la pathogenèse moléculaire de Shigella à l’aide d’analyses in vitro de l’adhérence, de l’invasion et de la réplication intracellulaire bactériennes dans les cellules épithéliales du côlon. Avant les analyses d’infection, le phénotype de virulence des colonies de Shigella a été vérifié par l’absorption du colorant rouge du Congo sur des plaques de gélose. Des milieux de laboratoire supplémentés peuvent également être envisagés pendant la culture bactérienne pour imiter les conditions in vivo . Les cellules bactériennes sont ensuite utilisées dans un protocole standardisé pour infecter les cellules épithéliales du côlon dans des plaques de culture tissulaire à une multiplicité d’infection établie avec des adaptations pour analyser chaque stade de l’infection. Pour les tests d’observance, les cellules de Shigella sont incubées avec des niveaux de milieu réduits pour favoriser le contact bactérien avec les cellules épithéliales. Pour les essais d’invasion et de réplication intracellulaire, la gentamicine est appliquée à différents intervalles de temps pour éliminer les bactéries extracellulaires et permettre l’évaluation de l’invasion et/ou la quantification des taux de réplication intracellulaire. Tous les protocoles d’infection dénombrent les bactéries adhérentes, envahies et/ou intracellulaires en diluant en série les lysats de cellules épithéliales infectées et en plaquant les unités formant des colonies bactériennes par rapport aux titres infectieux sur des plaques de gélose rouge du Congo. Ensemble, ces protocoles permettent une caractérisation et des comparaisons indépendantes pour chaque stade de l’infection à Shigella des cellules épithéliales afin d’étudier avec succès cet agent pathogène.
Introduction
Les maladies diarrhéiques causées par des bactéries pathogènes entériques constituent un fardeau important pour la santé mondiale. En 2016, les maladies diarrhéiques étaient responsables de 1,3 million de décès dans le monde et étaient la quatrième cause de décès chez les enfants de moins de cinq ans 1,2. Shigella, un pathogène bactérien entérique à Gram négatif, est l’agent causal de la shigellose, une cause majeure de décès diarrhéiques dans le monde3. La shigellose provoque une morbidité et une mortalité importantes chaque année chez les enfants des pays à revenu faible et intermédiaire 4,5, tandis que les infections dans les pays à revenu élevé sont liées aux épidémies d’origine alimentaire et hydrique dans les garderies 6,7,8,9. L’inefficacité de la mise au pointde vaccins 10 et l’augmentation des taux de résistance aux antimicrobiens (RAM)11,12 ont compliqué la gestion des épidémies de Shigella à grande échelle. Des données récentes des Centers for Disease Control and Prevention montrent que près de 46 % des infections à Shigella aux États-Unis présentaient une résistance aux médicaments en 202013,14, tandis que l’Organisation mondiale de la santé a déclaré Shigella comme un agent pathogène prioritaire contre la résistance aux antimicrobiens pour lequel de nouvelles thérapies sont nécessaires de toute urgence15.
Les infections à Shigella se transmettent facilement par voie fécale-orale lors de l’ingestion d’aliments ou d’eau contaminés, ou par contact humain direct. Shigella a évolué pour devenir un agent pathogène efficace et adapté à l’homme, avec une dose infectieuse de 10 à 100 bactéries suffisante pour provoquer une maladie16. Pendant le transit de l’intestin grêle, Shigella est exposée à des signaux environnementaux, tels qu’une température élevée et la bile17. La détection de ces signaux induit des changements transcriptionnels pour exprimer des facteurs de virulence qui améliorent la capacité de la bactérie à infecter le côlon humain 17,18,19. Shigella n’envahit pas l’épithélium colique à partir de la surface apicale, mais transite plutôt à travers la couche épithéliale après absorption dans des cellules microfold (cellules M) spécialisées présentant des antigènes dans l’épithélium associé au follicule 20,21,22. Après la transcytose, les cellules de Shigella sont phagocytées par les macrophages résidents. Shigella s’échappe rapidement du phagosome et déclenche la mort des cellules macrophages, entraînant la libération de cytokines pro-inflammatoires 5,23,24. Shigella envahit ensuite les cellules épithéliales du côlon par le côté basolatéral, lyse la vacuole macropinocytaire et établit une niche réplicative dans le cytoplasme 5,25. Les cytokines pro-inflammatoires, en particulier l’interleukine-8 (IL-8), recrutent des leucocytes polynucléaires neutrophiles (PMN) sur le site de l’infection, ce qui affaiblit les jonctions épithéliales serrées et permet l’infiltration bactérienne de la muqueuse épithéliale pour exacerber l’infection basolatérale5. Les NMP détruisent la muqueuse épithéliale infectée pour contenir l’infection, ce qui entraîne les symptômes caractéristiques de la dysenterie bacillaire (sanglante)5. Bien que les mécanismes d’invasion et de réplication intracellulaire aient été caractérisés de manière approfondie, de nouvelles recherches démontrent de nouveaux concepts importants dans l’infection à Shigella, notamment la régulation de la virulence pendant le transit gastro-intestinal (GI)17, l’observance19, l’amélioration de l’accès basolatéral grâce à la perméabilité de la barrière26 et le portage asymptomatique chez les enfants malnutris27.
La capacité de Shigella spp. à provoquer des maladies diarrhéiques est limitée aux humains et aux primates non humains (PNH)28. Des modèles d’infection intestinale à Shigella ont été développés pour le poisson-zèbre29, les souris30, les cochons d’Inde31, les lapins 21,32,33 et les porcs 34,35. Cependant, aucun de ces systèmes modèles ne peut reproduire avec précision les caractéristiques de la maladie observées lors de l’infection humaine36. Bien que des modèles de shigellose aient été établis pour étudier la pathogenèse de Shigella, ces systèmes modèles sont coûteux à mettre en œuvre et nécessitent des doses infectieuses artificiellement élevées, jusqu’à neuf ordres de grandeur supérieures à la dose infectieuse des humains 37,38,39,40,41,42. Ainsi, l’adaptation remarquable de Shigella à l’infection d’hôtes humains nécessite l’utilisation de cultures cellulaires d’origine humaine pour recréer des modèles physiologiquement pertinents pour une interrogation précise de la pathogenèse de Shigella.
Ici, des procédures détaillées sont décrites pour mesurer les taux d’adhérence, d’invasion et de réplication de Shigella dans les cellules épithéliales du côlon HT-29. Grâce à ces protocoles standardisés, les mécanismes moléculaires par lesquels les gènes de virulence bactérienne et les signaux environnementaux ont un impact sur chaque étape de l’infection à Shigella peuvent être interrogés pour mieux comprendre la relation dynamique hôte-pathogène.
Protocole
1. Préparation des réactifs et des matériaux
REMARQUE : Tous les volumes sont cohérents avec un test utilisant deux plaques à 6 puits.
- Milieu TSB : Ajouter 0,5 L d’eau désionisée (DI) à 15 g de milieu de bouillon de soja tryptique (TSB, voir le tableau des matières) et autoclave. Conserver à température ambiante.
- Milieu de sels biliaires (BST + BS) : Pour préparer du BST contenant 0,4 % (p/v) de sels biliaires, remettre en suspension 0,06 g de sels biliaires (BS, voir le tableau des matières) dans 15 mL de BSB autoclave. Stériliser le filtre à l’aide d’un filtre PES de 0,22 μm.
REMARQUE : Les sels biliaires sont constitués d’un mélange 1:1 de cholate de sodium et de désoxycholate de sodium. Préparez des supports frais immédiatement avant utilisation. - DMEM + 10 % (v/v) FBS : Ajouter 5 mL de sérum fœtal bovin (FBS) à 45 mL de milieu modifié Eagle de Dulbecco (DMEM). Conserver à 4 °C.
- DMEM + gentamicine : Dans un tube de 50 mL, ajouter 50 mL de DMEM et 50 μL de gentamicine à 50 mg/mL (voir la table des matières).
REMARQUE : Faire de l’aliquote fraîche et réchauffer au bain-marie à 37 °C avant chaque expérience. - PBS + 1 % (v/v) Triton X-100 : Ajouter 150 μL de Triton X-100 à 15 mL de solution saline tamponnée au phosphate (PBS).
REMARQUE : Faire de l’aliquote fraîche et réchauffer au bain-marie à 37 °C avant chaque expérience. - TSB + plaques indicatrices rouge Congo : Ajouter 15 g de TSB, 7,5 g de gélose sélectionnée et 0,125 g de colorant rouge Congo (voir tableau des matériaux) dans une bouteille de 1 L. Ajouter 0,5 L d’eau DI et autoclave. Verser 10 à 20 ml de milieu dans des boîtes de Pétri stériles individuelles (100 mm x 15 mm) et laisser se solidifier.
ATTENTION : Le rouge Congo est cancérigène et une toxine pour la reproduction. S’assurer que la manipulation du Congo rouge est effectuée à l’aide de l’équipement de protection individuelle approprié. Consultez la fiche de données de sécurité du produit pour plus d’informations.
REMARQUE : Environ 20 plaques sont fabriquées à partir de 0,5 L de support rouge Congo. Les assiettes peuvent être préparées 2 à 3 jours à l’avance et laissées à température ambiante jusqu’à utilisation. Pour un stockage à long terme, placez les plaques inversées dans des manchons en plastique à 4 °C jusqu’à 3 mois. - DMEM + 10 % (v/v) de FBS et 5 % (v/v) de diméthylsulfoxyde (DMSO) : Ajouter 42,5 mL de DMEM, 5 mL de FBS et 2,5 mL de DMSO dans un tube de 50 mL. Conserver à 4 °C.
2. Préparation des bactéries
REMARQUE : Tous les protocoles de culture et de stockage en laboratoire de Shigella sont adaptés de Payne, S. M.43.
ATTENTION : Shigella spp. sont des agents pathogènes du groupe de risque 244. Effectuer tous les travaux de laboratoire dans un environnement BSL-2, avec des mesures de sécurité supplémentaires prises pour limiter les expositions accidentelles dues à la faible dose infectieuse de Shigella spp.
- Croissance de Shigella à partir d’actions congelées
- Transférez une petite quantité de culture congelée du flacon cryogénique dans une plaque de gélose rouge TSB + Congo à l’aide d’un applicateur stérile.
- Stérilisez à la flamme une boucle d’inoculation et laissez-la refroidir. Striez l’inoculum d’avant en arrière sur un quadrant de la plaque. Flammez la boucle, laissez-la refroidir, puis passez du premier quadrant au deuxième quadrant de la plaque. Répéter l’opération pour faire des stries d’inoculum dans les troisième et quatrième quadrants de la plaque.
REMARQUE : Vous pouvez également faire des stries sur l’inoculum à l’aide d’un applicateur stérile frais entre chaque quadrant. - Retournez la plaque et incubez à 37 °C pendant la nuit.
NOTE : L’incubation à des températures ≥37 °C est nécessaire pour l’expression des facteurs de virulence de Shigella nécessaires à l’observation du phénotype45 Congo rouge positif (CR+). Les colonies avirulentes auront un aspect blanc et ne seront pas envahissantes. - Fermez la plaque avec un film de paraffine et conservez-la au réfrigérateur à 4 °C.
REMARQUE : Les colonies bactériennes resteront viables sur des plaques de gélose pendant 1 à 2 semaines.
- Croissance nocturne de Shigella en culture liquide
- Aliquote 3 mL de milieu TSB dans des tubes de culture stériles de 14 mL.
- Prélever une seule colonie rouge (CR+) bien isolée à l’aide d’un applicateur stérile et la remettre en suspension dans un milieu liquide.
- Incuber les cultures pendant la nuit (16-18 h) à 37 °C en agitant à 250 rotations par minute (tr/min).
3. Préparation des cellules eucaryotes HT-29
REMARQUE : Tous les volumes sont cohérents avec un test utilisant deux plaques à 6 puits. Les lignées cellulaires HT-29 ont été acquises à partir de l’American Type Culture Collection (ATCC). Les protocoles d’entretien HT-29 sont adaptés des recommandations46 de l’ATCC. Tous les milieux doivent être préchauffés au bain-marie à 37 °C avant utilisation. Tous les protocoles d’entretien du HT-29 doivent être effectués dans une enceinte de biosécurité. Évitez de produire des bulles lorsque vous mélangez ou travaillez avec des cellules HT-29 dans un milieu pour éviter des changements spectaculaires du pH.
- Décongélation des cellules HT-29 du stock congelé
- Décongeler le flacon de cellules HT-29 dans un bain-marie à 37 °C.
REMARQUE : Assurez-vous que le capuchon reste complètement au-dessus de l’eau pour éviter toute contamination. La décongélation devrait prendre moins de 2 minutes. - Retirez le flacon de l’eau immédiatement après la décongélation complète de la culture et décontaminez-le avec de l’éthanol à 70 %. Assurez-vous que toutes les étapes à partir de ce point sont effectuées à l’aide de techniques aseptiques.
- Ajouter tout le contenu du flacon dans un tube à centrifuger de 15 mL contenant 9 mL de DMEM + 10 % de FBS. Centrifuger à 125 x g pendant 5 min à température ambiante.
- Décanter le surnageant dans un conteneur à déchets et remettre le granulé en suspension dans 10 mL de DMEM chaud + 10 % FBS. Transférer les cellules remises en suspension dans une fiole de culture tissulaire de75 cm 2 (T75) contenant 10 mL de DMEM chaud + 10 % de FBS (volume total de 20 mL).
- Incuber les cellules à 37 °C avec 5 % de CO2 jusqu’à ce que les cellules atteignent 90 % de confluence (environ 6 à 7 jours).
REMARQUE : La confluence est estimée par approximation visuelle.
- Décongeler le flacon de cellules HT-29 dans un bain-marie à 37 °C.
- Ensemencement de cellules HT-29
- Préchauffer 20 mL de PBS et 50 mL de DMEM + 10 % FBS dans un bain-marie à 37 °C et préchauffer 3 mL de trypsine-EDTA à 0,25 % (p/v) à température ambiante.
- Une fois que les cellules HT-29 (de l’étape 3.1) atteignent 90 % de confluence, décantez les milieux de culture cellulaire HT-29 du ballon T75 dans un conteneur à déchets. Verser ~10 ml de PBS chaud dans la fiole et agiter doucement pour laver. Décanter le PBS dans un conteneur à déchets. Laver à nouveau avec du PBS chaud et décanter.
- Ajouter 2 à 3 mL de trypsine-EDTA à 0,25 % (p/v) et agiter doucement sur toute la surface. Incuber à 37 °C avec 5% de CO2 pendant 4 min.
- Retirez le flacon de l’incubateur et faites tourner doucement la trypsine-EDTA, en vous assurant visuellement que toutes les cellules se détachent de la surface.
- Ajouter immédiatement 6 mL de DMEM chaud + 10 % FBS pour désactiver la trypsine. Pipeter de haut en bas pour bien mélanger.
- Transférez tout le contenu dans un tube à centrifuger de 15 mL et faites-le tourner à 500 x g pendant 5 min à température ambiante.
- Décanter délicatement le surnageant dans un conteneur à déchets et remettre le granulé en suspension dans 6 mL de DMEM chaud + 10 % FBS.
- Immédiatement après la remise en suspension, transférer 10 μL de cellules HT-29 en suspension du milieu de la culture dans un tube PCR de 0,2 mL. Ajouter 10 μL de colorant bleu trypan dans le tube PCR et mélanger.
- Ajouter 10 μL de mélange de cellules HT-29/bleu de trypan à une lame de chambre de comptoir de cellules Countess jetable (voir le tableau des matériaux). Énumérer le nombre de cellules vivantes et calculer la viabilité des cellules.
REMARQUE : Lorsque vous documentez le nombre de cellules dans l’échantillon, lisez le nombre sous le nombre de cellules « vivantes », et non le nombre total de cellules. Alternativement, le dénombrement cellulaire peut être effectué manuellement à l’aide d’un hémocytomètre. - Grainez les cellules HT-29 remises en suspension dans un nouveau ballon T75 ou une plaque à 6 puits.
- Pour la fiole T75 :
- Pipeter doucement pour mélanger, puis transférer 2,5 x 106 cellules dans une fiole T75 fraîche selon l’équation ci-dessous :
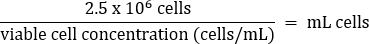
- Ajouter un milieu DMEM chaud + 10 % de FBS à un volume final de 20 mL (concentration finale de 1,25 x 105 cellules/mL).
- Répartissez les cellules uniformément dans le flacon en les balançant doucement d’avant en arrière.
- Incuber à 37 °C avec 5 % de CO2 jusqu’à ce que les cellules atteignent 80 % de confluence.
REMARQUE : Pour une croissance optimale, remplacez le milieu DMEM + 10 % FBS dans le flacon T75 tous les ~3 jours. Décanter le milieu dans un récipient à déchets et ajouter 10 ml de PBS chaud dans le flacon. Faites tourner doucement le PBS et décantez-le dans le conteneur à déchets. Ajouter ensuite 20 mL de DMEM frais et tiède + 10 % FBS dans le ballon et remettre dans l’incubateur à 37 °C, 5 % de CO2 .
- Pipeter doucement pour mélanger, puis transférer 2,5 x 106 cellules dans une fiole T75 fraîche selon l’équation ci-dessous :
- Pour une plaque à 6 puits :
- Pipeter doucement pour mélanger, puis transférer 5,85 x 106 cellules dans un tube conique frais de 50 mL selon l’équation ci-dessous :
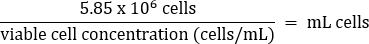
- Ajouter du DMEM chaud + 10 % de FBS dans un volume final de 26 mL (concentration finale de 2,25 x 105 cellules/mL).
- Pipeter doucement pour mélanger, puis distribuer 2 ml (4,5 x 105 cellules) dans des puits individuels de plaques à 6 puits.
- Dispersez les cellules uniformément dans le puits en les balançant doucement vers le haut/bas et vers la gauche/droite 2 à 3 fois.
- Incuber à 37 °C avec 5 % de CO2 jusqu’à ce que les cellules atteignent 80 % à 95 % de confluence (environ 3 à 4 jours).
REMARQUE : Une confluence de 85 % est recommandée pour les tests d’invasion et de réplication intracellulaire, tandis qu’une confluence de 90 % à 95 % est recommandée pour les tests d’observance. Les cellules devraient atteindre ~85 % de confluence après 48 h d’incubation avec une concentration finale d’environ 1 x 106 cellules/puits. Il peut être nécessaire d’ajuster le nombre de cellules ensemencées et la durée de l’incubation.
- Pipeter doucement pour mélanger, puis transférer 5,85 x 106 cellules dans un tube conique frais de 50 mL selon l’équation ci-dessous :
- Pour la fiole T75 :
- Fabrication de stocks HT-29 congelés
- Aliquote 1 mL de DMEM + 10 % FBS + 5 % de DMSO dans des flacons cryogéniques individuels.
- Ajouter 1 x 106 cellules HT-29 de l’étape 3.2.7 à chaque flacon. Calculez le volume des cellules selon la formule ci-dessous :
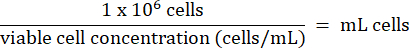
- Stockez les cellules HT-29 à long terme en dessous de -130 °C dans un congélateur à vapeur d’azote liquide.
4. Test d’observance
REMARQUE : Tous les volumes sont cohérents avec un test utilisant deux plaques à 6 puits.
- Sous-culture pendant la nuit Cultures de Shigella par dilution 1:50 dans un milieu frais.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
REMARQUE : Limitez le volume de culture à <20 % du volume du ballon ou du tube de culture pour assurer une bonne aération. - Incuber à 37 °C en agitant à 250 tr/min jusqu’à ce que les cellules atteignent une densité optique (OD600) de 0,7 (phase mi-log de la croissance de Shigella ) ; environ 2-2,5 h.
REMARQUE : Au cours de la sous-culture, aliquoter 50 mL de DMEM et un volume suffisant de PBS pour toutes les étapes de lavage et placer dans un bain-marie à 37 °C. Laissez le support atteindre 37 °C avant utilisation.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
- Transférer 2 x 108 unités formant colonies (UFC) Shigella sous-cultivées dans des tubes de microcentrifugation individuels de 2 mL.
REMARQUE : 2 x 108 UFC correspondent à environ 1 mL de cellules bactériennes à une DO600 de 0,7. Utilisez des lectures OD600 pour approximer la CFU/mL en fonction de l’étalonnage de chaque spectrophotomètre individuel. - Lavez chaque échantillon de Shigella 2x avec du PBS.
- Cellules à granulés par centrifugation à 17 000 x g pendant 2 min à température ambiante. Aspirer le surnageant, puis ajouter 1 mL de PBS chaud et bien remettre la pastille en suspension, en pipetant doucement l’échantillon de haut en bas jusqu’à ce que le mélange soit complètement homogène (8-10x).
- Répétez l’étape 4.3.1 1x fois plus longtemps.
- Centrifuger les granulés à 17 000 x g pendant 2 min à température ambiante, aspirer le surnageant et remettre les granulés en suspension dans 2 mL de DMEM chaud.
REMARQUE : La concentration finale de bactéries remises en suspension sera de 1 x 108 UFC/mL.
- Vortex, puis ajouter 1 mL (1 x 108 UFC) de Shigella remise en suspension dans chaque puits des monocouches épithéliales du côlon HT-29 préparées en plaques à 6 puits (à partir de l’étape 3.2.10.2).
REMARQUE : Les infections sont normalement réalisées à une multiplicité d’infection (MOI ; rapport entre les cellules bactériennes et épithéliales) de 100. Pour tester différents MOI, diluez Shigella en suspension dans du DMEM chaud à la concentration désirée, puis ajoutez 1 mL de bactéries diluées aux monocouches HT-29. Par exemple, pour tester un MOI de 10, diluez les bactéries 1:10 en ajoutant 150 μL de 1 x 108 UFC/mL de bactéries à 1,35 mL de DMEM chaud, puis appliquez 1 mL (1 x 107 UFC) sur les cellules HT-29. - Incuber les plaques à 6 puits à 37 °C avec 5% de CO2 pendant 3 h.
- Pendant l’incubation, déterminer le titre d’infection bactérienne.
- Préparer des dilutions en série de 10 fois de cellules Shigella remises en suspension (à partir de l’étape 4.3.3) dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-5 et 1 x 10-6 sur des plaques TSB + rouge Congo et incuber toute la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-5 et 1 x 10-6 correspond à un facteur de dilution final de 1 x 10-6 et 1 x 10-7, respectivement.
- Après l’incubation, lavez les monocouches 4 à 5 fois avec du PBS.
- Aspirer le milieu de chaque puits.
REMARQUE : Lors de l’aspiration de fluides à partir de plaques à 6 puits, guidez la pointe de l’aspirateur le long de la face inférieure des puits, en essayant d’éviter tout contact avec les cellules HT-29. - Ajouter 1 ml de PBS chaud dans chaque puits et laver délicatement.
REMARQUE : Pour laver délicatement les monocouches à 6 puits avec du PBS, déplacez la plaque de haut en bas et d’un côté à l’autre sur la paillasse. Le lavage des plaques dans un mouvement circulaire et/ou le retrait de la plaque de la surface de la paillasse peuvent entraîner l’élimination mécanique des cellules du plastique. - Répétez les étapes 4.7.1 et 4.7.2 4 fois de plus.
- Aspirer le milieu de chaque puits.
- Éliminer le PBS par aspiration et lyser les cellules HT-29 en ajoutant 1 mL de PBS + 1 % de Triton X-100 dans chaque puits.
- Incuber des plaques de 6 puits à 37 °C pendant 5 min.
- À l’aide d’un grattoir cellulaire ou d’une pointe de pipette courbée, grattez les cellules lysées du fond du puits et transférez la totalité de 1 ml dans un tube de microcentrifugation frais de 1,7 ml.
- Déterminez le nombre de bactéries associées aux cellules.
- Agiter chaque tube (à partir de l’étape 4.10) pendant au moins 30 s pour déplacer davantage Shigella des cellules eucaryotes lysées.
- Préparez des dilutions en série de 10 fois de lysats dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-2, 1 x 10-3 et 1 x 10-4 sur des plaques TSB + rouge Congo et incuber toute la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-2, 1 x 10-3 et 1 x 10-4 correspond à un facteur de dilution final de 1 x 10-3, 1 x 10-4 et 1 x 10-5, respectivement.
5. Essai d’invasion
REMARQUE : Tous les volumes sont cohérents avec un test utilisant deux plaques à 6 puits.
- Sous-culture pendant la nuit Cultures de Shigella par dilution 1:50 dans un milieu frais.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
REMARQUE : Limitez le volume de culture à <20 % du volume du ballon ou du tube de culture pour assurer une bonne aération. - Incuber à 37 °C en agitant à 250 tr/min jusqu’à ce que les cellules atteignent une OD600 de 0,7 (phase mi-log de croissance de Shigella ) ; environ 2-2,5 h.
REMARQUE : Au cours de la sous-culture, aliquoter 50 mL de DMEM + 50 mg/mL de gentamicine et un volume suffisant de PBS pour toutes les étapes de lavage et placer dans un bain-marie à 37 °C. Laissez le support atteindre 37 °C avant utilisation.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
- Transférer 2 x 108 UFC de Shigella sous-cultivée dans des tubes à microcentrifuger individuels de 2 ml.
REMARQUE : 2 x 108 UFC correspondent à environ 1 mL de cellules bactériennes à une DO600 de 0,7. Utilisez des lectures OD600 pour approximer la CFU/mL en fonction de l’étalonnage de chaque spectrophotomètre individuel. - Lavez les échantillons de Shigella 1x avec PBS.
- Cellules à granulés par centrifugation à 17 000 x g pendant 2 min à température ambiante. Aspirer le surnageant, puis ajouter 1 mL de PBS chaud et bien remettre la pastille en suspension, en pipetant doucement l’échantillon de haut en bas jusqu’à ce que le mélange soit complètement homogène (8-10x).
- Répétez l’étape 5.3.1. 1x temps supplémentaire.
- Centrifuger les granulés à 17 000 x g pendant 2 min à température ambiante, aspirer le surnageant et remettre les granulés en suspension dans 2 mL de DMEM chaud.
REMARQUE : La concentration finale de bactéries remises en suspension sera de 1 x 108 UFC/mL.
- Vortex, puis ajouter 1 mL (1 x 108 UFC) de Shigella remis en suspension plus 1 mL de DMEM dans chaque puits des monocouches épithéliales du côlon HT-29 préparées dans des plaques à 6 puits (à partir de l’étape 3.2.10.2).
REMARQUE : Les infections sont normalement réalisées à une multiplicité d’infection (MOI ; rapport entre les cellules bactériennes et épithéliales) de 100. Pour tester différentes MOI, diluez Shigella en suspension dans du DMEM à la concentration désirée, puis ajoutez 1 mL de bactéries diluées aux monocouches HT-29. Par exemple, pour tester un moment d’inertie de 10, diluez les bactéries 1:10 en ajoutant 150 μL de 1 x 108 UFC/mL de bactéries à 1,35 mL de DMEM, puis ajoutez 1 mL (1 x 107 UFC) aux cellules HT-29. - Pour favoriser le contact bactérien avec les cellules HT-29, centrifuger les plaques à 6 puits à 2 000 x g pendant 10 min à température ambiante ou 37 °C si le réglage de la température peut être ajusté.
REMARQUE : La centrifugation favorise le contact bactérien avec les cellules HT-29, ce qui contourne le besoin de facteurs d’adhérence et permet aux bactéries d’envahir rapidement les cellules. - Incuber des plaques de 6 puits à 37 °C avec 5 % de CO2 pendant 45 min.
- Pendant l’incubation, déterminer le titre d’infection bactérienne.
- Préparer des dilutions en série de 10 fois de cellules Shigella remises en suspension (à partir de l’étape 5.3.3) dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-5 et 1 x 10-6 sur des plaques TSB + rouge Congo et incuber toute la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-5 et 1 x 10-6 correspond à un facteur de dilution final de 1 x 10-6 et 1 x 10-7, respectivement.
- Laver soigneusement les cellules HT-29 infectées 3 fois avec 1 mL de PBS.
- Aspirer le milieu de chaque puits.
REMARQUE : Lors de l’aspiration de fluides à partir de plaques à 6 puits, guidez la pointe de l’aspirateur le long de la face inférieure des puits, en essayant d’éviter tout contact avec les cellules HT-29. - Ajouter 1 ml de PBS chaud dans chaque puits et laver délicatement.
REMARQUE : Pour laver délicatement les monocouches à 6 puits avec du PBS, déplacez la plaque de haut en bas et d’un côté à l’autre sur la paillasse. Le lavage des plaques dans un mouvement circulaire et/ou le retrait de la plaque de la surface de la paillasse peuvent entraîner l’élimination mécanique des cellules du plastique. - Répétez les étapes 5.8.1 et 5.8.2 2 fois de plus.
- Aspirer le milieu de chaque puits.
- Éliminer le PBS par aspiration, puis ajouter 2 mL de DMEM chaud complété par 50 μg/mL de gentamicine dans chaque puits et incuber pendant 30 min à 37 °C avec 5 % de CO2.
- Laver soigneusement les cellules HT-29 infectées 3 fois avec 1 mL de PBS.
- Répétez l’étape de lavage 5.8.
- Éliminer le PBS par aspiration, puis ajouter 2 mL de DMEM chaud complété par 50 μg/mL de gentamicine dans chaque puits et incuber pendant 60 min à 37 °C avec 5 % de CO2.
- Laver soigneusement les cellules HT-29 infectées 3 fois avec 1 mL de PBS.
- Répétez l’étape de lavage 5.8.
- Éliminer le PBS par aspiration et lyser les cellules HT-29 en ajoutant 1 mL de PBS + 1 % de Triton X-100 dans chaque puits.
- Incuber des plaques de 6 puits à 37 °C pendant 5 min.
- À l’aide d’un grattoir cellulaire ou d’une pointe de pipette courbée, grattez les cellules lysées du fond du puits et transférez la totalité de 1 ml dans un tube de microcentrifugation frais de 1,7 ml.
- Déterminez le nombre de bactéries intracellulaires.
- Vortex dans chaque tube (à partir de l’étape 5.15) pendant au moins 30 s pour déplacer davantage Shigella des cellules eucaryotes lysées.
- Préparez des dilutions en série de 10 fois de lysats dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-2 et 1 x 10-3 sur des plaques TSB + rouge Congo et incuber toute la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-2 et 1 x 10-3 correspond à un facteur de dilution final de 1 x 10-3 et 1 x 10-4, respectivement.
6. Test de réplication intracellulaire
REMARQUE : Tous les volumes sont cohérents avec un test utilisant deux plaques à 6 puits.
- Sous-culture pendant la nuit Cultures de Shigella par dilution 1:50 dans un milieu frais.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
REMARQUE : Limitez le volume de culture à <20 % du volume du ballon ou du tube de culture pour assurer une bonne aération. - Incuber à 37 °C en agitant à 250 tr/min jusqu’à ce que les cellules atteignent une OD600 de 0,7 (phase mi-log de croissance de Shigella ) ; environ 2-2,5 h.
REMARQUE : Au cours de la sous-culture, aliquoter 50 mL de DMEM + 50 mg/mL de gentamicine et un volume suffisant de PBS pour toutes les étapes de lavage et placer dans un bain-marie à 37 °C. Laissez le support atteindre 37 °C avant utilisation.
- Vortex, puis ajouter 100 μL de chaque culture de nuit à 5 mL de TSB ou TSB + BS frais dans un tube de culture de taille appropriée.
- Transférer 2 x 108 UFC de Shigella sous-cultivée dans des tubes à microcentrifuger individuels de 2 ml.
REMARQUE : 2 x 108 UFC correspondent à environ 1 mL de cellules bactériennes à une DO600 de 0,7. Utilisez des lectures OD600 pour approximer la CFU/mL en fonction de l’étalonnage de chaque spectrophotomètre individuel. - Lavez les échantillons de Shigella 1x avec PBS.
- Cellules à granulés par centrifugation à 17 000 x g pendant 2 min à température ambiante. Aspirer le surnageant, puis ajouter 1 mL de PBS chaud et bien remettre la pastille en suspension, en pipetant doucement l’échantillon de haut en bas jusqu’à ce que le mélange soit complètement homogène (8-10x).
- Répétez l’étape 6.3.1. 1x temps supplémentaire.
- Centrifuger les granulés à 17 000 x g pendant 2 min à température ambiante, aspirer le surnageant et remettre les granulés en suspension dans 2 mL de DMEM chaud.
REMARQUE : La concentration finale de bactéries remises en suspension sera de 1 x 108 UFC/mL.
- Vortex, puis ajouter 1 mL (1 x 108 UFC) de Shigella remise en suspension plus 1 mL de DMEM dans chaque puits de monocouches épithéliales du côlon HT-29 préparées dans des plaques à 6 puits (à partir de l’étape 3.2.10.2).
REMARQUE : Les infections sont normalement réalisées à une multiplicité d’infection (MOI ; rapport entre les cellules bactériennes et épithéliales) de 100. Pour tester différentes MOI, diluez Shigella en suspension dans du DMEM à la concentration désirée, puis ajoutez 1 mL de bactéries diluées aux monocouches HT-29. Par exemple, pour tester un MOI de 10, diluez les bactéries 1:10 en ajoutant 150 μL de 1 x 108 UFC/mL de bactéries à 1,35 mL de DMEM, puis appliquez 1 mL (1 x 107 UFC) sur les cellules HT-29. - Pour favoriser le contact bactérien avec les cellules HT-29, centrifuger les plaques à 6 puits à 2 000 x g pendant 10 min à température ambiante ou 37 °C si le réglage de la température peut être ajusté.
REMARQUE : La centrifugation favorise le contact bactérien avec les cellules HT-29, ce qui contourne le besoin de facteurs d’adhérence et permet aux bactéries d’envahir rapidement les cellules. - Incuber des plaques de 6 puits à 37 °C avec 5 % de CO2 pendant 45 min.
- Pendant l’incubation, déterminer le titre d’infection bactérienne.
- Préparer des dilutions en série de 10 fois de cellules Shigella remises en suspension (à partir de l’étape 6.3.3) dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-5 et 1 x 10-6 sur des plaques TSB + rouge Congo et incuber toute la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-5 et 1 x 10-6 correspond à un facteur de dilution final de 1 x 10-6 et 1 x 10-7, respectivement.
- Laver soigneusement les cellules HT-29 infectées 3 fois avec 1 mL de PBS.
- Aspirer le milieu de chaque puits.
REMARQUE : Lors de l’aspiration de fluides à partir de plaques à 6 puits, guidez la pointe de l’aspirateur le long de la face inférieure des puits, en essayant d’éviter tout contact avec les cellules HT-29. - Ajouter 1 ml de PBS chaud dans chaque puits et laver délicatement.
REMARQUE : Pour laver délicatement les monocouches à 6 puits avec du PBS, déplacez la plaque de haut en bas et d’un côté à l’autre sur la paillasse. Le lavage des plaques dans un mouvement circulaire et/ou le retrait de la plaque de la surface de la paillasse peuvent entraîner l’élimination mécanique des cellules du plastique. - Répétez les étapes 6.8.1 et 6.8.2 2 fois de plus.
- Aspirer le milieu de chaque puits.
- Éliminer le PBS par aspiration, puis ajouter 2 mL de DMEM chaud complété par 50 μg/mL de gentamicine dans chaque puits et incuber pendant 30 min à 37 °C avec 5 % de CO2.
- Laver soigneusement les cellules HT-29 infectées 3 fois avec 1 mL de PBS.
- Répétez l’étape de lavage 6.8.
- Éliminer le PBS par aspiration, puis ajouter 2 mL de DMEM chaud avec 50 μg/mL de gentamicine dans chaque puits des plaques à 6 puits et incuber à 37 °C avec 5 % de CO2 pendant la durée souhaitée pour permettre la réplication intracellulaire (jusqu’à 24 h).
- Laver soigneusement les cellules 2x avec 1 mL de PBS.
- Répétez l’étape de lavage 6.8.
- Éliminer le PBS par aspiration et lyser les cellules HT-29 en ajoutant 1 mL de PBS + 1 % de Triton X-100 dans chaque puits.
- Incuber des plaques de 6 puits à 37 °C pendant 5 min.
- Utilisez un grattoir cellulaire ou une pointe de pipette courbée pour gratter les cellules lysées du fond du puits et transférer la totalité de 1 mL dans un nouveau tube de microcentrifugation de 1,7 mL.
- Déterminez le nombre de bactéries intracellulaires.
- Agiter chaque tube (à partir de l’étape 6.15) pendant au moins 30 s pour déplacer davantage Shigella des cellules eucaryotes lysées.
- Préparez des dilutions en série de 10 fois de lysats dans du PBS.
- Déposer 100 μL des dilutions 1 x 10-2, 1 x 10-3 et 1 x 10-4 sur des plaques TSB + Congo Red et incuber pendant la nuit à 37 °C.
REMARQUE : Le placage de 100 μL à partir des dilutions 1 x 10-2, 1 x 10-3 et 1 x 10-4 correspond à un facteur de dilution final de 1 x 10-3, 1 x 10-4 et 1 x 10-5, respectivement.
Résultats
Des tests d’adhérence, d’invasion et de réplication intracellulaire ont été effectués en comparant S. flexneri 2457T de type sauvage (WT) à S. flexneri ΔVF (ΔVF), un mutant supposé réguler négativement la virulence de Shigella. Étant donné que Shigella utilise les sels biliaires comme signal pour réguler la virulence 17,18,47, des expériences ont été réalisées après une sous-culture bactérienne dans un milieu TSB ainsi qu’une supplémentation en sels...
Discussion
Ce protocole décrit un ensemble de trois tests standardisés pour étudier l’adhérence, l’invasion et la réplication intracellulaire de Shigella des cellules épithéliales intestinales. Bien que ces méthodes ne soient que des versions modifiées des tests classiques de gentamicine utilisés pour étudier l’invasion et la réplication intracellulaire de divers agents pathogènes bactériens dans les cellules hôtes 49,50,51, des considérations particulières doivent être appliquées lors de l’étud...
Déclarations de divulgation
Les auteurs ne déclarent aucun conflit d’intérêts.
Remerciements
Le soutien aux auteurs comprend le département de pédiatrie du Massachusetts General Hospital, la subvention 2022A009041 du Comité exécutif sur le financement provisoire du soutien à la recherche, la subvention R21AI146405 de l’Institut national des allergies et des maladies infectieuses et la subvention de l’Institut national du diabète et des maladies digestives et rénales Centre de recherche sur la nutrition et l’obésité à Harvard (NORCH) 2P30DK040561-26. Les bailleurs de fonds n’ont joué aucun rôle dans la conception de l’étude, la collecte et l’analyse des données, la décision de publier ou la préparation du manuscrit.
matériels
| Name | Company | Catalog Number | Comments |
| 0.22 μm PES filter | Millipore-Sigma | SCGP00525 | Sterile, polyethersulfone filter for sterilizing up to 50 mL media |
| 14 mL culture tubes | Corning | 352059 | 17 mm x 100 mm polypropylene test tubes with cap |
| 50 mL conical tubes | Corning | 430829 | 50 mL clear polypropylene conical bottom centrifuge tubes with leak-proof cap |
| 6-well tissue culture plates | Corning | 3516 | Plates are treated for optimal cell attachment |
| Bile salts | Sigma-Aldrich | B8756 | 1:1 ratio of cholate to deoxycholate |
| Congo red dye | Sigma-Aldrich | C6277 | A benzidine-based anionic diazo dye, >85% purity |
| Countess cell counting chamber slide | Invitrogen | C10283 | To be used with the Countess Automated Cell Counter |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D8418 | A a highly polar organic reagent |
| Dulbecco’s Modified Eagle Medium (DMEM) | Gibco | 10569-010 | DMEM is supplemented with high glucose, sodium pyruvate, GlutaMAX, and Phenol Red |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F4135 | Heat-inactivated, sterile |
| Gentamicin | Sigma-Aldrich | G3632 | Stock concentration is 50 mg/mL |
| HT-29 cell line | ATCC | HTB-38 | Adenocarcinoma cell line; colorectal in origin |
| Paraffin film | Bemis | PM999 | Laboratory sealing film |
| Petri dishes | Thermo Fisher Scientific | FB0875713 | 100 mm x 15 mm Petri dishes for solid media |
| Phosphate-buffered saline (PBS) | Thermo Fisher Scientific | 10010049 | 1x concentration; pH 7.4 |
| Select agar | Invitrogen | 30391023 | A mixture of polysaccharides extracted from red seaweed cell walls to make bacterial plating media |
| T75 flasks | Corning | 430641U | Tissue culture flasks |
| Triton X-100 | Sigma-Aldrich | T8787 | A common non-ionic surfactant and emulsifier |
| Trypan blue stain | Invitrogen | T10282 | A dye to detect dead tissue culture cells; only live cells can exclude the dye |
| Trypsin-EDTA | Gibco | 25200-056 | Reagent for cell dissociation for cell line maintenance and passaging |
| Tryptic Soy Broth (TSB) | Sigma-Aldrich | T8907 | Bacterial growth media |
Références
- Karambizi, N. U., McMahan, C. S., Blue, C. N., Temesvari, L. A. Global estimated Disability-Adjusted Life-Years (DALYs) of diarrheal diseases: A systematic analysis of data from 28 years of the global burden of disease study. PloS one. 16 (10), e0259077 (2021).
- WHO. WHO methods and data sources for country-level causes of death 2000-2016. World Health Organization. , (2018).
- Kotloff, K. L. Shigella infection in children and adults: a formidable foe. Lancet Glob Health. 5 (12), e1166-e1167 (2017).
- Kotloff, K. L., et al. Burden and aetiology of diarrhoeal disease in infants and young children in developing countries (the Global Enteric Multicenter Study, GEMS): A prospective, case-control study. Lancet. 382 (9888), 209-222 (2013).
- Schroeder, G. N., Hilbi, H. Molecular pathogenesis of Shigella spp.: Controlling host cell signaling, invasion, and death by type III secretion. Clin Microbiol Rev. 21 (1), 134-156 (2008).
- Arvelo, W., et al. Transmission risk factors and treatment of pediatric shigellosis during a large daycare center-associated outbreak of multidrug resistant shigella sonnei: Implications for the management of shigellosis outbreaks among children. Pediatr Infect Dis J. 28 (11), 976-980 (2009).
- Kozyreva, V. K., et al. Recent outbreaks of Shigellosis in California caused by two distinct populations of Shigella sonnei with either increased virulence or fluoroquinolone resistance. mSphere. 1 (6), 1-18 (2016).
- Bowen, A., et al. Importation and domestic transmission of Shigella sonnei resistant to ciprofloxacin - United States, May 2014-February 2015. MMWR Morb Mortal Wkly Rep. 64 (12), 318-320 (2015).
- Tansarli, G. S., et al. Genomic reconstruction and directed interventions in a multidrug-resistant Shigellosis outbreak in Seattle, WA, USA: a genomic surveillance study. Lancet. 3099 (22), 1-11 (2023).
- Barry, E. M., et al. Progress and pitfalls in Shigella vaccine research. Nat Rev Gastroenterol Hepatol. 10 (4), 245-255 (2013).
- Increase in Extensively Drug-Resistant Shigellosis in the United States. CDC Health Alert Network. Centers for Disease Control and Prevention Available from: https://emergency.cdc.gov/han/2023/han00486.asp?ACSTrackingID=USCDC_511-DM100260&ACSTrackingLabel=HAN%20486%20-%20General%20Public&deliveryName=USCDC_511-DM100260 (2023)
- Shiferaw, B., et al. Antimicrobial susceptibility patterns of Shigella isolates in Foodborne Diseases Active Surveillance Network (FoodNet) sites, 2000-2010. Clin Infect Dis. 54, S458-S463 (2012).
- Centers for Disease Control and Prevention. COVID-19: U.S. Impact on Antimicrobial Resistance, Special Report 2022. Atlanta, GA: U.S. Department of Health and Human Services. CDC. , (2022).
- Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019. CDC. 10 (1), (2019).
- WHO. Prioritization of pathogens to guide discovery, research and development of new antibiotics for drug-resistant bacterial infections, including tuberculosis. WHO. , (2017).
- DuPont, H. L., Levine, M. M., Hornick, R. B., Formal, S. B. Inoculum size in shigellosis and implications for expected mode of transmission. J Infect Dis. 159 (6), 1126-1128 (1989).
- Nickerson, K. P., et al. Analysis of Shigella flexneri resistance, biofilm formation, and transcriptional profile in response to bile salts. Infect Immun. 85 (6), 1-18 (2017).
- Faherty, C. S., Redman, J. C., Rasko, D. A. Shigella flexneri effectors OspE1 and OspE2 mediate induced adherence to the colonic epithelium following bile salts exposure. Mol Microbiol. 85 (1), 107-121 (2012).
- Chanin, R. B., et al. Shigella flexneri adherence factor expression in in vivo-like conditions. mSphere. 4 (6), e00751 (2019).
- Baranov, V., Hammarström, S. Carcinoembryonic antigen (CEA) and CEA-related cell adhesion molecule 1 (CEACAM1), apically expressed on human colonic M cells, are potential receptors for microbial adhesion. Histochem Cell Biol. 121 (2), 83-89 (2004).
- Wassef, J. S., Keren, D. F., Mailloux, J. L. Role of M cells in initial antigen uptake and in ulcer formation in the rabbit intestinal loop model of shigellosis. Infect Immun. 57 (3), 858-863 (1989).
- Sansonetti, P. J., Arondel, J., Cantey, J. R., Prévost, M. C., Huerre, M. Infection of rabbit Peyer's patches by Shigella flexneri: Effect of adhesive or invasive bacterial phenotypes on follicle-associated epithelium. Infect Immun. 64 (7), 2752-2764 (1996).
- Sansonetti, P. J., et al. Caspase-1 activation of IL-1beta and IL-18 are essential for Shigella flexneri-induced inflammation. Immunity. 12 (5), 581-590 (2000).
- Zychlinsky, A., Fitting, C., Cavaillon, J. M., Sansonetti, P. J. Interleukin 1 is released by murine macrophages during apoptosis induced by Shigella flexneri. J Clin Invest. 94 (3), 1328-1332 (1994).
- Sansonetti, P. J., Ryter, A., Clerc, P., Maurelli, A. T., Mounier, J. Multiplication of Shigella flexneri within HeLa cells: lysis of the phagocytic vacuole and plasmid-mediated contact hemolysis. Infect Immun. 51 (2), 461-469 (1986).
- Maldonado-Contreras, A., et al. Shigella depends on SepA to destabilize the intestinal epithelial integrity via cofilin activation. Gut Microbes. 8 (6), 544-560 (2017).
- Collard, J. -. M., et al. High prevalence of small intestine bacteria overgrowth and asymptomatic carriage of enteric pathogens in stunted children in Antananarivo, Madagascar. PLoS Negl Trop Dis. 16 (5), e0009849 (2022).
- Mattock, E., Blocker, A. J. How do the virulence factors of shigella work together to cause disease. Front Cell Infect Microbiol. 7, 1-24 (2017).
- Mostowy, S., et al. The zebrafish as a new model for the in vivo study of Shigella flexneri interaction with phagocytes and bacterial autophagy. PLoS Pathog. 9 (9), e1003588 (2013).
- Martinez-Becerra, F. J., et al. Parenteral immunization with IpaB/IpaD protects mice against lethal pulmonary infection by Shigella. Vaccine. 31 (24), 2667-2672 (2013).
- Shim, D. -. H., et al. New animal model of shigellosis in the Guinea pig: its usefulness for protective efficacy studies. J Immunol. 178 (4), 2476-2482 (2007).
- Marteyn, B., et al. Modulation of Shigella virulence in response to available oxygen in vivo. Nature. 465 (7296), 355-358 (2010).
- West, N. P., et al. Optimization of virulence functions through glucosylation of Shigella LPS. Science. 307 (5713), 1313-1317 (2005).
- Maurelli, A. T., et al. Shigella infection as observed in the experimentally inoculated domestic pig, Sus scrofa domestica. Microbial Pathog. 25 (4), 189-196 (1998).
- Jeong, K. -. I., Zhang, Q., Nunnari, J., Tzipori, S. A piglet model of acute gastroenteritis induced by Shigella dysenteriae Type 1. J Infect Dis. 201 (6), 903-911 (2010).
- Kim, Y. -. J., Yeo, S. -. G., Park, J. -. H., Ko, H. -. J. Shigella vaccine development: prospective animal models and current status. Curr Pharm Biotechnol. 14 (10), 903-912 (2013).
- Kent, T. H., Formal, S. B., LaBrec, E. H., Sprinz, H., Maenza, R. M. Gastric shigellosis in rhesus monkeys. Am J Pathol. 51 (2), 259-267 (1967).
- Shipley, S. T., et al. A challenge model for Shigella dysenteriae 1 in cynomolgus monkeys (Macaca fascicularis). Comp Med. 60 (1), 54-61 (2010).
- Higgins, R., Sauvageau, R., Bonin, P. Shigella flexneri Type 2 Infection in captive nonhuman primates. Can Vet J. 26 (12), 402-403 (1985).
- Oaks, E. V., Hale, T. L., Formal, S. B. Serum immune response to Shigella protein antigens in rhesus monkeys and humans infected with Shigella spp. Infect Immun. 53 (1), 57-63 (1986).
- Formal, S. B., et al. Protection of monkeys against experimental shigellosis with a living attenuated oral polyvalent dysentery vaccine. J Bacteriol. 92 (1), 17-22 (1966).
- Levine, M. M., Kotloff, K. L., Barry, E. M., Pasetti, M. F., Sztein, M. B. Clinical trials of Shigella vaccines: two steps forward and one step back on a long, hard road. Nat Rev Microbiol. 5 (7), 540-553 (2007).
- Payne, S. M. Laboratory cultivation and storage of Shigella. Curr Protoc Microbiol. 55 (1), 93 (2019).
- NIH Guidelines. NIH guidelines for research involving recombinant or synthetic nucleic acid molecules. NIH Guidelines. 2, 142 (2019).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Loss of pigmentation in Shigella flexneri 2a is correlated with loss of virulence and virulence-associated plasmid. Infect Immun. 43 (1), 397-401 (1984).
- HT-29 cell line product sheet. ATCC Available from: https://www.atcc.org/products/htb-38 (2023)
- Sistrunk, J. R., Nickerson, K. P., Chanin, R. B., Rasko, D. A., Faherty, C. S. Survival of the fittest: How bacterial pathogens utilize bile to enhance infection. Clin Microbiol Rev. 29 (4), 819-836 (2016).
- Stensrud, K. F., et al. Deoxycholate interacts with IpaD of Shigella flexneri in inducing the recruitment of IpaB to the type III secretion apparatus needle tip. J Biol Chem. 283 (27), 18646-18654 (2008).
- Mandell, G. L. Interaction of intraleukocytic bacteria and antibiotics. J Clin Invest. 52 (7), 1673-1679 (1973).
- Elsinghorst, E. A. Measurement of invasion by gentamicin resistance. Methods Enzymo. 236 (1979), 405-420 (1994).
- Elsinghorst, E. A., Weitz, J. A. Epithelial cell invasion and adherence directed by the enterotoxigenic Escherichia coli tib locus is associated with a 104-kilodalton outer membrane protein. Infect Immun. 62 (8), 3463-3471 (1994).
- Dorman, C. J., McKenna, S., Beloin, C. Regulation of virulence gene expression in Shigella flexneri, a facultative intracellular pathogen. Int J Med Microbiol. 291 (2), 89-96 (2001).
- Porter, M. E., Dorman, C. J. Positive regulation of Shigella flexneri virulence genes by integration host factor. J Bacteriol. 179 (21), 6537-6550 (1997).
- Maurelli, A. T., Blackmon, B., Curtiss, R. Temperature-dependent expression of virulence genes in Shigella species. Infect Immun. 43 (1), 195-201 (1984).
- Schuch, R., Maurelli, A. T. Virulence plasmid instability in Shigella flexneri 2a is induced by virulence gene expression. Infect Immun. 65 (9), 3686-3692 (1997).
- Formal, S. B., Hale, T. L., Sansonetti, P. J. Invasive enteric pathogens. Rev Infect Dis. 5, S702-S707 (1983).
- Pál, T., Hale, T. L. Plasmid-associated adherence of Shigella flexneri in a HeLa cell model. Infect Immun. 57 (8), 2580-2582 (1989).
- Noben, M., et al. Human intestinal epithelium in a dish: Current models for research into gastrointestinal pathophysiology. United European Gastroenterol J. 5 (8), 1073-1081 (2017).
- Liévin-Le Moal, V., Servin, A. L. Pathogenesis of human enterovirulent bacteria: lessons from cultured, fully differentiated human colon cancer cell lines. Microbiol Mol Biol Rev R. 77 (3), 380-439 (2013).
- Mitchell, D. M., Ball, J. M. Characterization of a spontaneously polarizing HT-29 cell line, HT-29/cl.f8. In Vitro Cell Dev Biol - Anim. 40 (10), 297-302 (2004).
- Gagnon, M., Zihler Berner, A., Chervet, N., Chassard, C., Lacroix, C. Comparison of the Caco-2, HT-29 and the mucus-secreting HT29-MTX intestinal cell models to investigate Salmonella adhesion and invasion. J Microbiol Methods. 94 (3), 274-279 (2013).
- Koestler, B. J., et al. Human intestinal enteroids as a model system of Shigella pathogenesis. Infect Immun. 87 (4), 00733 (2019).
- Ranganathan, S., et al. Evaluating Shigella flexneri pathogenesis in the human enteroid model. Infect Immun. 87 (4), (2019).
- Nickerson, K. P., et al. A versatile human intestinal organoid-derived epithelial monolayer model for the study of enteric pathogens. Microbiol Spectr. 9 (1), 1-17 (2021).
- Perlman, M., Senger, S., Verma, S., Carey, J., Faherty, C. S. A foundational approach to culture and analyze malnourished organoids. Gut Microbes. 15 (2), 2248713 (2023).
- Pope, L. M., Reed, K. E., Payne, S. M. Increased protein secretion and adherence to HeLa cells by Shigella spp. following growth in the presence of bile salts. Infect Immun. 63 (9), 3642-3648 (1995).
- Faherty, C. S., et al. The synthesis of OspD3 (ShET2) in Shigella flexneri is independent of OspC1. Gut Microbes. 7 (6), 486-502 (2016).
- Ridlon, J. M., Kang, D. -. J., Hylemon, P. B. Bile salt biotransformations by human intestinal bacteria. J Lipid Res. 47 (2), 241-259 (2006).
- Köseoğlu, V. K., Hall, C. P., Rodríguez-López, E. M., Agaisse, H. The Autotransporter IcsA promotes Shigella flexneri biofilm formation in the presence of bile salts. Infect Immun. 87 (7), 1-14 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon