Method Article
Quantificare le attività di Cis-Regolamentazione Elementi nella retina di topo Elettroporazione Espianto
In questo articolo
Riepilogo
Questo protocollo descrive un modo semplice ed economico per quantificare l'attività del cis-elementi normativi (ad esempio, enhancer / promoter), nel vivere retine del mouse tramite elettroporazione espianto. Preparazione di analisi del DNA, la dissezione della retina, elettroporazione, cultura espianto della retina, e post-fissazione e la quantificazione sono descritti.
Abstract
Fattori di trascrizione genetica all'interno di reti cellulari di controllo del modello spazio-temporali e livelli di espressione dei loro geni bersaglio legandosi a cis-normativo elementi (Cres), breve (~ 300-600 pb) tratti di DNA genomico che può mentire a monte, a valle, o entro gli introni dei geni che controllano. CRES (cioè, esaltatori / promotori) in genere costituiti da più siti raggruppati vincolante per entrambe le attivatori e repressori trascrizionali 1-3. Essi servono come integratori logica di input trascrizionale dare un rendimento unitario nella forma di attività promotore spatiotemporally preciso e quantitativo esatto. La maggior parte degli studi di mammiferi cis-regolazione fino ad oggi hanno fatto affidamento sulla transgenesi mouse come un mezzo per saggiare la funzione potenziatore del CRES 4-5. Questa tecnica richiede tempo, costosa e, a causa degli effetti di sito di inserimento, in gran parte non quantitativi. D'altra parte, analisi quantitative per mammiferi funzione CRE sono stati sviluppati in sistemi di coltura tissutale (ad esempio, test dual luciferasi), ma la rilevanza in vivo di questi risultati è spesso incerto.
Elettroporazione offre un'ottima alternativa per transgenesi mouse tradizionale in quanto consente sia la valutazione spazio-temporale e quantitativa di cis-regolamentazione dell'attività in tessuti viventi dei mammiferi. Questa tecnica è stata particolarmente utile nell'analisi di cis-regolazione del sistema nervoso centrale, specialmente nella corteccia cerebrale e della retina 6-8. Mentre elettroporazione del mouse della retina, sia in vivo ed ex vivo, è stato sviluppato e ampiamente descritto da Matsuda e Cepko 6-7,9, abbiamo recentemente sviluppato un approccio semplice per quantificare l'attività di fotorecettori specifici CRES in elettroporate topo retine 10. Dato che la quantità di DNA che viene introdotto nella retina mediante elettroporazione può variare da esperimento per esperimento, è necessario includere una co-elettroporate 'di controllo di carico' in tutti gli esperimenti. A questo proposito, la tecnica è molto simile al dosaggio doppio luciferasi utilizzato per quantificare l'attività promotore in cellule in coltura.
Quando il saggio fotorecettore cis-regolamentazione dell'attività, elettroporazione è di solito eseguita in topi neonati (giorno dopo la nascita 0, P0) che è il tempo del picco di produzione asta 11-12. Una volta che i tipi di cellule della retina diventano post-mitotico, elettroporazione è molto meno efficiente. Dato l'alto tasso di natalità asta nei topi appena nati e il fatto che aste costituiscono oltre il 70% delle cellule della retina topo adulto, la maggior parte delle cellule che sono elettroporate a P0 sono aste. Per questo motivo, bastoncelli sono le più facili tipo di cellule della retina per studiare tramite elettroporazione. La tecnica che descriviamo qui è soprattutto utile per quantificare l'attività del CRES fotorecettori.
Protocollo
1. Costruzione della camera di elettroporazione
- Ordina una microslide, BTX Modello 453 con un gap 3,2 mm (Harvard Apparatus # 45-0105) (Fig. 2A). I binari di metallo dovrebbero essere completamente sigillati alla parte inferiore della diapositiva.
- Utilizzare uno strumento di Dremel per tagliare fuori un manico di un rack di plastica provetta. Tagliare il manico in 5 piccoli pezzi rettangolari ognuno con le seguenti dimensioni: 0,8 centimetri di lunghezza, 0,6 centimetri altezza, larghezza 0,3 centimetri (Fig. 2B). Questi pezzi di plastica sono distanziatori riutilizzabili che verranno utilizzati per modellare singoli pozzi nella camera microslide.
- Inserire i distanziatori di plastica tra le rotaie metalliche del microslide a intervalli regolari. I distanziatori dovrebbero andare bene comodamente (Fig. 2C).
- Tagliare la punta fuori una punta P200 pipetta, inserire la punta di una siringa da 3 ml, e riempire la siringa con il 100% gomma di silicone sigillante acquario. Riempire i vuoti tra i distanziatori di plastica con sigillante. Assicurati di colmare le lacune dal basso verso l'alto in modo che non bolle si formano tra il tappo sigillante e la base del microslide.
- Inserire una barra di metallo sopra i distanziali in plastica e fissarlo con clip legante, in modo che i distanziali sono tenuti in posizione mentre si asciuga sigillante (Fig. 2D-E). Lasciate che il sigillante asciugare durante la notte.
- Rimuovere la clip legante, asta di metallo, e distanziali in plastica (Fig. 2F). Utilizzare una lama di bisturi per pulire il sigillante la parte superiore delle rotaie metalliche. Utilizzare un microscopio da dissezione per esaminare la microslide e assicurarsi che non siano presenti bolle alla parte inferiore delle dighe di silicone. Rimuovere eventuali pellicola sigillante che possono essersi formate in modo tale che i pozzi di metallo nudo è esposto all'interno del pozzetto. Riempire un pozzo con acqua e fare in modo che l'acqua non perde nel pozzo adiacente (s). Ripetere l'operazione per tutti i pozzetti.
- Il microslide finito si inserisce nel piatto di plastica con i poli di metallo vicino alla finestra sul lato del piatto (Fig. 2G), gli elettrodi saranno eventualmente fissato ai poli di metallo.
2. Estrazione del DNA
- Aggiungi DNA plasmidico in una provetta da microcentrifuga 1,5 ml su ghiaccio e portare il volume fino a 150μl con acqua distillata. Molteplici specie plasmide possono essere combinati per il co-elettroporazione (ad esempio, una costruzione sperimentale DsRed e un controllo costrutto GFP). Nel calcolare la quantità di DNA da aggiungere al tubo, tenere presente che il volume finale della aliquota DNA sarà 60μl. In genere, ogni costrutto è utilizzato ad una concentrazione finale di 0.5μg/μl.
- Precipitare il DNA con l'aggiunta di 15μl acetato di sodio 3M (pH 5.2) e 450μl di etanolo al 100%. Invertire o toccare il tubo più volte per mescolare.
- Rallenta il DNA a 4 ° C, 13.200 giri al minuto, per 30 minuti. Lavare il pellet con etanolo al 70%, poi girare di nuovo a 4 ° C, 13.200 giri al minuto, per 15 minuti. Far asciugare il pellet fino semitrasparente, circa 7 minuti, poi risospendere in 54μl di acqua sterile. Aggiungi 6μl sterile PBS 10X (pH 7,4) e mescolare.
3. Occhio raccolta
- Sterilizzare la camera di elettroporazione e tutti gli strumenti con il 70% di etanolo. Mentre la raccolta degli occhi e la dissezione non deve essere eseguita in una cappa di colture di tessuti, disinfettare i guanti e da banco con etanolo e tentativo di mantenere condizioni di sterilità durante tutta la procedura.
- Preparare Petri con dissezione medio (rapporto 1:1 di DMEM: F12, 100U/ml penicillina, streptomicina 100μg/ml, 0.29mg/ml L-glutamina, e l'insulina 5μg/ml): due piatti ciascuno con 35 millimetri di media da 3 ml e una piatto 60mm con 6 ml di media. Questa operazione deve essere eseguita in una cappa di coltura di tessuti.
- Disinfettare la testa e il collo di un neonato (giorno postnatale 0) topo cucciolo con il 70% di etanolo. Velocemente decapitare con le forbici e trasferire la testa per un piatto sterile 100mm.
- Tagliare via il cuoio capelluto con piccole forbici per esporre gli occhi. Utilizzare pinze curve per raccogliere delicatamente l'occhio fuori dall'orbita, e posto l'occhio in un piatto contenente 35 millimetri di media dissezione. Può essere utile per rimuovere gli occhi sotto un microscopio da dissezione a bassa potenza.
- Ripetere i passi 3.3 e 3.4 fino a quando tutti gli occhi sono stati raccolti. Tenere gli occhi a medio dissezione a temperatura ambiente, mentre dissezione. Avrete bisogno di 3-4 per gli occhi aliquota del DNA.
4. Dissezione della retina
- Utilizzare etanolo al 70% per disinfettare sia una lama di rasoio e l'involucro di una pipetta sterile trasferimento di plastica. Tagliare la punta fuori la pipetta con la lama in modo che possa aspirare un occhio intero. Conservare la pipetta nella confezione di plastica quando non in uso.
- Trasferire un occhio dal piatto 35mm a il piatto 60mm. Sotto il microscopio da dissezione ad alta potenza, usare una pinza sottile per rimuovere qualsiasi tessuto, come i muscoli extraoculari e grasso, dalla superficie dell'occhio. Quindi, rimuovere il nervo ottico da pizzicandola fuori alla base.
- Al fine di isolare la retina, colpire un piccolo foro in la sclera al limbus. Inserire una punta da entrambe le coppie di pinze nel foro (tangente alla superficie della retina) e delicatamente lacerare la sclera / RPE. Nei topi albini, la sclera e RPE appaiono relativamente lucido al tessuto della retina, che è un colore omogeneo grigio opaco. Nei topi pigmentati, la RPE è nero. Lascia la lente in posizione.
- Utilizzare la pipetta di trasferimento per spostare la retina sezionato nel piatto da 35 mm con altri media.
- Ripetere i passaggi 4,2-4,4 fino a quando tutti gli occhi sono stati sezionati.
- Conservare la retina in un incubatore a 37 ° la cultura del tessuto C fino a quando si è pronti a electroporate.
5. Preparazione per elettroporazione
- Preparare piatti 35 mm di media. Per ciascuna aliquota di DNA da elettroporate, sarà necessario un piatto dai media dissezione e un piatto di terreno di coltura (media dissezione più 10% FBS). Etichetta i piatti in modo appropriato.
- Utilizzare una pipetta P200 e sterile PBS 1X per lavare le camere nel piatto elettroporazione. Ogni camera ha un volume di 60-100μl. Lavare ogni camera tre volte.
- Riempire le camere con le aliquote del DNA. Camere a non utilizzata deve essere riempito con 60μl PBS 1X. Collegare gli elettrodi al piatto elettroporazione.
- Utilizzare le seguenti impostazioni sul elettroporatore: modalità, LV, la tensione, 30V, lunghezza di impulso, 50 msec, il numero di impulsi, 5; intervallo, 950 msec; polarità, unipolare.
6. Elettroporazione
- Utilizzare una pinza sottile di cogliere la retina dalla lente e trasferirli nelle camere di elettroporazione. Ogni camera può contenere fino a 3-4 topo retina (Fig. 3).
- Usare pinze per allineare la retina in modo che la lente si appoggia la barra di metallo collegato l'elettrodo positivo. Pulire la pinza con un Kimwipe dopo ogni trasferimento per evitare riporto di DNA da una camera all'altra.
- Una volta che tutte le retine sono allineati, premere il tasto "Start" sulla elettroporatore. Bollicine dovrebbero modulo a barra di metallo collegato l'elettrodo negativo.
- Scollegare gli elettrodi e spegnere il elettroporatore.
- Usare pinze per spostare delicatamente la retina lontano dalle pareti della camera.
- Utilizzare una pipetta sterile trasferimento di trasferire la retina dalle camere nei piatti contenenti 35 millimetri di media dissezione.
- Lavare ogni camera tre volte con PBS 1X sterile, quindi risciacquare con acqua sterile. Piatto a spruzzo con il 70% di etanolo.
7. Posizionamento retine sui filtri per la cultura
- Utilizzare una pipetta di trasferimento per trasferire la retina nei piatti 35 millimetri contenente terreno di coltura.
- Etichettare il pozzetti di una sterile 6 pozzetti cultura e riempire ogni bene con terreno di coltura da 3 ml.
- Usa pinza sterile per posizionare l'filtri Whatman Nuclepore, parte lucida in su, in cima alla media in ciascun pozzetto.
- Sotto un microscopio da dissezione, utilizzare una pipetta sterile trasferire per trasferire la retina sul filtro, lente-side-down. Se le terre retina lente-side-up, pick up con la pipetta e tentare di mettere di nuovo. Non mettere più di 4 retine su un filtro, e assicurarsi che le goccioline di mezzo circostante ogni retina rimanere separata dalle altre gocce. Si noti che la cultura la retina con la lente intatta, sebbene altri protocolli appello per la rimozione della lente prima di questo passo 9.
- Posizionare la piastra di coltura a 37 ° C di coltura tissutale incubatore (5% CO 2) e crescono per la quantità desiderata di tempo, di solito otto giorni. Nella nostra esperienza, cambiando il mezzo è inutili durante il periodo di otto cultura giorno, anche se un cambio medio può essere richiesto per periodi più lunghi cultura.
8. Raccolta e flatmounting fluorescenti espianti di retina
- Sostituire il terreno di coltura di ogni bene con il 4% paraformaldeide / PBS 1X. Se le retine rimangono attaccate al filtro, utilizzare pinze per capovolgere i filtri sopra e delicatamente sbucciare la retina il filtro. Incubare in paraformaldeide per 30 min. a temperatura ambiente. Proteggere la retina dalla luce per evitare di sbiancamento la fluorescenza.
- Sciacquare la retina due volte per 10 minuti in PBS 1X.
- Utilizzare una pipetta monouso per trasferire la retina su un vetrino in una piccola goccia di PBS. Sotto un microscopio a fluorescenza dissezione, utilizzare pinze per capovolgere la retina in modo che siano elettroporate-side-up (cioè, lente-side-down).
- Luogo di vetro "piedi" a base di schiacciata coprioggetto (frammenti di vetro di circa 1-2 mm di diametro) agli angoli della diapositiva; questi piedi prevenire appiattimento della retina. Inserire un coprioggetto intatta di vetro sopra la diapositiva in modo che copra la retina e poggia sui piedi. Se necessario, utilizzare una pipetta per aggiungere più PBS tra il vetrino ed il coprioggetto.
9. Imaging e quantificazione della fluorescenza in flatmount
- Utilizzare unmicroscopio a fluorescenza equipaggiato con una telecamera in bianco e nero per l'immagine della retina flatmounted a bassa potenza (obiettivo 4X) nei canali rosso e verde. Tutte le retine deve essere ripreso con il tempo di esposizione stesso per un dato canale fluorescente per consentire un confronto di intensità di fluorescenza. Assicurarsi che i pixel non sono saturi in qualsiasi immagine, altrimenti la quantificazione precisa sarà impossibile. Esportazione di immagini in scala di grigi formato TIFF.
- Aprite l'immagine impostata per una retina (cioè .., canale rosso e verde canale) in ImageJ software ( http://rsbweb.nih.gov/ij/ ). Per il bene di questo tutorial, il canale verde fluorescente (GFP proteine) è il costrutto di controllo che è costante in tutte le retine per l'esperimento. Il canale rosso fluorescente (proteine DsRed) è il costrutto sperimentale che varia per ogni serie di retine. Le immagini devono essere in scala di grigi.
- In ImageJ, selezionare l'immagine verde di controllo e specificare un cerchio di interesse con diametro di 100 unità (Analisi / Strumenti / ROI manager / Altri / specificare). Duplicare questo cerchio (ROI manager / Aggiungi) per creare otto cerchi totale. Spostare circoli 1-5 per selezionare cinque regioni che sono uniformemente elettroporate, evitando i bordi esterni della retina e la regione sovrastante la lente (Fig. 4A). Inoltre, selezionare tre regioni (cerchi 6-8) al di fuori della retina / obiettivo per misurare la fluorescenza di fondo. Selezionare l'immagine rossa, deselezionare la "Mostra tutto" nella casella ROI manager, e ri-selezionare la casella. Tutti gli otto cerchi dovrebbe apparire l'immagine rossa. De-selezionare tutte le coordinate in cerchio direttore ROI.
- Con l'immagine selezionata rosso, registrare il valore medio dei pixel per tutti i circoli di interesse (ROI direttore / Misura), misure 1-8 dovrebbe comparire, in cui 1-5 sono le misure rosso della retina e 6-8 sono le misure sfondo rosso. Selezionare l'immagine verde e registrare il valore medio dei pixel; misure 9-16 deve presentarsi, dove sono le misure 9-13 verde della retina e 14-16 sono le misure sfondo verde. Copiare i dati di misurazione in Excel per l'analisi (Fig. 5).
- Media delle tre misure di fondo sia il rosso e il canale verde. Sottrarre la media sfondo rosso da ciascuna delle cinque misure della retina nel canale rosso, ripetere per il canale verde. Per ogni regione della retina di interessi, dividere il background-sottratto misura dallo sfondo rosso-verde sottratto misura al fine di normalizzare il livello sperimentale rosso per il livello di controllo verde (Fig. 5).
- Determinare la media e deviazione standard di tutte le misurazioni normalizzato per un costrutto dato DsRed (ad esempio, 5 misure per 3 volte retina retina separato). Per confrontare quantitativamente i risultati di electroporations svolte in giorni diversi, sempre includere una precipitazione "standard" DsRed / GFP in ogni set elettroporazione. I valori relativi espressione attraverso gli esperimenti possono essere confrontati con la normalizzazione del livello di espressione di questo "standard".
10. Rappresentante dei risultati:
Un risultato elettroporazione buona espressione del costrutto di DNA (s) in 1 / 4 a 1 / 3 della superficie retinica (Fig. 4A). Dal bastoncelli, in particolare, vengono efficacemente trasdotte, questa tecnica è ideale per la quantificazione dei fotorecettori attività specifica promotore (Fig. 4B). In precedenza avevamo usato questo metodo per quantificare una serie di varianti promotore dallo stelo specifiche Rho e Gnat1 loci 10. Abbiamo scoperto che è possibile quantificare l'attività nel corso di un promotore quasi 300 volte gamma.
Figura 5 è un set di dati campione da un singolo elettroporate retina. In questo esempio particolare, la sperimentazione costruire pNrl (1.1kb)-DsRed è stata misurata nel canale rosso e il controllo costruire pNrl (3.2kb)-GFP è stata misurata nel canale verde. Un set di dati completi per la pNrl (1.1kb)-DsRed costruire consisterebbe di 6-9 retine misurato in questo modo, e la deviazione standard sarà calcolato sulla base di tutte "DsRed normalizzato a GFP" valori. Se dovessimo confrontare il livello di espressione di pNrl (1.1kb)-DsRed, per esempio, pNrl (0.8kb)-DsRed, allora entrambi i costrutti avrebbe bisogno di essere elettroporate con il controllo GFP stesso (ad esempio, pNrl (3.2kb) - GFP) e ripreso ai tempi di esposizione. E 'possibile raggruppare i dati raccolti in giorni diversi, se uno standard DsRed / elettroporazione GFP viene eseguita ogni giorno (ad esempio, pNrl (3.2kb)-DsRed + pNrl (3.2kb)-GFP). Per ciascun costrutto sperimentale, il livello normalizzato DsRed sarebbe successivamente normalizzata al livello normalizzato DsRed del "standard" (pNrl (3.2kb)-DsRed).
La tecnica che descriviamo qui è soprattutto utile per quantificare l'attività del CRES fotorecettori 10,13.Cell-specifico tipo cis-regolamentare l'attività può essere quantificato in più rari tipi di cellule retiniche come le cellule bipolari 14, ma questo di solito richiede che le aree di interesse da quantificare essere selezionata in verticale sezioni piuttosto che nelle preparazioni flatmount. Lo stesso vale per CRES quale unità espressione in molteplici tipi cellulari quali fotorecettori e cellule bipolari. Le procedure sperimentali sono altrimenti simili.
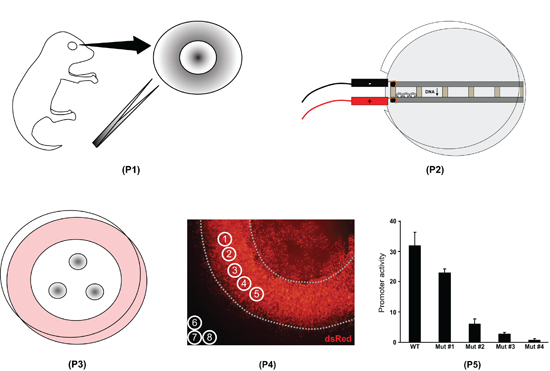
Figura 1. Panoramica della procedura di espianto della retina elettroporazione. In primo luogo, gli occhi sono interi isolati da postnatale giorno 0 cuccioli di topo e la retina sono sezionati (P1). In secondo luogo, le retine sono posti in camere piene di DNA e elettroporate (P2). In terzo luogo, le retine sono posti su filtri e in coltura per otto giorni (P3). Quarto, la espianti retinici sono fisse, montate su vetrini e immagine. Intensità di fluorescenza viene misurata con il software ImageJ (P4). Quinto, i dati ImageJ sono trattati in un foglio di calcolo per quantificare la differenza di attività dei diversi promotori (P5).

Figura 2. Costruzione del piatto elettroporazione. A) camera di microslide Unmodified di Harvard Apparatus, BTX modello 453 (catalogo # 45-0105). B) Uno strumento Dremel è usato per tagliare il manico fuori un rack tubo di plastica. Il manico è tagliato in distanziatori rettangolari con le seguenti dimensioni: 0,8 centimetri di lunghezza, 0,6 centimetri altezza, larghezza di 0,3 centimetri. C) I distanziatori di plastica sono montati nella camera microslide a intervalli uguali. Acquario sigillante viene iniettato nelle fessure tra i distanziatori (non mostrato). D) Una barra di metallo è posto sopra i distanziali. E) Il bar e distanziali sono fissati sul vetrino con clip legante per tenere tutto a posto come il sigillante si asciuga durante la notte. F) I distanziatori vengono rimossi ei pozzi sono testati per assicurare che siano a tenuta stagna. G) La slitta finito si inserisce nel piatto di plastica con le barre di metallo vicino alla finestra sul lato del piatto.

Figura 3. Schema del piatto elettroporazione con retine. Le camere sono piene di soluzioni di DNA (fino a cinque soluzioni diverse alla volta). Retine sono collocati nelle camere e orientata in modo che l'obiettivo è appoggiato al bancone di metallo collegato con l'elettrodo positivo, tre o quattro retina si adatta a ciascuna delle cinque Camere. La corrente elettrica provoca le molecole di DNA carichi negativamente per spostare nelle cellule della retina.

Figura 4. A) Misura ImageJ della retina livelli di fluorescenza in flatmount. Scala di grigi flatmount nel DsRed (sperimentale) e la GFP (controllo) i canali sono aperti nel software ImageJ; notare che queste immagini sono state colorate ha scopi puramente illustrativi. Cinque cerchi di misura (1 a 5) sono disposti sopra le regioni in modo uniforme elettroporate, evitando i bordi e lente (linee tratteggiate). Tre cerchi di misura (6 a 8) sono posti al di fuori della retina per determinare i livelli di fluorescenza di fondo. B) trasversale immagini di un espianto elettroporate retina ad alta potenza. L'espianto è stato fissato a 8 giorni dopo la nascita, crioprotetti nel 30% sucrose/1X PBS notte a 4 ° C, incorporato in ottobre, e crio-sezionati a 12μm. La fluorescenza costruisce pNrl (1.1kb)-DsRed e pNrl (3.2kb)-GFP sono espressi nelle cellule dei fotorecettori nello strato nucleare esterno (ONL). INL, strato interno nucleare; GCL, strato delle cellule gangliari.

Figura 5. Trattamento dei dati fluorescenza utilizzando Excel. Nella fase 1, il valore medio per ogni pixel cerchio misura è copiato nel foglio di calcolo (celle B3-B12, F3, F5, H3-H5). Misure # 1-5 sono i valori DsRed retina e # 6-8 sono i valori di fondo DsRed; misurazioni # 9-13 sono i valori GFP della retina e # 14-16 sono i valori di fondo GFP. Si noti che la misura # 1 e # 9 corrispondono al cerchio stessa misura, come fanno le misurazioni # 2 e # 10, e così via. Al passo 2, il valore di fondo media per i canali DsRed e GFP è calcolato (cellule F6, H6). Al punto 3, il fondo media è sottratto da ogni misurazione della retina (cellule C3-C12). Al punto 4, ogni sfondo, sottratto misura DsRed è normalizzato per la sua misura corrispondente GFP (celle D3-D7).
Discussione
Espianto elettroporazione è un mezzo semplice di quantificare cis-regolamentare l'attività del mouse nella retina di sviluppo. Rispetto al cis-normativo analisi tramite transgenesi mouse, elettroporazione è molto più economico, che richiede solo cuccioli di topo neonato, il DNA, strumenti di dissezione, ed elettroporazione / cultura materiale dei tessuti. E 'anche molto meno tempo di consumo: esperimento si richiede solo poche ore di tempo di preparazione, un periodo di coltura di circa otto giorni, e poche ore alla fine della sperimentazione per l'imaging e analisi dei dati. Espianto elettroporazione è anche superiore alla coltura cellulare a base di cis-regolamentazione analisi dal reale tessuto retinico viene utilizzato. La retina si sviluppa abbastanza normalmente in espianto cultura si forma tre strati distinti cellulare (strato esterno nucleare, nucleare strato interno e strato di cellule gangliari), anche se fotorecettori non riescono a elaborare segmenti esterni.
Un ulteriore vantaggio è che elettroporazione espianto è altamente riproducibile. I costrutti stesso elettroporate in retine diversi, anche in giorni diversi, in genere si traduce in livelli stessa espressione relativa. Inoltre, poiché i plasmidi elettroporate si pensa siano mantenuti episomally nel nucleo e non sono incorporati nei cromosomi, essi non sembrano essere soggette agli effetti stesso sito di integrazione che tormentano cis-normativo analisi effettuate nei topi transgenici.
Elettroporazione espianto ha diverse limitazioni. In primo luogo, solo le cellule che sono ancora nel ciclo cellulare può essere efficacemente trasdotte con elettroporazione 15. A P0, aste e altri più tardi, nato tipi di cellule della retina (cellule bipolari, cellule amacrine, M ller glia) sono i principali popolazioni di cellule bersaglio con questo metodo. Elettroporazione di fotorecettori cono da P0 elettroporazione è stato segnalato 16, ma l'efficacia sembra essere basso. Un secondo limite è che la cultura espianto di là di due risultati settimane malformazione progressiva della retina e non è pertanto raccomandato. Se quantificazione promotore è tenuto a timepoints tardi, tuttavia, un elettroporazione in vivo 9 possono essere eseguite, seguita da dissezione della retina al timepoint desiderato, piatto di montaggio della retina sezionato, e quantificazione come descritto nella sezione 9. Una terza limitazione è che questo saggio è solo moderatamente elevato throughput. A differenza dei saggi di cultura a base di cellule che possono testare centinaia di costruzioni in un singolo esperimento, la tecnica descritta in questo protocollo richiede un minimo di un retina del mouse tutto per costruire. Così, solo un paio di dozzine di costruzioni possono essere ragionevolmente elettroporate in un giorno.
Un avvertimento aggiuntivi rispetto alla quantificazione dell'attività di promotore con l'attuale approccio è che c'è un potenziale di 'sanguinare-through' di fluorescenza DsRed nel canale GFP, in particolare quando i promotori dosaggio molto forte. La ragione di questo è che lo spettro di emissione di DsRed in parte si sovrappone a quella di GFP. Per aggirare questo problema, filtri di emissioni ottimizzati dovrebbe essere utilizzato che riducano al minimo la sovrapposizione spettrale tra DsRed e GFP. Quando tali insiemi ottimizzato filtro non sono disponibili, un'altra possibile soluzione sarebbe quella di utilizzare un blu-spostato proteina fluorescente (per esempio, BFP o CFP) al posto di GFP.
Divulgazioni
Riconoscimenti
Gli autori desiderano ringraziare Karen Lawrence per il suo aiuto con sezione che descrive la costruzione della camera di elettroporazione.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
|---|---|---|---|
| Elettroporazione piatto, Microslide 453 | BTX Harvard Apparatus | 45-0105 | Vedere la sezione 1 del protocollo per le modifiche |
| 100% gomma siliconica acquario cemento | Perfecto Produzione | ||
| Microprovette cremagliera in plastica | Fisher Scientific | 05-541 | Solo il manico cremagliera saranno utilizzati |
| Dremel strumento | Per tagliare il manico fuori del rack tubo di plastica | ||
| DMEM | Gibco / Invitrogen | 11965 | |
| F12 | Gibco / Invitrogen | 11765 | |
| L-Glu/pen/strep | Gibco / Invitrogen | 10378-016 | Concentrazione 100X |
| Insulina | Sigma-Aldrich | I-6634 | Per magazzino 1000X, risospendere in 5mg/ml in 5mM HCl e filtro-sterilizzare |
| FBS | Gibco / Invitrogen | 26140-079 | |
| ECM 830 onda quadra elettroporatore | BTX Harvard Apparatus | ||
| Nuclepore filtri | Whatman | 110606 | 25mm, 0.2μm |
| Tessuto cultura incubatore | 37 ° C, 5% CO 2 | ||
| Coprioggetto di vetro # 1.5 | Fisher Scientific | 12-544E | 0,16 millimetri di spessore |
| Composto microscopio a fluorescenza dotato di telecamera | Fotocamera deve essere monocromatica (ad esempio, ORCA-ER fotocamera da Hamamatsu) | ||
| EGFP / DsRed set di filtri per il microscopio composto | Chroma Technology Corp. | 86007 | Questo bleedthrough set di filtri riduce al minimo tra il chanenels rosso e verde |
| ImageJ Software | NIH | http://rsbweb.nih.gov/ij/ |
Riferimenti
- Carroll, S. B., Grenier, J. K., Weatherbee, S. D. . From DNA to diversity : molecular genetics and the evolution of animal design. , (2005).
- Davidson, E. H. . Genomic regulatory systems : development and evolution. , (2001).
- Ptashne, M., Gann, A. . Genes & signals. , (2002).
- Blow, M. J. ChIP-Seq identification of weakly conserved heart enhancers. Nat Genet. 42, 806-810 (2010).
- Visel, A. ChIP-seq accurately predicts tissue-specific activity of enhancers. Nature. 457, 854-858 (2009).
- Matsuda, T., Cepko, C. L. Electroporation and RNA interference in the rodent retina in vivo and in vitro. Proc Natl Acad Sci U S A. 101, 16-22 (2004).
- Matsuda, T., Cepko, C. L. Controlled expression of transgenes introduced by in vivo electroporation. Proc Natl Acad Sci U S A. 104, 1027-1032 (2007).
- LoTurco, J., Manent, J. B., Sidiqi, F. New and improved tools for in utero electroporation studies of developing cerebral cortex. Cereb Cortex. 19, 120-125 (2009).
- Matsuda, T., Cepko, C. L. Analysis of gene function in the retina. Methods Mol Biol. 423, 259-278 (2008).
- Lee, J., Myers, C. A., Williams, N., Abdelaziz, M., Corbo, J. C. Quantitative fine-tuning of photoreceptor cis-regulatory elements through affinity modulation of transcription factor binding sites. Gene Ther. 17, 1390-1399 (2010).
- Carter-Dawson, L. D., LaVail, M. M. Rods and cones in the mouse retina. II. Autoradiographic analysis of cell generation using tritiated thymidine. J Comp Neurol. 188, 263-272 (1979).
- Young, R. W. Cell differentiation in the retina of the mouse. Anat Rec. 212, 199-205 (1985).
- Corbo, J. C. CRX ChIP-seq reveals the cis-regulatory architecture of mouse photoreceptors. Genome Res. 20, 1512-1525 (2010).
- Kim, D. S., Matsuda, T., Cepko, C. L. A core paired-type and POU homeodomain-containing transcription factor program drives retinal bipolar cell gene expression. J Neurosci. 28, 7748-7764 (2008).
- Karra, D., Dahm, R. Transfection techniques for neuronal cells. J Neurosci. 30, 6171-6177 (2010).
- Onishi, A., Peng, G. H., Chen, S., Blackshaw, S. Pias3-dependent SUMOylation controls mammalian cone photoreceptor differentiation. Nat Neurosci. 13, 1059-1065 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon