È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Microscopia di lievito di fissione del ciclo di vita sessuale
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
We provide a reproducible basic method for the long-term microscopy of the fission yeast sexual lifecycle. With minor adjustments described, the presented protocol allows research focus on different steps of the reproductive process.
Abstract
The fission yeast Schizosaccharomyces pombe has been an invaluable model system in studying the regulation of the mitotic cell cycle progression, the mechanics of cell division and cell polarity. Furthermore, classical experiments on its sexual reproduction have yielded results pivotal to current understanding of DNA recombination and meiosis. More recent analysis of fission yeast mating has raised interesting questions on extrinsic stimuli response mechanisms, polarized cell growth and cell-cell fusion. To study these topics in detail we have developed a simple protocol for microscopy of the entire sexual lifecycle. The method described here is easily adjusted to study specific mating stages. Briefly, after being grown to exponential phase in a nitrogen-rich medium, cell cultures are shifted to a nitrogen-deprived medium for periods of time suited to the stage of the sexual lifecycle that will be explored. Cells are then mounted on custom, easily built agarose pad chambers for imaging. This approach allows cells to be monitored from the onset of mating to the final formation of spores.
Introduzione
Anche se lo scambio genetico tra due cellule è l'evento centrale nella riproduzione sessuale, si basa su una catena di eventi che promuovono la differenziazione cellulare, permettono di scelta del partner, svolgono la fusione cellula-cellula e mantenere la stabilità genomica. Così il ciclo di vita sessuale si presenta come un sistema modello per studiare una serie di questioni biologiche riguardanti gli interruttori di sviluppo, risposta a stimoli estrinseci, fusione della membrana plasmatica, la segregazione dei cromosomi, ecc Esplorando il lievito di fissione ciclo sessuale di studiare questi fenomeni porta i vantaggi della genetica potenti del sistema modello, ben stabiliti approcci high-throughput e sofisticato microscopio. Sex in fissione lievito è un evento heterotypic tra una P-cellulare e una M-cellula tipi di accoppiamento distinti. I due tipi di cellule in modo differenziale esprimono un numero di geni 1,2, compresi quelli per la produzione del P secreto e M-feromoni, feromone-recettori map3 e MAM2 così come feromone-proteases Sxa1 e Sxa2. Ceppi Homothallic, come il ceppo H90 comunemente usato, trasportano le informazioni genetiche per entrambi i tipi di accoppiamento in un singolo genoma e cellule subiscono un complesso schema di commutazione tipo di accoppiamento nell'intero ciclo mitotico (valutata in Rif. 3). Molteplici isolati di heterothallic lievito di fissione che cambiano raramente o mai il tipo di accoppiamento sono comunemente utilizzati 4, il più prominente H + N (P-type) e h -S (tipo M) ceppi.
Nel lievito di fissione, l'ingresso nel ciclo di vita sessuale è sotto stretta regolamentazione nutrizionale. Solo le cellule di lievito di fissione azoto-fame arrestano riproduzione mitotico e producono feromoni diffusibili per segnalare la presenza di un partner di accoppiamento e promuovere ulteriori fasi del ciclo sessuale (rivisto in Rif. 5). Azoto privazione de-reprime il regolatore trascrizionale chiave di accoppiamento Ste11 che agisce come un interruttore di sviluppo e promuove eXpression di accoppiamento specifici geni tra cui il recettore feromone e il feromone geni di produzione 6,7. Impegno feromone-recettore attiva la proteina recettore accoppiato G-alfa ed a valle di segnalazione MAPK che migliora ulteriormente Ste11 attività trascrizionale 8-10, aumentando così la produzione di feromoni in un feedback positivo tra i partner di accoppiamento. Livelli di feromone sono cruciali per indurre diversi stati di polarizzazione delle cellule regolando l'organizzatore maestro di polarità delle cellule, la Rho-famiglia GTPasi Cdc42 11. Dopo l'esposizione a basse concentrazioni di feromoni, attiva Cdc42 viene visualizzato nella patch dinamici esplorare la periferia cellulare, e nessuna crescita cellulare si osserva in questa fase. I livelli aumentati di feromoni promuovere la stabilizzazione dell'attività Cdc42 ad una singola zona e la crescita di una proiezione polarizzata, chiamata shmoo, che porta le cellule socio a contatto. Successivamente, i due partner di accoppiamento aploidi si fondono per formare uno zigote diploide. Un recente lavoro rivela °e esistenza di una struttura actina nuova essenziale per la fusione che è assemblato dal formin accoppiamento indotta Fus1 12. Questa focalizzazione fusione concentra tipo V miosina processi dipendenti e posiziona il macchinario degrado parete cellulare, permettendo così rimodellamento della parete cellulare per consentire il contatto membrana plasmatica senza lisi cellulare 12. Al momento della fusione cellula-cellula, i nuclei vengono a contatto e sottoposti cariogamia. Un importante dynein-dipendente movimento avanti e indietro del nucleo all'interno della zigote (il movimento coda di cavallo) promuove quindi l'abbinamento di omologhi cromosomi 13,14, che è seguito da meiosi. Infine, i quattro prodotti della meiosi sono confezionati in singole spore durante sporulazione.
A causa della sua complessità e le numerose fasi, monitoraggio dettagliato di accoppiamento è stato impegnativo. Due difficoltà notevoli sono che l'intero processo richiede ben oltre quindici ore e che le cellule sono difficili da sincronizzare. questi difficulties sono aggirate da approcci di microscopia unicellulari. Qui un protocollo generale per indagare il ciclo di vita sessuale di lievito di fissione è presentato. Con piccoli aggiustamenti, questo protocollo permette lo studio di tutte le varie fasi del processo, vale a dire l'induzione di accoppiamento prodotto del gene, la polarizzazione delle cellule e abbinamento tra sorelle cellule dopo il passaggio da tipo di accoppiamento e tra i partner non-sorella, la fusione cellula-cellula, e post-fusione coda di cavallo il movimento, la meiosi e sporulazione. Questo metodo permette di 1) facilmente visualizzare fluorescently tag proteine nel corso del tempo pre, durante e post-fusione; 2) discriminare il comportamento delle cellule di tipo opposto accoppiamento; e 3) misura e quantificare parametri come shmooing, l'accoppiamento, la fusione o l'efficienza sporulazione.
Protocollo
Analisi al microscopio di lievito di fissione riproduzione sessuale
1. Carta Preparazione
- Preparare terreno minimo sporulazione (MSL-N) 15 mescolando i seguenti componenti: Glucosio: 10 g / L, KH 2 PO 4: 1 g / L, NaCl 0,1 g / L, MgSO 4 · 7H 2 O: 0,2 g / L. Aggiungere Oligoelementi (10.000X): 100 l / L, vitamine (1,000x): 1 ml / L, e 0,1 M CaCl 2 ml / L. Filtro-sterilizzare utilizzando un formato di filtro 0,22 micron pori e conservare a temperatura ambiente (RT).
- Utilizza il seguente Vitamine (1,000x) stock: pantotenato: 1 g / L, acido nicotinico: 10 g / L, inositolo: 10 g / L, biotina: 10 mg / l. Filtro-sterilizzare utilizzando un formato di filtro 0,22 micron pori e conservare a 4 ° C.
- Utilizzare i seguenti oligoelementi (10.000X) stock: Acido borico: 5 g / L, MnSO 4: 4 g / L, ZnSO4 · 7H 2 O: 4 g / L, FeCl 2 · 6H 2 O: 2 g / l , MoO 3: 0,4 g / L, KI: 1 g / L, CuSO 4· 5H 2 O: 0,4 g / L, acido citrico: 10 g / L. Filtro-sterilizzare utilizzando un formato di filtro 0,22 micron pori e conservare a 4 ° C.
- Preparare MSL-N agarosio 2% (usato per fare camere pad agarosio) combinando 10 ml di MSL-N con 0,2 g di agarosio. Melt per ~ 2 minuti ad alta potenza in un forno a microonde fino a quando si scioglie agarosio e aliquote da 0,5 ml in tubi microcentrifuga. Conservare a temperatura ambiente.
- Preparare terreno minimo sporulazione con azoto (MSL + N) da MSL-N aggiungendo (NH 4) 2 SO 4: 1 g / L, Leucina: 0,225 g / L, adenina: 0,225 g / L, Uracil: 0,225 g / L . Filtro-sterilizzare utilizzando un filtro dimensione dei pori di 0,22 micron. Conservare a temperatura ambiente.
Nota: leucina, adenina e uracile vengono aggiunti a questo mezzo per consentire la crescita di ceppi auxotrofi. Ulteriori supplemento di aminoacidi dovrebbe essere incluso se il ceppo utilizzato porta un auxotrofia distinta (per esempio his3Δ). Si prega di notare, inoltre, che il lavoro con ceppi completamente prototroph è consigliato, in quanto solotali ceppi permetteranno alta efficienza di accoppiamento. - Preparare 200 ml VALAP per la sigillatura della camera. In un becher aggiungere pesi uguali di lanolina, vaselina (o altro vaselina), e paraffina. miscela di calore a bassa temperatura e mescolare di tanto in tanto fino a quando completamente mescolato. Aliquota del mix in diversi piccoli capsule di Petri. Conservare a temperatura ambiente.
2. I ceppi Coltura lievito di fissione per esperimenti di accoppiamento (Figura 1).
- Giorno 1, Sera:
Seminare ceppi appena striate da un supporto solido in provette di coltura contenente 3 ml di MSL + N. Utilizzare tubi a fondo piatto di circa 2,5 cm di diametro. Utilizzare altri tubi di coltura o di boccette con volume di cultura adattato. Incubare per una notte (O / N) con agitazione a 25 ° C, 200 giri al minuto. Se si lavora con ceppi heterothallic, inoculare separatamente.- Diluire sospensioni cellulari in mezzi la mattina seguente per garantire che le culture hanno densità ottica misurata a 600 nm (OD 600) di 0,4-0,8 sera.
Nota:OD 600 misurazioni come proxy di concentrazione cellulare di lievito di fissione sono lineari nel range di circa 0,1-1,0. Per il nostro OD spettrofotometro 600 = 0.1 corrisponde a 1,4 x 10 6 cellule per ml. Così, se OD iniziale 600 legge sono al di fuori di questo intervallo campioni devono essere diluiti / concentrato per misurazioni affidabili.
- Diluire sospensioni cellulari in mezzi la mattina seguente per garantire che le culture hanno densità ottica misurata a 600 nm (OD 600) di 0,4-0,8 sera.
- 2 ° giorno, da sera:
Diluire le cellule in 20 ml di MSL + N media per OD 600 = 0,025 in 100 ml flaconi. Incubare ceppi O / N a 30 ° C, 200 giri al minuto.
Nota: colture di cellule wild-type dovrebbe raggiungere OD 600 ~ 0,8 la mattina seguente (15 ore più tardi). Se si lavora con ceppi con tempo più lungo di generazione regolare la diluizione di conseguenza. - 3 ° giorno, Mattina:
Misura OD 600 per verificare che le culture hanno densità cellulare di OD 600 ~ 0.8. Se si lavora con ceppi heterothallic, mescolare un numero uguale di cellule partner. Cellule pellet a 1.000 xg, trasferire in un 1,5 mltubo. Lavare le cellule tre volte in 1 ml di terreno MSL-N. Risospendere le cellule in 3 ml di terreno MSL-N e diluire le cellule ad OD 600 = 1.5.- Per monitorare l'accoppiamento tra le cellule sorelle montare direttamente le cellule per l'imaging (vedi protocollo sezione 2.4). Utilizzare ceppi H90 homothallic, in quale tipo di accoppiamento di commutazione avrà luogo durante le divisioni mitotiche che si verificano dopo deprivazione di azoto. montaggio immediato di celle del pad agarosio assicura che, dopo la divisione cellulare, sorella-cellule rimangono accanto all'altro.
- Per monitorare polarizzazione cellulare (dinamiche esplorative e shmooing) incubare 1-3 ml di coltura cellulare a 30 ° C, 200 rpm per 3-4 ore prima di celle di montaggio per l'imaging. All'interno di questi 3-4 ore, si sono verificati l'ultima divisione mitotica. A poche cellule saranno hanno avviato la polarizzazione, ma la maggior parte saranno solo iniziando la loro dinamica di polarizzazione esplorativi.
- Per monitorare la fusione cellula-cellula incubare 1-3 ml di coltura cellulare a 30 ° C, 200 rpm per 4-6 hr prio alle cellule di montaggio per l'imaging.
- Monitorare eventi post-fusione incubare 1-3 ml di colture cellulari a 30 ° C, 200 rpm per 8 ore prima di celle di montaggio per l'imaging.
- Per monitorare la risposta ad una concentrazione di feromone specifico (solo per ceppi heterothallic) incubare 3 ml di coltura cellulare a 30 ° C, 200 rpm per 3-4 ore prima di celle di montaggio per l'imaging.
- Rimuovere la provetta MSL-N-contenente dal blocco C di calore 95 ° (vedere la sezione Protocollo 2.4.1. Di seguito). Aggiungere P-factor o fattore M alla concentrazione desiderata direttamente in agarosio fuso MSL-N. Mescolare bene nel vortex prima della preparazione immediata pad.
- Dopo il montaggio cella, incubare il pad per 15-30 minuti a 25 ° C prima di imaging. P-factor e fattore M i feromoni sono stati utilizzati da una soluzione stock di 1 mg / ml in metanolo.
Nota: Se si lavora con mutante ceppi tempi di incubazione possono variare. Aggregazione delle cellule può verificarsi dopo incubazione prolungata in m liquidoedia e può essere particolarmente forte in alcuni ceppi mutanti. cellule aggregata non possono essere individuati sul pad agarosio in un unico strato e quindi sono difficili da autofocus e immagine. In caso di forte aggregazione cellulare montare celle pastiglie agarosio dopo lavaggi e risospensione in mezzi azoto privo (come nella sezione protocollo 2.3.1.) E incubare a 30 ° C prima di imaging.
- Le cellule di Imaging di montaggio:
- Preparare MSL-N pastiglie agarosio 16 fondendo una aliquota in un 95 ° C di calore-blocco per 10-15 minuti e aggiungendo 200 microlitri tra due vetrini separati da distanziali (Figura 1B). Utilizzare distanziatori con uno spessore di ~ 0,5 mm. Per rendere distanziatori, utilizzare strisce ritagliate di cartone, come quello fornito con enzimi di restrizione.
- Pellet 100 microlitri di cellule a 1.000 xg per 1 minuto, rimuovere il surnatante e risospendere le cellule in residuo medio 2-4 microlitri. NON risospendere in terreno fresco come ritardaaccoppiamento.
- Dopo 2-3 minuti rimuovere con cautela distanziali e il vetrino superiore. Se diversi ceppi devono essere montati, preparare la cellula si concentra prima di preparare il pad agarosio.
- Aggiungere 1 ml di cellule per il pad e attendere che il calo inizia l'essiccazione (~ 1 min) prima di coprire con il coperchio slip e sigillatura con VALAP.
- Lasciate che il pad riposare per almeno 30 minuti prima di imaging a 25 ° C o RT.
- Per ottenere una miscela di cellule in varie fasi di accoppiamento, fare un tampone subito dopo lavaggi in MSL-N (vedi Sezione 2.3.) E incubare a 18 ° CO / N (~ 15 ore). Questo approccio è utile per l'imaging cellule nelle fasi di accoppiamento distinte con alta risoluzione temporale o campioni con segnale debole.
Imaging 3. Live-cell di accoppiamento cellule di lievito
- Regola immagine Impostazioni di acquisizione.
Nota: le impostazioni di acquisizione di immagini qui presentate sono state ottimizzate per la piattaforma DeltaVision composto da una Olympus IX-71 personalizzatomicroscopio invertito con Plan Apo 60X / 1.42 NA o U-Plan Apo 100X / 1.4 obiettivi NA, una macchina fotografica CoolSnap HQ2 e un Insight SSI 7 colori unità combinata illuminatore. L'hardware è controllato da Softworx V4.1.2, che consente anche di autofocus digitale.- Assicurarsi che gli intervalli di autofocus non superano 15 min dal Z-deriva nel tempo più lungo può superare la capacità del sistema software autofocus. Se si utilizza un sistema di autofocus basato su hardware, utilizzare intervalli più grandi.
- Regolare intervallo di imaging. Prendere immagini ogni 10 min per ~ 15 hr come punto di partenza quando si lavora con un fluorescente contrassegnati proteine per la prima volta. Questo intervallo offre una buona panoramica di tutto il processo di accoppiamento senza vasta sbiancamento.
- Immagine con 5 min intervalli più brevi o più precisamente a eventi temporali come la fusione. intervalli più brevi richiedono fluorofori forti che permettono ridotti tempi di esposizione e poco sbiancamento. Per l'imaging con intervalli inferiori a 1 min evitare di O / N microscopiae invece preparare una miscela di tutte le fasi di accoppiamento come descritto nel protocollo sezione 2.4.6.
- Regolare il tempo di esposizione dell'immagine. tempi di esposizione di prova tra i 50 e 300 msec. Lo scopo di trovare un equilibrio tra il rapporto e photobleaching segnale-rumore da ottenere un elevato numero di punti temporali con un segnale percepibile (tipicamente oltre 100).
- Regolare Z-sezionamento. Sezioni ogni 0,3 micron che coprono 5 micron di fornire una buona profondità di imaging. Si noti che ulteriori aumenti foto-danni Z-sezionamento, che può essere minimizzata utilizzando il OAI (asse integrazione ottica - un'unica acquisizione spazzata di profondità campione totale). Buon sistema di messa a fuoco automatica riduce la necessità di molti Z-sezioni ed è altamente raccomandato.
- Suggerimenti per Studiare comportamenti di accoppiamento Tipo-specifici:
- Per i ceppi heterothallic, usa h- e h + cellule esprimenti fluorofori distinti, in modo che i due tipi di cellule possono essere distinti. Utilizzare qualsiasi fluoropho geneticamente codificatori espresso come una versione citosolica o fusione di una proteina endogena, anche se abbiamo avuto un buon successo con sfGFP, una variante veloce pieghevole di GFP 17. Proteine endogene di solito sono contrassegnati alla loro locus genomico endogeno per ricombinazione omologa 14.
- Per i ceppi homothallic, esprimere una proteina fluorescente citosolica geneticamente codificato sotto il controllo di promotori specifici tipo di accoppiamento come quelle dei geni dei recettori feromone map3 e MAM2 per guidare l'espressione in cellule P e M rispettivamente. I promotori di map3 e MAM2 sono inattivi durante la crescita vegetativa e sono indotte su di azoto fame 18,19. Costrutti guida l'espressione di GFP citosolico o mCherry sotto il controllo di tali promotori sono generalmente integrati in un locus genomico (ura4, leu1) che non interferisce con la normale progressione del ciclo sessuale 12.
- Usa tipo di accoppiamento specifici fluorofori citosoliche (come nel protocollo sezione 3.2.2) per determinare una tempistica precisa di fusione cellulare visualizzato come il trasferimento del segnale fluorescente da una cellula socio all'altro.
4. Quantificazione di accoppiamento e Fusion efficienze
- Per quantificare l'accoppiamento e la fusione efficienze 11,12, le cellule del punto subito dopo MSL-N lavare su MSL-N pad agarosio, come al punto 2.3.1, e lasciare per 24 ore a 25 ° C prima di imaging (Figura 1A). Usando questo protocollo una homothallic wild-type efficienza ceppo di accoppiamento raggiunge ~ 60% e l'efficienza di fusione del 99%. Per verificare se qualsiasi riduzione di questi valori osservati nei ceppi mutanti riflettono un fenotipo terminale o un ritardo, ripetere l'esperimento con un tempo di incubazione 36 ore.
- Calcolare l'efficienza di accoppiamento come
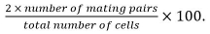 coppie di accoppiamento includono zigoti, ASCI, e le coppie di cellule non fuse identificati dala loro posizione e crescita verso ogni altri.
coppie di accoppiamento includono zigoti, ASCI, e le coppie di cellule non fuse identificati dala loro posizione e crescita verso ogni altri. - Calcolare l'efficienza fusione come
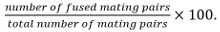
Nota: accoppiamenti accoppiamento fuso sono identificati dalla loro capacità di formare spore se è noto che il mutante in esame non influenza sporulazione. Se sporulazione non può essere utilizzato come read-out, la fusione è determinata osservando la diffusione di un marcatore citosolico espressa in un solo partner di accoppiamento in entrambi i partner.
Risultati
Lievito di fissione la crescita e l'accoppiamento Dynamics dopo la rimozione di una fonte di azoto
Come fame di azoto è un prerequisito per l'inizio della riproduzione sessuale in lievito di fissione, il ceppo selvatico homothallic H90 è stato monitorato su passaggio da medio di azoto-privato ricco di azoto (figura 2), seguendo il protocollo descritto nella Figura 1. In breve, le cellule sono state coltivate O / N a fase esponen...
Discussione
Condizioni ambientali, e la disponibilità di nutrienti, in particolare, influenzano pesantemente la fisiologia fissione lievito. L'azoto fame è necessario per l'impegno per la riproduzione sessuale e porta a notevoli cambiamenti nella progressione del ciclo cellulare mitotico (Rif. 21 e Figura 2) inizialmente. Alla rimozione azoto popolazione in crescita esponenziale, dimensione delle celle a divisione diminuisce rapidamente (Figura 2C) e la maggior parte delle cell...
Divulgazioni
The authors declare that they have no competing financial interests.
Riconoscimenti
AV è stato sostenuto da un lungo periodo borsa di studio postdottorato EMBO. La ricerca in laboratorio Martin è finanziato da una sovvenzione del CER di partenza (GeometryCellCycle) e una borsa di studio Science Foundation nazionale svizzero (31003A_155944) per SGM.
Materiali
| Name | Company | Catalog Number | Comments |
| Glucose | Sigma-Aldrich | G8270-10KG | |
| KH2PO4 | Sigma-Aldrich | 1.05108.0050 | |
| NaCl | Sigma-Aldrich | 71381 | |
| MgSO4•7H2O | Sigma-Aldrich | 63140 | |
| CaCl2 | Sigma-Aldrich | 12095 | |
| Pantothenate | AppliChem | A2088,0025 | |
| Nicotinic Acid | AppliChem | A0963,0100 | |
| Inositol | AppliChem | A1716,0100 | |
| Biotin | AppliChem | A0967,0250 | |
| Boric Acid | Sigma-Aldrich | B6768-1KG | |
| MnSO4 | AppliChem | A1038,0250 | |
| ZnSO4•7H2O | Sigma-Aldrich | Z4750 | |
| FeCl2•6H2O | AppliChem | A3514,0250 | |
| Molybdenum oxide (VI) (MoO3) | Sigma-Aldrich | 69850 | |
| KI | AppliChem | A3872,0100 | |
| CuSO4•5H2O | AppliChem | A1034,0500 | |
| Citric Acid | AppliChem | A2344,0500 | |
| Agarose | Promega | V3125 | |
| (NH4)2SO4 | Merck | 1.01217.1000 | |
| L-Leucine | Sigma-Aldrich | L8000-100G | |
| Adenine Hemisulfat Salt, mini 99% | Sigma-Aldrich | A9126-100G | |
| Uracil | Sigma-Aldrich | U0750 | |
| Lanolin | Sigma-Aldrich | L7387 | |
| Vaseline | Reactolab | 92045-74-4 | |
| Paraffin | Reactolab | 7005600 |
Riferimenti
- Mata, J., Bahler, J. Global roles of Ste11p, cell type, and pheromone in the control of gene expression during early sexual differentiation in fission yeast. Proc Natl Acad Sci U S A. 103 (42), 15517-15522 (2006).
- Xue-Franzen, Y., Kjaerulff, S., Holmberg, C., Wright, A., Nielsen, O. Genomewide identification of pheromone-targeted transcription in fission yeast. BMC Genomics. 7, 303 (2006).
- Klar, A. J. Lessons learned from studies of fission yeast mating-type switching and silencing. Annu Rev Genet. 41, 213-236 (2007).
- Beach, D. H., Klar, A. J. Rearrangements of the transposable mating-type cassettes of fission yeast. EMBO J. 3 (3), 603-610 (1984).
- Merlini, L., Dudin, O., Martin, S. G. Mate and fuse: how yeast cells do it. Open Biol. 3 (3), 130008 (2013).
- Mochizuki, N., Yamamoto, M. Reduction in the intracellular cAMP level triggers initiation of sexual development in fission yeast. Mol Gen Genet. 233 (1-2), 17-24 (1992).
- Sugimoto, A., Iino, Y., Maeda, T., Watanabe, Y., Yamamoto, M. Schizosaccharomyces pombe ste11+ encodes a transcription factor with an HMG motif that is a critical regulator of sexual development. Genes Dev. 5 (11), 1990-1999 (1991).
- Kjaerulff, S., Lautrup-Larsen, I., Truelsen, S., Pedersen, M., Nielsen, O. Constitutive activation of the fission yeast pheromone-responsive pathway induces ectopic meiosis and reveals ste11 as a mitogen-activated protein kinase target. Mol Cell Biol. 25 (5), 2045-2059 (2005).
- Obara, T., Nakafuku, M., Yamamoto, M., Kaziro, Y. Isolation and characterization of a gene encoding a G-protein alpha subunit from Schizosaccharomyces pombe: involvement in mating and sporulation pathways. Proc Natl Acad Sci U S A. 88 (13), 5877-5881 (1991).
- Toda, T., Shimanuki, M., Yanagida, M. Fission yeast genes that confer resistance to staurosporine encode an AP-1-like transcription factor and a protein kinase related to the mammalian ERK1/MAP2 and budding yeast FUS3 and KSS1 kinases. Genes Dev. 5 (1), 60-73 (1991).
- Bendezu, F. O., Martin, S. G. Cdc42 explores the cell periphery for mate selection in fission yeast. Curr Biol. 23 (1), 42-47 (2013).
- Dudin, O., et al. A formin-nucleated actin aster concentrates cell wall hydrolases for cell fusion in fission yeast. J Cell Biol. 208 (7), 897-911 (2015).
- Chikashige, Y., et al. Telomere-led premeiotic chromosome movement in fission yeast. Science. 264 (5156), 270-273 (1994).
- Bahler, J., et al. Heterologous modules for efficient and versatile PCR-based gene targeting in Schizosaccharomyces pombe. Yeast. 14 (10), 943-951 (1998).
- Egel, R., Willer, M., Kjaerulff, S., Davey, J., Nielsen, O. Assessment of pheromone production and response in fission yeast by a halo test of induced sporulation. Yeast. 10 (10), 1347-1354 (1994).
- Tran, P. T., Marsh, L., Doye, V., Inoue, S., Chang, F. A mechanism for nuclear positioning in fission yeast based on microtubule pushing. J Cell Biol. 153 (2), 397-411 (2001).
- Pedelacq, J. D., Cabantous, S., Tran, T., Terwilliger, T. C., Waldo, G. S. Engineering and characterization of a superfolder green fluorescent protein. Nat Biotechnol. 24 (1), 79-88 (2006).
- Kitamura, K., Shimoda, C. The Schizosaccharomyces pombe mam2 gene encodes a putative pheromone receptor which has a significant homology with the Saccharomyces cerevisiae Ste2 protein. EMBO J. 10 (12), 3743-3751 (1991).
- Tanaka, K., Davey, J., Imai, Y., Yamamoto, M. Schizosaccharomyces pombe map3+ encodes the putative M-factor receptor. Mol Cell Biol. 13 (1), 80-88 (1993).
- Motegi, F., Arai, R., Mabuchi, I. Identification of two type V myosins in fission yeast, one of which functions in polarized cell growth and moves rapidly in the cell. Mol Biol Cell. 12 (5), 1367-1380 (2001).
- Sajiki, K., Pluskal, T., Shimanuki, M., Yanagida, M. Metabolomic analysis of fission yeast at the onset of nitrogen starvation. Metabolites. 3 (4), 1118-1129 (2013).
- Miyawaki, A., Nagai, T., Mizuno, H. Mechanisms of protein fluorophore formation and engineering. Curr Opin Chem Biol. 7 (5), 557-562 (2003).
- Dyer, J. M., et al. Tracking shallow chemical gradients by actin-driven wandering of the polarization site. Curr Biol. 23 (1), 32-41 (2013).
- Beta, C., Bodenschatz, E. Microfluidic tools for quantitative studies of eukaryotic chemotaxis. Eur J Cell Biol. 90 (10), 811-816 (2011).
- Lee, S. S., et al. Quantitative and dynamic assay of single cell chemotaxis. Integr Biol (Camb). 4 (4), 381-390 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon