Method Article
Generazione in Vitro di Somite derivati da umani indotta da cellule staminali pluripotenti indotte
In questo articolo
Riepilogo
Presentiamo qui un protocollo per la differenziazione delle cellule staminali umane pluripotenti indotte in ogni derivato di somite (Miotomo, sclerotomo, dermatome e syndetome) in condizioni chimicamente definite, che ha applicazioni nella modellazione di malattia futura e terapie basate su cellule in chirurgia ortopedica.
Abstract
In risposta a segnali quali WNT, bone morphogenetic proteins (BMPs) e sonic hedgehog (SHH) secernuto dai tessuti circostanti, somiti (SMs) danno luogo a più tipi di cellule, tra cui il miotomo (MYO), sclerotomo (SCL), dermatome (D) e syndetome (SYN) , che a sua volta trasformarsi in muscolo scheletrico, scheletro assiale, derma dorsale e assiale del tendine/legamento, rispettivamente. Pertanto, la generazione di SMs e loro derivati da cellule staminali umane pluripotenti indotte (iPSCs) è fondamentale per ottenere cellule staminali pluripotenti (PSC) per applicazioni in medicina rigenerativa e per la ricerca di malattia nel campo della chirurgia ortopedica. Anche se i protocolli di induzione per MYO e SCL da PSC precedentemente sono stati segnalati da diversi ricercatori, nessuno studio ha ancora dimostrato l'induzione di SYN e D da iPSCs. Di conseguenza, induzione efficiente di SMs completamente competente rimane una sfida importante. Qui, abbiamo ricapitolare umano patterning di SM con iPSCs umane in vitro imitando l'ambiente segnalazione durante lo sviluppo di SM pulcino/mouse e relazione sui metodi di induzione sistematica dei derivati di SM (MYO, SCL, D e SYN) da iPSCs umane sotto chimicamente condizioni definite attraverso il mesoderm presomitic (PSM) e Stati di SM. Conoscenza per quanto riguarda lo sviluppo del pulcino/mouse SM è stato applicato con successo all'induzione di SMs con iPSCs umane. Questo metodo potrebbe essere un nuovo strumento per studiare umano somitogenesis e patterning senza l'uso di embrioni e per la modellazione di malattia e di terapia cellulare.
Introduzione
Lo sviluppo di un metodo di differenziazione diretto per un tipo di cella desiderata da sportelli unici è un passo necessario per tradurre lo studio di cellule derivanti dal PSC in applicazioni cliniche. Sovraespressione di geni chiave è una strategia promettente per organo-differenziamento da PSC e ha migliorato la nostra comprensione della regolazione genetica della determinazione del destino cellulare, la morfogenesi organo e organizzazione durante l'embriogenesi1. Inoltre, ricapitolare gli ambienti segnalazione endogeni, mediante lo sviluppo di embrioni di topo e pulcino come una tabella di marcia, è considerato essenziale per la differenziazione diretto degli sportelli unici. Tuttavia, la strategia di quest'ultima dato l'applicazione di cellule derivanti dal PSC in studi clinici come terapie basate sulle cellule, è più adatta perché non richiede la manipolazione genica.
Parecchi studi hanno riferito l'induzione del mesoderma dall'essere umano e del mouse PSC in condizioni definite chimicamente. In genere, questi metodi hanno invocato activin/nodale/trasformando la segnalazione di β (TGFβ) fattore di crescita e ossea morfogenetica (BMP) di segnalazione, creduto per eseguire la differenziazione meso-endoderma e mesoderma, conseguente a un'efficienza bassa induzione del il parassiale mesoderma (20% circa)2. In altre parole, il mesoderma PSC-derivato indotto da queste vie di segnalazione è stato principalmente mesoderma della piastra laterale e non il mesoderma parassiale. Recentemente, alcuni studi hanno dimostrato la produzione efficiente di mesoderma parassiale PSC-derivato basato su diverse strategie3,4,5,6,7,8 . In questi studi, gli sportelli unici sono stati coltivati con concentrazioni relativamente alte di chinasi 3 (GSK3) di glicogeno sintasi inibitori (attivatori di segnalazione WNT), di conseguenza l'efficienza di induzione del mesoderma parassiale raggiunto 70% – 95%6,7 .
In somitogenesis, il mesoderma parassiale prima forma mesoderm presomitic (PSM) posteriormente e quindi costituisce somiti (SMs) nella parte anteriore attraverso transizione mesenchima--epiteliale9,10. Ligando tacca del Delta-tipo 1 (DLL1) è noto per avere un ruolo fondamentale durante la somitogenesis, come controllo oscillatoria dell'espressione DLL1, sia a livello di mRNA e proteine, regola la segmentazione SM. SMs alla fine suddividere in due parti, dando luogo a dermomyotome (DM), dorsalmente e sclerotomo (SCL) ventralmente11. Successivamente, il DM si differenzia in dermatome (D), un precursore del derma e Miotomo (MYO), un precursore del muscolo scheletrico; Inoltre, una porzione ventrale del SCL forma syndetome (SYN), un precursore di tendini e legamenti12 (Figura 1). Alcuni ricercatori hanno segnalato l'induzione di derivati di PSC-derivato SM come MYO4,13 e SCL14; Tuttavia, ci sono diverse limitazioni in questi studi. In particolare, dal momento che la nostra conoscenza degli ambienti segnalazione di D e SYN è frammentario, protocolli di induzione per D e SYN non sono ancora state sistematicamente stabilite. Per dimostrare la competenza completa di SMs indotta da sportelli unici, è essenziale per mostrare la multi-differenziazione capacità di SMs indotta in tutti e quattro i derivati (D, MYO, SCL e SYN), mentre gli studi precedenti si sono concentrati solo su specifici derivati di SM. Qui, segnaliamo su come generare tutti i quattro derivati di SM, anche D e SYN, attraverso destini PSM e SM da umano iPSCs15. Siamo convinti che stabilire un metodo graduale in vitro che modella il processo di sviluppo di SM potrebbe contribuire allo studio della SM come umano si sviluppa durante l'embriogenesi, senza l'utilizzo di embrioni.
Protocollo
Tutti i protocolli sperimentali che coinvolgono iPSCs umane sono stati approvati dal comitato etico del dipartimento di medicina e Graduate School of Medicine, Università di Kyoto.
1. umani iPSCs preparazione prima di induzione
Nota: Cultura umana iPSCs (201B7-PAX3-GFP) sull'alimentatore SNL cellule16 con medium cellulare primate ES completati con 4 ng/mL di fattore di crescita fibroblastico ricombinante umano base (FGF2) e 0,5%, penicillina e streptomicina (in seguito denominato come mezzo di hESC, vedere Tabella 1). Quando il rapporto di confluenza raggiunge 70% – 80%, passaggio le celle come descritto in precedenza17.
-
Passaggio di umano iPSCs su SNL alimentatore cellule17
- Per passaggio, aggiungere PBS nella piastra di coltura delle cellule e sciacquare le cellule. Quindi, rimuovere il PBS (, questo processo sarà essere appresso come lavaggio con PBS).
- Aggiungere 1 mL di soluzione CTK (Vedi tabella 1) a temperatura ambiente (RT) e attendere che le cellule di alimentatore SNL iniziare lo scollegamento dalla parte inferiore del piatto.
- Rimuovere la soluzione CTK e poi lavare due volte con PBS.
- Aggiungere 1 mL di medium hESC (Vedi tabella 1) nel piatto, poi raschiare le celle utilizzando un raschietto e raccogliere in una provetta conica da 15 mL.
- Pipettare delicatamente il contenuto cinque volte usando una punta di 1.000 µ l e poi il trasferimento a un nuovo piatto riempito con il mezzo di hESC. Utilizzare un rapporto di divisione di 1:4 a 01:10, a seconda della percentuale di confluenza prima di passaggio. Inoltre, variare il volume del mezzo hESC a seconda dell'entità del piatto (ad esempio, 3 mL per un piatto di cm 6, 8ml per un piatto di 10 cm).
- Incubare le iPSCs umane a 37 ° C e 5% CO2.
- Cambiare il medio tutti i giorni (tranne il giorno dopo passaggio) e le cellule della coltura fino a quando la prossima procedura passaging.
-
Senza alimentatore coltura delle iPSCs umane prima di induzione di PSM
Nota: per ridurre al minimo l'effetto di fattori di crescita secernuta dalle cellule di alimentatore SNL, cultura il iPSCs umane nelle circostanze senza alimentatore con un mezzo di coltura privo di alimentatore cellulare (Vedi tabella 1) su soluzioni di matrice extracellulare (ECM) (Vedi tabella 1) piatto rivestito per 3 giorni prima di induzione di PSM.- Giorno -4 (4 giorni prima di iniziare ad induzione PSM)
- Per preparare il piatto di rivestite con soluzione di ECM, aggiungere 4 mL di soluzione di ECM su un piatto di 10 cm a 4 ° C durante la notte.
Nota: Mettere la soluzione ECM sul ghiaccio durante la preparazione.
- Per preparare il piatto di rivestite con soluzione di ECM, aggiungere 4 mL di soluzione di ECM su un piatto di 10 cm a 4 ° C durante la notte.
- Giorno -3
- Innanzitutto, rimuovere la soluzione ECM dal piatto e aggiungere 8 mL di mezzo di coltura cellulare privo di alimentatore.
- Per avviare l'alimentatore senza coltura, lavare una volta con PBS per lavare le cellule coltivate.
- Aggiungere 1 mL di soluzione CTK presso RT fino a quando le cellule di alimentatore SNL iniziano lo scollegamento dalla parte inferiore del piatto.
Nota: Utilizzare la microscopia per confermare che tutte le cellule di alimentatore sono staccate dal fondo. - Rimuovere la soluzione CTK e lavati con PBS due volte, affinché tutte le cellule di alimentatore SNL vengono completamente rimossi.
- Aggiungere 1 mL di mezzo di coltura privo di alimentatore cellulare nel piatto, poi raschiare le celle utilizzando un raschietto e raccoglierle in una provetta conica da 15 mL.
- Delicatamente Pipettare il contenuto tre volte usando una punta di 1.000 µ l, poi il trasferimento a un nuovo piatto di 10cm rivestite con soluzione di ECM (preparato durante passaggio 1.2.1). Utilizzare un rapporto di divisione di circa 1:2 a 1:4, a seconda della percentuale di confluenza prima coltura privo di alimentatore.
- Incubare le iPSCs umane a 37 ° C e 5% CO2 per 3 giorni, cambiando il mezzo giorno -1.
- Giorno -4 (4 giorni prima di iniziare ad induzione PSM)
2. PSM differenziazione e isolamento di fluorescenza-attivato delle cellule ordinano (FACS)
-
Differenziazione di PSM (giorno 0 – giorno 4)
- Aspirare il terreno di coltura cellulare privo di alimentatore e aggiungere 8 mL di mezzo di induzione di PSM (medium basale CDM supplementato con 10 µM SB431542, µM 10 CHIR99021, 2 µM DMH1 e 20 ng/mL FGF2, Vedi tabella 1).
Nota: Confluenza di cella all'inizio della differenziazione di PSM è fondamentale per l'efficienza di induzione. Utilizzare la microscopia per confermare che il rapporto confluente è circa il 30%. - Incubare le cellule a 37 ° C, con 5% di CO2, per 4 giorni, cambiando il mezzo il giorno 3.
- Raccogliere le cellule per FACS il giorno 4 (sezione 2.2, sotto).
- Aspirare il terreno di coltura cellulare privo di alimentatore e aggiungere 8 mL di mezzo di induzione di PSM (medium basale CDM supplementato con 10 µM SB431542, µM 10 CHIR99021, 2 µM DMH1 e 20 ng/mL FGF2, Vedi tabella 1).
-
Isolamento di cellule positive di PSM DLL1 di fluorescenza-attivato delle cellule ordinano (FACS)
Nota: qui di seguito è una procedura per la preparazione delle cellule prima FACS l'ordinamento delle cellule positive DLL1. Eseguire un FACS ordinamento utilizzando un citometro a flusso, secondo il protocollo del produttore.- Aspirare il mezzo, quindi lavare con PBS. Successivamente, aggiungere 1 mL di reagente di dissociazione delle cellule e lasciare per 3 minuti a TA.
- Aggiungere 4 mL di sostanza basale CDM, raschiare le celle utilizzando un raschietto e raccoglierle in una provetta conica da 15 mL.
- Contare il numero di celle utilizzando un contatore di cellule automatizzato, quindi centrifugare a 280 x g per 3 min.
- Attentamente l'aspirazione per rimuovere il supernatante e risospendere le cellule in buffer di FACS (Vedi tabella 1) ad una concentrazione di 1,0 x 107 cellule/mL. Per un campione di controllo negativo (controllo di isotipo, o in convenzione senza anticorpo), trasferire 50 µ l in una provetta conica da 15 mL e quindi sospendere con 450 µ l di tampone di FACS.
- Aggiungere DLL1 anticorpo (Vedi Tabella materiali) in un rapporto di 1/200. Proteggere il tubo da luce e tenere il ghiaccio per 30 min.
- Centrifuga a 280 x g per 3 min.
- Attentamente, aspirare il supernatante e risospendere in tampone di FACS (1.0 x 107 cellule/mL) completati con 1 mg/mL DAPI.
- Trasferimento in una provetta di raccolta, incorporata con una maglia di nylon 35 µm nel tappo per il filtraggio, quindi posizionare il tubo sul ghiaccio fino a quando l'ordinamento è completo. Eseguire la stessa procedura con il campione di controllo negativo (passo 2.2.4).
- Eseguire l'ordinamento utilizzando un citometro a flusso secondo il protocollo del produttore.
- Raccogliere le cellule positive DLL1 ordinate in una provetta conica da 15 mL, contenente 4 mL di medium basale CDM supplementato con 10 µM di Y27632. Per estrazione di RNA totale, centrifugare a 280 x g per 3 minuti poi risospendere in buffer di lisi di RNA e conservare a-30 ° C. Vedere l'estrazione del RNA, retrotrascrizione e RT-qPCR procedure (vedere paragrafo 5.1) per informazioni più dettagliate.
- Eseguire la differenziazione di SM usando le cellule ordinate secondo la sotto protocollo (sezione 3).
3. SM differenziazione da PSM
-
Differenziazione di SM da cellule positive PSM DLL1 ordinate (giorno 4 – giorno 8)
Nota: preparare le piastre 12-pozzetti rivestite con soluzione di ECM il giorno prima dell'ordinamento FACS. Per preparare un piatto 12-pozzetti rivestite con soluzione di ECM, aggiungere 1 mL di soluzione di ECM in ogni pozzetto a 4 ° C e lasciare durante la notte. Mantenere la soluzione ECM sul ghiaccio durante la preparazione.- Seguendo passo 2.2.10, centrifugare a 280 x g per 3 min.
- Attentamente aspirare il supernatante e risospendere in 1 mL di mezzo di induzione di SM (medium basale CDM supplementato con 10 µM SB431542 e 5 µM CHIR99021, Vedi tabella 1).
- Contare il numero di celle utilizzando un contatore di cellule automatizzato.
- Cellule di seme 1.0 x 105 su ciascun pozzetto delle contenente 1 mL di medium di induzione SM supplementato con 10 µM di Y27632 piastre 12-pozzetti rivestite con soluzione ECM.
- Incubare a 37 ° C con 5% CO2 per 4 giorni fino al giorno 8. Modificare il mezzo non contenenti Y27632 il giorno 5 (il giorno dopo l'ordinamento FACS) e giorno 7.
- Eseguire la differenziazione di derivati SM utilizzando cellule indotte SM secondo i seguenti protocolli. Per estrazione di RNA totale da cellule indotte SM, raccogliere le cellule in una provetta conica da 15 mL e centrifugare a 280 x g per 3 min, poi risospendere in buffer di lisi di RNA e conservare a-30 ° C.
4. SM derivati (DM, MYO, D, SCL, SYN) differenziazione da SM
Nota: Per dimostrare la competenza completa delle cellule di SM, in primo luogo eseguire DM (dermomyotome) e SCL (sclerotomo) induzione di conseguenza utilizzando cellule di SM iPSC-derivate. Successivamente, eseguire MYO (Miotomo) e D (dermatome) induzione usando le cellule di DM e condurre ad induzione di SYN (syndetome) usando le cellule SCL. Di seguito sono i protocolli per l'induzione di ogni derivato (DM, MYO, D, SCL e SYN) da cellule SM indotte in vitro.
-
DM differenziazione dalle cellule SM (giorno 8 – giorno 11)
- Aspirare il mezzo, quindi aggiungere 1 mL di mezzo di induzione di DM (medium basale CDM completate con 5 µM CHIR99021 e 10 ng/mL BMP4, Vedi tabella 1).
- Incubare le cellule a 37 ° C, con 5% di CO2, per 3 giorni fino al giorno 11. Modificare il mezzo il giorno 10 (giorno 2 del DM induzione).
- Eseguire MYO e D differenziazione utilizzo hanno indotto le cellule DM secondo i seguenti protocolli.
-
Differenziazione di MYO dalle cellule DM (giorno 11 – giorno 41)
- Aspirare il mezzo, quindi aggiungere 1 mL di mezzo di induzione di MYO (medium basale CDM completate con 5 µM CHIR99021, Vedi tabella 1).
- Incubare le cellule a 37 ° C, con 5% di CO2, per 30 giorni fino al giorno 41. Modificare il mezzo ogni 3 giorni.
-
Differenziazione dalle cellule DM (giorno 11 – giorno 20) D
- Aspirare il mezzo, quindi aggiungere 1 mL di mezzo di induzione D (medium basale CDM completate con 5 µM CHIR99021 e 10 ng/mL BMP4, Vedi tabella 1).
- Incubare le cellule a 37 ° C, con 5% di CO2, per 9 giorni fino al giorno 20. Modificare il mezzo ogni 3 giorni.
-
Differenziazione di SCL dalle cellule SM (giorno 8 – giorno 11)
- Aspirare il mezzo, quindi aggiungere 1 mL di mezzo di induzione di SCL (medium basale CDM completate con 100 nM SAG e 0,6 µM LDN193189, Vedi tabella 1)14.
- Incubare le cellule a 37 ° C, con 5% di CO2, per 3 giorni. Modificare il mezzo il giorno 10 (induzione di giorno 2 di SCL).
- Eseguire la differenziazione di SYN utilizzando cellule indotte SCL secondo il protocollo sottostante.
-
Differenziazione di SYN da cellule SCL (giorno giorno 11 – 32)
Nota: preparare le piastre 24 pozzetti rivestite con soluzione di ECM il giorno prima dell'inizio di induzione di SYN. Per preparare un piatto 24 pozzetti rivestite con soluzione di ECM, aggiungere 0,5 mL di soluzione di ECM in ogni pozzetto a 4 ° C e lasciare durante la notte. Mantenere la soluzione ECM sul ghiaccio durante la preparazione.- Aspirare il mezzo e poi lavare con PBS, poi aggiungere 0,2 mL di reagente di dissociazione delle cellule in ciascun pozzetto e lasciare per 3 minuti a TA.
- Aggiungere 0,8 mL di sostanza basale CDM in ciascun pozzetto quindi raschiare e raccogliere tutte le cellule in una provetta conica da 15 mL.
- Centrifuga a 280 x g per 3 min.
- Attentamente aspirare il supernatante e risospendere in 1 mL di SYN induzione media-1 (media basale CDM addizionata di 20 ng/mL FGF8, Vedi tabella 1), poi contare il numero di celle utilizzando un contatore di cellule automatizzato.
- 4 cellule di seme 5,0 x 10 in tutti i pozzetti delle contenente 1 mL di SYN induzione media-1 piastre 24 pozzetti rivestite con soluzione ECM.
- Incubare a 37 ° C, con 5% di CO2, per 3 giorni.
- Il giorno 14 (induzione di giorno 3 di SYN), sostituire il mezzo con SYN induzione medio-2 (medium basale CDM supplementato con 10 ng/mL BMP7 e 10 ng/mL TGFβ3, Vedi tabella 1).
- Incubare a 37 ° C, con 5% di CO2, per 18 giorni fino al giorno 32. Modificare il mezzo ogni 3 giorni.
5. caratterizzazione dei derivati dell'iPSC
Nota: Differenziamento, caratterizzano iPSCs umana derivati mediante PCR quantitativa in tempo reale (RT-qPCR), immunocitochimica (ICC), analisi enzima-collegate dell'immunosorbente (ELISA) e le analisi di stimolazione meccanica elasticizzato, di conseguenza.
-
Colture di cellule, estrazione di RNA totale, trascrizione d'inversione e analisi quantitativa in tempo reale di PCR (RT-qPCR)
- Raccogliere i campioni di cellule (procedure 2.2.10, 3.1.6, 5.4.3) in una provetta da 1,5 mL, poi Centrifugare a 280 x g per 3 min.
- Eliminare il surnatante, quindi risospendere in 350 µ l di tampone di lisi di RNA, fornito da un apposito kit di estrazione di RNA totale.
- Estrarre RNA totale usando il kit secondo il protocollo del produttore.
- Retromarcia trascrivere l'isolato 1 µ g di RNA totale di cDNA, secondo il protocollo del produttore.
- Eseguire RT-qPCR utilizzando enzimi adatti, reagenti e primer secondo il protocollo del produttore. Le sequenze dell'iniettore utilizzate in questo studio sono riportate nella tabella 2.
-
Immunocitochimica (ICC)
- Prima di eseguire immunocitochimica con anticorpi, fissare le cellule con 2% paraformaldeide al 4 ° C per 10 min e lavare due volte con PBS.
- Per la permeabilizzazione, incubare con metanolo 0,2% o 0,2% polisorbato 20/PBS (in appresso denominato "PBS-T") a 4 ° C per 15 min.
- Rimuovere i reagenti di permeabilizzazione e trattare le cellule con un adeguato blocco buffer o 1% bovina siero albumina/PBS a 4 ° C per 60 min.
- Aggiungere il primo anticorpo diluito con il tampone bloccante 10% in PBS-T e posto su una macchina d'agitazione a 4 ° C durante la notte.
- Lavare tre volte con PBS-T (aggiungere PBS-T e appoggiare sulla macchina d'agitazione a temperatura ambiente per 10 min).
- Aggiungere il secondo anticorpo, diluito con il tampone bloccante 10% in PBS-T e posto sulla macchina agitazione a temperatura ambiente per 60 min. Gli anticorpi primi e la secondo per ICC utilizzato in questo studio sono riportati nella tabella 3.
Nota: Da questo punto in poi, è necessario proteggere il piatto/piatto dalla luce avvolgendolo in un foglio. - Lavare due volte con PBS-T.
- Per la colorazione del contatore, aggiungere 1/5000 DAPI diluito con PBS e posto sulla macchina agitazione a temperatura ambiente per 5 min.
- Rimuovere la soluzione DAPI e aggiungere PBS in ciascun pozzetto.
- Osservare la colorazione delle cellule usando un microscopio a fluorescenza. In alternativa, è possibile memorizzare il piatto/piatto a 4 ° C per 1 mese.
-
Enzima-collegata dell'immunosorbente (ELISA) per l'analisi funzionale di iPSC-derivato D
Nota: cellule di fibroblasti cutanei umani (HDF) sono disponibili in commercio. Cultura di HDF in DMEM completati con 10% siero bovino fetale (Vedi tabella 1).- Le cellule di seme 1.0 x 105 di iPSC-derivato D e HDF sui 24 pozzetti contenente 1 mL di ogni mezzo di coltura (mezzo di induzione: d: D, HDF: DMEM completati con 10% siero bovino fetale).
- Dopo 3 giorni di coltura cellulare, raccogliere 100 µ l di ciascun mezzo, posto in una provetta da 1,5 mL e conservare a 4 ° C.
- Eseguire la serie di procedure, come l'aggiunta di rilevamento anticorpi e anticorpi secondari, secondo le istruzioni del fabbricante e quantificare il numero di bersagli generando una curva standard contro la concentrazione del controllo campioni di analita.
-
Analisi di stimolazione stretch meccanico per l'analisi funzionale di SYN iPSC-derivato
Nota: tenociti umani adulti sono commercialmente disponibili (Vedi Tabella materiali). Cultura umana iPSC-derivato SYN e tenociti adulto umano su una cella che si estende di dispositivo per il test di stimolazione meccanica elasticizzato come descritto di seguito18,19. Terreno uso SYN induzione medio-2 e tenociti crescita (Vedi Tabella materiali) come mezzo di coltura per ogni derivato iPSC SYN e tenociti umano adulto, rispettivamente.- A 24 h prima dell'allungamento, piastra 1.0 x 105 cellule di SYN iPSC-derivato e tenociti umano sulla gomma di pozzo-tipo silicone multi rivestite con soluzione ECM chambers, ciascuno con una superficie di cultura di 1,5 x 1,5 cm (Vedi Tabella materiali).
- Impostare gli alloggiamenti sul dispositivo per l'allungamento delle cellule e forza deformazione ciclica monoassiale (0,5 Hz, 5%) per 12 h.
- Per estrazione di RNA totale, 350 µ l di tampone di lisi del RNA, poi raschiare e raccogliere le cellule in una provetta da 1,5 mL per estrazione di RNA totale e la successiva analisi di RT-qPCR (vedi procedura 5.1).
Risultati
Tutte le figure in questo rapporto sono state ottenute con 201B7-PAX3-GFP iPSCs, in cui EGFP sostituisce un allele della sequenza di codificazione PAX3 in essone 1. Istituzione di 201B7-PAX3-GFP iPSCs sarà descritto altrove (H. Sakurai, comunicazione personale). La significatività statistica è stata valutata utilizzando il software statistico. Valori di P inferiori a 0,05 sono stati considerati significativi.
Caratterizzazione di cellule umane derivate iPSC PSM e SM
Per valutare la differenziazione delle iPSCs umane verso SM attraverso lo stato PSM (Figura 2A), analisi di FACS, sono state eseguite analisi di ICC e l'analisi di RT-qPCR. Come illustrato nella Figura 2B, oltre l'85% delle cellule erano positive per DLL1, un marker di PSM, ma la negazione per PAX3, un indicatore di SM, dopo 4 giorni di induzione di PSM con iPSCs umane. Successivamente, questa popolazione è diventato PAX3 cellule positive di SM dopo 4 giorni di induzione di SM. La transizione di PSM-SM è stata confermata anche da ICC (Figura 2) e RT-qPCR (Figura 2D). TBX6, MSGN1 e WNT3A, marcatori PSM erano espressa presso lo stato PSM (giorno 4), ma non espressa presso lo stato di SM (giorno 8). PARAXIS, MEOX1 e PAX3, marcatori di SM, erano espressi al SM, ma non espresse a PSM. Inoltre, solo la macchiatura di CDH11, un indicatore di SM epithelialized, accumulato allo svincolo cellula-cellula, dopo l'aggiunta di SB431542 con CHIR99021 (Figura 2E).
Caratterizzazione dei derivati di SM indotta da cellule SM iPSC-derivate essere umano
Per valutare la potenza di differenziazione di SM iPSC-derivati umani, differenziazione verso DM, MYO, D, SCL e SYN (Figura 3A) è stata valutata tramite analisi di ICC e PAX3 (GFP)-fluorescenza. Come illustrato in Figura 3B, DM differenziazione è stata confermata da ALX4 ed EN1 macchiatura e PAX3 (GFP)-fluorescenza; Differenziazione di MYO è stata confermata dalla catena pesante MYOD, MYOG e della miosina (MHC) colorazione; D differenziazione è stata confermata da EN1 e PDGFRa colorazione; Differenziazione di SCL è stata confermata da PAX1, PAX9 e NKX3.2 colorazione; e differenziazione di SYN è stata confermata da SCX, MKX, COL1A1 e COL1A2 macchiatura.
Caratterizzazione di indotto D e SYN
1. enzima-collegata dell'immunosorbente (ELISA) per analisi funzionale dell'iPSC-derivato D
Nel corpo umano, una delle funzioni primarie di fibroblasti cutanei è di secernere proteine della matrice extracellulare (ECM), come il collagene e acido ialuronico che idrata la pelle e contribuire a sostenere la struttura della pelle. Per dimostrare che una quantità paragonabile di proteine di collagene di tipo 1 e acido ialuronico sono stati secernuti nel terreno di coltura di iPSC-derivato D e HDF, ELISA è stata eseguita, come mostrato in Figura 4A.
2. analisi di stimolazione per l'analisi funzionale di SYN iPSC-derivato stretch meccanico
Come diversi studi hanno già segnalato, stimolazione meccanica colpisce lo sviluppo di tendine prima e dopo la nascita e promuove la differenziazione dei tenociti dal precursore cellule18,19. Pertanto, è ben noto che la reattività allo stress meccanico è una delle caratteristiche di tenociti. Per dimostrare la reattività paragonabile di SYN iPSC-derivato umano e umano adulto tenociti, è stato eseguito un dosaggio di stimolazione meccanica elasticizzato come mostrato in Figura 4B.
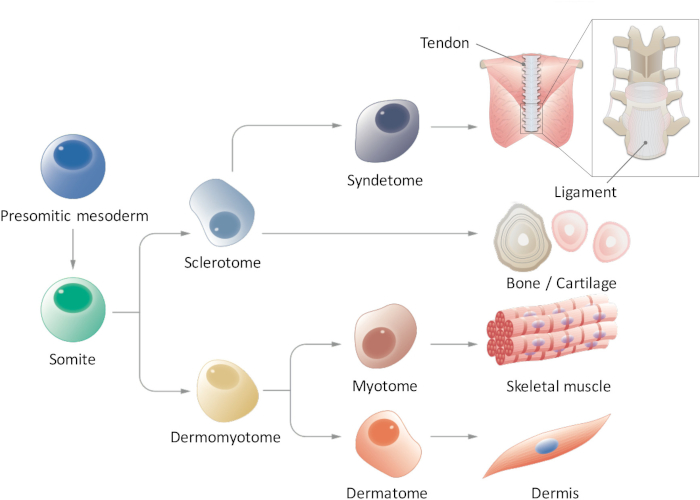
Figura 1: rappresentazione schematica di differenziazione gerarchica del mesoderma parassiale. Mesoderm presomitic è una popolazione di cellule che transitoriamente emerge durante l'embriogenesi precoce e subisce la segmentazione per somiti forma. Somiti sono una popolazione di cellule staminali transitoria che dà luogo a diversi tipi di cellule, come cellule sclerotomo, dermomyotome, syndetome, dermatome e Miotomo, che alla fine si differenziano in tendine/legamento, osso/cartilagine, muscolo scheletrico e derma cellule. Clicca qui per visualizzare una versione più grande di questa figura.
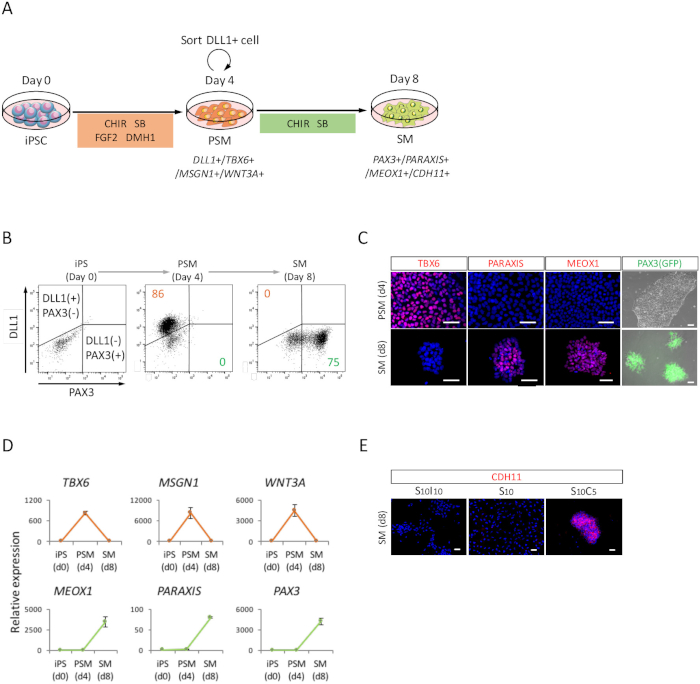
Figura 2: analisi FACS, RT-qPCR e ICC di PSM iPSC-derivati umani e cm. (A) rappresentazione schematica di un protocollo per il differenziamento di SM attraverso PSM. (B) trama punto rappresentativo di colorazione DLL1 e PAX3 (GFP)-fluorescenza il giorno 4 (giorno 8 da iPSC) di induzione di SM e 4 di induzione di PSM. (C) rappresentante immunocytochemical immagini e PAX3 (GFP)-fluorescenza il giorno 4 (giorno 8 da iPSC) di induzione di SM e 4 di induzione di PSM. Le cellule sono state colorate con anti-TBX6, PARAXIS e MEOX1 anticorpi (rosso) e rilevati con PAX3 (GFP) o co-macchiato con DAPI (blu)-fluorescenza (verde). (D) analisi di RT-qPCR di marcatori per PSM e SM presso iPSC, PSM e SM. L'errore di standard ± di mezzi (S.E.) da tre serie di esperimenti sono mostrati. (E) rappresentante immunocytochemical immagini il giorno 4 (giorno 8 da iPSC) di SM, coltivate in S10I10 (combinazione di SB431542 e IWR1, un inibitore di segnalazione WNT), S10 (SB431542) e S10C5 le condizioni (combinazione di SB431542 e CHIR99021). Le cellule erano macchiate con l'anticorpo anti-CDH11 (rosse) e co-macchiate con DAPI (blu). iPS, cellule staminali pluripotenti indotte; PSM, mesoderm presomitic; SM, somite; S10, SB431542 10 ΜM; C5, CHIR99021 5 ΜM; I10, IWR1 10 ΜM; Scala bar = 50 μm. Questa figura è stata modificata da Nakajima et al (2018)15. Clicca qui per visualizzare una versione più grande di questa figura.
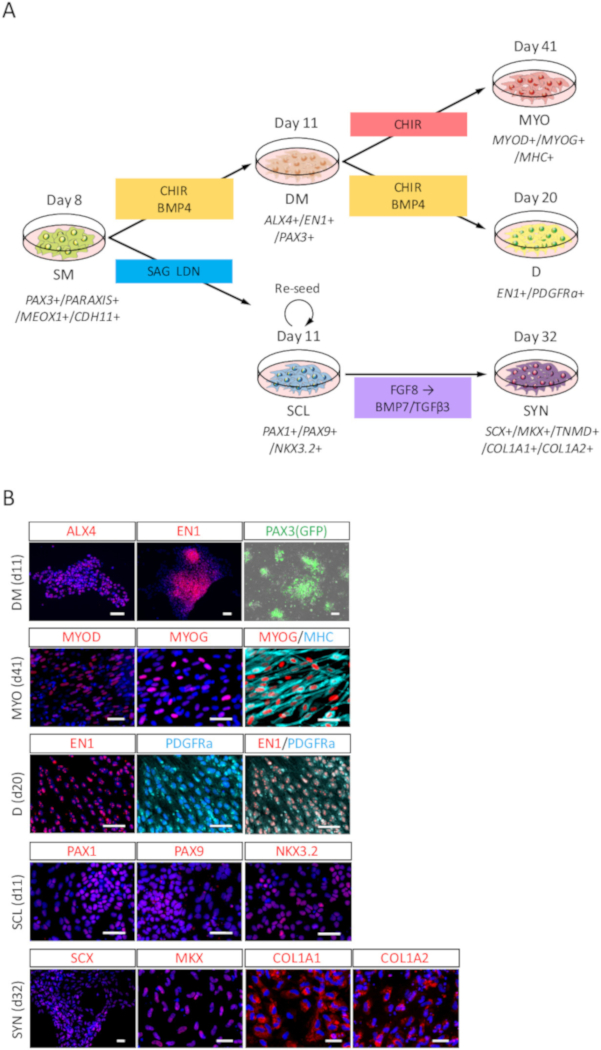
Figura 3: analisi di ICC di DM, MYO, D, SCL e SYN differenziato da mq. iPSC-derivati umani (A) rappresentazione schematica dei protocolli per il differenziamento di derivati di SM. (B) rappresentante immunocytochemical immagini e PAX3 (GFP)-fluorescenza il giorno 3 (giorno 11 da iPSC) di induzione di DM, giorno 30 (giorno 41 da iPSC) di induzione di MYO, giorno 9 (giorno 20 da iPSC) di induzione D, giorno 3 (giorno 11 da iPSC) di induzione SCL e giorno 21 (giorno 32 da iPSC) di induzione di SYN. DM, le cellule erano macchiate di EN1 anticorpi (rosso) e anti-ALX4 e co-macchiate con DAPI (blu) o rilevate con PAX3 (GFP)-fluorescenza (verde); MYO, le cellule sono state colorate con anti-MYOD, MYOG (rosso) e gli anticorpi di MHC (ciano), anche co-macchiati con DAPI (blu); S, cellule erano macchiate con gli anticorpi PDGFRa (ciano) e anti-EN1 (rosso) e co-macchiate con DAPI (blu); SCL, le cellule erano macchiati con anti-PAX1, PAX9 e NKX3.2 anticorpi (rossi) e co-macchiato con DAPI (blu); SYN, le cellule erano macchiati con anti-SCX, MKX, COL1A1 e COL1A2 anticorpi (rossi) e co-macchiato con DAPI (blu). DM, dermomyotome; MYO, Miotomo; D, dermatome; SCL, sclerotomo; SYN, syndetome; Scala bar = 50 μm. Questa figura è stata modificata da Nakajima et al (2018)15. Clicca qui per visualizzare una versione più grande di questa figura.
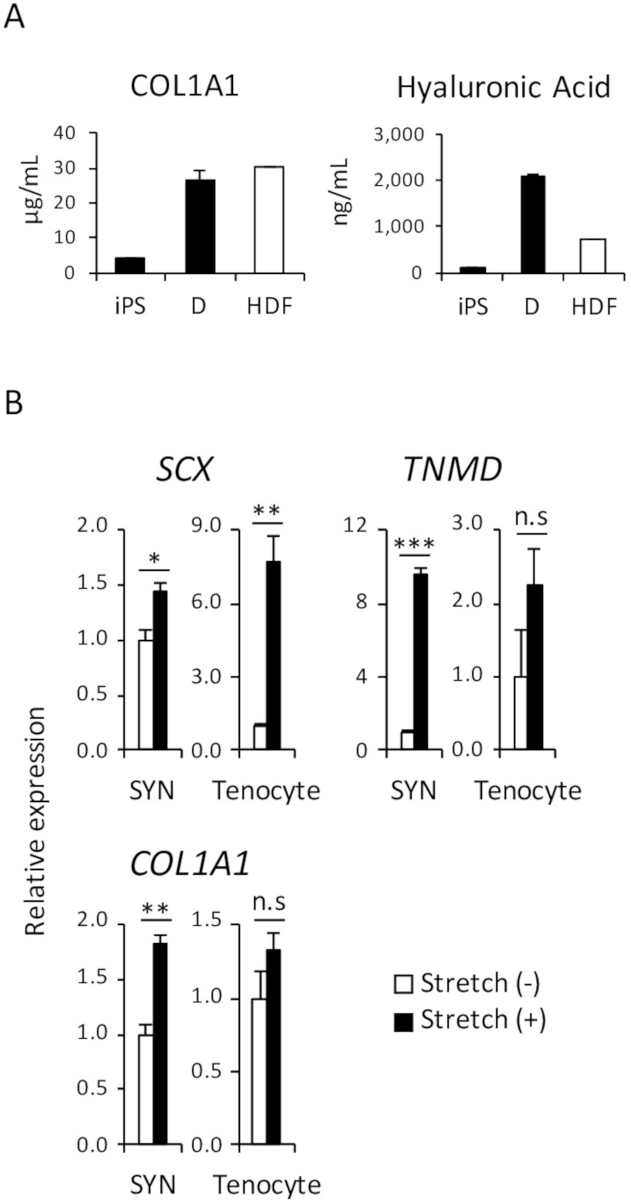
Figura 4: analisi funzionale di indotto D e SYN. (A) la quantità di collagene di tipo 1 e acido ialuronico proteine nel terreno di coltura sono stati analizzati da ELISA. (B) l'effetto di stiramento meccanico stimolazione indotta SYN e tenociti adulto umano è stata valutata da RT-qPCR. L'errore di standard ± di mezzi (S.E.) da tre serie di esperimenti sono mostrati. * p < 0,05; * * p < 0.01; p < 0,001 di Dunnett più confronti t-test rispetto al tratto (-); Nardo, non significativo, HDF, umano adulto dei fibroblasti cutanei. Questa figura è stata modificata da Nakajima et al (2018)15. Clicca qui per visualizzare una versione più grande di questa figura.
| Medio/soluzione | Reagant | Concentrazione |
| Medio basale CDM | Iscove per volta F12 medio/prosciutto di Dulbecco | 1:1 |
| Penicillina/streptomicina | 0,5% | |
| Concentrato di lipidi di costituzione chimica definita | 1% | |
| APO-transferrina | 15 mg/mL | |
| Monothioglycerol | 450 mM | |
| Albumina di siero bovino | 5 mg/mL | |
| Insulina | 7 mg/mL | |
| Soluzione CTK | Acqua | - |
| Tripsina | 0,25% | |
| Collagenasi IV | 0,1 mg/mL | |
| Cloruro di calcio | 1 mM | |
| Knockout SR | 20% | |
| Mezzo di induzione di D | Medio basale CDM | - |
| CHIR99021 | 5 ΜM | |
| BMP4 | 10 ng/mL | |
| Mezzo di induzione di DM | Medio basale CDM | - |
| CHIR99021 | 5 ΜM | |
| BMP4 | 10 ng/mL | |
| Soluzione di ECM | Matrice extracellulare artificiale | 0,3 mg/mL |
| DMEM/F12 | - | |
| Buffer di FACS | PBS | - |
| Albumina di siero bovino | 0,1% | |
| Mezzo di coltura cellulare privo di alimentatore | mTeSR1 | - |
| Penicillina/streptomicina | 0,5% | |
| Terreno di coltura HDF | DMEM | - |
| Siero bovino fetale | 10% | |
| hESC medio | Mezzo di primate ES cellulare | - |
| Penicillina/streptomicina | 0,5% | |
| FGF2 | 4 ng/mL | |
| Mezzo di induzione di MYO | Medio basale CDM | - |
| CHIR99021 | 5 ΜM | |
| Mezzo di induzione di PSM | Medio basale CDM | - |
| SB431542 | 10 ΜM | |
| CHIR99021 | 10 ΜM | |
| DMH1 | 2 ΜM | |
| FGF2 | 20 ng/mL | |
| Mezzo di induzione di SCL | Medio basale CDM | - |
| SAG | 100 nM | |
| LDN193189 | 0,6 ΜM | |
| Mezzo di induzione di SM | Medio basale CDM | - |
| SB431542 | 10 ΜM | |
| CHIR99021 | 5 ΜM | |
| SYN induzione media-1 | Medio basale CDM | - |
| FGF8 | 20 ng/mL | |
| SYN induzione medio-2 | Medio basale CDM | - |
| BMP7 | 10 ng/mL | |
| TGFΒ3 | 10 ng/mL |
Tabella 1: Ricette di Media e soluzione.
| NOME | Avanti | Invertire |
| ACTB | CACCATTGGCAATGAGCGGTTC | AGGTCTTTGCGGATGTCCACGT |
| COL1A1 | GGACACAGAGGTTTCAGTGGT | GCACCATCATTTCCACGAGC |
| MEOX1 | GAGATTGCGGTAAACCTGGA | GAACTTGGAGAGGCTGTGGA |
| MSGN1 | GGAGAAGCTCAGGATGAGGA | GTCTGTGAGTTCCCCGATGT |
| PARAXIS | TCCTGGAGAGCTGTGAGGAT | CACACCCTGTCACCAACAGT |
| PAX3 | AGGAAGGAGGCAGAGGAAAG | CAGCTGTTCTGCTGTGAAGG |
| SCX | CCCAAACAGATCTGCACCTTC | GCGAATCGCTGTCTTTCTGTC |
| TBX6 | AGCCTGTGTCTTTCCATCGT | AGGCTGTCACGGAGATGAAT |
| TNMD | CCCTTCATGCTGAAGCCACTT | CTCACTTTCAGCAGAATTGGGG |
| WNT3A | CAAGATTGGCATCCAGGAGT | ATGAGCGTGTCACTGCAAAG |
Tabella 2: Sequenze Primer per l'analisi di RT-qPCR.
| Concentrazione | ||
| 1 ° Anticorpo | ALX4_Goat | 1/50 |
| CDH11_Mouse | 1/1000 | |
| COL1A1_Rabbit | 1/100 | |
| COL2A1_Mouse | 1-2 μg/mL | |
| EN1_Rabbit | 1/50 | |
| MEOX1_Rabbit | 1/50 | |
| MHC_Rabbit | 1/200 | |
| MKX_Rabbit | 1/50 | |
| MYOD_Rabbit | 1/500 | |
| MYOG_Mouse | 1/400 | |
| NKX3.2_Rabbit | 1/50 | |
| PARAXIS_Rabbit | 1/50 | |
| PAX1_Rabbit | 1/50 | |
| PAX9_Rabbit | 1/50 | |
| PDGFRa_Goat | 1/100 | |
| SCX_Rabbit | 1/50 | |
| TBX6_Goat | 1/50 | |
| 2 ° Anticorpo | Asino anti-capra IgG(H+L) secondaria antibody555 | 1/500 |
| Asino anti-capra IgG(H+L) secondaria antibody647 | 1/500 | |
| Capra anti-Mouse IgG(H+L) secondaria antibody555 | 1/500 | |
| Capra anti-coniglio IgG(H+L) secondaria antibody555 | 1/500 | |
| Capra anti-coniglio IgG(H+L) secondaria antibody647 | 1/500 |
Tabella 3: Primo e secondo gli anticorpi per ICC.
Discussione
Un metodo ben noto per l'induzione del PSC-derivato SM attraverso PSM è la combinazione di CHIR99021 + A83-01 (inibitore di TGFβ) durante l'induzione di PSM dal PSC, ma non durante il processo di maturazione PSM6. Nello studio presente, la segnalazione WNT/beta-catenina è stata inibita utilizzando C59 per indurre SM da PSM. Tuttavia, abbiamo introdotto l'uso di CHIR99021 per attivare la via di WNT durante la differenziazione di SM. Questa decisione è stata fatta basato sulla constatazione che diversi Wnt sono espressi nei tessuti circostanti di SM e dato il fatto che i giornalisti WNT sono attivi nella SM20. Di conseguenza, abbiamo osservato epithelialization, una caratteristica della SM in vivo solo sotto la condizione con CHIR99021, basato sull'accumulo di CDH11 in giunzioni della cellula-cellula (Figura 2E). Questa osservazione indica il coinvolgimento critico di WNT segnalazione durante la differenziazione di PSM ed epithelialization SM, quindi il nostro protocollo può meglio ricapitolare l'ambiente segnalazione endogena. Tuttavia, essa implica anche un'ulteriore possibilità di messa a punto la durante il differenziamento, la via di segnalazione WNT/beta-catenina perché la robustezza e l'efficienza della differenziazione potrebbe variare notevolmente a seconda i tipi cellulari, linee cellulari e vari composti chimici di WNT-induttori utilizzati da ciascun ricercatore.
Questo metodo permette anche di generare tutti i quattro SM derivati, MYO, D, SCL e SYN, da iPSCs umane. I nostri protocolli graduale utilizzando CDM possono essere utilizzati per identificare i requisiti di segnalazione durante umano somitogenesis/somite patterning e forniscono le comprensioni importanti sullo sviluppo dei SM. Ad esempio, i nostri metodi potrebbero essere utili per studiare i meccanismi di segmentazione dell'orologio, un sistema di oscillazione molecolare che regola la formazione di SM. È stato accuratamente studiato in topi e pulcini in zebrafish, ma non in esseri umani a causa della mancanza di adeguati strumenti sperimentali.
Inoltre, il nostro metodo può essere applicabile a future terapie basate sulle cellule cliniche. Ad esempio, D iPSC-derivati umani o SYN possono essere trapiantati in pelle gravemente danneggiata o tendini per la rigenerazione e trattamento. Tuttavia, numerose limitazioni devono essere risolti prima che questo metodo può essere applicato praticamente. Anche se nel presente studio, abbiamo usato le cellule di alimentatore SNL per iPSC manutenzione e soluzione di ECM, che viene estratta dal sarcoma di topo Engelbreth-Holm-Swarm, come un rivestimento superficiale sul piatto durante l'induzione, questi reagenti di derivazione animale non umano dovrebbero essere rimosso per migliorare la qualità clinica. Inoltre, la cella quantità e qualità, che comprende la purezza e la maturazione delle cellule desiderate, inoltre deve essere migliorati. Inoltre, non solo il numero di cellulare, ma anche la forza delle cellule è una caratteristica importante per la rigenerazione del legamento/del tendine. Inoltre, lo sviluppo di marcatori di superficie per purificazione e un metodo novello per la ricostituzione 3D sono indispensabili al fine di avanzare i nostri protocolli alla clinica delle terapie basate sulle cellule.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Vorremmo ringraziare il Dr. Junya Toguchida (CiRA) per il suo aiuto con la gestione del progetto e finanziamento acquisizione, signor Mitsuaki Shibata (CiRA) e Sig. ra Mei Terashima (CiRA) per la loro assistenza tecnica, Dr. Yayoi Toyooka (CiRA) e Dr. Daisuke Kamiya (CiRA) per loro revisione del manoscritto e Mr. Masaya Todani (CiRA) per fornire l'illustrazione (Figura 1). Ringraziamo anche tutti i membri dei laboratori Ikeya e Toguchida (CiRA) per il loro supporto durante questo studio. Questo lavoro è stato supportato da localizzativi per la ricerca scientifica della Japan Society per la promozione della scienza (JSPS) (26670661), il programma per le cellule iPS insolubile malattie ricerca utilizzando malattia-specifica dal Giappone scienza e tecnologia Agenzia (JST) e l'Agenzia giapponese per la ricerca medica e sviluppo (AMED), il centro di Core per iPS Cell Research il centro della rete di ricerca per la realizzazione di medicina rigenerativa (JST/AMED) e iPS Cell Research Fund (in parte a Makoto Ikeya e Junya Toguchida). Makoto Ikeya è stato anche supportato da localizzativi per ricerca scientifica (JSPS) (16H 05447) e il programma di accelerazione per la ricerca di malattie intrattabili che utilizzano le cellule iPS specifici di malattia (AMED).
Materiali
| Name | Company | Catalog Number | Comments |
| ALX4_Goat antibody | Santacruz | sc-22066 | |
| Apo-transferrin | Sigma | T1147 | |
| BMP4 | R&D | 314-BP-010 | |
| BMP7 | R&D | 354-BP-010 | |
| Bovine serum albumin | Sigma | A8806 | |
| Calcium chloride | Nacalai tesque | 067730-15 | |
| CDH11_Mouse antibody | Cell signaling | 13577 | |
| Cell streching device | Strex | STB-140 | |
| Chemically defined lipid concentrate | Gibco | 11905-031 | |
| CHIR99021 | Axon | 1386 | |
| COL1A1_Rabbit antibody | Abcam | ab34710 | |
| COL2A1_Mouse antibody | Thermo scientific | MS-235 | |
| Collagenase IV | Thermofisher | 17104019 | |
| DLL1 APC-conjugated_Mouse antibody | R&D | FAB1818A | For FACS |
| DMEM | Sigma | D6046 | |
| DMEM/F12 | Gibco | 11320-082 | |
| DMH1 | Tocris | 4126 | |
| EN1_Rabbit antibody | Abcam | ab70993 | |
| Fetal bovine serum | Nichirei | 171012 | |
| FGF2 | Wako | 060-04543 | |
| FGF8 | Peprotech | 100-25 | |
| Human dermal fibroblast | Cell applications | 160-05a | |
| Human tenocyte | Angio proteomie | cAP-0041 | |
| Insulin | Wako | 090-06474 | |
| Iscove’s modified Dulbecco’s medium/Ham’s F12 | Gibco | 21056023 | |
| Knockout SR | Gibco | 10828028 | |
| LDN193189 | Axon | 1509 | |
| Matrigel | BD bioscience | 354230 | Artificial extracellular matrix |
| MEOX1_Rabbit antibody | Abcam | ab75895 | |
| MHC_Rabbit antibody | Santacruz | sc-20641 | |
| MKX_Rabbit antibody | Atlas antibodies | A83377 | |
| Monothioglycerol | Sigma | M6145 | |
| mTeSR1 | Stemcell tech | 85850 | |
| Multi well-type silicon rubber chamber | Strex | STB-CH-4W | |
| MYOD_Rabbit antibody | Abcam | ab133627 | |
| MYOG_Mouse antibody | Santacruz | sc-12732 | |
| NKX3.2_Rabbit antibody | Sigma | HPA027564 | |
| Novex Donkey anti Goat IgG(H+L) secondary antibody555 | Invitrogen | A21432 | |
| Novex Donkey anti Goat IgG(H+L) secondary antibody647 | Invitrogen | A21447 | |
| Novex Goat anti Mouse IgG(H+L) secondary antibody555 | Invitrogen | A21422 | |
| Novex Goat anti Rabbit IgG(H+L) secondary antibody555 | Invitrogen | A21428 | |
| Novex Goat anti Rabbit IgG(H+L) secondary antibody647 | Invitrogen | A21245 | |
| PARAXIS_Rabbit antibody | Santacruz | sc-98796 | |
| PAX1_Rabbit antibody | Abcam | ab95227 | |
| PAX9_Rabbit antibody | Gene tex | GTX104454 | |
| PBS | - | - | |
| PDGFRa_Goat | R&D | AF307 | |
| Penicillin/Streptomycin | Invitrogen | 15140-122 | |
| Primate ES cell medium | Reprocell | RCHEMD001 | |
| SAG | Calbiochem | 566661 | |
| SB431542 | Selleckchem | SEL-S1067-10 | |
| SCX_Rabbit antibody | Abcam | ab58655 | |
| TBX6_Goat antibody | R&D | AF4744 | |
| Tendon cell growth medium | Angio-proteomie | cAP-40 | Tenocytes growth medium |
| TGFβ3 | R&D | 243-B3-200 | |
| Trypsin | Gibco | 15090046 |
Riferimenti
- Tanaka, A., et al. Efficient and reproducible myogenic differentiation from human iPS cells: prospects for modeling Miyoshi Myopathy in vitro. PLoS One. 8 (4), e61540 (2013).
- Sakurai, H., et al. In vitro modeling of paraxial mesodermal progenitors derived from induced pluripotent stem cells. PLoS One. 7 (10), e47078 (2012).
- Chal, J., et al. Generation of human muscle fibers and satellite-like cells from human pluripotent stem cells in vitro. Nature Protocl. 11 (10), 1833-1850 (2016).
- Chal, J., et al. Differentiation of pluripotent stem cells to muscle fiber to model Duchenne muscular dystrophy. Nature Biotechnology. 33 (9), 962-969 (2015).
- Umeda, K., et al. Human chondrogenic paraxial mesoderm, directed specification and prospective isolation from pluripotent stem cells. Scientific Reports. 2, 455 (2012).
- Loh, K. M., et al. Mapping the Pairwise Choices Leading from Pluripotency to Human Bone, Heart, and Other Mesoderm Cell Types. Cell. 166 (2), 451-467 (2016).
- Xi, H., et al. In Vivo Human Somitogenesis Guides Somite Development from hPSCs. Cell Reports. 18 (6), 1573-1585 (2017).
- Chal, J., Pourquie, O. Making muscle: skeletal myogenesis in vivo and in vitro. Development. 144 (12), 2104-2122 (2017).
- Tam, P. P., Beddington, R. S. The formation of mesodermal tissues in the mouse embryo during gastrulation and early organogenesis. Development. 99 (1), 109-126 (1987).
- Aulehla, A., Pourquie, O. Signaling gradients during paraxial mesoderm development. Cold Spring Harbor Perspectives in Biology. 2 (2), a000869 (2010).
- Christ, B., Scaal, M. Formation and differentiation of avian somite derivatives. Advances in Experimental Medicine and Biology. 638, 1-41 (2008).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Sakurai, H., et al. Bidirectional induction toward paraxial mesodermal derivatives from mouse ES cells in chemically defined medium. Stem Cell Research. 3 (2-3), 157-169 (2009).
- Zhao, J., et al. Small molecule-directed specification of sclerotome-like chondroprogenitors and induction of a somitic chondrogenesis program from embryonic stem cells. Development. 141 (20), 3848-3858 (2014).
- Nakajima, T., et al. Modeling human somite development and fibrodysplasia ossificans progressiva with induced pluripotent stem cells. Development. 145 (16), (2018).
- McMahon, A. P., Bradley, A. The Wnt-1 (int-1) proto-oncogene is required for development of a large region of the mouse brain. Cell. 62 (6), 1073-1085 (1990).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Suzuki, H., et al. targeting of the transcription factor Mohawk in rats causes heterotopic ossification of Achilles tendon via failed tenogenesis. Proceedings of the National Academy of Sciences of the United States of America. 113 (28), 7840-7845 (2016).
- Marturano, J. E., Arena, J. D., Schiller, Z. A., Georgakoudi, I., Kuo, C. K. Characterization of mechanical and biochemical properties of developing embryonic tendon. Proceedings of the National Academy of Sciences of the United States of America. 110 (16), 6370-6375 (2013).
- Maretto, S., et al. Mapping Wnt/beta-catenin signaling during mouse development and in colorectal tumors. Proceedings of the National Academy of Sciences of the United States of America. 100 (6), 3299-3304 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon