È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Uso di Crystal Violet per migliorare la lettura basata sull'effetto citopatico visivo per la titolazione virale utilizzando i saggi TCID50
In questo articolo
Riepilogo
Questo protocollo mostra un approccio accurato e oggettivo per visualizzare le titolazioni virali utilizzando il viola cristallino, confrontandolo con la microscopia ottica e la colorazione immunocitochimica.
Abstract
La titolazione virale è un test chiave per la ricerca virologica. Il rilevamento dell'effetto citopatico (CPE) tramite saggi TCID50 e saggi di unità di formazione della placca (PFU) sono i due metodi principali per calcolare il titolo di un virus e sono spesso basati sul rilevamento al microscopio o sulla colorazione cellulare per la visualizzazione. Nel caso del test TCID50, la visualizzazione oggettiva si basa comunemente sulla colorazione immunocitochimica (ICC) del virus intracellulare per calcolare i titoli combinati con il rilevamento visivo cpe tramite microscopia. Tuttavia, la colorazione ICC è costosa e richiede tempo. In questo studio, abbiamo confrontato l'osservazione visiva della CPE tramite microscopia, la colorazione ICC e la colorazione viola cristallina per determinare i titoli di due virus che formano CPE, il virus dell'influenza A (IAV) di origine suina e il virus della sindrome riproduttiva e respiratoria suina (PRRSV). Mostriamo che sia la colorazione viola cristallo che icc sono più accurate del rilevamento visivo CPE, presentando livelli di precisione quasi identici sia su IAV che su PRRSV. Per questo motivo, qui presentiamo la colorazione viola cristallina come un modo più rapido e conveniente per determinare le titolazioni virali su un test TCID50 per virus che formano CPE titolati in linee cellulari.
Introduzione
La titolazione virale tramite test TCID50 è una tecnica comunemente usata nella ricerca sulle malattie infettive1. Sebbene nel tempo siano state proposte variazioni sulla matematica alla base di questo metodo1,2,3,4, i metodi attualmente applicati di rilevamento delle infezioni si basano sulla conferma visiva attraverso la presenza di effetto citopatico (CPE) utilizzando la microscopia5. Per confermare la visualizzazione CPE in modo più obiettivo sui saggi TCID50, la colorazione intracellulare immunocitochimica (ICC) che prende di mira le proteine del virus è uno dei metodi più comunemente usati6 in quanto diversi virus possono produrre varie forme di CPE. Nel nostro caso, i cambiamenti morfologici cellulari sono simili quando infettati sia dal virus dell'influenza A (IAV) che dal virus della sindrome riproduttiva e respiratoria suina (PRRSV), in cui le cellule infette si arrotondano e si staccano dalla piastra. Nel caso del PRRSV, provoca un CPE noto come "distruzione totale", in cui tutte le cellule finiscono per staccarsi dal pozzo. IAV, d'altra parte, può presentare sia la distruzione totale che un CPE aggiuntivo noto come "distruzione subtotale" in cui un piccolo numero di cellule non si stacca dopo l'infezione7. Tuttavia, questa tecnica richiede molto tempo per essere eseguita e richiede l'uso di reagenti relativamente costosi. È importante notare che ICC non etichetta CPE, ma piuttosto il numero di cellule infettate con successo dal virus. Ciò implica che le cellule che sono state infettate con successo alla fine dell'incubazione saranno viste come positive anche se l'infezione non ha ancora causato CPE e, quindi, si prevede una percentuale più elevata di cellule POSITIVE ICC rispetto a CPE. Per questo motivo, in questo studio descriviamo un metodo complementare di rilevamento visivo del CPE in un test TCID50 basato sul viola cristallino, una sostanza chimica con una carica positiva che si attacca alle membrane cellulari e viene utilizzata per macchiare le cellule aderenti. Il viola cristallino è spesso utilizzato nella ricerca virologica per misurare le unità di formazione della placca, tra gli altri8.
In questo studio, confrontiamo la sensibilità del rilevamento CPE al microscopio non colorato con la colorazione viola cristallina e la colorazione immunocitochimica basata sul riconoscimento delle proteine virali, che è noto per essere più obiettivo a causa della sua elevata sensibilità. Questo studio dimostra che sia la colorazione viola cristallina che quella immunocitochimica sono più accurate del rilevamento CPE basato sulla microscopia visiva e possono essere utilizzate per identificare oggettivamente i pozzetti infetti in una titolazione TCID50. Data la loro capacità di raggiungere un livello quasi identico di accuratezza sui virus citopatici testati in linee cellulari, il viola cristallino è presentato come un modo più veloce e conveniente per determinare le titolazioni virali su un test TCID50. Il metodo proposto che utilizza la colorazione viola cristallina richiede un tempo totale di 40 minuti per l'esecuzione, con 15 minuti per l'incubazione della paraformaldeide (PFA), 5 minuti per l'incubazione del viola cristallino e un massimo di 15 minuti per la preparazione del materiale, i lavaggi tampone e l'essiccazione. Il protocollo immunocitochimico applicato per il confronto richiede un tempo medio di 4 h 30 min ed è stato eseguito come descritto in precedenza9,10. Il metodo proposto mira a visualizzare una titolazione virale completata. I tempi di infezione e incubazione possono essere eseguiti con layout diversi a seconda del virus. Qui abbiamo testato due virus a RNA con effetto citopatico sulle linee cellulari.
Access restricted. Please log in or start a trial to view this content.
Protocollo
1. Protocollo di titolazione
NOTA: Utilizzare un virus citopatico che infetta le cellule aderenti. Per questa dimostrazione sono stati utilizzati il virus dell'influenza A (IAV) di origine suina (A/California/07/2009/(H1N1)) e il virus della sindrome riproduttiva e respiratoria suina (PRRSV) di tipo 2, ceppo NC 1-7-4.
- Titolare questi virus in 96 piastre di pozzo per 7 giorni in un armadio di biosicurezza situato in un laboratorio di livello di biosicurezza 2 (BSL-2).
- Per eseguire queste titolazioni, seminare 96 piastre di pozzo con la linea cellulare richiesta. Per PRRSV, utilizzare la linea cellulare MA-104 e per IAV utilizzare la linea cellulare MDCK. Per la coltura cellulare, utilizzare il mezzo DMEM integrato con il 10% di FBS, L-Glutammina e Penicillina-Streptomicina e far crescere le cellule alla confluenza.
- Prima dell'infezione, lavare le cellule usando 200 μL di PBS.
- Diluire le scorte virali utilizzando serie di diluizioni 10 volte mescolando 900 μL di mezzi e 100 μL di virus. Assicurarsi di ruotare correttamente il tubo per garantire una corretta miscelazione del mezzo e del virus ed evitare errori di diluizione.
- Segna la disposizione del piatto sul coperchio. Lavare i pozzetti con 1x tampone salino di fosfato (PBS). Aggiungere 50 μL di inoculo nei pozzetti corrispondenti seguendo i metodi di titolazione descritti in precedenza2,3.
- Incubare a 37 °C in un incubatore a CO2 al 5% per 7 giorni.
2. Valutazione dell'effetto citopatico (CPE) tramite microscopia
- Dopo l'incubazione di 7 giorni, lavare tutti i pozzetti con 200 μL di 1x PBS due volte.
- Valutare tutti i pozzetti della piastra al microscopio ottico per rilevare visivamente cpe. Nel caso sia di PRRSV che di IAV, il loro CPE consiste nella morte cellulare e nel successivo distacco dalla piastra, che porta alla rottura del monostrato. Tuttavia, altri virus possono presentare diversi tipi di CPE.
3. Protocollo di colorazione
NOTA: L'effetto citopatico (CPE) è stato valutato tramite colorazione viola cristallina.
- Dopo l'incubazione di 7 giorni, lavare tutti i pozzetti usando 200 μL di 1x PBS due volte.
- Fissare le cellule aggiungendo 50 μL/pozzetto di paraformaldeide al 4% (PFA) in 1x PBS e incubare per 15 minuti a temperatura ambiente (RT).
- Dopo l'incubazione, lavare le cellule due volte con 200 μL di 1x PBS. Quindi, aggiungere 50 μL / pozzetto di cristallo viola diluito al 4% in acqua e incubare per 5 minuti a RT.
NOTA: Il cristallo viola chimico macchia le cellule che rimangono attaccate alla piastra al momento della fissazione, lasciando le sezioni del pozzo in cui le cellule si sono staccate come non macchiate. - Infine, aspirare il cristallo viola dai pozzetti e, facoltativamente, lasciare asciugare le piastre all'aria per 2-5 minuti a RT o lavare la piastra con 200 μL di acqua per rimuovere la macchia in eccesso prima della visualizzazione.
- Utilizzare metodi descritti in precedenza per calcolare matematicamente il titolo. Qui, la formula di Karber e la formula di Muench sono state applicate rispettivamente per PRRSV e IAV2,3. I dettagli di queste equazioni sono presentati nella sezione dei risultati rappresentativi.
4. Etichettatura immunocitochimica (ICC)
NOTA: l'etichettatura immunocitochimica per entrambi i virus è stata eseguita seguendo i metodi descritti in precedenza9,10,11.
- Dopo l'incubazione di 7 giorni della titolazione, fissare le cellule usando PFA come descritto nel passaggio 3.2.
- Lavare le piastre con una soluzione composta da 100 mL di 1 M Tris cloridrato, 1 g di Saponina e 8,5 g di NaCl in 900 mL di H2O. Quindi, completare due ulteriori lavaggi con una miscela di 1x PBS e 5% fetale di siero bovino (FBS) e incubare con quella soluzione di Tris-Saponin-NaCl a temperatura ambiente (RT) per 20 minuti.
- Incubare tutti i pozzetti con l'anticorpo primario per 2 ore a RT.
NOTA: Il volume di anticorpi primari per ciascun virus era di 100 μL ed è stato preparato utilizzando una diluizione 1:300 del 2% FBS in 1x PBS. - Lavare le cellule due volte con la soluzione di Tris, Saponina e NaCl preparata nel passaggio 4.2.
- Incubare tutti i pozzetti con l'anticorpo secondario per 1 ora a RT.
NOTA: Il volume di anticorpi secondari era di 100 μL ed è stato preparato utilizzando una diluizione 1:250 del 2% FBS in 1x PBS. - Lavare le cellule con la diluizione Tris, Saponina e NaCl come descritto nel passaggio 4.2.
- Aspirare la precedente diluizione e incubare le piastre con 200 μL/pozzetto di soluzione di aminoetilptazolo (AEC) ad una diluizione finale di 1:50 in acqua seguendo le indicazioni del produttore per 30 minuti a RT.
- Dopo l'incubazione, scartare la soluzione AEC e aggiungere 100 μL/pozzetto di 1x PBS per l'imaging al microscopio.
- Derivare matematicamente il titolo come descritto nel passaggio 3.5.
Access restricted. Please log in or start a trial to view this content.
Risultati
Le equazioni utilizzate per calcolare matematicamente il titolo erano state precedentemente descritte2,3.
In breve, per PRRSV, applichiamo il metodo Karber:
Titolo (TCID50) = 10 T + 1.3 dove:
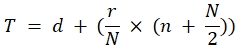
In questa formula d = log negativo dell'ultima diluizione con risposta al virus compl...
Access restricted. Please log in or start a trial to view this content.
Discussione
Le titolazioni virali sono abitualmente utilizzate nella ricerca virologica, con il rilevamento PFU e i saggi TCID50 più comunemente usati1,2,3,4. Entrambi i metodi si basano sul rilevamento di CPE nelle cellule infette e, anche se possono essere valutati visivamente tramite microscopia, di solito viene applicata una colorazione per ottenere un risultato più obiettivo o addirittura p...
Access restricted. Please log in or start a trial to view this content.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Gli autori vorrebbero ringraziare il Dr. Frank Scholle per i suoi utili commenti nel manoscritto, Chloe Mariant per il suo aiuto con le immagini al microscopio e Teresa M. Tiedge per la sua utile revisione in inglese.
Access restricted. Please log in or start a trial to view this content.
Materiali
| Name | Company | Catalog Number | Comments |
| 96-well cell culture plates | Genesee | 25-221 | Clear, flat bottom |
| AEC solution | Thermo Fisher | 1122 | |
| Crystal violet | Thermo Fisher | C581-25; C581-100 | |
| DMEM | Corning | 10-017-CV | |
| Fetal bovine serum | BioWest | S1480 | |
| Paraformaldehide | Thermo Fisher | J19943 | |
| Primary Influenza Antibody | Bioss | BS-0344R | |
| Primary PRRSV Antibody | Bioss | BS-10043R | |
| Saponin | Thermo Scientific | AAA1882014 | |
| Secondaty antibody | Invitrogen | 31460 | |
| Tris Hydrochloride | Thermo Scientific | AM9856 |
Riferimenti
- Kärber, G. Contribution to the collective treatment of pharmacological series experiments. Naunyn-Schmiedeberg's Archive for Experimental Pathology and Pharmacology. 162 (4), 480-483 (1931).
- Ramakrishnan, M. A. Determination of 50% endpoint titer using a simple formula. World Journal of Virology. 5 (2), 85-86 (2016).
- Reed, L. J., Muench, H. A simple method of estimating fifty per cent endpoints. American Journal of Epidemiology. 27 (3), 493-497 (1938).
- Spearman, C. The method of right and wrong cases (constant stimuli) without Gauss's formulae. British Journal of Psychology. 2 (3), 227(1908).
- Darling, A. J., Boose, J. A., Spaltro, J. Virus assay methods: accuracy and validation. Biologicals. 26 (2), 105-110 (1998).
- Kim, J., Chae, C. A comparison of virus isolation, polymerase chain reaction, immunohistochemistry, and in situ hybridization for the detection of porcine circovirus 2 and porcine parvovirus in experimentally and naturally coinfected pigs. Journal of Veterinary Diagnostic Investigation. 16 (1), 45-50 (2004).
- Suchman, E., Blair, C. Cytopathic effects of viruses protocols. American Society of Microbiology. , (2007).
- Karakus, U., Crameri, M., Lanz, C., Yángüez, E. Influenza Virus. , Springer. 59-88 (2018).
- Tingstedt, J. -E., Nielsen, J. Cellular immune responses in the lungs of pigs infected in utero with PRRSV: an immunohistochemical study. Viral Immunology. 17 (4), 558-564 (2004).
- Guarner, J., et al. Immunohistochemical and in situ hybridization studies of influenza A virus infection in human lungs. American Journal of Clinical Pathology. 114 (2), 227-233 (2000).
- Nicholls, J. M., et al. Detection of highly pathogenic influenza and pandemic influenza virus in formalin fixed tissues by immunohistochemical methods. Journal of Virological Methods. 179 (2), 409-413 (2012).
Access restricted. Please log in or start a trial to view this content.
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon