Method Article
In vivo Imaging di criceti infetti da Leishmania infantum mediante inoculazione gengivale di amastigoti axenici che esprimono luciferasi
In questo articolo
Riepilogo
Qui presentiamo una valutazione longitudinale di criceti dorati infettati per via intraperitoneale (IP) o per via intragengivale (IG) con L. infantum-Luc mediante imaging bioluminescente e mediante PCR. I criceti sono stati valutati 1 giorno dopo l'infezione (1 dpi), 1 settimana dopo l'infezione (8 dpi) e 3 settimane dopo l'infezione (22 dpi) e soppressi al 50° dpi e 8 mesi dopo l'infezione.
Abstract
La leishmaniosi tegumentaria americana (ATL) e la leishmaniosi viscerale (VL) sono considerate trascurate dall'Organizzazione Mondiale della Sanità. La VL può essere letale se non trattata; I farmaci utilizzati nel trattamento sono tossici e ci sono casi di resistenza. I test preclinici possono rappresentare un collo di bottiglia nella scoperta di nuovi farmaci per il trattamento, a seconda del modello animale, del ceppo utilizzato e della via dell'inoculo. Il criceto dorato si distingue per la sua elevata suscettibilità ai sottogeneri Viannia e Leishmania , mostrando molti dei processi clinici e immunopatologici osservati nelle malattie umane.
Per l'anatomia del criceto, che ha una coda e arti corti, la via intracardiaca è solitamente la scelta per l'iniezione endovenosa di Leishmania. Tuttavia, è un inoculo che può portare al sanguinamento e alla fine alla morte degli animali. Pertanto, abbiamo standardizzato una via di inoculazione endovenosa alternativa per l'infezione alla vena gengivale, che è minimamente invasiva, consente un facile accesso venoso e provoca poche lesioni locali e sistemiche all'animale. Pertanto, i criceti infettati per via intraperitoneale (IP) o intragengivale (IG) con Leishmania Infantum che esprime luciferasi (Luc) sono stati seguiti per 22 giorni dal sistema di imaging a bioluminescenza e 50 giorni e 8 mesi dopo l'infezione mediante PCR.
Dopo l'inoculazione gengivale sia di amastigoti axenici che di promastigoti di L. infantum-Luc, la bioluminescenza è stata limitata per almeno 2 settimane nel sito di iniezione, che è un indicatore di infezione nei tessuti intorno al plesso gengivale. I criceti infettati per via intraperitoneale con L. infantum-Luc hanno mostrato bioluminescenza dispersa in tutto l'addome, come previsto. Tuttavia, con il sistema di imaging a bioluminescenza l'infezione è diminuita fino al 50° dpi ed è stata rilevabile solo mediante PCR. Gli amastigoti axenici hanno mostrato un'infezione migliore rispetto ai promastigoti, come valutato mediante PCR. Infatti, 8 mesi dopo l'infezione, i parassiti sono stati rilevati mediante PCR nel fegato di animali inoculati con amastigoti axenici per via endovenosa, che può essere una caratteristica del ceppo di riferimento di L. infantum MHOM/BR/1974/PP75, la cui infezione progredisce lentamente e mostra un basso carico di parassiti, inferiore alla risoluzione di imaging bioluminescente. Pertanto, gli amastigoti axenici possono essere una scelta migliore per l'infezione e il follow-up rispetto ai promastigoti e l'inoculo gengivale è una via fattibile per l'iniezione endovenosa di Leishmania e altri agenti patogeni.
Introduzione
Le leishmaniosi sono considerate malattie trascurate e riemergenti causate da più di 20 specie di Leishmania, endemiche in diversi paesi nelle quattro regioni eco-epidemiologiche centrali: America Latina, Africa settentrionale e orientale e Asia occidentale e sud-orientale1. Possono essere raggruppati in leishmaniosi tegumentaria (TL) e viscerale (VL), che è letale se non trattata. L'agente eziologico della VL in Brasile è la Leishmania infantum e il trattamento viene effettuato utilizzando antimoniali pentavalenti o amfotericina B. Questi farmaci vengono somministrati per via endovenosa, hanno un'elevata tossicità, mostrano reazioni avverse e ci sono casi di resistenza2.
Pertanto, è necessario investire nella ricerca di una nuova chemioterapia. I test preclinici sono, infatti, un collo di bottiglia nella scoperta di nuovi farmaci per il trattamento della VL, a seconda del modello animale, del ceppo utilizzato, della via dell'inoculo e di altri fattori logistici, tecnici e operativi. Il criceto dorato si distingue per la sua elevata suscettibilità alle specie dei sottogeneri Viannia e Leishmania, mostrando molti dei processi clinici e immunopatologici osservati nella malattia umana come osservato in precedenti studi con Leishmania braziliensis 3,4. Il criceto infettato da L. infantum sviluppa anche la maggior parte dei processi immunopatologici caratteristici della VL nell'uomo e nei cani5, come anemia, leucopenia, trombocitopenia ed epatosplenomegalia. Inoltre, il criceto dorato è un animale di razza e non mostra una risposta uniforme, riproducendo l'eterogeneità delle manifestazioni cliniche osservate nell'uomo3.
Un altro aspetto da considerare per l'esito dell'infezione è il ceppo di L. infantum e la via di inoculazione. Diversi ceppi di L. infantum differiscono per background genetico e suscettibilità al trattamento 2,6,7. Alcuni ceppi hanno una bassa carica parassitaria nel fegato e nella milza dopo l'infezione da promastigoti8, e gli amastigoti axenici possono essere un'alternativa per migliorare l'infezione che non è molto esplorata. Infatti, la via endovenosa favorisce l'infezione e aumenta la frequenza degli animali con segni clinici; Ma l'inoculazione intraperitoneale è la più utilizzata. La via intracardiaca è la scelta per l'infezione endovenosa con L. infantum 5,8,9. Tuttavia, nei criceti, l'inoculazione intragengivale è una via alternativa per l'iniezione endovenosa, non descritta come sito di infezione. Nonostante sia stata segnalata, la venipuntura gengivale è minimamente invasiva, consente un facile accesso venoso e causa poche lesioni locali e sistemiche10. La puntura della vena gengivale è quella che concorda maggiormente con le raccomandazioni per massimizzare la qualità e l'applicabilità dei risultati, preservando il benessere degli animali11.
La valutazione preclinica dei composti per la VL con metodi tradizionali richiede più animali, che devono essere soppressi per l'analisi istopatologica e la valutazione del carico di parassiti nei tessuti. Al contrario, il sistema di imaging bioluminescente può accelerare gli studi preclinici e ridurre il numero di animali. I siti bioluminescenti nei tessuti infetti possono essere seguiti in tempo reale nello stesso animale per diverse settimane. Diversi studi sulla standardizzazione di questo cruciale strumento tecnologico hanno dimostrato la sua applicazione in studi con topi infettati da Trypanosoma cruzi, Leishmania spp. e Toxoplasma gondii 12,13,14,15. Tuttavia, a seconda del carico di parassiti nei tessuti, la bioluminescenza può essere sottorilevata dal sistema di imaging in vivo, che richiede una valutazione mediante PCR quantitativa degli organi colpiti. Pertanto, proponiamo di sviluppare una metodologia basata sull'iniezione endovenosa di L. infantum che esprime luciferasi nella vena gengivale di criceti dorati per il follow-up da parte del sistema di imaging bioluminescente e PCR.
Protocollo
I protocolli che coinvolgono i criceti hanno seguito le linee guida dell'Instituto Oswaldo Cruz/Comitato Etico del CIO per la Ricerca Animale (approvazione: CEUA/IOC L-015/2022).
1. Clonazione del gene della luciferasi Firefly nel plasmide di espressione di Leishmania
- Digerire il plasmide pLEXSY-hyg2 con l'endonucleasi BglII (10 U/μL) e NotI (10 U/μL). Digerire prima con BglII: aggiungere 50 μl di plasmide (9 μg di DNA), 30 μl di tampone enzimatico di restrizione, 3 μl di albumina sierica bovina (BSA), 3 μl di BglII e acqua priva di nucleasi fino a un volume finale di 300 μl. Incubare per 3 ore a 37 °C.
- Digerire il plasmide TOPOII/LUC con BamHI (10 U/μL) e NotI per rilasciare il frame di lettura aperto della luciferasi della lucciola con la sequenza di Kozak (Kozak-LUC), clonata nel vettore TOPOII16. Digerire prima con BamHI: mescolare 100 μL di plasmide (8,5 μg di DNA) con 30 μL di tampone enzimatico di restrizione; 3 μL di BSA, 3 μL di BamHI e acqua priva di nucleasi fino a 300 μL di volume finale. Incubare per 3 ore a 37 °C.
- Pulisci ogni plasmide su una membrana di silice ed eluisci il DNA due volte con 50 μL di acqua per un volume finale di 100 μL.
- Doppia digestione dei plasmidi pLEXSY-hyg2 e TOPOII/LUC precedentemente linearizzati con NotI. Miscelare 100 μl di DNA eluito dalla membrana di silice con 15 μl di tampone D, 2 μl di NotI e acqua priva di nucleasi fino a 150 μl di volume finale. Incubare per 1 ora a 37 °C.
- Il gel purifica il vettore a doppia digestione (pLEXSY-hyg2) e l'inserto, Kozak-LUC, da un gel di agarosio all'1% dopo elettroforesi con tampone EDTA (TAE) di tris-acetato. Accisa le bande di ~8 kb e 1,6kb con un bisturi. Pesare le fette, aggiungere tre volumi di tampone a 1 volume di gel, incubare a 50 °C per sciogliere il gel, trasferire sulla membrana di silice e lavare ed eluire il DNA due volte con 20 μl di acqua, con un volume finale di 40 μl.
- Legare il vettore a doppia digestione (pLEXSY-hyg2) e l'inserto, Kozak-LUC, con la ligasi T4: miscelare 2 μL di vettore e inserto, entrambi a 55 ng/μL, rapporto molare di 1:5; aggiungere 5 μL di tampone di legatura; 1 μL di DNA ligasi T4 (1U/mL), volume finale di 10 μL. Incubare per una notte a 16 °C.
- Aggiungere 40 μL di acqua priva di nucleasi ai 10 μL della reazione di legatura e precipitare il DNA con 10 volumi di butanolo. Centrifugare alla massima velocità per 10 minuti in una microcentrifuga, lavare il pellet con etanolo al 70%, centrifugare alla massima velocità per 10 minuti, lasciare asciugare il pellet sul banco e aggiungere 4 μL di acqua.
- Mescolare 4 μL della reazione di legatura con batteri elettrocompetenti, JM109 (40 μL), trasferire la miscela in una cuvetta (d = 0,2 cm) e tenerla in ghiaccio. Elettroporare a 2.500 V, 200 Ω, 25 μF, mettere la cuvetta nel ghiaccio e aggiungere 1 mL di mezzo LB. Trasferire immediatamente la miscela in una provetta conica da 15 mL e incubare a 37 °C per 60 min e 100 giri/min.
- Disporre la miscela su due piastre LB di agar/ampicillina (100 μg/mL): una da 200 μL e un'altra con i batteri elettroporosi interi. Concentrare il volume centrifugando i batteri trasformati a 2.000 × g per 10 minuti, risospendere il pellet in 100 μl e impiattare l'intero volume/pellet. Incubare le piastre per 24-30 ore a 30 °C e attendere che i cloni crescano.
- Selezionare i cloni con uno stuzzicadenti e trasferirli in provette coniche da 50 mL contenenti 5 mL di terreno LB/ampicillina (100 μg/mL). Incubare per 20-24 ore a 30 °C e 100 giri/min per consentire la crescita delle colture miniprep.
- Purificare il plasmide con il kit miniprep ed eluire il DNA con 30 μL di acqua. Cloni di schermo contenenti il costrutto pLESXY-LUC-hyg2 mediante digestione con BamHI.
- Mescolare 10 μL di DNA da miniprep; 2 μL di tampone enzimatico di restrizione; 0,2 μL di BSA: 1 μL di BamHI e acqua priva di nucleasi fino a 20 μL di volume finale. Incubare per 1 ora a 37 °C-i frammenti attesi di cloni positivi in elettroforesi su gel di agarosio all'1%: 3.450 e 6.290 paia di basi.
- Preparare una coltura maxiprep per purificare il costrutto pLESXY-LUC-hyg2 su scala più ampia. Aggiungere un Erlenmeyer (1 L), 250 mL di terreno LB integrato con ampicillina e 1 mL di JM109 trasfettato con miniprep e incubare per 18-20 ore a 30 °C e 200 giri/min.
2. Produzione e selezione di Leishmania infantum che esprime luciferasi
- Per rilasciare la cassetta di integrazione di Leishmania , digerire il plasmide pLESXY-LUC-hyg2 fino al completamento con SwaI. Aggiungere 40 μl di plasmide (50 μg), 10 μl di tampone a digestione rapida, 4 μl di SwaI e acqua priva di nucleasi al volume finale di 100 μl. Incubare per una notte a 30 °C.
- Precipitare con etanolo, lavare una volta con etanolo al 70%, risospendere con 50 μL di acqua e tenere in ghiaccio.
- Centrifuga Leishmania MHOM/BR/1974/PP75 promastigote a 1.000 × g per 10 minuti nella fase di crescita esponenziale nel terreno di Schneider. Lavare il pellet con un mezzo per infusione cerebrale e cardiaca (BHI) e centrifugare a 1.000 × g per 10 minuti. Risospendere il pellet a 108 promastigoti/mL in BHI.
- Miscelare i promastigoti di L. infantum (500 μL) con la cassetta di integrazione pLESXY-LUC-hyg2 (50 μg) nella cuvetta di elettroporazione (d = 0,4 cm), su ghiaccio. Elettroporato con due impulsi di 1.500 V/25 micro F e intervalli di 10 s. Dopo il polso, tenere in ghiaccio per 10 minuti.
- Trasferire i promastigoti trasfettati in un matraccio con terreno di Schneider addizionato con FCS al 20% a 26 °C. Dopo 24 ore, aggiungere prima l'igromicina a 50 μg/mL, quindi aumentare a 100 μg/mL dopo 48 ore e 150 μg/mL dopo 1 settimana.
- Il clone ha trasfettato i promastigoti di L. infantum-Luc limitando la diluizione in una piastra a 96 pozzetti con Schneider addizionato con FCS al 20% e 150 μg/mL di igromicina. Incubare a 26 °C per la crescita dei cloni.
- Screening dei cloni per selezionare L. infantum-Luc geneticamente omogeneo e altamente espressivo mediante unità di luminescenza relativa all'emissione bioluminescente (RLU), nel lettore di micropiastre.
- In breve, miscelare 106 promastigoti in una piastra bianca a 96 pozzetti con 100 μL di substrato D-luciferina (300 μg/mL) in tampone composto da 25 mM HEPES pH 7,8, 15 mM MgCl2, 4 mM EGTA, 1 mM DTT, 1 μg/mL BSA, 2,5 mM ATP, 0,1% Triton X-100 e 300 μM Coenzima A, immediatamente valutati nel lettore di micropiastre.
3. PCR per valutare l'integrazione genomica nel locus ribosomiale 18S rRNA (ssu)
- Purificare il DNA genomico da promastigoti che esprimono luciferasi e wild type (10,8). Centrifugare i promastigoti a 1.000 × g per 10 minuti in provette coniche da 15 mL, aggiungere soluzione salina tamponata con fosfato (PBS) al pellet, contare con una camera Neubauer e centrifugare a 1.000 × g per 10 minuti. Risospendere il pellet con tampone di lisi: 100 mM di NaCl, 10 mM di Tris, 25 mM di EDTA, 0,5% di SDS e 0,1 mg/mL di proteinasi K alla densità di 108/mL. Incubare per una notte a 56 °C.
- Eseguire due cicli di purificazione del DNA con un volume di fenolo/cloroformio/alcol isoamilico, 25:24:1 (v/v) e con un volume di cloroformio. Tra ogni ciclo di estrazione, trasferire la fase acquosa in nuove provette e centrifugare a 2.000 × g per 10 minuti.
- Far precipitare il DNA con due volumi di etanolo e centrifugare a 5.000 × g, 10 min, 4 °C. Aggiungere 1 mL di etanolo al 70%, trasferire in una microprovetta e centrifugare a 15.000 × g per 5 minuti.
- Eseguire un secondo lavaggio con etanolo al 70%. Dopo la centrifugazione, scartare il surnatante e asciugare il pellet sul banco.
- Reidratare il DNA genomico con 50-100 μL di acqua ultrapura a bagnomaria a 65 °C per 30 minuti, quantificare il DNA e conservarlo a -20 °C.
- Eseguire la PCR diagnostica per valutare l'integrazione della cassetta pLEXSY nel genoma di Leishmania impostando la seguente reazione PCR: 10 μL di tampone PCR, 0,2 mM di dNTP, 1 U di DNA polimerasi, 1 μg di DNA genomico, 0,5 μM di ciascun primer e acqua priva di nucleasi fino a 50 μL di volume finale.
- Selezionare i primer che ibridano nel vettore pLEXSY e nel genoma di Leishmania: (1) aprt reverse primer A1715 5'-TATTCGTTGTCAGATGGCGCAC-3', ibridare nella regione utr1 (aprt); e (2) ssu forward primer F3001 5'-GATCTGGTTGATTCTGCCAGTAG-3', nel locus cromosomico 18SrRNA (ssu) della Leishmania.
- Utilizzare il seguente protocollo di amplificazione: Un ciclo di denaturazione a 94 °C per 2 min; 30 cicli di amplificazione, 30 s a 94 °C, 30 s a 60 °C, 1 min a 72 °C; allungamento finale di 10 min a 72 °C. La dimensione prevista del prodotto PCR è di 1,1 Kb.
- Utilizzare un secondo set di primer: (1) hyg forward primer A3804 5'-CCGATGGCTGTGTAGAAGTACTCG-3, e (2) ssu reverse primer F3002 5'-CTGCAGGTTCACCTACAGCTAC-3', che si ibridano nel marcatore di resistenza del vettore pLEXSY e nel locus 18SrRNA (ssu) del genoma di Leishmania . Utilizzare lo stesso protocollo PCR descritto al punto 3.6.2, ad eccezione della temperatura di ricottura di 53 °C, anziché di 60 °C. Il prodotto atteso è di 1.8 kb (hyg - 3'ssu).
- Analizzare i prodotti della PCR mediante elettroforesi su gel di agarosio all'1% per verificare se vi fosse integrazione nel locus ribosomiale 18S rRNA del genoma di L. infantum .
4. Promastigote metaciclico di L. infantum- Luc e differenziazione dell'amastigote axenico
- Coltivare L. infantum-LucMHOM/BR/1974/PP75, che esprime costitutivamente la luciferasi, in terreno BHI, integrato con 25 mg/L di emina e 20% di siero fetale di vitello (FCS).
NOTA: L'igromicina è stata ritirata dal mezzo di transfettanti stabili. - Per ottenere promastigoti metaciclici di L. infantum-Luc, seminare 10 promastigoti da6/mL in fiaschi da 75 cm2 , aggiungere 20 mL di terreno BHI integrato con il 20% di FCS a 26 oC e mantenere in posizione orizzontale per 4 giorni o leggermente inclinata fino alla fase stazionaria di crescita. Dopo 6-8 giorni consecutivi, il pallone contiene un aumento dei promastigoti metaciclici; centrifugare i promastigoti a 1.000 × g per 10 minuti a 4 oC.
- Risospendere il pellet di promastigoti con 20 mL di DMEM a basso contenuto di glucosio, contare utilizzando la camera Neubauer e centrifugare a 1.000 × g per 10 minuti a 4 oC.
- Aggiungere DMEM a basso contenuto di glucosio al pellet a 3-5 × 107 promastigoti/mL di densità. Versare 5 mL di promastigoti su un cuscino di Ficoll da 10 mL, diluito in DMEM, 2:1 (v/v). Centrifugare la pendenza a 100 × g per 15 min a 4 oC, con il freno disinserito e l'accelerazione lenta.
- Raccogliere i promastigoti metaciclici all'interfaccia, sulla parte superiore del cuscino di Ficoll, contare utilizzando la camera di Neubauer e centrifugare a 1.000 × g per 10 minuti a 4 oC. Lavare il pellet con PBS per rimuovere eventuali residui di Ficoll, contare utilizzando la camera di Neubauer e centrifugare a 1.000 × g per 10 minuti a 4 oC.
- Risospendere il pellet in PBS a 2 × 109/mL. Tenere i promastigoti sul ghiaccio prima dell'inoculazione.
- Promastigoti differenziati di L. infantum-Luc in amastigoti axenici in vitro, in terreno modificato per amastigote axenicamente cresciuto (MAA)17.
- Preparare la MAA aggiungendo 9,55 g/L di terreno 199 con sali di Early e glutammina, 0,5% di tripto-caseina, 3 mM di L-cisteina, 15 mM di D-glucosio, 4 mM di NaHCO3, 0,33 mM di Na2HPO4, 25 mM di HEPES fino a un pH finale di 6,5 e integrare con il 20% di siero fetale di vitello (FCS).
- Iniziare le colture in fiasche medie (75cm2) con 9 mL di MAA media e 1 mL di promastigoti nella fase stazionaria di crescita (7° giorno), 2-3 × 107 promastigoti/mL. Incubare per 24-48 ore a 32 °C per consentire la differenziazione dell'amastigote.
- Mantenere gli amastigoti cresciuti assinicamente per due o tre passaggi a settimana fino a quando non raggiungono una densità di 2-3 × 107/mL.
- Aumentare la coltura di amastigoti axenici per l'infezione da criceto. Preparare2 matracci da 150 cm con un'alta densità di amastigoti (5 × 106/mL) in 20 mL di terreno MAA integrato con il 20% di FCS.
- Dopo 24 ore, centrifugare a 1.000 × g per 10 minuti a 4 oC, lavare con lo stesso volume di PBS, contare con la camera Neubauer e centrifugare a 1.000 × g per 10 minuti a 4 oC. Aggiungere PBS per preparare l'inoculo di 108 amastigoti in 50 μL.
5. Animali
- Procurati criceti dorati maschi all'età di 4-6 settimane e tienili in quarantena per 2 settimane. Assicurati che abbiano 6-8 settimane e pesino 80-100 g quando sono infettati da L. infantum MHOM/BR/1974/PP75.
NOTA: Questo protocollo si applica sia ai criceti maschi che alle femmine. Tuttavia, a causa della loro disponibilità all'epoca, in questo studio sono stati utilizzati solo criceti maschi. - Dividere i criceti in quattro gruppi: gruppo 1, infettato intraperitonealmente con amastigote (n = 3); gruppo 2, infettato intraperitonealmente da promastigoti (n = 3); gruppo 3, infettato nella vena gengivale con amastigote (n = 2); gruppo 4, infettato nella vena gengivale con promastigote (n = 2); Gruppo 5, criceti non infetti come controllo (n = 2).
- Tenere gli animali in rastrelliere ventilate, dotate di sistema di ventilazione controllata (10-15 ricambi aria/h), ciclo luce/buio 12 h, a 21 ± 2 oC e 40-60% di umidità. Fornire arricchimento ambientale, come carta, fieno, cotone idrofobo, rotoli di cartone, acqua sterilizzata in autoclave e cibo ad libitum.
6. Infezione per via intraperitoneale
- Trattiene fisicamente il criceto sulla griglia della gabbia, usando il pollice e l'indice come pinzette per pizzicare la pelle sul retro dai lati vicino alla testa, e le altre dita tirano la pelle lungo la schiena, cercando di ottenere quanta più pelle possibile.
- Allineare il sito dell'inoculo nel quadrante addominale in basso a destra con i muscoli posteriori della coscia.
- Inoculare con il corpo dell'animale inclinato di 45º, la testa più bassa dell'addome e l'ago posizionato a 15°-20°.
- Inoculare amastigoti axenici o promastigoti, 50 μL (108) in PBS, con un ago da 13 x 0,45 mm accoppiato a una siringa da 1 mL. Inserire 0,5 cm oltre lo smusso. Dopo aver inserito l'ago nell'addome, tirare lo stantuffo per verificare che non sia stato inserito nella vena e iniettare il liquido con attenzione.
7. Infezione endovenosa mediante inoculazione gengivale
- Trattiene il criceto seguendo la sezione 6. Somministrare per via intraperitoneale una miscela di 200 mg/kg di ketamina cloridrato al 10% e 10 mg/kg di xilazina cloridrato al 2%, volume finale di 1,3 mL, in una siringa da 3 mL accoppiata a un ago da 13 x 0,45 mm.
- Pizzicare la zampa bloccando la base interdigitale del cuscino per assicurarsi che l'animale sia anestetizzato.
- Trattenere il criceto anestetizzato in posizione supina con entrambe le mani e tirare delicatamente verso il basso il labbro inferiore con i pollici per esporre la gengiva e la vena gengivale
- Posizionare un ago più sottile (8 x 0,30 mm) accoppiato a una siringa da 1 ml sotto gli incisivi inferiori lungo la linea mediana tra la coppia di denti con un angolo di 25º e inserire 2-4 mm nella vena labiale mandibolare.
- Per confermare che l'inoculo era endovenoso e che l'ago era inserito nella vena labiale mandibolare, aspirare il sangue fino al fusto dell'ago (parte gialla).
- Iniettare lentamente e con attenzione l'inoculo di 50 μL (10,8) di amastigoti o promastigoti in PBS per 1 minuto fino a quando l'intero volume è stato inoculato per evitare la fuoriuscita dell'inoculo e del sangue dalla vena all'ambiente esterno e consentire la dispersione nel flusso sanguigno.
- Prima di rimuovere l'ago, applicare una leggera pressione con un batuffolo di cotone. Contemporaneamente, rimuovere l'ago dal recipiente tenendo il batuffolo di cotone per favorire l'emostasi nel sito, applicando una leggera pressione per 1 minuto per evitare sanguinamento e perdita dell'inoculo
8. Eutanasia mediante dissanguamento con puntura cardiaca
- Trattenere l'animale per l'anestesia, come descritto nella sezione 7. Eseguire la puntura cardiaca con un ago da 20 x 0,55 mm accoppiato a una siringa da 5 ml, inserita con un angolo di 45º e leggermente inclinata a sinistra, appena sotto la cartilagine xifoidea.
- Prelevare 1 ml di sangue. Dopo il prelievo di sangue terminale, rimuovere la siringa ma mantenere l'ago nel cuore.
- Se l'animale sta già respirando e mostra un battito cardiaco, somministrare tiopentale sodico al 5% (200 mg/kg) con una siringa da 5 ml e accoppiarlo all'ago da 20 x 0,55 mm che si trova già nel cuore.
- Dopo 5 minuti, con uno stetoscopio, verificare se ci sono movimenti respiratori e battito cardiaco.
9. Estrazione di DNA da organi e tessuti
- Dopo l'eutanasia, raccogliere la milza, i linfonodi mesenterici e i frammenti del fegato.
- Tagliare i fazzoletti congelandoli su ghiaccio secco, scongelandoli e tagliandoli con due lame di bisturi. Digerire 20 mg di tessuti tritati in 600 μL di tampone di lisi con 17,5 μL di proteinasi K (20 mg/mL) e incubare a 55 °C durante la notte.
- Centrifugare a 15.000 × g per 4 minuti per rimuovere i frammenti di tessuto che non vengono lisati.
- Aggiungere 200 μl di soluzione di precipitazione proteica, mescolare energicamente per 20 s, incubare per 5 minuti su ghiaccio e centrifugare a 15.000 × g per 4 minuti.
- Trasferire il surnatante in un'altra provetta, aggiungere 600 μl di isopropanolo e mescolare delicatamente fino alla precipitazione del DNA. Centrifugare il DNA a 15.000 × g per 2 minuti e lavare il pellet con etanolo al 70% per 30 minuti in un agitatore inclinabile e centrifugare.
- Scartare l'etanolo ed essiccare il pellet a temperatura ambiente. Reidratare il DNA con 50-200 μL di acqua ultrapura a 65 °C per 30 minuti, quantificare e diluire il DNA a 0,1 μg/mL e conservare a -20 °C.
10. Valutazione dell'infezione nei tessuti e negli organi mediante PCR
- Valutare la carica di parassiti nel DNA genomico di tessuti e organi mediante PCR convenzionale: 2,5 μL di tampone PCR, 1,5 mM di MgSO4, 0,2 mM di dNTP, 1 U di DNA polimerasi, 300 ng di DNA genomico, 0,2 μM di ciascun primer e acqua priva di nucleasi fino a un volume finale di 25 μL.
- Amplificare utilizzando il protocollo di L. infantum kDNA18: Un ciclo di denaturazione a 94 °C per 2 min; 39 cicli di amplificazione, 30 s a 94 °C, 15 s a 60 °C, 30 s a 72 °C, estensione finale di 5 min a 72 °C. La dimensione prevista del prodotto PCR è di 145 bp. (1) Fw kDNA (RV1) 5'-CTTTTCTGGTCCCGCGGGTAGG-3'; (2) Rv kDNA (RV2) 5'-CCACCTGGCCTATTTTACACCA-3'.
- Amplificare il criceto GAPDH, il controllo endogeno della PCR3: un ciclo di denaturazione a 94 °C per 2 min, 39 cicli di amplificazione, 30 s a 94 °C, 15 s a 58 °C, 30 s a 72 °C, estensione finale di 5 min a 72 °C. (1) Fw GAPDH criceto 5'- GGTTGCCAAACCTTATCAGAAATG-3'; (2) Criceto Rv GAPDH 5'- TTCACCTGTTCCACAGCCTTG -3'.
- Analizzare i prodotti PCR mediante elettroforesi su gel di agarosio all'1%.
11. Follow-up del criceto mediante imaging a bioluminescenza in vivo
- Valutare la progressione dell'infezione nel tempo mediante l'emissione di bioluminescenza dell'intero animale nel sistema di imaging in vivo a bioluminescenza, che consiste in una telecamera raffreddata ad accoppiamento di carica (CCD) montata su una camera a tenuta di luce. Mantenere l'animale anestetizzato durante l'acquisizione dell'immagine con un'erogazione del cono nasale.
- Prima dell'acquisizione della bioluminescenza e 5 minuti prima di indurre l'anestesia, iniettare D-luciferina (150 mg/kg) per via intraperitoneale nei criceti seguendo la sezione 6. Preparare una soluzione madre di sale di potassio di D-luciferina 15 mg/mL in PBS, filtrare-sterilizzare e conservare a -80 oC.
- Cinque minuti dopo aver iniettato agli animali la D-luciferina, anestetizzarli con isoflurano al 2% in una camera di induzione ricca di ossigeno. Dopo altri 5 minuti, acquisire immagini in bioluminescenza utilizzando la telecamera CCD. Ottenere immagini di criceti in posizione ventrale 2 ore e 24 ore dopo l'infezione, 8 e 22 giorni dopo l'infezione (dpi).
NOTA: Mantenere l'anestesia durante il processo di imaging con il dispositivo di erogazione di isofluorano-ossigeno. - In base al livello di emissione di bioluminescenza, utilizzare i seguenti parametri: tempo di esposizione, variabile da 30 s a 5 min, Binning medio o grande, f/stop 1 e campo visivo D (12,5 cm).
12. Quantificazione della bioluminescenza in animali infettati da L. infantum-Luc
- Utilizza lo strumento di misurazione manuale per identificare le regioni di interesse (ROI), le dimensioni e l'area della testa e del corpo dell'animale. Quantifica la bioluminescenza acquisita dalla telecamera CCD nel ROI selezionato.
- Misurare il segnale di fondo con il ROI medio di fondo e sottrarre dall'emissione bioluminescente ottenuta dal ROI manuale.
- Esprimere l'emissione di bioluminescenza in radianza, un'unità normalizzata per tempo e area: photons.sec-1.cm-2.sr-1
Risultati
Espressione stabile della luciferasi in L. infantum
L. infantum geneticamente modificato è stato prodotto utilizzando il plasmide della linea pLEXSY, che si integra nel genoma di Leishmania nel locus ribosomiale 18S rRNA (ssu), la cui trascrizione è guidata dalla RNA polimerasi I. Pertanto, i cloni di L. infantum-Luc sono stati valutati per l'integrazione plasmidica nel genoma di Leishmania e per l'espressione stabile mediante emissione di bioluminescenza in vitro. Il clone altamente esprimente che mostra bioluminescenza >120 volte sopra lo sfondo è stato scelto per la valutazione dell'integrazione genomica mediante PCR. Vedere la Figura 1 per l'elettroforesi su gel di agarosio dei prodotti PCR per valutare l'integrazione plasmidica nel genoma e l'emissione di bioluminescenza (RLU) dei promastigoti del clone di L. infantum-Luc. Da ciascuna PCR sono stati ottenuti frammenti delle dimensioni previste; un prodotto di circa 1,1 kb (5'ssu - utr1) e un altro di 1,8 kb (hyg-3'ssu) sono stati amplificati dal genoma di L. infantum-Luc (Figura 1A), confermando l'integrazione della cassetta plasmidica e del gene della luciferasi nel locus ssu del genoma di L. infantum.
L'espressione della luciferasi della lucciola è stata valutata anche nei promastigoti di L. infantum-Luc mediante emissione di bioluminescenza (RLU) nel lettore di micropiastre, come descritto nei protocolli, sezione 2. Anche dopo diversi passaggi in coltura e in topi BALB/c per 5 giorni, ha mantenuto il livello di bioluminescenza; 569,3 ± 19,5 per il fondo wild type, e 59361,9 ± 2673,3 (n = 2) per il clonato L. infantum-Luc (Figura 1B). Così, il clone di L. infantum-Luc, che esprime stabilmente la luciferasi della lucciola, è stato utilizzato per infettare i criceti attraverso le vie intragengivali o intraperitoneali.
Inoculo endovenoso nella vena gengivale
Per inoculare la Leishmania nel flusso sanguigno dei criceti, è necessario prestare attenzione per ridurre al minimo la perforazione delle vene, il sanguinamento e la fuoriuscita dell'inoculo. Pertanto, il labbro inferiore deve essere tirato delicatamente verso il basso per esporre la vena gengivale (Figura 2A); e un ago da 30 G di calibro più piccolo deve essere utilizzato per evitare un'eccessiva perforazione della vena. L'ago deve essere posizionato con la lunetta rivolta verso l'alto per inserirsi nella vena con un'angolazione corretta (Figura 2B). Infatti, per garantire che l'ago sia stato iniettato nel vaso, la vena labiale mandibolare, lo stantuffo della siringa deve essere tirato verso il basso fino a quando il sangue non viene aspirato nel cilindro dell'ago (Figura 2C). Prima della rimozione dell'ago, è necessario applicare una leggera pressione con un batuffolo di cotone per favorire l'emostasi (Figura 2D).
Valutazione longitudinale mediante imaging a bioluminescenza
I criceti infettati per via intraperitoneale (IP) o per via intragengivale (IG) con L. infantum-Luc sono stati seguiti fino al 50° dpi e sono stati valutati mediante imaging a bioluminescenza fino a 22 dpi (Figura 3). Le immagini sono state acquisite 2 ore dopo l'infezione intraperitoneale con 108 parassiti nella cavità peritoneale; Le immagini sono state acquisite per 30 s o 1 minuto di esposizione binning medium. Il segnale di bioluminescenza era >65 volte più intenso nell'addome negli animali infetti da amastigote (4,6 × 105 ± 3,7 × 105) rispetto a quelli infetti da promastigoti (6,8 × 103 ± 3,8 × 103) (Tabella 1), il che dimostra che gli amastigoti differenziati in vitro sono più bioluminescenti dei promastigoti metaciclici in fase stazionaria e purificati nel cuscinetto di Ficoll.
Un giorno dopo l'infezione (1 dpi), sono state acquisite immagini in bioluminescenza per 3 minuti di esposizione (Figura 3). C'è stata una diminuzione del 45% del segnale di bioluminescenza nella regione addominale nei criceti infetti da promastigoti e del 70% in quelli infetti da amastigoti (Figura 3), il che suggerisce che gli amastigoti si erano degradati in misura maggiore rispetto ai promastigoti metaciclici (Tabella 1 e Figura 4). Una settimana dopo l'infezione (8 dpi), gli animali infetti da promastigoti hanno sostenuto il segnale di bioluminescenza (3,3 × 103 ± 5 × 103). Tuttavia, l'emissione di bioluminescenza nei criceti infetti da amastigote è diminuita del 95%, da 1,3 × 105 ± 1,1 × 105 a 6,7 × 103 ± 7,5 × 103 (Tabella 1) e ha raggiunto lo stesso livello dei criceti infetti da promastigote. Tre settimane dopo l'infezione (22 dpi), è stato acquisito un segnale di bioluminescenza per 5 minuti di esposizione e binning large (Figura 3); il segnale era molto più basso per i promastigoti e gli animali infetti da amastigoti (Tabella 1 e Figura 4).
Un altro gruppo di criceti è stato infettato per via intragengivale con amastigoti e promastigoti di L. infantum-Luc (10,8); è stata osservata un'emissione di bioluminescenza nella regione mascellare (Figura 3). Il follow-up è iniziato 1 giorno dopo l'infezione e i criceti infetti da amastigote hanno mostrato più segnale di bioluminescenza e radiosità (7,3 × 103 ± 4,1 × 103) rispetto a quelli infetti da promastigoti (1 × 103 ± 5,7 × 102). Una settimana dopo l'infezione (8 dpi), è stato osservato un calo del 36% del segnale di bioluminescenza negli animali infetti da promastigoti e del 90% nei criceti infetti da amastigote; La radianza variava da 7,3 ×10 3 ± 4,1 × 103 a 7,8 × 102 ± 5,6 × 102 (Tabella 1). Tre settimane dopo l'infezione (22 dpi), è stato acquisito anche un segnale di bioluminescenza per 5 minuti di esposizione e binning large (Figura 3). Il segnale di bioluminescenza era simile e basso per gli animali infetti da promastigote e amastigoti (Tabella 1 e Figura 4) nella testa degli animali infettati alla gengiva e non è stata osservata dispersione dell'infezione nella regione addominale da parte del segnale di bioluminescenza.
Valutazione dell'infezione nei tessuti e negli organi mediante PCR
Abbiamo eseguito la PCR convenzionale per studiare l'infezione in organi specifici, come fegato, milza e linfonodi, che potrebbero essere al di sotto del limite di rilevamento dell'imaging in vivo . La regione bersaglio del kDNA era più specifica per l'amplificazione del DNA di L. infantum nei tessuti e negli organi infetti, e la PCR per l'enzima GAPDH dal criceto era un controllo dell'integrità del DNA e della reazione PCR. Nell'analisi sono stati presi in considerazione solo i campioni che sono stati amplificati per GAPDH . Così, mediante PCR, due dei tre criceti infettati per via intraperitoneale con amastigoti axenici hanno mostrato un'infezione nei tessuti e negli organi; l'animale due (A2) nella milza e l'animale tre (A3) nel fegato, a 50 dpi (Figura 5A). Un criceto è stato infettato per via intraperitoneale con promastigoti axenici; l'animale tre (A3) (Figura 5B) ha mostrato amplificazione nel linfonodo. I criceti inoculati per via intragengivale con promastigoti o amastigoti axenici a 50 dpi non potevano mostrare una chiara amplificazione: solo una banda nel fegato di un animale (A1) infetto da promastigoti (Figura 5C). In particolare, abbiamo avuto tre animali mantenuti per 8 mesi il cui inoculo somministrato per via intragengivale con amastigoti ha perso leggermente durante l'iniezione. Due dei tre animali hanno mostrato una chiara infezione nel fegato, gli animali uno e due (Figura 5D).
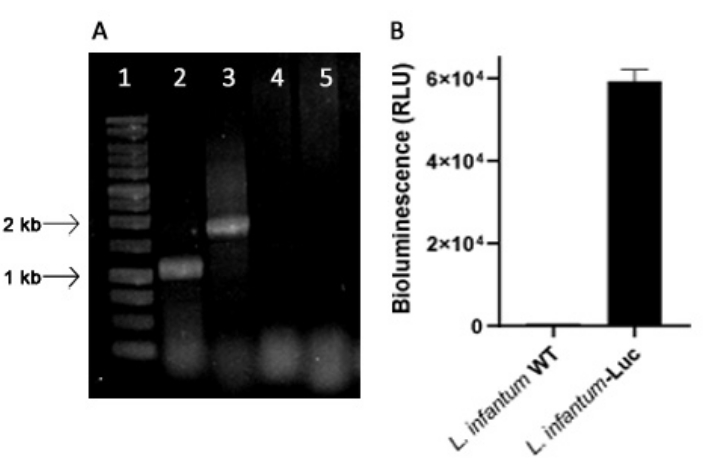
Figura 1: Valutazione del clone di Leishmania infantum-Luc mediante PCR ed emissione di bioluminescenza. (A) Elettroforesi su gel di agarosio di prodotti PCR per valutare l'integrazione plasmidica nel genoma: corsia 1 - 1 kb scala del DNA; PCR del DNA genomico di L. infantum-Luc, corsia 2 - 5'ssu - utr1 (1.1 kb) e corsia 3- hyg- 3'ssu (1.8 kb); PCR del DNA genomico di L. infantum-wt, corsie 4 e 5. (B) Emissione di bioluminescenza (RLU) di promastigoti di clone di L. infantum-Luc (10,6) nel lettore di micropiastre. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Iniezione endovenosa di Leishmania infantum-Luc nella vena gengivale. (A) Il criceto è stato posto in decubito dorsale e il labbro inferiore è stato abbassato. (B) Un ago più sottile (8 x 0,30 mm) accoppiato a una siringa da 1 ml è stato posizionato sotto gli incisivi inferiori lungo la linea mediana tra la coppia di denti con un angolo di 25º e inserito 2-4 mm nella vena mandibolare labiale. (C) Inoculazione di 50 μl (10,8) di amastigoti o promastigoti in PBS. (D) Emostasi utilizzando un batuffolo di cotone e applicando una leggera pressione sul sito di inoculazione. Clicca qui per visualizzare una versione più grande di questa figura.
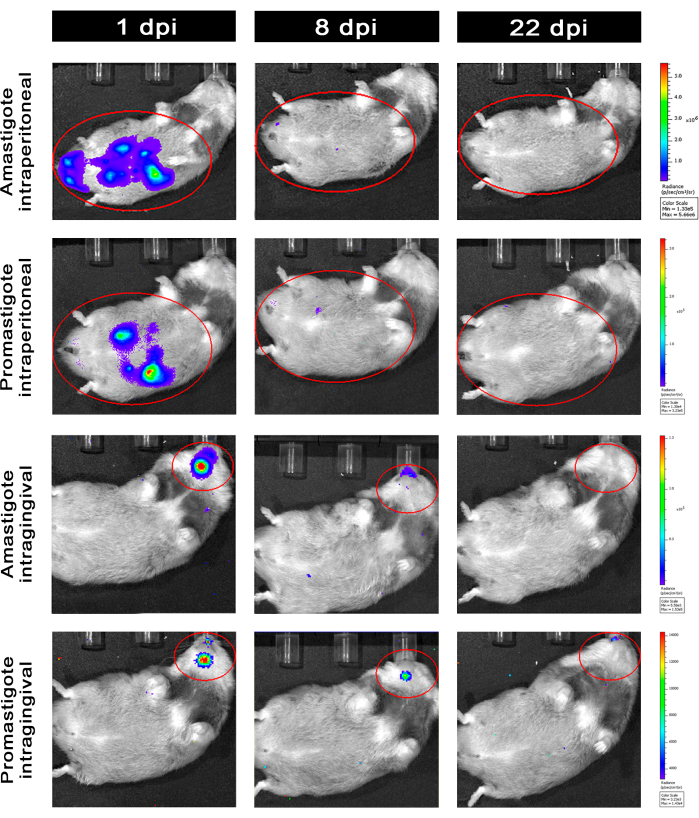
Figura 3: Follow-up mediante imaging a bioluminescenza in vivo . Immagini rappresentative di un animale per gruppo: Infettato tramite intraperitoneale (pannelli superiori) o intragengivale (pannelli inferiori), con amastigoti o promastigoti di L. infantum-Luc, per 1, 8 e 22 dpi. ROI rosso che rappresenta le regioni sondate all'addome e alla testa, rispettivamente per l'infezione intraperitoneale o intragengivale. I dati mostrano che a 1 dpi, tutti gli animali mostravano un segnale di bioluminescenza nell'addome o nella mandibola. Il segnale diminuiva dopo 8 dpi ed era quasi impercettibile in qualsiasi gruppo a 22 dpi. Clicca qui per visualizzare una versione più grande di questa figura.
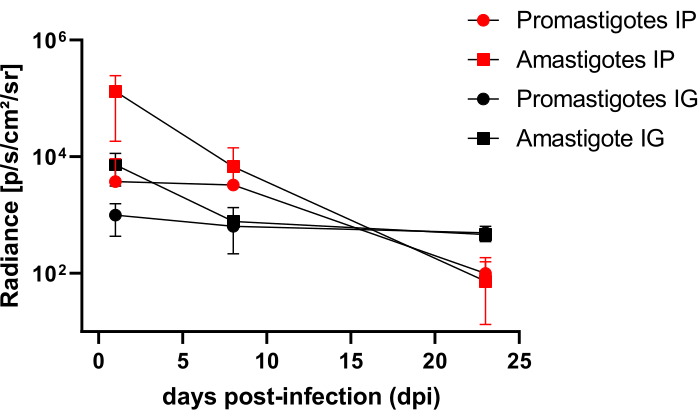
Figura 4: Analisi comparativa della radianza da immagini in bioluminescenza. La quantificazione della radianza photons.sec-1.cm-2.sr-1 è stata eseguita nell'addome o nella testa dei criceti con strumenti manuali di misurazione del ROI. Il ROI medio di fondo è stato sottratto dal ROI della misurazione per rimuovere qualsiasi segnale spurio. Clicca qui per visualizzare una versione più grande di questa figura.
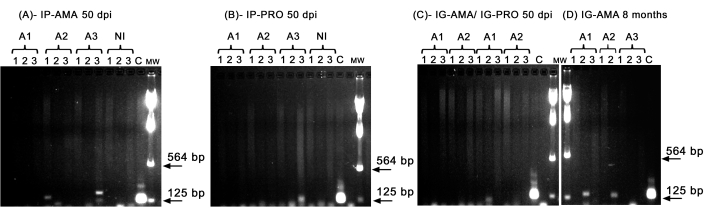
Figura 5: Amplificazione PCR del kDNA. (A) IP-AMA, criceti infettati per via intraperitoneale con amastigote, 50 dpi (n = 3); (B) IP-PRO, infettato per via intraperitoneale con promastigoti, 50 dpi (n = 3); (C) IG-AMA, infettato per via intragengivale con amastigote (n = 2), IG-PRO, infettato per via intragengivale con promastigote (n = 2); (D) IG-AMA, infettato per via intragengivale con amastigote, 8 mesi dopo l'infezione (n = 3). NI, criceti non infetti come controllo negativo (n = 2); C- DNA genomico di L. infantum-Luc, controllo positivo della PCR. Tessuti e organi: 1- milza, 2- fegato, 3- linfonodi. MW- Marcatore di peso molecolare, le frecce indicano le bande di peso molecolare più basse. A1- animale uno, A2- animale due e A3- animale tre. Clicca qui per visualizzare una versione più grande di questa figura.
| Dpi | Amastigotes IP | Promastigotes IP | Amastigotes IG | Promastigotes IG | ||||||||
| Significare | SD | N | Significare | SD | N | Significare | SD | N | Significare | SD | N | |
| 0 | 4,6 x 105 | 3,7 x 105 | 3 | 6,8 x 103 | 3,8 x 103 | 3 | - | - | - | - | - | - |
| 1 | 1,3 x 105 | 1,1 x 105 | 3 | 3,8 x 103 | 5,5 x 103 | 3 | 7,3 x 103 | 4,1 x 103 | 2 | 1,0 x 103 | 5,7 x 102 | 2 |
| 8 | 6,7 x 103 | 7,5 x 103 | 3 | 3,3 x 103 | 5,0 x 103 | 3 | 7,8 x 102 | 5,6 x 102 | 2 | 6,4 x 102 | 8,2 x 101 | 2 |
| 22 | 7,3 x 101 | 8,5 x 101 | 3 | 9,9 x 101 | 8,6 x 101 | 3 | 4,6 x 102 | 7,5 x 101 | 2 | 5,0 x 102 | 1,5 x 102 | 2 |
Tabella 1: Dati grezzi dall'analisi comparativa della radianza delle immagini di bioluminescenza. Quantificazione della radianza fotoni medi.sec-1.cm-2.sr-1 per gruppo e percorso. Abbreviazioni: dpi = giorni dopo l'infezione; SD = deviazione standard; N = dimensione del campione.
Discussione
La raccolta del sangue o l'iniezione endovenosa di sostanze nei criceti è necessaria per vari studi scientifici. Sono stati sviluppati diversi metodi per accedere a diversi percorsi di raccolta o inoculazione direttamente correlati agli obiettivi della ricerca19. A causa dell'anatomia del criceto - una coda corta e arti - la via intracardiaca è solitamente la scelta per l'iniezione endovenosa di Leishmania. A seconda del ceppo utilizzato, la via intracardiaca si è rivelata vantaggiosa come ceppo di riferimento L. infantum MHOM/BR/1974/PP75, la cui infezione si verifica a lungo termine, 6-9 mesi5. Tuttavia, è un inoculo che può portare al sanguinamento e alla morte dell'animale. Pertanto, abbiamo standardizzato una via di inoculazione endovenosa alternativa per l'infezione al plesso gengivale, la vena labiale mandibolare, che causa meno danni all'animale. Gli animali sono stati infettati con il ceppo di riferimento geneticamente modificato L. infantum MHOM/BR/1974/PP75, che ha espresso stabilmente la luciferasi della lucciola anche dopo diversi passaggi in coltura e topi (Figura 1), come correlato per altre specie di Leishmania trasfettate dallo stesso plasmide integrativo20.
La vena mandibolare labiale o vena gengivale è un percorso migliore per il prelievo di sangue e per la raccolta multipla di sangue10,11. Tuttavia, questa è la prima dimostrazione che la vena gengivale è un sito possibile per l'infezione endovenosa da Leishmania nei criceti. A differenza del prelievo di sangue che di solito utilizza un ago da 26 G ad alto calibro per evitare l'emolisi del sangue10, questo calibro dell'ago non era appropriato per l'inoculazione della Leishmania, a causa della perforazione venosa, del sanguinamento e della perdita dell'inoculo. Per l'infezione da Leishmania attraverso la vena mandibolare mascellare, era essenziale un ago da 30 G di calibro inferiore. Un altro aspetto che differenzia la puntura venosa dall'infezione attraverso la vena gengivale è la velocità di somministrazione, a circa 1 μL/s; e per garantire che venga iniettato nel vaso, la vena labiale mandibolare, e non si depositi nella mucosa, sottocutanea o intradermica. A causa del basso ricambio ematico del plesso gengivale, i 50 μL di un inoculo ad alta densità di amastigoti axenici o promastigoti di L. infantum-Luc, 2 x 109 parassiti/mL, dovevano essere inoculati lentamente (~ 1 min), e l'ago doveva essere rimosso mantenendo il tampone premuto per 1 min per consentire la dispersione dell'inoculo nel flusso sanguigno (Figura 2).
Per la valutazione longitudinale dell'infezione, i criceti sono stati infettati per via intraperitoneale (IP) o per via intragengivale (IG) con L. infantum-Luc e sono stati seguiti per 50 dpi dal sistema di imaging a bioluminescenza fino all'eutanasia. Considerando che il ceppo di riferimento PP75 potrebbe essere meno virulento di per sé e che la super-espressione della luciferasi potrebbe anche influire sull'efficacia dell'infezione e mantenere l'infezione a lungo termine, per l'infezione è stato utilizzato un inoculo alto di 108 parassiti. Dopo l'inoculazione gengivale di amostigoti e promastigoti di L. infantum-Luc e la valutazione da parte del sistema di imaging a bioluminescenza, la bioluminescenza è stata limitata alla regione mascellare dei criceti 24 ore dopo l'infezione. Infatti, i criceti infettati per via intraperitoneale con amastigoti e promastigoti di L. infantum-Luc mostravano bioluminescenza dispersa in tutto l'addome (Figura 3, 1 dpi). La continua diminuzione dell'emissione bioluminescente nel tempo, dal primo giorno di infezione fino all'8° giorno e fino al 22° dpi nei criceti infettati da L. infantum-Luc, era indipendente dalla via di inoculazione (Tabella 1 e Figura 4).
Tuttavia, quando il carico di parassiti nei tessuti animali è basso, può essere inferiore al limite di rilevamento del sistema di imaging a bioluminescenza, ma può essere rilevato e quantificato mediante PCR o qPCR. Come già riportato, l'infezione causata dal ceppo PP75 è effettivamente inferiore a quella di altri ceppi5 e solo pochi hanno sviluppato segni clinici della malattia a causa della variabilità genetica degli animali. In questo studio, nonostante il piccolo numero di animali e la bassa virulenza di questo ceppo, gli amastigoti axenici hanno dimostrato un vantaggio a 50 dpi, mostrando un'infezione migliore rispetto ai promastigoti, come dimostrato dalla PCR (Figura 5). Otto mesi dopo l'infezione da amastigoti per via gengivale, i parassiti potevano essere rilevati mediante PCR nel fegato (Figura 5) e mostravano anche piloerezione moderata, tenuta orbitale e postura arcuata.
Gli amastigoti axenici possono essere una scelta migliore per l'infezione e il follow-up rispetto ai promastigoti21 e hanno il vantaggio di essere facili da produrre su larga scala. L'inoculo gengivale è fattibile e rappresenta una via migliore per l'inoculazione endovenosa di composti e per l'infezione da Leishmania e altri agenti patogeni, senza danni o gonfiore nel sito di applicazione nella mandibola o per la salute degli animali.
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Fundação de Apoio ao Desenvolvimento do Ensino, Ciência e Tecnologia do Estado de Mato Grosso do Sul- FUNDECT. PPSUS/Decit-MS/CNPq/SES ha fornito sostegno finanziario a questa ricerca. Grazie a Monique Ribeiro de Lima per i suoi consigli sulle vie di inoculazione. Questo progetto è stato sviluppato nell'ambito dell'Accordo diCooperazione n. 258/2017 tra FIOCRUZ e l'Universidade Federal do Rio de Janeiro- UFRJ. Il team è sinceramente grato ai produttori video Ricardo Baptista Schmidt e Genilton José Vieira del Science Popularization Center (IOC) per il loro inestimabile supporto e assistenza nelle riprese dei protocolli e nella conduzione delle interviste.
Materiali
| Name | Company | Catalog Number | Comments |
| 1 kb ladder | Promega | G5711 | |
| 1-Butanol | Sigma-Aldrich | B7906 | |
| Acetyl coenzyme A | Sigma-Aldrich | A2181 | |
| Agarose, LE, Analytical Grade | Promega | V3125 | |
| aprt reverse primer A1715 aprt reverse primer A1715 | Jena Bioscience | PM-111 | |
| BamHI 10 U/mL | Promega | R6021 | |
| BglII 10 U/mL | Promega | R6071 | |
| brain and heart infusion (BHI) | Sigma-Aldrich | 53286 | |
| Cetamin (Ketamine hydrochloride 10%) | Syntec | - | Veterinary use. Anesthetic. Injectable solution containing a 10 mL vial of 10% ketamine hydrochloride. |
| Dextrose Glucose, BD Diagnostics | Difco | 215530 | |
| D-luciferin potassium salt | Promega | E1601 | |
| DMEM low glucose | Sigma-Aldrich | D6046 | |
| eletroporador Gene Pulser Xcell | BioRad Laboratories | ||
| Fetal calf serum (FCS) | vitrocell/embriolife | ||
| Ficoll-plaqueTM PLUS | Cytiva | 17144003 | |
| Gene Pulser/MicroPulser Electroporation Cuvettes, 0.2 cm gap 1652082 | Bio-Rad | 1652082 | |
| Gene Pulser/MicroPulser Electroporation Cuvettes, 0.4 cm gap 1652081 | Bio-Rad | 1652081 | |
| GoTaq Platinum polymerase | Fischer Scientific | 10-966-034 | |
| GoTaq DNA Polymerase | Promega | M3001 | |
| Hemin powder | inlab | ||
| HEPES buffer | Sigma-Aldrich | H3375 Sigma-Aldrich | |
| hyg forward primer A3804 | Jena Bioscience | PM-109 | |
| Hygromycin | Sigma-Aldrich | H3274 | |
| ISOFORINE | Cristalia | Inhalation solution in packs containing 1 bottle of 100 and 240 mL of isoflurane | |
| IVIS Lumina | Perkin Elmer | ISO838N4625 | |
| JM109 Competent Cells | Promega | L2005 | |
| L- Glutamin | Sigma-Aldrich | G8540 Sigma-Aldrich | |
| Lambda DNA/HindIII Marker | Thermo Fischer Scientific | SM0101 | |
| L-cystein | Sigma-Aldrich | 168149 Sigma-Aldrich | |
| Living Image software | Perkin Elmer | - | |
| NotI 10 U/mL | Promega | R6431 | |
| Phenol/Chloroform/Isoamyl Alcohol, 25:24:1 (v/v), Molecular Biology Grade | Sigma-Aldrich | 516726 | |
| Phosphate-buffered saline (DPBS) | Gibco | 14190 | |
| plasmid pLEXSY-hyg2 | Jena Bioscience | EGE-232 | |
| Proteinase K | Promega | V3021 | |
| QIAGEN Plasmid Midi Kit | Qiagen | 12143 | |
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | |
| QIAquick PCR & Gel Cleanup Kit | Promega | A9281 | |
| Schneider's medium | Gibco | 21720-024 | |
| Sodium bicarbonate | Sigma-Aldrich | S6014 | |
| Sodium phosphate dibasic | Sigma-Aldrich | S9763 Sigma-Al | |
| SpectraMax2 microplate reader | Applied Biosystems | ||
| ssu forward primer F3001 primer F3001 | Jena Bioscience | PM-105 | |
| ssu reverse primer F3002 ssu reverse primer F3002 | Jena Bioscience | PM-104 | |
| Steady-Glo Luciferase Assay System | Promega | E2510 | |
| SwaI 10 U/mL | Thermo Scientific | ER1241 | |
| T4 DNA ligase 1 U/mL | Promega | M1801 | |
| T4 fast ligation system | Promega | M8221 | |
| Thermal cycler | Applied Biosystems | Veritiy 96 well plate | |
| TRITON X-100 | Sigma-Aldrich | T8787 Sigma-Aldrich | |
| Tryptic Soy Broth (Soybean-Casein Digest Medium) | Difco-BD | 211823 | |
| Ventilated racks | Alesco | ||
| With Earle′s salts and L-glutamine, without sodium bicarbonate, powder, | Merck | M5017 | |
| Wizard SV Gel and PCR Clean-Up | Promega | A9282 | |
| Xilazina (Xylazine hydrochloride 2%) | Syntec | - | Veterinary use. Sedative, analgesic and myorelaxant. Injectable solution containing a 10 mL vial of 2% xylazine hydrochloride. |
| Zero Blunt TOPO PCR Cloning Kit | Thermo Fischer | 451245 |
Riferimenti
- Ruiz-Postigo, J. A., et al. Global leishmaniasis surveillance, 2022: assessing trends over the past 10 years. WHO, Weekly epidemiological record n° 40. , (2023).
- Faraut-Gambarelli, F., et al. In vitro and in vivo resistance of Leishmania infantum to meglumine antimoniate: a study of 37 strains collected from patients with visceral leishmaniasis. Antimicrob Agents Chemother. 41 (4), 827-830 (1997).
- Ribeiro-Romão, R. P., et al. Comparative evaluation of lesion development, tissue damage, and cytokine expression in golden hamsters (Mesocricetus auratus) infected by inocula with different Leishmania (Viannia) braziliensis concentrations. Infect Immun. 82 (12), 5203-5213 (2014).
- Gomes-Silva, A., et al. hamster (Mesocricetus auratus) as an experimental model for Leishmania (Viannia) braziliensis infection. Parasitology. 140 (6), 771-779 (2013).
- Moreira, N. D., et al. hematological and biochemical alterations in hamster (Mesocricetus auratus) experimentally infected with Leishmania infantum through different routes of inoculation. Parasit Vectors. 9 (1), 181-194 (2016).
- Carnielli, J. B. T., et al. A Leishmania infantum genetic marker associated with miltefosine treatment failure for visceral leishmaniasis. EBioMedicine. 36, 83-91 (2018).
- Carnielli, J. B. T., et al. Natural resistance of Leishmania infantum to miltefosine contributes to the low efficacy in the treatment of visceral leishmaniasis in Brazil. Am J Trop Med Hyg. 101 (4), 789-794 (2019).
- Moreira, D., et al. Impact of continuous axenic cultivation in Leishmania infantum virulence. PLoS Negl Trop Dis. 6 (1), e1469 (2012).
- Fortin, A., et al. Efficacy and tolerability of oleylphosphocholine (OlPC) in a laboratory model of visceral leishmaniasis. J Antimicrob Chemother. 67 (11), 2707-2712 (2012).
- Rodrigues, M. V., et al. The gingival vein is a minimally traumatic site for multiple blood sampling in guinea pigs and hamsters. PLoS ONE. 12 (5), e0177967 (2017).
- Oliveira, D. T., et al. Technical report: Gingival vein punction: A new simple technique for drug administration or blood sampling in rats and mice. Scand J Lab Anim Sci. 36 (2), 109-113 (2009).
- Saeij, J. P., et al. Bioluminescence imaging of Toxoplasma gondii infection in living mice reveals dramatic differences between strains. Infect Immun. 73 (2), 695-702 (2005).
- Thalhofer, C. J., et al. In vivo imaging of transgenic Leishmania parasites in a live host. J Vis Exp. 41, e1980 (2010).
- Andriani, G., et al. Activity in vivo of anti-Trypanosoma cruzi compounds selected from a high throughput screening. PLoS Negl Trop Dis. 5 (8), e1298 (2011).
- Henriques, C., et al. In vivo imaging of mice infected with bioluminescent Trypanosoma cruzi unveils novel sites of infection. Parasit Vectors. 7, 89 (2014).
- Henriques, C., et al. Bioluminescent imaging of Trypanosoma cruzi infection in Rhodnius prolixus. Parasit Vectors. 26 (5), 214 (2012).
- Sereno, D., Lemesre, J. L. Axenically cultured amastigote forms as an in vitro model for investigation of antileishmanial agents. Antimicrob Agents Chemother. 41 (5), 972-976 (1997).
- Lachaud, L., et al. Value of two PCR methods for the diagnosis of canine visceral leishmaniasis and the detection of asymptomatic carriers. Parasitology. , 197-207 (2002).
- . Blood sampling Microsite Available from: https://www.nc3rs.org.uk/3rs-resources/blood-sampling/blood-sampling-hamster (2021)
- Bolhassani, A., et al. Fluorescent Leishmania species: development of stable GFP expression and its application for in vitro and in vivo studies. Exp Parasitol. 127 (3), 637-645 (2011).
- Mendes Costa, D., et al. Murine infection with bioluminescent Leishmania infantum axenic amastigotes applied to drug discovery. Sci Rep. 9 (1), 18989 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon