このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ガスクロマトグラフィー-質量分析に基づくハードコーラル試料のターゲットメタボロミクス
要約
ここでは、ガスクロマトグラフィー質量分析のために、サンゴのホロビオントからの極性および半極性代謝産物の抽出と調製、ならびに分離されたサンゴの宿主組織とSymbiodiniaceae細胞分画を紹介します。
要約
ガスクロマトグラフィー質量分析(GC-MS)ベースのアプローチは、刺胞動物と渦鞭毛藻の共生の代謝基盤と、サンゴがストレス(つまり、温度による白化)にどのように応答するかを解明するのに強力であることが証明されています。刺胞動物の宿主とそれに関連する微生物(Symbiodiniaceaeと他の原生生物、細菌、古細菌、真菌、ウイルス)で構成されるサンゴのホロビオントの定常状態代謝物プロファイリングは、サンゴの全体的な代謝状態を特徴付けるために、環境条件およびストレス条件下でうまく適用されています。
しかし、共生相互作用をめぐる疑問に答えるためには、サンゴの宿主とその藻類共生生物の代謝物プロファイルを独立して解析する必要があり、これは組織の物理的な分離と単離、それに続く独立した抽出と分析によってのみ達成できます。メタボロミクスの応用はサンゴ分野では比較的新しいものですが、研究グループの継続的な努力により、サンゴの宿主組織と藻類共生生物の分離など、サンゴの代謝物を分析するための堅牢な方法が開発されました。
このホワイトペーパーでは、GC-MS 分析のためのホロビオント分離と代謝物の抽出について、検討すべき重要な最適化ステップを含め、ステップバイステップで説明します。2つの分画(サンゴとシンビオディニア科)の複合代謝物プロファイルが全体(ホロビオント)のプロファイルと類似していることを、独立して分析すると、組織を分離することで、全体だけでは得られない2つのパートナーの代謝と相互作用に関する重要な情報も得ることができることを実証します。
概要
代謝産物は細胞プロセスの最終生成物を表し、メタボロミクス(特定の生物または生態系によって生成される一連の代謝産物の研究)は、生物の機能の直接的な尺度を提供できます1。これは、ほとんどの管理戦略の目標は特定の生態系サービス機能を維持(または回復)することであるため、生態系、共生相互作用、および回復ツールを探求するために特に重要です2。サンゴ礁は、共生相互作用を解明し、サンゴの生理学的応答をコミュニティレベルおよび生態系レベルの影響に結びつけるためのメタボロミクスの潜在的な価値を実証する水生生態系の1つです3。ハイスループットガスクロマトグラフィー質量分析(GC-MS)のアプリケーションは、幅広い代謝物クラスを高い選択性と感度で同時に迅速に分析し、スペクトルライブラリが利用可能な場合に迅速な化合物同定を提供し、サンプルあたりのコストが比較的低く、高レベルの再現性と精度を提供する能力があるため、特に高く評価されています。
サンゴは、サンゴ動物、光合成渦鞭毛藻類の内部共生生物(科:共生虫科4)、および複雑なマイクロバイオーム5,6からなるホロビオントです。全体として、ホロビオントの適応度は、主に各メンバーの代謝機能をサポートするための低分子および元素の交換によって維持される7,8,9,10。メタボロミクス的アプローチは、サンゴの共生特異性9,11、熱ストレスに対する白化応答7,8,12,13、疾患応答14、汚染曝露応答15、光順化16、化学シグナル伝達17の代謝基盤を解明し、バイオマーカーの発見18を支援するのに特に強力であることが証明されています、19。さらに、メタボロミクスは、DNAおよびRNAベースの技術から推測される結論の貴重な確認を提供することができます9,20。したがって、ストレスの代謝バイオマーカーの検出など、サンゴ礁の健康状態を評価し、サンゴ礁保全のためのツールを開発するためのメタボロミクスの使用にはかなりの可能性があります18,19栄養補助金21などの積極的な管理戦略の可能性を検討します。
宿主細胞と共生生物細胞を分離し、それらの代謝物プロファイルをホロビオントとして一緒に分析するのではなく、独立して分析することで、パートナーの相互作用、独立した生理学的および代謝状態、および適応のための潜在的な分子メカニズムについてより多くの情報を得ることができます11,12,22,23,24.サンゴと共生藻科を分離しなければ、複雑なゲノム再構成と代謝モデリングを除いて、サンゴおよび/または共生虫科の寄与と代謝を独立して解明することはほとんど不可能である25が、これはサンゴと渦鞭毛藻の共生にはまだ適用されていない。さらに、宿主や藻類の共生生物の個々の代謝に関する情報をホロビオントの代謝物プロファイルから抽出しようとすると、誤解を招く可能性があります。
例えば、サンゴやホロビオント組織からの抽出物中のC18:3n-6、C18:4n-3、およびC16多価不飽和脂肪酸の存在は、サンゴがオメガ3(ω3)脂肪酸の生産に不可欠なωx脱サチュラーゼを持っていないと考えられていたため、最近まで藻類共生生物に由来すると考えられていました。しかし、最近のゲノム証拠は、複数の刺胞動物がω3 PUFA de novoを産生し、さらにω3長鎖PUFAを生合成する能力を持っていることを示唆しています26。GC-MSと安定同位体標識(例:13 C-重炭酸塩、NaH 13CO 3)を組み合わせることで、制御条件下および外部ストレス要因への応答の両方で、サンゴのホロビオント代謝ネットワークを介して光合成的に固定された炭素の運命を追跡することができます27,28。しかし、13Cの運命を追跡する上で重要なステップは、藻類細胞からのサンゴ組織の分離であり、サンゴの宿主画分に13C標識化合物が存在する場合にのみ、サンゴに転座したシンビオディニア科由来の代謝物または転座した標識化合物の下流生成物として明確に割り当てることができます。この技術は、光合成が共生生物から宿主へと移動する主要な形態はグリセロールであるという長年の仮定に異議を唱え29、白化中にパートナー間の栄養フラックスがどのように変化するかを解明することによってその力を実証しました27,28および互換性のないSymbiodiniaceae種11。
組織を分離する決定は、主に研究課題によって左右されますが、このアプローチの実用性、信頼性、および潜在的な代謝への影響を考慮することが重要です。ここでは、ホロビオントから代謝物を抽出するための詳細で実証された方法、および宿主と共生生物の分画を分離します。宿主と共生生物の代謝物プロファイルを独立して比較し、これらのプロファイルをホロビオントの代謝物プロファイルとどのように比較するか。
プロトコル
注:実験計画、サンプルの収集および保管については、他の場所で詳細に説明されています2,30,31。野生サンゴの採集許可は、採集と実験の前に取得する必要があります。ここでのサンプルは、バタビアサンゴ農場(ワシントン州ジェラルトン)から輸入されたMontipora mollis(緑色のカラーモルフ)のコロニーから採取されたもので、もともとはアブロロス諸島(西オーストラリア州、南緯28度52分43.3秒、東経114度00分17.0秒)沖のサンゴ礁から採取され、水産養殖ライセンスAQ1643の下で水深1mで採取されました。サンプリングに先立ち、コロニーを800Lの水槽、35PSU、青色および白色光、150μmol光子・m・2・s-1、25°C±0.5°Cで3ヶ月間保持した。サンゴの破片(~5 cm2、N = 6)を液体窒素中で急速凍結し、処理するまで-80°Cで保存しました。
1. 抽出液と装置の準備
- サンゴ組織を除去する少なくとも1日前に、抽出液と機器を準備します。
- 界面活性剤を含まない清潔なガラス器具で超純水を4°Cで予冷します。
- 100% LC グレードのメタノールを、最終濃度 10 μg·mL-1 の適切な内部標準試料( 例:13C6 ソルビトール)と混合します。
- 半分が100%LCグレードのメタノール、半分が超純水を使用して、50%メタノール抽出溶液を作成します。両方のメタノール溶液を-20°Cで保存します。
注:代謝物の分解を防ぐために、サンプル処理ステップを一度に 5 つのサンゴ断片のバッチで実行し、1 つの生物学的(水のみ)ブランク(合計サンプル N = 6)を追加することをお勧めします。各サンゴサンプルが2つのフラクション(サンゴの宿主組織、以下「宿主」、微細藻類細胞、以下「共生菌」)に分離されると、1つの処理バッチのサンプルの総数は12になります。
2.サンゴの新陳代謝の癒やす
注:実験計画、サンプルの収集および保管については、他の場所で詳細に説明されています2,30,31。しかしながら、代謝を鎮めるのに要する時間(すなわち、サンゴ試料の採取と保存の間の時間)は、元の応答を捕捉するために重要であることに留意すべきである30。サンプルの分解または非標的生理学的応答による代謝物組成の変化を防ぐために、採取後できるだけ早くサンプルを保存する32。
- サンゴの破片を滅菌サンプル採取バッグに入れ、余分な海水をできるだけ排出します。サンプルを液体窒素に最低30秒間浸します。サンプルはできるだけ早く-80°Cの冷凍庫に移して保管してください。
注:サンプルは、凍結融解サイクルを回避するため、処理するまで-80°Cでライトブロック容器で凍結できます。
3.骨格からのサンゴ組織の除去
注:サンプルは常に氷上(4°C)に保管して、進行中の代謝を防ぎながら同時に液体の形になるようにする必要があります。
- 清潔で滅菌済みのサンプル採取バッグを氷の上に置き、バッグが安定して浅いウェルの氷の上に開いていて、氷に沈まないようにします。バッグに冷水(4°C)10mLを加えます。
注:これは、冷たい加圧空気と周囲の氷によるサンゴの破片の繰り返しの凍結融解を回避するのに役立ちます。 - 滅菌したピンセットでサンゴの破片を選び、滅菌パスツールピペットを使用して冷水(4°C)の超純水で海水の残留物がなくなるまですすぎます。すすいだサンゴの破片を、10mLの超純水を入れた袋に沈めます。
注:このすすぎは、下流の分析を妨げる残留塩分を除去するために重要です。サンプルを4°Cに維持するために、バッグを通して水やサンゴの破片に手が触れないようにしてください。 - 滅菌した1 mLのピペットチップをエアガンの端に電気テープで固定し、チップの端を~5 mm切り取ります(図1A)。
- バッグを半分密封し、空気の流れを低培地にして、エアガンをサンゴの破片に向け、サンゴの破片の上を水を円を描くように促して組織を静かに取り除きます。
- ~3分後、またはすべての組織が骨格から除去されたように見える場合は、空気を止め、エアブラシを取り外します。バッグを完全に密封します。
- 取り除いたすべてのサンゴ組織をバッグの下隅に押し込みます。反対側の角を切り取り、バッグの内容物を氷上の15mLチューブにそっと注ぎます。
4.オプションの均質化
注:一部のサンゴ種は他の種よりも粘性が高いため、エアブラシはスラリーではなく塊で組織を除去します。エアブラシで塗布したホモジネートに組織の塊が見られる場合は、すべてのサンプルに4°Cのホモジナイズステップを追加できます。
- 機械式ノコギリ歯型ホモジナイザーを 4 °C の 70% メタノールで 2 回洗浄し、最後に 4 °C の超純水で洗浄します。
- サンゴサンプルを15 mLチューブ中で1~1分間、サンプルが完全に均質化され、塊が見えなくなるまでホモジナイズします。
- ステップ 4.1 と同様に、各サンプル間のホモジナイザーを洗浄します。サンプル間で均質化時間を一定に保ちます。
5. 正規化のためのサンプル採取
- ホモジナイズした組織から1,000 μLのアリコートを採取し、Symbiodiniaceaeの細胞数、サンゴの宿主組織タンパク質含有量分析、およびクロロフィル a 推定を行います。分析の準備が整うまで-20°Cで保存します(セクション10)。
6. 任意サンゴの宿主のティッシュSymbiodiniaceaeの細胞分離
- サンゴホモジネートを2,500 × g で4°Cで5分間遠心分離します。
注:この速度は、上清に懸濁している宿主組織から、細胞壁をそのまま維持しながら、より重いSymbiodiniaceae細胞を分離するのに最適です。 - ホスト物質を含む上清を取り除き、新しい15 mLチューブに入れます。
注:宿主組織由来の脂質は、通常、共生細胞の上に細いピンク/白の層を形成します。この層は、ピペッティング により 可溶性宿主上清とともに回収できます(図1B)。 - ホストをちょうど1分間激しくボルテックスします。藻類ペレットサンプルと宿主上清サンプルを氷上に保管してください。
- 藻類ペレットに4°Cの超純水2mLを加えます。正確に2分間激しくボルテックスして、ペレットを再懸濁します。
注:Symbiodiniaceaeのジェノタイピングのためにサンゴのコロニーから1cmの個々の断片が収集されなかった場合は、Symbiodiniaceae細胞懸濁液の200 μLアリコートをここで収集し、好ましいDNA緩衝液に保存し、Thurber et al.30 のSymbiodiniaceaeジェノタイピングの記載に従って保存することができます(例えば、González-Pech et al.12による)。 - 手順6.1〜6.4をもう一度繰り返します。
注:宿主と共生生物の確実な分離は、一部の種が他の種よりも粘性が高い可能性があるため、サンゴのバイオマスと種によって異なります。最低 3 つの洗浄ステップが推奨されますが、分離の成功によっては、このステップを増やすことができます。宿主画分の底部にSymbiodiniaceae細胞が見られなくなり、Symbiodiniaceae画分に宿主物質が目に見えなくなるまで(例えば、上部に白い層がなくなるまで)、洗浄ステップ4.7〜4.9を繰り返します(図1)。 - ホスト物質を含む上清を取り除き、新しい15 mLチューブに入れます。
- 共生生物ペレットを 15 mL チューブに保持します。
7. サンプルの乾燥
- ホロビオントホモジネートのいずれか、または分離した宿主とSymbiodiniaceae画分の両方を、-80°Cで~120分間凍結します。サンプルを -85 °C で 0.01 mbar 真空で一晩凍結乾燥します。
注:凍結乾燥中のサンプルの損失を避けるために、滅菌済み25 Gニードルを使用して慎重に~2 mmの穴を開けた別の滅菌チューブまたは滅菌パラフィルムから切り取った蓋を使用することをお勧めします。 - 乾燥したら、実験室の天びんを使用して、次のいずれかを計量します:1)25mgのホロビオント。2)共生生物画分15mg;または3)各サンプルから30 mgの宿主組織を別々の2 mL可塑剤を含まない微量遠心チューブに入れます。
注:重要なステップ:十分なシグナルを確保しながらGC-MSが過負荷にならないようにするには、抽出用のバイオマスの最適化が不可欠です。乾燥した珊瑚の材料は非常に静的です。サンプルの損失を防ぐには、帯電防止装置を使用して、サンプルと計量容器から静電気を除去します。簡単で費用対効果の高い代替手段は、サンプルチューブの下にランドリードライヤーシートを置くことです。乾燥共生菌ペレットは、滅菌ブレードを使用して所望の重量に切断することができる。
8. 細胞内代謝物抽出
- 凍結乾燥ホロビオントからの細胞内代謝産物抽出:
- 400 μL の 100% 冷(−20 °C)メタノールを内部標準試料で添加します(IS;13名C6ソルビトールおよび/または13C5-15 Nバリン、10μMで)を各チューブに注入した。
- 各サンプルに少量の710-1,180 μmの酸洗浄ガラスビーズ(~10 mg)を加えます。50Hzのビーズミルに、あらかじめ冷やした(-20°C)ビーズミルインサートに3分間入れます。
- さらに600 μLの100%冷(-20°C)メタノールとIS(13C6 ソルビトールおよび/または 13C5-15 Nバリン、10μM)を各チューブに加えます。
- ボルテックスで1分間混ぜます。4°Cのロティサリーシェーカーに30分間置きます。
- 分離された凍結乾燥したSymbiodiniaceae細胞からの細胞内代謝産物抽出:
- 乾燥したシンビオディニア科の材料に、100%冷(-20°C)メタノール200 μLとIS(13C6 ソルビトールおよび/または 13C5-15 Nバリン、10μM)を加えます。
- 710-1,180 μmの酸洗浄ガラスビーズ(~10 mg)を少数添加します。50Hzのビーズミルに、あらかじめ冷やした(-20°C)ビーズミルインサートに3分間入れます。
- さらに800 μLの100%冷(-20°C)メタノールとISを加え、30秒間ボルテックスします。
- 分離された凍結乾燥宿主組織からの細胞内代謝物抽出:
- ISを含む100%コールド(−20°C)LCグレードメタノール1 mL(13C6 ソルビトールおよび/または 13C5-15 Nバリン、10μM)を乾燥したホスト材料に加えます。
- 20秒間混合するボルテックス。フローティングチューブホルダーに入れ、4°Cに設定された超音波処理槽に30分間入れます。
9. 代謝物質のエキスの浄化
- サンプル(ホロビオント/宿主/共生生物)を3,000 × g で4°Cで30分間遠心分離します。
- すべての上清を新しい2 mL微量遠心チューブに移し、細胞破片ペレットを乱さないように注意します。
注:これらは半極性抽出物です。これらは一時的に氷上に保管できますが、暗所で-80°Cで長期間保管できます。 - 残りの細胞破片に、1,000 μL の 50% 冷(-20 °C)メタノールを加えます。1分間激しくボルテックスして再懸濁します。
- サンプルを3,000 × g で4°Cで30分間遠心分離します。
- 上清(極性抽出物)を同じサンプルから半極性抽出物と収集してプールします。
注:細胞破片は-80°Cで保存し、タンパク質含量の正規化(セクション11)に使用できます。 - プールした抽出物を16,100 g で15分間遠心分離して沈殿物をすべて除去し、上清を新しい可塑剤フリーの微量遠心チューブ(2 mL)に移します。
注:サンプル抽出物は、暗所で-80°Cで保存できます。 - 分析の準備ができたら、各抽出物 50 μL をガラスインサートに分注します。真空濃縮器を使用して30°Cで30分間濃縮します。さらに4回繰り返します(全乾燥抽出物250μLの場合)。
注:乾燥したサンプルは、分析まで乾燥剤条件下で室温で保存できます。
10. 代謝物誘導体化
注:極性代謝物のメトキシム化およびトリメチルシリル化には、2段階のオンライン誘導体化プロセスが使用されます。
- 各サンプルに塩酸メトキシアミン 25 μL(ピリジン中 30 mg/mL)を加えます。
- 750rpmに設定したオービタルシェーカーで37°Cで2時間撹拌します。
- 各サンプルに25μLの N,O-ビス(トリメチルシリル)トリフルオロアセトアミド+トリメチルクロロシランを加えます。
- 再び37°C、750rpmで1時間撹拌する。
- サンプルを室温で 1 時間平衡化してから、1 μL を 1:10 の分割比で GC に注入します。
11. ガスクロマトグラフ質量分析
注:質量分析計は、トリス-(パーフルオロブチル)-アミン(CF43)を使用して、メーカーの推奨に従って調整する必要があります。
- キャリアガスには超高純度ヘリウムを使用し、カラム流量は1mL/minの一定です。
- 膜厚1 μm、内径0.25 mmの30 mのDB-5カラムをご使用ください。
- GCオーブンプログラム
- 入口温度を280°Cに設定します。
- オーブン温度 100 °C の注入を開始し、4 分間保持します。
- 温度を10°C/分上げて320°Cにし、11分間保持します。
- 質量分析計のパラメータ
- MS トランスファーラインを 280 °C に設定し、イオン源を 200 °C に調整します。
- アルゴンをコリジョンセルガスとして使用して、マルチプル反応モニタリング(MRM)プロダクトイオンを生成します。
- MRMターゲットを含む時間セグメント化されたMRMライブラリーに対する代謝物検出を実現します。
12. 共生虫科の細胞数、サンゴの宿主組織タンパク質含量分析、クロロフィル推定
- Symbiodiniaceaeの細胞数:
- サンゴ組織ホモジネートのアリコートを取ります。
- サンプルを2,000 × g で遠心分離し、藻類をペレット化します。
- ~200 μLの上清を藻類ペレットから取り出し、新しい微量遠心チューブに入れます。
注:これは、データを正規化するために使用されるタンパク質サンプルになります。必要に応じて、分析前に-20°Cで保存してください。 - 藻類ペレットをろ過した海水1 mLに、ピペッティングで静かに上下させ、再懸濁します。必要に応じて、藻類懸濁液を希釈して細胞のカウントを容易にします。
- チャンバーの1つに10μLを添加することにより、光学顕微鏡下で血球計算盤を使用して細胞数を実施します。サンプルごとに8〜10カウントを完了します。
注:藻類細胞をカウントするための代替方法も、利用可能な場合に適用することができます(例:フローサイトメトリー、ハイスループット共焦点顕微鏡法)。 - 共生菌細胞の濃度(mL-1)を、使用する希釈係数を考慮して計算します。
- 蛋白質含量の試金
- サンプルのタンパク質含有量を定量化します(例えば、Bradfordらによって最初に記述されたBradfordの比色アッセイ33、または他の場所で刺胞動物についてプロトコルが記述されているLowryアッセイ34,35を介して36)。
- クロロフィル抽出
- ~200、000の細胞の細胞の餌を、凍らせか、または新しい使用しなさい。
- 各藻類ペレットをガラスシンチレーションバイアル中のジメチルホルムアミド(DMF)2 mLに移し、暗所で4°Cで48時間インキュベートします。
注:DMFは有毒で発がん性があるため、サンプル調製はできるだけ暗いドラフトの下で氷上で行う必要があります。<200,000 セルがある場合は、使用する DMF を減らします。 - 16,000 × gで3分間遠心分離します。
- 200 mL を UV-96 ウェルプレートに移し、測光測定を行います。各サンプルを DMF をブランクとして 3 回に分けて分析します。
- 波長(E)663.8nm、646nm、750nmの吸光度を測定します。他の両方の波長の吸光度から750nmの吸光度を差し引きます。
注:750 nmで測定すると、サンプル内の散乱や濁度が補正されます。 - 式(1)を使用してクロロフィルa濃度(μg/mL)を計算します。
Chl a濃度(μg/mL) = (12.00 × E 663.8) - (3.11 × E646.8) (1)
13. 正常化のための代謝物抽出後の細胞バイオマスの定量化
注:以下に説明する細胞バイオマスの定量には、修正ブラッドフォード比色法を使用したバイオマスに関連するタンパク質の定量と、細胞破片乾燥重量の測定の2つのオプションがあります。どちらの方法も細胞バイオマスの正確な定量を提供するため、使用するのが適切です。
- 細胞残骸のタンパク質含有量
- 凍結した細胞破片を1 mLの0.2 M NaOHで再懸濁し、サンプルを98°Cで20分間インキュベートします。
- サンプルを氷上で~10分間冷却し、周囲温度で3,000 × g で5分間遠心分離します。
- サンプルのタンパク質含有量を定量します(例:Bradford et al.33 によって最初に記述され、Smart et al.37 によって修正された Bradford の比色アッセイを介して)。
- 細胞破片乾燥重量の測定
- 細胞内代謝物抽出からの細胞破片を二重蒸留水(~10 mL)に再懸濁します。
- あらかじめ計量したメンブレンフィルター(孔径0.22 μm、47 mm)を使用して、真空下で溶液をろ過します。
- バイオマスの入ったチューブを超純水で2回洗浄し、バイオマスがメンブレンフィルターに完全に移行するようにします。
- バイオマスを含むメンブレンフィルターを取り外し、電子レンジ(低電力、~250W、20分間)を使用して乾燥させます。
- ろ紙をデシケーターに一晩保管します。濾紙の乾燥重量を記録し、総重量から乾式メンブレンフィルターの重量を差し引いてバイオマスの乾燥重量を計算します(サンプルフィルターと一緒に乾燥させた清潔な乾式メンブレンフィルターを使用)。
14. データ分析
- 代謝物データベースを使用して代謝物ターゲットを解析し、各ターゲットは定量化子と修飾子 MRM で構成されています。
- 検出された代謝物ターゲットを目視で検査し、必要に応じて手動で統合します。
- 代謝物のピーク面積を使用して、各グループの各サンプルの相対存在量を計算します。値はブランク補正され、正規化されて内部標準ピーク面積をサンプリングし、次に Smart et al.37 に従って細胞破片タンパク質含有量をサンプリングします。
- すべての治療群で相対標準偏差が35%を超える代謝物を廃棄します(N = 23代謝物)。
- データ(立方根など)を変換し、それらを平均中心化します。正規分布と分散の均一性を確認します。
- データ分析(ANOVAとヒートマップの構築、https://www.metaboanalyst.ca などを使用)38を実行します。パッケージ "cluster"、"factoextra"、および "klustR" を使用して、サンプルをクラスター化し、処理内の変動を調べます。R の関数 "clusGap" を使用してギャップ統計量 (クラスター39 の最適な数を決定する方法) を計算し、R パッケージ "tidyverse" を使用してプロットします。PERMANOVAを実行して、治療代謝物プロファイル間の分離における重要性を調べます(例:.、プライマー)。
結果
この作業中に生成されたすべてのデータは、補足情報で入手できます。
宿主と共生生物の分離
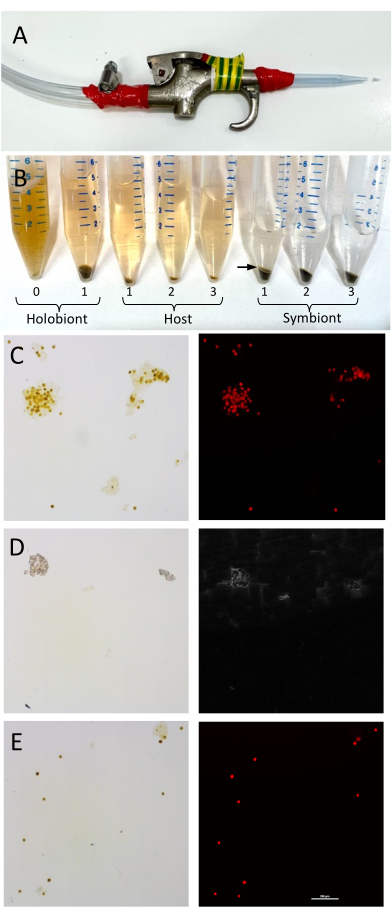
図1:サンゴの宿主組織と共生痊科細胞の分離のセットアップと検証。 (A)サンゴの骨格からサンゴ組?...
ディスカッション
宿主と共生生物の分離は、簡単な遠心分離によって簡単かつ迅速に達成でき、ここでの結果は、フラクションを分離することで、特定のホロビオントメンバーの寄与を示す貴重な情報が得られ、サンゴの健康の機能分析に貢献できることを示しています。成体のサンゴでは、脂質合成は主に常在する藻類共生生物40によって行われ、脂質(例えば、トリアシルグリセロ?...
開示事項
著者には開示すべき利益相反はありません。
謝辞
J.L.M.は、UTS Chancellor's Research Fellowshipの支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| 100% LC-grade methanol | Merck | 439193 | LC grade essential |
| 2 mL microcentrifuge tubes, PP | Eppendorf | 30121880 | Polypropylene provides high resistance to chemicals, mechanical stress and temperature extremes |
| 2030 Shimadzu gas chromatograph | Shimadzu | GC-2030 | |
| 710-1180 µm acid-washed glass beads | Merck | G1152 | This size is optimal for breaking the Symbiodiniaceae cells |
| AOC-6000 Plus Multifunctional autosampler | Shimadzu | AOC6000 | |
| Bradford reagent | Merck | B6916 | Any protein colourimetric reagent is acceptable |
| Compressed air gun | Ozito | 6270636 | Similar design acceptable. Having a fitting to fit a 1 mL tip over is critical. |
| DB-5 column with 0.25 mm internal diameter column and 1 µm film thickness | Agilent | 122-5013 | |
| DMF | Merck | RTC000098 | |
| D-Sorbitol-6-13C and/or 13C5–15N Valine | Merck | 605514/ 600148 | Either or both internal standards can be added to the methanol. |
| Flat bottom 96-well plate | Merck | CLS3614 | |
| Glass scintillation vials | Merck | V7130 | 20 mL, with non-plastic seal |
| Immunoglogin G | Merck | 56834 | if not availbe, Bovine Serum Albumin is acceptable |
| Primer | v4 | ||
| R | v4.1.2 | ||
| Shimadzu LabSolutions Insight software | v3.6 | ||
| Sodium Hydroxide | Merck | S5881 | Pellets to make 1 M solution |
| tidyverse | v1.3.1 | R package | |
| TissueLyser LT | Qiagen | 85600 | Or similar |
| TQ8050NX triple quadrupole mass spectrometer | Shimadzu | GCMS-TQ8050 NX | |
| UV-96 well plate | Greiner | M3812 | |
| Whirl-Pak sample bag | Merck | WPB01018WA | Sample collection bag; Size: big enough to house a ~5 cm coral fragment, but not too big that the water is too spread |
参考文献
- Bundy, J. G., Davey, M. P., Viant, M. R. Environmental metabolomics: A critical review and future perspectives. Metabolomics. 5 (1), 3-21 (2008).
- Matthews, J. L., Beale, D. J., Hillyer, K. E., Warden, A. C., Jones, O. A. H., et al. The metabolic significance of symbiont community composition in the coral-algal symbiosis. Applied Environmental Metabolomics. , 211-229 (2022).
- Lawson, C. A., van Oppen, M. J. H., Aranda Lastra, M., et al. Informing coral reef conservation through metabolomic approaches. Coral Reef Conservation and Restoration in the Omics Age. Coral Reefs of the World. , 179-202 (2022).
- LaJeunesse, T. C., et al. Systematic revision of Symbiodiniaceae highlights the antiquity and diversity of coral endosymbionts. Current Biology. 28 (16), 2570-2580 (2018).
- Rohwer, F., Seguritan, V., Azam, F., Knowlton, N. Diversity and distribution of coral-associated bacteria. Marine Ecology Progress Series. 243, 1-10 (2002).
- Maire, J., et al. Intracellular bacteria are common and taxonomically diverse in cultured and in hospite algal endosymbionts of coral reefs. The ISME Journal. 15 (7), 2028-2042 (2021).
- Hillyer, K. E., et al. Metabolite profiling of symbiont and host during thermal stress and bleaching in the coral Acropora aspera. Coral Reefs. 36, 105-118 (2016).
- Hillyer, K. E., Tumanov, S., Villas-Bôas, S., Davy, S. K. Metabolite profiling of symbiont and host during thermal stress and bleaching in a model cnidarian-dinoflagellate symbiosis. Journal of Experimental Biology. 219 (4), 516-527 (2016).
- Matthews, J. L., et al. Optimal nutrient exchange and immune responses operate in partner specificity in the cnidarian-dinoflagellate symbiosis. Proceedings of the National Academy of Sciences of the United States of America. 114 (50), 13194-13199 (2017).
- Rosset, S. L., et al. The molecular language of the cnidarian-dinoflagellate symbiosis. Trends in Microbiology. 29 (4), 320-333 (2020).
- Matthews, J. L., et al. Partner switching and metabolic flux in a model cnidarian-dinoflagellate symbiosis. Royal Society. 285 (1892), 20182336 (2018).
- González-Pech, R. A., et al. Physiological factors facilitating the persistence of Pocillopora aliciae and Plesiastrea versipora in temperate reefs of south-eastern Australia under ocean warming. Coral Reefs. 41, 1239-1253 (2022).
- Williams, A., et al. Metabolomic shifts associated with heat stress in coral holobionts. Science Advances. 7 (1), (2021).
- Deutsch, J. M., et al. Metabolomics of healthy and stony coral tissue loss disease affected Montastraea cavernosa corals. Frontiers in Marine Science. 8, 1421 (2021).
- Stien, D., et al. A unique approach to monitor stress in coral exposed to emerging pollutants. Scientific Reports. 10 (1), 9601 (2020).
- Lohr, K. E., et al. Resolving coral photoacclimation dynamics through coupled photophysiological and metabolomic profiling. Journal of Experimental Biology. 222 (8), (2019).
- Jorissen, H., et al. Coral larval settlement preferences linked to crustose coralline algae with distinct chemical and microbial signatures. Scientific Reports. 11 (1), 14610 (2021).
- Roach, T. N., Dilworth, J., Jones, A. D., Quinn, R. A., Drury, C. Metabolomic signatures of coral bleaching history. Nature Ecology & Evolution. 5 (4), 495-503 (2021).
- Parkinson, J. E., et al. Molecular tools for coral reef restoration: Beyond biomarker discovery. Conservation Letters. 13 (1), 12687 (2020).
- Jiang, J., et al. How Symbiodiniaceae meets the challenges of life during coral bleaching. Coral Reefs. 40, 1339-1353 (2021).
- Guerra, F. D., Attia, M. F., Whitehead, D. C., Alexis, F. Nanotechnology for environmental remediation: materials and applications. Molecules. 23 (7), 1760 (2018).
- Matthews, J. L., et al. Metabolite pools of the reef building coral Montipora capitata are unaffected by Symbiodiniaceae community composition. Coral Reefs. 39, 1727-1737 (2020).
- Papina, M., Meziane, T., van Woesik, R. Symbiotic zooxanthellae provide the host-coral Montipora digitata with polyunsaturated fatty acids. Comparative Biochemistry and Physiology Part B: Biochemistry and Molecular Biology. 135 (3), 533-537 (2003).
- Kellogg, R., Patton, J. Lipid droplets, medium of energy exchange in the symbiotic anemone Condylactis gigantea: A model coral polyp. Marine Biology. 75, 137-149 (1983).
- Ankrah, N. Y., Chouaia, B., Douglas, A. E. The cost of metabolic interactions in symbioses between insects and bacteria with reduced genomes. mBio. 9 (5), e01433 (2018).
- Kabeya, N., et al. Genes for de novo biosynthesis of omega-3 polyunsaturated fatty acids are widespread in animals. Science Advances. 4 (5), (2018).
- Hillyer, K. E., Dias, D., Lutz, A., Roessner, U., Davy, S. K. 13C metabolomics reveals widespread change in carbon fate during coral bleaching. Metabolomics. 14 (1), 12 (2018).
- Hillyer, K. E., Dias, D. A., Lutz, A., Roessner, U., Davy, S. K. Mapping carbon fate during bleaching in a model cnidarian symbiosis: the application of 13C metabolomics. New Phytologist. 214 (4), 1551-1562 (2017).
- Burriesci, M. S., Raab, T. K., Pringle, J. R. Evidence that glucose is the major transferred metabolite in dinoflagellate-cnidarian symbiosis. Journal of Experimental Biology. 215 (19), 3467-3477 (2012).
- Thurber, R. V., et al. Unified methods in collecting, preserving, and archiving coral bleaching and restoration specimens to increase sample utility and interdisciplinary collaboration. PeerJ. 10, 14176 (2022).
- Grottoli, A. G., et al. Increasing comparability among coral bleaching experiments. Ecological Applications. 31 (4), 02262 (2020).
- Mushtaq, M. Y., Choi, Y. H., Verpoorte, R., Wilson, E. G. Extraction for metabolomics: access to the metabolome. Phytochemical Analysis. 25 (4), 291-306 (2014).
- Bradford, M. M. A rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1), 248-254 (1976).
- Peterson, G. L., et al. A simplification of the protein assay method of Lowry et al. which is more generally applicable. Analytical Biochemistry. 83 (2), 346-356 (1977).
- Lowry, O. H., Rosebrough, N. J., Farr, A. L., Randall, R. J. Protein measurement with the Folin phenol reagent. Journal of Biological Chemistry. 193 (1), 265-275 (1951).
- Zamer, W. E., Shick, J. M., Tapley, D. W. Protein measurement and energetic considerations: Comparisons of biochemical and stoichiometric methods using bovine serum albumin and protein isolated from sea anemones. Limnology and Oceanography. 34 (1), 256-263 (1989).
- Smart, K. F., Aggio, R. B., Van Houtte, J. R., Villas-Boas, S. G. Analytical platform for metabolome analysis of microbial cells using methyl chloroformate derivatization followed by gas chromatography-mass spectrometry. Nature Protocols. 5 (10), 1709-1729 (2010).
- Pang, Z., et al. Using MetaboAnalyst 5.0 for LC-HRMS spectra processing, multi-omics integration and covariate adjustment of global metabolomics data. Nature Protocols. 17 (8), 1735-1761 (2022).
- Tibshirani, R., Walther, G., Hastie, T. Estimating the number of clusters in a data set via the gap statistic. Journal of the Royal Statistical Society: Series B (Statistical Methodology). 63 (2), 411-423 (2001).
- Chen, W. -. N., et al. Diel rhythmicity of lipid-body formation in a coral-Symbiodinium endosymbiosis). Coral Reefs. 31 (2), 521-534 (2012).
- Imbs, A. Fatty acids and other lipids of corals: composition, distribution, and biosynthesis. Russian Journal of Marine Biology. 39 (3), 153-168 (2013).
- Rosset, S., et al. Lipidome analysis of Symbiodiniaceae reveals possible mechanisms of heat stress tolerance in reef coral symbionts. Coral Reefs. 38 (6), 1241-1253 (2019).
- Carreón-Palau, L., Parrish, C. C., Del Angel-Rodriguez, J. A., Perez-Espana, H. Seasonal shifts in fatty acids and sterols in sponges, corals, and bivalves, in a southern Gulf of Mexico coral reef under river influence. Coral Reefs. 40 (2), 571-593 (2021).
- Imbs, A. B., Dang, L. T. Seasonal dynamics of fatty acid biomarkers in the soft coral Sinularia flexibilis, a common species of Indo-Pacific coral reefs. Biochemical Systematics and Ecology. 96, 104246 (2021).
- Oku, H., Yamashiro, H., Onaga, K., Sakai, K., Iwasaki, H. Seasonal changes in the content and composition of lipids in the coral Goniastrea aspera. Coral Reefs. 22 (1), 83-85 (2003).
- Weis, V. M. Cell biology of coral symbiosis: foundational study can inform solutions to the coral reef crisis. Integrative and Comparative Biology. 59 (4), 845-855 (2019).
- Oakley, C., Davy, S., van Oppen, M., Lough, J. Cell biology of coral bleaching. Coral Bleaching. , 189-211 (2018).
- Lu, W., et al. Metabolite measurement: Pitfalls to avoid and practices to follow. Annual Review of Biochemistry. 86, 277-304 (2017).
- Lawson, C. A., et al. Heat stress decreases the diversity, abundance and functional potential of coral gas emissions. Global Change Biology. 27 (4), 879-891 (2021).
- Olander, A., et al. Comparative volatilomics of coral endosymbionts from one-and comprehensive two-dimensional gas chromatography approaches. Marine Biology. 168 (5), 76 (2021).
- Wuerz, M., et al. Symbiosis induces unique volatile profiles in the model cnidarian Aiptasia. Journal of Experimental Biology. 225 (19), (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved